Method Article
הנדסת חלבונים על ידי תצוגת פני שטח שמרים
In This Article
Summary
פרוטוקול זה מתאר את השלבים החיוניים לביצוע קמפיינים לבחירת פני השטח של שמרים להעשרת וריאנטים חלבוניים הנקשרים לאנטיגן מעניין.
Abstract
הנדסת חלבונים מאפשרת שיפור תפקודים קיימים של חלבון נתון או יצירת פונקציות חדשות. אחד הכלים הנפוצים והמגוונים ביותר בתחום הנדסת החלבונים הוא תצוגת פני השטח של שמרים, שבה מאגר של חלבונים אקראיים מבוטא על פני השטח של שמרים. הקישור בין פנוטיפ (למשל, קשירת החלבון המוצג בשמרים לאנטיגן המעניין) וגנוטיפ (קידוד פלסמיד לגרסת החלבון) מאפשר בחירה של ספרייה זו עבור התכונות הרצויות ולאחר מכן ריצוף של וריאנטים מועשרים. על ידי שילוב של בחירת חרוזים מגנטיים עם מיון ציטומטרי זרימה, ניתן לבחור ולהעשיר גרסאות חלבונים עם קשירה משופרת לאנטיגן מטרה. יש לציין כי בנוסף להבשלת האהדה, ניתן להשיג קשירה למטרה גם ללא כל זיקה מחייבת ראשונית. כאן, אנו מספקים פרוטוקול שלב אחר שלב המכסה את כל החלקים החיוניים של קמפיין בחירת תצוגת משטח שמרים ומספק דוגמאות לתוצאות תצוגת משטח שמרים טיפוסיות. אנו מראים כי תצוגת פני השטח של שמרים היא שיטה ישימה וחזקה שניתן לבסס בכל מעבדה לביולוגיה מולקולרית עם גישה לציטומטריית זרימה.
Introduction
תצוגת פני שטח של שמרים היא אחת הטכנולוגיות המרכזיות בתחום הנדסת החלבונים. הוא מאפשר בחירה של גרסאות חלבון עם תכונות רצויות כגון זיקה משופרת או יציבות. היא הוצגה לראשונה בשנת 19971, והיא אחת מטכנולוגיות התצוגה הנפוצות ביותר מלבד תצוגת פאגים 2,3, תצוגת ריבוזום4 ותצוגת תאי יונקים 5,6,7. חלבון העניין (POI) מוצג על פני השטח של תאי שמרים על ידי התכה שלו כדי לעגן חלבונים. קיים מגוון של חלבוני עוגן שונים, ובדרך כלל, נקודת העניין מאוחה למסוף C של חלבון ההזדווגות אגלוטינין שמרים Aga2p 1,8. נוסף על כך, נקודת העניין בדרך כלל מוקפת בשני תגים, כגון תג המגלוטינין (HA-tag) ותג c-myc, המאפשר זיהוי של רמת התצוגה באמצעות נוגדנים מסומנים פלואורסצנטית וציטומטריית זרימה (איור 1A). קמפיינים טיפוסיים לבחירת שמרים כוללים שילוב של בחירת חרוזים מגנטיים ומיון ציטומטרי זרימה. בחירת החרוזים מאפשרת טיפול במספרי תאים גדולים והעשרה של וריאנטים חלבוניים הנקשרים לאנטיגן המטרה גם עם זיקות נמוכות, מאחר שאינטראקציות רב-ערכיות עם החרוזים הטעונים באנטיגן מובילות להשפעות אווידיות, ולכן מונעות אובדן של וריאנטים בעלי זיקה נמוכה (איור 1B). ניתוח ציטומטרי זרימה ובחירה מציעים את היתרון של הדמיה של קשירת גרסאות POI המוצגות לאנטיגן המסומן. כתוצאה מכך, ניתן למיין ולטפח את אוכלוסיות הקשירה, מה שמוביל להעשרת וריאנטים חלבוניים בעלי תכונות רצויות לאורך מספר סבבי מיון. יתר על כן, ניתן לבצע סבבים נוספים של מוטגנזה אקראית כדי להגדיל עוד יותר את המגוון, ומכאן, את הסבירות למצוא מוטציות נוספות התורמות לזיקה ו / או יציבות של החלבון.
תצוגת פני השטח של שמרים מציגה יתרונות מסוימים, כגון (א) מנגנון ביטוי אאוקריוטי, המאפשר קיפול חלבונים חמצוני וכן שינויים אאוקריוטים לאחר תרגום (כגון N-גליקוזילציה), (ב) נורמליזציה של ביטוי עקב זיהוי שני תגי הפפטיד הצמודים לחלבון, (ג) בדיקה חזותית של התקדמות הבחירה על ידי ציטומטריית זרימה (למשל, אחוז התאים הקושרים ועוצמת הקשירה) ו-(ד) האפשרות לנתח מוטציות חלבוניות בודדות על שמרים (למשל, ניתוח יציבות תרמית כמו גם זיקה), המציגים חלופה חוסכת זמן לביטוי וטיהור חלבון מייגע9. למעשה, הן זיקות (ערכי KD) והן יציבויות (ערכי T50) של חלבונים המוצגים בפני השטח של שמרים הראו מתאם טוב עם נתונים שהתקבלו בשיטות ביופיזיקליות וחלבונים מסיסים 9,10,11,12. תצוגת פני השטח של שמרים שימשה להנדסה של מגוון חלבונים, למשל, מקטעי נוגדנים 13,14,15,16, תחום פיברונקטין סוג III10 17,18, rcSso7d19,20, או קשרטין21. באופן דומה, מחקר מקיף נערך כדי לייעל את עיצובי ספריית השמרים על ידי שינוי המיקומים האקראיים, כמו גם שימוש בקודון חומצות אמינו 17,22,23. תצוגת פני השטח של שמרים הוכחה כמוצלחת בהנדסת יציבות 14,15,24,25, זיקה 18,26,27, פעילות אנזימטית 28,29,30,31 וביטוי חלבונים 32. בנוסף, יישומים מתוחכמים יותר כמו קשירה מותנית בנוכחות או היעדר מולקולה קטנה הושגו באמצעות תצוגת משטח שמרים20.
בפרוטוקול זה, אנו מתארים את כל השלבים החיוניים למסע בחירה עם תצוגת משטח שמרים עם הדוגמה של ספריית G4 (המבוססת על תחום פיברונקטין סוג III10, Fn3) שנבחרה כנגד חלבון קושר רטינול אנושי אנטיגן 4 (hRBP4) בנוכחות המולקולה הקטנה A112020. סלקציה זו נערכה כדי להניב אינטראקציה חלבון-חלבון התלויה במולקולה קטנה שיכולה לשמש כמתג מולקולרי. יש לציין כי בעוד שגישות חלופיות אפשריות עם תצוגת משטח שמרים, בחירות שמרים טיפוסיות בדרך כלל שואפות להיקשר לאנטיגן מטרה ללא כל זיקה קודמת לקשירה. אנו מכסים את כל השלבים של קמפיין בחירת שמרים, הכולל טיפוח ספריית שמרים, בחירת חרוזים, מיון ציטומטרי זרימה והבשלת זיקה על ידי PCR נוטה לשגיאות (epPCR). לכן, פרוטוקול זה משלים פרוטוקולים קודמים של תצוגת פני שטח של שמרים33,34 וניתן להשתמש בו כבסיס לבחירות תצוגה של פני השטח של שמרים (איור 1) עם כל ספריית שמרים נתונה ואנטיגן מטרה לפי בחירה.
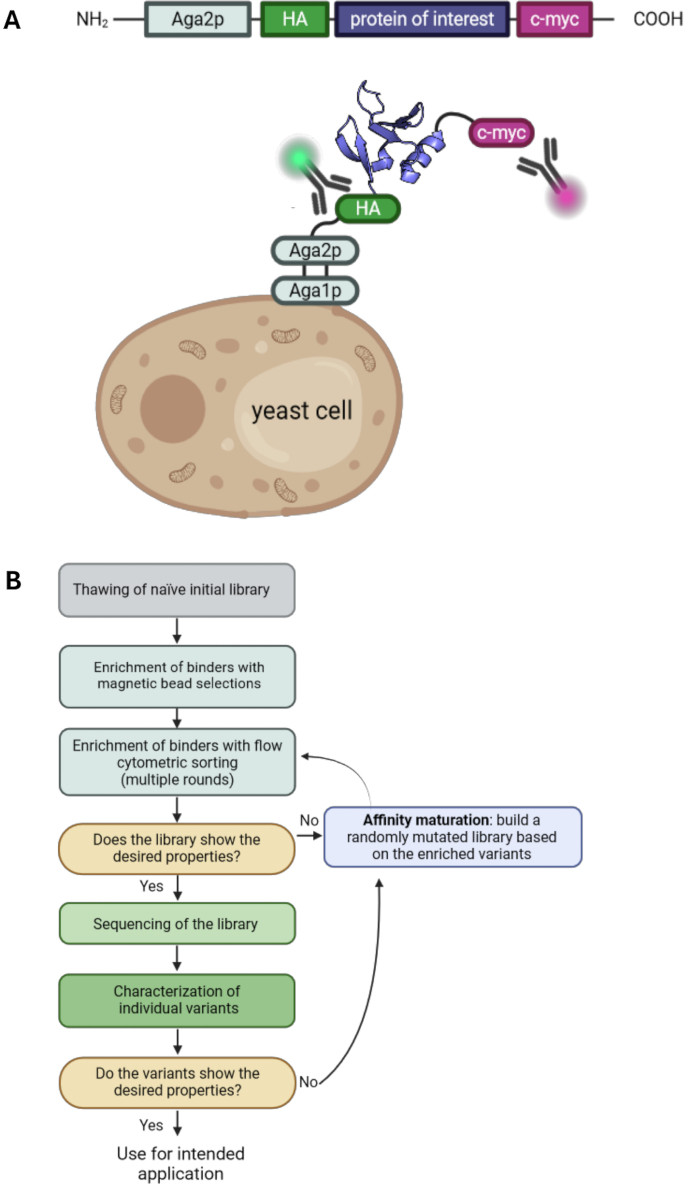
איור 1: עקרון תצוגת פני השטח של שמרים ותהליך עבודה טיפוסי עבור בחירות של משטחי שמרים. (A) נקודת העניין משוכפלת לתוך וקטור תצוגת פני שטח שמרים ובדרך כלל מוקפת בתג N-terminal HA- ובתג c-myc מסוף C. המבנה מאוחה לחלבון ההזדווגות של השמרים Aga2p לתצוגה על פני השטח. החלבון המתואר הוא הקלסר המהונדס "RS3" ממזהה PDB: 6QBA20. (B) תרשים זרימה הממחיש תהליך עבודה טיפוסי עבור קמפיינים לבחירת משטחי שמרים, המשלבים העשרה של גרסאות חלבון עם תכונות רצויות על ידי בחירת חרוזים ומיון ציטומטרי זרימה, כמו גם epPCR להבשלת אהדה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Protocol
1. הפשרה וטיפוח ספריות שמרים
- הפשירו מספיק אליציטוטים של ספריית השמרים הקפואים ב-RT ודיללו מיד ב-SD-CAA (ראו טבלה 1) ל-OD600 של כ-1 (זה מתאים ל-1 x 107 תאים/מ"ל).
הערה: הקפד לכסות את מגוון הספרייה פי 10 לפחות. לכן, אם גודל הספרייה מהווה 2.5 x 108 שיבוטים (זהו המגוון של ספריית G4 המשמשת בדוגמה זו), חסן מינימום של 2.5 x 109 תאים ב 250 מ"ל של SD-CAA כדי להגיע OD600 של 1. פרוטוקול זה יכול גם להתחיל מתרביות נוזליות (תאי שמרים שזה עתה עברו טרנספורמציה עם ספרייה). - הערך את מספר התאים בני קיימא.
- קח aliquot 100 μL מתרחיף שמרים זה (ב OD600 של 1) ולבצע סדרת דילול 1:10 עם 900 μL של SD-CAA בינוני צינורות microcentrifuge.
- הכינו דילולים עד 10-5 וצלחת 100 מיקרוליטר של דילולים 10-3, 10-4 ו-10-5 על לוחות SD-CAA (טבלה 1). לדגור את הצלחות ב 30 ° C במשך 2-3 ימים. ודא שמספר התאים בני קיימא לאחר ההפשרה עולה על מגוון הספרייה לפחות פי 10.
- בינתיים, המשיכו עם מתלה התאים שנותר וטפחו את תרבית השמרים המכילה את ספריית G4 בצלוחיות זכוכית למשך הלילה בטמפרטורה של 30 מעלות צלזיוס תוך כדי רעידות (180 סל"ד). לאחר הדגירה הלילית ממשיכים עם סעיף 2 (השראת ביטוי חלבונים על פני השמרים).
- לאחר 2-3 ימים של דגירה, לספור את המושבות על לוחות SD-CAA על ידי בדיקה חזותית. בעת חישוב המספר הסופי של תאים ברי קיימא לאחר הפשרה, יש לקחת בחשבון את גורם הדילול ואת היחס בין המצופה (כלומר, 100 מיקרוליטר) לעומת נפח התרבית המקורי (כלומר, 250 מ"ל).
תאים קיימא/מ"ל = מספר מושבות על צלחת × גורם דילול
תאים בני קיימא (סה"כ) = (תאים בני קיימא/מ"ל) × נפח תרבית
| בינוני/חיץ | רכיב | ריכוז [גר'/ל'] | הערות/תיאור | |||
| SD-CAA | D-גלוקוז | 20 | ממיסים את כל רכיבי המדיה ב-1000 מ"ל ddH2O ומסננים סטריליים עם מסננים סטריליים חד-פעמיים של 0.22 מיקרומטר. | |||
| בסיס חנקן שמרים | 6.7 | |||||
| חומצות קסמינו | 5 | |||||
| חומצת לימון מונוהידראט | 7.4 | |||||
| טרי-נתרן ציטראט דיהידראט | 10.83 | |||||
| SG-CAA | די-גלקטוז | 20 | ממיסים את כל רכיבי המדיה ב-1000 מ"ל ddH2O ומסננים סטריליים עם מסננים סטריליים חד-פעמיים של 0.22 מיקרומטר. | |||
| D-גלוקוז | 2 | |||||
| בסיס חנקן שמרים | 6.7 | |||||
| חומצות קאמינו | 5 | |||||
| די-נתרן מימן פוספט heptahydrate | 10.2 | |||||
| נתרן דימימן פוספט מונוהידראט | 8.56 | |||||
| לוחות SD-CAA | סורביטול | 182 | להמיס סורביטול, די-נתרן מימן פוספט heptahydrate, נתרן dihydrogen פוספט monohydrate ו agar-agar ב 900 מ"ל ddH2O ו autoclave. ממיסים ומסננים סטריליים את שאר הרכיבים ב-100 מ"ל ddH2O ומוסיפים כאשר המדיה האוטומטית פושרת. | |||
| די-נתרן מימן פוספט heptahydrate | 10.2 | |||||
| נתרן דימימן פוספט מונוהידראט | 7.44 | |||||
| אגר-אגר | 15 | |||||
| D-גלוקוז | 20 | |||||
| בסיס חנקן שמרים | 6.7 | |||||
| חומצות קאמינו | 5 | |||||
| YPD | פפטון | 20 | הכינו ציר 10x D-גלוקוז (200 גרם לליטר) ותסנין סטרילי עם מסננים סטריליים חד פעמיים של 0.22 מיקרומטר. ממיסים פפטון ותמצית שמרים ב 900 מ"ל ddH2O ו autoclave. כאשר פושר, להוסיף 100 מ"ל 10x D-גלוקוז. | |||
| תמצית שמרים | 10 | |||||
| D-גלוקוז | 20 | |||||
| צלחות YPD | פפטון | 20 | הכינו ציר 10x D-גלוקוז (200 גרם לליטר) ותסנין סטרילי עם מסננים סטריליים חד פעמיים של 0.22 מיקרומטר. ממיסים פפטון, תמצית שמרים ואגר-אגר ב 900 מ"ל ddH2O ו autoclave. כאשר פושר, להוסיף 100 מ"ל 10x D-גלוקוז. | |||
| תמצית שמרים | 10 | |||||
| D-גלוקוז | 20 | |||||
| אגר-אגר | 15 | |||||
| PBSA | BSA | 1 | ממיסים BSA ב-PBS ומסננים סטריליים עם מסננים סטריליים חד-פעמיים של 0.22 מיקרומטר. | |||
טבלה 1: מדיה והרכב חיץ.
2. אינדוקציה של ביטוי חלבון על פני השמרים
- לאחר דגירה של לילה, תרביות השמרים מגיעות בדרך כלל ל-OD600 בין 2-20. מדוד את OD600 עם פוטומטר כדי לקבוע את צפיפות תרבית השמרים.
הערה: מדידות OD הן ליניאריות בלבד בטווח של 0.1-1. לכן, בצע דילולים (בדרך כלל 1:20 ו- 1:50) ב- SD-CAA כדי לקבוע את OD600.- דללו את התאים ב-SD-CAA ל-OD600 של 1 וודאו שהמגוון מכוסה פי 10 (כלומר, 2.5 x 109 תאים משמשים לדילול ספריית G4). המשך הדגירה ב 30 ° C תוך כדי רעד כדי לאפשר לתאים להגיע לשלב צמיחה אקספוננציאלי.
- הכינו מלאי גליצרול (המכסה את המגוון פי 50) של ספריות שמרים (ב-SD-CAA המכילות 15% גליצרול) מתרבית הלילה. לשם כך, צנטריפוגה את המספר הנדרש של תאי שמרים (2000 x גרם, 3 דקות, 4 ° C), להשליך את supernatant, ולהשהות מחדש את גלולת התא ב 1 מ"ל של מדיום SD-CAA בתוספת 15% גליצרול. מיד להקפיא מלאי גליצרול ב -80 ° C.
הערה: הכדאיות של תאי שמרים גדלה אם תרביות שמרים גדלות לשלב נייח לפני ההקפאה. בסבבי בחירה מאוחרים יותר, כאשר מגוון הספריות נמוך יותר, בדרך כלל מספיק להשתמש ב-700 מיקרוליטר של תרבית השמרים ולערבב אותה עם 300 מיקרוליטר של SD-CAA בתוספת 50% גליצרול. כאן, תרביות שמרים מראות בדרך כלל ערכי OD600 של 15 בפאזה הנייחת, אך הערך בפועל עשוי להיות תלוי בפוטומטר שבשימוש. לפיכך, ניתן לקבוע את השלב הנייח על ידי מדידות OD600 חוזרות. אם הערך OD600 אינו גדל במהלך תקופת דגירה של 2 שעות, מגיע לשלב נייח.
- צנטריפוגה נפח מתאים של תאים כדי לכסות את המגוון 10x ב 2000 x גרם במשך 5 דקות, 4-6 שעות לאחר דילול של תרבית שמרים (ערכי OD600 טיפוסיים הם בין 4-6), ולהשליך את supernatant.
- השהה מחדש את גלולת התא ב- SG-CAA (ראה טבלה 1) כדי להפיק OD600 של 1 והעבר אותה לצלוחית זכוכית טרייה.
הערה: תרחיף בתווך המכיל גלקטוז גורם לביטוי של החלבון על פני השטח על ידי פרומוטור המושרה בגלקטוז. - יש לדגור על תרביות שמרים למשך הלילה בטמפרטורה של 20 מעלות צלזיוס תוך כדי טלטול ב-180 סל"ד כדי לגרום לביטוי פני השטח של נקודת העניין. לאחר השראת לילה, תרביות שמרים יגיעו בדרך כלל לערכי OD600 בין 2-5; אם טמפרטורת האינדוקציה מועלית ל 37 °C, צפו לערכי OD600 של 4-10.
הערה: הפחתת הטמפרטורה ל-20°C מאטה את צמיחת התאים ובדרך כלל מניבה רמות תצוגה טובות יותר. עם זאת, אינדוקציה בטמפרטורה של 37°C עשויה להיות שימושית על מנת להגביר את לחץ הבחירה כלפי מוטנטים מיוצבים35.
3. סבב בחירת חרוזים ראשון של ספריות שמרים (בחירה חיובית)
הערה: הליך בחירת חרוזים סטנדרטי כולל 6 שלבים (טבלה 2).
| יום | צעד | |
| 0 | תרבות לילה | |
| 1 | השראת ביטוי חלבונים על פני תאי השמרים | |
| 2 | בחירת חרוזים ראשונה עם בחירה חיובית אחת | |
| 3 | הוצאת החרוזים, מעבר, השראת ביטוי חלבונים על פני תאי השמרים והקפאת הספרייה | |
| 4 | בחירת חרוזים שנייה עם 3 בחירה שלילית ובחירה חיובית אחת | |
| 5 | הוצאת החרוזים והקפאת הספרייה | |
טבלה 2: ציר זמן אופייני להולכה של בחירות חרוזים בספריית שמרים.
- הכנת החרוזים
- הכינו את החרוזים (ראו טבלת חומרים) לבחירת החרוזים הראשונה: עבור כל צינור, השהו מחדש 10 μL של חרוזים מגנטיים קושרי ביוטין (4 × 105 חרוזים/μL) ב-990 מיקרוליטר PBSA (טבלה 1) לכביסה, הניחו את הצינור על מדף מגנטי למשך 2 דקות כשהמכסה פתוח. בזהירות להסיר את supernatant. חזור על שלב הכביסה עם 1 מ"ל PBSA.
הערה: אם יש צורך בשפופרות מרובות של חרוזים עם אותו אנטיגן ביוטינילציה, ניתן להכין אותן בשפופרת אחת בשלב זה כדי להניב תמיסת אנטיגן-חרוזים אחידה. במקרה זה, כמות האנטיגן צריכה להיות מוגברת בהתאם בשלב הבא. - להשהות מחדש את החרוזים בנפח כולל של 1 מ"ל PBSA עם 6.7-33 pmoles של אנטיגן biotinylated (כאן: 6.86 μL של hRBP4 עם ריכוז של 0.125 מ"ג / מ"ל) בצינור מיקרוצנטריפוגה 1.5 מ"ל.
הערה: בעוד 6.7 pmoles של אנטיגן מספיקים, 33 pmoles יש להשתמש אם האנטיגן אינו מגביל. אם הבחירה מבוצעת עבור אנטיגן הקשור לתרופה מולקולה קטנה או ליגנד, כמו במקרה זה, תרופה / ליגנד זה חייב להיות נוכח במהלך הבחירה ויש להוסיף בשלב זה (כאן: 5 מיקרומטר A1120). - יש לדגור בטמפרטורה של 4°C למשך שעתיים עם רעידות או סיבוב עדינים.
הערה: ניתן להכין תאים במהלך שלב זה. - לאחר הדגירה ולפני החיבור לתאים, הניחו את הצינור על מדף מגנטי למשך 2 דקות כשהמכסה פתוח. הסר את supernatant ולשטוף את חרוזים טעון אנטיגן עם 1 מ"ל של PBSA כמתואר לעיל. חזרו על שלב הכביסה.
הערה: אם נבחר אנטיגן הקשור לתרופה או ליגנד, מאגר הכביסה צריך להכיל את התרופה או הליגנד בעודף (כאן: 5 מיקרומטר A1120). - להשעות מחדש את החרוזים טעונים אנטיגן ב 50 μL של PBSA (בנוכחות התרופה או ליגנד, אם רלוונטי).
הערה: אם אותם חרוזים טעונים אנטיגן הוכנו עבור צינורות מרובים, להתאים את עוצמת הקול בהתאם (עבור ספריית G4, הנפח הסופי כאן הוא 100 μL מניב חרוזים עבור שני צינורות).
- הכינו את החרוזים (ראו טבלת חומרים) לבחירת החרוזים הראשונה: עבור כל צינור, השהו מחדש 10 μL של חרוזים מגנטיים קושרי ביוטין (4 × 105 חרוזים/μL) ב-990 מיקרוליטר PBSA (טבלה 1) לכביסה, הניחו את הצינור על מדף מגנטי למשך 2 דקות כשהמכסה פתוח. בזהירות להסיר את supernatant. חזור על שלב הכביסה עם 1 מ"ל PBSA.
- הכנת תאי השמרים
- יש למדוד OD600 של תרבית השמרים המושרה כמתואר לעיל.
- גלולה מספיק תאים כדי לכסות פי 10 את המגוון של הספרייה (2000 x גרם, 10 דקות, 4 ° C). עבור ספריית G4, השתמש 2.5 x 109 תאים עבור צנטריפוגה. הסר את supernatant ולשטוף את התאים עם 10 מ"ל של PBSA.
- צנטריפוגה שוב (2000 x גרם, 10 דקות, 4 ° C) ולהשהות מחדש את התאים לריכוז של 1.25 x 109 תאים / מ"ל בנפח המתאים של PBSA (כאן: 2 מ"ל) ולהעביר צינור מיקרוצנטריפוגה.
הערה: עוצמת הקול של PBSA תלויה במספר התאים. - צנטריפוגה (2000 x גרם, 3 דקות, 4 ° C), והשעיה מחדש ב 950 μL של PBSA.
- דגירה של תאי שמרים עם חרוזים טעונים אנטיגן
- הוסף 50 μL של חרוזים טעונים אנטיגן 950 μL של תרחיף התא ודגור במשך 2 שעות ב 4 ° C תוך כדי ניעור או סיבוב.
- לאחר הדגירה, הניחו את תמיסת התא-אנטיגן-חרוזים על מדף מגנטי כשהמכסה פתוח. נוזל פיפטה מהמכסה לצינור כדי לחשוף את כל תרבית השמרים לברירה מגנטית. המתן 2 דקות לפני שתשליך בזהירות תאים לא קשורים. מיד לשטוף עם 1 מ"ל של PBSA.
הערה: אם הבחירות מתבצעות בנוכחות תרופה/ליגנד - בדוגמה זו A1120 - זה צריך להיות נוכח ב- PBSA. - לאחר שלב השטיפה עם PBSA, השהה מחדש את התאים במהירות ב -1 מ"ל של SD-CAA כדי למנוע מהם להתייבש.
- הוסף 200 מ"ל של SD-CAA ובצע דילולים סדרתיים כמתואר לעיל. צלחת 100 μL על לוחות SD-CAA ודגרה במשך 2-3 ימים ב 30 ° C כדי לקבוע את המגוון המרבי החדש. כאן, המגוון של ספריית G4 לאחר בחירת החרוזים הראשונה הוא 1.25 x106 תאים.
הערה: מכיוון שהספרייה המקורית נדגמה פי 10 בסבב בחירה זה, סביר להניח שרוב השיבוטים יהיו נוכחים מספר פעמים במאגר המועשר לאחר הבחירה. כתוצאה מכך, המגוון האמיתי יהיה נמוך יותר, ומספר זה צריך להיחשב כערך מקסימלי. בדרך כלל, המגוון מצטמצם לפחות פי 100 בסבב הבחירה הראשון הזה. לדגור על יתרת התרבית (200 מ"ל) ב 30 ° C עם רעד (180 סל"ד) במשך 16 שעות לפחות. ניתן להשהות את הפרוטוקול לאחר דגירה של לילה על ידי מיקום התאים ב -4 מעלות צלזיוס.
4. הסרת החרוזים וטיפוחם לפני סבב בחירת החרוזים הבא
- מדוד OD600 כמתואר לעיל. גלולה מספיק תאים כדי לכסות את המגוון על ידי לפחות 10x (2000 x גרם, 3 דקות, RT). השליכו את תאי הסופרנאטנט והשהו מחדש ב-1 מ"ל של SD-CAA.
- מעבירים לצינור מיקרוצנטריפוגה ומניחים על מדף מגנטי למשך 2 דקות כשהמכסה פתוח. מעבירים את התאים הלא קשורים לצינור מיקרוצנטריפוגה חדש וחוזרים על תהליך ההפרדה.
- לדלל את התאים הלא קשורים ב 100 מ"ל של SD-CAA ולדגור ב 30 ° C עם רעד ב 180 סל"ד עד OD600 של 3-4 הוא הגיע.
הערה: אם OD600 נמוך מ-4 לפני הסרת החרוזים, ניתן להשרות את התאים מיד לאחר מכן ללא דגירה. - כאשר מגיעים ל-OD600 של 3-4, צנטריפוגו מספר מספיק של תאים כדי לכסות את המגוון לפחות פי 10 (2000 x גרם, 3 דקות, RT) והשהינו מחדש ב-SG-CAA ל-OD600 מתוך 1 להשראת ביטוי חלבונים ודגרו ב-20 מעלות צלזיוס תוך רעידות ב-180 סל"ד במשך הלילה.
- כאשר תרבית השמרים הנותרת של SD-CAA הגיעה לשלב הצמיחה הנייח (OD600 מעל 15), יש מספיק תאים כדי לכסות את המגוון פי 50 (2000 x גרם, 3 דקות, RT). יש להקפיא את התאים באמצעות צנטריפוגה (2000 x גרם, 3 דקות), ולאחר מכן השעיה של גלולת התא ב-SD-CAA המכילה 15% גליצרול ואחסון בטמפרטורה של -80°C.
5. סיבוב בחירת חרוזים שני עם 3 בחירה שלילית ובחירה חיובית אחת
- הכינו את התאים ואת חרוזי האנטיגן כמתואר לעיל. הכינו תמיסה של חרוזים חשופים (מבלי להוסיף את האנטיגן) לבחירות השליליות.
הערה: מכיוון שמבוצעות 3 בחירות שליליות, ניתן להכין את החרוזים יחד לכל שלושת הסבבים. - לאחר השטיפה, להשעות מחדש את חרוזי האנטיגן ב 50 μL של PBSA ולהשעות מחדש חרוזים חשופים ב 150 μL של PBSA (אם הם היו מוכנים לכל שלוש בחירות שליליות).
- עבור הבחירה השלילית הראשונה, הוסף 50 μL של חרוזים חשופים שטופים ל 950 μL של תאים שטופים PBSA ודגור במשך 1.5 שעות ב 4 ° C.
- לאחר הדגירה, הניחו את הצינורות המכילים את מתלי תאי החרוזים החשופים על מדף מגנטי כשהמכסה פתוח. פיפטה כל נוזל במכסה לתוך הצינור ולחכות 2 דקות. מעבירים את התאים הלא קשורים לצינור מיקרוצנטריפוגה טרי ומוסיפים 50 מיקרוליטר של חרוזים חשופים שטופים.
- חזור על התהליך לשני סבבים נוספים של בחירה שלילית. בינתיים, הכינו חרוזים עמוסי אנטיגן לבחירה החיובית.
- לאחר 3 סבבי בחירה שליליים, הוסף 50 μL של תמיסת חרוזים טעונה אנטיגן לתאים ודגור במשך 2 שעות ב 4 ° C.
- הניחו את התאים המכילים את החרוזים הטעונים באנטיגן על מדף מגנטי כשהמכסה פתוח, והכניסו לצינור את כל הנוזלים שנמצאים במכסה. המתן 2 דקות לפני השלכת תאים לא מאוגדים.
- בצע את כל השלבים הנותרים כמתואר עבור בחירת חרוזי האנטיגן הראשונה. ההבדל היחיד לבחירה החיובית הראשונה (שלב 3.3.2) הוא מספר שלבי הכביסה: במקום לשטוף פעם אחת עם PBSA, שטפו את מתלה תאי החרוז פעמיים עם PBSA כדי להגביר את חומרת הבחירה.
הערה: בדרך כלל, המגוון מצטמצם שוב פי 10-100. המגוון המרבי של ספריית G4 לאחר בחירת החרוזים השנייה היה 3.45 x 105 תאים. כפי שצוין לעיל, המגוון בפועל צפוי להיות נמוך יותר בשל דגימת יתר של הספרייה.
6. בחירת ספריות באמצעות מיון ציטומטרי זרימה
- לאחר אינדוקציה לילית של ביטוי פני השטח ב-SG-CAA, יש למדוד OD600 כמתואר לעיל. גלולה מספיק תאים כדי לכסות 10x מגוון (2000 x גרם, 5 דקות, RT). השליכו את הסופרנטנט.
- השהה מחדש את הגלולה ב- PBSA והעבר אותה לצינורות מיקרוצנטריפוגה. השתמש 3 x 107 תאים עבור צביעה עבור כל צינור. הכינו כמה שיותר צינורות, בהתאם לגיוון. הכינו צינור בקרה אחד להכתמה ללא האנטיגן.
הערה: לדוגמה, אם יש למיין 30 x 106 תאים, גלולה 300 x 106 תאים כדי להשיג גלולת תא גלויה ולמנוע אובדן תאים בשלבים הבאים. Resuspend ב 5 מ"ל של PBSA ולהעביר 3 עד 4 aliquots של 500 μL כל אחד לתוך צינורות microcentrifuge. הכינו בערך פי 3 יותר תאים מהמספר שיש למיין מכיוון שחלק קטן של תאים יאבד במהלך תהליך הצביעה. אם הבחירה מבוצעת בנוכחות תרופה/ליגנד, הכינו בקרה נוספת עם האנטיגן בלבד אך ללא התרופה/ליגנד. - צנטריפוגה שוב (2000 x גרם, 5 דקות, RT), להשעות מחדש את הגלולה ב 200 μL של PBSA המכיל את האנטיגן (hRBP4 עם 5 μM A1120) ולדגור במשך 1 שעה ב 4 ° C.
הערה: כל השלבים מנקודה זו ואילך צריכים להתבצע על קרח ועם ריאגנטים קרים. כדי למנוע דלדול נוגדנים או אנטיגן, יש להוסיף את כל הריאגנטים בעודף סטויכיומטרי בהשוואה למספר הכולל של חלבונים המוצגים בשמרים בצינור. אחרת, עלול להיות סיכון של הגבלת הקישור של וריאנטים המוצגים שמרים בשל הזמינות המוגבלת של אנטיגנים ו / או נוגדנים. בדרך כלל, 5 x 104 מולקולות לכל תא שמרים משמשים לחישוב, ומומלץ להוסיף לפחות עודף סטוכומטרי פי 10 של אנטיגן ונוגדנים. אם האנטיגן מוגבל מאוד, ניתן להפחית את העודף פי 5. הסבר מפורט יותר, כולל דוגמאות לחישובים למניעת דלדול ליגנד, ניתן בפרוטוקול קודם9. - לאחר הדגירה, תאי צנטריפוגות (2000 x גרם, 5 דקות, 4 ° C) ולשטוף עם 1 מ"ל של PBSA (אם תרופה / ליגנד נוכח במהלך הבחירות - כמו A1120 במקרה זה - זה צריך להיות נוכח במאגר הכביסה).
- להשהות מחדש את התאים ב 100 μL של PBSA קר המכיל את הנוגדנים עבור צביעת תצוגה וזיהוי של האנטיגן כרוך לדגור במשך 30 דקות ב 4 ° C.
- השתמש בנוגדן anti-c-myc (דילול סופי 1:100) כדי לזהות נקודות עניין מוצגות באורך מלא. השתמש בנוגדן אנטי-HA (דילול סופי 1:50) כדי לזהות את הכמות הכוללת של החלבונים המוצגים. השתמש בנוגדן Penta-His (דילול סופי 1:20) או סטרפטאווידין (דילול סופי 1:200) כדי לבחור תאי שמרים הנקשרים לאנטיגן המסיס המכיל תג HIS פי 6 או ביוטין, בהתאמה.
- החליפו בין ריאגנטים לגילוי בין סבבי מיון כדי למנוע העשרה של וריאנטים חלבוניים הנקשרים למגיב המשני.
הערה: ניתן להשתמש בשילובים שונים של פלואורופורים, אך Alexa Fluor 488 ו- Alexa Fluor 647 הן אפשרויות טובות מכיוון שהן אינן דורשות פיצוי בציטומטריית זרימה. כדי למנוע דלדול נוגדנים, יש להוסיף אותו עודף סטויכיומטרי בהשוואה למספר הכולל של חלבונים המוצגים שמרים בצינור. בדרך כלל, החישובים מבוססים על 5 x 104 מולקולות לכל תא שמרים. הסבר מפורט יותר ניתן בפרוטוקולים קודמים9.
- לאחר הדגירה, תאי צנטריפוגות (2000 x גרם, 5 דקות, 4 ° C) ומוסיפים 1 מ"ל של PBSA (המכיל את התרופה/ליגנד, עבור ספריית G4 שנבחרה hRBP4 5 מיקרומטר של A1120) לגלולה ולצנטריפוגה שוב. הסר את רוב supernatant ולשמור רק 20-30 μL כדי למנוע את הגלולה להתייבש.
- יש להשהות מחדש את הכדורית ב-PBSA קר רק ממש לפני המיון ולמיין את תאי השמרים המציגים מולקולות Fn3 בעלות התכונות הרצויות (כלומר, להיקשר ל-hRBP4 בנוכחות A1120).
הערה: אסטרטגיית ה-gating מתוארת באיור 2A. - מיין את התאים ישירות למדיום SD-CAA, הוסף עוד מדיום SD-CAA לאחר המיון, ודגר ב-30°C עם טלטול ב-180 סל"ד.
הערה: במקרה שהנפח גדל באופן משמעותי לאחר המיון, יש לבצע שלב צנטריפוגה (2000 x גרם, 5 דקות, RT) כדי להסיר את נוזל הנדן העודף, והתאים מושהים מחדש בתווך SD-CAA טרי. רשמו את מספר התאים שמוינו לתוך הצינור החדש, מכיוון שזה הופך למגוון המקסימלי החדש לסבב המיון הבא. כפי שצוין לעיל, המגוון בפועל של הספרייה יהיה נמוך יותר עקב דגימת יתר של הספרייה. - מיונים ציטומטריים של זרימה חוזרת במשך כמה סבבים כדי לראות העשרה של גרסאות חלבון קושרות בספריית השמרים (איור 2B).
הערה: כדאי להשוות את הספריות זו לצד זו בניסוי ציטומטרי של זרימה אנליטית. בסבבי בחירה מאוחרים יותר, הורדת ריכוז האנטיגן יכולה להועיל בהבחנה בין קושרי זיקה נמוכים וגבוהים יותר, ובמיון ספציפי עבור וריאנטים חלבוניים שמראים אות קשירה גבוה יותר למטרה בריכוזים נמוכים (איור 2C). בסבבי בחירה מאוחרים יותר, ניתן גם למיין ליציבות תרמית מוגברת. לשם כך בוצעה מוטגנזה אקראית של הספרייה על ידי epPCR, כמתואר בסעיף 7. הכינו תאים בדומה לסבבי מיון ציטומטריים אחרים של זרימה על ידי אינדוקציה, קביעת OD600 ושטיפת תאים. הניחו תאי שמרים ב-PBSA על קרח למשך 10 דקות, ולאחר מכן דגירה של 10 דקות בתרמוסייקלר להלם חום בטמפרטורה גבוהה. לאחר מכן, הניחו אותם שוב על קרח למשך 10 דקות והמשיכו בפרוטוקול הצביעה. וריאנטים מיוצבים יתנגדו לדנטורציה תרמית, ולכן יראו קשירה מתמשכת לאחר דגירה חום. ניתן לבדוק טמפרטורות שונות להלם חום, בהתאם ליציבות התרמו-יציבה הראשונית של חלבון Wildtype. בדרך כלל, טמפרטורות הנעות בין 40-80 מעלות צלזיוס מתאימות. עם זאת, מעל 48 מעלות צלזיוס, הכדאיות של תאי שמרים תרד באופן דרסטי. לכן, יש צורך לחלץ את פלסמידים לאחר מיון ספריית שמרים הלם חום, כפי שנדון בשלב 7.1. במקרה זה, תאי EBY100 צריכים לעבור טרנספורמציה חדשה עם וריאנטים מועשרים לפני שימשיכו לסבב המיון הבא36. פרוטוקול מפורט יותר להגברת היציבות התרמית ניתן למצוא במקום אחר37. אם הדנטורציה של החלבון המוצג הפיכה, שיטה זו לא תעבוד. לחלופין, השראת ביטוי חלבון ב-37°C במקום ב-20°C ביום שלפני המיון ומיון עבור גרסאות חלבון שעדיין מציגות תצוגה יעילה באורך מלא.
7. הבשלת זיקה עם epPCR כדי להציג מוטציות אקראיות
הערה: ניתן לבצע הבשלת זיקה באמצעות epPCR לפני סבב המיון הציטומטרי הראשון של הזרימה או בין סבבי מיון ציטומטרי של זרימה. לבחירת ספריית G4 עם hRBP4 בנוכחות A1120, הבשלת זיקה נערכה לפני הסבב הראשון של מיון ציטומטרי זרימה. הדבר תלוי גם בגודל הספרייה לאחר בחירת החרוזים ובאות המחייב שניתן לזהות באמצעות ציטומטריית זרימה. בפרט, במקרים בהם זיקות לאחר בחירת חרוזים אינן מספיקות כדי לקבל אות בניסויים ציטומטריים של זרימה (מכיוון שהאנטיגן מתנתק במהירות במהלך שלבי השטיפה), epPCR יכול ליצור וריאנטים משופרים שלאחר מכן ניתן לזהות ולבחור באמצעות ציטומטריית זרימה.
- בצע מיצוי DNA זעיר של שמרים מספריית השמרים המועשרת באמצעות ערכת הפלסמיד המסחרית miniprep II (ראה טבלת חומרים) עם השינויים הבאים:
- למדוד את צפיפות התאים של תרבית לילה, לדלל OD600 של 0.2 בבוקר, ולדגור ב 30 ° C עם רעיד.
- כאשר OD600 מגיע ל -0.6, קצור 3 מ"ל של תרבית על ידי צנטריפוגה של התאים ב 600 x גרם במשך 2 דקות.
- הוסף 200 μL של תמיסה 1 לכדור. הוסף 6 μL של Zymolyase. יש להשהות מחדש את הגלולה על ידי הבהוב או מערבולות קלות ולדגור בטמפרטורה של 37°C למשך 3 שעות.
- מוסיפים 200 μL של תמיסה 2 ומערבבים בעדינות. מוסיפים 400 μL של תמיסה 3 ומערבבים בעדינות.
- צנטריפוגה במהירות מרבית למשך 3 דקות.
- מעבירים את הסופרנאטנט לטור ספין-I, צנטריפוגה במהירות מרבית למשך 30 שניות, ומשליכים את הזרימה.
- יש לשטוף עם 550 μL של Wash Buffer המכיל אתנול וצנטריפוגה במהירות מרבית למשך 2 דקות.
- העבירו את העמוד לצינור מיקרוצנטריפוגה וצנטריפוגה נקיים במהירות מרבית למשך 2 דקות כדי להסיר את שאריות חיץ השטיפה.
- מעבירים את העמוד לצינור מיקרוצנטריפוגה חדש, מוסיפים 10 μL של מים נטולי נוקלאז, ומסתובבים במשך דקה אחת במהירות מרבית כדי לנטרל את הדנ"א.
- בצע PCR ראשוני כדי להגביר את התבנית עבור epPCR.
- להגביר את ה- DNA על ידי PCR באמצעות פריימרים חישול בצד החיצוני של הכנס על וקטור התצוגה של משטח השמרים.
הערה: מכיוון שהרבה דנ"א של שמרים גנומיים מדולל יחד עם הפלסמיד המעניין במהלך הכנת השמרים, שלב זה מומלץ להשיג מקטע DNA נקי שישמש כתבנית עבור epPCR. - עבור pCTCON2, השתמש בפריימרים fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGAG
GGTCGGCTAGC ו rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC עם רכיבי התגובה המתוארים בטבלה 3 ותנאי הרכיבה המתוארים בטבלה 4. - לטהר את ה-PCR עם ערכת ניקוי PCR ו-DNA. Elute ב 20 μL של H2O ללא נוקלאז.
- להגביר את ה- DNA על ידי PCR באמצעות פריימרים חישול בצד החיצוני של הכנס על וקטור התצוגה של משטח השמרים.
- בצע epPCR כדי להציג מוטציות אקראיות.
- בצע epPCR עם אנלוגים נוקלאוטידים עם המרכיבים שצוינו (טבלה 5) ותנאים (טבלה 6).
- הפעל את מוצר epPCR כולו על ג'ל אגרוז מכין 1% ב 120 V למשך 45 דקות. כלול סמן והשאר נתיב אחד בין מוצר epPCR לבין הסולם. הבלו את הרצועה הרצויה המתאימה לגודל הצפוי של העלון, לטהר אותו באמצעות ערכת מיצוי ג'ל, ולמדוד את ריכוז ה- DNA.
- להגביר את ה- DNA האקראי על ידי PCR.
- להגביר 50 ng של DNA עם Q5 פולימראז. הגדר 2 x 100 μL של תגובת PCR עבור כל אלקטרופורציה (טבלה 7) עם התנאים שצוינו (טבלה 8). השתמש באותם פריימרים ששימשו עבור PCR הראשון.
הערה: כמויות גדולות של DNA נדרשות עבור אלקטרופורציה לתוך שמרים. - לנתח 5 μL של מוצר PCR על ג'ל agarose 1%, לרוץ עם 120 V במשך 45 דקות.
- להגביר 50 ng של DNA עם Q5 פולימראז. הגדר 2 x 100 μL של תגובת PCR עבור כל אלקטרופורציה (טבלה 7) עם התנאים שצוינו (טבלה 8). השתמש באותם פריימרים ששימשו עבור PCR הראשון.
- לטהר את הדנ"א המוגבר עם משקעים אתנול.
- בריכה 2 x 100 μL יחד. הוסף 20 μL (10% מהנפח) של סטרילי מסונן 3 M נתרן אצטט, pH 5.2.
הערה: ניתן להכין מאגר זה מראש ולאחסן אותו למשך מספר חודשים ב- RT. - הוסף 440 μL של 100% אתנול (או לפחות 2 x נפח) ודגור ב RT במשך 2 דקות.
- צנטריפוגה ב 20,000 x גרם במשך 5 דקות ב RT. להסיר את supernatant, להוסיף 500 μL של 70% אתנול ולערבב לזמן קצר.
- צנטריפוגה ב 20,000 x גרם במשך 5 דקות ב RT. להסיר את supernatant, להוסיף 500 μL של 100% אתנול ולערבב לזמן קצר.
- צנטריפוגה ב 20,000 x גרם במשך 5 דקות ב RT. להסיר את supernatant ולתת גלולת DNA להתייבש מתחת מכסה זרימה למינרית עד שכל האתנול מתאדה.
הערה: השתמש בבלוק חום ב- 48 ° C כדי להאיץ תהליך זה. ייתכן שכדורית הדנ"א אינה נראית לעין כל הזמן. - ממיסים את כדורית הדנ"א המיובשת ב-10 מיקרוליטר של H2O נטול נוקלאז.
הערה: יש להמיס DNA בנפח קטן ככל האפשר. אין למדוד את ריכוז הדנ"א.
- בריכה 2 x 100 μL יחד. הוסף 20 μL (10% מהנפח) של סטרילי מסונן 3 M נתרן אצטט, pH 5.2.
| נפח [μL] | ריכוז סופי | |
| משפר 5x Q5 | 10 | 1x |
| מאגר 5x Q5 | 10 | 1x |
| פריימר fwd 10 מיקרומטר | 2.5 | 0.5 מיקרומטר |
| פריימר rev 10 מיקרומטר | 2.5 | 0.5 מיקרומטר |
| dNTPs 10 mM | 1 | 200 מיקרומטר |
| Q5 פולימראז | 0.5 | 20 U/מ"ל |
| DNA ממיניפרפ שמרים | 10 | |
| ללא Nuclease H2O | 13.5 |
טבלה 3: תנאים ל-PCR שלבראשון להגברה של גנים POI ממיני-הכנה של שמרים מבודדים.
| צעד | טמפרטורה | זמן |
| דנטורציה ראשונית | 98 °C | 30 שניות |
| 25 מחזורים | 98 °C | 10 שניות |
| 72 °C | 30 שניות | |
| 72 °C | 30 שניות | |
| הארכה סופית | 72 °C | 2 דקות |
| אחז | 4 °C |
טבלה 4: תנאי מחזור עבור PCR שלבראשון להגברה של גנים POI ממיניפרפ שמרים מבודד.
| נפח [μL] | ריכוז סופי | |
| ללא Nuclease H2O | עד 50 | |
| 10x חיץ תרמופול | 5 | 1x |
| Primer_fwd (10 מיקרומטר) | 2.5 | 0.5 מיקרומטר |
| Primer_rev (10 מיקרומטר) | 2.5 | 0.5 מיקרומטר |
| dNTPs (10 mM) | 1 | 200 מיקרומטר |
| 8-אוקסו-dGTP (100 מיקרומטר) | 1 | 2 מיקרומטר |
| dPTP (100 מיקרומטר) | 1 | 2 מיקרומטר |
| מוצר PCR מבית 1st PCR | XX | 50 ng |
| Taq DNA פולימראז | 0.5 | 0.05 U/μL |
טבלה 5: תנאים עבור epPCR המבוצע לאחר הגברה של DNA POI עם PCR שלב1 .
| צעד | טמפרטורה | זמן |
| דנטורציה ראשונית | 94°C | 30 שניות |
| 15 מחזורים | 94°C | 45 שניות |
| 60°C | 30 שניות | |
| 72 °C | 1 דקות | |
| הארכה סופית | 72 °C | 10 דק' |
| אחז | 4 °C |
טבלה 6: תנאי רכיבה על אופניים עבור epPCR.
| נפח [μL] | ריכוז סופי | |
| משפר 5x Q5 | 20 | 1x |
| מאגר 5x Q5 | 20 | 1x |
| פריימר fwd 10 מיקרומטר | 5 | 0.5 מיקרומטר |
| פריימר rev 10 מיקרומטר | 5 | 0.5 מיקרומטר |
| dNTPs 10 mM | 1 | 200 מיקרומטר |
| Q5 פולימראז | 1 | 20 U/מ"ל |
| 50 ננוגרם דנ"א | XX | |
| ddH20 | עד 100 |
טבלה 7: תנאים לשלב השני PCR להגברה של מוצר epPCR לפני אלקטרופורציה של תאי EBY100.
| צעד | טמפרטורה | זמן |
| דנטורציה ראשונית | 98 °C | 30 שניות |
| 25 מחזורים | 98 °C | 10 שניות |
| 72 °C | 30 שניות | |
| 72 °C | 30 שניות | |
| הארכה סופית | 72 °C | 2 דקות |
| אחז | 4 °C |
טבלה 8: תנאי רכיבה על אופניים עבור PCR שלבשני להגברה של מוצר epPCR.
8. ליניאריזציה של וקטור תצוגת שמרים לאלקטרופורציה
- ליניאריזציה של וקטור תצוגת פני השטח של שמרים pCTCON2 (פלסמיד Addgene #41843).
- בצע עיכול בקנה מידה גדול והשתמש בווקטור ליניארי מעוכל זה עבור כל אלקטרופורציות נוספות.
- לעכל 2 x 200 מיקרוגרם של pCTCON2 פלסמיד DNA עם Sal I במשך 24 שעות ב 37 ° C עבור ליניאריזציה ראשונית (טבלה 9).
- יש לעכל עם NheI ו-BamHI במשך 24 שעות ב-37°C כדי להוציא את הבלו (טבלה 10).
- לבסוף, להוסיף 5 μL של כל אנזים לצינור (Sal I, NheI, BamHI) ולדגור שוב במשך 24 שעות ב 37 ° C.
- הפעל את הווקטור המעוכל על ג'ל אגרוז מכין. הבלו על הרצועות וטיהרו עם ערכת מיצוי ג'ל. לבודד את הווקטור ב 20 μL, למדוד את ריכוז ה- DNA ולהתאים אותו בערך 2 μg / μL.
הערה: עיכול הפלסמיד לאורך זמן מבטיח פיצול מספיק של האינסרט ויגרום לפחות רקע מהווקטור לאחר התחשמלות.
| דנ א | 200 מיקרוגרם |
| 10x CutSmartBuffer | 50 מיקרוליטר |
| סאל I-HF (NEB) | 30 מיקרוליטר (60 U) |
| ח2O | עד 500 μL |
טבלה 9: תנאים לשלב הראשון של תקציר בקנה מידה גדול של וקטור תצוגת פני השטח של שמרים pCTCON2.
| pCTCON2 (סאל עיכלתי) | 500 μL |
| 10x CutSmartBuffer | 37.5 מיקרוליטר |
| NheI-HF (NEB) | 15 מיקרוליטר (30 U) |
| BamHI-HF (NEB) | 15 מיקרוליטר (30 U) |
| ח2O | עד 875 μL |
טבלה 10: תנאים לשלב השני של תקציר בקנה מידה גדול של וקטור התצוגה של משטח השמרים pCTCON2.
9. אלקטרופורציה של EBY100 עם DNA אקראי וקטור ליניארי
- הכינו את תאי השמרים EBY100 (ATCC, ראו טבלת חומרים) על ידי הוצאתם ממלאי גליצרול 3 ימים לפני ההתחשמלות. מאחר שתאי EBY100 שלא עברו טרנספורמציה אינם מכילים את וקטור תצוגת פני השטח של שמרים, השתמש בלוחות YPD במדיה מלאה (טבלה 1). לדגור ב 30 °C (75 °F).
- יום אחד לפני ההתחשמלות, יש לחסן מושבה אחת בודדת של EBY100 במדיום YPD טרי של 30 מ"ל (טבלה 1). יש לדגור למשך הלילה ב-30°C תוך רעידות ב-180 סל"ד.
הערה: מדיום YPD לא צריך להיות מעל חודש אחד. - למחרת בבוקר, יש לדלל את תרבית הלילה EBY100 ל-OD600 של 0.2 ב-100 מ"ל של מדיום YPD ולדגור ב-30°C תוך רעידות ב-180 סל"ד.
הערה: נפח תרבית של 50 מ"ל מספיק עבור 2 אלקטרופורציות. בדרך כלל, 100 מ"ל מוכנים להכיל מספיק תאים לבקרות (תאים בלבד ותאים בתוספת וקטור). - כאשר מגיעים ל-OD600 של 1.3-1.5, מכניסים את התאים לצינורות של 50 מ"ל ב-2000 x גרם למשך 3 דקות ומסירים את הסופרנטנט.
- להשהות מחדש את הגלולה ב 25 מ"ל של 100 mM סטרילי מסונן ליתיום אצטט (ראה טבלה של חומרים). הוסף DTT סטרילי מסונן טרי (1 M) (ראה טבלת חומרים) לריכוז סופי של 10 mM.
הערה: נפח הליתיום אצטט צריך להיות מחצית מהנפח המקורי של YPD. הקטן או הגדל במידת הצורך. ליתיום אצטט (100 מילימול) ניתן להכין מראש ולאחסן במשך כמה חודשים ב- RT, אך יש להכין את מלאי ה- DTT של 1 M ממש לפני השימוש ולשמור אותו על קרח. - יש לדגור על התאים בטמפרטורה של 30°C תוך כדי ניעור במשך 10 דקות.
- צנטריפוגה ב 2000 x גרם במשך 3 דקות, להשליך את supernatant, ומניחים תאים על קרח.
הערה: יש לבצע את כל השלבים הבאים על קרח ועם ריאגנטים מקוררים, קוביטות וצינורות מיקרוצנטריפוגה. - להשהות את הגלולה ב 25 מ"ל של קריר סטרילי H2O. צנטריפוגה ב 2000 x גרם במשך 3 דקות.
הערה: אמצעי האחסון H2O שבו נעשה שימוש צריך להיות מחצית מאמצעי האחסון המקורי של YPD. - Resuspend ב 350 μL של סטרילי H2O.
הערה: הנפח הכולל לאחר השעיה צריך להיות 500 μL. הוסף עוד H2O במידת הצורך. - הכינו את ספריית הדנ"א במקביל: הניחו את צינורות המיקרוצנטריפוגות המכילות את התוספות המטוהרות באתנול על קרח והוסיפו 4 מיקרוגרם של וקטור pCTCON2 ליניארי.
- הוסף 250 μL של תאים לדנ"א וערבב בעדינות על ידי חיתוך. מעבירים לקובט אלקטרופורציה 2 מ"מ מקורר מראש ושומרים על קרח עד להתחשמלות.
הערה: הוסף פקדים (תאים בלבד ללא DNA ותאים עם וקטור ליניארי בלבד ללא החדרה) כדי לקבוע את הצלחת האלקטרופורציה. - אלקטרופורטט באמצעות קוביות אלקטרופורציה 2 מ"מ (ראה טבלת חומרים) בתנאים הבאים: פרוטוקול גל מרובע, פולס יחיד, 500 וולט, משך פולס 15 אלפיות השנייה.
הערה: צניחות אופייניות הן 3%-9%. ניתן להשתמש באלקטרופורטורים אחרים, אך התנאים שצוינו מותאמים למכשיר המשמש במחקר זה. - להציל את התאים עם 1 מ"ל של YPD מחומם מראש, להעביר צינור microcentrifuge שחומם מראש, לדגור בלי לרעוד במשך 1 שעה ב 30 ° C.
הערה: מקם את בלוק החימום ליד האלקטרופורטור כדי להבטיח שצינורות השחזור יישארו בטמפרטורה הנכונה. - צלחת 100 μL של 10-3, 10-4, 10-5, ו 10-6 דילול של תאים על לוחות SD-CAA (ראה טבלה 1) ולאפשר צמיחה של טרנספורמנטים ב 30 ° C.
הערה: מגוון הספרייה התיאורטית מתקבל על ידי חישוב מספר הטרנספורמנטים לנפח התרבות המקורי, כאן 1 מ"ל. בדרך כלל, פרוטוקול טרנספורמציה זה מניב בערך 107-10 8 תאים מותמרים. - משחררים את התאים הנותרים ב-2000 x גרם למשך 3 דקות, מרחפים מחדש ב-200 מ"ל SD-CAA ודגרים ב-30°C עם רעידות ב-180 סל"ד למשך הלילה. התאים שעברו טרנספורמציה המכילים את הספרייה האקראית epPCR יכולים לעבור ב- SD-CAA ולגרום להם לביטוי פני השטח ב- SG-CAA למחרת או לאחסן ב- 4 ° C עד לשימוש נוסף. להקפיא מלאי גליצרול של הספרייה לאחר epPCR לאחר קביעת המגוון. מכיוון שלמחרת חלק קטן מהתאים עדיין יהיו תאים שלא עברו טרנספורמציה והועברו בהליך הטרנספורמציה ללא קבלת פלסמיד, מומלץ לעבור לפחות פי 20 ממגוון הספרייה בשלב המעבר הראשון כדי לפצות על השפעה זו.
10. ריצוף ספריות שמרים לאחר מספר סבבי בחירה
- חלצו את הדנ"א של השמרים על ידי ביצוע מיני-הכנה של שמרים כמתואר לעיל.
- אלקטרופורטים מסחריים E. coli 10-beta תאים אלקטרו-מוכשרים (ראה טבלת חומרים) עם DNA מבודד.
- הניחו את קוביות האלקטרופורציה (1 מ"מ, ראו טבלת חומרים) ואת צינורות המיקרוצנטריפוגות על קרח. צינורות מיקרוצנטריפוגות טרום חם עם 950 μL של תווך צמיחה ב 37 ° C כדי לשחזר את התאים לאחר אלקטרופורציה.
- הפשירו את התאים על קרח וערבבו על ידי הזזת הצינור בעדינות. מעבירים 50 μL של התאים לצינור מיקרוצנטריפוגה צונן. הוסף 1 μL של ה- DNA miniprep שמרים.
- מעבירים בזהירות את תערובת התא-דנ"א לקובט אלקטרופורציה צונן.
הערה: הימנעו מבועות וודאו שהתאים שוקעים בתחתית הקובט. - אלקטרופורטט עם מכשיר אלקטרופורציה באמצעות התנאים הבאים: 2.0 קילו וולט; 200 Ω; 25 מיקרופ.
הערה: קבועי זמן אופייניים הם 4.8-5.1 אלפיות השנייה. ניתן להשתמש באלקטרופורטורים אחרים, אך התנאים שצוינו מותאמים למכשיר המשמש במחקר זה. - יש להוסיף מיד 950 μL של 37°C מחומם מראש (ראו טבלת חומרים) לקובטות, לערבב בעדינות ולהעביר בחזרה לצינורות המיקרוצנטריפוגות החמים.
הערה: מקם את בלוק החימום ליד האלקטרופורטור כדי להבטיח שצינורות השחזור יישארו בטמפרטורה הנכונה. - יש לדגור במשך 40 דקות ב-37°C וב-300 סל"ד. צלחת 10 μL, 100 μL, ואת שאר התאים (לאחר צנטריפוגה) על צלחת LB סלקטיבית שחוממה מראש (ראה טבלה 1). לדגור את הצלחות לילה ב 37 °C (77 °F).
הערה: עבור וקטור pCTCON2, אמפיצילין משמש לבחירת אנטיביוטיקה. - הכינו צלחת העתקה עם אגר LB והאנטיביוטיקה הנדרשת (כאן אמפיצילין משמש): פיפטה 200 μL של אגר LB עם 100 מיקרוגרם / מ"ל אמפיצילין בכל באר של צלחת באר 96 ולתת לו להתמצק.
- הכינו צלחת ריצוף 96 היטב עם 50 μL של LB עם 100 מיקרוגרם / מ"ל אמפיצילין לכל באר.
- בחרו מושבה בודדת מהצלחות, וחוסנו תחילה את לוחית ההעתקה ואת אותה באר של צלחת הריצוף.
הערה: לוחית ההעתקה מבטיחה שניתן יהיה להשיג בקלות את הפלסמידים הרצויים על ידי חיסון תרבית נוזלית וביצוע מיניפרפ. - לדגור על צלחת ההעתקה למשך הלילה ב 37 ° C ולאחר מכן לאחסן ב 4 ° C.
- יש לדגור על צלחת הרצף למשך 3-4 שעות בטמפרטורה של 37°C תוך כדי ניעור (180 סל"ד) מכוסה ברדיד אלומיניום או פלסטיק.
- אטמו את לוח הריצוף ושלחו אותו לריצוף יחד עם 500 μL של פריימר ריצוף pCTCON2 בגודל 10 מיקרומטר (5'-CGTTTGTCAGTAATTGCGGTTCTC).
- לניתוח הרצפים, תרגם את רצפי ה- DNA המתקבלים לרצפי חומצות אמינו באמצעות תוכנית מתאימה לבחירה, לדוגמה, EMBOSS Transeq38. לאחר מכן, השתמשו ב-Multiple Sequence Alignment (MUSCLE)39 או בכלי יישור דומה כדי ליישר את רצפי הווריאנטים זה לזה ולחלבון Wildtype ההורי.
הערה: על ידי ביצוע ריצוף ספריות של סבבי בחירה שונים (לדוגמה, לאחר סבב בחירה קודם, וכן לאחר סבבי בחירה נוספים עם ריכוזי אנטיגן נמוכים יותר להעשרת וריאנטים בעלי זיקה גבוהה), ניתן לזהות וריאנטים שונים. הדבר תלוי מאוד בחומרת השערים המיושמים במהלך סבבי המיון, ומכאן בגיוון הספריות.
תוצאות
ספריית G4 נבחרה כנגד האנטיגן hRBP4 הקשור לתרופה המולקולה הקטנה A1120. צביעת הספריות לצורך מיון ציטומטרי של זרימה בוצעה כמתואר בשיטה 6, ואסטרטגיית הגאטינג המיושמת מוצגת באיור 2A. השער הראשוני כלל את כל התאים בהתבסס על מורפולוגיה של התא, והשער השני (היסטוגרמה של FSC-Width) הראה אסטרטגיית gating מחמירה שהוחלה לבחירת תאים בודדים ולהסרת צברי תאים. השער השלישי והאחרון הראה תצוגה של וריאנטים חלבוניים (ציר x) לעומת קשירת אנטיגן (ציר y). תאי שמרים המציגים אותות תצוגה וקשירה מוינו. חשוב לציין, שער המיון נקבע בצורה מחמירה כדי להעשיר תחומי קשירה באות מחייב גבוה ולכן זיקה גבוהה. הבחירה הקפדנית הזו הניבה העשרה של הצגת תאי שמרים שנקשרים באופן ספציפי לאנטיגן המטרה לאורך כל מסע הבחירה (איור 2B). בסבבי מיון ציטומטריים מאוחרים יותר, ריכוז האנטיגן ירד פי 10 (מ-100 ננומטר ל-10 ננומטר). לכן, אות הקשירה הכולל הופחת, ורק קלסרים עם זיקה גבוהה עדיין היו ניתנים לזיהוי ולמיון (איור 2C).
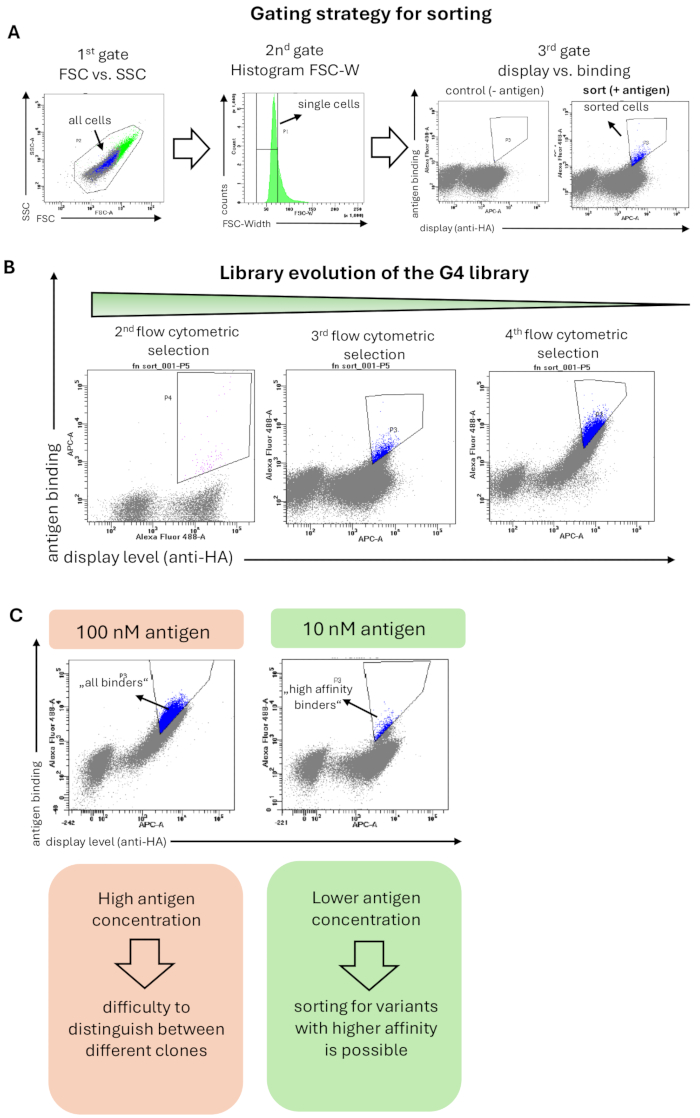
איור 2: תוצאות מייצגות מבחירת משטח שמרים של ספריית G4 מבוססת Fn3 לצורך קשירה לאנטיגן (hRBP4 בנוכחות A1120). (A) אסטרטגיית הגאטינג הכללית למיון ספריות שמרים. השער הראשון (FSC לעומת SSC) הוא לבחור את כל תאי השמרים ולא לכלול אירועי פיזור; השער השני (היסטוגרמה של FSC-W) נועד להסיר צברי תאים ולבחור רק תאי שמרים בודדים. השער השלישי משרטט את רמת תצוגת פני השטח (זיהוי תג HA או c-myc) לעומת קשירה לאנטיגן (כאן hRBP4 בנוכחות 5 מיקרומטר A1120, שזוהה על ידי נוגדן אנטי-שלו). בנוסף, הספרייה הוכתמה בנוגדנים משניים בלבד (ללא אנטיגן), שם לא צפויה קשירת אנטיגן. תאים ממוינים מסומנים בכחול. (B) אבולוציה של ספריית G4 לאורך 3 סבבים של מיון ציטומטרי זרימה. ניתן לצפות בהעשרת האוכלוסייה המחייבת בכל סבב סלקציה. (C) השימוש בריכוזי אנטיגן נמוכים יותר מאפשר בחירה של וריאנטים חלבוניים בעלי זיקה גבוהה יותר לאנטיגן המטרה. עם הפחתת ריכוז האנטיגן (כאן hRBP4) פי 10, מופיעים אלכסונים שונים, המעידים על נוכחות של שיבוטים בעלי זיקה גבוהה יותר (תאים ממוינים, כחולים) או נמוכה יותר. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Discussion
תצוגת פני השטח של שמרים התפתחה כאחת משיטות המפתח המשמשות בהנדסת חלבונים. למרות שהוא משמש בדרך כלל להנדסת זיקה 1,18,40,41, ביטוי/יציבות 24,27,42,43 ופעילות 28,44, שימושים נוספים כמו מיפוי אפיטופ45,46 או אפיון מוטנטים בודדים על פני תאי שמרים9 אפשריים גם כן. בפרוטוקול זה, אנו מספקים את השלבים הבסיסיים להתחלת קמפיין בחירת תצוגת משטח שמרים, כולל הבחירה בחרוזים מגנטיים ועל ידי מיון ציטומטרי של זרימה וכן גיוון ספריית השמרים על ידי epPCR להבשלת זיקה.
אחת הדרישות החיוניות לבחירות קונבנציונליות של משטחי שמרים היא זמינותו של חלבון מסיס באיכות מספקת. החל מחלבון מטרה מקופל היטב עם טוהר גבוה ומצב אוליגומריזציה מוגדר (כלומר, חלבון מונומרי צריך להיות נוכח רק כמונומר) מספק את שיעור ההצלחה הגבוה ביותר לבחירה עבור וריאנט חלבון הנקשר לאנטיגן המטרה עם זיקה גבוהה. חלופה לחלבוני מטרה קשים לביטוי היא בחירות מבוססות תאים, המציגות אסטרטגיה סבירה לעקוף מגבלה זו47. עם זאת, תצוגת פני השטח של השמרים מציעה יתרונות רבים, כגון האפשרות לאפיין גרסאות חלבוניות המתקבלות ישירות על פני השטח של שמרים ללא צורך לבצע שיבוט מייגע וגוזל זמן, ביטוי בפורמט מסיס וטיהור חלבונים. הן את הזיקה והן את יציבות הווריאנטים ניתן לנתח ישירות על משטח שמרים9.
בפרוטוקול זה, אנו מראים כיצד ספריית G4 של גרסאות חלבון, באופן ספציפי יותר של תחום 10סוג III של פיברונקטין אנושי, נבחרה להיקשר לאנטיגן hRBP4 בנוכחות המולקולה הקטנה A1120. השילוב של בחירת חרוזים ומיון ציטומטרי של זרימה הניב העשרה של וריאנטים, שהראו קשירה מוגברת לאנטיגן המטרה לאורך כל סבבי הבחירה (איור 2B). הראינו ששימוש בריכוזים נמוכים יותר של אנטיגן מאפשר בחירה של וריאנטים חלבוניים בעלי זיקה גבוהה (איור 2C). בדרך כלל, זיקות שניתן להשיג עם בחירת תצוגות שמרים הן בתחום הננו-מולארי או אפילו הפיקומולרי18. הזיקות הסופיות תלויות באנטיגן המטרה, במספר סבבי הבחירה והבשלת האהדה, בפיגום המחייב שנעשה בו שימוש ובאסטרטגיית הגאטינג המיושמת. האפיון של וריאנטים חלבוניים בודדים אינו מכוסה בפרוטוקול זה, אך מוסבר בפירוט בעבודתנו הקודמת9. למרות שתצוגת שמרים שימשה במקור להנדסה של מקטעי נוגדנים כגון scFvs 1,40, השיטה נמצאת בשימוש נרחב גם עבור חלבונים שאינם מבוססי נוגדנים10.
לסיכום, תצוגת פני השטח של שמרים היא כלי רב עוצמה להנדסת חלבונים המאפשר יצירת גרסאות חלבון בעלות תכונות חדשניות או משופרות, כגון קשירה כמעט לכל חלבון מטרה ו/או יציבות מוגברת.
Disclosures
M.W.T מקבלת מימון מ-Miltenyi Biotec. כל המחברים הם ממציאים של בקשות פטנט על טכנולוגיות וחלבונים מהונדסים שפותחו באמצעות תצוגת משטח שמרים.
Acknowledgements
עבודה זו נתמכה על ידי הקרן האוסטרית למדע (FWF Project W1224 - Doctoral Program on Biomolecular Technology of Proteins - BioToP and FWF Project ESP 465-B), המשרד הפדרלי לעניינים דיגיטליים וכלכליים של אוסטריה, הקרן הלאומית למחקר, טכנולוגיה ופיתוח של אוסטריה לאגודה הנוצרית לחקר דופלר (Christian Doppler Laboratory for Next Generation CAR T Cells), ועל ידי תרומות פרטיות למכון סנט אנה לחקר סרטן ילדים (וינה, אוסטריה). א.ס. זכה במלגת DOC של האקדמיה האוסטרית למדעים במכון סנט אנה לחקר סרטן ילדים. דמויות נוצרו עם BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
References
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved