Method Article
Ingénierie des protéines par affichage de surface de levure
Dans cet article
Résumé
Ce protocole décrit les étapes essentielles pour mener des campagnes de sélection de présentation de surface de levure afin d’enrichir les variants protéiques se liant à un antigène d’intérêt.
Résumé
L’ingénierie des protéines permet d’améliorer les fonctions existantes d’une protéine donnée ou de générer de nouvelles fonctions. L’un des outils les plus utilisés et les plus polyvalents dans le domaine de l’ingénierie des protéines est l’affichage de la surface de la levure, où un pool de protéines aléatoires est exprimé à la surface de la levure. La liaison du phénotype (p. ex., liaison de la protéine affichée par la levure à l’antigène d’intérêt) et du génotype (le plasmide codant pour la variante protéique) permet de sélectionner cette bibliothèque pour les propriétés souhaitées et de séquencer ultérieurement les variantes enrichies. En combinant la sélection de billes magnétiques avec le tri cytométrique en flux, il est possible de sélectionner et d’enrichir des variants protéiques avec une liaison améliorée à un antigène cible. Notamment, en plus de la maturation de l’affinité, la liaison à une cible peut également être réalisée sans aucune affinité de liaison initiale. Ici, nous fournissons un protocole étape par étape qui couvre toutes les parties essentielles d’une campagne de sélection d’affichage de surface de levure et donne des exemples de résultats typiques d’affichage de surface de levure. Nous démontrons que l’affichage de la surface de la levure est une méthode largement applicable et robuste qui peut être établie dans n’importe quel laboratoire de biologie moléculaire ayant accès à la cytométrie en flux.
Introduction
L’affichage de la surface de la levure est l’une des technologies clés dans le domaine de l’ingénierie des protéines. Il permet de sélectionner des variantes de protéines présentant les propriétés souhaitées telles qu’une affinité ou une stabilité améliorées. Introduite pour la première fois en 19971, c’est l’une des technologies d’affichage les plus couramment utilisées avec l’affichage des phages 2,3, l’affichage des ribosomes4 et l’affichage des cellules de mammifères 5,6,7. La protéine d’intérêt (POI) est affichée à la surface des cellules de levure en la fusionnant pour ancrer des protéines. Une gamme de protéines d’ancrage différentes est disponible, et le plus souvent, le POI est fusionné à l’extrémité C-terminale de la protéine d’accouplement de l’agglutinine de levure Aga2p 1,8. De plus, le POI est généralement flanqué de deux marqueurs, tels qu’un marqueur d’hémagglutinine (HA-tag) et un marqueur c-myc, qui permettent de détecter le niveau d’affichage à l’aide d’anticorps marqués par fluorescence et de la cytométrie en flux (Figure 1A). Les campagnes typiques de sélection de levures impliquent une combinaison de sélections de billes magnétiques et de tri cytométrique en flux. Les sélections de billes permettent de gérer un grand nombre de cellules et d’enrichir les variants protéiques se liant à l’antigène cible, même avec de faibles affinités, car les interactions multivalentes avec les billes chargées d’antigène entraînent des effets d’avidité et, par conséquent, empêchent la perte de variants de faible affinité (Figure 1B). L’analyse et la sélection par cytométrie en flux offrent l’avantage de visualiser la liaison des variants POI affichés à l’antigène marqué. Par conséquent, les populations de liaison peuvent être triées et cultivées, ce qui permet d’enrichir les variantes protéiques avec les caractéristiques souhaitées tout au long de plusieurs cycles de tri. De plus, des cycles supplémentaires de mutagénèse aléatoire peuvent être effectués pour augmenter davantage la diversité et, par conséquent, la probabilité de trouver des mutations supplémentaires qui contribuent à l’affinité et/ou à la stabilité de la protéine.
L’affichage de la surface de la levure présente certains avantages, tels que (a) la machinerie d’expression eucaryote, permettant le repliement oxydatif des protéines ainsi que les modifications post-traductionnelles eucaryotes (telles que la N-glycosylation), (b) la normalisation de l’expression due à la détection des deux marqueurs peptidiques flanquant la protéine, (c) l’inspection visuelle de la progression de la sélection par cytométrie en flux (par exemple, le pourcentage de cellules de liaison et l’intensité de liaison) et (d) la possibilité d’analyser les mutants protéiques individuels sur levure (par exemple, en analysant la thermostabilité ainsi que l’affinité), présentant une alternative permettant de gagner du temps à l’expression et à la purification laborieuses des protéines9. En fait, les affinités (valeurs KD) ainsi que les stabilités (valeurs T50) des protéines de surface de la levure ont montré de bonnes corrélations avec les données obtenues par des méthodes biophysiques et les protéines solubles 9,10,11,12. L’affichage de la surface de la levure a été utilisé pour l’ingénierie d’une variété de protéines, par exemple, les fragments d’anticorps13, 14, 15, 16, le 10e domaine de fibronectine de type III17, 18, rcSso7d19, 20 ou les nœuds21. De même, des recherches approfondies ont été entreprises pour optimiser la conception des banques de levures en modifiant les positions aléatoires ainsi que l’utilisation des codons d’acides aminés 17,22,23. L’affichage de la surface de la levure s’est avéré efficace pour l’ingénierie de la stabilité 14,15,24,25, de l’affinité 18,26,27, de l’activité enzymatique 28,29,30,31 et de l’expression des protéines32. De plus, des applications plus sophistiquées telles que la liaison conditionnelle en présence ou en l’absence d’une petite molécule ont été réalisées à l’aide de l’affichage de surface de levure20.
Dans ce protocole, nous décrivons toutes les étapes essentielles à une campagne de sélection avec affichage de surface chez la levure avec l’exemple de la banque G4 (basée sur le 10ème domainede fibronectine de type III, Fn3) sélectionnée contre l’antigène de la protéine de liaison au rétinol humain 4 (hRBP4) en présence de la petite molécule A112020. Cette sélection a été menée pour produire une interaction protéine-protéine qui dépend d’une petite molécule pouvant être utilisée comme interrupteur moléculaire. Il convient de noter que, bien que d’autres approches soient possibles avec l’affichage de la surface de la levure, les sélections typiques de levures visent généralement à se lier à un antigène cible sans aucune affinité de liaison préalable. Nous couvrons toutes les étapes d’une campagne de sélection de levures, impliquant la culture d’une bibliothèque de levures, la sélection de billes, le tri cytométrique en flux et la maturation d’affinité par PCR sujette aux erreurs (epPCR). Par conséquent, ce protocole complète les protocoles précédents d’affichage de surface de levure33,34 et peut être utilisé comme base pour les sélections d’affichage de surface de levure (Figure 1) avec n’importe quelle bibliothèque de levure donnée et antigène cible de choix.
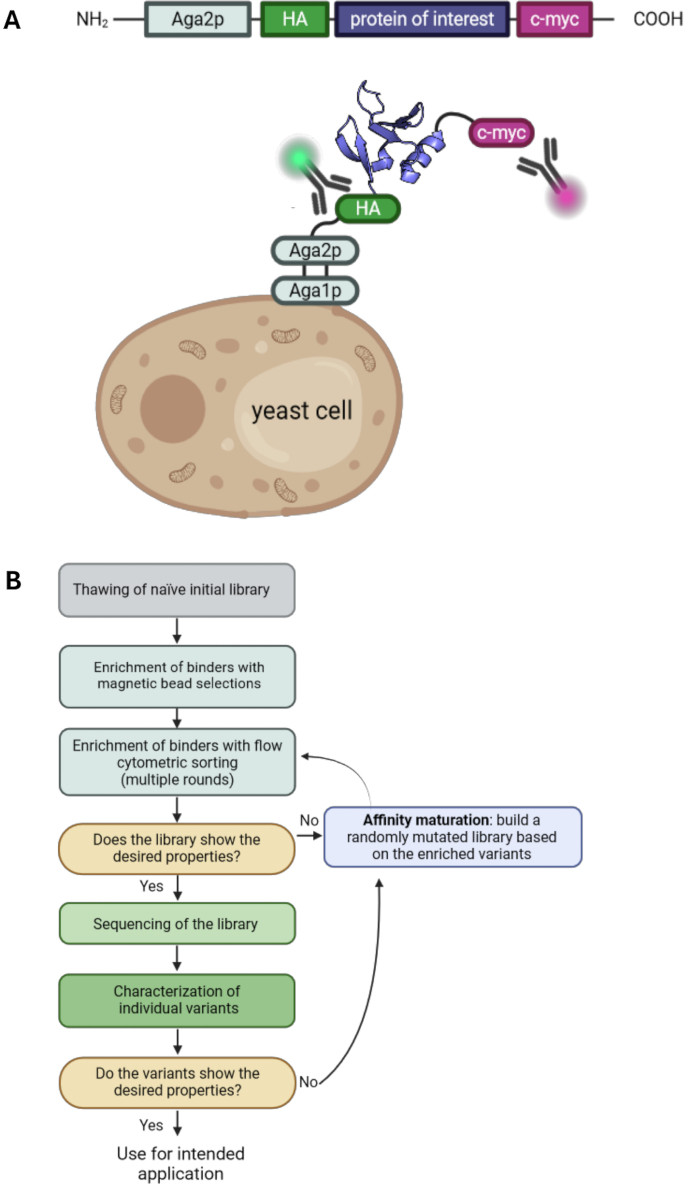
Figure 1 : Principe de l’affichage de la surface de la levure et un flux de travail typique pour les sélections d’affichage de la surface de la levure. (A) Le POI est cloné dans un vecteur d’affichage de surface de levure et généralement flanqué d’un marqueur N-terminal HA- et d’un C-terminal c-myc-tag. La construction est fusionnée à la protéine d’accouplement de levure Aga2p pour être affichée à la surface. La protéine représentée est le liant technique « RS3 » de l’ID PDB : 6QBA20. (B) Organigramme illustrant un flux de travail typique pour les campagnes de sélection d’affichage de surface de levure, qui combinent l’enrichissement des variantes protéiques avec les propriétés souhaitées par sélections de billes et le tri cytométrique en flux, ainsi que l’epPCR pour la maturation d’affinité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Décongélation et culture de banques de levures
- Décongeler suffisamment d’aliquotes de la banque de levures congelées à la RT et diluer immédiatement dans du SD-CAA (voir le tableau 1) jusqu’à une DO600 d’environ 1 (cela correspond à 1 x 107 cellules/mL).
REMARQUE : Assurez-vous de couvrir la diversité de la bibliothèque d’au moins 10 fois. Ainsi, si la taille de la bibliothèque représente 2,5 x 108 clones (c’est la diversité de la bibliothèque G4 utilisée dans cet exemple), inoculez un minimum de 2,5 x 109 cellules dans 250 mL de SD-CAA pour atteindre une OD600 de 1. Ce protocole peut également être lancé à partir de cultures liquides (cellules de levure fraîchement transformées avec une bibliothèque). - Évaluez le nombre de cellules viables.
- Prélever une aliquote de 100 μL de cette suspension de levure (àOD 600 de 1) et effectuer une série de dilutions 1:10 avec 900 μL de milieu SD-CAA dans des tubes de microcentrifugation.
- Préparer des dilutions jusqu’à 10-5 et des plaques de 100 μL de dilutions 10-3, 10-4 et 10-5 sur des plaques SD-CAA (tableau 1). Incuber les plaques à 30 °C pendant 2-3 jours. Assurez-vous que le nombre de cellules viables après décongélation dépasse la diversité de la banque d’au moins 10 fois.
- Pendant ce temps, continuer avec la suspension cellulaire restante et cultiver la culture de levure contenant la banque G4 dans des flacons en verre pendant une nuit à 30 °C en agitant (180 tr/min). Après l’incubation d’une nuit, poursuivre avec la section 2 (Induction de l’expression des protéines à la surface de la levure).
- Après 2-3 jours d’incubation, comptez les colonies sur les plaques SD-CAA par inspection visuelle. Pour calculer le nombre final de cellules viables après décongélation, il faut tenir compte du facteur de dilution et du rapport entre le volume de culture d’origine (c.-à-d. 100 μL) et le volume de culture d’origine (c.-à-d. 250 ml).
Cellules viables/mL = nombre de colonies sur plaque × facteur de dilution
Cellules viables (totales) = (cellules viables/ml) × volume de culture
| Support/tampon | Composant | Concentration [g/L] | Commentaires/Description | |||
| SD-CAA | D-glucose | 20 | Dissoudre tous les composants du média dans 1000 mL de ddH2O et un filtrat stérile avec des filtres stériles jetables de 0,22 μm. | |||
| Base azotée de levure | 6.7 | |||||
| Acides casmino | 5 | |||||
| Acide citrique monohydraté | 7.4 | |||||
| Citrate tri-sodique dihydraté | 10.83 | |||||
| SG-CAA | D-galactose | 20 | Dissoudre tous les composants du média dans 1000 mL de ddH2O et un filtrat stérile avec des filtres stériles jetables de 0,22 μm. | |||
| D-glucose | 2 | |||||
| Base azotée de levure | 6.7 | |||||
| Acides aminés | 5 | |||||
| hydrogénophosphate disodique heptahydraté | 10.2 | |||||
| Dihydrogénophosphate de sodium monohydraté | 8.56 | |||||
| Plaques SD-CAA | Sorbitol | 182 | Dissoudre le sorbitol, l’hydrogénophosphate disodique heptahydraté, le dihydrogénophosphate de sodium monohydraté et l’agar-agar dans 900 mL dejdH2O et l’autoclave. Dissoudre et filtrer stérilement les composants restants dans 100 mL de ddH2O et les ajouter lorsque le milieu autoclavé est tiède. | |||
| hydrogénophosphate disodique heptahydraté | 10.2 | |||||
| Dihydrogénophosphate de sodium monohydraté | 7.44 | |||||
| Agar-agar | 15 | |||||
| D-glucose | 20 | |||||
| Base azotée de levure | 6.7 | |||||
| Acides aminés | 5 | |||||
| YPD | Peptone | 20 | Préparez un stock de D-glucose 10x (200 g/L) et un filtrat stérile avec des filtres stériles jetables de 0,22 μm. Dissoudre la peptone et l’extrait de levure dans 900 mL de jjdH2O et l’autoclave. Lorsqu’il est tiède, ajoutez 100 ml de D-glucose 10x. | |||
| Extrait de levure | 10 | |||||
| D-glucose | 20 | |||||
| Plaques YPD | Peptone | 20 | Préparez un stock de D-glucose 10x (200 g/L) et un filtrat stérile avec des filtres stériles jetables de 0,22 μm. Dissoudre la peptone, l’extrait de levure et l’agar-agar dans 900 mL de jjdH2 O et d’autoclave. Lorsqu’il est tiède, ajoutez 100 ml de D-glucose 10x. | |||
| Extrait de levure | 10 | |||||
| D-glucose | 20 | |||||
| Agar-agar | 15 | |||||
| La | BSA | 1 | Dissoudre le BSA dans le PBS et le filtrat stérile à l’aide de filtres stériles jetables de 0,22 μm. | |||
Tableau 1 : Composition des supports et des tampons.
2. Induction de l’expression des protéines à la surface de la levure
- Après une nuit d’incubation, les cultures de levures atteignent généralement une OD600 entre 2 et 20. Mesurez la DO600 à l’aide d’un photomètre pour déterminer la densité de la culture de levure.
REMARQUE : Les mesures de diamètre extérieur ne sont linéaires que dans la plage de 0,1 à 1. Par conséquent, effectuez des dilutions (généralement 1:20 et 1:50) dans SD-CAA pour déterminer la DO600.- Diluez les cellules dans SD-CAA à une DO600 de 1 et assurez-vous que la diversité est couverte 10x (c’est-à-dire que 2,5 x 109 cellules sont utilisées pour la dilution de la banque G4). Poursuivre l’incubation à 30 °C en agitant pour permettre aux cellules d’atteindre une phase de croissance exponentielle.
- Préparez des stocks de glycérol (couvrant la diversité 50x) de banques de levures (en SD-CAA contenant 15 % de glycérol) à partir de la culture de nuit. Pour ce faire, centrifugez le nombre requis de cellules de levure (2000 x g, 3 min, 4 °C), jetez le surnageant et remettez en suspension la pastille cellulaire dans 1 mL de milieu SD-CAA complété par 15 % de glycérol. Congelez immédiatement les stocks de glycérol à -80 °C.
REMARQUE : La viabilité des cellules de levure est augmentée si les cultures de levure sont cultivées jusqu’à une phase stationnaire avant la congélation. Lors des cycles de sélection ultérieurs, lorsque la diversité des banques est plus faible, il suffit généralement d’utiliser 700 μL de culture de levure et de la mélanger avec 300 μL de SD-CAA complété par 50 % de glycérol. Ici, les cultures de levure présentent généralement des valeurs OD600 de 15 dans la phase stationnaire, mais la valeur réelle peut dépendre du photomètre utilisé. Ainsi, la phase stationnaire peut être déterminée par des mesures répétées de OD600 . Si la valeur de DO600 n’augmente pas sur une période d’incubation de 2 h, une phase stationnaire est atteinte.
- Centrifuger un volume approprié de cellules pour couvrir la diversité 10x à 2000 x g pendant 5 min, 4-6 h après la dilution de la culture de levure (les valeurs typiques de OD600 sont comprises entre 4-6), et jeter le surnageant.
- Remettre la pastille en suspension dans le SG-CAA (voir tableau 1) pour obtenir une OD600 de 1 et la transférer dans une fiole en verre frais.
REMARQUE : La mise en suspension dans un milieu contenant du galactose induit l’expression de la protéine à la surface par le promoteur inductible au galactose. - Incuber les cultures de levure pendant la nuit à 20 °C tout en agitant à 180 tr/min pour induire l’expression en surface du POI. Après une induction nocturne, les cultures de levure atteignent généralement des valeurs OD600 comprises entre 2 et 5 ; si la température d’induction est élevée à 37 °C, attendez-vous à des valeurs OD600 de 4 à 10.
REMARQUE : La réduction de la température à 20°C ralentit la croissance cellulaire et donne généralement de meilleurs niveaux d’affichage. Cependant, l’induction à 37 °C pourrait être utile afin d’augmenter la pression de sélection vers les mutants stabilisés35.
3. Première sélection de billes de banques de levures (sélection positive)
REMARQUE : Une procédure standard de sélection de cordon comporte 6 étapes (Tableau 2).
| Jour | Pas | |
| 0 | Culture du jour au lendemain | |
| 1 | Induction de l’expression des protéines à la surface des cellules de levure | |
| 2 | Première sélection de talons avec 1 sélection positive | |
| 3 | Élimination des billes, passage, induction de l’expression des protéines à la surface des cellules de levure et congélation de la bibliothèque | |
| 4 | Deuxième sélection de perles avec 3 négatifs et 1 positif | |
| 5 | Retrait des billes et congélation de la bibliothèque | |
Tableau 2 : Chronologie typique de la réalisation des sélections de billes d’une bibliothèque de levures.
- Préparation des perles
- Préparez les billes (voir le tableau des matériaux) pour la première sélection de billes : Pour chaque tube, remettre en suspension 10 μL de billes magnétiques de liant de biotine (4 × 105 billes /μL) dans 990 μL de PBSA (tableau 1) pour le lavage, placez le tube sur une grille magnétique pendant 2 min avec le couvercle ouvert. Retirez délicatement le surnageant. Répétez l’étape de lavage avec 1 mL de PBSA.
REMARQUE : Si plusieurs tubes de billes contenant le même antigène biotinylé sont nécessaires, ils peuvent être préparés dans un seul tube à cette étape pour obtenir une solution uniforme d’antigène-billes. Dans ce cas, la quantité d’antigène doit être augmentée en conséquence à l’étape suivante. - Remettre les billes en suspension dans un volume total de 1 mL de PBSA avec 6,7 à 33 pmoles d’antigène biotinylé (ici : 6,86 μL de hRBP4 avec une concentration de 0,125 mg/mL) dans un tube de microcentrifugation de 1,5 mL.
REMARQUE : Bien que 6,7 pmoles d’antigène soient suffisants, 33 pmoles doivent être utilisés si l’antigène n’est pas limitant. Si la sélection est effectuée pour un antigène lié à une petite molécule médicamenteuse ou à un ligand, comme dans ce cas, ce médicament/ligand doit être présent lors de la sélection et doit être ajouté dans cette étape (ici : 5 μM A1120). - Incuber à 4 °C pendant 2 h en secouant doucement ou en tournant.
REMARQUE : Les cellules peuvent être préparées au cours de cette étape. - Après l’incubation et avant l’ajout aux cellules, placez le tube sur une grille magnétique pendant 2 min avec le couvercle ouvert. Retirer le surnageant et laver les billes chargées d’antigène avec 1 mL de PBSA comme décrit ci-dessus. Répétez l’étape de lavage.
REMARQUE : Si un antigène lié à un médicament ou à un ligand est sélectionné, le tampon de lavage doit contenir ce médicament ou ligand en excès (ici : 5 μM A1120). - Remettre en suspension les billes chargées d’antigène dans 50 μL de PBSA (en présence du médicament ou du ligand, le cas échéant).
REMARQUE : Si les mêmes billes chargées d’antigène ont été préparées pour plusieurs tubes, ajustez le volume en conséquence (pour la bibliothèque G4, le volume final est de 100 μL de billes pour deux tubes).
- Préparez les billes (voir le tableau des matériaux) pour la première sélection de billes : Pour chaque tube, remettre en suspension 10 μL de billes magnétiques de liant de biotine (4 × 105 billes /μL) dans 990 μL de PBSA (tableau 1) pour le lavage, placez le tube sur une grille magnétique pendant 2 min avec le couvercle ouvert. Retirez délicatement le surnageant. Répétez l’étape de lavage avec 1 mL de PBSA.
- Préparation des cellules de levure
- Mesurer la DO600 de la culture de levure induite comme décrit ci-dessus.
- Granulez suffisamment de cellules pour couvrir 10 fois la diversité de la bibliothèque (2000 x g, 10 min, 4 °C). Pour la bibliothèque G4, utilisez des cellules 2,5 x 109 pour la centrifugation. Retirer le surnageant et laver les cellules avec 10 mL de PBSA.
- Centrifuger à nouveau (2000 x g, 10 min, 4 °C) et remettre les cellules en suspension à une concentration de 1,25 x 109 cellules/mL dans le volume approprié de PBSA (ici : 2 mL) et transférer dans un tube de microcentrifugation.
REMARQUE : Le volume de PBSA dépend du nombre de cellules. - Centrifuger (2000 x g, 3 min, 4 °C), et remettre en suspension dans 950 μL de PBSA.
- Incubation de cellules de levure avec des billes chargées d’antigène
- Ajouter 50 μL de billes chargées d’antigène à 950 μL de suspension cellulaire et incuber pendant 2 h à 4 °C en agitant ou en tournant.
- Après l’incubation, placez la solution cellule-antigène-billes sur une grille magnétique avec le couvercle ouvert. Pipetez le liquide du couvercle au tube pour soumettre l’ensemble de la culture de levure à une sélection magnétique. Attendez 2 minutes avant de jeter soigneusement les cellules non liées. Laver immédiatement avec 1 mL de PBSA.
REMARQUE : Si les sélections sont effectuées en présence d’un médicament ou d’un ligand - dans cet exemple A1120 - celui-ci doit être présent dans le PBSA. - Après l’étape de lavage avec PBSA, remettre rapidement les cellules en suspension dans 1 mL de SD-CAA pour éviter qu’elles ne se dessèchent.
- Ajouter 200 ml de SD-CAA et effectuer les dilutions en série comme décrit ci-dessus. Plaque de 100 μL sur des plaques SD-CAA et incubation pendant 2 à 3 jours à 30 °C pour déterminer la nouvelle diversité maximale. Ici, la diversité de la bibliothèque G4 après la première sélection de billes est de 1,25 x106 cellules.
REMARQUE : Étant donné que la bibliothèque d’origine a été suréchantillonnée 10 fois lors de ce cycle de sélection, il est raisonnable de supposer que la plupart des clones seront présents plusieurs fois dans le pool enrichi après la sélection. En conséquence, la diversité réelle sera plus faible, et ce nombre doit être considéré comme une valeur maximale. Habituellement, la diversité est réduite d’au moins 100 fois au cours de ce premier tour de sélection. Incuber le reste de la culture (200 ml) à 30 °C en agitant (180 tr/min) pendant au moins 16 h. Le protocole peut être interrompu après une nuit d’incubation en plaçant les cellules à 4 °C.
4. Retrait des billes et culture avant le prochain tour de sélection des billes
- Mesurez OD600 comme décrit ci-dessus. Granulez suffisamment de cellules pour couvrir la diversité d’au moins 10x (2000 x g, 3 min, RT). Jeter le surnageant et remettre les cellules en suspension dans 1 mL de SD-CAA.
- Transférez dans un tube de microcentrifugation et placez-le sur une grille magnétique pendant 2 min avec le couvercle ouvert. Transférez les cellules non liées dans un nouveau tube de microcentrifugation et répétez le processus de séparation.
- Diluer les cellules non liées dans 100 mL de SD-CAA et incuber à 30 °C en agitant à 180 tr/min jusqu’à ce qu’une DO600 de 3-4 soit atteinte.
REMARQUE : Si la DO600 est inférieure à 4 avant l’élimination des billes, les cellules peuvent être induites directement après sans incubation. - Lorsqu’une OD600 de 3-4 est atteinte, centrifuger un nombre suffisant de cellules pour couvrir la diversité au moins 10x (2000 x g, 3 min, RT) et remettre en suspension dans SG-CAA à une OD600 de 1 pour l’induction de l’expression des protéines et incuber à 20 °C tout en agitant à 180 tr/min pendant la nuit.
- Lorsque la culture de levure SD-CAA restante a atteint la phase de croissance stationnaire (OD600 au-dessus de 15), granulez suffisamment de cellules pour couvrir la diversité 50x (2000 x g, 3 min, RT). Congeler les cellules par centrifugation (2000 x g, 3 min), puis remettre en suspension la pastille cellulaire dans du SD-CAA contenant 15 % de glycérol et la stocker à -80 °C.
5. Deuxième tour de sélection de perles avec 3 négatifs et 1 positif
- Préparez les cellules et les billes d’antigène comme décrit ci-dessus. Préparez une solution de billes nues (sans ajouter l’antigène) pour les sélections négatives.
REMARQUE : Étant donné que 3 sélections négatives sont effectuées, les perles peuvent être préparées ensemble pour les trois tours. - Après le lavage, remettre en suspension les billes d’antigène dans 50 μL de PBSA et remettre en suspension les billes nues dans 150 μL de PBSA (si elles ont été préparées pour les trois sélections négatives).
- Pour la première sélection négative, ajouter 50 μL de billes nues lavées à 950 μL de cellules lavées dans du PBSA et incuber pendant 1,5 h à 4 °C.
- Après l’incubation, placez les tubes contenant les suspensions à billes nues sur un support magnétique avec le couvercle ouvert. Pipetez le liquide du couvercle dans le tube et attendez 2 min. Transférez les cellules non liées dans un tube de microcentrifugation frais et ajoutez 50 μL de billes nues lavées.
- Répétez la procédure pour deux autres tours de sélection négative. Pendant ce temps, préparez des billes chargées d’antigène pour la sélection positive.
- Après 3 cycles de sélection négative, ajouter 50 μL de solution de billes chargées d’antigène dans les cellules et incuber pendant 2 h à 4 °C.
- Placez les cellules contenant les billes chargées d’antigène sur un support magnétique avec le couvercle ouvert, et pipetez tout liquide qui se trouve dans le couvercle dans le tube. Attendez 2 minutes avant de jeter les cellules non liées.
- Effectuez toutes les étapes restantes comme décrit pour la première sélection d’antigène-bille. La seule différence par rapport à la première sélection positive (étape 3.3.2) est le nombre d’étapes de lavage : au lieu de laver une fois avec du PBSA, lavez la suspension à billes deux fois avec du PBSA pour augmenter la rigueur de la sélection.
REMARQUE : Habituellement, la diversité est à nouveau réduite de 10x-100x. La diversité maximale de la bibliothèque G4 après cette deuxième sélection de billes était de 3,45 x 105 cellules. Comme indiqué ci-dessus, la diversité réelle devrait être plus faible en raison du suréchantillonnage de la bibliothèque.
6. Sélection des banques par tri cytométrique en flux
- Après l’induction pendant la nuit de l’expression de surface dans SG-CAA, mesurer la DO600 comme décrit ci-dessus. Granulez suffisamment de cellules pour couvrir 10 fois la diversité (2000 x g, 5 min, RT). Jetez le surnageant.
- Remettez la pastille en suspension dans du PBSA et transférez-la dans des tubes de microcentrifugation. Utilisez 3 x 107 cellules pour la coloration de chaque tube. Préparez autant de tubes que nécessaire, en fonction de la diversité. Préparez un tube de contrôle pour la coloration sans l’antigène.
REMARQUE : Par exemple, si 30 x 106 cellules doivent être triées, granulez 300 x 106 cellules pour obtenir une pastille cellulaire visible et éviter la perte de cellules dans les étapes suivantes. Remettre en suspension dans 5 mL de PBSA et transférer 3 à 4 aliquotes de 500 μL chacune dans des tubes de microcentrifugation. Préparez environ 3 fois plus de cellules que le nombre à trier, car une fraction des cellules sera perdue pendant le processus de coloration. Si la sélection est effectuée en présence d’un médicament/ligand, préparer un contrôle supplémentaire avec l’antigène uniquement mais sans le médicament/ligand. - Centrifuger à nouveau (2000 x g, 5 min, RT), remettre en suspension la pastille dans 200 μL de PBSA contenant l’antigène (hRBP4 avec 5 μM A1120) et incuber pendant 1 h à 4 °C.
REMARQUE : Toutes les étapes à partir de ce moment doivent être effectuées sur de la glace et avec des réactifs froids. Pour éviter l’épuisement des anticorps ou de l’antigène, tous les réactifs doivent être ajoutés en excès stœchiométrique par rapport au nombre total de protéines présentées par la levure dans le tube. Sinon, il pourrait y avoir un risque de limiter la liaison des variants présentés par la levure en raison de la disponibilité limitée des antigènes et/ou des anticorps. Habituellement, 5 x 104 molécules par cellule de levure sont utilisées pour le calcul, et l’ajout d’un excès stochiométrique d’antigène et d’anticorps d’au moins 10 fois est conseillé. Si l’antigène est très limité, l’excès peut être réduit à 5 fois. Une explication plus détaillée, y compris des exemples de calculs pour éviter l’épuisement des ligands, est fournie dans un protocole précédent9. - Après l’incubation, centrifuger les cellules (2000 x g, 5 min, 4 °C) et les laver avec 1 mL de PBSA (si un médicament/ligand est présent lors des sélections - comme A1120 dans ce cas - il doit être présent dans le tampon de lavage).
- Remettre les cellules en suspension dans 100 μL de PBSA froid contenant les anticorps pour la coloration et la détection de l’antigène lié et incuber pendant 30 min à 4 °C.
- Utilisez un anticorps anti-c-myc (dilution finale 1:100) pour détecter le POI affiché sur toute la longueur. Utilisez un anticorps anti-HA (dilution finale 1:50) pour détecter la quantité totale de protéines affichées. Utilisez l’anticorps Penta-His (dilution finale 1:20) ou la streptavidine (dilution finale 1:200) pour sélectionner les cellules de levure se liant à l’antigène soluble contenant soit une étiquette HIS 6x, soit de la biotine, respectivement.
- Alternez entre les réactifs de détection entre les cycles de tri pour éviter l’enrichissement des variants protéiques qui se lient au réactif secondaire.
REMARQUE : Différentes combinaisons de fluorophores peuvent être utilisées, mais Alexa Fluor 488 et Alexa Fluor 647 sont de bonnes options car elles ne nécessitent pas de compensation en cytométrie en flux. Pour éviter l’épuisement des anticorps, il doit être ajouté en excès stœchiométrique par rapport au nombre total de protéines affichées par la levure dans le tube. Habituellement, les calculs sont basés sur 5 x 104 molécules par cellule de levure. Une explication plus détaillée est fournie dans les protocoles précédents9.
- Après l’incubation, centrifuger les cellules (2000 x g, 5 min, 4 °C) et ajouter 1 mL de PBSA (contenant le médicament/ligand, pour la banque G4 sélectionnée par hRBP4 5 μM de A1120) à la pastille et centrifuger à nouveau. Retirez la majeure partie du surnageant et ne conservez que 20 à 30 μL pour éviter que la pastille ne se dessèche.
- Remettre la pastille en suspension dans du PBSA froid juste avant le tri et le tri des cellules de levure présentant des molécules Fn3 avec les propriétés souhaitées (c’est-à-dire se liant à hRBP4 en présence de A1120).
REMARQUE : La stratégie de contrôle est illustrée à la figure 2A. - Triez les cellules directement dans le milieu SD-CAA, ajoutez d’autres milieux SD-CAA après le tri et incubez à 30 °C en secouant à 180 tr/min.
REMARQUE : Dans le cas où le volume augmente considérablement après le tri, une étape de centrifugation doit être effectuée (2000 x g, 5 min, RT) pour éliminer l’excès de liquide de gaine, et les cellules sont remises en suspension dans un nouveau milieu SD-CAA. Notez le nombre de cellules triées dans le nouveau tube, car cela devient la nouvelle diversité maximale pour le prochain tour de tri. Comme indiqué ci-dessus, la diversité réelle de la bibliothèque sera plus faible en raison du suréchantillonnage de la bibliothèque. - Répétez les tris cytométriques en flux pendant plusieurs cycles pour voir un enrichissement des variantes de protéines de liaison dans la bibliothèque de levures (Figure 2B).
REMARQUE : Il peut être utile de comparer les banques côte à côte dans une expérience analytique de cytométrie en flux. Lors des cycles de sélection ultérieurs, la réduction de la concentration de l’antigène peut être bénéfique pour distinguer les liants de faible et de haute affinité et pour trier spécifiquement les variants protéiques qui montrent un signal de liaison plus élevé à la cible à de faibles concentrations (Figure 2C). Lors des tours de sélection ultérieurs, il est également possible de trier pour une meilleure thermostabilité. À cette fin, une mutagénèse aléatoire de la banque par epPCR a été effectuée, comme décrit dans la section 7. Préparez des cellules de la même manière que d’autres cycles de tri par cytométrie en flux par induction, détermination dela DO 600 et lavage des cellules. Placez les cellules de levure dans le PBSA sur de la glace pendant 10 min, suivies d’une incubation de 10 min dans un thermocycleur pour le choc thermique à une température élevée. Ensuite, placez-les à nouveau sur de la glace pendant 10 min et procédez au protocole de coloration. Les variantes stabilisées résisteront à la dénaturation thermique et, par conséquent, montreront une liaison maintenue après l’incubation thermique. Différentes températures peuvent être testées pour le choc thermique, en fonction de la thermostabilité initiale de la protéine de type sauvage. En règle générale, des températures allant de 40 à 80 °C conviennent. Cependant, au-dessus de 48 °C, la viabilité des cellules de levure diminuera considérablement. Par conséquent, il est nécessaire d’extraire les plasmides après avoir trié la banque de levures choquées thermiquement, comme indiqué à l’étape 7.1. Dans ce cas, les cellules EBY100 doivent être fraîchement transformées avec des variants enrichis avant de passer au prochain tri36. Un protocole plus détaillé pour augmenter la thermostabilité peut être trouvé ailleurs37. Si la dénaturation de la protéine affichée est réversible, cette méthode ne fonctionnera pas. Alternativement, induire l’expression des protéines à 37 °C au lieu de 20 °C la veille du tri et trier pour les variantes de protéines montrant encore un affichage efficace sur toute la longueur.
7. Maturation de l’affinité avec l’epPCR pour introduire des mutations aléatoires
REMARQUE : La maturation par affinité à l’aide de l’epPCR peut être effectuée soit avant le premier tour de tri par cytométrie en flux, soit entre les cycles de tri par cytométrie en flux. Pour la sélection de la banque G4 avec hRBP4 en présence d’A1120, la maturation d’affinité a été réalisée avant le premier tour de tri cytométrique en flux. Cela dépend également de la taille de la banque après la sélection des billes et du signal de liaison qui peut être détecté par cytométrie en flux. En particulier, dans les cas où les affinités après sélection des billes ne sont pas suffisantes pour obtenir un signal dans les expériences de cytométrie en flux (parce que l’antigène se dissocie rapidement pendant les étapes de lavage), l’epPCR peut générer des variants améliorés qui peuvent ensuite être détectés et sélectionnés par cytométrie en flux.
- Effectuez une extraction d’ADN miniprep de levure à partir de la bibliothèque de levure enrichie à l’aide du kit commercial plasmidique miniprep II (voir la table des matériaux) avec les modifications suivantes :
- Mesurez la densité cellulaire d’une culture de nuit, diluez à une OD600 de 0,2 le matin et incubez à 30 °C en agitant.
- Lorsque la DO600 atteint 0,6, récolter 3 mL de culture en centrifugeant les cellules à 600 x g pendant 2 min.
- Ajouter 200 μL de la solution 1 à la pastille. Ajouter 6 μL de Zymolyase. Remettez la pastille en suspension par effleurement ou par un léger tourbillon et incubez à 37 °C pendant 3 h.
- Ajouter 200 μL de la solution 2 et mélanger délicatement. Ajouter 400 μL de la solution 3 et mélanger délicatement.
- Centrifuger à vitesse maximale pendant 3 min.
- Transférez le surnageant dans la colonne de spin-I, centrifugez à vitesse maximale pendant 30 s et jetez le flux.
- Laver avec 550 μL de tampon de lavage contenant de l’éthanol et centrifuger à vitesse maximale pendant 2 min.
- Transférez la colonne dans un tube de microcentrifugation propre et centrifugez à vitesse maximale pendant 2 min pour éliminer le tampon de lavage résiduel.
- Transférez la colonne dans un nouveau tube de microcentrifugation, ajoutez 10 μL d’eau sans nucléase et tournez pendant 1 min à vitesse maximale pour éluer l’ADN.
- Effectuez une PCR initiale pour amplifier le modèle d’epPCR.
- Amplifier l’ADN par PCR à l’aide d’amorces recuites à l’extérieur de l’insert sur le vecteur d’affichage de la surface de la levure.
REMARQUE : Comme une grande quantité d’ADN génomique de levure est éluée avec le plasmide d’intérêt lors de la mini-préparation de levure, cette étape est recommandée pour obtenir un fragment d’ADN propre à utiliser comme matrice pour l’epPCR. - Pour pCTCON2, utilisez les amorces fwd 5'- GGCTCTGGTGGAGGCGGGGTAGCGGAGGCGGAG
GGTCGGCTAGC et rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTTTGTTCGGATCC avec les composants de réaction décrits au tableau 3 et les conditions de cycle décrites au tableau 4. - Purifiez la PCR avec un kit de nettoyage PCR et ADN. Éluer dans 20 μL de H2O sans nucléases.
- Amplifier l’ADN par PCR à l’aide d’amorces recuites à l’extérieur de l’insert sur le vecteur d’affichage de la surface de la levure.
- Effectuez une epPCR pour introduire des mutations aléatoires.
- Effectuer une epPCR avec des analogues nucléotidiques avec les ingrédients indiqués (tableau 5) et les conditions (tableau 6).
- Exécutez l’ensemble du produit epPCR sur un gel d’agarose préparatif à 1 % à 120 V pendant 45 min. Incluez un marqueur et laissez une voie entre le produit epPCR et l’échelle. Excisez la bande souhaitée correspondant à la taille attendue de l’insert, purifiez-la à l’aide d’un kit d’extraction de gel et mesurez la concentration d’ADN.
- Amplifiez l’ADN randomisé par PCR.
- Amplifiez 50 ng de l’ADN avec une polymérase Q5. Mettre en place une réaction de PCR de 2 x 100 μL pour chaque électroporation (tableau 7) dans les conditions indiquées (tableau 8). Utilisez les mêmes amorces que celles utilisées pour la première PCR.
REMARQUE : De grandes quantités d’ADN sont nécessaires pour l’électroporation en levure. - Analyser 5 μL du produit PCR sur un gel d’agarose à 1 %, passer avec 120 V pendant 45 min.
- Amplifiez 50 ng de l’ADN avec une polymérase Q5. Mettre en place une réaction de PCR de 2 x 100 μL pour chaque électroporation (tableau 7) dans les conditions indiquées (tableau 8). Utilisez les mêmes amorces que celles utilisées pour la première PCR.
- Purifiez l’ADN amplifié avec une précipitation d’éthanol.
- Pool de 2 x 100 μL ensemble. Ajouter 20 μL (10 % du volume) d’acétate de sodium 3 M filtré stérile, pH 5,2.
REMARQUE : Ce tampon peut être préparé à l’avance et stocké pendant quelques mois chez RT. - Ajouter 440 μL d’éthanol à 100 % (ou au moins 2 x volume) et incuber à RT pendant 2 min.
- Centrifuger à 20 000 x g pendant 5 min à RT. Retirer le surnageant, ajouter 500 μL d’éthanol à 70 % et mélanger brièvement.
- Centrifuger à 20 000 x g pendant 5 min à RT. Retirer le surnageant, ajouter 500 μL d’éthanol à 100 % et mélanger brièvement.
- Centrifuger à 20 000 x g pendant 5 min à RT. Retirer le surnageant et laisser sécher la pastille d’ADN sous une hotte à flux laminaire jusqu’à ce que tout l’éthanol se soit évaporé.
REMARQUE : Utilisez un bloc de chaleur à 48 °C pour accélérer ce processus. La pastille d’ADN peut ne pas être visible à tout moment. - Dissoudre la pastille d’ADN séchée dans 10 μL de H2O sans nucléases.
REMARQUE : L’ADN doit être dissous dans le plus petit volume possible. Ne mesurez pas la concentration d’ADN.
- Pool de 2 x 100 μL ensemble. Ajouter 20 μL (10 % du volume) d’acétate de sodium 3 M filtré stérile, pH 5,2.
| Volume [μL] | Concentration finale | |
| 5x rehausseur Q5 | 10 | 1x |
| 5x tampon Q5 | 10 | 1x |
| Amorce fwd 10 μM | 2.5 | 0,5 μM |
| Amorce rev 10 μM | 2.5 | 0,5 μM |
| dNTP 10 mM | 1 | 200 μM |
| Q5 polymérase | 0.5 | 20 U/mL |
| ADN de levure miniprep | 10 | |
| Sans nucléase H2O | 13.5 |
Tableau 3 : Conditions de la 1èreétape de la PCR pour l’amplification des gènes POI à partir de la miniprep de levure isolée.
| Pas | Température | Heure |
| Dénaturation initiale | 98 °C | 30 secondes |
| 25 cycles | 98 °C | 10 secondes |
| 72 °C | 30 secondes | |
| 72 °C | 30 secondes | |
| Extension finale | 72 °C | 2 min |
| Tenir | 4 °C |
Tableau 4 : Conditions de cyclage de la PCR de la1ère étape pour l’amplification des gènes POI à partir du miniprep de levure isolé.
| Volume [μL] | Concentration finale | |
| Sans nucléase H2O | jusqu’à 50 | |
| 10x tampon Thermopol | 5 | 1x |
| Primer_fwd (10 μM) | 2.5 | 0,5 μM |
| Primer_rev (10 μM) | 2.5 | 0,5 μM |
| dNTP (10 mM) | 1 | 200 μM |
| 8-oxo-dGTP (100 μM) | 1 | 2 μM |
| dPTP (100 μM) | 1 | 2 μM |
| Produit PCR à partir de la 1ère PCR | XX | 50 ng |
| Taq ADN polymérase | 0.5 | 0,05 U/μL |
Tableau 5 : Conditions pour l’epPCR qui est réalisée après amplification de l’ADN du POI avec la PCR de la1ère étape.
| Pas | Température | Heure |
| Dénaturation initiale | 94 °C | 30 secondes |
| 15 cycles | 94 °C | 45 secondes |
| 60 °C | 30 secondes | |
| 72 °C | 1 minute | |
| Extension finale | 72 °C | Durée : 10 minutes |
| Tenir | 4 °C |
Tableau 6 : Conditions de cyclisme pour l’epPCR.
| Volume [μL] | Concentration finale | |
| 5x rehausseur Q5 | 20 | 1x |
| 5x tampon Q5 | 20 | 1x |
| Amorce fwd 10 μM | 5 | 0,5 μM |
| Amorce rev 10 μM | 5 | 0,5 μM |
| dNTP 10 mM | 1 | 200 μM |
| Q5 polymérase | 1 | 20 U/mL |
| 50 ng d’ADN | XX | |
| jjH20 | jusqu’à 100 |
Tableau 7 : Conditions de la PCR de 2ème étape pour l’amplification du produit epPCR avant électroporation des cellules EBY100.
| Pas | Température | Heure |
| Dénaturation initiale | 98 °C | 30 secondes |
| 25 cycles | 98 °C | 10 secondes |
| 72 °C | 30 secondes | |
| 72 °C | 30 secondes | |
| Extension finale | 72 °C | 2 min |
| Tenir | 4 °C |
Tableau 8 : Conditions de cyclage de la PCR de la 2eétape pour l’amplification du produit epPCR.
8. Linéarisation du vecteur d’affichage de la levure pour l’électroporation
- Linéariser le vecteur d’affichage de surface de la levure pCTCON2 (plasmide Addgene #41843).
- Effectuez une synthèse à grande échelle et utilisez ce vecteur linéarisé digéré pour toutes les électroporations ultérieures.
- Digérer 2 x 200 μg d’ADN plasmidique pCTCON2 avec Sal I pendant 24 h à 37 °C pour une linéarisation initiale (tableau 9).
- Digestion avec NheI et BamHI pendant 24 h à 37 °C pour exciser l’insert (Tableau 10).
- Enfin, ajoutez 5 μL de chaque enzyme dans le tube (Sal I, NheI, BamHI) et incubez à nouveau pendant 24 h à 37 °C.
- Exécutez le vecteur digéré sur un gel d’agarose préparateur. Exciser les bandes et purifier à l’aide d’un kit d’extraction de gel. Éluer le vecteur en 20 μL, mesurer la concentration d’ADN et l’ajuster à environ 2 μg/μL.
REMARQUE : La digestion prolongée du plasmide assure un clivage suffisant de l’insert et entraînera une diminution du bruit de fond du vecteur après électroporation.
| ADN | 200 μg |
| 10x CutSmartBuffer | 50 μL |
| Sal I-HF (NEB) | 30 μL (60 U) |
| H2O | Jusqu’à 500 μL |
Tableau 9 : Conditions de la première étape de la digestion à grande échelle du vecteur d’affichage de surface de la levure pCTCON2.
| pCTCON2 (Sal que j’ai digéré) | 500 μL |
| 10x CutSmartBuffer | 37,5 μL |
| NheI-HF (NEB) | 15 μL (30 U) |
| BamHI-HF (NEB) | 15 μL (30 U) |
| H2O | jusqu’à 875 μL |
Tableau 10 : Conditions de la deuxième étape de la digestion à grande échelle du vecteur d’affichage de surface de la levure pCTCON2.
9. Électroporation d’EBY100 avec de l’ADN randomisé et un vecteur linéarisé
- Préparez les cellules de levure EBY100 (ATCC, voir tableau des matériaux) en les frappant d’un stock de glycérol 3 jours avant l’électroporation. Étant donné que les cellules EBY100 non transformées ne contiennent pas le vecteur d’affichage de surface de levure, utilisez des plaques YPD à plein milieu (tableau 1). Incuber à 30 °C.
- La veille de l’électroporation, inoculer une seule colonie d’EBY100 dans 30 mL de milieu YPD fraîchement préparé (tableau 1). Incuber une nuit à 30 °C en secouant à 180 tr/min.
REMARQUE : Le support YPD ne doit pas avoir plus de 1 mois. - Le lendemain matin, diluer la culture de nuit EBY100 à une DO600 de 0,2 dans 100 mL de milieu YPD et incuber à 30 °C en agitant à 180 tr/min.
REMARQUE : Un volume de culture de 50 mL est suffisant pour 2 électroporations. En règle générale, 100 mL sont préparés pour avoir suffisamment de cellules pour les témoins (cellules seulement et cellules plus vecteur). - Lorsqu’une OD600 de 1,3-1,5 est atteinte, granulez les cellules dans des tubes de 50 mL à 2000 x g pendant 3 min et retirez le surnageant.
- Remettre la pastille en suspension dans 25 mL d’acétate de lithium filtré stérile de 100 mM (voir le tableau des matériaux). Ajouter du DTT filtré stérile fraîchement préparé (1 M) (voir tableau des matériaux) à une concentration finale de 10 mM.
REMARQUE : Le volume d’acétate de lithium doit être la moitié du volume d’origine de YPD. Réduire ou augmenter la taille si nécessaire. L’acétate de lithium (100 mM) peut être préparé à l’avance et stocké pendant quelques mois à RT, mais le stock DTT de 1 M doit être préparé juste avant utilisation et conservé sur de la glace. - Incuber les cellules à 30 °C en agitant pendant 10 min.
- Centrifugez à 2000 x g pendant 3 min, jetez le surnageant et placez les cellules sur de la glace.
REMARQUE : Toutes les étapes suivantes doivent être effectuées sur de la glace et avec des réactifs refroidis, des cuvettes et des tubes de microcentrifugation. - Remettre la pastille en suspension dans 25 mL de H2O. Centrifugeuse à 2000 x g pendant 3 min.
REMARQUE : Le volume H2O utilisé doit être la moitié du volume YPD d’origine. - Mettre en suspension dans 350 μL de H2O stérile.
REMARQUE : Le volume total après remise en suspension doit être de 500 μL. Ajouter plus de H2O si nécessaire. - Préparez la banque d’ADN en parallèle : placez les tubes de microcentrifugation contenant les inserts purifiés à l’éthanol sur de la glace et ajoutez 4 μg de vecteur pCTCON2 linéarisé.
- Ajoutez 250 μL de cellules à l’ADN et mélangez doucement en coupant. Transférer dans une cuvette d’électroporation pré-refroidie de 2 mm et conserver sur de la glace jusqu’à l’électroporation.
REMARQUE : Ajoutez des contrôles (cellules uniquement sans ADN et cellules avec vecteur linéarisé uniquement sans insert) pour déterminer le succès de l’électroporation. - Électroporat à l’aide de cuvettes d’électroporation de 2 mm (voir Tableau des matériaux) dans les conditions suivantes : protocole d’onde carrée, impulsion unique, 500 V, durée d’impulsion de 15 ms.
REMARQUE : Les affaissement typiques sont de 3 % à 9 %. D’autres électroporateurs peuvent être utilisés, mais les conditions indiquées sont optimisées pour l’instrument utilisé dans cette étude. - Récupérez les cellules avec 1 mL de YPD préchauffé, transférez-les dans un tube de microcentrifugation préchauffé et incubez sans agiter pendant 1 h à 30 °C.
REMARQUE : Placez le bloc chauffant à côté de l’électroporateur pour vous assurer que les tubes de récupération restent à la bonne température. - Plaque 100 μL de dilutions 10-3, 10-4, 10-5 et 10-6 de cellules sur des plaques SD-CAA (voir le tableau 1) et permettre la croissance des transformants à 30 °C.
REMARQUE : La diversité théorique de la bibliothèque est obtenue en recalculant le nombre de transformants au volume de culture d’origine, ici 1 mL. En règle générale, ce protocole de transformation produit environ 107-10 8 cellules transformées. - Granulez les cellules restantes à 2000 x g pendant 3 min, mettez-les en suspension dans 200 ml de SD-CAA et incubez à 30 °C en agitant à 180 tr/min pendant la nuit. Les cellules transformées contenant la banque randomisée epPCR peuvent soit être passées dans SD-CAA et induites pour l’expression en surface dans SG-CAA le lendemain, soit stockées à 4 °C jusqu’à une utilisation ultérieure. Congeler les stocks de glycérol de la bibliothèque après epPCR après avoir déterminé la diversité. Étant donné que le lendemain, une fraction des cellules sera encore constituée de cellules non transformées qui ont été transférées dans le processus de transformation sans obtenir de plasmide, il est recommandé de passer au moins 20 fois la diversité de la banque lors de la première étape de passage pour compenser cet effet.
10. Séquençage des banques de levures après plusieurs cycles de sélection
- Extrayez l’ADN de la levure en effectuant une mini-préparation de levure comme décrit ci-dessus.
- Électroporatez des cellules électrocompétentes commerciales 10-bêta d’E . coli (voir Tableau des matériaux) avec l’ADN isolé.
- Placez les cuvettes d’électroporation (1 mm, voir tableau des matériaux) et les tubes de microcentrifugation sur de la glace. Préchauffer les tubes de microcentrifugation avec 950 μL de milieu de croissance à 37 °C pour récupérer les cellules après électroporation.
- Décongelez les cellules sur de la glace et mélangez en effleurant doucement le tube. Transférez 50 μL des cellules dans un tube de microcentrifugation réfrigéré. Ajouter 1 μL d’ADN miniprep de levure.
- Transférez soigneusement le mélange cellule-ADN dans une cuvette d’électroporation réfrigérée.
REMARQUE : Évitez les bulles et assurez-vous que les cellules se déposent au fond de la cuvette. - Électroporat avec un dispositif d’électroporation dans les conditions suivantes : 2,0 kV ; 200 Ω; 25 μF.
REMARQUE : Les constantes de temps typiques sont de 4,8 à 5,1 ms. D’autres électroporateurs peuvent être utilisés, mais les conditions indiquées sont optimisées pour l’instrument utilisé dans cette étude. - Ajouter immédiatement 950 μL de milieu de croissance préchauffé à 37 °C (voir le tableau des matériaux) dans les cuvettes, mélanger doucement et transférer dans les tubes de microcentrifugation chauds.
REMARQUE : Placez le bloc chauffant à côté de l’électroporateur pour vous assurer que les tubes de récupération restent à la bonne température. - Incuber pendant 40 min à 37 °C et 300 tr/min. Déposer des plaques de 10 μL, 100 μL et le reste des cellules (après centrifugation) sur une plaque LB sélective préchauffée (voir tableau 1). Incuber les plaques toute la nuit à 37 °C.
REMARQUE : Pour le vecteur pCTCON2, l’ampicilline est utilisée pour la sélection des antibiotiques. - Préparez une plaque de copie avec la gélose LB et l’antibiotique requis (ici l’ampicilline est utilisée) : pipetez 200 μL de gélose LB avec 100 μg/mL d’ampicilline dans chaque puits d’une plaque de 96 puits et laissez-la se solidifier.
- Préparez une plaque de séquençage de 96 puits avec 50 μL de LB avec 100 μg/mL d’ampicilline par puits.
- Prélevez une seule colonie dans les plaques, et inoculez d’abord la plaque de copie et le même puits de la plaque de séquençage.
REMARQUE : La plaque de copie garantit que les plasmides souhaités peuvent être facilement obtenus en inoculant une culture liquide et en effectuant une mini-préparation. - Incuber la plaque de copie pendant la nuit à 37 °C, puis la conserver à 4 °C.
- Incuber la plaque de séquençage pendant 3-4 h à 37 °C en l’agitant (180 tr/min) recouverte d’une feuille de plastique ou d’aluminium.
- Scellez la plaque de séquençage et envoyez-la pour le séquençage avec 500 μL d’amorce de séquençage 10 μM pCTCON2 (5'-CGTTTGTCAGTAATTGCGGTTCTC).
- Pour l’analyse des séquences, traduisez les séquences d’ADN obtenues en séquences d’acides aminés à l’aide d’un programme approprié de votre choix, par exemple, EMBOSS Transeq38. Ensuite, utilisez l’alignement de séquences multiples (MUSCLE)39 ou un outil d’alignement similaire pour aligner les séquences variantes les unes sur les autres et sur la protéine parentale de type sauvage.
REMARQUE : En effectuant le séquençage de banques de différents cycles de sélection (par exemple, après un cycle de sélection antérieur, ainsi qu’après des cycles supplémentaires de sélection avec des concentrations d’antigènes plus faibles pour enrichir les variants de haute affinité), il est possible d’identifier différents variants. Cela dépend fortement de la rigueur des portes appliquées lors des rondes de tri et, par conséquent, de la diversité des bibliothèques.
Résultats
La banque G4 a été sélectionnée contre l’antigène hRBP4 lié à la petite molécule médicament A1120. La coloration des banques pour le tri cytométrique en flux a été effectuée comme décrit dans la méthode 6, et la stratégie de contrôle appliquée est illustrée à la figure 2A. Une première porte comprenait toutes les cellules en fonction de la morphologie cellulaire, et la deuxième porte (histogramme de FSC-Width) a montré une stratégie de contrôle stricte qui a été appliquée pour sélectionner des cellules uniques et éliminer les agrégats de cellules. La troisième et dernière porte a montré l’affichage des variantes protéiques (axe x) par rapport à la liaison à l’antigène (axe y). Les cellules de levure présentant à la fois des signaux d’affichage et de liaison ont été triées. Il est important de noter que la porte de tri a été réglée de manière stricte pour enrichir les domaines de liaison avec un signal de liaison élevé et donc une affinité élevée. Cette sélection rigoureuse a permis d’obtenir un enrichissement de cellules de levure qui se lient spécifiquement à l’antigène cible tout au long de la campagne de sélection (Figure 2B). Lors de cycles ultérieurs de tri par cytométrie en flux, la concentration d’antigène a été divisée par 10 (de 100 nM à 10 nM). Par conséquent, le signal de liaison global a été réduit, et seuls les liants ayant une affinité élevée étaient encore détectables et triés (Figure 2C).
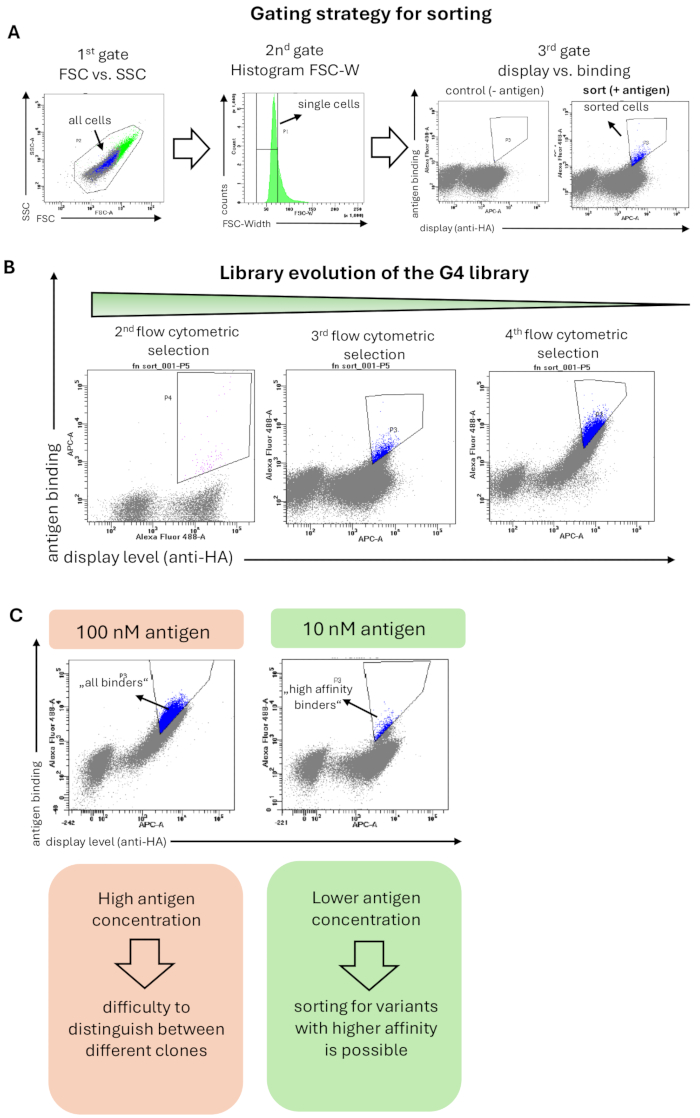
Figure 2 : Résultats représentatifs d’une sélection d’affichage à la surface de la levure de la banque G4 basée sur Fn3 pour la liaison à l’antigène (hRBP4 en présence de A1120). (A) La stratégie générale de contrôle pour le tri des banques de levures. La première étape (FSC vs. SSC) consiste à sélectionner toutes les cellules de levure et à exclure les événements de diffusion ; la deuxième porte (histogramme de FSC-W) vise à éliminer les agrégats cellulaires et à ne sélectionner que des cellules de levure uniques. La troisième porte représente le niveau d’affichage de surface (détection de l’étiquette HA ou c-myc) par rapport à la liaison à l’antigène (ici hRBP4 en présence de 5 μM A1120, détecté par l’anticorps anti-HIS). La banque a en outre été colorée avec des anticorps secondaires uniquement (sans antigène), alors qu’aucune liaison à l’antigène n’est attendue. Les cellules triées sont surlignées en bleu. (B) Evolution de la banque G4 à travers 3 cycles de tri cytométrique en flux. L’enrichissement de la population de liaison peut être observé à chaque cycle de sélection. (C) L’utilisation de concentrations d’antigènes plus faibles permet de sélectionner des variantes de protéines ayant une plus grande affinité envers l’antigène cible. Lors de la réduction de la concentration de l’antigène (ici hRBP4) de 10 fois, différentes diagonales apparaissent, indiquant la présence de clones ayant une affinité supérieure (cellules triées, bleue) ou plus faible. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
L’affichage de la surface de la levure est devenu l’une des principales méthodes utilisées dans l’ingénierie des protéines. Bien qu’il soit couramment utilisé pour l’ingénierie de l’affinité 1,18,40,41, de l’expression/stabilité 24,27,42,43 et de l’activité 28,44, d’autres utilisations comme la cartographie des épitopes45,46 ou la caractérisation des mutants individuels à la surface des cellules de levure9 sont également possibles. Dans ce protocole, nous fournissons les étapes de base pour démarrer une campagne de sélection d’affichage de surface de levure, y compris la sélection par billes magnétiques et par tri cytométrique en flux ainsi que la diversification de la bibliothèque de levures par epPCR pour la maturation par affinité.
Une condition essentielle pour les sélections conventionnelles de présentation de la surface des levures est la disponibilité de protéines solubles de qualité suffisante. Commencer avec une protéine cible bien repliée avec une pureté élevée et un état d’oligomérisation défini (c’est-à-dire que la protéine monomère ne doit être présente que sous forme de monomère) offre le taux de réussite le plus élevé pour sélectionner une variante protéique se liant à l’antigène cible avec une affinité élevée. Une alternative pour les protéines cibles difficiles à exprimer est la sélection cellulaire, qui présente une stratégie raisonnable pour contourner cette limitation47. Cependant, l’affichage de la surface de la levure offre de nombreux avantages, tels que la possibilité de caractériser les variantes protéiques résultantes directement à la surface de la levure sans avoir à effectuer un clonage laborieux et chronophage, l’expression dans un format soluble et la purification des protéines. L’affinité et la stabilité des variantes peuvent être analysées directement à la surface de la levure9.
Dans ce protocole, nous montrons comment la bibliothèque G4 de variants protéiques, plus précisément du 10e domaine de type III de la fibronectine humaine, a été sélectionnée pour se lier à l’antigène hRBP4 en présence de la petite molécule A1120. La combinaison de la sélection des billes et du tri cytométrique en flux a permis d’enrichir les variants, ce qui a montré une liaison accrue à l’antigène cible tout au long des cycles de sélection (figure 2B). Nous avons montré que l’utilisation de concentrations plus faibles d’antigène permet de sélectionner des variants protéiques de haute affinité (Figure 2C). En règle générale, les affinités qui peuvent être obtenues avec des sélections d’affichage de levure sont de l’ordre nanomolaire ou même picomolaire18. Les affinités finales dépendent de l’antigène cible, du nombre de cycles de sélection et de maturation de l’affinité, de l’échafaudage de liaison utilisé et de la stratégie de déclenchement appliquée. La caractérisation de variants protéiques individuels n’est pas couverte dans ce protocole mais est expliquée en détail dans nos travaux précédents9. Bien que l’affichage de levure ait été utilisé à l’origine pour l’ingénierie de fragments d’anticorps tels que scFvs 1,40, la méthode a également été largement utilisée pour les protéines non basées sur des anticorps10.
Pour résumer, l’affichage de la surface de la levure est un puissant outil d’ingénierie des protéines qui permet de générer des variantes de protéines avec des propriétés nouvelles ou améliorées, telles que la liaison à presque toutes les protéines cibles et/ou une stabilité accrue.
Déclarations de divulgation
M.W.T. reçoit un financement de Miltenyi Biotec. Tous les auteurs sont des inventeurs de demandes de brevet pour des technologies et des protéines modifiées qui ont été développées en utilisant l’affichage de surface de levure.
Remerciements
Ce travail a été soutenu par le Fonds autrichien pour la science (projet FWF W1224 - Programme doctoral sur la technologie biomoléculaire des protéines - BioToP et projet FWF ESP 465-B), le ministère fédéral autrichien du Numérique et des Affaires économiques, la Fondation nationale autrichienne pour la recherche, la technologie et le développement à l’Association de recherche Christian Doppler (Christian Doppler Laboratory for Next Generation CAR T Cells), et par des dons privés à l’Institut de recherche sur le cancer des enfants St. Anna (Vienne, Autriche). E.S. est récipiendaire d’une bourse DOC de l’Académie autrichienne des sciences à l’Institut de recherche sur le cancer des enfants St. Anna. Les figurines ont été créées avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
Références
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon