Method Article
酵母表面ディスプレイによるタンパク質工学
要約
このプロトコールは、目的の抗原に結合するタンパク質変異体を濃縮するための酵母表面ディスプレイ選択キャンペーンを実施するための重要なステップを説明しています。
要約
タンパク質工学は、特定のタンパク質の既存の機能を改善したり、新しい機能を生成したりすることを可能にします。タンパク質工学分野で最も広く使用され、汎用性の高いツールの1つは、ランダム化されたタンパク質のプールが酵母の表面に発現する酵母表面ディスプレイです。表現型(例えば、酵母に表示されたタンパク質の目的の抗原への結合)と遺伝子型(タンパク質バリアントをコードするプラスミド)の連鎖により、目的の特性のためのこのライブラリーの選択と、その後の濃縮バリアントのシーケンシングが可能になります。マグネティックビーズの選択とフローサイトメトリーソーティングを組み合わせることで、標的抗原への結合が強化されたタンパク質バリアントを選択し、濃縮することができます。注目すべきは、アフィニティーの成熟に加えて、ターゲットへの結合も初期結合アフィニティーなしで達成できることです。ここでは、酵母表面ディスプレイ選択キャンペーンのすべての重要な部分をカバーし、典型的な酵母表面ディスプレイ結果の例を示すステップバイステップのプロトコルを提供します。私たちは、酵母表面ディスプレイが、フローサイトメトリーにアクセスできるあらゆる分子生物学研究室で確立できる、広く適用可能で堅牢な方法であることを示しています。
概要
酵母表面ディスプレイは、タンパク質工学の分野における重要な技術の1つです。これにより、親和性や安定性の向上など、必要な特性を持つタンパク質バリアントの選択が可能になります。1997年に初めて導入された1、ファージディスプレイ2,3、リボソームディスプレイ4、および哺乳動物細胞ディスプレイ5,6,7以外で最も一般的に使用されるディスプレイ技術の1つです。目的タンパク質(POI)は、酵母細胞を融合させてアンカータンパク質にすることで、酵母細胞の表面に表示されます。さまざまなアンカータンパク質が利用可能であり、最も一般的には、POIは酵母凝集体交配タンパク質Aga2p 1,8のC末端に融合しています。さらに、POIの側面には通常、ヘマグルチニンタグ(HAタグ)やc-mycタグなどの2つのタグがあり、蛍光標識抗体やフローサイトメトリーを使用してディスプレイレベルを検出することができます(図1A)。一般的な酵母選択キャンペーンには、磁気ビーズ選択とフローサイトメトリーソーティングの組み合わせが含まれます。ビーズの選択により、抗原をロードしたビーズとの多価相互作用が結合効果をもたらし、したがって低親和性変異体の喪失を防ぐことができるため、標的抗原に結合するタンパク質変異体の濃縮と、大きな細胞数の処理が可能になります(図1B)。フローサイトメトリーの解析と選択には、表示されたPOIバリアントと標識抗原の結合を視覚化できるという利点があります。その結果、結合集団を選別および培養することができ、数回の選別ラウンドを通じて所望の特性を持つタンパク質変異体を濃縮することができます。さらに、ランダム突然変異誘発の追加ラウンドを行うことで、多様性をさらに高め、したがって、タンパク質の親和性および/または安定性に寄与する追加の突然変異を見つける可能性を高めることができます。
酵母表面ディスプレイには、(a)酸化的タンパク質のフォールディングおよび真核生物の翻訳後修飾(N-グリコシル化など)を可能にする真核生物の発現機構、(b)タンパク質に隣接する2つのペプチドタグの検出による発現の正常化、(c)フローサイトメトリーによる選択進行の目視検査(例えば、結合細胞の割合や結合強度)、(d)個々のタンパク質変異体を解析する可能性など、特定の利点があります。酵母(例えば、熱安定性や親和性の解析)は、面倒なタンパク質の発現と精製に代わる時間節約の代替手段を提供します9。実際、酵母表面表示タンパク質の親和性(KD値)および安定性(T50値)は、生物物理学的方法および可溶性タンパク質9,10,11,12を用いて得られたデータと良好な相関を示している。酵母表面ディスプレイは、種々のタンパク質、例えば、抗体断片13、14、15、16、10番目のIII型フィブロネクチンドメイン17、18、rcSso7d19、20、またはノッチン21のエンジニアリングに採用されている.同様に、ランダム化された位置およびアミノ酸コドンの使用量17,22,23を変更することにより、酵母ライブラリーの設計を最適化するための広範な研究が行われている。酵母表面ディスプレイは、安定性14,15,24,25、親和性18,26,27、酵素活性28,29,30,31、およびタンパク質発現32の工学に成功したことが証明されています.さらに、低分子の存在下または非存在下での条件付き結合のようなより洗練されたアプリケーションは、酵母表面ディスプレイ20を使用して達成された。
このプロトコルでは、小分子A1120 20の存在下で抗原ヒトレチノール結合タンパク質4(hRBP4)に対して選択されたG4ライブラリ(10番目のIII型フィブロネクチンドメイン、Fn3に基づく)の例を使用して、酵母表面ディスプレイを使用した選択キャンペーンのすべての重要なステップについて説明します20。この選択は、分子スイッチとして使用できる小分子に依存するタンパク質間相互作用を生じさせるために行われました。注目すべきは、酵母表面ディスプレイでは別のアプローチが可能ですが、一般的な酵母の選択は通常、以前の結合親和性なしに標的抗原に結合することを目的としています。酵母ライブラリーの培養、ビーズの選択、フローサイトメトリーソーティング、エラー発生しやすいPCR(epPCR)によるアフィニティー成熟など、酵母選択キャンペーンのすべてのステップをカバーしています。したがって、このプロトコルは、以前の酵母表面表示プロトコル33,34を補完し、任意の所与の酵母ライブラリーおよび選択された標的抗原を用いた酵母表面表示選択(図1)の基礎として使用することができる。
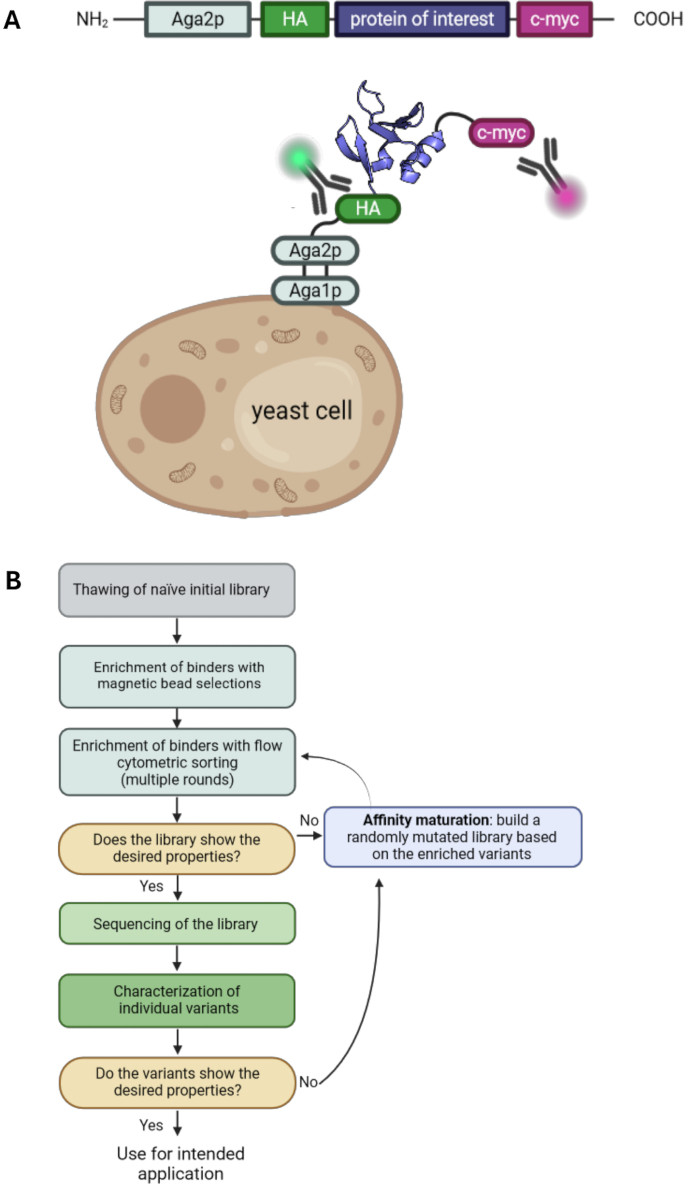
図1:酵母表面ディスプレイの原理と酵母表面ディスプレイ選択の典型的なワークフロー。 (A)POIは、酵母表面表示ベクトルにクローニングされ、通常はN末端HAおよびC末端c-myc-タグが隣接しています。このコンストラクトは、酵母の交配タンパク質Aga2pに融合して表面に表示します。描かれているタンパク質は、PDB ID:6QBA20の改変バインダー「RS3」です。(B)ビーズ選択とフローサイトメトリーソーティングによるタンパク質バリアントの濃縮と所望の特性、およびアフィニティー成熟のためのepPCRを組み合わせた酵母表面ディスプレイ選択キャンペーンの典型的なワークフローを示すフローチャート。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
1. 酵母ライブラリーの解凍と培養
- 凍結酵母ライブラリーの十分なアリコートをRTで解凍し、直ちにSD-CAA( 表1参照)で約1のOD600 (1 x 107 細胞/mLに相当)に希釈します。
注:ライブラリの多様性を少なくとも10倍カバーするようにしてください。したがって、ライブラリサイズが 2.5 x 108 クローン(これがこの例で使用されている G4 ライブラリの多様性)を占める場合、250 mL の SD-CAA に最小 2.5 x 109 細胞を接種して、OD600 1 に到達します。このプロトコールは、液体培養物(ライブラリーで新たに形質転換された酵母細胞)からも開始できます。 - 生細胞の数を評価します。
- この酵母懸濁液(OD600 of 1)から100 μLのアリコートを取り出し、900 μLのSD-CAA培地を微量遠心チューブに入れて1:10希釈シリーズを行います。
- 10-5までの希釈液を調製し、10-3、10-4、10-5の希釈液100 μLをSD-CAAプレートにプレート化します(表1)。プレートを30°Cで2〜3日間インキュベートします。解凍後の生細胞数がライブラリの多様性を少なくとも10倍超えていることを確認してください。
- その間、残りの細胞懸濁液を継続し、G4ライブラリーを含む酵母培養物をガラスフラスコで30°C(180rpm)に振とうしながら一晩培養します。一晩インキュベーションした後、セクション2(酵母表面でのタンパク質発現の誘導)に進みます。
- インキュベーションの2〜3日後、目視検査によりSD-CAAプレート上のコロニーをカウントします。解凍後の生細胞の最終数を計算する際には、希釈係数と、めっきされた細胞(100 μL)と元の培養容量(250 mL)の比率を考慮してください。
生細胞数/mL = プレート上のコロニー数 × 希釈係数
生細胞数(合計)=(生細胞数/mL)×培養量
| ミディアム/バッファ | コンポーネント | 濃度 [g/L] | コメント/説明 | |||
| SD-CAAの | D-グルコース | 20 | すべての培地成分を1000 mL ddH2Oおよび滅菌濾液に溶解し、使い捨ての0.22 μm滅菌フィルターで滅菌します。 | |||
| 酵母窒素ベース | 6.7 | |||||
| カスミノ酸 | 5 | |||||
| クエン酸一水和物 | 7.4 | |||||
| 三クエン酸ナトリウム二水和物 | 10.83 | |||||
| SG-CAAの | D-ガラクトース | 20 | すべての培地成分を1000 mL ddH2Oおよび滅菌濾液に溶解し、使い捨ての0.22 μm滅菌フィルターで滅菌します。 | |||
| D-グルコース | 2 | |||||
| 酵母窒素ベース | 6.7 | |||||
| カサミノ酸 | 5 | |||||
| リン酸二ナトリウム水素七水和物 | 10.2 | |||||
| リン酸二水素ナトリウム一水和物 | 8.56 | |||||
| SD-CAAプレート | ソルビトール | 182 | ソルビトール、リン酸二ナトリウム水素七水和物、リン酸二水素ナトリウム一水和物、寒天を900mLのddH2Oおよびオートクレーブに溶解します。残りの成分を100 mL ddH2Oに溶解して滅菌ろ過し、オートクレーブ処理した培地がぬるま湯になったら加えます。 | |||
| リン酸二ナトリウム水素七水和物 | 10.2 | |||||
| リン酸二水素ナトリウム一水和物 | 7.44 | |||||
| 寒天 | 15 | |||||
| D-グルコース | 20 | |||||
| 酵母窒素ベース | 6.7 | |||||
| カサミノ酸 | 5 | |||||
| YPD(イーピーディー) | ペプトン | 20 | 10x D-グルコースストック(200 g/L)と滅菌濾液を、使い捨ての0.22 μm滅菌フィルターで調製します。ペプトンと酵母抽出物を900mLのddH2Oおよびオートクレーブに溶解します。ぬるま湯になったら、100 mLの10x D-グルコースを加えます。 | |||
| 酵母エキス | 10 | |||||
| D-グルコース | 20 | |||||
| YPDプレート | ペプトン | 20 | 10x D-グルコースストック(200 g/L)と滅菌濾液を、使い捨ての0.22 μm滅菌フィルターで調製します。ペプトン、酵母抽出物、寒天培地を900mLのddH2Oおよびオートクレーブに溶解します。ぬるま湯になったら、100 mLの10x D-グルコースを加えます。 | |||
| 酵母エキス | 10 | |||||
| D-グルコース | 20 | |||||
| 寒天 | 15 | |||||
| PBSAの | BSAの | 1 | BSAをPBSに溶解し、使い捨ての0.22μm滅菌フィルターで滅菌ろ液を溶解します。 | |||
表1:メディアとバッファーの構成。
2. 酵母表面でのタンパク質発現の誘導
- 一晩のインキュベーション後、酵母培養物は通常、2〜20のOD600 に達します。OD600 を光度計で測定し、酵母培養物の密度を決定します。
注:OD測定値は、0.1〜1の範囲でのみ線形です。したがって、SD-CAAで希釈(通常は1:20および1:50)を実行して、OD600を決定します。- SD-CAAの細胞をOD600 1に希釈し、多様性が10倍に覆われていることを確認します(つまり、G4ライブラリの希釈には2.5 x 109 個の細胞が使用されます)。30°Cでインキュベーションを続け、振とうしながら細胞を指数関数的な成長段階に到達させます。
- 酵母ライブラリー(15%グリセロールを含むSD-CAA中)のグリセロールストック(多様性50倍をカバー)を一晩培養した状態で調製します。この目的のために、必要な数の酵母細胞(2000 x g、3分、4°C)を遠心分離し、上清を廃棄し、15%グリセロールを添加したSD-CAA培地1 mLに細胞ペレットを再懸濁します。グリセロールストックを-80°Cで直ちに凍結します。
注:酵母培養物を凍結前に固定相まで増殖すると、酵母細胞の生存率が向上します。その後の選択ラウンドでは、ライブラリーの多様性が低い場合、通常は700 μLの酵母培養物を使用し、50%グリセロールを添加した300 μLのSD-CAAと混合するだけで十分です。ここで、酵母培養物は通常、固定相でOD600 の値を15に示しますが、実際の値は使用中の光度計によって異なる場合があります。したがって、固定相はOD600 の繰り返し測定によって決定できます。OD600 の値が2時間のインキュベーション期間にわたって増加しない場合、固定相に達します。
- 酵母培養液の希釈後4〜6時間後(一般的なOD600値は4〜6時間)に、2000 x gで多様性を10倍に覆うように適切な量の細胞を遠心分離し、上清を廃棄します。
- 細胞ペレットをSG-CAA( 表1参照)に再懸濁してOD600 1を得られ、新しいガラスフラスコに移します。
注:ガラクトース含有培地での再懸濁は、ガラクトース誘導性プロモーターによって表面上のタンパク質の発現を誘導します。 - 酵母培養物を20°Cで一晩インキュベートし、180rpmで振とうして、POIの表面発現を誘導します。一晩の誘導後、酵母培養物は通常、2〜5のOD600 値に達します。誘導温度が37°Cに上昇した場合、OD600 値は4〜10です。
注:温度を20°Cに下げると、細胞の成長が遅くなり、通常は表示レベルが向上します。しかし、37°Cでの誘導は、安定化変異体35に向けた選択圧を高めるために有用であり得る。
3. 酵母ライブラリーの第1回ビーズ選抜ラウンド(ポジティブセレクション)
注意: 標準的なビード選択手順には 6 つのステップが含まれます (表 2)。
| 日 | 歩 | |
| 0 | オーバーナイトカルチャー | |
| 1 | 酵母細胞表面におけるタンパク質発現の誘導 | |
| 2 | 1つの正の選択を持つ最初のビーズ選択 | |
| 3 | ビーズの除去、継代、酵母細胞表面でのタンパク質発現の誘導、およびライブラリーの凍結 | |
| 4 | 3つのネガティブ選択と1つのポジティブ選択による2番目のビーズ選択 | |
| 5 | ビーズの除去とライブラリの凍結 | |
表2:酵母ライブラリーのビーズ選択の伝導に関する典型的なタイムライン。
- ビーズの準備
- 最初のビーズ選択のためにビーズを準備します( 材料の表を参照):各チューブについて、10μLのビオチンバインダー磁気ビーズ(4 × 105 ビーズ/μL)を990μLのPBSA(表1)に再懸濁して洗浄し、蓋を開けた状態でチューブを磁気ラックに2分間置きます。上清を慎重に取り除きます。1mLのPBSAで洗浄ステップを繰り返します。
注:同じビオチン化抗原を含むビーズの複数のチューブが必要な場合は、このステップで1つのチューブに調製して、均一な抗原ビーズ溶液を得ることができます。この場合、次のステップで抗原の量をそれに応じて増やす必要があります。 - 1.5 mLの微量遠心チューブに、6.7-33 pmolesのビオチン化抗原(ここでは、0.125 mg/mLのhRBP4 6.86 μL)を含むPBSAの総量1 mLにビーズを再懸濁します。
注:6.7 pmolesの抗原で十分ですが、抗原が制限されていない場合は33 pmolesを使用する必要があります。.このケースのように、低分子薬物またはリガンドに結合した抗原に対して選択を行う場合、この薬物/リガンドは選択中に存在しなければならず、このステップで追加する必要があります(ここでは、5 μM A1120)。 - 4°Cで2時間インキュベートし、穏やかに振とうまたは回転させます。
注:このステップで細胞を調製できます。 - インキュベーション後、細胞に添加する前に、蓋を開けた状態でチューブを磁気ラックに2分間置きます。上清を取り除き、抗原を充填したビーズを上記のようにPBSA1 mLで洗浄します。洗浄手順を繰り返します。
注:薬物またはリガンドに結合した抗原を選択した場合、洗浄バッファーにはこの薬物またはリガンドを過剰に含む必要があります(ここでは、5μM A1120)。 - 抗原をロードしたビーズを50 μLのPBSAに再懸濁します(薬物またはリガンドが存在する場合)。
注:同じ抗原負荷ビーズを複数のチューブに調製した場合は、それに応じて容量を調整します(G4ライブラリの場合、ここでの最終容量は2本のチューブで100 μLのビーズを生成します)。
- 最初のビーズ選択のためにビーズを準備します( 材料の表を参照):各チューブについて、10μLのビオチンバインダー磁気ビーズ(4 × 105 ビーズ/μL)を990μLのPBSA(表1)に再懸濁して洗浄し、蓋を開けた状態でチューブを磁気ラックに2分間置きます。上清を慎重に取り除きます。1mLのPBSAで洗浄ステップを繰り返します。
- 酵母細胞の調製
- 上記のように誘導された酵母培養物のOD600 を測定します。
- ライブラリーの多様性の10倍をカバーするのに十分な細胞をペレット化します(2000 x g、10分、4°C)。G4ライブラリの場合、遠心分離には2.5 x 109 細胞を使用します。上清を取り除き、10mLのPBSAで細胞を洗浄します。
- 再度遠心分離(2000 x g、10分、4°C)し、細胞を1.25 x 109 細胞/mLの濃度まで適量のPBSA(ここでは2 mL)に再懸濁し、微量遠心チューブに移します。
注:PBSAの量はセルの数によって異なります。 - 遠心分離(2000 x g、3分、4°C)し、950 μLのPBSAに再懸濁します。
- 抗原をロードしたビーズによる酵母細胞のインキュベーション
- 950 μLの細胞懸濁液に50 μLの抗原負荷ビーズを添加し、振とうまたは回転させながら4°Cで2時間インキュベートします。
- インキュベーション後、細胞抗原ビーズ溶液を蓋を開けた状態で磁気ラックに置きます。蓋からチューブに液体をピペットで移し、酵母培養物全体を磁気選択にかけます。2分間待ってから、結合されていない細胞を慎重に破棄します。すぐにPBSA1mLで洗浄してください。
注:薬物/リガンド(この例ではA1120)の存在下で選択が行われる場合、これはPBSAに存在する必要があります。 - PBSAによる洗浄ステップの後、細胞が乾燥するのを防ぐために、細胞を1 mLのSD-CAAに急速に再懸濁します。
- 200 mLのSD-CAAを加え、上記のように段階希釈を行います。SD-CAAプレートに100 μLをプレートし、30°Cで2〜3日間インキュベートして、新しい最大ダイバーシティを決定します。ここで、最初のビーズ選択後のG4ライブラリの多様性は1.25 x106 セルです。
注:この選択ラウンドでは、元のライブラリが10倍にオーバーサンプリングされたため、ほとんどのクローンは選択後に濃縮プールに複数回存在すると考えるのが妥当です。結果として、実際の多様性は低くなり、この数値は最大値と見なす必要があります。通常、多様性は、この最初の選択ラウンドで少なくとも100倍減少します。残りの培養物(200mL)を30°Cで、振とう(180rpm)しながら少なくとも16時間インキュベートします。プロトコールは、細胞を4°Cに置くことにより、一晩のインキュベーション後に一時停止することができます。
4. ビーズの除去と次回のビーズ選択ラウンド前の培養
- 上記のようにOD600 を測定します。多様性を少なくとも10倍(2000 x g、3分、RT)カバーするのに十分な細胞をペレット化します。上清を捨て、細胞を1mLのSD-CAAに再懸濁します。
- 微量遠心チューブに移し、蓋を開けた状態で磁気ラックに2分間置きます。結合していない細胞を新しい微量遠心チューブに移し、分離プロセスを繰り返します。
- 非結合細胞を100 mLのSD-CAAで希釈し、OD600 が3-4に達するまで180 rpmで振とうしながら30°Cでインキュベートします。
注:ビーズの除去前にOD600 が4未満の場合、細胞はインキュベーションせずに直後に誘導できます。 - OD600 が3-4に達したら、多様性を少なくとも10倍(2000 x g、3分、RT)カバーするのに十分な数の細胞を遠心分離し、SG-CAAでOD600 を1に再懸濁してタンパク質発現を誘導し、180 rpmで一晩振とうしながら20°Cでインキュベートします。
- 残りのSD-CAA酵母培養物が定常増殖段階(OD600 以上15)に達したら、多様性を50倍(2000 x g、3分、RT)に覆うのに十分な細胞をペレット化します。細胞を遠心分離(2000 x g、3分)で凍結した後、15%グリセロールを含むSD-CAAに細胞ペレットを再懸濁し、-80°Cで保存します。
5. 3つのネガティブセレクションと1つのポジティブセレクションによる2回目のビーズセレクションラウンド
- 上記のように細胞および抗原ビーズを調製する。ネガティブセレクションのために、(抗原を添加せずに)ベアビーズの溶液を調製します。
注:3つのネガティブセレクションが実行されるため、ビーズは3ラウンドすべてで一緒に調製できます。 - 洗浄後、抗原ビーズを50 μLのPBSAに再懸濁し、ベアビーズを150 μLのPBSAに再懸濁します(3つのネガティブセレクションすべてに対して調製された場合)。
- 最初のネガティブセレクションでは、PBSA中の洗浄細胞950 μLに洗浄したベアビーズ50 μLを加え、4°Cで1.5時間インキュベートします。
- インキュベーション後、ベアビーズ細胞懸濁液を含むチューブを、蓋を開けた状態で磁気ラックに置きます。蓋の中の液体をチューブにピペットで入れ、2分間待ちます。結合していない細胞を新しい微量遠心チューブに移し、洗浄したベアビーズ50μLを加えます。
- この手順を繰り返して、さらに 2 ラウンドのネガティブ選択を行います。その間、ポジティブセレクションのために抗原を充填したビーズを準備します。
- ネガティブセレクションラウンドを3回行った後、抗原をロードしたビーズ溶液50 μLを細胞に加え、4°Cで2時間インキュベートします。
- 抗原をロードしたビーズを含む細胞を、蓋を開けたマグネットラックに置き、蓋の中にある液体をチューブにピペットで入れます。2分間待ってから、結合されていないセルを破棄します。
- 最初の抗原ビーズ選択で説明したように、残りのすべてのステップを実行します。最初のポジティブセレクション(ステップ3.3.2)との唯一の違いは、洗浄ステップの数です:PBSAで1回洗浄する代わりに、ビーズ細胞懸濁液をPBSAで2回洗浄して、選択の厳しさを高めます。
注:通常、ダイバーシティは再び10倍から100倍に減少します。この2回目のビーズ選択後のG4ライブラリの最大多様性は、3.45 x 105 セルでした。上記のように、実際の多様性は、ライブラリのオーバーサンプリングにより低くなると予想されます。
6. フローサイトメトリーソーティングによるライブラリーの選択
- SG-CAAで表面発現を一晩誘導した後、上記のようにOD600 を測定します。10倍の多様性(2000 x g、5分、RT)をカバーするのに十分な細胞をペレット化します。上清を捨てます。
- ペレットをPBSAに再懸濁し、微量遠心チューブに移します。各チューブの染色には、3 x 107 細胞を使用します。チューブは、多様性に応じて必要な数だけ用意してください。抗原なしで染色するためのコントロールチューブを1本用意します。
注:たとえば、30 x 106 セルを選別する必要がある場合は、300 x 106 セルをペレット化して、目に見えるセルペレットを実現し、後続のステップでセルが失われるのを防ぎます。5 mLのPBSAに再懸濁し、500 μLのアリコートをそれぞれ3〜4アリコートを微量遠心チューブに移します。染色プロセス中に細胞の一部が失われるため、選別する数よりも約3倍多くの細胞を準備します。薬物/リガンドの存在下で選択を行う場合は、抗原のみを使用し、薬物/リガンドを含まない追加のコントロールを調製します。 - 再度遠心分離(2000 x g、5分間、RT)し、抗原を含むPBSAの200 μL(hRBP4と5 μM A1120)にペレットを再懸濁し、4°Cで1時間インキュベートします。
注:この時点以降のすべての手順は、氷上および冷たい試薬を使用して実行する必要があります。抗体または抗原の枯渇を防ぐために、すべての試薬は、チューブ内の酵母表示タンパク質の総数と比較して、化学量論的に過剰に添加する必要があります。そうしないと、抗原や抗体の利用可能性が限られているため、酵母に表示された変異体の結合が制限されるリスクがある可能性があります。通常、酵母細胞あたり5 x 104 分子が計算に使用され、少なくとも10倍以上の抗原と抗体をストキオメトリックに添加することをお勧めします。抗原が非常に限られている場合、過剰量を5倍に減らすことができます。リガンドの枯渇を避けるための計算例を含むより詳細な説明は、以前のプロトコル9で提供されています。 - インキュベーション後、細胞(2000 x g、5分、4°C)を遠心分離し、1 mLのPBSAで洗浄します(選択中に薬物/リガンドが存在する場合、この場合はA1120など)、これを洗浄バッファー中に存在する必要があります)。
- 抗体を含む100 μLの冷PBSAに細胞を再懸濁し、結合抗原の表示染色および検出を行い、4°Cで30分間インキュベートします。
- 抗c-myc抗体(最終希釈率1:100)を使用して、表示された全長のPOIを検出します。抗HA抗体(最終希釈率1:50)を使用して、表示されたタンパク質の総量を検出します。Penta-His抗体(最終希釈1:20)またはストレプトアビジン(最終希釈1:200)を使用して、それぞれ6x HISタグまたはビオチンを含む可溶性抗原に結合する酵母細胞を選択します。
- ソーティングラウンド間で検出試薬を交互に使用して、二次試薬に結合するタンパク質バリアントの濃縮を防ぎます。
注:さまざまな蛍光色素の組み合わせを使用できますが、Alexa Fluor 488とAlexa Fluor 647は、フローサイトメトリーでのコンペンセーションを必要としないため、優れた選択肢です。抗体の枯渇を防ぐために、チューブ内の酵母表示タンパク質の総数と比較して、化学量論的に過剰に添加する必要があります。通常、計算は酵母細胞あたり5 x 104 分子に基づいています。より詳細な説明は、前のプロトコル9で提供されている。
- インキュベーション後、細胞(2000 x g、5分、4°C)を遠心分離し、1 mLのPBSA(hRBP4選択G4ライブラリー5 μMのA1120の薬物/リガンドを含む)をペレットに加え、再度遠心分離します。ペレットが乾燥するのを防ぐために、ほとんどの上清を取り除き、20〜30μLだけに保ちます。
- ソーティングの直前にのみペレットを冷たいPBSAに再懸濁し、目的の特性(すなわち、A1120の存在下でhRBP4に結合する)を持つFn3分子を示す酵母細胞をソーティングします。
注: ゲート方式を 図 2A に示します。 - 細胞を直接SD-CAA培地に選別し、選別後にSD-CAA培地をさらに追加し、180rpmで振とうしながら30°Cでインキュベートします。
注:選別後に容量が大幅に増加した場合は、遠心分離ステップ(2000 x g、5分、RT)を実行して余分なシース液を除去し、細胞を新鮮なSD-CAA培地に再懸濁する必要があります。新しいチューブにソーティングされたセルの数を書き留めます。これは、次のソーティングラウンドの新しい最大多様性になります。前述のように、ライブラリの実際の多様性は、ライブラリのオーバーサンプリングにより低くなります。 - フローサイトメトリーによるソートを数回繰り返して、酵母ライブラリー中の結合タンパク質変異体の濃縮を確認します(図2B)。
注:分析フローサイトメトリー実験でライブラリを並べて比較すると便利な場合があります。後の選択ラウンドでは、抗原の濃度を下げることは、低親和性結合剤と高親和性結合剤を区別し、低濃度でターゲットに対して高い結合シグナルを示すタンパク質変異体を特異的に選別するのに有益です(図2C)。その後の選抜ラウンドでは、熱安定性を高めるために選別することも可能です。この目的のために、セクション7に記載されているように、epPCRによるライブラリのランダム突然変異誘発を行った。他のフローサイトメトリーソーティングラウンドと同様に、誘導、OD600 の測定、および細胞の洗浄により細胞を調製します。酵母細胞をPBSAに氷上に10分間置き、続いてサーモサイクラーで10分間インキュベーションし、高温でのヒートショックを行います。その後、それらを再び氷上に10分間置き、染色プロトコルを進めます。安定化された変異体は熱変性に抵抗するため、加熱インキュベーション後も結合が維持されます。熱ショックについては、野生型タンパク質の初期の熱安定性に応じて、さまざまな温度を試験できます。通常、40〜80°Cの範囲の温度が適しています。しかし、48°Cを超えると、酵母細胞の生存率は急激に低下します。したがって、ステップ7.1で説明したように、熱ショックを受けた酵母ライブラリーを選別した後にプラスミドを抽出する必要があります。この場合、EBY100細胞は、次のソーティングラウンド36に進む前に、濃縮された変異体で新たに形質転換する必要があります。熱安定性を高めるためのより詳細なプロトコルは、他の場所で見つけることができる37。表示されたタンパク質の変性が可逆的である場合、この方法は機能しません。あるいは、ソーティングの前日に20°Cではなく37°Cでタンパク質発現を誘導し、効率的な全長表示を示すタンパク質バリアントをソーティングします。
7. ランダム変異を導入するためのepPCRによるアフィニティー成熟
注:epPCRを用いたアフィニティー成熟は、最初のフローサイトメトリーソーティングラウンドの前に、またはフローサイトメトリーソーティングラウンドの間に行うことができます。A1120の存在下でhRBP4を用いたG4ライブラリーを選択するために、フローサイトメトリーソーティングの最初のラウンドの前にアフィニティー成熟を行いました。これは、ビーズ選択後のライブラリサイズと、フローサイトメトリーで検出できる結合シグナルにも依存します。特に、ビーズ選択後の親和性がフローサイトメトリー実験でシグナルを得るのに十分でない場合(抗原が洗浄ステップ中に急速に解離するため)、epPCRは改良されたバリアントを生成し、その後フローサイトメトリーを介して検出および選択することができます。
- 市販のプラスミドminiprep IIキット( 材料表を参照)を使用して、濃縮酵母ライブラリーから酵母miniprep DNA抽出を行い、以下の変更を加えます。
- 一晩培養した細胞密度を測定し、朝にOD600 を0.2に希釈し、振とうしながら30°Cでインキュベートします。
- OD600 が0.6に達したら、細胞を600 x g で2分間遠心分離することにより、3 mLの培養物を回収します。
- ペレットに200μLの溶液1を加えます。6 μLのザイモリアーゼを加えます。フリックまたはマイルドボルテックスによりペレットを再懸濁し、37°Cで3時間インキュベートします。
- 200μLの溶液2を加え、穏やかに混合します。400 μLの溶液3を加え、穏やかに混合します。
- 最高速度で3分間遠心分離します。
- 上清をスピンIカラムに移し、最高速度で30秒間遠心分離し、フロースルーを廃棄します。
- エタノールを含む550μLのWash Bufferで洗浄し、最高速度で2分間遠心分離します。
- カラムを清潔な微量遠心チューブに移し、最高速度で2分間遠心分離して、残留洗浄バッファーを除去します。
- カラムを新しい微量遠心チューブに移し、10 μL のヌクレアーゼフリー水を加え、最高速度で 1 分間遠心して DNA を溶出します。
- 初期PCRを実行して、epPCRのテンプレートを増幅します。
- 酵母表面表示ベクターのインサートの外側にアニーリングしたプライマーを用いてPCRによりDNAを増幅します。
注:酵母ミニプレップ中には、目的のプラスミドとともに多くのゲノム酵母DNAが溶出されるため、このステップでは、epPCRのテンプレートとして使用するクリーンなDNAフラグメントを取得することをお勧めします。 - pCTCON2 には、プライマー fwd 5'- GGCTCTGGTGGAGGAGGCGGTAGCGGAGGCGGAGG を使用します。
GGTCGGCTAGCおよびrev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC を 、表 3 に記載の反応成分と 表 4 に示すサイクリング条件で測定します。 - PCRおよびDNAクリーンアップキットでPCRを精製します。20 μL のヌクレアーゼフリー H2O で溶出します。
- 酵母表面表示ベクターのインサートの外側にアニーリングしたプライマーを用いてPCRによりDNAを増幅します。
- epPCRを実施して、ランダムな変異を導入します。
- ヌクレオチド類似体を用いて、示された成分(表5)および条件(表6)を用いてepPCRを実施します。
- 調製済みの1%アガロースゲル上で、120 Vで45分間、全epPCR産物を泳動します。マーカーを含め、epPCR産物とラダーの間に1レーンを残します。インサートの予想されるサイズに対応する目的のバンドを切り出し、ゲル抽出キットを使用して精製し、DNA濃度を測定します。
- 無作為化されたDNAをPCRで増幅します。
- 50 ngのDNAをQ5ポリメラーゼで増幅します。示された条件(表8)で、各エレクトロポレーション(表7)に対して2 x 100 μLのPCR反応をセットアップします。最初のPCRに使用したものと同じプライマーを使用してください。
注:酵母へのエレクトロポレーションには大量のDNAが必要です。 - 5 μL の PCR 産物を 1% アガロースゲルで 120 V で 45 分間分析します。
- 50 ngのDNAをQ5ポリメラーゼで増幅します。示された条件(表8)で、各エレクトロポレーション(表7)に対して2 x 100 μLのPCR反応をセットアップします。最初のPCRに使用したものと同じプライマーを使用してください。
- 増幅したDNAをエタノール沈殿で精製します。
- プール 2 x 100 μL 一緒に。滅菌ろ過された3 M酢酸ナトリウム(pH 5.2)を20 μL(容量の10%)加えます。
注:このバッファーは事前に準備し、RTで数ヶ月間保存することができます。 - 100%エタノール440μL(または容量×2以上)を加え、室温で2分間インキュベートします。
- 室温で20,000 x g で5分間遠心分離し、上清を取り除き、500 μLの70%エタノールを加えて短時間混合します。
- 20,000 x g でRTで5分間遠心分離し、上清を取り除き、100%エタノール500μLを加えて短時間混合します。
- 室温で20,000 x g で5分間遠心分離し、上清を取り除き、DNAペレットを層流フードの下ですべてのエタノールが蒸発するまで乾燥させます。
注:このプロセスを高速化するには、48°Cのヒートブロックを使用します。DNAペレットは常に見えるとは限りません。 - 乾燥させたDNAペレットを10μLのヌクレアーゼフリーH2Oに溶解します。
注:DNAはできるだけ少量で溶解する必要があります。DNA濃度を測定しないでください。
- プール 2 x 100 μL 一緒に。滅菌ろ過された3 M酢酸ナトリウム(pH 5.2)を20 μL(容量の10%)加えます。
| 容量 [μL] | 最終濃度 | |
| 5x Q5エンハンサー | 10 | 1倍 |
| 5x Q5バッファ | 10 | 1倍 |
| プライマー fwd 10 μM | 2.5 | 0.5 μM |
| プライマー rev 10 μM | 2.5 | 0.5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Q5ポリメラーゼ | 0.5 | 20 U/mL |
| 酵母ミニプレップ由来DNA | 10 | |
| ヌクレアーゼフリーH2O | 13.5 |
表3:単離された酵母ミニプレップからのPOI遺伝子の増幅のための1st ステップPCRの条件。
| 歩 | 温度 | 時間 |
| 初期変性 | 98°C | 30秒 |
| 25サイクル | 98°C | 10秒 |
| 72°C | 30秒 | |
| 72°C | 30秒 | |
| 最終的な延長 | 72°C | 2分間 |
| 持つ | 4 °C |
表4:単離された酵母ミニプレップからのPOI遺伝子の増幅のための1st ステップPCRのサイクリング条件。
| 容量 [μL] | 最終濃度 | |
| ヌクレアーゼフリーH2O | 50まで | |
| 10x Thermopolバッファー | 5 | 1倍 |
| Primer_fwd (10 μM) | 2.5 | 0.5 μM |
| Primer_rev (10 μM) | 2.5 | 0.5 μM |
| dNTP(10 mM) | 1 | 200 μM |
| 8-oxo-dGTP (100 μM) | 1 | 2 μM |
| dPTP(100μM) | 1 | 2 μM |
| 1st PCRからのPCR産物 | XXさん | 50 ng |
| Taq DNAポリメラーゼ | 0.5 | 0.05 U/μL |
表5:第1ステップPCR でPOI DNAを増幅した後に実行されるepPCRの条件。
| 歩 | 温度 | 時間 |
| 初期変性 | 94°C | 30秒 |
| 15サイクル | 94°C | 45秒 |
| 60°C | 30秒 | |
| 72°C | 1分間 | |
| 最終的な延長 | 72°C | 10分間 |
| 持つ | 4 °C |
表6:epPCRのサイクリング条件。
| 容量 [μL] | 最終濃度 | |
| 5x Q5エンハンサー | 20 | 1倍 |
| 5x Q5バッファ | 20 | 1倍 |
| プライマー fwd 10 μM | 5 | 0.5 μM |
| プライマー rev 10 μM | 5 | 0.5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Q5ポリメラーゼ | 1 | 20 U/mL |
| 50 ng のDNA | XXさん | |
| ddH20 | 100まで |
表7:EBY100細胞のエレクトロポレーション前のepPCR産物の増幅のための2nd ステップPCRの条件。
| 歩 | 温度 | 時間 |
| 初期変性 | 98°C | 30秒 |
| 25サイクル | 98°C | 10秒 |
| 72°C | 30秒 | |
| 72°C | 30秒 | |
| 最終的な延長 | 72°C | 2分間 |
| 持つ | 4 °C |
表8:epPCR産物の増幅のための2nd ステップPCRのサイクリング条件。
8. エレクトロポレーションのための酵母ディスプレイベクトルの線形化
- pCTCON2酵母表面表示ベクター (Addgene plasmid #41843) を線状化します。
- 大規模なダイジェストを行い、このダイジェストされた直鎖状化ベクターをその後のすべてのエレクトロポレーションに使用します。
- 2 x 200 μg の pCTCON2 プラスミド DNA を Sal I と共に 37 °C で 24 時間消化し、初期直進化を行います (表 9)。
- NheIおよびBamHIで37°Cで24時間消化し、インサートを切除します(表10)。
- 最後に、各酵素(Sal I、NheI、BamHI)5 μLをチューブに加え、再度37°Cで24時間インキュベートします。
- 調製したベクターを調製アガロースゲルで実行します。バンドを切除し、ゲル抽出キットで精製します。ベクターを20 μLで溶出し、DNA濃度を測定して約2 μg/μLに調整します。
注:プラスミドを長時間消化すると、インサートの十分な切断が確保され、エレクトロポレーション後のベクターからのバックグラウンドが少なくなります。
| デオキシリボ核酸 | 200μgの |
| 10x CutSmartBuffer | 50 μL |
| サルI-HF(NEB) | 30 μL (60 U) |
| H2O | 500μLまで |
表9:酵母表面表示ベクターpCTCON2の大規模消化の最初のステップの条件。
| pCTCON2 (サル I 消化) | 500μL |
| 10x CutSmartBuffer | 37.5μL |
| NheI-HF (NEB) | 15 μL (30 U) |
| BamHI-HF (NEB) | 15 μL (30 U) |
| H2O | 875μLまで |
表10:酵母表面表示ベクターpCTCON2の大規模消化の第2ステップの条件。
9. ランダム化DNAと直鎖状化ベクターを用いたEBY100のエレクトロポレーション
- EBY100酵母細胞(ATCC、 材料表を参照)を、エレクトロポレーションの3日前にグリセロールストックから打って調製します。形質転換されていないEBY100細胞には酵母表面表示ベクターが含まれていないため、フル培地のYPDプレートを使用してください(表1)。30°Cでインキュベートします。
- エレクトロポレーションの1日前に、1つのEBY100コロニーを新たに調製したYPD培地30mLに接種します(表1)。180 rpmで振とうしながら、30°Cで一晩インキュベートします。
注:YPD培地は1か月以上経過してはなりません。 - 翌朝、EBY100を一晩培養し、100 mLのYPD培地中でOD 600を0.2のOD600 に希釈し、180 rpmで振とうしながら30°Cでインキュベートします。
注:2回のエレクトロポレーションには50mLの培養量で十分です。通常、100 mLは、コントロール(細胞のみおよび細胞とベクター)に十分な細胞を持つように調製されます。 - OD600 が1.3-1.5に達したら、細胞を50 mLチューブに2000 x g で3分間ペレット化し、上清を除去します。
- ペレットを25 mLの100 mM滅菌ろ過酢酸リチウムに再懸濁します( 材料の表を参照)。調製したばかりの滅菌ろ過済みDTT(1 M)( 材料表を参照)を最終濃度10 mMまで加えます。
注:酢酸リチウムの体積は、YPDの元の体積の半分である必要があります。必要に応じてスケールダウンまたはスケールアップします。酢酸リチウム(100 mM)は事前に調製してRTで数ヶ月間保存することができますが、1 M DTTストックは使用直前に調製し、氷上に保管する必要があります。 - 細胞を30°Cで10分間振とうしながらインキュベートします。
- 2000 x g で3分間遠心分離し、上清を捨てて細胞を氷上に置きます。
注:以下のすべての手順は、氷の上で、冷却された試薬、キュベット、および微量遠心チューブを使用して実行する必要があります。 - ペレットを25 mLの冷冷滅菌H2Oに再懸濁し、2000 x g で3分間遠心分離します。
注: 使用する H2O ボリュームは、元の YPD ボリュームの半分にする必要があります。 - 350 μL の滅菌 H2O に再懸濁します。
注:再懸濁後の総容量は500 μLです。必要に応じてH2Oをさらに追加します。 - DNAライブラリーを並行して調製する:エタノール精製インサートを含む微量遠心チューブを氷上に置き、4 μgの直鎖状pCTCON2ベクターを添加します。
- DNAに250μLの細胞を加え、切り取って穏やかに混合します。あらかじめ冷やした2mmエレクトロポレーションキュベットに移し、エレクトロポレーションまで氷上に保ちます。
注:コントロール(DNAを含まない細胞のみ、および挿入なしの線形化ベクターのみを持つ細胞)を追加して、エレクトロポレーションの成功を判断します。 - 2 mmエレクトロポレーションキュベット( 材料表を参照)を使用し、矩形波プロトコル、シングルパルス、500 V、パルス幅15 msの条件でエレクトロポレーションします。
注:典型的なドループは3%〜9%です。他のエレクトロポレーターも使用できますが、示されている条件は、この研究で使用した機器に最適化されています。 - 予熱したYPD1 mLで細胞をレスキューし、予め加温した微量遠心チューブに移し、30°Cで1時間振とうせずにインキュベートします。
注意: エレポレーターの隣に加熱ブロックを配置して、回復チューブが適切な温度に保たれるようにします。 - SD-CAAプレート(表1を参照)に10-3、10-4、10-5、および10-6希釈液の細胞を100μLプレートし(表1を参照)、30°Cで形質転換体を増殖させます。
注:理論上のライブラリーの多様性は、形質転換体の数を元の培養量(ここでは1 mL)に計算することによって得られます。典型的には、この形質転換プロトコルは、約107〜108 形質転換細胞を生じる。 - 残りの細胞を2000 x g で3分間ペレット化し、200 mL SD-CAAに再懸濁し、180 rpmで一晩振とうしながら30°Cでインキュベートします。epPCRランダム化ライブラリーを含む形質転換細胞は、SD-CAAで継代して翌日SG-CAAで表面発現するように誘導するか、さらに使用するまで4°Cで保存することができます。多様性を決定した後、epPCR後にライブラリのグリセロールストックを凍結します。翌日には、細胞の一部は、プラスミドを取得せずに形質転換手順で持ち越された未形質転換細胞であるため、この影響を補うために、最初の継代ステップでライブラリの多様性の少なくとも20倍を継代することが推奨されます。
10. 数回の選択ラウンド後の酵母ライブラリーのシーケンシング
- 上記のように酵母ミニプレップを行うことにより、酵母DNAを抽出します。
- 市販の 大腸菌 10-βエレクトロコンピテントセル( 材料表を参照)を単離されたDNAでエレクトロポレートします。
- エレクトロポレーションキュベット(1 mm、 材料表を参照)と微量遠心チューブを氷の上に置きます。エレクトロポレーション後の細胞を回収するために、950 μLの伸長培地を37°Cで予温します。
- 氷の上で細胞を解凍し、チューブをやさしくフリックして混合します。50 μLの細胞を冷却した微量遠心チューブに移します。酵母miniprep DNAを1 μL加えます。
- 細胞-DNAミックスを冷却したエレクトロポレーションキュベットに慎重に移します。
注意: 気泡を避け、細胞がキュベットの底に沈着することを確認してください。 - 以下の条件を用いてエレクトロポレーション装置を用いてエレクトロポレーションする:2.0kV;200 Ω;25μF。
注:一般的な時定数は4.8〜5.1ミリ秒です。他のエレクトロポレーターも使用できますが、示されている条件は、この研究で使用した機器に最適化されています。 - 直ちに950 μLの37 °C温めた伸長培地( 材料表を参照)をキュベットに加え、穏やかに混合し、温めた微量遠心チューブに戻します。
注意: エレポレーターの隣に加熱ブロックを配置して、回復チューブが適切な温度に保たれるようにします。 - 37°C、300rpmで40分間インキュベートします。10 μL、100 μL、および残りの細胞(遠心分離後)を、予め加温した選択的LBプレートにプレートします( 表1を参照)。プレートを37°Cで一晩インキュベートします。
注:pCTCON2ベクターの場合、抗生物質の選択にはアンピシリンが使用されます。 - LB寒天培地と必要な抗生物質(ここではアンピシリンを使用)を含むコピープレートを準備します:96ウェルプレートの各ウェルに200μLのLB寒天と100μg/mLのアンピシリンをピペットで入れ、固化させます。
- ウェルあたり50 μLのLBと100 μg/mLのアンピシリンを含む96ウェルシーケンシングプレートを調製します。
- プレートから1つのコロニーを選択し、最初にコピープレートとシーケンシングプレートの同じウェルに接種します。
注:コピープレートは、液体培養物に接種し、ミニプレップを行うことで、目的のプラスミドを容易に取得できることを保証します。 - コピープレートを37°Cで一晩インキュベートし、4°Cで保存します。
- シーケンシングプレートを37°Cで3〜4時間インキュベートし、プラスチックまたはアルミホイルで覆ったシェーク(180rpm)します。
- シーケンシングプレートをシールし、500 μL の 10 μM pCTCON2 シーケンシングプライマー (5'-CGTTTGTCAGTAATTGCGGTTCTCTC) と一緒にシーケンシングに送ります。
- 配列の解析には、得られたDNA配列を、EMBOSS Transeq38などの適切なプログラムを用いてアミノ酸配列に翻訳します。次に、Multiple Sequence Alignment(MUSCLE)39 または同様のアライメントツールを使用して、バリアント配列を相互に、および親の野生型タンパク質にアラインメントする。
注:異なる選択ラウンドのライブラリのシーケンシングを行うことにより(例えば、以前の選択ラウンドの後、および高親和性バリアントを濃縮するための抗原濃度の低い追加の選択ラウンドの後)、異なるバリアントを同定することが可能です。これは、ソーティングラウンド中に適用されるゲートの厳しさ、したがって図書館の多様性に大きく依存します。
結果
G4ライブラリーは、低分子医薬品A1120に結合した抗原hRBP4に対して選択されました。フローサイトメトリーソーティングのためのライブラリーの染色は、方法6に記載したように行い、適用されたゲーティング戦略を 図2Aに示します。最初のゲートには、細胞形態に基づくすべての細胞が含まれ、2番目のゲート(FSC-Widthのヒストグラム)は、単一細胞を選択し、細胞凝集体を除去するために適用された厳格なゲーティング戦略を示しました。3番目と最後のゲートは、タンパク質変異体(x軸)と抗原結合(y軸)の表示を示しました。ディスプレイシグナルと結合シグナルの両方を示す酵母細胞を選別しました。重要なことに、ソーティングゲートは、高い結合シグナル、したがって高い親和性を持つ結合ドメインを濃縮するために、厳密な方法で設定されました。この厳格な選択により、選択キャンペーン全体を通じて標的抗原に特異的に結合する酵母細胞を表示することの濃縮がもたらされました(図2B)。その後のフローサイトメトリーソーティングラウンドでは、抗原濃度が10倍(100 nMから10 nM)に減少しました。したがって、全体的な結合シグナルは減少し、親和性の高いバインダーのみが依然として検出可能で選別されました(図2C)。
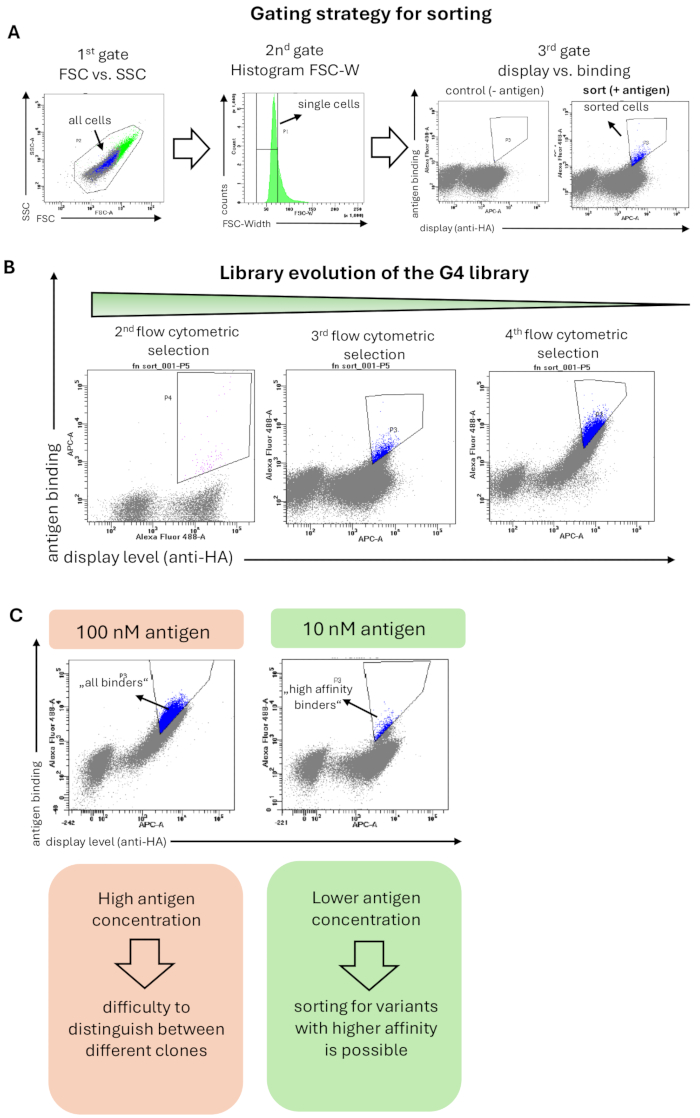
図2:抗原(A1120の存在下でのhRBP4)に結合するためのFn3ベースのG4ライブラリーの酵母表面表示選択からの代表的な結果。 (A)酵母ライブラリーのソーティングのための一般的なゲーティング戦略。最初のゲート(FSCとSSC)は、すべての酵母細胞を選択し、散乱イベントを除外することです。2番目のゲート(FSC-Wのヒストグラム)は、細胞凝集体を除去し、単一の酵母細胞のみを選択することを目的としています。3 番目のゲートは、表面の表示レベル(HA タグまたは c-myc-タグの検出)と抗原への結合(ここでは 5 μM A1120 存在下での hRBP4、抗 His 抗体によって検出)をプロットします。ライブラリーは、抗原結合が期待されない二次抗体のみ(抗原なし)で追加的に染色しました。ソートされたセルは青色で強調表示されます。(B)3ラウンドのフローサイトメトリーソーティングにおけるG4ライブラリーの進化。結合集団の濃縮は、各選択ラウンドで観察できます。(C)より低い抗原濃度の使用により、標的抗原に対する親和性が高いタンパク質変異体の選択が可能になる。抗原濃度(ここではhRBP4)を10倍に減少させると、異なる対角線が現れ、親和性が高い(選別された細胞、青)またはより低い親和性を持つクローンが存在することを示します。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
酵母表面ディスプレイは、タンパク質工学で使用される主要な方法の1つとして進化してきました。これは、親和性1,18,40,41、発現/安定性24,27,42,43および活性28,44の工学に一般的に使用されるが、さらに、エピトープマッピング45,46や酵母細胞9の表面上の個々の変異体の特性評価のような使用も行われるも可能です。このプロトコールでは、マグネティックビーズによる選択やフローサイトメトリーソーティングによる選択、アフィニティー成熟のためのepPCRによる酵母ライブラリーの多様化など、酵母表面ディスプレイ選択キャンペーンを開始するための基本的なステップを提供します。
従来の酵母表面ディスプレイの選択に不可欠な要件の1つは、十分な品質の可溶性タンパク質の利用可能性です。高純度で明確なオリゴマー化状態を持つ(つまり、モノマータンパク質はモノマーとしてのみ存在すべき)十分に折り畳まれた標的タンパク質から始めると、高い親和性で標的抗原に結合するタンパク質バリアントを選択するための成功率が最も高いことができます。発現が困難な標的タンパク質の代替手段は、細胞ベースの選択であり、これはこの制限を回避するための合理的な戦略を提示する47。しかし、酵母表面ディスプレイには、手間と時間のかかるクローニング、可溶性フォーマットでの発現、タンパク質精製を行うことなく、得られたタンパク質変異体を酵母の表面で直接特性評価できるなど、多くの利点があります。変異体の親和性および安定性は、いずれも酵母表面上で直接分析することができる9。
このプロトコルでは、タンパク質変異体のG4ライブラリー、より具体的にはヒトフィブロネクチンの10番目のIII型ドメインが、低分子A1120の存在下で抗原hRBP4に結合するためにどのように選択されたかを示します。ビーズ選択とフローサイトメトリーソーティングの組み合わせにより、バリアントが濃縮され、選択ラウンド全体で標的抗原への結合が増加したことが示されました(図2B)。低濃度の抗原を使用することで、高親和性タンパク質バリアントの選択が可能になることを示しました(図2C)。典型的には、酵母ディスプレイ選択で達成され得る親和性は、ナノモルまたはピコモルの範囲18にある。最終的なアフィニティーは、標的抗原、選択ラウンド数とアフィニティー成熟度、使用する結合足場、および適用されるゲーティング戦略によって異なります。個々のタンパク質変異体の特性評価は、このプロトコルではカバーされていませんが、以前の研究9で詳細に説明されています。酵母ディスプレイはもともとscFvs 1,40などの抗体断片のエンジニアリングに用いられたが、この方法は非抗体ベースのタンパク質にも広く使用されている10。
要約すると、酵母表面ディスプレイは、ほぼすべての標的タンパク質への結合や安定性の向上など、新規または改良された特性を持つタンパク質バリアントの生成を可能にする強力なタンパク質エンジニアリングツールです。
開示事項
M.W.T.はMiltenyi Biotecから資金提供を受けています。すべての著者は、酵母表面ディスプレイを使用して開発された技術および改変タンパク質の特許出願の発明者です。
謝辞
この研究は、オーストリア科学基金(FWF Project W1224 - Doctoral Program on Biomolecular Technology of Proteins - BioToP and FWF Project ESP 465-B)、オーストリア連邦デジタル経済省、オーストリア国立研究技術開発財団、クリスチャン・ドップラー研究協会(Christian Doppler Laboratory for Next Generation CAR T Cells)の支援を受けて行われました。 St. Anna Children's Cancer Research Institute(オーストリア、ウィーン)への個人寄付による。E.S.は、St. Anna Children's Cancer Research Instituteのオーストリア科学アカデミーのDOCフェローシップを受賞しています。フィギュアは BioRender.com で作成しました。
資料
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
参考文献
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved