Method Article
Protein-Engineering durch Hefe-Oberflächendisplay
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die wesentlichen Schritte für die Durchführung von Auswahlkampagnen für Hefeoberflächendisplays zur Anreicherung von Proteinvarianten, die an ein Antigen von Interesse binden.
Zusammenfassung
Protein-Engineering ermöglicht die Verbesserung bestehender Funktionen eines gegebenen Proteins oder die Generierung neuer Funktionen. Eines der am weitesten verbreiteten und vielseitigsten Werkzeuge im Bereich des Protein-Engineerings ist die Hefeoberflächenanzeige, bei der ein Pool randomisierter Proteine auf der Oberfläche der Hefe exprimiert wird. Die Verknüpfung von Phänotyp (z. B. Bindung des in der Hefe gezeigten Proteins an das interessierende Antigen) und Genotyp (das Plasmid, das für die Proteinvariante kodiert) ermöglicht die Auswahl dieser Bibliothek für die gewünschten Eigenschaften und die anschließende Sequenzierung angereicherter Varianten. Durch die Kombination von magnetischer Bead-Selektion mit durchflusszytometrischer Sortierung können Proteinvarianten mit verstärkter Bindung an ein Zielantigen selektiert und angereichert werden. Bemerkenswert ist, dass neben der Affinitätsreifung auch die Bindung an ein Ziel ohne anfängliche Bindungsaffinität erreicht werden kann. Hier stellen wir ein Schritt-für-Schritt-Protokoll zur Verfügung, das alle wesentlichen Teile einer Auswahlkampagne für Hefeoberflächendisplays abdeckt und Beispiele für typische Ergebnisse der Hefeoberflächenpräsentation enthält. Wir zeigen, dass die Hefeoberflächenanzeige eine breit anwendbare und robuste Methode ist, die in jedem molekularbiologischen Labor mit Zugang zur Durchflusszytometrie etabliert werden kann.
Einleitung
Das Display der Hefeoberfläche ist eine der Schlüsseltechnologien im Bereich des Protein-Engineerings. Es ermöglicht die Auswahl von Proteinvarianten mit gewünschten Eigenschaften wie verbesserter Affinität oder Stabilität. Es wurde erstmals 19971 eingeführt und ist neben dem Phagen-Display 2,3, dem Ribosomen-Display4 und dem Säugetierzell-Display 5,6,7 eine der am häufigsten verwendeten Display-Technologien. Das Protein of Interest (POI) wird auf der Oberfläche von Hefezellen dargestellt, indem es mit Verankerungsproteinen fusioniert wird. Es steht eine Reihe verschiedener Ankerproteine zur Verfügung, und am häufigsten wird der POI mit dem C-Terminus des Hefe-Agglutinin-Paarungsproteins Aga2p 1,8 fusioniert. Darüber hinaus wird der POI in der Regel von zwei Tags flankiert, wie z. B. einem Hämagglutinin-Tag (HA-Tag) und einem c-myc-Tag, was die Detektion des Anzeigepegels unter Verwendung von fluoreszenzmarkierten Antikörpern und Durchflusszytometrie ermöglicht (Abbildung 1A). Typische Hefeauswahlkampagnen beinhalten eine Kombination aus magnetischer Bead-Selektion und durchflusszytometrischer Sortierung. Die Bead-Selektionen ermöglichen den Umgang mit großen Zellzahlen und die Anreicherung von Proteinvarianten, die an das Zielantigen binden, auch bei geringen Affinitäten, da multivalente Wechselwirkungen mit den antigenbeladenen Beads zu Aviditätseffekten führen und somit den Verlust von Varianten mit niedriger Affinität verhindern (Abbildung 1B). Die durchflusszytometrische Analyse und Selektion bietet den Vorteil, dass die Bindung der angezeigten POI-Varianten an das markierte Antigen sichtbar gemacht werden kann. Folglich können die Bindungspopulationen sortiert und kultiviert werden, was zur Anreicherung von Proteinvarianten mit gewünschten Eigenschaften über mehrere Sortierrunden hinweg führt. Darüber hinaus können zusätzliche Runden der zufälligen Mutagenese durchgeführt werden, um die Diversität und damit die Wahrscheinlichkeit, zusätzliche Mutationen zu finden, die zur Affinität und/oder Stabilität des Proteins beitragen, weiter zu erhöhen.
Die Darstellung der Hefeoberfläche bietet bestimmte Vorteile, wie z.B. (a) eukaryotische Expressionsmaschinerie, die sowohl oxidative Proteinfaltung als auch eukaryotische posttranslationale Modifikationen (wie N-Glykosylierung) ermöglicht, (b) Expressionsnormalisierung aufgrund der Detektion der beiden Peptid-Tags, die das Protein flankieren, (c) visuelle Inspektion des Selektionsfortschritts durch Durchflusszytometrie (z. B. Prozentsatz der Bindungszellen und Bindungsintensität) und (d) die Möglichkeit, einzelne Proteinmutanten auf Hefe (z. B. Analyse der Thermostabilität sowie der Affinität), die eine zeitsparende Alternative zur aufwendigen Proteinexpression und -reinigung darstellt9. Tatsächlich haben sowohl die Affinitäten (KD-Werte) als auch die Stabilitäten (T50-Werte) von Proteinen, die auf der Hefeoberfläche angezeigt werden, gute Korrelationen mit Daten gezeigt, die mit biophysikalischen Methoden und löslichen Proteinen gewonnen wurden 9,10,11,12. Das Hefeoberflächendisplay wurde für das Engineering einer Vielzahl von Proteinen eingesetzt, z. B. für die Antikörperfragmente 13,14,15,16, die 10. Typ-III-Fibronektindomäne 17,18, rcSso7d19,20 oder Knottine21. In ähnlicher Weise wurden umfangreiche Forschungen durchgeführt, um das Design von Hefebibliotheken zu optimieren, indem die randomisierten Positionen sowie die Verwendung von Aminosäure-Codons geändert wurden 17,22,23. Die Anzeige der Hefeoberfläche hat sich als erfolgreich für die Entwicklung der Stabilität 14,15,24,25, der Affinität 18,26,27, der enzymatischen Aktivität 28,29,30,31 und der Proteinexpression32 erwiesen. Darüber hinaus wurden anspruchsvollere Anwendungen, wie z. B. die bedingte Bindung in Gegenwart oder Abwesenheit eines kleinen Moleküls, unter Verwendung der Hefeoberflächenanzeige20 durchgeführt.
In diesem Protokoll beschreiben wir alle wesentlichen Schritte für eine Selektionskampagne mit Hefeoberflächendarstellung am Beispiel der G4-Bibliothek (basierend auf der 10. Typ III Fibronektindomäne, Fn3), die gegen das Antigen humanes Retinol-bindendes Protein 4 (hRBP4) in Gegenwart des kleinen Moleküls A112020 selektiert wurde. Diese Selektion wurde durchgeführt, um eine Protein-Protein-Interaktion zu erhalten, die von einem kleinen Molekül abhängt, das als molekularer Schalter verwendet werden kann. Bemerkenswert ist, dass zwar alternative Ansätze mit der Darstellung der Hefeoberfläche möglich sind, typische Hefeselektionen jedoch in der Regel auf die Bindung an ein Zielantigen ohne vorherige Bindungsaffinität abzielen. Wir decken alle Schritte einer Hefeauswahlkampagne ab, die die Kultivierung einer Hefebibliothek, die Kügelchenauswahl, die durchflusszytometrische Sortierung und die Affinitätsreifung durch fehleranfällige PCR (epPCR) umfasst. Daher ergänzt dieses Protokoll frühere Protokolle zur Anzeige von Hefeoberflächen33, 34 und kann als Grundlage für die Auswahl der Hefeoberflächenanzeige (Abbildung 1) mit einer beliebigen Hefebibliothek und einem Zielantigen der Wahl verwendet werden.
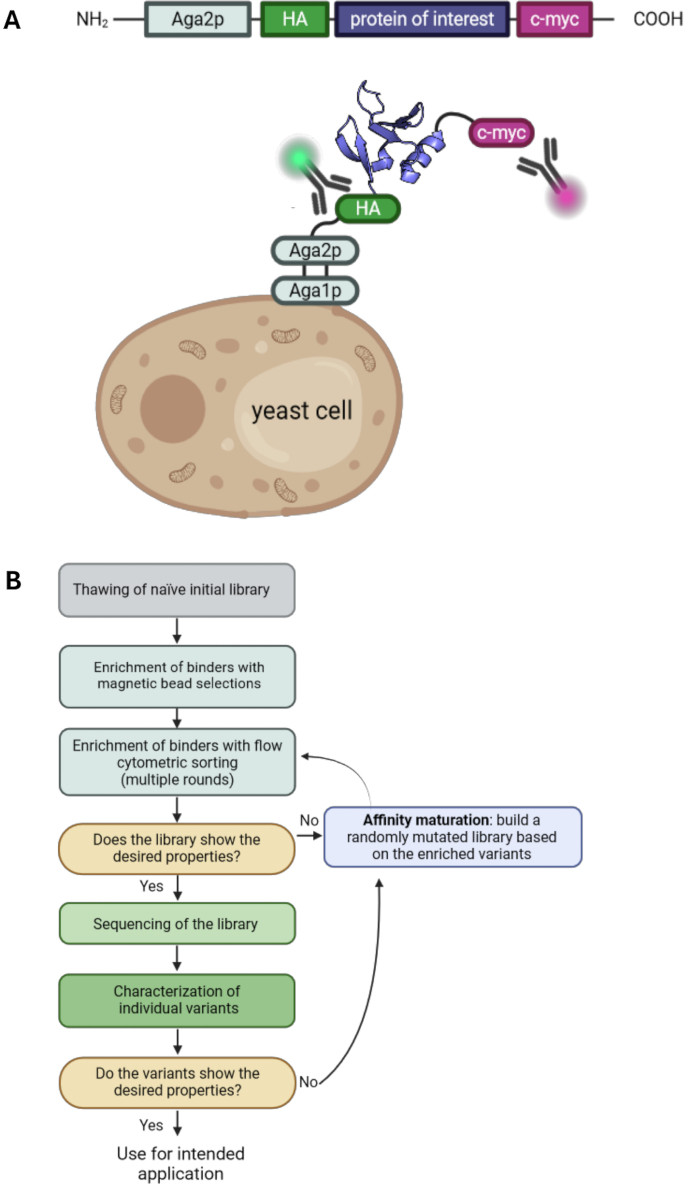
Abbildung 1: Prinzip der Hefeoberflächenanzeige und ein typischer Arbeitsablauf für die Auswahl der Hefeoberflächenanzeige. (A) Der POI wird in einen Hefeoberflächen-Anzeigevektor kloniert und typischerweise von einem N-terminalen HA- und einem C-terminalen c-myc-Tag flankiert. Das Konstrukt ist mit dem Hefe-Paarungsprotein Aga2p fusioniert, um es auf der Oberfläche anzuzeigen. Bei dem abgebildeten Protein handelt es sich um das technisch hergestellte Binder "RS3" von PDB ID: 6QBA20. (B) Flussdiagramm, das einen typischen Arbeitsablauf für Auswahlkampagnen für Hefeoberflächendisplays veranschaulicht, die die Anreicherung von Proteinvarianten mit den gewünschten Eigenschaften durch Kügelchenauswahl und durchflusszytometrische Sortierung sowie epPCR für die Affinitätsreifung kombinieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Auftauen und Kultivieren von Hefebibliotheken
- Ausreichende Aliquots der gefrorenen Hefebibliothek bei RT auftauen und sofort in SD-CAA (siehe Tabelle 1) auf einen OD600 von etwa 1 verdünnen (dies entspricht 1 x 107 Zellen/ml).
HINWEIS: Stellen Sie sicher, dass Sie die Vielfalt der Bibliothek mindestens 10x abdecken. Wenn also die Bibliotheksgröße 2,5 x 108 Klone ausmacht (dies ist die Vielfalt der in diesem Beispiel verwendeten G4-Bibliothek), inokulieren Sie mindestens 2,5 x 109 Zellen in 250 ml SD-CAA, um einen OD600 von 1 zu erreichen. Dieses Protokoll kann auch von Flüssigkulturen (Hefezellen, die mit einer Bibliothek frisch transformiert werden) gestartet werden. - Beurteilen Sie die Anzahl lebensfähiger Zellen.
- Nehmen Sie ein 100 μl Aliquot aus dieser Hefesuspension (bei OD600 von 1) und führen Sie eine 1:10-Verdünnungsreihe mit 900 μl SD-CAA-Medium in Mikrozentrifugenröhrchen durch.
- Bereiten Sie Verdünnungen bis zu 10-5 vor und plattieren Sie 100 μl der Verdünnungen 10-3, 10-4 und 10-5 auf SD-CAA-Platten (Tabelle 1). Inkubieren Sie die Platten bei 30 °C für 2-3 Tage. Stellen Sie sicher, dass die Anzahl lebensfähiger Zellen nach dem Auftauen die Bibliotheksvielfalt um mindestens das 10-fache übersteigt.
- In der Zwischenzeit wird mit der restlichen Zellsuspension fortgefahren und die Hefekultur mit der G4-Bibliothek in Glaskolben über Nacht bei 30 °C unter Schütteln (180 U/min) kultiviert. Nach der Inkubation über Nacht fahren Sie mit Abschnitt 2 (Induktion der Proteinexpression auf der Hefeoberfläche) fort.
- Nach 2-3 Tagen Inkubation die Völker auf den SD-CAA-Platten durch Sichtprüfung zählen. Bei der Berechnung der endgültigen Anzahl lebensfähiger Zellen nach dem Auftauen ist der Verdünnungsfaktor und das Verhältnis der plattierten (d. h. 100 μl) zum ursprünglichen Kulturvolumen (d. h. 250 ml) zu berücksichtigen.
Lebensfähige Zellen/ml = Anzahl der Kolonien auf der Platte × Verdünnungsfaktor
Lebensfähige Zellen (insgesamt) = (lebensfähige Zellen/ml) × Kulturvolumen
| Medium/Puffer | Bestandteil | Konzentration [g/L] | Kommentare/Beschreibung | |||
| SD-CAA | D-Glukose | 20 | Alle Medienkomponenten werden in 1000 mL ddH2O und sterilem Filtrat mit 0,22 μm Einweg-Sterilfiltern gelöst. | |||
| Hefestickstoffbasis | 6.7 | |||||
| Casminosäuren | 5 | |||||
| Zitronensäure-Monohydrat | 7.4 | |||||
| Trinatriumcitrat-Dihydrat | 10.83 | |||||
| SG-CAA | D-Galaktose | 20 | Alle Medienkomponenten werden in 1000 mL ddH2O und sterilem Filtrat mit 0,22 μm Einweg-Sterilfiltern gelöst. | |||
| D-Glukose | 2 | |||||
| Hefestickstoffbasis | 6.7 | |||||
| Casaminosäuren | 5 | |||||
| Dinatriumhydrogenphosphat-Heptahydrat | 10.2 | |||||
| Natriumdihydrogenphosphat-Monohydrat | 8.56 | |||||
| SD-CAA-Platten | Sorbit | 182 | Sorbit, Dinatriumhydrogenphosphat-Heptahydrat, Natriumdihydrogenphosphat-Monohydrat und Agar-Agar in 900 mL ddH2O auflösen und autoklavieren. Die restlichen Bestandteile werden in 100 mL ddH2O aufgelöst, sterilfiltriert und bei lauwarmem autoklaviertem Medium zugegeben. | |||
| Dinatriumhydrogenphosphat-Heptahydrat | 10.2 | |||||
| Natriumdihydrogenphosphat-Monohydrat | 7.44 | |||||
| Agar-Agar | 15 | |||||
| D-Glukose | 20 | |||||
| Hefestickstoffbasis | 6.7 | |||||
| Casaminosäuren | 5 | |||||
| YPD | Pepton | 20 | Bereiten Sie eine 10-fache D-Glukose-Brühe (200 g/L) und ein steriles Filtrat mit 0,22 μm Einweg-Sterilfiltern vor. Pepton und Hefeextrakt in 900 mL ddH2O auflösen und autoklavieren. Wenn es lauwarm ist, fügen Sie 100 mL 10x D-Glukose hinzu. | |||
| Hefeextrakt | 10 | |||||
| D-Glukose | 20 | |||||
| YPD-Platten | Pepton | 20 | Bereiten Sie eine 10-fache D-Glukose-Brühe (200 g/L) und ein steriles Filtrat mit 0,22 μm Einweg-Sterilfiltern vor. Pepton, Hefeextrakt und Agar-Agar in 900 mL ddH2O auflösen und autoklavieren. Wenn es lauwarm ist, fügen Sie 100 mL 10x D-Glukose hinzu. | |||
| Hefeextrakt | 10 | |||||
| D-Glukose | 20 | |||||
| Agar-Agar | 15 | |||||
| PBSA | BSA | 1 | BSA in PBS und sterilem Filtrat mit 0,22 μm Einweg-Sterilfiltern auflösen. | |||
Tabelle 1: Zusammensetzung von Medien und Puffern.
2. Induktion der Proteinexpression auf der Hefeoberfläche
- Nach der Inkubation über Nacht erreichen die Hefekulturen typischerweise einen OD600 zwischen 2 und 20. Messen Sie den OD600 mit einem Photometer, um die Dichte der Hefekultur zu bestimmen.
HINWEIS: Außendurchmessermessungen sind nur im Bereich von 0,1-1 linear. Führen Sie daher Verdünnungen (typischerweise 1:20 und 1:50) in SD-CAA durch, um den OD600 zu bestimmen.- Verdünnen Sie die Zellen in SD-CAA auf einen OD600 von 1 und stellen Sie sicher, dass die Diversität 10x abgedeckt ist (d.h. 2,5 x 109 Zellen werden für die Verdünnung der G4-Bibliothek verwendet). Die Inkubation bei 30 °C unter Schütteln fortsetzen, damit die Zellen eine exponentielle Wachstumsphase erreichen können.
- Bereiten Sie Glycerin-Brühen (die die 50-fache Diversität abdecken) von Hefebibliotheken (in SD-CAA mit 15 % Glycerin) aus der Übernachtkultur vor. Zu diesem Zweck zentrifugieren Sie die erforderliche Anzahl von Hefezellen (2000 x g, 3 min, 4 °C), verwerfen den Überstand und resuspendieren das Zellpellet in 1 mL SD-CAA-Medium, das mit 15 % Glycerin versetzt ist. Glycerinvorräte sofort bei -80 °C einfrieren.
HINWEIS: Die Lebensfähigkeit von Hefezellen wird erhöht, wenn Hefekulturen vor dem Einfrieren in eine stationäre Phase gezüchtet werden. In späteren Auswahlrunden, wenn die Diversität der Bibliotheken geringer ist, reicht es in der Regel aus, 700 μl der Hefekultur zu verwenden und sie mit 300 μl SD-CAA, ergänzt mit 50 % Glycerin, zu mischen. Hier zeigen Hefekulturen typischerweise OD600-Werte von 15 in der stationären Phase, aber der tatsächliche Wert kann vom verwendeten Photometer abhängen. Somit kann die stationäre Phase durch wiederholte OD600-Messungen bestimmt werden. Wenn der OD600-Wert über eine Inkubationszeit von 2 h nicht ansteigt, wird eine stationäre Phase erreicht.
- Zentrifugieren Sie ein geeignetes Zellvolumen, um die Diversität 10x bei 2000 x g für 5 min, 4-6 h nach der Verdünnung der Hefekultur abzudecken (typische OD600-Werte liegen zwischen 4-6), und verwerfen Sie den Überstand.
- Das Zellpellet wird in SG-CAA (siehe Tabelle 1) resuspendiert, um einen OD600 von 1 zu erhalten, und in einen frischen Glaskolben überführt.
HINWEIS: Die Resuspension in einem Galaktose-haltigen Medium induziert die Expression des Proteins auf der Oberfläche durch den Galaktose-induzierbaren Promotor. - Inkubieren Sie Hefekulturen über Nacht bei 20 °C und schütteln Sie sie bei 180 U/min, um die Oberflächenexpression des POI zu induzieren. Nach der Induktion über Nacht erreichen Hefekulturen in der Regel OD600-Werte zwischen 2 und 5; Wenn die Induktionstemperatur auf 37 °C angehoben wird, ist mit OD600-Werten von 4-10 zu rechnen.
HINWEIS: Eine Reduzierung der Temperatur auf 20 °C verlangsamt das Zellwachstum und führt in der Regel zu besseren Anzeigewerten. Eine Induktion bei 37 °C könnte jedoch nützlich sein, um den Selektionsdruck in Richtung stabilisierter Mutantenzu erhöhen 35.
3. Erste Kügelchenauswahlrunde der Hefebibliotheken (Positivauswahl)
HINWEIS: Ein Standardverfahren zur Auswahl der Sippen umfasst 6 Schritte (Tabelle 2).
| Tag | Schritt | |
| 0 | Übernachtungskultur | |
| 1 | Induktion der Proteinexpression auf der Oberfläche der Hefezellen | |
| 2 | Erste Perlenauswahl mit 1 positiven Auswahl | |
| 3 | Entfernung der Kügelchen, Passage, Induktion der Proteinexpression auf der Oberfläche der Hefezellen und Einfrieren der Bibliothek | |
| 4 | Zweite Perlenauswahl mit 3 negativen und 1 positiven Auswahl | |
| 5 | Entfernen der Kügelchen und Einfrieren der Bibliothek | |
Tabelle 2: Typischer Zeitplan für die Durchführung der Kügelchenauswahl einer Hefebibliothek.
- Vorbereitung der Kügelchen
- Bereiten Sie die Kügelchen (siehe Materialtabelle) für die erste Perlenauswahl vor: Für jedes Röhrchen resuspendieren Sie 10 μl Biotin-Bindemittel-Magnetkügelchen (4 × 105 Kügelchen/μl) in 990 μl PBSA (Tabelle 1) zum Waschen und legen Sie das Röhrchen 2 Minuten lang bei geöffnetem Deckel auf ein Magnetgestell. Entfernen Sie vorsichtig den Überstand. Wiederholen Sie den Waschschritt mit 1 mL PBSA.
HINWEIS: Wenn mehrere Röhrchen mit Kügelchen mit demselben biotinylierten Antigen benötigt werden, können diese in diesem Schritt in einem Röhrchen hergestellt werden, um eine einheitliche Antigen-Beads-Lösung zu erhalten. In diesem Fall muss die Antigenmenge im nächsten Schritt entsprechend erhöht werden. - Resuspendieren Sie die Kügelchen in einem Gesamtvolumen von 1 mL PBSA mit 6,7-33 μmol biotinyliertem Antigen (hier: 6,86 μl hRBP4 mit einer Konzentration von 0,125 mg/ml) in einem 1,5 mL Mikrozentrifugenröhrchen.
HINWEIS: Während 6,7 pmole Antigen ausreichend sind, sollten 33 pmoles verwendet werden, wenn das Antigen nicht limitiert. Wenn die Selektion für ein Antigen durchgeführt wird, das an ein niedermolekulares Arzneimittel oder einen Liganden gebunden ist, wie in diesem Fall, muss dieses Medikament/dieser Ligand während der Auswahl vorhanden sein und sollte in diesem Schritt hinzugefügt werden (hier: 5 μM A1120). - 2 h bei 4 °C unter leichtem Schütteln oder Wenden inkubieren.
HINWEIS: In diesem Schritt können Zellen vorbereitet werden. - Nach der Inkubation und vor der Zugabe zu den Zellen legen Sie das Röhrchen 2 Minuten lang bei geöffnetem Deckel auf ein Magnetgestell. Entfernen Sie den Überstand und waschen Sie die antigenbeladenen Kügelchen mit 1 mL PBSA wie oben beschrieben. Wiederholen Sie den Waschschritt.
HINWEIS: Wenn ein Antigen ausgewählt wird, das an ein Arzneimittel oder einen Liganden gebunden ist, sollte der Waschpuffer dieses Arzneimittel oder diesen Liganden im Überschuss enthalten (hier: 5 μM A1120). - Resuspendieren Sie die antigenbeladenen Kügelchen in 50 μl PBSA (in Gegenwart des Arzneimittels oder des Liganden, falls zutreffend).
HINWEIS: Wenn dieselben antigenbeladenen Kügelchen für mehrere Röhrchen hergestellt wurden, passen Sie das Volumen entsprechend an (für die G4-Bibliothek beträgt das endgültige Volumen hier 100 μl, was Beads für zwei Röhrchen ergibt).
- Bereiten Sie die Kügelchen (siehe Materialtabelle) für die erste Perlenauswahl vor: Für jedes Röhrchen resuspendieren Sie 10 μl Biotin-Bindemittel-Magnetkügelchen (4 × 105 Kügelchen/μl) in 990 μl PBSA (Tabelle 1) zum Waschen und legen Sie das Röhrchen 2 Minuten lang bei geöffnetem Deckel auf ein Magnetgestell. Entfernen Sie vorsichtig den Überstand. Wiederholen Sie den Waschschritt mit 1 mL PBSA.
- Vorbereitung der Hefezellen
- Messen Sie OD600 der induzierten Hefekultur wie oben beschrieben.
- Pelletieren Sie ausreichend Zellen, um die 10-fache Diversität der Bibliothek abzudecken (2000 x g, 10 min, 4 °C). Verwenden Sie für die G4-Bibliothek 2,5 x 109 Zellen für die Zentrifugation. Entfernen Sie den Überstand und waschen Sie die Zellen mit 10 mL PBSA.
- Erneut zentrifugieren (2000 x g, 10 min, 4 °C) und die Zellen auf eine Konzentration von 1,25 x 109 Zellen/mL im entsprechenden PBSA-Volumen (hier: 2 mL) resuspendieren und in ein Mikrozentrifugenröhrchen überführen.
HINWEIS: Das Volumen von PBSA hängt von der Anzahl der Zellen ab. - Zentrifugieren (2000 x g, 3 min, 4 °C) und in 950 μl PBSA resuspendieren.
- Inkubation von Hefezellen mit antigenbeladenen Kügelchen
- 50 μl antigenbeladene Kügelchen zu 950 μl Zellsuspension geben und 2 h bei 4 °C unter Schütteln oder Rotation inkubieren.
- Nach der Inkubation die Zell-Antigen-Kügelchenlösung bei geöffnetem Deckel auf ein Magnetgestell stellen. Pipettieren Sie Flüssigkeit vom Deckel in das Röhrchen, um die gesamte Hefekultur einer magnetischen Selektion zu unterziehen. Warten Sie 2 Minuten, bevor Sie ungebundene Zellen vorsichtig entsorgen. Sofort mit 1 mL PBSA waschen.
HINWEIS: Wenn die Selektion in Gegenwart eines Medikaments/Liganden durchgeführt wird - in diesem Beispiel A1120 - muss dies in PBSA vorhanden sein. - Nach dem Waschschritt mit PBSA resuspendieren Sie die Zellen schnell in 1 ml SD-CAA, um ein Austrocknen zu verhindern.
- Fügen Sie 200 mL SD-CAA hinzu und führen Sie serielle Verdünnungen wie oben beschrieben durch. 100 μl auf SD-CAA-Platten auffüllen und 2-3 Tage bei 30 °C inkubieren, um die neue maximale Diversität zu bestimmen. Hier beträgt die Diversität der G4-Bibliothek nach der ersten Perlenauswahl 1,25 x106 Zellen.
HINWEIS: Da die ursprüngliche Bibliothek in dieser Auswahlrunde um das 10-fache übersampelt wurde, kann davon ausgegangen werden, dass die meisten Klone nach der Auswahl mehrfach im angereicherten Pool vorhanden sind. Infolgedessen ist die tatsächliche Diversität geringer, und diese Zahl sollte als Maximalwert betrachtet werden. In der Regel reduziert sich die Diversität innerhalb dieser ersten Auswahlrunde um mindestens das 100-fache. Die verbleibende Kultur (200 ml) wird bei 30 °C unter Schütteln (180 U/min) mindestens 16 Stunden lang inkubiert. Das Protokoll kann nach der Inkubation über Nacht pausiert werden, indem die Zellen bei 4 °C platziert werden.
4. Entnahme der Perlen und Kultivierung vor der nächsten Perlenauswahlrunde
- Messen Sie OD600 wie oben beschrieben. Pelletieren Sie ausreichend Zellen, um die Diversität um mindestens das 10-fache (2000 x g, 3 min, RT) abzudecken. Verwerfen Sie den Überstand und resuspendieren Sie die Zellen in 1 ml SD-CAA.
- In ein Mikrozentrifugenröhrchen umfüllen und bei geöffnetem Deckel 2 Minuten lang auf ein Magnetgestell stellen. Übertragen Sie die ungebundenen Zellen in ein neues Mikrozentrifugenröhrchen und wiederholen Sie den Trennvorgang.
- Die ungebundenen Zellen werden in 100 mL SD-CAA verdünnt und bei 30 °C unter Schütteln bei 180 U/min inkubiert, bis ein OD600 von 3-4 erreicht ist.
HINWEIS: Wenn der OD600 vor der Entnahme der Kügelchen niedriger als 4 ist, können die Zellen direkt danach ohne Inkubation induziert werden. - Wenn ein OD600 von 3-4 erreicht ist, zentrifugieren Sie eine ausreichende Anzahl von Zellen, um die Diversität mindestens 10x (2000 x g, 3 min, RT) abzudecken, und resuspendieren Sie in SG-CAA zu einem OD600 von 1 zur Induktion der Proteinexpression und inkubieren Sie bei 20 °C unter Schütteln bei 180 U/min über Nacht.
- Wenn die verbleibende SD-CAA-Hefekultur die stationäre Wachstumsphase erreicht hat (OD600 über 15), pelletieren Sie ausreichend Zellen, um die Diversität 50x (2000 x g, 3 min, RT) abzudecken. Frieren Sie die Zellen durch Zentrifugation (2000 x g, 3 min) ein, gefolgt von der Resuspension des Zellpellets in SD-CAA mit 15 % Glycerin und Lagerung bei -80 °C.
5. Zweite Perlenauswahlrunde mit 3 negativen und 1 positiven Auswahl
- Bereiten Sie die Zellen und die Antigenkügelchen wie oben beschrieben vor. Bereiten Sie eine Lösung aus nackten Kügelchen (ohne Zugabe des Antigens) für die negative Selektion vor.
HINWEIS: Da 3 negative Selektionen durchgeführt werden, können die Perlen zusammen für alle drei Runden vorbereitet werden. - Nach dem Waschen resuspendieren Sie die Antigenkügelchen in 50 μl PBSA und resuspendieren Sie die blanken Kügelchen in 150 μl PBSA (wenn sie für alle drei negativen Selektionen vorbereitet wurden).
- Für die erste negative Selektion werden 50 μl gewaschene blanke Kügelchen zu 950 μl gewaschenen Zellen in PBSA hinzugefügt und 1,5 Stunden bei 4 °C inkubiert.
- Nach der Inkubation legen Sie die Röhrchen mit den blanken Bead-Cell-Suspensionen bei geöffnetem Deckel auf ein Magnetgestell. Pipettieren Sie die Flüssigkeit im Deckel in das Röhrchen und warten Sie 2 Minuten. Übertragen Sie die ungebundenen Zellen in ein frisches Mikrozentrifugenröhrchen und fügen Sie 50 μl gewaschene blanke Kügelchen hinzu.
- Wiederholen Sie den Vorgang für zwei weitere Runden der negativen Auswahl. Bereiten Sie in der Zwischenzeit antigenbeladene Kügelchen für die Positivselektion vor.
- Nach 3 negativen Selektionsrunden werden 50 μl antigenbeladene Bead-Lösung in die Zellen gegeben und 2 h bei 4 °C inkubiert.
- Legen Sie die Zellen mit den antigenbeladenen Kügelchen bei geöffnetem Deckel auf ein Magnetgestell und pipettieren Sie die Flüssigkeit, die sich im Deckel befindet, in das Röhrchen. Warten Sie 2 Minuten, bevor Sie ungebundene Zellen verwerfen.
- Führen Sie alle verbleibenden Schritte wie für die erste Antigen-Bead-Auswahl beschrieben durch. Der einzige Unterschied zur ersten positiven Selektion (Schritt 3.3.2) ist die Anzahl der Waschschritte: Statt einmal mit PBSA zu waschen, wird die Kügelchen-Suspension, zweimal mit PBSA gewaschen, um die Stringenz der Selektion zu erhöhen.
HINWEIS: In der Regel wird die Diversität nochmals um das 10-100-fache reduziert. Die maximale Diversität der G4-Bibliothek nach dieser zweiten Perlenauswahl betrug 3,45 x 105 Zellen. Wie oben erwähnt, wird erwartet, dass die tatsächliche Vielfalt aufgrund der Überstichprobenziehung der Bibliothek geringer sein wird.
6. Auswahl der Bibliotheken über durchflusszytometrische Sortierung
- Nach Induktion der Oberflächenexpression in SG-CAA über Nacht ist OD600 wie oben beschrieben zu messen. Pelletieren Sie ausreichend Zellen, um die 10-fache Diversität (2000 x g, 5 min, RT) abzudecken. Entsorgen Sie den Überstand.
- Resuspendieren Sie das Pellet in PBSA und geben Sie es in Mikrozentrifugenröhrchen. Verwenden Sie 3 x 107 Zellen zum Färben für jedes Röhrchen. Bereiten Sie je nach Sorte so viele Röhrchen vor, wie Sie benötigen. Bereiten Sie ein Kontrollröhrchen für die Färbung ohne Antigen vor.
HINWEIS: Wenn beispielsweise 30 x 106 Zellen sortiert werden sollen, pelletieren Sie 300 x 106 Zellen, um ein sichtbares Zellpellet zu erzielen und den Verlust von Zellen in den nachfolgenden Schritten zu verhindern. In 5 mL PBSA resuspendieren und 3 bis 4 Aliquots à 500 μl in Mikrozentrifugenröhrchen überführen. Bereiten Sie etwa 3-mal mehr Zellen vor, als sortiert werden sollten, da ein Bruchteil der Zellen während des Färbeprozesses verloren geht. Wenn die Selektion in Gegenwart eines Arzneimittels/Liganden durchgeführt wird, ist eine zusätzliche Kontrolle nur mit dem Antigen, aber ohne das Arzneimittel/den Liganden vorzubereiten. - Erneut zentrifugieren (2000 x g, 5 min, RT), das Pellet in 200 μl PBSA, das das Antigen enthält (hRBP4 mit 5 μM A1120), resuspendieren und 1 h bei 4 °C inkubieren.
HINWEIS: Alle Schritte ab diesem Zeitpunkt sollten auf Eis und mit kalten Reagenzien durchgeführt werden. Um eine Depletion von Antikörpern oder Antigenen zu verhindern, sollten alle Reagenzien in einem stöchiometrischen Überschuss im Vergleich zur Gesamtzahl der in Hefe angezeigten Proteine im Röhrchen zugegeben werden. Andernfalls besteht die Gefahr, dass die Bindung von Hefevarianten aufgrund der begrenzten Verfügbarkeit von Antigenen und/oder Antikörpern eingeschränkt wird. In der Regel werden 5 x 104 Moleküle pro Hefezelle für die Berechnung verwendet, und es wird empfohlen, mindestens einen 10-fachen stochiometrischen Überschuss an Antigen und Antikörpern hinzuzufügen. Ist das Antigen sehr begrenzt, kann der Überschuss auf das 5-fache reduziert werden. Eine detailliertere Erklärung, einschließlich Beispielen für Berechnungen zur Vermeidung von Ligandenverarmungen, findet sich in einem früheren Protokoll9. - Nach der Inkubation die Zellen zentrifugieren (2000 x g, 5 min, 4 °C) und mit 1 ml PBSA waschen (wenn während der Selektion ein Medikament/Ligand vorhanden ist - wie in diesem Fall A1120 - muss dieser im Waschpuffer vorhanden sein).
- Die Zellen werden in 100 μl kaltem PBSA, das die Antikörper für die Display-Färbung und den Nachweis des gebundenen Antigens enthält, resuspendiert und 30 Minuten bei 4 °C inkubiert.
- Verwenden Sie einen Anti-c-myc-Antikörper (endgültige Verdünnung 1:100), um angezeigte POI in voller Länge zu erkennen. Verwenden Sie einen Anti-HA-Antikörper (endgültige Verdünnung 1:50), um die Gesamtmenge der angezeigten Proteine nachzuweisen. Verwenden Sie Penta-His-Antikörper (endgültige Verdünnung 1:20) oder Streptavidin (endgültige Verdünnung 1:200), um Hefezellen auszuwählen, die an das lösliche Antigen binden, das entweder einen 6-fachen HIS-Tag bzw. Biotin enthält.
- Wechseln Sie zwischen den Sortierrunden zwischen den Detektionsreagenzien, um eine Anreicherung von Proteinvarianten zu verhindern, die an das Sekundärreagenz binden.
HINWEIS: Es können verschiedene Fluorophor-Kombinationen verwendet werden, aber Alexa Fluor 488 und Alexa Fluor 647 sind gute Optionen, da sie keine Kompensation in der Durchflusszytometrie erfordern. Um eine Depletion der Antikörper zu verhindern, sollte es im stöchiometrischen Überschuss im Vergleich zur Gesamtzahl der in Hefe angezeigten Proteine im Röhrchen zugegeben werden. In der Regel basieren die Berechnungen auf 5 x 104 Molekülen pro Hefezelle. Eine ausführlichere Erläuterung findet sich in den vorangegangenen Protokollen9.
- Nach der Inkubation zentrifugieren Sie die Zellen (2000 x g, 5 min, 4 °C) und geben Sie 1 ml PBSA (mit dem Wirkstoff/Liganden, für die hRBP4-selektierte G4-Bibliothek 5 μM von A1120) in das Pellet und zentrifugieren Sie erneut. Entfernen Sie den größten Teil des Überstands und bewahren Sie nur 20-30 μl auf, um ein Austrocknen des Pellets zu verhindern.
- Resuspendieren Sie das Pellet erst unmittelbar vor der Sortierung in kaltem PBSA und sortieren Sie nach den Hefezellen, die Fn3-Moleküle mit den gewünschten Eigenschaften aufweisen (d.h. Bindung an hRBP4 in Gegenwart von A1120).
HINWEIS: Die Gating-Strategie ist in Abbildung 2A dargestellt. - Sortieren Sie die Zellen direkt in SD-CAA-Medium, fügen Sie nach der Sortierung weiteres SD-CAA-Medium hinzu und inkubieren Sie bei 30 °C und Schütteln bei 180 U/min.
HINWEIS: Falls das Volumen nach dem Sortieren erheblich zunimmt, sollte ein Zentrifugationsschritt (2000 x g, 5 min, RT) durchgeführt werden, um die überschüssige Mantelflüssigkeit zu entfernen, und die Zellen werden in einem frischen SD-CAA-Medium resuspendiert. Notieren Sie sich die Anzahl der in das neue Röhrchen einsortierten Zellen, da dies die neue maximale Diversität für die nächste Sortierrunde wird. Wie oben erwähnt, wird die tatsächliche Vielfalt der Bibliothek aufgrund der Überstichprobe der Bibliothek geringer sein. - Wiederholen Sie die durchflusszytometrischen Sortierungen über mehrere Runden, um eine Anreicherung der bindenden Proteinvarianten in der Hefebibliothek zu sehen (Abbildung 2B).
HINWEIS: Es kann nützlich sein, die Bibliotheken in einem analytischen durchflusszytometrischen Experiment nebeneinander zu vergleichen. In späteren Selektionsrunden kann eine Verringerung der Konzentration des Antigens von Vorteil sein, um zwischen Bindern mit niedrigerer und höherer Affinität zu unterscheiden und spezifisch nach den Proteinvarianten zu sortieren, die bei niedrigen Konzentrationen ein höheres Bindungssignal an das Ziel zeigen (Abbildung 2C). In späteren Auswahlrunden ist es auch möglich, für eine erhöhte Thermostabilität zu sortieren. Zu diesem Zweck wurde eine zufällige Mutagenese der Bibliothek mittels epPCR durchgeführt, wie in Abschnitt 7 beschrieben. Bereiten Sie die Zellen ähnlich wie bei anderen durchflusszytometrischen Sortierrunden durch Induktion, Bestimmung von OD600 und Waschen der Zellen vor. Legen Sie Hefezellen in PBSA für 10 Minuten auf Eis, gefolgt von einer 10-minütigen Inkubation in einem Thermocycler für den Hitzeschock bei erhöhter Temperatur. Legen Sie sie anschließend erneut für 10 Minuten auf Eis und fahren Sie mit dem Färbeprotokoll fort. Stabilisierte Varianten widerstehen der thermischen Denaturierung und zeigen daher nach der Wärmeinkubation eine gleichbleibende Bindung. Für einen Hitzeschock können unterschiedliche Temperaturen getestet werden, abhängig von der anfänglichen Thermostabilität des Wildtyp-Proteins. In der Regel eignen sich Temperaturen von 40-80 °C. Oberhalb von 48 °C nimmt die Lebensfähigkeit der Hefezellen jedoch drastisch ab. Daher ist es notwendig, die Plasmide nach dem Sortieren der hitzegeschockten Hefebibliothek zu extrahieren, wie in Schritt 7.1 beschrieben. In diesem Fall müssen EBY100-Zellen frisch mit angereicherten Varianten transformiert werden, bevor sie in die nächste Sortierrunde36 übergehen. Ein ausführlicheres Protokoll zur Erhöhung der Thermostabilität findet sich an anderer Stelle37. Wenn die Denaturierung des angezeigten Proteins reversibel ist, funktioniert diese Methode nicht. Alternativ können Sie die Proteinexpression am Tag vor der Sortierung bei 37 °C statt bei 20 °C induzieren und nach Proteinvarianten sortieren, die noch eine effiziente Anzeige in voller Länge aufweisen.
7. Affinitätsreifung mit epPCR zur Einführung von Zufallsmutationen
HINWEIS: Die Affinitätsreifung mittels epPCR kann entweder vor der ersten durchflusszytometrischen Sortierrunde oder zwischen den durchflusszytometrischen Sortierrunden durchgeführt werden. Für die Selektion der G4-Bibliothek mit hRBP4 in Gegenwart von A1120 wurde eine Affinitätsreifung vor der ersten Runde der durchflusszytometrischen Sortierung durchgeführt. Dies hängt auch von der Bibliotheksgröße nach der Auswahl der Beads und dem Bindungssignal ab, das mit der Durchflusszytometrie nachgewiesen werden kann. Insbesondere in Fällen, in denen die Affinitäten nach der Selektion der Kügelchen nicht ausreichen, um in durchflusszytometrischen Experimenten ein Signal zu erhalten (da das Antigen während der Waschschritte schnell dissoziiert), kann die epPCR verbesserte Varianten erzeugen, die anschließend mittels Durchflusszytometrie nachgewiesen und selektiert werden können.
- Führen Sie eine Hefe-Miniprep-DNA-Extraktion aus der angereicherten Hefebibliothek unter Verwendung des kommerziellen Plasmid-Miniprep-II-Kits (siehe Materialtabelle) mit den folgenden Modifikationen durch:
- Die Zelldichte der Übernachtkultur messen, morgens auf einen OD600 von 0,2 verdünnen und bei 30 °C unter Schütteln inkubieren.
- Wenn OD600 0,6 erreicht, ernten Sie 3 ml Kultur, indem Sie die Zellen 2 Minuten lang bei 600 x g zentrifugieren.
- Geben Sie 200 μl Lösung 1 in das Pellet. Fügen Sie 6 μl Zymolyase hinzu. Das Pellet durch Schnippen oder leichtes Vortexen resuspendieren und 3 h bei 37 °C inkubieren.
- 200 μl Lösung 2 zugeben und vorsichtig mischen. 400 μl Lösung 3 zugeben und vorsichtig mischen.
- Zentrifugieren Sie 3 Minuten lang bei maximaler Drehzahl.
- Der Überstand wird in die Spin-I-Säule überführt, 30 s lang bei maximaler Geschwindigkeit zentrifugiert und der Durchfluss verworfen.
- Mit 550 μl ethanolhaltigem Waschpuffer waschen und 2 Minuten bei maximaler Geschwindigkeit zentrifugieren.
- Übertragen Sie die Säule in ein sauberes Mikrozentrifugenröhrchen und zentrifugieren Sie sie 2 Minuten lang bei maximaler Drehzahl, um den restlichen Waschpuffer zu entfernen.
- Übertragen Sie die Säule in ein neues Mikrozentrifugenröhrchen, fügen Sie 10 μl nukleasefreies Wasser hinzu und drehen Sie sie 1 Minute lang mit maximaler Geschwindigkeit, um die DNA zu eluieren.
- Führen Sie eine erste PCR durch, um das Template für epPCR zu amplifizieren.
- Amplifizieren Sie die DNA durch PCR mit Primern, die an der Außenseite des Inserts auf dem Anzeigevektor der Hefeoberfläche geglüht werden.
HINWEIS: Da während der Hefe-Minivorbereitung viel genomische Hefe-DNA zusammen mit dem interessierenden Plasmid eluiert wird, wird dieser Schritt empfohlen, um ein sauberes DNA-Fragment zu erhalten, das als Template für die epPCR verwendet werden kann. - Für pCTCON2 verwenden Sie Primer fwd 5'- GGCTGGTGGAGGCGGTAGCGGAGGCGGAG
GGTCGGCTAGC und rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC mit den in Tabelle 3 beschriebenen Reaktionskomponenten und den in Tabelle 4 dargestellten Zyklusbedingungen. - Reinigen Sie die PCR mit einem PCR- und DNA-Aufreinigungskit. Eluieren Sie in 20 μl nukleasefreiem H2O.
- Amplifizieren Sie die DNA durch PCR mit Primern, die an der Außenseite des Inserts auf dem Anzeigevektor der Hefeoberfläche geglüht werden.
- Führen Sie eine epPCR durch, um zufällige Mutationen einzuführen.
- Führen Sie eine epPCR mit Nukleotidanaloga mit den angegebenen Inhaltsstoffen (Tabelle 5) und Bedingungen (Tabelle 6) durch.
- Lassen Sie das gesamte epPCR-Produkt 45 Minuten lang auf einem präparativen 1%igen Agarose-Gel bei 120 V laufen. Fügen Sie einen Marker hinzu und lassen Sie eine Spur zwischen dem epPCR-Produkt und der Leiter. Exzidieren Sie die gewünschte Bande, die der erwarteten Größe des Einsatzes entspricht, reinigen Sie sie mit einem Gelextraktionskit und messen Sie die DNA-Konzentration.
- Amplifizieren Sie die randomisierte DNA durch PCR.
- Amplifizieren Sie 50 ng der DNA mit einer Q5-Polymerase. Richten Sie 2 x 100 μl PCR-Reaktion für jede Elektroporation ein (Tabelle 7) mit den angegebenen Bedingungen (Tabelle 8). Verwenden Sie die gleichen Primer, die für die erste PCR verwendet wurden.
HINWEIS: Für die Elektroporation in Hefe werden große Mengen an DNA benötigt. - Analysieren Sie 5 μl des PCR-Produkts auf einem 1%igen Agarose-Gel und führen Sie es 45 Minuten lang mit 120 V durch.
- Amplifizieren Sie 50 ng der DNA mit einer Q5-Polymerase. Richten Sie 2 x 100 μl PCR-Reaktion für jede Elektroporation ein (Tabelle 7) mit den angegebenen Bedingungen (Tabelle 8). Verwenden Sie die gleichen Primer, die für die erste PCR verwendet wurden.
- Reinigen Sie die amplifizierte DNA mit Ethanolfällung.
- Pools 2 x 100 μL zusammen. 20 μl (10 % des Volumens) steril filtriertes 3 M Natriumacetat mit einem pH-Wert von 5,2 zugeben.
HINWEIS: Dieser Puffer kann im Voraus vorbereitet und einige Monate bei RT gelagert werden. - Fügen Sie 440 μL 100% Ethanol (oder mindestens 2 x Volumen) hinzu und inkubieren Sie 2 Minuten lang bei RT.
- Bei 20.000 x g für 5 min bei RT zentrifugieren. Überstand entfernen, 500 μL 70%iges Ethanol zugeben und kurz mischen.
- Bei 20.000 x g 5 min bei RT zentrifugieren, den Überstand entfernen, 500 μL 100%iges Ethanol zugeben und kurz mischen.
- Zentrifugieren Sie bei 20.000 x g für 5 min bei RT. Entfernen Sie den Überstand und lassen Sie das DNA-Pellet unter einer Laminar-Flow-Haube trocknen, bis das gesamte Ethanol verdampft ist.
HINWEIS: Verwenden Sie einen Heizblock bei 48 °C, um diesen Vorgang zu beschleunigen. Das DNA-Kügelchen ist möglicherweise nicht immer sichtbar. - Das getrocknete DNA-Pellet wird in 10 μl nukleasefreiem H2O aufgelöst.
HINWEIS: Die DNA sollte in so wenig Volumen wie möglich aufgelöst werden. Messen Sie nicht die DNA-Konzentration.
- Pools 2 x 100 μL zusammen. 20 μl (10 % des Volumens) steril filtriertes 3 M Natriumacetat mit einem pH-Wert von 5,2 zugeben.
| Volumen [μL] | Endkonzentration | |
| 5x Q5 Verstärker | 10 | 1x |
| 5x Q5 Puffer | 10 | 1x |
| Grundierung fwd 10 μM | 2.5 | 0,5 μM |
| Primer rev 10 μM | 2.5 | 0,5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Q5-Polymerase | 0.5 | 20 U/ml |
| DNA aus Hefe-Miniprep | 10 | |
| Nukleasefreies H2O | 13.5 |
Tabelle 3: Bedingungen für die PCR im 1. Schritt zur Amplifikation der POI-Gene aus dem isolierten Hefe-Miniprep.
| Schritt | Temperatur | Zeit |
| Anfängliche Denaturierung | 98 °C | 30 Sek. |
| 25 Zyklen | 98 °C | 10 Sek. |
| 72 °C | 30 Sek. | |
| 72 °C | 30 Sek. | |
| Letzte Verlängerung | 72 °C | 2 Minuten |
| Halten | 4 °C |
Tabelle 4: Zyklusbedingungen für die PCR im 1. Schritt zur Amplifikation der POI-Gene aus dem isolierten Hefe-Miniprep.
| Volumen [μL] | Endkonzentration | |
| Nukleasefreies H2O | bis zu 50 | |
| 10x Thermopol Puffer | 5 | 1x |
| Primer_fwd (10 μM) | 2.5 | 0,5 μM |
| Primer_rev (10 μM) | 2.5 | 0,5 μM |
| dNTPs (10 mM) | 1 | 200 μM |
| 8-Oxo-dGTP (100 μM) | 1 | 2 μM |
| dPTP (100 μM) | 1 | 2 μM |
| PCR-Produkt aus der 1. PCR | XX | 50 ng |
| Taq-DNA-Polymerase | 0.5 | 0,05 U/μL |
Tabelle 5: Bedingungen für die epPCR, die nach Amplifikation der POI-DNA mit der PCR des 1. Schritts durchgeführt wird.
| Schritt | Temperatur | Zeit |
| Anfängliche Denaturierung | 94 °C | 30 Sek. |
| 15 Zyklen | 94 °C | 45 Sek. |
| 60 °C | 30 Sek. | |
| 72 °C | ca. 1 min | |
| Letzte Verlängerung | 72 °C | ca. 10 Minuten |
| Halten | 4 °C |
Tabelle 6: Zyklusbedingungen für die epPCR.
| Volumen [μL] | Endkonzentration | |
| 5x Q5 Verstärker | 20 | 1x |
| 5x Q5 Puffer | 20 | 1x |
| Grundierung fwd 10 μM | 5 | 0,5 μM |
| Primer rev 10 μM | 5 | 0,5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Q5-Polymerase | 1 | 20 U/ml |
| 50 ng DNA | XX | |
| ddH20 | bis zu 100 |
Tabelle 7: Bedingungen für die PCR der 2. Stufe zur Amplifikation des epPCR-Produkts vor der Elektroporation von EBY100-Zellen.
| Schritt | Temperatur | Zeit |
| Anfängliche Denaturierung | 98 °C | 30 Sek. |
| 25 Zyklen | 98 °C | 10 Sek. |
| 72 °C | 30 Sek. | |
| 72 °C | 30 Sek. | |
| Letzte Verlängerung | 72 °C | 2 Minuten |
| Halten | 4 °C |
Tabelle 8: Zyklusbedingungen für die PCR der 2. Stufe zur Amplifikation des epPCR-Produkts.
8. Linearisierung des Hefe-Display-Vektors für die Elektroporation
- Linearisieren Sie den pCTCON2-Hefeoberflächen-Anzeigevektor (Addgene-Plasmid #41843).
- Führen Sie einen großflächigen Aufschluss durch und verwenden Sie diesen verdauten linearisierten Vektor für alle weiteren Elektroporationen.
- Verdau von 2 x 200 μg pCTCON2-Plasmid-DNA mit Sal I für 24 h bei 37 °C für eine initiale Linearisierung (Tabelle 9).
- Mit NheI und BamHI 24 h bei 37 ΰC aufschließen, um die Beilage herauszuschneiden (Tabelle 10).
- Zum Schluss werden von jedem Enzym 5 μl in das Röhrchen gegeben (Sal I, NheI, BamHI) und erneut 24 h bei 37 °C inkubiert.
- Führen Sie den verdauten Vektor auf einem präparativen Agarosegel aus. Schneiden Sie die Banden heraus und reinigen Sie sie mit einem Gel-Extraktionskit. Eluieren Sie den Vektor in 20 μL, messen Sie die DNA-Konzentration und stellen Sie sie auf etwa 2 μg/μL ein.
HINWEIS: Das Verdauen des Plasmids über einen längeren Zeitraum gewährleistet eine ausreichende Spaltung des Einsatzes und führt nach der Elektroporation zu weniger Hintergrund aus dem Vektor.
| DNS | 200 μg |
| 10x SchnittSmartPuffer | 50 μL |
| Sal I-HF (NEB) | 30 μL (60 HE) |
| H2Ω | Bis zu 500 μl |
Tabelle 9: Bedingungen für den ersten Schritt des großskaligen Aufschlusses des Hefeoberflächenanzeigevektors pCTCON2.
| pCTCON2 (Sal I verdaut) | 500 μL |
| 10x SchnittSmartPuffer | 37,5 μL |
| NheI-HF (NEB) | 15 μl (30 HE) |
| BamHI-HF (NEB) | 15 μl (30 HE) |
| H2Ω | bis zu 875 μL |
Tabelle 10: Bedingungen für den zweiten Schritt des großskaligen Aufschlusses des Hefeoberflächenanzeigevektors pCTCON2.
9. Elektroporation von EBY100 mit randomisierter DNA und linearisiertem Vektor
- Bereiten Sie die EBY100-Hefezellen (ATCC, siehe Materialtabelle) vor, indem Sie sie 3 Tage vor der Elektroporation aus einem Glycerinvorrat schlagen. Da nicht transformierte EBY100-Zellen den Anzeigevektor der Hefeoberfläche nicht enthalten, verwenden Sie Vollmedien-YPD-Platten (Tabelle 1). Inkubieren bei 30 °C.
- Einen Tag vor der Elektroporation inokulieren Sie eine einzelne EBY100-Kolonie in 30 mL frisch zubereitetes YPD-Medium (Tabelle 1). Über Nacht bei 30 °C unter Schütteln bei 180 U/min inkubieren.
HINWEIS: Das YPD-Medium sollte nicht älter als 1 Monat sein. - Am nächsten Morgen verdünnen Sie die EBY100-Übernachtkultur auf einen OD600 von 0,2 in 100 mL YPD-Medium und inkubieren Sie bei 30 °C unter Schütteln bei 180 U/min.
HINWEIS: Ein Kulturvolumen von 50 mL ist ausreichend für 2 Elektroporationen. In der Regel werden 100 ml vorbereitet, um genügend Zellen für die Kontrollen zu haben (nur Zellen und Zellen plus Vektor). - Wenn ein OD600 von 1,3-1,5 erreicht ist, werden die Zellen in 50-ml-Röhrchen bei 2000 x g für 3 min pelletiert und der Überstand entfernt.
- Resuspendieren Sie das Pellet in 25 mL steril filtriertem 100 mM Lithiumacetat (siehe Materialtabelle). Frisch zubereitetes steril filtriertes DVB-T (1 M) (siehe Materialtabelle) wird bis zu einer Endkonzentration von 10 mM zugegeben.
HINWEIS: Das Lithiumacetat-Volumen sollte halb so groß sein wie das ursprüngliche Volumen von YPD. Skalieren Sie bei Bedarf nach unten oder oben. Lithiumacetat (100 mM) kann im Voraus aufbereitet und einige Monate bei RT gelagert werden, aber der 1 M DTT-Vorrat sollte direkt vor der Verwendung vorbereitet und auf Eis aufbewahrt werden. - Inkubieren Sie die Zellen bei 30 °C und schütteln Sie sie 10 Minuten lang.
- Bei 2000 x g für 3 min zentrifugieren, den Überstand verwerfen und die Zellen auf Eis legen.
HINWEIS: Alle folgenden Schritte sollten auf Eis und mit gekühlten Reagenzien, Küvetten und Mikrozentrifugenröhrchen durchgeführt werden. - Das Pellet wird in 25 mL kaltem, sterilem H2O resuspendiert. Zentrifugieren Sie es 3 Minuten lang bei 2000 x g .
HINWEIS: Das verwendete H2O-Volumen sollte die Hälfte des ursprünglichen YPD-Volumens betragen. - In 350 μl sterilem H2O resuspendieren.
HINWEIS: Das Gesamtvolumen nach der Resuspension sollte 500 μl betragen. Bei Bedarf wird mehr H2O zugegeben. - Bereiten Sie die DNA-Bibliothek parallel vor: Legen Sie die Mikrozentrifugenröhrchen mit den ethanolgereinigten Einsätzen auf Eis und fügen Sie 4 μg linearisierten pCTCON2-Vektor hinzu.
- Geben Sie 250 μl der Zellen zur DNA und mischen Sie sie vorsichtig durch Schneiden. In eine vorgekühlte 2 mm Elektroporationsküvette umfüllen und bis zur Elektroporation auf Eis stellen.
HINWEIS: Fügen Sie Steuerelemente hinzu (Zellen nur ohne DNA und Zellen mit linearisiertem Vektor nur ohne Insert), um den Erfolg der Elektroporation zu bestimmen. - Elektroporat mit 2 mm Elektroporationsküvetten (siehe Materialtabelle) unter folgenden Bedingungen: Rechteckprotokoll, Einzelimpuls, 500 V, 15 ms Impulsdauer.
HINWEIS: Typische Einbrüche betragen 3 % bis 9 %. Andere Elektroporatoren können verwendet werden, aber die angegebenen Bedingungen sind für das in dieser Studie verwendete Instrument optimiert. - Die Zellen werden mit 1 ml vorgewärmtem YPD gerettet, in ein vorgewärmtes Mikrozentrifugenröhrchen umgefüllt und 1 h lang bei 30 °C ohne Schütteln inkubiert.
HINWEIS: Platzieren Sie den Heizblock neben dem Elektroporator, um sicherzustellen, dass die Rückgewinnungsrohre auf der richtigen Temperatur bleiben. - Platte 100 μl von 10-3, 10-4, 10-5 und 10-6 Verdünnungen von Zellen auf SD-CAA-Platten (siehe Tabelle 1) und ermöglicht das Wachstum von Transformanten bei 30 °C.
HINWEIS: Die theoretische Bibliotheksdiversität ergibt sich, indem die Anzahl der Transformanten auf das ursprüngliche Kulturvolumen, hier 1 ml, zurückgerechnet wird. Typischerweise ergibt dieses Transformationsprotokoll etwa 10 7-10 8 transformierte Zellen. - Die restlichen Zellen werden 3 min lang bei 2000 x g pelletiert, in 200 mL SD-CAA resuspendiert und bei 30 °C mit Schütteln bei 180 U/min über Nacht inkubiert. Die transformierten Zellen, die die epPCR-randomisierte Bibliothek enthalten, können entweder in SD-CAA passageiert und am nächsten Tag für die Oberflächenexpression in SG-CAA induziert oder bis zur weiteren Verwendung bei 4 °C gelagert werden. Frieren Sie die Glycerinbestände der Bibliothek nach der epPCR nach Bestimmung der Diversität ein. Da es sich am nächsten Tag bei einem Teil der Zellen immer noch um nicht-transformierte Zellen handelt, die im Transformationsverfahren ohne Gewinnung eines Plasmids übernommen wurden, empfiehlt es sich, im ersten Passageschritt mindestens das 20-fache der Bibliotheksdiversität zu passieren, um diesen Effekt zu kompensieren.
10. Sequenzierung von Hefebibliotheken nach mehreren Auswahlrunden
- Extrahieren Sie die Hefe-DNA, indem Sie eine Hefe-Miniprep wie oben beschrieben durchführen.
- Elektroporate kommerzieller E. coli 10-beta elektrokompetenter Zellen (siehe Materialtabelle) mit der isolierten DNA.
- Legen Sie die Elektroporationsküvetten (1 mm, siehe Materialtabelle) und die Mikrozentrifugenröhrchen auf Eis. Mikrozentrifugenröhrchen mit 950 μl Auswuchsmedium bei 37 °C vorwärmen, um die Zellen nach der Elektroporation zurückzugewinnen.
- Tauen Sie die Zellen auf Eis auf und mischen Sie, indem Sie die Tube leicht schnippen. 50 μl der Zellen in ein gekühltes Mikrozentrifugenröhrchen geben. Fügen Sie 1 μl der Hefe-Miniprep-DNA hinzu.
- Den Zell-DNA-Mix vorsichtig in eine gekühlte Elektroporationsküvette überführen.
HINWEIS: Vermeiden Sie Blasen und achten Sie darauf, dass sich die Zellen am Boden der Küvette ablagern. - Elektroporat mit einer Elektroporationsvorrichtung unter folgenden Bedingungen: 2,0 kV; 200 Ω; 25 μF.
HINWEIS: Typische Zeitkonstanten sind 4,8-5,1 ms. Andere Elektroporatoren können verwendet werden, aber die angegebenen Bedingungen sind für das in dieser Studie verwendete Instrument optimiert. - Sofort 950 μl 37 μC vorgewärmtes Auswachsmedium (siehe Materialtabelle) in die Küvetten geben, vorsichtig mischen und zurück in die warmen Mikrozentrifugenröhrchen geben.
HINWEIS: Platzieren Sie den Heizblock neben dem Elektroporator, um sicherzustellen, dass die Rückgewinnungsrohre auf der richtigen Temperatur bleiben. - 40 min bei 37 °C und 300 U/min inkubieren. Platte 10 μl, 100 μl und den Rest der Zellen (nach der Zentrifugation) auf eine vorgewärmte selektive LB-Platte geben (siehe Tabelle 1). Die Platten über Nacht bei 37 °C inkubieren.
HINWEIS: Für den pCTCON2-Vektor wird Ampicillin zur Antibiotikaauswahl verwendet. - Bereiten Sie eine Kopierplatte mit LB-Agar und dem erforderlichen Antibiotikum vor (hier wird Ampicillin verwendet): Pipettieren Sie 200 μl LB-Agar mit 100 μg/ml Ampicillin in jede Vertiefung einer 96-Well-Platte und lassen Sie es verfestigen.
- Bereiten Sie eine 96-Well-Sequenzierungsplatte mit 50 μl LB und 100 μg/ml Ampicillin pro Well vor.
- Nehmen Sie eine einzelne Kolonie aus den Platten und inokulieren Sie zuerst die Kopierplatte und die gleiche Vertiefung der Sequenzierplatte.
HINWEIS: Die Kopierplatte stellt sicher, dass die gewünschten Plasmide durch Beimpfen einer Flüssigkultur und Durchführen eines Miniprep leicht erhalten werden können. - Inkubieren Sie die Kopierplatte über Nacht bei 37 °C und lagern Sie sie dann bei 4 °C.
- Inkubieren Sie die Sequenzierplatte für 3-4 h bei 37 °C unter Schütteln (180 U/min) und ziehen Sie sie mit einer Plastik- oder Aluminiumfolie bedeckt auf.
- Verschließen Sie die Sequenzierplatte und senden Sie sie zusammen mit 500 μl 10 μM pCTCON2-Sequenzierprimer (5'-CGTTTGTCAGTAATTGCGGTTCTC) zur Sequenzierung.
- Für die Analyse der Sequenzen werden die erhaltenen DNA-Sequenzen mit einem geeigneten Programm Ihrer Wahl, z. B. EMBOSS Transeq38, in Aminosäuresequenzen übersetzt. Verwenden Sie als Nächstes Multiple Sequence Alignment (MUSCLE)39 oder ein ähnliches Alignment-Tool, um die Variantensequenzen aneinander und an das elterliche Wildtyp-Protein auszurichten.
HINWEIS: Durch die Sequenzierung von Bibliotheken verschiedener Selektionsrunden (z. B. nach einer früheren Selektionsrunde sowie nach zusätzlichen Selektionsrunden mit niedrigeren Antigenkonzentrationen zur Anreicherung von Varianten mit hoher Affinität) ist es möglich, verschiedene Varianten zu identifizieren. Dies hängt stark von der Stringenz der Gates ab, die während der Sortierrunden angewendet werden, und damit von der Vielfalt der Bibliotheken.
Ergebnisse
Die G4-Bibliothek wurde gegen das Antigen hRBP4 selektiert, das an den niedermolekularen Wirkstoff A1120 gebunden ist. Die Färbung der Bibliotheken für die durchflusszytometrische Sortierung wurde wie in Methode 6 beschrieben durchgeführt, und die angewandte Gating-Strategie ist in Abbildung 2A dargestellt. Ein erstes Gate umfasste alle Zellen basierend auf der Zellmorphologie, und das zweite Gate (Histogramm der FSC-Breite) zeigte eine stringente Gating-Strategie, die angewendet wurde, um einzelne Zellen auszuwählen und Zellaggregate zu entfernen. Das dritte und letzte Gate zeigte die Darstellung von Proteinvarianten (x-Achse) versus Antigenbindung (y-Achse). Hefezellen, die sowohl Display- als auch Bindungssignale zeigten, wurden sortiert. Wichtig ist, dass das Sortiergatter stringent eingestellt wurde, um Bindungsdomänen mit hohem Bindungssignal und damit hoher Affinität anzureichern. Diese stringente Selektion führte zu einer Anreicherung von Hefezellen, die während der gesamten Selektionskampagne spezifisch an das Zielantigen binden (Abbildung 2B). In späteren durchflusszytometrischen Sortierrunden wurde die Antigenkonzentration um das 10-fache (von 100 nM auf 10 nM) gesenkt. Dadurch wurde das Gesamtbindungssignal reduziert, und nur Bindemittel mit einer hohen Affinität waren noch nachweisbar und sortiert (Abbildung 2C).
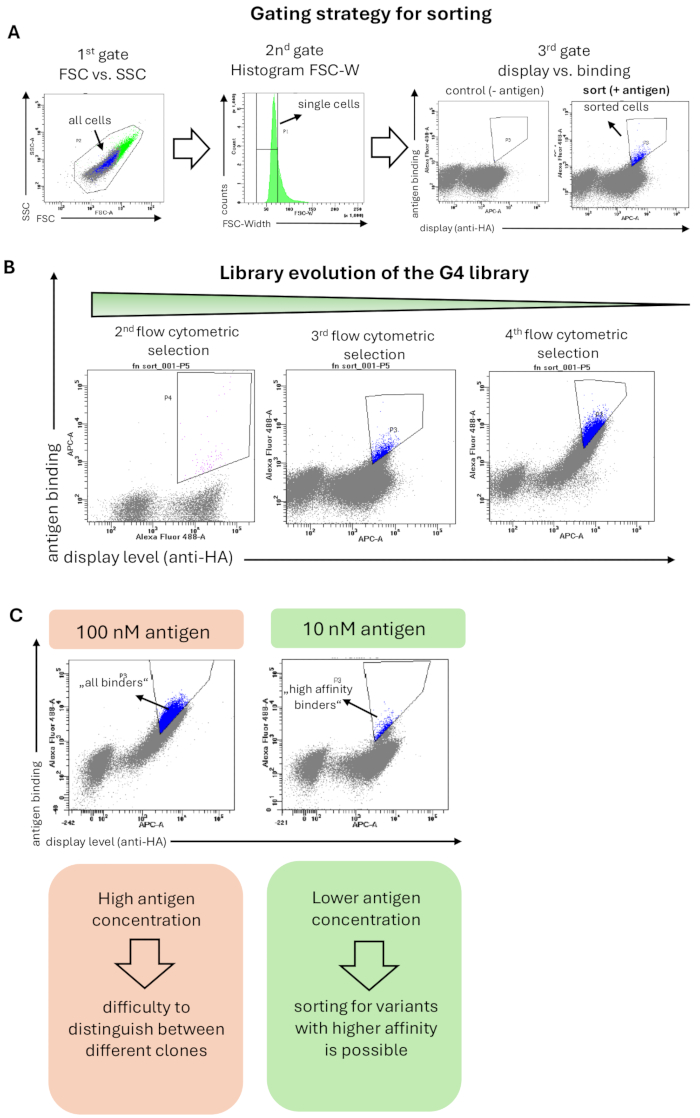
Abbildung 2: Repräsentative Ergebnisse einer Selektion der Fn3-basierten G4-Bibliothek zur Bindung an das Antigen (hRBP4 in Gegenwart von A1120). (A) Die allgemeine Gating-Strategie zur Sortierung von Hefebibliotheken. Das erste Gatter (FSC vs. SSC) besteht darin, alle Hefezellen auszuwählen und Streuereignisse auszuschließen; Das zweite Gate (Histogramm von FSC-W) zielt darauf ab, Zellaggregate zu entfernen und nur einzelne Hefezellen auszuwählen. Das dritte Gate stellt das Oberflächenanzeigeniveau (Nachweis des HA- oder c-myc-Tags) vs. die Bindung an das Antigen dar (hier hRBP4 in Gegenwart von 5 μM A1120, nachgewiesen durch einen Anti-His-Antikörper). Die Bibliothek wurde zusätzlich nur mit Sekundärantikörpern (ohne Antigen) gefärbt, bei denen keine Antigenbindung zu erwarten ist. Sortierte Zellen werden blau hervorgehoben. (B) Entwicklung der G4-Bibliothek über 3 Runden der durchflusszytometrischen Sortierung. Eine Anreicherung der Bindungspopulation kann mit jeder Auswahlrunde beobachtet werden. (C) Die Verwendung niedrigerer Antigenkonzentrationen ermöglicht die Selektion von Proteinvarianten mit einer höheren Affinität zum Zielantigen. Bei 10-facher Reduktion der Antigenkonzentration (hier hRBP4) erscheinen unterschiedliche Diagonalen, die auf das Vorhandensein von Klonen mit höherer (sortierte Zellen, blau) oder niedrigerer Affinität hinweisen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Darstellung der Hefeoberfläche hat sich zu einer der wichtigsten Methoden des Protein-Engineerings entwickelt. Obwohl es häufig für das Engineering der Affinität 1,18,40,41, der Expression/Stabilität 24,27,42,43 und der Aktivität28,44 eingesetzt wird, sind weitere Anwendungen wie das Epitop-Mapping45,46 oder die Charakterisierung der einzelnen Mutanten auf der Oberfläche von Hefezellen9 sind ebenfalls möglich. In diesem Protokoll stellen wir die grundlegenden Schritte für den Start einer Auswahlkampagne für Hefeoberflächendisplays vor, einschließlich der Selektion mit magnetischen Beads und durch durchflusszytometrische Sortierung sowie der Diversifizierung der Hefebibliothek durch epPCR für die Affinitätsreifung.
Eine wesentliche Voraussetzung für die Auswahl herkömmlicher Hefeoberflächen ist die Verfügbarkeit von löslichem Protein von ausreichender Qualität. Ausgehend von einem gut gefalteten Zielprotein mit hoher Reinheit und einem definierten Oligomerisierungszustand (d.h. monomeres Protein sollte nur als Monomer vorliegen) bietet die höchste Erfolgsrate bei der Selektion einer Proteinvariante, die an das Zielantigen mit hoher Affinität bindet. Eine Alternative für schwer zu exprimierende Zielproteine ist die zellbasierte Selektion, die eine vernünftige Strategie darstellt, um diese Einschränkung zu umgehen47. Die Darstellung der Hefeoberfläche bietet jedoch viele Vorteile, wie z.B. die Möglichkeit, resultierende Proteinvarianten direkt auf der Oberfläche der Hefe zu charakterisieren, ohne dass eine aufwändige und zeitintensive Klonierung, Expression in einem löslichen Format und Proteinaufreinigung durchgeführt werden müssen. Sowohl die Affinität als auch die Stabilität der Varianten können direkt an der Hefeoberflächeanalysiert werden 9.
In diesem Protokoll zeigen wir, wie die G4-Bibliothek von Proteinvarianten, genauer gesagt der 10. Typ-III-Domäne von humanem Fibronektin, für die Bindung an das Antigen hRBP4 in Gegenwart des kleinen Moleküls A1120 ausgewählt wurde. Die Kombination von Bead-Selektion und durchflusszytometrischer Sortierung führte zu einer Anreicherung der Varianten, die eine erhöhte Bindung an das Zielantigen während der gesamten Selektionsrunden zeigte (Abbildung 2B). Wir zeigten, dass die Verwendung niedrigerer Antigenkonzentrationen die Selektion von Proteinvarianten mit hoher Affinität ermöglicht (Abbildung 2C). Typischerweise liegen Affinitäten, die mit der Auswahl von Hefedisplays erreicht werden können, im nanomolaren oder sogar pikomolaren Bereich18. Die endgültigen Affinitäten hängen vom Zielantigen, der Anzahl der Selektionsrunden und der Affinitätsreifung, dem verwendeten Bindungsgerüst und der angewandten Gating-Strategie ab. Die Charakterisierung einzelner Proteinvarianten wird in diesem Protokoll nicht behandelt, aber in unserer vorherigen Arbeitausführlich erläutert 9. Obwohl das Hefedisplay ursprünglich für die Entwicklung von Antikörperfragmenten wie scFvs 1,40 eingesetzt wurde, wurde das Verfahren auch für nicht-antikörperbasierte Proteine weit verbreitet10.
Zusammenfassend lässt sich sagen, dass das Hefeoberflächendisplay ein leistungsstarkes Protein-Engineering-Werkzeug ist, das die Generierung von Proteinvarianten mit neuartigen oder verbesserten Eigenschaften ermöglicht, wie z. B. die Bindung an fast jedes Zielprotein und/oder eine erhöhte Stabilität.
Offenlegungen
M.W.T. wird von Miltenyi Biotec gefördert. Alle Autoren sind Erfinder von Patentanmeldungen für Technologien und technisch hergestellte Proteine, die unter Verwendung von Hefeoberflächendisplays entwickelt wurden.
Danksagungen
Diese Arbeit wurde unterstützt durch den österreichischen Wissenschaftsfonds (FWF-Projekt W1224 - Doktoratsprogramm zur biomolekularen Technologie von Proteinen - BioToP und FWF-Projekt ESP 465-B), das Bundesministerium für Digitalisierung und Wirtschaftsstandort Österreichs, die Nationale Stiftung für Forschung, Technologie und Entwicklung Österreichs bis hin zur Christian Doppler Forschungsvereinigung (Christian Doppler Labor für Next Generation CAR T-Zellen), und durch private Spenden an das St. Anna Kinderkrebsforschungsinstitut (Wien, Österreich). E.S. ist Stipendiatin eines DOC Fellowships der Österreichischen Akademie der Wissenschaften am St. Anna Kinderkrebsforschungsinstitut. Die Figuren wurden mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
Referenzen
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten