Method Article
Engenharia de proteínas por exibição de superfície de levedura
Neste Artigo
Resumo
Este protocolo descreve as etapas essenciais para a realização de campanhas de seleção de exibição de superfície de levedura para enriquecer as variantes de proteína que se ligam a um antígeno de interesse.
Resumo
A engenharia de proteínas permite a melhoria das funções existentes de uma determinada proteína ou a geração de novas funções. Uma das ferramentas mais utilizadas e versáteis no campo da engenharia de proteínas é a exibição da superfície da levedura, onde um conjunto de proteínas aleatórias é expresso na superfície da levedura. A ligação do fenótipo (por exemplo, ligação da proteína exibida pela levedura ao antígeno de interesse) e genótipo (o plasmídeo que codifica a variante da proteína) permite a seleção desta biblioteca para as propriedades desejadas e subsequente sequenciamento de variantes enriquecidas. Ao combinar a seleção de esferas magnéticas com a classificação por citometria de fluxo, variantes de proteínas com ligação aprimorada a um antígeno alvo podem ser selecionadas e enriquecidas. Notavelmente, além da maturação da afinidade, a ligação a um alvo também pode ser alcançada sem qualquer afinidade de ligação inicial. Aqui, fornecemos um protocolo passo a passo que cobre todas as partes essenciais de uma campanha de seleção de exibição de superfície de levedura e fornece exemplos de resultados típicos de exibição de superfície de levedura. Demonstramos que a exibição da superfície da levedura é um método amplamente aplicável e robusto que pode ser estabelecido em qualquer laboratório de biologia molecular com acesso à citometria de fluxo.
Introdução
A exibição da superfície da levedura é uma das principais tecnologias no campo da engenharia de proteínas. Ele permite a seleção de variantes de proteínas com propriedades desejadas, como afinidade ou estabilidade aprimoradas. Introduzido pela primeira vez em 19971, é uma das tecnologias de exibição mais comumente usadas além da exibição de fagos 2,3, exibição de ribossomos4 e exibição de células de mamíferos 5,6,7. A proteína de interesse (POI) é exibida na superfície das células de levedura, fundindo-a para ancorar proteínas. Uma variedade de diferentes proteínas âncora está disponível e, mais comumente, o POI é fundido ao terminal C da proteína de aglutinina de levedura Aga2p 1,8. Além disso, o POI é normalmente flanqueado por duas tags, como uma tag de hemaglutinina (HA-tag) e uma tag c-myc, que permite a detecção do nível de exibição usando anticorpos marcados com fluorescência e citometria de fluxo (Figura 1A). As campanhas típicas de seleção de leveduras envolvem uma combinação de seleções de esferas magnéticas e classificação por citometria de fluxo. As seleções de grânulos permitem o manuseio de grandes números de células e o enriquecimento de variantes de proteínas que se ligam ao antígeno alvo também com baixas afinidades, uma vez que as interações multivalentes com os grânulos carregados de antígenos levam a efeitos de avidez e, portanto, evitam a perda de variantes de baixa afinidade ( Figura 1B ). A análise e seleção de citometria de fluxo oferecem a vantagem de visualizar a ligação das variantes de POI exibidas ao antígeno marcado. Consequentemente, as populações de ligação podem ser classificadas e cultivadas, levando ao enriquecimento de variantes proteicas com características desejadas ao longo de várias rodadas de classificação. Além disso, rodadas adicionais de mutagênese aleatória podem ser realizadas para aumentar ainda mais a diversidade e, portanto, a probabilidade de encontrar mutações adicionais que contribuam para a afinidade e/ou estabilidade da proteína.
A exibição da superfície da levedura apresenta certas vantagens, como (a) maquinário de expressão eucariótica, permitindo o dobramento oxidativo de proteínas, bem como modificações pós-traducionais eucarióticas (como N-glicosilação), (b) normalização da expressão devido à detecção das duas marcas peptídicas flanqueando a proteína, (c) inspeção visual do progresso da seleção por citometria de fluxo (por exemplo, porcentagem de células de ligação e intensidade de ligação) e (d) a possibilidade de analisar mutantes proteicos individuais em levedura (por exemplo, analisando a termoestabilidade e a afinidade), apresentando uma alternativa que economiza tempo para a laboriosa expressão e purificação de proteínas9. De fato, tanto as afinidades (valores de KD) quanto as estabilidades (valores de T50) das proteínas exibidas na superfície da levedura mostraram boas correlações com os dados obtidos por métodos biofísicos e proteínas solúveis 9,10,11,12. A exibição da superfície da levedura tem sido empregada para a engenharia de uma variedade de proteínas, por exemplo, fragmentos de anticorpos 13,14,15,16, o10º domínio de fibronectina tipo III17,18, rcSso7d19,20 ou nós21. Da mesma forma, uma extensa pesquisa foi realizada para otimizar os projetos de bibliotecas de leveduras, alterando as posições aleatórias, bem como o uso de códons de aminoácidos 17,22,23. A exibição da superfície da levedura provou ser bem-sucedida para a engenharia de estabilidade 14,15,24,25, afinidade 18,26,27, atividade enzimática 28,29,30,31 e expressão de proteína32. Além disso, aplicações mais sofisticadas, como ligação condicional na presença ou ausência de uma molécula pequena, foram realizadas usando a exibição da superfície da levedura20.
Neste protocolo, descrevemos todas as etapas essenciais para uma campanha de seleção com exibição da superfície da levedura com o exemplo da biblioteca G4 (baseada no10º domínio da fibronectina tipo III, Fn3) selecionada contra o antígeno proteína de ligação ao retinol humano 4 (hRBP4) na presença da pequena molécula A112020. Essa seleção foi conduzida para produzir uma interação proteína-proteína que depende de uma pequena molécula que pode ser usada como um interruptor molecular. É importante observar que, embora abordagens alternativas sejam possíveis com a exibição da superfície da levedura, as seleções típicas de levedura geralmente visam a ligação a um antígeno alvo sem qualquer afinidade de ligação anterior. Cobrimos todas as etapas de uma campanha de seleção de leveduras, envolvendo o cultivo de uma biblioteca de leveduras, seleções de esferas, classificação por citometria de fluxo e maturação por afinidade por PCR propenso a erros (epPCR). Portanto, este protocolo complementa os protocolos anteriores de exibição da superfície da levedura33,34 e pode ser usado como base para seleções de exibição da superfície da levedura (Figura 1) com qualquer biblioteca de levedura e antígeno alvo de escolha.
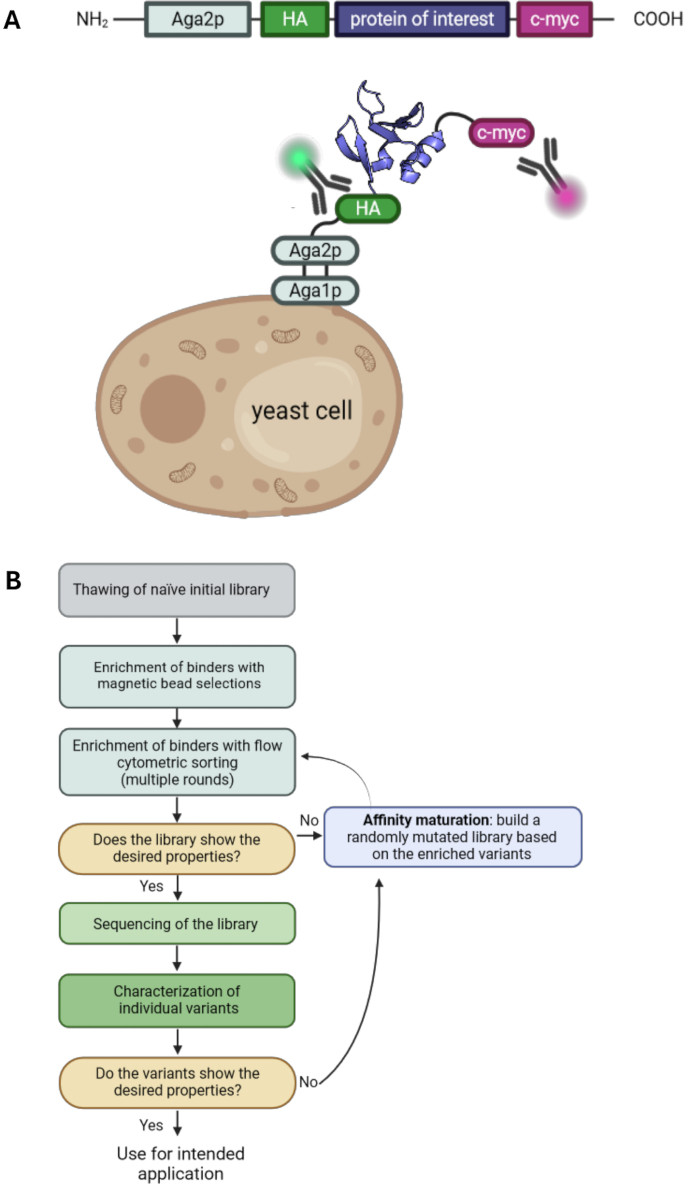
Figura 1: Princípio da exibição da superfície da levedura e um fluxo de trabalho típico para seleções de exibição da superfície da levedura. (A) O POI é clonado em um vetor de exibição de superfície de levedura e normalmente flanqueado por uma tag HA- N-terminal e c-myc-tag. A construção é fundida à proteína de acasalamento de levedura Aga2p para exibição na superfície. A proteína representada é o aglutinante projetado "RS3" do PDB ID: 6QBA20. (B) Fluxograma ilustrando um fluxo de trabalho típico para campanhas de seleção de exibição de superfície de levedura, que combinam enriquecimento de variantes de proteínas com propriedades desejadas por seleções de esferas e classificação por citometria de fluxo, bem como epPCR para maturação de afinidade. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Descongelamento e cultivo de bibliotecas de leveduras
- Descongelar alíquotas suficientes da biblioteca de leveduras congeladas em RT e diluir imediatamente em SD-CAA (ver Tabela 1) até uma DO600 de aproximadamente 1 (isso corresponde a 1 x 107 células / mL).
NOTA: Certifique-se de cobrir a diversidade da biblioteca em pelo menos 10x. Assim, se o tamanho da biblioteca for responsável por 2,5 x 108 clones (esta é a diversidade da biblioteca G4 usada neste exemplo), inocule um mínimo de 2,5 x 109 células em 250 mL de SD-CAA para atingir um OD600 de 1. Este protocolo também pode ser iniciado a partir de culturas líquidas (células de levedura recém-transformadas com uma biblioteca). - Avalie o número de células viáveis.
- Tomar uma alíquota de 100 μL desta suspensão de levedura (a OD600 de 1) e realizar uma série de diluições de 1:10 com 900 μL de meio SD-CAA em tubos de microcentrífuga.
- Prepare diluições de até 10-5 e coloque 100 μL de diluições 10-3, 10-4 e 10-5 em placas de SD-CAA (Tabela 1). Incubar as placas a 30 °C durante 2-3 dias. Certifique-se de que o número de células viáveis após o descongelamento exceda a diversidade da biblioteca em pelo menos 10 vezes.
- Enquanto isso, continuar com a suspensão celular restante e cultivar a cultura de levedura contendo a biblioteca G4 em frascos de vidro durante a noite a 30 °C agitando (180 rpm). Após a incubação durante a noite, continue com a seção 2 (Indução da expressão de proteínas na superfície da levedura).
- Após 2-3 dias de incubação, conte as colônias nas placas SD-CAA por inspeção visual. Ao calcular o número final de células viáveis após o descongelamento, considere o fator de diluição e a proporção do plaqueado (ou seja, 100 μL) versus o volume de cultura original (ou seja, 250 mL).
Células viáveis/ml = número de colónias na placa × factor de diluição
Células viáveis (total) = (células viáveis/mL) × volume de cultura
| Médio/tampão | Componente | Concentração [g/L] | Comentários/Descrição | |||
| SD-CAA | D-glicose | 20 | Dissolva todos os componentes do meio em 1000 mL ddH2O e filtrado estéril com filtros estéreis descartáveis de 0,22 μm. | |||
| Base de nitrogênio de levedura | 6.7 | |||||
| Ácidos de Casmino | 5 | |||||
| Ácido cítrico monohidratado | 7.4 | |||||
| Citrato trissódico di-hidratado | 10.83 | |||||
| SG-CAA | D-galactose | 20 | Dissolva todos os componentes do meio em 1000 mL ddH2O e filtrado estéril com filtros estéreis descartáveis de 0,22 μm. | |||
| D-glicose | 2 | |||||
| Base de nitrogênio de levedura | 6.7 | |||||
| Ácidos de Casamino | 5 | |||||
| hidrogenofosfato dissódico hepta-hidratado | 10.2 | |||||
| Di-hidrogenofosfato de sódio mono-hidratado | 8.56 | |||||
| Placas SD-CAA | Sorbitol | 182 | Dissolver sorbitol, hidrogenofosfato dissódico heptahidratado, dihidrogenofosfato monohidratado e ágar-ágar em 900 mL ddH2O e autoclave. Dissolva e filtre estéril os componentes restantes em 100 mL ddH2O e adicione quando o meio autoclavado estiver morno. | |||
| hidrogenofosfato dissódico hepta-hidratado | 10.2 | |||||
| Di-hidrogenofosfato de sódio mono-hidratado | 7.44 | |||||
| Ágar-ágar | 15 | |||||
| D-glicose | 20 | |||||
| Base de nitrogênio de levedura | 6.7 | |||||
| Ácidos de Casamino | 5 | |||||
| YPD | Peptona | 20 | Prepare um estoque de D-glicose 10x (200 g/L) e filtrado estéril com filtros estéreis descartáveis de 0,22 μm. Dissolva a peptona e o extrato de levedura em 900 mL ddH2O e autoclave. Quando morno, adicione 100 mL 10x D-glicose. | |||
| Extrato de levedura | 10 | |||||
| D-glicose | 20 | |||||
| Placas YPD | Peptona | 20 | Prepare um estoque de D-glicose 10x (200 g/L) e filtrado estéril com filtros estéreis descartáveis de 0,22 μm. Dissolva a peptona, o extrato de levedura e o ágar-ágar em 900 mL ddH2O e autoclave. Quando morno, adicione 100 mL 10x D-glicose. | |||
| Extrato de levedura | 10 | |||||
| D-glicose | 20 | |||||
| Ágar-ágar | 15 | |||||
| PBAS | BSA | 1 | Dissolva o BSA em PBS e filtrado estéril com filtros estéreis descartáveis de 0,22 μm. | |||
Tabela 1: Composição de mídia e buffer.
2. Indução da expressão proteica na superfície da levedura
- Após a incubação durante a noite, as culturas de levedura normalmente atingem um OD600 entre 2-20. Meça o OD600 com um fotômetro para determinar a densidade da cultura de levedura.
NOTA: As medições de diâmetro externo são lineares apenas na faixa de 0,1-1. Portanto, execute diluições (normalmente 1:20 e 1:50) em SD-CAA para determinar o OD600.- Dilua as células em SD-CAA para um OD600 de 1 e certifique-se de que a diversidade seja coberta 10x (ou seja, 2,5 x 109 células são usadas para a diluição da biblioteca G4). Continuar a incubação a 30 °C agitando para permitir que as células atinjam uma fase de crescimento exponencial.
- Prepare estoques de glicerol (cobrindo a diversidade 50x) de bibliotecas de leveduras (em SD-CAA contendo 15% de glicerol) da cultura noturna. Para isso, centrifugue o número necessário de células de levedura (2000 x g, 3 min, 4 ° C), descarte o sobrenadante e ressuspenda o pellet celular em 1 mL de meio SD-CAA suplementado com 15% de glicerol. Congele imediatamente os estoques de glicerol a -80 °C.
NOTA: A viabilidade das células de levedura é aumentada se as culturas de levedura forem cultivadas até uma fase estacionária antes do congelamento. Em rodadas de seleção posteriores, quando a diversidade das bibliotecas é menor, normalmente é suficiente usar 700 μL da cultura de levedura e misturá-la com 300 μL de SD-CAA suplementado com 50% de glicerol. Aqui, as culturas de levedura normalmente mostram valores de OD600 de 15 na fase estacionária, mas o valor real pode depender do fotômetro em uso. Assim, a fase estacionária pode ser determinada por medições repetidas de OD600 . Se o valor OD600 não aumentar durante um período de incubação de 2 h, uma fase estacionária é atingida.
- Centrifugue um volume apropriado de células para cobrir a diversidade 10x a 2000 x g por 5 min, 4-6 h após a diluição da cultura de levedura (os valores típicos de OD600 estão entre 4-6) e descarte o sobrenadante.
- Ressuspenda o pellet celular em SG-CAA (ver Tabela 1) para obter um OD600 de 1 e transfira-o para um novo frasco de vidro.
NOTA: A ressuspensão em um meio contendo galactose induz a expressão da proteína na superfície pelo promotor induzível por galactose. - Incubar culturas de levedura durante a noite a 20 °C enquanto agita a 180 rpm para induzir a expressão superficial do POI. Após a indução durante a noite, as culturas de levedura normalmente atingem valores de OD600 entre 2-5; se a temperatura de indução for elevada para 37 °C, espere valores de OD600 de 4-10.
NOTA: Reduzir a temperatura para 20 ° C retarda o crescimento celular e normalmente produz melhores níveis de exibição. No entanto, a indução a 37 °C pode ser útil para aumentar a pressão de seleção em direção a mutantes estabilizados35.
3. Primeira rodada de seleção de esferas de bibliotecas de levedura (seleção positiva)
NOTA: Um procedimento padrão de seleção de contas envolve 6 etapas (Tabela 2).
| Dia | Passo | |
| 0 | Cultura da noite para o dia | |
| 1 | Indução da expressão proteica na superfície das células de levedura | |
| 2 | Primeira seleção de contas com 1 seleção positiva | |
| 3 | Remoção dos grânulos, passagem, indução da expressão proteica na superfície das células de levedura e congelamento da biblioteca | |
| 4 | Segunda seleção de grânulos com 3 seleções negativas e 1 seleção positiva | |
| 5 | Remoção das contas e congelamento da biblioteca | |
Tabela 2: Linha do tempo típica para a condução de seleções de esferas de uma biblioteca de leveduras.
- Preparação das contas
- Prepare os grânulos (consulte a Tabela de Materiais) para a primeira seleção de esferas: Para cada tubo, ressuspenda 10 μL de esferas magnéticas de aglutinante de biotina (4 ×10 5 contas/μL) em 990 μL de PBSA (Tabela 1) para lavagem, coloque o tubo em um suporte magnético por 2 min com a tampa aberta. Remova cuidadosamente o sobrenadante. Repita a etapa de lavagem com 1 mL de PBSA.
NOTA: Se forem necessários vários tubos de esferas com o mesmo antígeno biotinilado, eles podem ser preparados em um tubo nesta etapa para produzir uma solução uniforme de antígeno-grânulo. Nesse caso, a quantidade de antígeno precisa ser aumentada de acordo na próxima etapa. - Ressuspenda os grânulos em um volume total de 1 mL de PBSA com 6,7-33 pmoles de antígeno biotinilado (aqui: 6,86 μL de hRBP4 com concentração de 0,125 mg / mL) em um tubo de microcentrífuga de 1,5 mL.
NOTA: Embora 6,7 pmoles de antígeno sejam suficientes, 33 pmoles devem ser usados se o antígeno não for limitante. Se a seleção for realizada para um antígeno ligado a um medicamento ou ligante de molécula pequena, como neste caso, esse medicamento/ligante deve estar presente durante a seleção e deve ser adicionado nesta etapa (aqui: 5 μM A1120). - Incubar a 4 °C durante 2 h com agitação suave ou rotação.
NOTA: As células podem ser preparadas durante esta etapa. - Após a incubação e antes da adição às células, coloque o tubo em um rack magnético por 2 min com a tampa aberta. Remova o sobrenadante e lave os grânulos carregados de antígeno com 1 mL de PBSA conforme descrito acima. Repita a etapa de lavagem.
NOTA: Se um antígeno ligado a um medicamento ou ligante for selecionado, o tampão de lavagem deve conter esse medicamento ou ligante em excesso (aqui: 5 μM A1120). - Ressuspenda as esferas carregadas de antígeno em 50 μL de PBSA (na presença do medicamento ou ligante, se aplicável).
NOTA: Se os mesmos grânulos carregados de antígeno foram preparados para vários tubos, ajuste o volume de acordo (para a biblioteca G4, o volume final aqui é de 100 μL produzindo grânulos para dois tubos).
- Prepare os grânulos (consulte a Tabela de Materiais) para a primeira seleção de esferas: Para cada tubo, ressuspenda 10 μL de esferas magnéticas de aglutinante de biotina (4 ×10 5 contas/μL) em 990 μL de PBSA (Tabela 1) para lavagem, coloque o tubo em um suporte magnético por 2 min com a tampa aberta. Remova cuidadosamente o sobrenadante. Repita a etapa de lavagem com 1 mL de PBSA.
- Preparação das células de levedura
- Meça OD600 da cultura de levedura induzida conforme descrito acima.
- Pellet células suficientes para cobrir 10x a diversidade da biblioteca (2000 x g, 10 min, 4 °C). Para a biblioteca G4, use 2,5 x 109 células para centrifugação. Remova o sobrenadante e lave as células com 10 mL de PBSA.
- Centrifugue novamente (2000 x g, 10 min, 4 ° C) e ressuspenda as células a uma concentração de 1,25 x 109 células / mL no volume apropriado de PBSA (aqui: 2 mL) e transfira para um tubo de microcentrífuga.
NOTA: O volume de PBSA depende do número de células. - Centrifugar (2000 x g, 3 min, 4 °C) e ressuspender em 950 μL de PBSA.
- Incubação de células de levedura com grânulos carregados de antígenos
- Adicionar 50 μL de esferas carregadas de antigénio a 950 μl de suspensão celular e incubar durante 2 h a 4 °C com agitação ou rotação.
- Após a incubação, coloque a solução célula-antígeno-grânulo em um rack magnético com a tampa aberta. Pipetar o líquido da tampa para o tubo para submeter toda a cultura de levedura à seleção magnética. Aguarde 2 minutos antes de descartar cuidadosamente as células não ligadas. Lave imediatamente com 1 mL de PBSA.
NOTA: Se as seleções forem realizadas na presença de um medicamento/ligante - neste exemplo A1120 - isso precisa estar presente no PBSA. - Após a etapa de lavagem com PBSA, ressuspenda as células rapidamente em 1 mL de SD-CAA para evitar que sequem.
- Adicione 200 mL de SD-CAA e execute diluições seriadas conforme descrito acima. Colocar 100 μL em placas SD-CAA e incubar durante 2-3 dias a 30 °C para determinar a nova diversidade máxima. Aqui, a diversidade da biblioteca G4 após a primeira seleção de contas é de 1,25 x106 células.
NOTA: Como a biblioteca original foi sobreamostrada 10 vezes nesta rodada de seleção, é razoável supor que a maioria dos clones estará presente várias vezes no pool enriquecido após a seleção. Como consequência, a diversidade real será menor, e esse número deve ser considerado como um valor máximo. Normalmente, a diversidade é reduzida em pelo menos 100x nesta primeira rodada de seleção. Incubar a cultura restante (200 ml) a 30 °C com agitação (180 rpm) durante, pelo menos, 16 h. O protocolo pode ser pausado após a incubação durante a noite, colocando as células a 4 °C.
4. Remoção dos grânulos e cultivo antes da próxima rodada de seleção de grânulos
- Meça OD600 conforme descrito acima. Pellet células suficientes para cobrir a diversidade em pelo menos 10x (2000 x g, 3 min, RT). Descarte o sobrenadante e ressuspenda as células em 1 mL de SD-CAA.
- Transfira para um tubo de microcentrífuga e coloque em um rack magnético por 2 min com a tampa aberta. Transfira as células não ligadas para um novo tubo de microcentrífuga e repita o processo de separação.
- Diluir as células não ligadas em 100 ml de SD-CAA e incubar a 30 °C com agitação a 180 rpm até atingir uma DO600 de 3-4.
NOTA: Se o OD600 for inferior a 4 antes da remoção dos grânulos, as células podem ser induzidas diretamente depois sem incubação. - Quando for atingido um OD600 de 3-4, centrifugar um número suficiente de células para cobrir a diversidade pelo menos 10x (2000 x g, 3 min, RT) e ressuspender em SG-CAA a um OD600 de 1 para indução da expressão proteica e incubar a 20 °C agitando a 180 rpm durante a noite.
- Quando a cultura de levedura SD-CAA restante atingir a fase de crescimento estacionário (OD600 acima de 15), granular células suficientes para cobrir a diversidade 50x (2000 x g, 3 min, RT). Congelar as células por centrifugação (2000 x g, 3 min), seguido de ressuspensão do pellet celular em SD-CAA contendo 15% de glicerol e armazenamento a -80 °C.
5. Segunda rodada de seleção de contas com 3 seleções negativas e 1 positiva
- Preparar as células e as esferas de antigénio como acima descrito. Prepare uma solução de grânulos nus (sem adicionar o antígeno) para as seleções negativas.
NOTA: Como são realizadas 3 seleções negativas, as contas podem ser preparadas juntas para as três rodadas. - Após a lavagem, ressuspenda as esferas de antígeno em 50 μL de PBSA e ressuspenda as esferas nuas em 150 μL de PBSA (se elas foram preparadas para todas as três seleções negativas).
- Para a primeira seleção negativa, adicione 50 μL de grânulos nus lavados a 950 μL de células lavadas em PBSA e incube por 1,5 h a 4 ° C.
- Após a incubação, coloque os tubos contendo as suspensões de células nuas em um rack magnético com a tampa aberta. Pipete qualquer líquido da tampa para o tubo e aguarde 2 min. Transfira as células não ligadas para um novo tubo de microcentrífuga e adicione 50 μL de esferas nuas lavadas.
- Repita o procedimento por mais duas rodadas de seleção negativa. Enquanto isso, prepare grânulos carregados de antígeno para a seleção positiva.
- Após 3 rodadas de seleção negativa, adicione 50 μL de solução de esferas carregada de antígeno às células e incube por 2 h a 4 ° C.
- Coloque as células que contêm as esferas carregadas de antígeno em um rack magnético com a tampa aberta e pipete qualquer líquido que esteja na tampa para dentro do tubo. Aguarde 2 minutos antes de descartar as células não ligadas.
- Execute todas as etapas restantes conforme descrito para a primeira seleção do grânulo de antígeno. A única diferença para a primeira seleção positiva (etapa 3.3.2) é o número de etapas de lavagem: em vez de lavar uma vez com PBSA, lave a suspensão de célula de talão duas vezes com PBSA para aumentar o rigor da seleção.
NOTA: Normalmente, a diversidade é novamente reduzida em 10x-100x. A diversidade máxima da biblioteca G4 após esta segunda seleção de contas foi de 3,45 x 105 células. Como observado acima, espera-se que a diversidade real seja menor devido à sobreamostragem da biblioteca.
6. Seleção de bibliotecas por meio de classificação por citometria de fluxo
- Após a indução noturna da expressão superficial em SG-CAA, meça OD600 conforme descrito acima. Pellet células suficientes para cobrir 10x a diversidade (2000 x g, 5 min, RT). Descarte o sobrenadante.
- Ressuspenda o pellet em PBSA e transfira-o para tubos de microcentrífuga. Use 3 x 107 células para coloração para cada tubo. Prepare quantos tubos forem necessários, dependendo da diversidade. Preparar um tubo de controlo para a coloração sem o antigénio.
NOTA: Por exemplo, se 30 x 106 células devem ser classificadas, faça um pellet de 300 x 106 células para obter um pellet de célula visível e evitar a perda de células nas etapas subsequentes. Ressuspenda em 5 mL de PBSA e transfira 3 a 4 alíquotas de 500 μL cada para tubos de microcentrífuga. Prepare aproximadamente 3 vezes mais células do que o número que deve ser classificado, pois uma fração das células será perdida durante o processo de coloração. Se a seleção for realizada na presença de um medicamento/ligante, prepare um controle adicional apenas com o antígeno, mas sem o medicamento/ligante. - Centrifugar novamente (2000 x g, 5 min, RT), ressuspender o pellet em 200 μL de PBSA contendo o antígeno (hRBP4 com 5 μM A1120) e incubar por 1 h a 4 °C.
NOTA: Todas as etapas a partir deste ponto devem ser realizadas no gelo e com reagentes frios. Para evitar a depleção de anticorpos ou antígenos, todos os reagentes devem ser adicionados em excesso estequiométrico em comparação com o número total de proteínas exibidas por leveduras no tubo. Caso contrário, pode haver o risco de limitar a ligação de variantes exibidas por leveduras devido à disponibilidade limitada de antígenos e/ou anticorpos. Normalmente, 5 x 104 moléculas por célula de levedura são usadas para o cálculo, e é aconselhável adicionar pelo menos um excesso estequiométrico de 10 vezes de antígeno e anticorpos. Se o antígeno for muito limitado, o excesso pode ser reduzido para 5 vezes. Uma explicação mais detalhada, incluindo exemplos de cálculos para evitar a depleção do ligante, é fornecida em um protocolo anterior9. - Após a incubação, centrifugue as células (2000 x g, 5 min, 4 ° C) e lave com 1 mL de PBSA (se um medicamento / ligante estiver presente durante as seleções - como A1120 neste caso - ele precisa estar presente no tampão de lavagem).
- Ressuspenda as células em 100 μL de PBSA frio contendo os anticorpos para coloração de exibição e detecção do antígeno ligado e incube por 30 min a 4 ° C.
- Use o anticorpo anti-c-myc (diluição final 1:100) para detectar o POI exibido em todo o comprimento. Use anticorpo anti-HA (diluição final 1:50) para detectar a quantidade total de proteínas exibidas. Use o anticorpo Penta-His (diluição final 1:20) ou estreptavidina (diluição final 1:200) para selecionar células de levedura que se ligam ao antígeno solúvel contendo uma marca HIS 6x ou biotina, respectivamente.
- Alterne entre os reagentes de detecção entre as rodadas de classificação para evitar o enriquecimento de variantes de proteínas que se ligam ao reagente secundário.
NOTA: Diferentes combinações de fluoróforos podem ser usadas, mas Alexa Fluor 488 e Alexa Fluor 647 são boas opções, pois não requerem compensação na citometria de fluxo. Para evitar a depleção de anticorpos, ele deve ser adicionado em excesso estequiométrico em comparação com o número total de proteínas exibidas por levedura no tubo. Normalmente, os cálculos são baseados em 5 x 104 moléculas por célula de levedura. Uma explicação mais detalhada é fornecida em protocolos anteriores9.
- Após a incubação, centrifugue as células (2000 x g, 5 min, 4 ° C) e adicione 1 mL de PBSA (contendo o medicamento / ligante, para a biblioteca G4 selecionada por hRBP4 5 μM de A1120) ao pellet e centrifugue novamente. Remova a maior parte do sobrenadante e mantenha apenas 20-30 μL para evitar que o pellet seque.
- Ressuspenda o pellet em PBSA frio apenas antes da classificação e classifique as células de levedura que exibem moléculas de Fn3 com as propriedades desejadas (ou seja, ligando-se a hRBP4 na presença de A1120).
NOTA: A estratégia de gating é representada na Figura 2A. - Classifique as células diretamente em meio SD-CAA, adicione mais meio SD-CAA após a classificação e incube a 30 ° C com agitação a 180 rpm.
NOTA: Caso o volume aumente significativamente após a classificação, deve ser realizada uma etapa de centrifugação (2000 x g, 5 min, RT) para remover o excesso de fluido da bainha e as células são ressuspensas em um meio SD-CAA fresco. Anote o número de células classificadas no novo tubo, pois isso se torna a nova diversidade máxima para a próxima rodada de classificação. Conforme observado acima, a diversidade real da biblioteca será menor devido à sobreamostragem da biblioteca. - Repita as classificações de citometria de fluxo por várias rodadas para ver um enriquecimento das variantes de proteínas de ligação na biblioteca de leveduras (Figura 2B).
NOTA: Pode ser útil comparar as bibliotecas lado a lado em um experimento analítico de citometria de fluxo. Em rodadas de seleção posteriores, a redução da concentração do antígeno pode ser benéfica na discriminação entre ligantes de afinidade mais baixa e mais alta e na classificação especificamente para as variantes de proteínas que mostram um sinal de ligação mais alto ao alvo em baixas concentrações ( Figura 2C ). Em rodadas de seleção posteriores, também é possível classificar para aumentar a termoestabilidade. Para isso, foi realizada mutagênese aleatória da biblioteca por epPCR, conforme descrito na Seção 7. Preparar células semelhantes a outras rondas de classificação por citometria de fluxo por indução, determinação de OD600 e lavagem de células. Coloque as células de levedura em PBSA no gelo por 10 minutos, seguido de uma incubação de 10 minutos em um termociclador para o choque térmico a uma temperatura elevada. Em seguida, coloque-os novamente no gelo por 10 min e prossiga com o protocolo de coloração. As variantes estabilizadas resistirão à desnaturação térmica e, portanto, apresentarão ligação mantida após a incubação de calor. Diferentes temperaturas podem ser testadas para choque térmico, dependendo da termoestabilidade inicial da proteína do tipo selvagem. Normalmente, temperaturas que variam de 40 a 80 °C são adequadas. No entanto, acima de 48 °C, a viabilidade das células de levedura diminuirá drasticamente. Portanto, é necessário extrair os plasmídeos após classificar a biblioteca de leveduras em choque térmico, conforme discutido na etapa 7.1. Nesse caso, as células EBY100 precisam ser transformadas com variantes enriquecidas antes de prosseguir para a próxima rodadade classificação 36. Um protocolo mais detalhado para aumentar a termoestabilidade pode ser encontrado em outro lugar37. Se a desnaturação da proteína exibida for reversível, este método não funcionará. Em alternativa, induzir a expressão proteica a 37 °C em vez de 20 °C no dia anterior à triagem e classificar as variantes proteicas que ainda apresentem uma visualização eficiente a todo o comprimento.
7. Maturação de afinidade com epPCR para introduzir mutações aleatórias
NOTA: A maturação por afinidade usando epPCR pode ser realizada antes da primeira rodada de classificação por citometria de fluxo ou entre as rodadas de classificação por citometria de fluxo. Para a seleção da biblioteca G4 com hRBP4 na presença de A1120, a maturação por afinidade foi realizada antes da primeira rodada de classificação por citometria de fluxo. Isso também depende do tamanho da biblioteca após a seleção do cordão e do sinal de ligação que pode ser detectado com citometria de fluxo. Em particular, nos casos em que as afinidades após as seleções de grânulos não são suficientes para obter um sinal em experimentos de citometria de fluxo (porque o antígeno se dissocia rapidamente durante as etapas de lavagem), a epPCR pode gerar variantes aprimoradas que podem ser posteriormente detectadas e selecionadas por citometria de fluxo.
- Realize uma extração de DNA de minipreparação de levedura da biblioteca de levedura enriquecida usando o kit miniprep II de plasmídeo comercial (consulte a Tabela de Materiais) com as seguintes modificações:
- Medir a densidade celular da cultura durante a noite, diluir até uma DO600 de 0,2 de manhã e incubar a 30 °C com agitação.
- Quando OD600 atingir 0,6, colha 3 mL de cultura centrifugando as células a 600 x g por 2 min.
- Adicione 200 μL da Solução 1 ao pellet. Adicione 6 μL de Zymolyase. Ressuspender o pellet por sacudidas ou vórtices suaves e incubar a 37 °C durante 3 h.
- Adicione 200 μL da Solução 2 e misture suavemente. Adicione 400 μL da Solução 3 e misture suavemente.
- Centrifugue na velocidade máxima por 3 min.
- Transferir o sobrenadante para a coluna de rotação I, centrifugar à velocidade máxima durante 30 s e rejeitar o escoamento.
- Lave com 550 μL de tampão de lavagem contendo etanol e centrifugue na velocidade máxima por 2 min.
- Transfira a coluna para um tubo de microcentrífuga limpo e centrifugue na velocidade máxima por 2 min para remover o tampão de lavagem residual.
- Transfira a coluna para um novo tubo de microcentrífuga, adicione 10 μL de água livre de nuclease e gire por 1 min na velocidade máxima para eluir o DNA.
- Execute uma PCR inicial para amplificar o modelo para epPCR.
- Amplificar o DNA por PCR usando primers recozimento na parte externa da inserção no vetor de exibição da superfície da levedura.
NOTA: Como muito DNA genômico de levedura é eluído junto com o plasmídeo de interesse durante a minipreparação de levedura, esta etapa é recomendada para obter um fragmento de DNA limpo para ser usado como modelo para o epPCR. - Para pCTCON2, use primers fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGAG
GGTCGGCTAGC e rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC com os componentes de reação descritos na Tabela 3 e as condições de ciclagem descritas na Tabela 4. - Purifique o PCR com um kit de limpeza de PCR e DNA. Eluir em 20 μL de H2O livre de nuclease.
- Amplificar o DNA por PCR usando primers recozimento na parte externa da inserção no vetor de exibição da superfície da levedura.
- Realize epPCR para introduzir mutações aleatórias.
- Realize uma epPCR com análogos de nucleotídeos com os ingredientes indicados (Tabela 5) e condições (Tabela 6).
- Execute todo o produto de epPCR em um gel preparativo de agarose a 1% a 120 V por 45 min. Inclua um marcador e deixe uma faixa entre o produto epPCR e a escada. Extirpar a banda desejada correspondente ao tamanho esperado da inserção, purificá-la com um kit de extração de gel e medir a concentração de DNA.
- Amplificar o ADN aleatório por PCR.
- Amplificar 50 ng do ADN com uma polimerase Q5. Configure 2 x 100 μL de reação de PCR para cada eletroporação (Tabela 7) com as condições indicadas (Tabela 8). Use os mesmos primers que foram usados para a primeira PCR.
NOTA: Grandes quantidades de DNA são necessárias para a eletroporação em levedura. - Analise 5 μL do produto de PCR em um gel de agarose a 1%, executado com 120 V por 45 min.
- Amplificar 50 ng do ADN com uma polimerase Q5. Configure 2 x 100 μL de reação de PCR para cada eletroporação (Tabela 7) com as condições indicadas (Tabela 8). Use os mesmos primers que foram usados para a primeira PCR.
- Purifique o DNA amplificado com precipitação de etanol.
- Pool 2 x 100 μL juntos. Adicione 20 μL (10% do volume) de acetato de sódio 3 M filtrado estéril, pH 5,2.
NOTA: Este buffer pode ser preparado com antecedência e armazenado por alguns meses no RT. - Adicione 440 μL de etanol a 100% (ou pelo menos 2 x volume) e incube em RT por 2 min.
- Centrifugue a 20.000 x g por 5 min em RT. Remova o sobrenadante, adicione 500 μL de etanol a 70% e misture brevemente.
- Centrifugue a 20.000 x g por 5 min em RT. Remova o sobrenadante, adicione 500 μL de etanol 100% e misture brevemente.
- Centrifugue a 20.000 x g por 5 min em RT. Remova o sobrenadante e deixe o pellet de DNA secar sob uma capela de fluxo laminar até que todo o etanol evapore.
NOTA: Use um bloco de calor a 48 °C para acelerar este processo. O pellet de DNA pode não ser visível o tempo todo. - Dissolver o sedimento de ADN seco em 10 μL de H2O isento de nuclease.
NOTA: O DNA deve ser dissolvido no menor volume possível. Não meça a concentração de DNA.
- Pool 2 x 100 μL juntos. Adicione 20 μL (10% do volume) de acetato de sódio 3 M filtrado estéril, pH 5,2.
| Volume [μL] | Concentração final | |
| 5x aprimorador Q5 | 10 | 1x |
| 5x buffer Q5 | 10 | 1x |
| Primer fwd 10 μM | 2.5 | 0,5 μM |
| Primer rev 10 μM | 2.5 | 0,5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Polimerase Q5 | 0.5 | 20 U/mL |
| DNA de minipreparação de levedura | 10 | |
| H2O livre de nuclease | 13.5 |
Tabela 3: Condições para a1ª etapa de PCR para amplificação dos genes POI da minipreparação de levedura isolada.
| Passo | Temperatura | Hora |
| Desnaturação inicial | 98 °C | 30 s |
| 25 ciclos | 98 °C | 10 s |
| 72 °C | 30 s | |
| 72 °C | 30 s | |
| Extensão final | 72 °C | 2 min |
| Segurar | 4 °C |
Tabela 4: Condições de ciclagem para a1ª etapa de PCR para amplificação dos genes POI da minipreparação de levedura isolada.
| Volume [μL] | Concentração final | |
| H2O livre de nuclease | até 50 | |
| 10x tampão Thermopol | 5 | 1x |
| Primer_fwd (10 μM) | 2.5 | 0,5 μM |
| Primer_rev (10 μM) | 2.5 | 0,5 μM |
| dNTPs (10 mM) | 1 | 200 μM |
| 8-oxo-dGTP (100 μM) | 1 | 2 μM |
| dPTP (100 μM) | 1 | 2 μM |
| Produto PCR da 1ª PCR | XX | 50 ng |
| Taq DNA polimerase | 0.5 | 0,05 U/μL |
Tabela 5: Condições para a epPCR que é realizada após a amplificação do DNA POI com a PCR da1ª etapa.
| Passo | Temperatura | Hora |
| Desnaturação inicial | 94 °C | 30 s |
| 15 ciclos | 94 °C | 45 s |
| 60 °C | 30 s | |
| 72 °C | 1 minuto | |
| Extensão final | 72 °C | 10 minutos |
| Segurar | 4 °C |
Tabela 6: Condições de ciclagem para a epPCR.
| Volume [μL] | Concentração final | |
| 5x aprimorador Q5 | 20 | 1x |
| 5x buffer Q5 | 20 | 1x |
| Primer fwd 10 μM | 5 | 0,5 μM |
| Primer rev 10 μM | 5 | 0,5 μM |
| dNTPs 10 mM | 1 | 200 μM |
| Polimerase Q5 | 1 | 20 U/mL |
| 50 ng de DNA | XX | |
| ddH20 | até 100 |
Tabela 7: Condições para a2ª etapa de PCR para amplificação do produto de epPCR antes da eletroporação de células EBY100.
| Passo | Temperatura | Hora |
| Desnaturação inicial | 98 °C | 30 s |
| 25 ciclos | 98 °C | 10 s |
| 72 °C | 30 s | |
| 72 °C | 30 s | |
| Extensão final | 72 °C | 2 min |
| Segurar | 4 °C |
Tabela 8: Condições de ciclagem para a PCR da2ª etapa para amplificação do produto epPCR.
8. Linearização do vetor de exibição de levedura para eletroporação
- Linearize o vetor de exibição da superfície da levedura pCTCON2 (plasmídeo Addgene # 41843).
- Execute uma digestão em larga escala e use este vetor linearizado digerido para todas as eletroporações posteriores.
- Digerir 2 x 200 μg de DNA do plasmídeo pCTCON2 com Sal I por 24 h a 37 ° C para uma linearização inicial (Tabela 9).
- Digerir com NheI e BamHI durante 24 h a 37 °C para extirpar a bula (quadro 10).
- Por fim, adicionar 5 μL de cada enzima ao tubo (Sal I, NheI, BamHI) e incubar novamente durante 24 h a 37 °C.
- Execute o vetor digerido em um gel preparativo de agarose. Extirpar as bandas e purificar com um kit de extração de gel. Eluir o vetor em 20 μL, medir a concentração de DNA e ajustá-la para aproximadamente 2 μg/μL.
NOTA: Digerir o plasmídeo por um tempo prolongado garante clivagem suficiente da inserção e resultará em menos fundo do vetor após a eletroporação.
| ADN | 200 μg |
| 10x CutSmartBuffer | 50 μL |
| Sal I-HF (NEB) | 30 μL (60 U) |
| H2O | Até 500 μL |
Tabela 9: Condições para a primeira etapa da digestão em larga escala do vetor de exibição da superfície da levedura pCTCON2.
| pCTCON2 (Sal que digeri) | 500 μL |
| 10x CutSmartBuffer | 37,5 μL |
| NheI-HF (NEB) | 15 μL (30 U) |
| BamHI-HF (NEB) | 15 μL (30 U) |
| H2O | até 875 μL |
Tabela 10: Condições para a segunda etapa da digestão em larga escala do vetor de exibição da superfície da levedura pCTCON2.
9. Eletroporação de EBY100 com DNA randomizado e vetor linearizado
- Prepare as células de levedura EBY100 (ATCC, consulte a Tabela de Materiais) retirando-as de um estoque de glicerol 3 dias antes da eletroporação. Como as células EBY100 não transformadas não contêm o vetor de exibição da superfície da levedura, use placas YPD de mídia completa (Tabela 1). Incubar a 30 °C.
- Um dia antes da eletroporação, inocule uma única colônia EBY100 em 30 mL de meio YPD recém-preparado (Tabela 1). Incubar durante a noite a 30 °C agitando a 180 rpm.
NOTA: O meio YPD não deve ter mais de 1 mês. - Na manhã seguinte, diluir a cultura EBY100 durante a noite para uma DO600 de 0,2 em 100 mL de meio YPD e incubar a 30 ° C enquanto agita a 180 rpm.
NOTA: Um volume de cultura de 50 mL é suficiente para 2 eletroporações. Normalmente, 100 mL são preparados para ter células suficientes para os controles (apenas células e células mais vetor). - Quando um OD600 de 1,3-1,5 for atingido, pulverize as células em tubos de 50 mL a 2000 x g por 3 min e remova o sobrenadante.
- Ressuspenda o pellet em 25 mL de acetato de lítio filtrado estéril 100 mM (consulte a Tabela de Materiais). Adicionar TDT filtrada estéril recém-preparada (1 M) (ver Tabela de Materiais) até uma concentração final de 10 mM.
NOTA: O volume de acetato de lítio deve ser metade do volume original de YPD. Reduza ou aumente a escala vertical, se necessário. O acetato de lítio (100 mM) pode ser preparado com antecedência e armazenado por alguns meses em RT, mas o estoque de 1 M TDT deve ser preparado logo antes do uso e mantido em gelo. - Incubar as células a 30 °C agitando durante 10 min.
- Centrifugue a 2000 x g por 3 min, descarte o sobrenadante e coloque as células no gelo.
NOTA: Todas as etapas a seguir devem ser executadas no gelo e com reagentes, cubetas e tubos de microcentrífuga resfriados. - Ressuspenda o pellet em 25 mL de H2O. Estéril frio Centrifugue a 2000 x g por 3 min.
NOTA: O volume H2O usado deve ser metade do volume YPD original. - Ressuspender em 350 μL de H2O estéril.
NOTA: O volume total após a ressuspensão deve ser de 500 μL. Adicione mais H2O, se necessário. - Prepare a biblioteca de DNA em paralelo: Coloque os tubos de microcentrífuga contendo as inserções purificadas com etanol no gelo e adicione 4 μg de vetor pCTCON2 linearizado.
- Adicione 250 μL das células ao DNA e misture suavemente cortando. Transferir para uma cubeta de eletroporação de 2 mm pré-refrigerada e manter no gelo até à eletroporação.
NOTA: Adicione controles (somente células sem DNA e células com vetor linearizado apenas sem inserção) para determinar o sucesso da eletroporação. - Eletroporar usando cubetas de eletroporação de 2 mm (ver Tabela de Materiais) com as seguintes condições: protocolo de onda quadrada, pulso único, 500 V, duração do pulso de 15 ms.
NOTA: As quedas típicas são de 3% a 9%. Outros eletroporadores podem ser utilizados, mas as condições indicadas são otimizadas para o instrumento utilizado neste estudo. - Resgate as células com 1 mL de YPD pré-aquecido, transfira para um tubo de microcentrífuga pré-aquecido e incube sem agitar por 1 h a 30 ° C.
NOTA: Coloque o bloco de aquecimento próximo ao eletroporador para garantir que os tubos de recuperação permaneçam na temperatura adequada. - Placa de 100 μL de diluições de 10-3, 10-4, 10-5 e 10-6 de células em placas SD-CAA (ver Tabela 1) e permitir o crescimento de transformantes a 30 ° C.
NOTA: A diversidade da biblioteca teórica é obtida calculando o número de transformantes para o volume de cultura original, aqui 1 mL. Normalmente, esse protocolo de transformação produz aproximadamente 10células transformadas 7-10 8 . - Pulverize as células restantes a 2000 x g por 3 min, ressuspenda em 200 mL SD-CAA e incube a 30 ° C com agitação a 180 rpm durante a noite. As células transformadas contendo a biblioteca aleatória de epPCR podem ser passadas em SD-CAA e induzidas para expressão de superfície em SG-CAA no dia seguinte ou armazenadas a 4 ° C até uso posterior. Congelar os estoques de glicerol da biblioteca após epPCR após determinar a diversidade. Como no dia seguinte uma fração de células ainda será células não transformadas que foram transportadas no procedimento de transformação sem obter um plasmídeo, recomenda-se passar pelo menos 20 vezes a diversidade da biblioteca na primeira etapa de passagem para compensar esse efeito.
10. Sequenciamento de bibliotecas de leveduras após várias rodadas de seleção
- Extraia o DNA da levedura realizando uma minipreparação da levedura conforme descrito acima.
- Células eletroporadas comerciais de E. coli 10-beta eletrocompetentes (ver Tabela de Materiais) com o DNA isolado.
- Coloque as cubetas de eletroporação (1 mm, consulte a Tabela de Materiais) e os tubos de microcentrífuga no gelo. Pré-aqueça os tubos de microcentrífuga com 950 μL de meio de crescimento a 37 °C para recuperar as células após a eletroporação.
- Descongele as células no gelo e misture sacudindo o tubo suavemente. Transfira 50 μL das células para um tubo de microcentrífuga resfriado. Adicione 1 μL do DNA da minipreparação de levedura.
- Transfira cuidadosamente a mistura célula-DNA para uma cubeta de eletroporação resfriada.
NOTA: Evite bolhas e certifique-se de que as células se depositem no fundo da cubeta. - Eletroporar com um dispositivo de eletroporação usando as seguintes condições: 2,0 kV; 200 Ω; 25 μF.
NOTA: As constantes de tempo típicas são de 4,8 a 5,1 ms. Outros eletroporadores podem ser utilizados, mas as condições indicadas são otimizadas para o instrumento utilizado neste estudo. - Adicione imediatamente 950 μL de meio de crescimento pré-aquecido a 37 °C (consulte a Tabela de Materiais) às cubetas, misture delicadamente e transfira de volta para os tubos de microcentrífuga quentes.
NOTA: Coloque o bloco de aquecimento próximo ao eletroporador para garantir que os tubos de recuperação permaneçam na temperatura adequada. - Incubar durante 40 min a 37 °C e 300 rpm. Coloque 10 μL, 100 μL e o resto das células (após centrifugação) em uma placa LB seletiva pré-aquecida (ver Tabela 1). Incubar as placas durante a noite a 37 °C.
NOTA: Para o vetor pCTCON2, a ampicilina é usada para a seleção de antibióticos. - Prepare uma placa de cópia com ágar LB e o antibiótico necessário (aqui a ampicilina é usada): pipete 200 μL de ágar LB com 100 μg / mL de ampicilina em cada alvéolo de uma placa de 96 poços e deixe solidificar.
- Prepare uma placa de sequenciamento de 96 poços com 50 μL de LB com 100 μg / mL de ampicilina por poço.
- Escolha uma única colônia das placas e primeiro inocule a placa de cópia e o mesmo poço da placa de sequenciamento.
NOTA: A placa de cópia garante que os plasmídeos desejados possam ser facilmente obtidos inoculando uma cultura líquida e realizando uma minipreparação. - Incubar a placa de cópia durante a noite a 37 °C e conservar em seguida a 4 °C.
- Incubar a placa sequencial durante 3-4 h a 37 °C agitando (180 rpm) coberta com uma folha de plástico ou alumínio.
- Sele a placa de sequenciamento e envie-a para sequenciamento junto com 500 μL de primer de sequenciamento pCTCON2 10 μM (5'-CGTTTGTCAGTAATTGCGGTTCTC).
- Para análise das sequências, traduza as sequências de DNA obtidas em sequências de aminoácidos usando um programa adequado de sua escolha, por exemplo, EMBOSS Transeq38. Em seguida, use o Alinhamento de Sequência Múltipla (MUSCLE)39 ou uma ferramenta de alinhamento semelhante para alinhar as sequências variantes entre si e com a proteína do tipo selvagem parental.
NOTA: Ao realizar o sequenciamento de bibliotecas de diferentes rodadas de seleção (por exemplo, após uma rodada anterior de seleção, bem como após rodadas adicionais de seleção com concentrações de antígenos mais baixas para enriquecer variantes de alta afinidade), é possível identificar diferentes variantes. Isso depende muito do rigor dos portões aplicados durante as rodadas de classificação e, portanto, das diversidades das bibliotecas.
Resultados
A biblioteca G4 foi selecionada contra o antígeno hRBP4 ligado ao medicamento de molécula pequena A1120. A coloração das bibliotecas para classificação por citometria de fluxo foi realizada conforme descrito no Método 6, e a estratégia de gating aplicada é mostrada na Figura 2A. Um portão inicial incluía todas as células com base na morfologia celular, e o segundo portão (histograma de FSC-Width) mostrou uma estratégia de gating rigorosa que foi aplicada para selecionar células individuais e remover agregados de células. A terceira e última porta mostrou a exibição de variantes proteicas (eixo x) versus ligação ao antígeno (eixo y). As células de levedura que mostram sinais de exibição e ligação foram classificadas. É importante ressaltar que a porta de classificação foi configurada de maneira rigorosa para enriquecer os domínios de ligação com alto sinal de ligação e, portanto, alta afinidade. Essa seleção rigorosa produziu um enriquecimento da exibição de células de levedura que se ligam especificamente ao antígeno alvo durante a campanha de seleção (Figura 2B). Em rodadas posteriores de classificação por citometria de fluxo, a concentração de antígeno diminuiu 10 vezes (de 100 nM para 10 nM). Portanto, o sinal geral de ligação foi reduzido e apenas os ligantes com alta afinidade ainda eram detectáveis e classificados ( Figura 2C ).
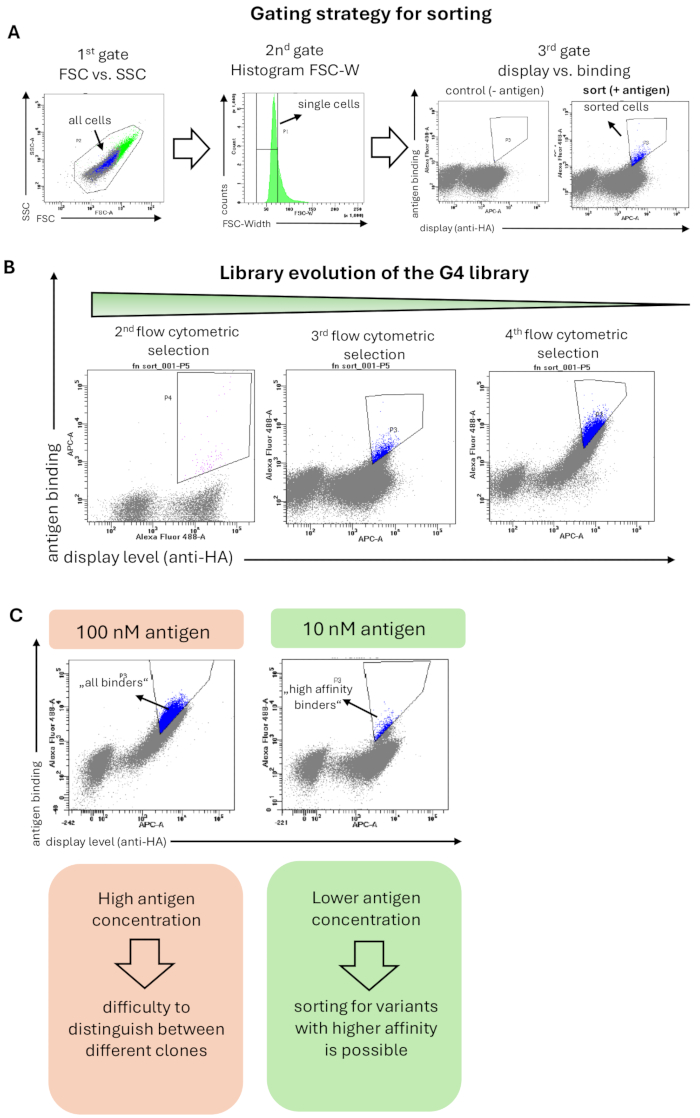
Figura 2: Resultados representativos de uma seleção de exibição de superfície de levedura da biblioteca G4 baseada em Fn3 para ligação ao antígeno (hRBP4 na presença de A1120). (A) A estratégia geral de gating para classificação de bibliotecas de leveduras. A primeira porta (FSC vs. SSC) é selecionar todas as células de levedura e excluir eventos de dispersão; a segunda porta (histograma do FSC-W) visa remover agregados celulares e selecionar apenas células de levedura individuais. A terceira porta traça o nível de exibição da superfície (detecção da marca HA- ou c-myc) vs. ligação ao antígeno (aqui hRBP4 na presença de 5 μM A1120, detectado pelo anticorpo anti-His). A biblioteca foi adicionalmente corada apenas com anticorpos secundários (sem antígeno), onde nenhuma ligação ao antígeno é esperada. As células classificadas são destacadas em azul. (B) Evolução da biblioteca G4 ao longo de 3 rodadas de classificação por citometria de fluxo. O enriquecimento da população de ligação pode ser observado a cada rodada de seleção. (C) O uso de concentrações mais baixas de antígeno permite a seleção de variantes proteicas com maior afinidade pelo antígeno alvo. Após a redução da concentração de antígeno (aqui hRBP4) em 10 vezes, diferentes diagonais aparecem, indicando a presença de clones com maior (células classificadas, azul) ou menor afinidade. Clique aqui para ver uma versão maior desta figura.
Discussão
A exibição da superfície da levedura evoluiu como um dos principais métodos usados na engenharia de proteínas. Embora seja comumente empregado para a engenharia de afinidade 1,18,40,41, expressão / estabilidade 24,27,42,43 e atividade 28,44, outros usos como mapeamento de epítopos45,46 ou caracterização dos mutantes individuais na superfície de células de levedura9 também são possíveis. Neste protocolo, fornecemos as etapas básicas para iniciar uma campanha de seleção de exibição de superfície de levedura, incluindo a seleção com esferas magnéticas e por classificação por citometria de fluxo, bem como a diversificação da biblioteca de leveduras por epPCR para maturação de afinidade.
Um requisito essencial para as seleções convencionais de exibição de superfície de levedura é a disponibilidade de proteína solúvel de qualidade suficiente. Começar com uma proteína-alvo bem dobrada com alta pureza e um estado de oligomerização definido (ou seja, a proteína monomérica deve estar presente apenas como monômero) fornece a maior taxa de sucesso para selecionar uma variante de proteína que se liga ao antígeno alvo com alta afinidade. Uma alternativa para proteínas-alvo difíceis de expressar são as seleções baseadas em células, que apresentam uma estratégia razoável para contornar essa limitação47. No entanto, a exibição da superfície da levedura oferece muitas vantagens, como a possibilidade de caracterizar variantes proteicas resultantes diretamente na superfície da levedura sem a necessidade de realizar clonagem trabalhosa e demorada, expressão em formato solúvel e purificação de proteínas. Tanto a afinidade quanto a estabilidade das variantes podem ser analisadas diretamente na superfície da levedura9.
Neste protocolo, mostramos como a biblioteca G4 de variantes proteicas, mais especificamente do10º domínio tipo III da fibronectina humana, foi selecionada para ligação ao antígeno hRBP4 na presença da pequena molécula A1120. A combinação de seleções de grânulos e classificação por citometria de fluxo produziu um enriquecimento de variantes, que mostraram uma ligação aumentada ao antígeno alvo ao longo das rodadas de seleção ( Figura 2B ). Mostramos que o uso de concentrações mais baixas de antígeno permite a seleção de variantes proteicas de alta afinidade (Figura 2C). Normalmente, as afinidades que podem ser alcançadas com as seleções de exibição de levedura estão na faixa nanomolar ou mesmo picomolar18. As afinidades finais dependem do antígeno alvo, do número de rodadas de seleção e maturação de afinidade, do andaime de ligação usado e da estratégia de gating aplicada. A caracterização de variantes proteicas individuais não é abordada neste protocolo, mas é explicada em detalhes em nosso trabalho anterior9. Embora a exibição de leveduras tenha sido originalmente empregada para a engenharia de fragmentos de anticorpos, como scFvs 1,40, o método também tem sido amplamente utilizado para proteínas não baseadas em anticorpos10.
Resumindo, a exibição da superfície da levedura é uma poderosa ferramenta de engenharia de proteínas que permite a geração de variantes de proteínas com propriedades novas ou aprimoradas, como ligação a quase qualquer proteína-alvo e/ou maior estabilidade.
Divulgações
M.W.T. recebe financiamento da Miltenyi Biotec. Todos os autores são inventores em pedidos de patentes para tecnologias e proteínas projetadas que foram desenvolvidas usando exibição de superfície de levedura.
Agradecimentos
Este trabalho foi apoiado pelo Fundo Austríaco de Ciência (Projeto FWF W1224 - Programa de Doutorado em Tecnologia Biomolecular de Proteínas - BioToP e Projeto FWF ESP 465-B), o Ministério Federal de Assuntos Digitais e Econômicos da Áustria, a Fundação Nacional de Pesquisa, Tecnologia e Desenvolvimento da Áustria para a Associação Cristã de Pesquisa Doppler (Laboratório Doppler Cristão para Células T CAR de Próxima Geração), e por doações privadas ao St. Anna Children's Cancer Research Institute (Viena, Áustria). E.S. recebeu uma bolsa DOC da Academia Austríaca de Ciências no St. Anna Children's Cancer Research Institute. As figuras foram criadas com BioRender.com.
Materiais
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
Referências
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados