Method Article
Visualización in vivo de transitorios de calcio durante la fertilización y el desarrollo temprano en C. elegans
En este artículo
Resumen
Aquí, presentamos protocolos y herramientas para visualizar el calcio durante la fertilización y la embriogénesis temprana utilizando un reportero de calcio codificado genéticamente expresado en la línea germinal del nematodo modelo C. elegans.
Resumen
El calcio es una importante molécula de señalización durante la transición del ovocito al embrión (OET) y la embriogénesis temprana. El nematodo hermafrodita Caenorhabditis elegans proporciona varias ventajas únicas para el estudio de la OET, ya que es transparente y tiene una gónada ordenada que produce un ovocito maduro cada ~23 min a 20 °C. Hemos modificado el indicador de calcio codificado genéticamente jGCaMP7s para que indique fluorescentemente el momento de la fecundación dentro de un organismo vivo. Hemos denominado a este reportero "CaFE" para el Calcio durante la Fertilización en C. elegans. El reportero CaFE fue diseñado en un locus de puerto seguro en una sola copia, no tiene un impacto significativo en la fisiología o la fecundidad, y produce una señal robusta tras la fertilización. Aquí, se presentan una serie de protocolos para utilizar el reportero CaFE como herramienta in vivo para diseccionar el OET y la embriogénesis. Incluimos métodos para sincronizar gusanos, examinar los efectos de la eliminación de ARNi, montar gusanos para imágenes y visualizar el calcio en ovocitos y embriones. Además, presentamos la generación de cepas de gusanos adicionales para ayudar en este tipo de análisis. Demostrando la utilidad del reportero CaFE para visualizar el momento de la fertilización, informamos que la doble ovulación ocurre cuando ipp-5 es el objetivo del ARNi y que solo el primer ovocito se somete a la fertilización inmediata. Además, aquí se informa del descubrimiento de transitorios de calcio unicelulares durante la embriogénesis temprana, lo que demuestra que el reportero de CaFE persiste en el desarrollo temprano. Es importante destacar que el reportero de CaFE en gusanos es lo suficientemente simple como para usarlo para incorporarlo a las clases de laboratorio de investigación de pregrado (CURE) basadas en cursos. El reportero de CaFE, junto con la gónada ordenada y la facilidad de ARNi en los gusanos, facilita la investigación de la dinámica célula-célula necesaria para regular la fertilización interna y la embriogénesis temprana.
Introducción
La fertilización marca el comienzo de un nuevo ciclo de vida, pero definir el momento preciso de la fertilización es un desafío. Una característica conservada de la fertilización es una onda de calcio a través del ovocito inmediatamente después de la fusión de espermatozoides. Aunque la naturaleza de la onda de calcio, en términos de frecuencia y velocidad, difiere entre especies, casi todos los organismos exhiben un aumento transitorio de calcio intracelular después de la fertilización. La onda de calcio desempeña un papel fundamental en el bloqueo de la polispermia, la activación de los óvulos y otros eventos celulares importantes2. Dado que la onda de calcio se inicia en el sitio de fusión de los espermatozoides, el calcio sirve como marcador para la fertilización3.
Caenorhabditis elegans es un organismo modelo ideal para estudiar el desarrollo temprano. Los gusanos son hermafroditas transparentes que son genéticamente tratables4. Lo más importante para el estudio de la fertilización es que los adultos de C. elegans producen continuamente ovocitos utilizando una gónada estrictamente ordenada5. De hecho, los ovocitos son las únicas células nuevas producidas, ya que el soma es postmitótico en la edad adulta6. En la figura 1 se destaca la gónada, que consta de 2 tubos simétricos en forma de U de ovocitos en desarrollo. El ovocito más cercano a la espermateca (órgano de almacenamiento de espermatozoides) se denomina ovocito -1. Este diseño de línea de montaje de la gónada del gusano ovula un ovocito maduro en la espermateca cada ~23 min a 20 °C en adultos jóvenes7. Luego, el embrión recién fertilizado se mueve a un útero compartido antes de ser depositado a través de una sola vulva.
Las técnicas previas para visualizar el calcio durante la fertilización en C. elegans se basaban en la microinyección de colorantes sensibles al calcio 3,8. Para ver más fácilmente la onda de calcio en la fusión espermatozoide-óvulo, se insertó un indicador de calcio codificado genéticamente basado en jGCaMP7s en una copia única en un locus de puerto seguro utilizando repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR)7,9. El reportero se denominó CaFE para el calcio durante la fertilización en C. elegans. El reportero no muestra defectos significativos en fisiología o fecundidad.
En este trabajo se presentan los protocolos para la visualización de la onda de calcio en los ovocitos y embriones de C. elegans utilizando el reportero CaFE. En combinación con la miríada de herramientas disponibles en la comunidad de gusanos, como el ARN de interferencia (ARNi) y los marcadores de gónadas mCherry, el informe CaFE facilita la investigación sobre la regulación de los eventos internos de fertilización, en particular la competencia de fertilización y el momento de la fertilización. Además, el reportero CaFE persiste en el desarrollo temprano y es una herramienta única para sondear la embriogénesis.
Protocolo
1. Mantenimiento de C. elegans
- Preparación de la placa del medio de crecimiento de nematodos (NGM)
- Combine 3 g de cloruro de sodio (NaCl), 2,5 g de bacto-peptona, 20 g de agar y 1 l de agua desionizada en un matraz de 4 L (consulte la tabla de materiales). Cubra la parte superior del matraz con papel de aluminio antes de esterilizarlo en autoclave.
- Autoclave para esterilizar utilizando los ajustes del ciclo de esterilización líquida. Se recomienda un ciclo de esterilización ajustado a 121 °C durante 30 minutos.
- Deje que la mezcla se enfríe a ~50 °C, aproximadamente 20 minutos a temperatura ambiente (RT).
- Añadir 25 mL de 1 M KH2PO4 estéril estéril (disuelto en agua, pH = 6), 1 mL de CaCl2 1 M estéril (disuelto en agua), 1 mL de 1 M MgSO4 estéril (disuelto en agua) y 1 mL de colesterol 5 mg/mL (disuelto en etanol al 100%), agitando suavemente después de cada adición (ver Tabla de Materiales).
- Utilizando una técnica estéril, transfiera una porción de la mezcla a un vaso de precipitados estéril de 300 ml para facilitar el vertido. Vierta el medio tibio para cubrir el fondo de la placa.
NOTA: Aproximadamente 10 mL por placa en placas de Petri estériles de 60 mm x 15 mm (consulte la Tabla de materiales) producirán 100 placas. Alternativamente, se pueden usar placas de Petri estériles de 35 mm x 10 mm, donde ~4 mL por placa producirán ~250 placas. - Deje que las placas NGM se sequen en RT durante al menos 1 día antes de sembrar con bacterias. Guarde las placas boca abajo a 4 °C para su uso futuro.
- Preparación de césped bacteriano OP50
- Prepare el caldo Luria (LB) combinando 5 g de extracto de levadura, 10 g de Bacto-triptona, 5 g de NaCl y 1 L de agua desionizada en un matraz de 2 L (ver Tabla de Materiales). Con una placa para revolver y una barra para revolver, mezcle hasta que se disuelva.
- Alícuota LB en biberones y autoclave utilizando los ajustes del ciclo de esterilización de líquidos. Se recomienda un ciclo de esterilización ajustado a 121 °C durante 30 minutos. Si usa un recipiente con tapón de rosca, afloje el tapón de rosca un cuarto de vuelta para una ventilación adecuada durante el ciclo del autoclave.
- Deje que el caldo se enfríe a RT antes de usarlo.
- Utilizando una técnica estéril, inocular 50 mL de LB en un matraz Erlenmeyer de 250 mL con la bacteria OP50 y crecer durante la noche a 37 °C con agitación.
- Utilizando una técnica estéril, transfiera 300 μL de cultivo saturado al centro de una placa NGM de 60 mm x 15 mm. Para placas de 35 mm x 10 mm, se recomiendan 100 μL de cultivo saturado.
- Deje que las placas se sequen a RT durante 1-3 días hasta que la mancha bacteriana OP50 ya no sea líquida. Asegúrese de que las placas estén secas y libres de contaminación antes de usarlas. Guarde las placas a 4 °C para su uso futuro. Almacenar las placas boca abajo reduce la condensación en la superficie de la placa.
- Construcción de pico de tornillo sin fin
- Afloje el collar plateado hacia la cabeza del mango del sinfín (consulte la Tabla de materiales) desenroscándolo suavemente para que las cuatro secciones se separen.
- Corte 1.5 pulgadas (aproximadamente la longitud del pulgar) de alambre de platino (consulte la Tabla de materiales) con unas tijeras y colóquelo en el medio de las cuatro secciones; Aproximadamente una pulgada sobresaldrá del mango.
- Sostenga el cable en el centro mientras aprieta el collar; Las cuatro secciones se unirán en el medio y mantendrán el alambre de platino en su lugar.
- Dale forma cuidadosamente al alambre con un par de alicates de punta plana. Una forma de arco iris o arco tiende a funcionar mejor para la recolección.
- Cree un "pie" de aproximadamente 2-3 mm en el extremo del alambre sujetándolo repetidamente con los alicates de punta plana. Use este "pie" para recoger los gusanos y mantenga el "pie" plano a la superficie del plato.
NOTA: Si se sujeta con demasiada fuerza, se cortará el alambre de platino. Evite crear bordes afilados con los alicates de punta plana (o equivalentes) para evitar perforar los gusanos o las placas.
- Manipulación de gusanos
- Sostenga el pico de gusano similar a un lápiz y esterilice el "pie" colocando el cable en la llama de una lámpara de etanol al 95% hasta que esté de color naranja brillante.
- Coloque una placa con semillas OP50 en la platina de un telescopio de disección y retire la tapa. Toque suavemente el "pie" de la púa en un área del césped bacteriano y use un movimiento suave de deslizamiento para recolectar las bacterias en la parte inferior. Las bacterias que están pegadas al pico (conocidas como "pegajosas") actuarán como un pegamento para recoger los gusanos.
- Mueva una placa de gusanos a la etapa del visor de disección y retire la tapa mientras evita que el pico toque otras superficies. Toque rápida y suavemente el "pie" del pico sobre un gusano para recogerlo. Esta técnica permite que varios gusanos se muevan entre placas simultáneamente.
- Toque suavemente el "pie" del pico con la parte sin semillas de un plato nuevo para transferir los gusanos; Normalmente, se trata de la misma placa de la que se extrajeron las bacterias en la sección 1.4.2.
- Esterilice el palillo entre cada placa y/o cepa de gusano para evitar la contaminación y permitir que el palillo se enfríe después de la esterilización.
NOTA: Tocar un gusano con un pico caliente estresará o matará al animal. El recogedor de lombrices se enfriará después de tocar el césped bacteriano para recoger la "sustancia viscosa" para la transferencia de lombrices.
- Mantener los gusanos a 20 °C en placas NGM (paso 1.1) sembradas con la bacteria OP50 Escherichia coli (paso 1.2). Pase las lombrices de 2 a 3 veces por semana transfiriendo varios adultos a platos frescos (60 mm x 15 mm) con un palillo de lombrices (pasos 1.3-1.4) para evitar la inanición, que puede alterar la fisiología.
NOTA: Las cepas de C. elegans y la bacteria OP50 están disponibles a través del Centro de Genética de Caenorhabditis (CGC).
2. Sincronización del desarrollo de C. elegans
- Sincronice la edad de los gusanos utilizando una puesta de huevos cronometrada (descrita aquí). Si se necesita una gran cantidad de gusanos para un aula u otro propósito, realice la sincronización de lejía como se describe en Golden et al.10.
- Transfiera los gusanos adultos a una nueva placa OP50 sembrada utilizando un pico de alambre de platino (descrito en la sección 1). El número de gusanos está dictado por la necesidad experimental. Los gusanos del día 1 ponen aproximadamente 3 huevos por hora. Por lo tanto, 10 lombrices generarán aproximadamente 30 huevos en 1 h. Preferiblemente, use lombrices el primer día de la edad adulta (Día 1).
- Después de aproximadamente 1 h, retire los gusanos adultos del plato, dejando solo los huevos.
NOTA: Dejar un gusano inadvertidamente es un error común. Los gusanos que permanezcan continuarán poniendo huevos e interrumpirán la sincronización. Los gusanos maduran hasta convertirse en adultos del día 1 aproximadamente 72 horas después de ser puestos cuando se incuban a 20 ° C.
3. Montaje de gusanos para la obtención de imágenes
- Al obtener imágenes de los eventos de fertilización, use adultos del día 1 para maximizar las oportunidades de visualización.
NOTA: En los adultos del día 1, se fertiliza un ovocito cada ~ 23 min. Sin embargo, la ovulación se ralentiza con la edad, y un hermafrodita típico dejará de poner huevos después del día 4. - Preparar una solución de agarosa al 3% en agua mezclando 3 g de agarosa en 100 mL de agua. Caliente la solución de agarosa en un microondas hasta que la agarosa se disuelva por completo. Para reutilizar una solución solidificada, caliente hasta que se disuelva por completo. Después de varios usos, genere una solución nueva.
- Coloque un portaobjetos de microscopio en blanco para la obtención de imágenes entre otros dos portaobjetos de microscopio que tengan una sola capa de cinta de laboratorio a lo largo del eje largo del portaobjetos (consulte la Tabla de materiales). La cinta crea un espaciador para la almohadilla de agarosa, lo que permite una distribución uniforme y un ancho uniforme. Asegúrese de que los tres portaobjetos estén en fila con los lados largos tocándose: dos portaobjetos con cinta adhesiva que flanquean un portaobjetos de microscopio en blanco.
- Deje caer la solución de agarosa calentada al 3% (~100-150 μL) en el centro del portaobjetos del microscopio que se encuentra entre los portaobjetos pegados con cinta adhesiva con una pipeta de 1 mL.
- Coloque rápidamente un nuevo portaobjetos de microscopio sobre la gota de agarosa, asegurándose de que el nuevo portaobjetos esté perpendicular al portaobjetos con la gota de agarosa y que descanse sobre los portaobjetos adyacentes pegados con cinta adhesiva.
- Separe suavemente los portaobjetos. Evite rasgar la almohadilla.
- Prepare las almohadillas de agarosa frescas cada día de imagen. Se recomienda hacer varias almohadillas cuando se prepare para la toma de imágenes, pero absténgase de exponer más de una almohadilla al mismo tiempo. Las almohadillas expuestas se secarán rápidamente.
- Con una pipeta de 20 μL, agregue una pequeña gota (~7 μL) de 1 mM de levamisol en tampón M9 al centro de la almohadilla de agarosa para paralizar los gusanos. Utilice una solución madre de 100 mM de levamisol esterilizada por filtro para hacer la solución de trabajo de 1 mM.
- Para hacer tampón M9, combine 6 g de Na2HPO4, 3 g de KH2PO4, 0,5 g de NaCl, 1 g de NH4Cl y 1 L de agua desionizada en un recipiente adecuado (consulte la tabla de materiales).
- Esterilizar la solución en autoclave utilizando los ajustes del ciclo de esterilización líquida y dejar que la solución se enfríe hasta RT antes de usarla. Si usa un recipiente con tapón de rosca, afloje el tapón de rosca aproximadamente un cuarto de vuelta para una ventilación adecuada. Se recomienda un ciclo de esterilización ajustado a 121 °C durante 30 minutos.
- Elija al menos 5 lombrices sincronizadas y toque suavemente la gota de levamisol con el "pie" para transferir todas las lombrices. Use al menos 5 gusanos por portaobjetos, ya que la orientación del cuerpo puede hacer que el intestino oscurezca la línea germinal, bloqueando la visualización de la onda de calcio en el ovocito.
NOTA: Asegúrese de que los gusanos se transfieran rápidamente, ya que el levamisol se evaporará si se deja reposando en la almohadilla de agarosa durante más de 5 minutos. - Cubra con cuidado la almohadilla de agarosa con un cubreobjetos. Verifique la ubicación de los gusanos, ya que el cubreobjetos puede causar la dispersión de los gusanos desde la ubicación inicial. Etiquete el portaobjetos del microscopio con los detalles apropiados, como el nombre de la cepa de gusano (consulte la Tabla de materiales).
NOTA: Asegúrese de que las imágenes se realicen lo antes posible después de montar los gusanos, ya que la exposición prolongada al levamisol es tóxica.
4. Imágenes del calcio durante la fertilización en C. elegans
- Localice los gusanos utilizando un objetivo de baja potencia, como 4x, en el microscopio de su elección. Utilice iluminación de campo claro.
- Utilice un mínimo de un objetivo de aire de 20x para visualizar claramente la onda de calcio en los ovocitos. Utilice un objetivo de inmersión de 40x o más.
- Dependiendo del sistema que se utilice, la intensidad de excitación suele ser un valor fijo. Para una visualización óptima de los reporteros fluorescentes, ajuste la ganancia o el tiempo de exposición.
NOTA: La luz continua de alta intensidad dañará la muestra y es perjudicial para la salud de los gusanos. Si lo desea, apague el láser mientras espera un evento de fertilización para evitar la iluminación GFP continua. Véanse las características distintivas de un evento de fertilización en las secciones 4.8 - 4.9. - Asegúrese de que el canal GFP de 488 nm esté configurado a una potencia láser en la que la señal reportera de CaFE sea fácilmente visible.
- Para un confocal de escaneo láser Nikon ECLIPSE Ti2, detecte el reportero CaFE a una potencia láser del 30% para el canal GFP y una ganancia de 75. El tamaño del orificio para el confocal de escaneo láser Nikon ECLIPSE Ti2 es de 30 μm.
- Para el confocal de disco giratorio Andor Dragonfly utilizando la cámara Zyla, detecte el reportero de CaFE a una potencia láser del 30% para el canal GFP. El tamaño del agujero de alfiler para el confocal del disco giratorio Andor Dragonfly es de 40 μm.
- Ajuste la configuración del láser y el tiempo de exposición para cada muestra examinada.
- Si se utiliza el EAG28 (CaFE; PH-mCherry) o EAG25 (CaFE; H2B-mCherry), asegúrese de que el canal rojo esté configurado a una potencia láser en la que la señal mCherry sea fácilmente visible además del canal GFP.
- Para el confocal de escaneo láser ECLIPSE Ti2 de Nikon, detecte el reportero mCherry a una potencia láser de 15 para el canal RFP y una ganancia de 130.
- Para el confocal de disco giratorio Andor Dragonfly utilizando la cámara Zyla, detecte el reportero mCherry a una potencia láser del 30% para el canal RFP.
- Ajuste la configuración del láser y el tiempo de exposición para cada muestra examinada.
- Establezca la velocidad de fotogramas o fotogramas por segundo (fps) tan rápido como lo permita el sistema.
NOTA: El reportero CaFE se observa normalmente a aproximadamente 1 fps para el confocal de escaneo láser Nikon ECLIPSE Ti2 y a 10 fps para el confocal de disco giratorio Andor Dragonfly con la cámara Zyla. - Seleccione el protocolo de lapso de tiempo o serie de tiempo y ajústelo a la configuración deseada. Adquiera imágenes de lapso de tiempo durante 30 minutos o menos, con la opción de repeticiones establecida en '1'.
NOTA: Si el evento de fertilización ocurre antes de que se complete el intervalo de tiempo, detenga el video para preservar el tamaño del archivo. - Ubique los gusanos en el portaobjetos y seleccione un gusano donde la gónada proximal sea visible. El intestino y la gónada se retuercen uno alrededor del otro dentro del gusano. Como tal, un brazo gonadal a menudo está oscurecido por el intestino, lo que impide la visualización de la fertilización. Si el ovocito -1 está oscurecido, seleccione otro gusano para su visualización. Para aumentar la probabilidad de que el gusano seleccionado tenga un brazo gonadal visible, monte un mínimo de 5 gusanos por portaobjetos. Utilice los adultos tempranos del día 1 para visualizar la fertilización.
- Examine cada ovocito -1 en los brazos de las gónadas del gusano para determinar si un evento de fertilización es inminente.
NOTA: Antes de la fecundación, durante la maduración meiótica, el núcleo del ovocito -1 migrará a la parte posterior del ovocito y la envoltura nuclear se disolverá. Estos eventos son visibles en los canales Nomarksi y GFP utilizando el reportero CaFE, y tales eventos indican que la ovulación ocurrirá dentro de los próximos ~ 10 minutos. - Después de la ruptura de la envoltura nuclear, el citoesqueleto del ovocito -1 más proximal se reorganizará, adoptará una apariencia redondeada y se separará del ovocito -2. En este punto, la ovulación ocurrirá de manera inminente. Comience a grabar el video inmediatamente si se observan estos eventos. Graba vídeos en un entorno de campo amplio.
NOTA: Si el movimiento del núcleo o el redondeo del ovocito no es visible después de 20 minutos, es poco probable que ocurra un evento de ovulación. Concéntrese en un gusano diferente y/o prepare una diapositiva nueva.
5. Cuantificación de transitorios de calcio en la fertilización de C. elegans
- Cuantifique los transitorios de calcio a partir de videos de fertilización utilizando el software del sistema de microscopio como Nikon NIS Elements (descrito aquí). ImageJ/FIJI también son adecuados para el análisis de imágenes11.
- Para cuantificar el cambio en la fluorescencia de los videos de fertilización, utilice los siguientes valores de señal: la señal en el tiempo 0, la señal del marco de interés y la señal de referencia.
- Identificar el momento y el marco en el que se capta el primer estallido de fluorescencia que se produce en el ovocito -1. Defina este fotograma como tiempo 0.
- Para obtener la señal del marco de interés, dibuje la región de interés (ROI) para que abarque las siguientes áreas: el ovocito -1 antes de la fertilización, la espermateca y el ovocito recién fertilizado en el útero. La señal mostrada en el ROI se denomina F1.
NOTA: Asegúrese de que el ROI cubra toda el área traficada por el ovocito. El ROI debe ser idéntico en cada fotograma de la grabación de lapso de tiempo que se está analizando. - Defina un área en blanco de la imagen sin fluorescencia utilizando las mismas dimensiones que el ROI definido en el paso 5.4 para calcular la señal de fondo en cada fotograma. Resta la señal de fondo de cada fotograma.
- Para obtener la señal de línea de base (F0), ubique la trama en un mínimo de 15 tramas antes del tiempo 0. Utilizando el mismo ROI que en el paso 5.4, observe la señal en cada fotograma hasta el tiempo 0. Promedie las señales (con sustracción de fondo) desde el fotograma inicial hasta el tiempo 0 para calcular la señal de referencia.
- Calcule el cambio en la fluorescencia (ΔF) usando la siguiente fórmula:
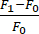 . F1 se define en cada trama como la señal en el ROI sin la señal de fondo. La F0 es la señal fluorescente de referencia promediada de ≥15 fotogramas antes del tiempo 0, calculada en la sección 5.6.
. F1 se define en cada trama como la señal en el ROI sin la señal de fondo. La F0 es la señal fluorescente de referencia promediada de ≥15 fotogramas antes del tiempo 0, calculada en la sección 5.6. - Determine la tasa de cambio entre fotogramas restando el cambio de fluorescencia entre 2 fotogramas y dividiendo por el tiempo.
6. ARNi en C. elegans
- Preparación de placas de ARNi
- Prepare las placas de acuerdo con el paso 1.1 y agregue los siguientes reactivos adicionales para permitir la eliminación de genes a través de ARNi en el paso 6.1.4.
- Añadir 1 mL de isopropil β-d-1-tiogalactopiranósido (IPTG) 1 M de isopropil filtrado estéril (IPTG) (disuelto en agua desionizada) para obtener una concentración final de 1 mM de IPTG en el medio (ver Tabla de Materiales).
- Añadir 1 mL de 100 mg/mL de ampicilina filtrada estéril (disuelta en agua desionizada) para una concentración final de 100 μg/mL de ampicilina en el medio (ver Tabla de Materiales). Alternativamente, use 40 μg/mL de carbenicilina12.
- Revuelva suave pero minuciosamente para mezclar y verter las placas como se describe en los pasos 1.1.4 - 1.1.5.
- Preparación de cultivos saturados de ARNi
- Calcule la cantidad de cultivo saturado multiplicando el número de placas de ARNi sembradas necesarias por 300 μL cuando se utilizan placas de 60 mm x 15 mm o por 100 μL cuando se utilizan placas de 35 mm x 10 mm.
- Utilizando una técnica estéril, cultive las bacterias ARNi deseadas en LB (descritas en los pasos 1.2.1-1.2.3) que contengan 100 μg/mL de ampicilina durante la noche a 37 °C hasta que estén saturadas. Incluya una cepa bacteriana que contenga el vector vacío L4440 como control negativo y una cepa bacteriana que contenga un control positivo de ARNi (por ejemplo, huevo-5) para verificar que el ARNi está funcionando. Además, incluya una condición de "blanco" que contenga solo LB para verificar que no haya contaminación en el LB de la culata.
- Añadir IPTG filtrado estérilmente hasta una concentración final de 1 mM al cultivo saturado durante la noche para la inducción de la expresión de dsRNA e incubar durante al menos 2 h 30 min a 37 °C antes de la siembra. Alternativamente, diluya el cultivo saturado durante la noche fuera de la fase logarítmica antes de inducir con IPTG.
NOTA: IPTG se incluye tanto en las placas como en el cultivo saturado para una expresión óptima de dsRNA. - Utilizando una técnica estéril, transfiera 300 μL del cultivo inducido al centro de una placa de ARNi de 60 mm x 15 mm (solidificada). Alternativamente, use 100 μL de cultivo saturado para una placa de 35 mm x 10 mm.
- Deje que las placas se sequen a RT durante 1-3 días hasta que la mancha bacteriana de ARNi ya no sea líquida. Asegúrese de que las placas estén secas y libres de contaminación antes de usarlas. Guarde las placas a 4 °C boca abajo para su uso futuro.
NOTA: Utilice placas de ARNi lo antes posible para una eliminación óptima, ya que la eficacia del ARNi disminuye con el tiempo. Deséchelo si tiene >1 mes de edad.
- Como se describe en la sección 2, sincronice los gusanos en placas de ARNi.
- Revise las lombrices 1-2 días después de la sincronización de la puesta de huevos para ver si hay retrasos en el desarrollo.
- Algunos ARNi pueden causar defectos en el desarrollo cuando el derribo comienza en la puesta de huevos. Si es necesario, coloque gusanos en las placas de ARNi en una etapa posterior, como L3.
- Como se describe en las secciones 3 y 4, monte los gusanos y prepárese para la obtención de imágenes utilizando cualquier sistema de microscopio equipado para detectar la fluorescencia GFP.
7. Obtención de imágenes del calcio durante la embriogénesis temprana en C. elegans
- Sincronice los gusanos como se describe en la sección 2. Utilice la cepa EAG28, que combina el reportero CaFE y un marcador mCherry de dominio de homología de pleckstrina que resalta el límite de la membrana plasmática. Alternativamente, para visualizar embriones fertilizados ex útero, corte suavemente el gusano a lo largo del útero con una aguja de jeringa estéril para liberar los embriones.
- Prepare los gusanos o embriones para la obtención de imágenes como se describe en la sección 3 y siga las mismas instrucciones descritas en la sección 4 para establecer los parámetros de obtención de imágenes.
- Centra el área de interés en el útero del gusano para visualizar embriones o un huevo puesto.
- En el caso de los embriones en el útero, asegúrese de que el útero no esté detrás del intestino, que el útero no esté abarrotado y que las células individuales del embrión estén claras en el plano focal.
- En el caso de los óvulos ex útero, asegúrese de que las etapas de las células no hayan pasado la etapa de coma del ciclo de vida del gusano.
- Seleccione el protocolo de lapso de tiempo o serie de tiempo y ajústelo a la configuración deseada.
- Para una visualización óptima de los transitorios de calcio en el embrión, aumente los ajustes de tiempo para películas prolongadas. Tenga en cuenta que la exposición constante al láser dañará al embrión durante largos períodos de tiempo.
- Capture videos o imágenes enfocándose en las células de los embriones en la etapa celular deseada.
- Utilice un objetivo de inmersión de 40x o superior para obtener imágenes, aunque los transitorios se detectan fácilmente con un objetivo de aire de 20x.
Resultados
Utilizando los protocolos descritos en este manuscrito, se observaron los patrones dinámicos de señalización de calcio en la fertilización y la embriogénesis en C. elegans.
En la Figura 2 se muestra una secuencia típica de fertilización en lombrices que contienen el reportero CaFE. Para facilitar el análisis, se utilizó la cepa EAG28 que combina el reportero CaFE con un transgén13 en el dominio de homología de pleckstrina (PH-mCherry). El marcador PH-mCherry se localiza en la membrana plasmática y permite una visualización más fácil de los límites celulares, particularmente en el brazo de la gónada. La cepa EAG28 se creó cruzando la cepa EAG16 que contiene el reportero CaFE con la cepa reportera OD70 PH-mCherry13. En C. elegans, la ovulación y la fertilización ocurren al mismo tiempo. El tiempo 0 de la fecundación se considera el primer fotograma en el que se detecta una señal de fluorescencia en el ovocito ovulante (Figura 2). Se produce un estallido brillante de fluorescencia en el sitio de fusión de espermatozoides tan pronto como el borde delantero del ovocito ingresa a la espermateca, el órgano de almacenamiento de espermatozoides7. La señal aparece antes de que se complete la ovulación. La onda de calcio es bifásica, con un rápido estallido inicial seguido de una onda de fluorescencia desde el punto de entrada hacia el polo opuesto. Todo el ovocito se vuelve fluorescente en <30 s.
Tenga en cuenta que el reportero de CaFE está en una sola copia y, por lo tanto, la señal no es tan brillante como la de otros transgenes. No se recomienda el lapso de tiempo de pila Z durante la ovulación a menos que haya un confocal de disco giratorio disponible y optimizado. Las imágenes de la pila Z se pueden obtener antes o después de una película para capturar los cambios en 3D en la morfología de las gónadas. Aunque las imágenes aquí se tomaron con un microscopio confocal, los transitorios también se han observado utilizando microscopía de fluorescencia de campo amplio, que es más común y asequible que la confocal. Usando un confocal de escaneo láser, la velocidad de fotogramas es de aproximadamente 1 fps. Usando un disco giratorio confocal, la velocidad de fotogramas es de 10 fps o más rápida. La señal de fondo del reportero de CaFE es suficiente para iluminar los ovocitos en maduración en el brazo de la gónada. Tras la fecundación, se suele observar un aumento de 1,5-2 veces en la intensidad de la señal GFP.
Existen varios métodos alternativos para la cuantificación. Por ejemplo, el software de análisis de imágenes Imaris, creado por Oxford Instruments (la misma empresa matriz que el microscopio confocal de disco giratorio Andor Dragonfly), tiene la capacidad de cuantificar la señal en cada fotograma solo del ovocito en tránsito en lugar de un ROI mayor. Sin embargo, el software de Imaris no es gratuito. Además, Takayama, Fujita y Onami han descrito con excelente detalle una estrategia detallada de análisis de imágenes para medir las ondas de calcio en el ovocito y utiliza ImageJ, que está disponible gratuitamente14. Alternativamente, los quimógrafos para ilustrar la onda de señal fluorescente a través de un solo ovocito requieren una manipulación de imagen más extensa que la descrita aquí, pero se describen en Takayama y Onami3.
Una ventaja distintiva de C. elegans es su capacidad para eliminar la expresión de casi cualquier gen alimentando a los gusanos con bacterias que expresan dsRNA para desencadenar el ARNiendógeno 15,16. Para una expresión óptima de dsRNA, tanto las placas de ARNi como el cultivo de ARNi saturado contienen IPTG. El dsRNA se produce expresando el gen de interés con promotores flanqueantes. Aquí, hemos utilizado la biblioteca de Ahringer, que se dirige a la mayor parte del genoma del gusano mediante el uso de 16.256 cepas bacterianas17. Alternativamente, el laboratorio Vidal creó un ORFeome de ~ 11,000 clones de ARNi utilizando el sistema Gateway y está disponible para su compra a través de Horizon Discovery18.
Para demostrar la utilidad del indicador CaFE en combinación con el ARNi, examinamos la ovulación prematura. Una deleción parcial en ipp-5 induce un evento de doble ovulación en el que tanto el ovocito -1 como el ovocito -2 entran en la espermateca durante la misma ovulación19. IPP-5 es una fosfatasa que actúa sobre IP3 y disminuye eficazmente los niveles del segundo mensajero IP3. Descubrimos que la eliminación de ARNi de ipp-5 provoca un fenotipo de doble ovulación similar al mutante.
El análisis del reportero de CaFE durante la doble ovulación inducida por la eliminación del ARNi de ipp-5 reveló aspectos novedosos del OET. En primer lugar, se observó una señal de calcio inmediatamente después de la entrada en la espermateca en el ovocito -1 principal, pero no en el ovocito -2 posterior (Figura 3). Aunque se ovulan 2 ovocitos, el ovocito -1 sigue siendo el único ovocito que muestra signos de maduración adecuada, en particular, la ruptura de la envoltura nuclear (NEBD). Estos datos sugieren que, aunque el ovocito -2 esté en presencia de espermatozoides, no es competente para ser fecundado ya que aún no ha madurado adecuadamente. En segundo lugar, el ovocito -2 exhibe una onda de calcio retardada, generalmente cuando el ovocito sale de la espermateca hacia el útero. Aunque la maduración del ovocito es normalmente un requisito previo para la ovulación, estos datos sugieren que la maduración del ovocito y la competencia para la fertilización aún pueden ocurrir después de la ovulación. La visualización de la onda de calcio retardada durante el derribo de ipp-5 destaca las ventajas del reportero CaFE para interrogar la competencia y el tiempo de fertilización.
Además, hemos descubierto que el reportero de CaFE es detectable en los huevos puestos y revela transitorios de calcio unicelulares durante la embriogénesis temprana en C. elegans. Embriones de adultos sincronizados día 1 EAG28 (CaFE; PH-mCherry) se fotografiaron utilizando los protocolos de las secciones 1-5 con el disco giratorio Andor Dragonfly confocal utilizando la cámara Zyla (Figura 4). Los transitorios de calcio se limitan a células individuales y tardan menos tiempo que la onda de calcio de fertilización en completarse (~ 9 s, n = 11). Sin embargo, la onda de calcio durante la embriogénesis no parece ser bifásica. No se observaron transitorios de calcio antes de la etapa de 8 células. Cabe destacar que el calcio no se localiza en el surco de escote en C. elegans, como se observa en muchos otros organismos, incluidos los humanos y Xenopus 20,21,22. Los transitorios de calcio unicelulares se observaron mucho después de la gastrulación (~200 min después de la fertilización) y en los huevos puestos. Un solo embrión muestra múltiples transitorios de calcio a lo largo del tiempo, pero en diferentes células y, por lo general, una célula a la vez. Tenga en cuenta que la exposición continua a la estimulación láser dañará los embriones. Se recomienda una potencia de LED/láser más baja o una exposición discontinua. Sin embargo, dado que las ondas de calcio son relativamente cortas a ~ 9 s, no recomendamos más de 2 s entre eventos de estimulación LED / láser.
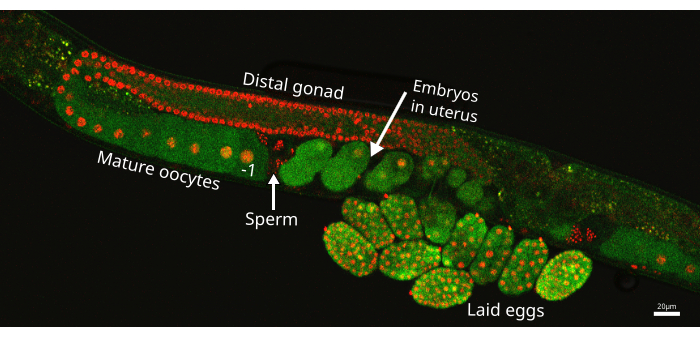
Figura 1: Brazo de la gónada de C. elegans . Imagen de un brazo gonadal de la cepa de gusano EAG25, que muestra el GFP reportero de CaFE (verde) y el marcador de histona 2B-mCherry (rojo), que resalta los núcleos de la línea germinal. El ovocito proximal se denota por el -1 justo antes de la espermateca, donde se almacenan los espermatozoides (flecha). El embrión situado más cerca de la espermateca, pero dentro del útero, es el embrión más reciente. La imagen fue tomada utilizando el confocal de escaneo láser Nikon ECLIPSE Ti2. Barra de escala = 20 μm. Haga clic aquí para ver una versión más grande de esta figura.
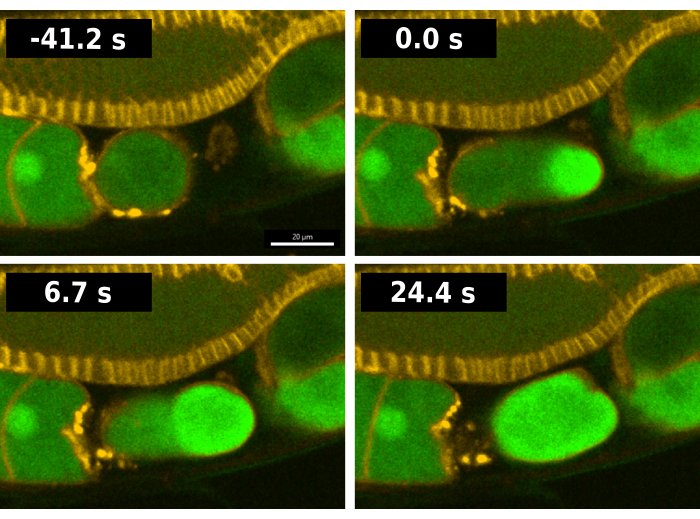
Figura 2: Visualización de la onda de calcio durante la fecundación. Imágenes de series temporales de fluorescencia que reflejan el calcio intracelular durante la fertilización en la cepa de gusano EAG28. La cepa EAG28 contiene tanto el reportero CaFE (verde) como un dominio de homología pleckstrina-fusión mCherry (amarillo), que resalta las membranas plasmáticas. El tiempo 0 indica el primer fotograma que muestra un claro aumento de la señal fluorescente en el ovocito. El tiempo 24,4 s refleja el primer fotograma que muestra la fluorescencia del ovocito completo. Las imágenes de series de tiempo se tomaron utilizando el disco giratorio Andor Dragonfly confocal con una cámara Zyla. Barra de escala = 20 μm. Haga clic aquí para ver una versión más grande de esta figura.
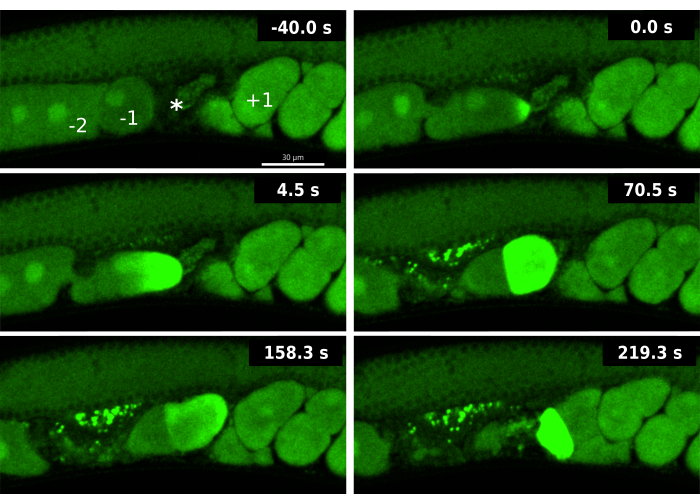
Figura 3: Ondas de calcio durante un evento de doble ovulación inducido por ipp-5 RNAi. Imágenes time-lapse de ondas secuenciales de calcio del ovocito -1 y del ovocito -2 en una cepa EAG16 (CaFE en verde GFP) expuestas al ARNi ipp-5 . El marco -40.0 s incluye etiquetas para el ovocito -1 (-1), el ovocito -2 (-2), la espermateca (*) y el embrión +1 (+1). El tiempo 0 s muestra el aumento inicial de la fluorescencia de la onda de calcio del ovocito -1. El tiempo 4,5 s muestra la onda de calcio extendiéndose hacia el polo opuesto en el ovocito -1. El tiempo 70,5 s muestra fluorescencia de célula completa en el ovocito -1 acompañado por el ovocito -2 en la señal GFP basal; Ambos ovocitos se encuentran en la espermateca. El ovocito -1 fecundado y el ovocito -2 no fecundado entran en el útero a los 158,3 s. A los 219,3 s, el ovocito -2 muestra una onda de calcio fluorescente tardía. Las imágenes fueron tomadas usando el disco giratorio Andor Dragonfly confocal con una cámara Zyla. Barra de escala = 30 μM Haga clic aquí para ver una versión más grande de esta figura.
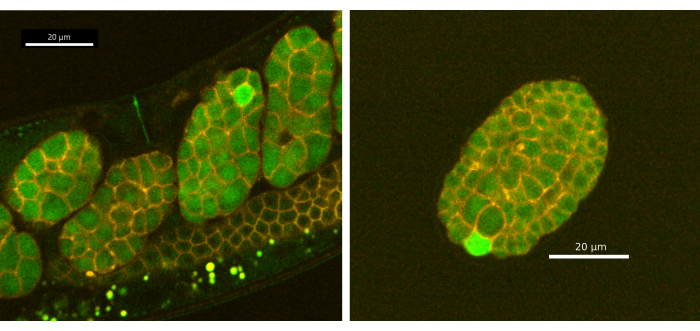
Figura 4: Transitorios de calcio unicelular en embriogénesis temprana. (A) Los embriones en el útero y (B) ex útero (huevo puesto) mostraron una señal fluorescente en una sola célula durante la embriogénesis. Las imágenes fueron tomadas usando el disco giratorio Andor Dragonfly confocal con una cámara Zyla. Barra de escala = 20 μm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Una herramienta simple con un protocolo robusto es una combinación potente para abordar preguntas científicas difíciles. Aquí, se presentan los métodos para la visualización del calcio como un proxy fácilmente detectable para la fertilización utilizando el reportero CaFE. Este mismo reportero persiste en la embriogénesis temprana y también permite la visualización de transitorios de calcio más adelante en el desarrollo. La señalización del calcio sirve como un segundo mensajero crítico que demarca cambios importantes en la función celular, particularmente para la biología del desarrollo. En el ovocito, la primera fase de estallido de la onda de calcio marca no solo el momento de la fertilización, sino también el sitio de fusión de los espermatozoides3. En C. elegans, el eje A-P también está determinado por el sitio de fusión de los espermatozoides23. Por lo tanto, la capacidad de visualizar el calcio en ovocitos y embriones permite la investigación de preguntas complejas que son fundamentales para la biología celular y del desarrollo.
El método descrito aquí con el reportero CaFE debería ser lo suficientemente sencillo para los novatos en nematodos. Los métodos previos para detectar ondas de calcio en ovocitos de C. elegans se basaban en la inyección de colorante 3,8,14. Si bien estos estudios fueron importantes y esclarecedores, la inyección de tinte requiere mucha mano de obra y el equipo de inyección no está disponible en todos los laboratorios.
Los protocolos presentados aquí se han optimizado para gusanos sanos. Para maximizar las posibilidades de éxito, asegúrese de que no haya contaminación en el medio de lombrices o en el césped de bacterias del que se alimentan las lombrices. No expongas a las lombrices a condiciones de estrés, como la temperatura, ya que afectarán la ovulación y la fertilización. Además, los controles de la eficacia del ARNi deben incluirse en cada experimento, ya que la eficacia disminuye con el tiempo. Utilice un control positivo para la eficacia del ARNi como el huevo-5, que genera embriones, pero no progenie viable ya que la formación de la cáscara del huevo se ve comprometida24. Además, los parámetros de captura de imágenes deben estar optimizados para cada sistema de microscopía. Nuestras especificaciones se incluyen aquí como referencia, pero es de esperar que haya desviaciones. Aunque este sistema detecta fácilmente el calcio citoplasmático como un proxy de la fertilización, no representa necesariamente un evento de fusión de espermatozoides-ovocitos de buena fe.
La amplia gama de herramientas y mutantes distribuidos libremente dentro de la comunidad de gusanos mejora la utilidad del reportero CaFE. El reportero está integrado en el genoma del gusano y se cruza fácilmente con otras cepas mutantes o reporteras de C. elegans 25. Se reporta la creación de EAG25 que expresa el reportero CaFE con un marcador de histona H2B-mCherry para visualizar núcleos (Figura 1) y EAG28 con el reportero de CaFE y un marcador de dominio de homología de pleckstrina-mCherry, que resalta la periferia celular (Figura 2)13,26. Ambas cepas ayudan en la visualización de las células en la línea germinal y durante la embriogénesis. Además, la instalación de ARNi en
C. elegans, cuando se utiliza con el reportero CaFE, ha revelado nuevos conocimientos sobre la competencia de fertilización. Como se muestra en la doble ovulación inducida por la caída del ARNi ipp-5 en la Figura 3, la presencia de espermatozoides y un ovocito ovulado es insuficiente para estimular un evento de fertilización.
Estos resultados indican que debe existir otra señal, o la ausencia de un inhibidor, que permita que el ovocito sea fecundado. El ovocito ovulado prematuramente muestra un retraso transitorio de calcio cuando el ovocito se mueve hacia el útero. Esta onda tardía de calcio sugiere que el ovocito -2 ovulado prematuramente puede desarrollar la competencia de fertilización con el tiempo. Anticipamos que los estudios sobre el momento de la fertilización, particularmente con respecto a la señalización y regulación célula-célula, serán ayudados por el uso del reportero CaFE. Además, el reportero de CaFE persiste en la embriogénesis y muestra transitorios de calcio de una sola célula. Esta señalización embrionaria de calcio también se reportó dentro de las primeras 24 horas del desarrollo del pez cebra27. Se desconoce el papel de los transitorios de calcio, pero la presencia sugiere un evento de señalización celular durante el desarrollo que aún no se ha explorado. En particular, los transitorios de calcio no se observaron en los cigotos recién fertilizados. Por lo tanto, el calcio no se localiza en el surco de escisión como se ha documentado en varios otros organismos, incluidos Xenopus y humanos 20,21,22.
Es importante destacar que el reportero CaFE es lo suficientemente fácil como para ser utilizado por estudiantes universitarios con una capacitación mínima. Hemos diseñado y ejecutado un laboratorio CURE (experiencia de investigación de pregrado basada en cursos) de 1 crédito para estudiantes de biología con las cepas y protocolos descritos aquí. En el transcurso de un semestre, la clase se reunió una vez a la semana durante 3 horas o dos veces por semana durante 1 hora y 30 minutos cada una. A los estudiantes se les dio la opción de trabajar solos o en grupos de 2. Cada estudiante/pareja seleccionó un gen diferente para estudiar de una lista seleccionada. Realizaron ARNi contra el gen elegido en la cepa EAG28 y examinaron los efectos de los gusanos sobre la fecundidad, la fertilización y/o la morfología de las gónadas. Con base en sus resultados y en sus lecturas de fondo utilizando literatura primaria, los estudiantes desarrollaron hipótesis que podrían probar en experimentos posteriores. Este diseño iterativo fue fundamental para aumentar la participación de los estudiantes28. Los estudiantes obtuvieron una auténtica experiencia en investigación y adquirieron habilidades en organismos modelo, pantallas genéticas y microscopía de fluorescencia. Dada la facilidad de uso del reportero CaFE, los estudiantes sin experiencia en investigación pudieron tener éxito. Posteriormente, los estudiantes expresaron abrumadoramente una preferencia por el formato CURE sobre las clases de laboratorio tradicionales, y muchos estudiantes expresaron su deseo de continuar con la investigación. En conjunto, estas herramientas y protocolos ayudan tanto en la educación como en la investigación de los procesos de desarrollo temprano.
Divulgaciones
Los autores declaran no tener intereses contrapuestos ni financieros.
Agradecimientos
KSKG fue financiado por una subvención del Instituto Nacional de Salud Infantil y Desarrollo Humano Eunice Kennedy Shriver (R15HD111986). Algunas cepas fueron proporcionadas por el CGC, que está financiado por la Oficina de Programas de Infraestructura de Investigación de los NIH (P40 OD4010440). Agradecemos a WormBase.
Materiales
| Name | Company | Catalog Number | Comments |
| Agar | Fisher Scientific | DF0140-07-4 | 2 kg; Powder dissolves easier than flakes |
| Agarose | MidSci | BE-A125 | 500 g |
| Alcohol lamp | Fisher Scientific | S13475 | Use with 95% ethanol |
| Ammonium chloride (NH4Cl) | Fisher Scientific | AAA1500030 | 250 g |
| Ampicillin | Fisher Scientific | BP1760-5 | 5 g |
| AMSCO 400 Series Small Steam Sterilizer | Steris Healthcare | N/A | |
| Bacto-peptone | Fisher Scientific | BP1420-500 | 500 g |
| Bacto-tryptone | Fisher Scientific | DF0123-17-3 | 500 g |
| Calcium chloride (CaCl2) | Fisher Scientific | C69-500 | 500 g |
| Cholesterol | Thermo Scientific | A11470.18 | 50 g |
| Dragonfly 200 spinning disk confocal | Oxford Instruments Andor | N/A | Used with Leica microscope |
| Fisherbrand Superfrost Cytogenics Microscope Slides | Fisher Scientific | 22-035-900 | 144 slides per pack |
| Flat Nose Pliers, Smooth Jaw | Home Depot | 305530604 | Ensure pliers are smooth jaw |
| Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Fisher Scientific | BP1755-10 | 10 g; dioxane-free |
| Laboratory tape | Fisher Scientific | 15-901-10R | 0.5 inch tape is used to tape microscope slides |
| Levamisole | Fisher Scientific | AC187870100 | 10 g |
| Magnesium sulfate (MgSO4) | Fisher Scientific | M63-500 | 500 g |
| Microscope cover glass | Fisher Scientific | 12541016 | 1 oz pack |
| Nikon ECLIPSE Ti2 laser scanning confocal | Nikon | N/A | |
| Nikon NIS Elements software | Nikon | N/A | Confocal |
| OP50 Escherichia coli | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Platinum Wire | TriTech | PT-9010 | |
| Potassium phosphate dibasic (K2HPO4) | Fisher Scientific | P288-500 | 500 g |
| Potassium phosphate monobasic (KH2PO4) | Fisher Scientific | AA1159436 | 500 g |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | 500 g |
| Sodium phosphate dibasic heptahydrate (Na2HPO4) | Fisher Scientific | S471-3 | 3 kg |
| Stereo microscope | Leica | KL300 LED | |
| Sterile Petri dish (35 mm x 10 mm) | CellTreat | 229638 | 960 Petri dishes per case |
| Sterile Petri dish (60 mm x 15 mm) | CellTreat | 229665 | 500 Petri dishes per case |
| Strain EAG16 spn-4p::jGCaMP7s::pie-1u | Caenorhabditis Genetics Center (CGC) | EAG16 | Created by Kim Guisbert Lab |
| Strain EAG25 spn-4p::jGCaMP7s::pie-1u; ujIs113 II. | Caenorhabditis Genetics Center (CGC) | EAG25 | Created by Kim Guisbert Lab |
| Strain EAG28 spn-4p::jGCaMP7s::pie-1u; unc-119(ed3) III; ltIs44 V. | Caenorhabditis Genetics Center (CGC) | EAG28 | Created by Kim Guisbert Lab |
| Strain JIM113 ujIs113 II [pie-1p::mCherry::H2B::pie-1 3'UTR + nhr-2p::his-24::mCherry::let-858 3'UTR + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | JIM113 | Created by E. Preston - Murray Lab |
| Strain OD70 unc-119(ed3) III; ltIs44 V [pie-1p::mCherry::PH(PLC1delta1) + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | OD70 | Created by Audhya/Oegema - Greenstein Lab |
| Tritech Worm Pick Handle | TriTech | TWPH1 | |
| Yeast extract | IBI Scientific | IB49160 | 500 g |
Referencias
- Stein, P., Savy, V., Williams, A. M., Williams, C. J. Modulators of calcium signalling at fertilization. Open Biol. 10 (7), 200118(2020).
- McAvey, B. A., Wortzman, G. B., Williams, C. J., Evans, J. P. Involvement of calcium signaling and the actin cytoskeleton in the membrane block to polyspermy in mouse eggs. Biolo Reprod. 67 (4), 1342-1352 (2002).
- Takayama, J., Onami, S. The sperm TRP-3 channel mediates the onset of a Ca 2+ wave in the fertilized C. elegans oocyte. Cell Rep. 15 (3), 625-637 (2016).
- Yamamoto, I., Kosinski, M. E., Greenstein, D. Start me up: Cell signaling and the journey from oocyte to embryo in C. elegans. Dev Dyn. 235 (3), 571-585 (2006).
- Hubbard, E. J. A., Greenstein, D. The Caenorhabditis elegans gonad: A test tube for cell and developmental biology. Dev Dyn. 218 (1), 2-22 (2000).
- Korta, D. Z., Hubbard, E. J. A. Soma-germline interactions that influence germline proliferation in Caenorhabditis elegans. Dev Dyn. 239 (5), 1449-1459 (2010).
- Toperzer, K. M., Brennan, S. J., Carroll, D. J., Guisbert, E. A., Kim Guisbert,, S, K. Visualization of the biphasic calcium wave during fertilization in Caenorhabditis elegans using a genetically encoded calcium indicator. Biol Open. 12 (9), 059832(2023).
- Samuel, A. D., Murthy, V. N., Hengartner, M. O. Calcium dynamics during fertilization in C. elegans. BMC Dev Biol. 1 (1), 8(2001).
- Dana, H., et al. High-performance calcium sensors for imaging activity in neuronal populations and microcompartments. Nat Methods. 16 (7), 649-657 (2019).
- Golden, N. L., Plagens, R. N., Kim Guisbert, K. S., Guisbert, E. Standardized methods for measuring induction of the heat shock response in Caenorhabditis elegans. J Vis Exp. (161), e61030(2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamath, R., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Kachur, T. M., Audhya, A., Pilgrim, D. B. UNC-45 is required for NMY-2 contractile function in early embryonic polarity establishment and germline cellularization in C. elegans. Dev Biol. 314 (2), 287-299 (2008).
- Takayama, J., Fujita, M., Onami, S. In vivo live imaging of calcium waves and other cellular processes during fertilization in Caenorhabditis elegans. Bio Protoc. 7 (7), e2205(2017).
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391 (6669), 806-811 (1998).
- Conte, D., MacNeil, L. T., Walhout, A. J. M., Mello, C. C. RNA Interference in Caenorhabditis elegans. Curr Protoc Mol Biol. 109, 1-30 (2015).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421 (6920), 231-237 (2003).
- Reboul, J., et al. elegans ORFeome version 1.1: experimental verification of the genome annotation and resource for proteome-scale protein expression. Nat Genet. 34 (1), 35-41 (2003).
- Bui, Y. K., Sternberg, P. W. Caenorhabditis elegans inositol 5-phosphatase homolog negatively regulates inositol 1,4,5-triphosphate signaling in ovulation. Mol Biol Cell. 13 (5), 1641-1651 (2002).
- Muto, A., Kume, S., Inoue, T., Okano, H., Mikoshiba, K. Calcium waves along the cleavage furrows in cleavage-stage Xenopus embryos and its inhibition by heparin. J Cell Biol. 135 (1), 181-190 (1996).
- Paudel, S., Sindelar, R., Saha, M. Calcium signaling in vertebrate development and its role in disease. Int J Mol Sci. 19 (11), 3390(2018).
- Paudel, S., Yue, M., Nalamalapu, R., Saha, M. S. Deciphering the calcium code: A review of calcium activity analysis methods employed to identify meaningful activity in early neural development. Biomolecules. 14 (1), 138(2024).
- Goldstein, B., Hird, S. N. Specification of the anteroposterior axis in Caenorhabditis elegans. Development. 122 (5), 1467-1474 (1996).
- Johnston, W. L., Dennis, J. W. The eggshell in the C. elegans oocyte-to-embryo transition. Genesis. 50 (4), 333-349 (2012).
- Stevenson, Z. C., Moerdyk-Schauwecker, M. J., Jamison, B., Phillips, P. C. Rapid self-selecting and clone-free integration of transgenes into engineered CRISPR safe harbor locations in Caenorhabditis elegans. G3. 10 (10), Bethesda. 3775-3782 (2020).
- Zacharias, A. L., Walton, T., Preston, E., Murray, J. I. Quantitative differences in nuclear β-catenin and TCF pattern embryonic cells in C. elegans. PLoS Genet. 11 (10), e1005585(2015).
- Webb, S. E., Miller, A. L. Calcium signalling during zebrafish embryonic development. BioEssays. 22 (2), 113-123 (2000).
- Wiseman, E., Carroll, D. J., Fowler, S. R., Guisbert, E. Iteration in an inquiry-based undergraduate laboratory strengthens student engagement and incorporation of scientific skills. J Scholarsh Teach Learn. 20 (2), 99-112 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados