Method Article
In vivo Visualisierung von Calciumtransienten während der Befruchtung und frühen Entwicklung bei C. elegans
In diesem Artikel
Zusammenfassung
Hier stellen wir Protokolle und Werkzeuge zur Visualisierung von Kalzium während der Befruchtung und der frühen Embryogenese unter Verwendung eines genetisch kodierten Kalziumreporters vor, der in der Keimbahn des Modellfadenwurms C . elegans exprimiert wird.
Zusammenfassung
Calcium ist ein wichtiges Signalmolekül während des Übergangs von der Eizelle zum Embryo (OET) und der frühen Embryogenese. Der hermaphroditische Fadenwurm Caenorhabditis elegans bietet mehrere einzigartige Vorteile für die Untersuchung des OET, da er transparent ist und eine geordnete Gonade hat, die alle ~23 min bei 20 °C eine reife Eizelle produziert. Wir haben den genetisch kodierten Kalziumindikator jGCaMP7s so modifiziert, dass er fluoreszierend den Zeitpunkt der Befruchtung in einem lebenden Organismus anzeigt. Wir haben diesen Reporter "CaFE" für Calcium während der Befruchtung bei C. elegans genannt. Der CaFE-Reporter wurde in einer einzigen Kopie in einen Safe-Harbor-Locus umgewandelt, hat keinen signifikanten Einfluss auf die Physiologie oder Fruchtbarkeit und erzeugt ein robustes Signal bei der Befruchtung. In dieser Arbeit wird eine Reihe von Protokollen zur Verwendung des CaFE-Reporters als in vivo-Werkzeug zur Dissektion der OET und der Embryogenese vorgestellt. Wir umfassen Methoden zur Synchronisierung von Würmern, zur Untersuchung der Auswirkungen des RNAi-Knockdowns, zur Montage von Würmern für die Bildgebung und zur Visualisierung von Kalzium in Eizellen und Embryonen. Darüber hinaus stellen wir die Generierung zusätzlicher Wurmstämme vor, um diese Art der Analyse zu unterstützen. Um die Nützlichkeit des CaFE-Reporters zur Visualisierung des Zeitpunkts der Befruchtung zu demonstrieren, berichten wir, dass der doppelte Eisprung auftritt, wenn ipp-5 von RNAi angegriffen wird, und dass nur die erste Eizelle sofort befruchtet wird. Darüber hinaus wird hier über die Entdeckung von einzelligen Kalziumtransienten während der frühen Embryogenese berichtet, was zeigt, dass der CaFE-Reporter bis in die frühe Entwicklung fortbesteht. Wichtig ist, dass der CaFE-Reporter in Würmern einfach genug ist, um ihn in kursbasierte Forschungslaborkurse (CURE) zu integrieren. Der CaFE-Reporter, gekoppelt mit der geordneten Gonade und der Leichtigkeit von RNAi in Würmern, erleichtert die Untersuchung der Zell-Zell-Dynamik, die zur Regulierung der internen Befruchtung und der frühen Embryogenese erforderlich ist.
Einleitung
Die Befruchtung markiert den Beginn eines neuen Lebenszyklus, aber die Bestimmung des genauen Zeitpunkts der Befruchtung ist eine Herausforderung. Ein konserviertes Merkmal der Befruchtung ist eine Welle von Kalzium über die Eizelle unmittelbar nach der Spermienfusion1. Obwohl die Art der Kalziumwelle in Bezug auf Häufigkeit und Geschwindigkeit von Spezies zu Art unterschiedlich ist, zeigen fast alle Organismen nach der Befruchtung einen vorübergehenden Anstieg des intrazellulären Kalziums. Die Kalziumwelle spielt eine entscheidende Rolle bei der Blockade der Polyspermie, der Eizellaktivierung und anderer wichtiger zellulärer Ereignisse2. Da die Kalziumwelle an der Stelle der Spermienfusion beginnt, dient Kalzium als Marker für die Befruchtung3.
Caenorhabditis elegans ist ein idealer Modellorganismus für die Untersuchung der frühen Entwicklung. Bei den Würmern handelt es sich um durchsichtige Hermaphroditen, die genetisch manipulierbar sind4. Am wichtigsten für die Untersuchung der Befruchtung ist, dass erwachsene C. elegans-Erwachsene kontinuierlich Eizellen mit einer streng geordneten Gonade produzieren5. Tatsächlich sind Eizellen die einzigen neuen Zellen, die produziert werden, da das Soma im Erwachsenenalter postmitotisch ist6. Abbildung 1 zeigt die Gonade, die aus 2 symmetrischen U-förmigen Röhren mit sich entwickelnden Eizellen besteht. Die Eizelle, die der Spermathek (Speicherorgan für Spermien) am nächsten liegt, wird als -1-Eizelle bezeichnet. Dieses Fließbanddesign der Wurmgonade ovuliert bei jungen Erwachsenen alle ~23 min bei 20 °C eine reife Eizelle in die Spermatheca7. Der frisch befruchtete Embryo wandert dann in eine gemeinsame Gebärmutter, bevor er durch eine einzelne Vulva gelegt wird.
Frühere Techniken zur Visualisierung von Calcium während der Befruchtung bei C. elegans beruhten auf der Mikroinjektion von calciumempfindlichen Farbstoffen 3,8. Um die Kalziumwelle bei der Fusion von Spermien und Eizellen besser betrachten zu können, wurde ein genetisch kodierter Kalziumindikator auf Basis von jGCaMP7s in einer Einzelkopie an einem Safe-Harbor-Locus unter Verwendung von Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR) eingefügt7,9. Der Reporter wurde als CaFE für Calcium während der Befruchtung bei C. elegans bezeichnet. Der Reporter zeigt keine signifikanten Mängel in der Physiologie oder Fruchtbarkeit.
In dieser Arbeit werden Protokolle zur Visualisierung der Kalziumwelle in den Eizellen und Embryonen von C. elegans mit Hilfe des CaFE-Reporters vorgestellt. In Kombination mit den unzähligen Werkzeugen, die in der Wurmgemeinschaft zur Verfügung stehen, wie z. B. RNA-Interferenz (RNAi) und mCherry-Gonadenmarkern, erleichtert der CaFE-Reporter die Untersuchung der Regulation interner Befruchtungsereignisse, insbesondere der Befruchtungskompetenz und des Zeitpunkts der Befruchtung. Darüber hinaus bleibt der CaFE-Reporter bis in die frühe Entwicklung bestehen und ist ein einzigartiges Werkzeug zur Untersuchung der Embryogenese.
Protokoll
1. Pflege von C. elegans
- Vorbereitung der Nematoden-Wachstumsmedium (NGM)-Platte
- Kombinieren Sie 3 g Natriumchlorid (NaCl), 2,5 g Bacto-Pepton, 20 g Agar und 1 l deionisiertes Wasser in einem 4-l-Kolben (siehe Materialtabelle). Decken Sie die Oberseite des Kolbens vor dem Autoklavieren mit Alufolie ab.
- Autoklav zum Sterilisieren mit den Einstellungen des flüssigen Sterilisationszyklus. Es wird ein Sterilisationszyklus empfohlen, der 30 Minuten lang auf 121 °C eingestellt ist.
- Lassen Sie die Mischung auf ~50 °C abkühlen, ca. 20 min bei Raumtemperatur (RT).
- 25 mL steriles 1 M KH2PO4 (in Wasser gelöst, pH = 6), 1 mL steriles 1 M CaCl2 (in Wasser gelöst), 1 mL steriles 1 M MgSO4 (in Wasser gelöst) und 1 mL 5 mg/ml Cholesterin (gelöst in 100 % Ethanol) zugeben und nach jeder Zugabe vorsichtig schwenken (siehe Materialtabelle).
- Übertragen Sie einen Teil der Mischung mit einer sterilen Technik in ein steriles 300-ml-Becherglas, um das Ausgießen zu erleichtern. Gießen Sie das warme Medium ein, um den Boden des Tellers zu bedecken.
HINWEIS: Ungefähr 10 ml pro Platte in 60 mm x 15 mm sterilen Petriplatten (siehe Materialtabelle) ergeben 100 Platten. Alternativ können sterile Petrischalen mit einer Größe von 35 mm x 10 mm verwendet werden, wobei ~4 mL pro Platte ~250 Platten ergeben. - Lassen Sie die NGM-Platten mindestens 1 Tag bei RT trocknen, bevor Sie sie mit Bakterien aussäen. Lagern Sie die Platten für die zukünftige Verwendung kopfüber bei 4 °C.
- OP50 bakterielle Rasenvorbereitung
- Stellen Sie Luria-Brühe (LB) her, indem Sie 5 g Hefeextrakt, 10 g Bacto-Trypton, 5 g NaCl und 1 l entionisiertes Wasser in einem 2-Liter-Kolben kombinieren (siehe Materialtabelle). Mit einer Rührplatte und einem Rührstab mischen, bis es sich aufgelöst hat.
- Aliquotieren Sie LB in Flaschen und autoklavieren Sie mit den Einstellungen des Flüssigkeitssterilisationszyklus. Es wird ein Sterilisationszyklus empfohlen, der 30 Minuten lang auf 121 °C eingestellt ist. Wenn Sie einen Behälter mit Schraubverschluss verwenden, lösen Sie den Schraubverschluss um eine Vierteldrehung, um eine gute Belüftung während des Autoklavenzyklus zu gewährleisten.
- Lassen Sie die Brühe vor der Verwendung auf RT abkühlen.
- In steriler Technik werden 50 ml LB in einem 250-ml-Erlenmeyerkolben mit OP50-Bakterien inokuliert und über Nacht bei 37 °C unter Schütteln gezüchtet.
- Übertragen Sie mit steriler Technik 300 μl gesättigte Kultur auf die Mitte einer 60 mm x 15 mm großen NGM-Platte. Für 35 mm x 10 mm große Platten werden 100 μl gesättigte Kultur empfohlen.
- Lassen Sie die Platten 1-3 Tage bei RT trocknen, bis der OP50-Bakterienfleck nicht mehr flüssig ist. Stellen Sie sicher, dass die Platten trocken und frei von Verunreinigungen sind, bevor Sie sie verwenden. Lagern Sie die Platten für die zukünftige Verwendung bei 4 °C. Die Lagerung der Platten auf dem Kopf reduziert die Kondensatbildung auf der Plattenoberfläche.
- Konstruktion der Schneckenhacke
- Lösen Sie den silbernen Kragen in Richtung des Kopfes des Schneckenpickelgriffs (siehe Materialtabelle), indem Sie ihn vorsichtig abschrauben, so dass sich die vier Teile auseinander lösen.
- Schneiden Sie mit einer Schere 1,5 Zoll (etwa die Länge des Daumens) Platindraht (siehe Materialtabelle) ab und legen Sie ihn in die Mitte der vier Abschnitte; Etwa ein Zentimeter ragt aus dem Griff heraus.
- Halten Sie den Draht in der Mitte, während Sie den Kragen festziehen. Die vier Abschnitte kommen in der Mitte zusammen und halten den Platindraht an Ort und Stelle.
- Forme den Draht vorsichtig mit einer Flachzange. Eine Regenbogen- oder Bogenform eignet sich am besten zum Pflücken.
- Schaffen Sie etwa einen 2-3 mm "Fuß" am Ende des Drahtes, indem Sie ihn wiederholt mit der Flachzange festklemmen. Benutze diesen "Fuß", um die Würmer aufzunehmen und halte den "Fuß" flach an der Oberfläche der Platte.
HINWEIS: Wenn Sie zu fest klemmen, wird der Platindraht durchtrennt. Vermeiden Sie scharfe Kanten mit der Flachzange (oder einer gleichwertigen Zange), um ein Durchstechen der Würmer oder der Platten zu vermeiden.
- Umgang mit Schnecken
- Halten Sie den Wurmpickel wie einen Bleistift und sterilisieren Sie den "Fuß", indem Sie den Draht in die Flamme einer 95%igen Ethanollampe legen, bis er leuchtend orange ist.
- Legen Sie eine OP50-Saatplatte auf den Tisch eines Präparierfernrohrs und entfernen Sie den Deckel. Berühren Sie mit dem "Fuß" des Pickels vorsichtig einen Bereich des Bakterienrasens und sammeln Sie mit einer sanften Wischbewegung die Bakterien auf dem Boden. Die Bakterien, die an der Spitzhacke kleben (als "Glibber" bezeichnet), fungieren als Klebstoff, um Würmer aufzunehmen.
- Schieben Sie eine Platte mit Würmern auf den Präparierzielfernrohr und entfernen Sie den Deckel, während Sie darauf achten, dass das Plektrum andere Oberflächen nicht berührt. Berühre schnell und vorsichtig den "Fuß" der Spitzhacke auf einen Wurm, um ihn aufzuheben. Diese Technik ermöglicht es, mehrere Würmer gleichzeitig zwischen den Platten zu bewegen.
- Berühren Sie vorsichtig den "Fuß" des Pickels mit dem nicht gesäten Teil einer neuen Platte, um die Würmer zu übertragen; In der Regel handelt es sich dabei um dieselbe Platte, aus der in Abschnitt 1.4.2 die Bakterien entnommen wurden.
- Sterilisieren Sie das Meißel zwischen den einzelnen Platten und/oder Wurmstämmen, um eine Kontamination zu vermeiden und das Meißel nach dem Sterilisieren abkühlen zu lassen.
HINWEIS: Das Berühren eines Wurms mit einer scharfen Spitzhacke stresst oder tötet das Tier. Der Wurmpickel kühlt ab, nachdem er den Bakterienrasen berührt hat, um "Schleim" für die Wurmübertragung aufzunehmen.
- Die Würmer werden bei 20 °C auf NGM-Platten (Schritt 1.1) gehalten, die mit OP50-Escherichia coli-Bakterien (Schritt 1.2) besiedelt sind. Passage der Würmer 2 - 3 Mal pro Woche, indem mehrere erwachsene Tiere mit einem Wurmpickel (Schritte 1.3-1.4) auf frische Platten (60 mm x 15 mm) umgeladen werden, um Hunger zu vermeiden, der die Physiologie verändern kann.
HINWEIS: C. elegans-Stämme und OP50-Bakterien sind über das Caenorhabditis Genetics Center (CGC) erhältlich.
2. Synchronisation der Entwicklung von C. elegans
- Synchronisieren Sie das Alter von Würmern mit einer zeitgesteuerten Eiablage (hier beschrieben). Wenn eine große Anzahl von Würmern für ein Klassenzimmer oder einen anderen Zweck benötigt wird, führen Sie eine Bleichsynchronisation durch, wie in Golden et al.10 beschrieben.
- Übertragen Sie adulte Würmer mit einem Platindrahtpickel (beschrieben in Abschnitt 1) auf eine neue, ausgesäte OP50-Platte. Die Anzahl der Würmer wird durch den experimentellen Bedarf bestimmt. Tag 1 Würmer legen etwa 3 Eier pro Stunde. Daher erzeugen 10 Würmer in 1 h etwa 30 Eier. Verwenden Sie Würmer vorzugsweise am ersten Tag des Erwachsenenalters (Tag 1).
- Nach ca. 1 h die adulten Würmer von der Platte nehmen, so dass nur die Eier übrig bleiben.
HINWEIS: Das versehentliche Zurücklassen eines Wurms ist ein häufiger Fehler. Die verbleibenden Würmer legen weiterhin Eier und stören die Synchronisation. Die Würmer reifen etwa 72 Stunden nach der Ablage zu adulten Tieren am Tag 1 heran, wenn sie bei 20 °C inkubiert werden.
3. Montage von Schnecken für die Bildgebung
- Verwenden Sie bei der Abbildung von Befruchtungsereignissen Erwachsene am Tag 1, um die Visualisierungsmöglichkeiten zu maximieren.
HINWEIS: Bei Erwachsenen an Tag 1 wird alle ~23 Minuten eine Eizelle befruchtet. Der Eisprung verlangsamt sich jedoch mit zunehmendem Alter, und ein typischer Hermaphrodit hört nach dem 4. Tag auf, Eier zu legen. - Bereiten Sie eine Lösung von 3% Agarose in Wasser vor, indem Sie 3 g Agarose in 100 ml Wasser mischen. Die Agaroselösung in der Mikrowelle erhitzen, bis sich die Agarose vollständig aufgelöst hat. Um eine verfestigte Lösung wiederzuverwenden, erhitzen, bis sie sich vollständig aufgelöst hat. Erzeugen Sie nach mehrmaliger Anwendung eine frische Lösung.
- Legen Sie einen leeren Objektträger für die Bildgebung zwischen zwei andere Objektträger, die eine einzelne Schicht Laborklebeband über die Längsachse des Objektträgers haben (siehe Materialtabelle). Das Band bildet einen Abstandshalter für das Agarose-Pad, das eine gleichmäßige Verteilung und eine gleichmäßige Breite ermöglicht. Achten Sie darauf, dass die drei Objektträger in einer Reihe stehen und sich die langen Seiten berühren: zwei Objektträger mit Klebeband, die einen leeren Objektträger flankieren.
- Lassen Sie die erhitzte 3%ige Agaroselösung (~100-150 μL) mit einer 1-ml-Pipette auf die Mitte des Objektträgers fallen, die sich zwischen den geklebten Objektträgern befindet.
- Platzieren Sie schnell einen neuen Objektträger auf dem Agarose-Tröpfchen und stellen Sie sicher, dass der neue Objektträger senkrecht zum Objektträger mit dem Agarose-Tropfen steht und auf den angrenzenden Objektträgern aufliegt.
- Trennen Sie die Objektträger vorsichtig. Vermeiden Sie es, das Pad zu zerreißen.
- Bereiten Sie die Agarose-Pads jeden Tag der Bildgebung frisch vor. Es wird empfohlen, bei der Vorbereitung auf die Bildgebung mehrere Pads herzustellen, aber nicht mehr als ein Pad gleichzeitig zu belichten. Freiliegende Ballen trocknen schnell aus.
- Geben Sie mit einer 20-μl-Pipette einen kleinen Tropfen (~7 μl) von 1 mM Levamisol in M9-Puffer in die Mitte des Agarose-Pads, um die Würmer zu lähmen. Verwenden Sie eine filtersterilisierte 100 mM Stammlösung von Levamisol, um die 1 mM Arbeitslösung herzustellen.
- Zur Herstellung von M9-Puffer werden 6 g Na2HPO4, 3 g KH2PO4, 0,5 g NaCl, 1 g NH4Cl und 1 l entionisiertes Wasser in einem geeigneten Behälter kombiniert (siehe Materialtabelle).
- Autoklavieren Sie die Lösung mit den Einstellungen des Flüssigkeitssterilisationszyklus und lassen Sie die Lösung vor der Verwendung auf RT abkühlen. Wenn Sie einen Behälter mit Schraubverschluss verwenden, lösen Sie den Schraubverschluss etwa eine Vierteldrehung, um eine gute Belüftung zu gewährleisten. Es wird ein Sterilisationszyklus empfohlen, der 30 Minuten lang auf 121 °C eingestellt ist.
- Wählen Sie mindestens 5 synchronisierte Würmer aus und berühren Sie vorsichtig den "Fuß" mit dem Levamisol-Tröpfchen, um alle Würmer zu übertragen. Verwenden Sie mindestens 5 Würmer pro Objektträger, da die Ausrichtung des Körpers dazu führen kann, dass der Darm die Keimbahn verdunkelt und die Visualisierung der Kalziumwelle in der Eizelle blockiert.
HINWEIS: Stellen Sie sicher, dass die Würmer schnell übertragen werden, da das Levamisol verdunstet, wenn es länger als 5 Minuten auf dem Agarose-Pad liegt. - Decken Sie das Agarose-Pad vorsichtig mit einem Deckglas ab. Überprüfen Sie die Position der Würmer, da das Deckglas dazu führen kann, dass sich die Würmer von der ursprünglichen Platzierung aus verteilen. Beschriften Sie den Objektträger mit entsprechenden Details, wie z. B. dem Namen des Wurmstamms (siehe Materialtabelle).
HINWEIS: Stellen Sie sicher, dass die Bildgebung so schnell wie möglich nach dem Aufsteigen von Würmern durchgeführt wird, da eine längere Exposition gegenüber Levamisol giftig ist.
4. Bildgebung von Calcium während der Befruchtung bei C. elegans
- Lokalisieren Sie die Würmer mit einem Objektiv mit geringer Leistung, z. B. 4x, am Mikroskop Ihrer Wahl. Verwenden Sie Hellfeldbeleuchtung.
- Verwenden Sie mindestens ein 20-faches Luftobjektiv, um die Kalziumwelle in den Eizellen deutlich sichtbar zu machen. Verwenden Sie ein Immersionsobjektiv von 40x oder mehr.
- Je nach verwendetem System ist die Anregungsintensität in der Regel ein fester Wert. Für eine optimale Visualisierung der Fluoreszenzreporter passen Sie die Verstärkung oder Belichtungszeit an.
HINWEIS: Kontinuierliches Licht mit hoher Intensität schädigt die Probe und ist schädlich für die Gesundheit der Würmer. Falls gewünscht, schalten Sie den Laser aus, während Sie auf ein Befruchtungsereignis warten, um eine kontinuierliche GFP-Beleuchtung zu vermeiden. Siehe die Kennzeichen eines Befruchtungsereignisses in den Abschnitten 4.8 - 4.9. - Stellen Sie sicher, dass der 488-nm-GFP-Kanal auf eine Laserleistung eingestellt ist, bei der das CaFE-Reportersignal gut sichtbar ist.
- Bei einem Nikon ECLIPSE Ti2 Laserscanning-Konfokal wird der CaFE-Reporter mit einer Laserleistung von 30 % für den GFP-Kanal und einer Verstärkung von 75 % detektiert. Die Lochblendengröße für das konfokale Laserscanning Nikon ECLIPSE Ti2 beträgt 30 μm.
- Für die konfokale Spinning-Disk der Andor Dragonfly mit der Zyla-Kamera ist der CaFE-Reporter mit einer Laserleistung von 30 % für den GFP-Kanal zu detektieren. Die Lochblendengröße für die konfokale Spinning-Scheibe der Andor Dragonfly beträgt 40 μm.
- Passen Sie die Lasereinstellungen und die Belichtungszeit für jede untersuchte Probe an.
- Bei Verwendung des EAG28 (CaFE; PH-mCherry) oder EAG25 (CaFE; H2B-mCherry) stellen Sie sicher, dass der rote Kanal auf eine Laserleistung eingestellt ist, bei der das mCherry-Signal zusätzlich zum GFP-Kanal gut sichtbar ist.
- Für das Nikon ECLIPSE Ti2 Laserscanning-Confocal wird der mCherry-Reporter mit einer Laserleistung von 15 für den RFP-Kanal und einer Verstärkung von 130 detektiert.
- Für die konfokale rotierende Scheibe Andor Dragonfly mit der Zyla-Kamera wird der mCherry-Reporter mit einer Laserleistung von 30 % für den RFP-Kanal detektiert.
- Passen Sie die Lasereinstellungen und die Belichtungszeit für jede untersuchte Probe an.
- Stellen Sie die Bildrate oder Bilder pro Sekunde (fps) so schnell ein, wie es das System zulässt.
HINWEIS: Der CaFE-Reporter wird in der Regel mit ca. 1 fps für die konfokale Nikon ECLIPSE Ti2 Laserscanning-Kamera und 10 fps für die konfokale Spinning-Disk-Kamera der Andor Dragonfly beobachtet. - Wählen Sie Zeitraffer- oder Zeitreihenprotokoll und passen Sie die gewünschten Einstellungen an. Nehmen Sie Zeitrafferbilder für 30 Minuten oder weniger auf, wobei die Wiederholungsoption auf '1' eingestellt ist.
HINWEIS: Wenn das Befruchtungsereignis vor Ablauf des Zeitintervalls auftritt, stoppen Sie das Video, um die Dateigröße zu erhalten. - Lokalisieren Sie die Würmer auf der Folie und wählen Sie einen Wurm aus, bei dem die proximale Gonade sichtbar ist. Der Darm und die Keimdrüse drehen sich im Wurm umeinander. Daher wird ein Gonadenarm oft vom Darm verdeckt, was die Visualisierung der Befruchtung verhindert. Wenn die -1-Eizelle verdeckt ist, wählen Sie einen anderen Wurm zur Visualisierung aus. Um die Wahrscheinlichkeit zu erhöhen, dass der ausgewählte Wurm einen sichtbaren Gonadenarm hat, montieren Sie mindestens 5 Würmer pro Objektträger. Verwenden Sie frühe Erwachsene am Tag 1, um die Befruchtung zu visualisieren.
- Untersuchen Sie jede -1 Eizelle in den Gonadenarmen des Wurms, um festzustellen, ob ein Befruchtungsereignis unmittelbar bevorsteht.
HINWEIS: Vor der Befruchtung, während der meiotischen Reifung, wandert der Kern in der -1-Eizelle in den hinteren Teil der Eizelle und die Kernhülle löst sich auf. Diese Ereignisse sind sowohl im Nomarksi- als auch im GFP-Kanal mit dem CaFE-Reporter sichtbar, und solche Ereignisse deuten darauf hin, dass der Eisprung innerhalb der nächsten ~10 Minuten stattfinden wird. - Nach dem Zusammenbruch der Kernhülle ordnet sich das Zytoskelett der proximalsten -1-Eizelle neu an, nimmt ein abgerundetes Aussehen an und trennt sich von der -2-Eizelle. Zu diesem Zeitpunkt steht der Eisprung unmittelbar bevor. Beginnen Sie sofort mit der Aufnahme des Videos, wenn diese Ereignisse beobachtet werden. Nehmen Sie Videos in einer Weitwinkel-Umgebung auf.
HINWEIS: Wenn die Bewegung des Zellkerns oder die Rundung der Eizelle nach 20 Minuten nicht sichtbar ist, ist es unwahrscheinlich, dass ein Eisprungereignis auftritt. Konzentrieren Sie sich auf einen anderen Wurm und/oder bereiten Sie eine frische Folie vor.
5. Quantifizierung von Calciumtransienten bei der Befruchtung von C. elegans
- Quantifizieren Sie Kalziumtransienten aus Befruchtungsvideos mit der Mikroskopsystemsoftware wie Nikon NIS Elements (hier beschrieben). ImageJ/FIJI eignen sich auch für die Bildanalyse11.
- Um die Änderung der Fluoreszenz von Befruchtungsvideos zu quantifizieren, verwenden Sie die folgenden Signalwerte: das Signal zum Zeitpunkt 0, das Signal aus dem interessierenden Frame und das Basissignal.
- Identifizieren Sie den Zeitpunkt und den Rahmen, der den ersten Fluoreszenzschub in der -1-Eizelle erfasst. Definieren Sie diesen Frame als Zeit 0.
- Um das Signal aus dem Frame of Interest zu erhalten, zeichnen Sie die Region of Interest (ROI) so, dass sie die folgenden Bereiche umfasst: die -1-Eizelle vor der Befruchtung, die Spermatheca und die neu befruchtete Eizelle in der Gebärmutter. Das Signal, das in der ROI angezeigt wird, wird als F1 bezeichnet.
HINWEIS: Stellen Sie sicher, dass der ROI den gesamten Bereich abdeckt, der von der Eizelle frequentiert wird. Der ROI sollte in jedem Frame der zu analysierenden Zeitrafferaufnahme identisch sein. - Definieren Sie einen leeren Bereich des Bildes ohne Fluoreszenz mit den gleichen Abmessungen wie der in Schritt 5.4 definierte ROI, um das Hintergrundsignal in jedem Frame zu berechnen. Subtrahieren Sie das Hintergrundsignal von jedem Frame.
- Um das Basissignal (F0) zu erhalten, lokalisieren Sie das Bild in mindestens 15 Bildern vor der Zeit 0. Notieren Sie bei gleichem ROI wie in Schritt 5.4 das Signal bei jedem Frame bis zum Zeitpunkt 0. Mittelung der Signale (mit Hintergrundsubtraktion) vom Startbild bis zum Zeitpunkt 0, um das Basissignal zu berechnen.
- Berechnen Sie die Änderung der Fluoreszenz (ΔF) mit der folgenden Formel:
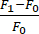 . F1 wird in jedem Frame als das Signal in der ROI ohne das Hintergrundsignal definiert. he F0 ist das gemittelte Basisfluoreszenzsignal von ≥15 Bildern vor der Zeit 0, wie in Abschnitt 5.6 berechnet.
. F1 wird in jedem Frame als das Signal in der ROI ohne das Hintergrundsignal definiert. he F0 ist das gemittelte Basisfluoreszenzsignal von ≥15 Bildern vor der Zeit 0, wie in Abschnitt 5.6 berechnet. - Bestimmen Sie die Änderungsrate zwischen den Bildern, indem Sie die Änderung der Fluoreszenz zwischen 2 Bildern subtrahieren und durch die Zeit dividieren.
6. RNAi in C. elegans
- RNAi-Platten vorbereiten
- Bereiten Sie die Platten gemäß Schritt 1.1 vor und fügen Sie die folgenden zusätzlichen Reagenzien hinzu, um den Gen-Knockdown über RNAi in Schritt 6.1.4 zu ermöglichen.
- 1 ml 1 M sterilfiltriertes Isopropyl β-d-1-thiogalactopyranosid (IPTG) (gelöst in deionisiertem Wasser) für eine Endkonzentration von 1 mM IPTG in das Medium geben (siehe Materialtabelle).
- 1 ml sterilfiltriertes Ampicillin mit 100 mg/ml (gelöst in entionisiertem Wasser) für eine Endkonzentration von 100 μg/ml Ampicillin in das Medium geben (siehe Materialtabelle). Alternativ können Sie 40 μg/ml Carbenicillin12 verwenden.
- Vorsichtig aber gründlich schwenken, um die Platten zu mischen und zu gießen, wie in den Schritten 1.1.4 - 1.1.5 beschrieben.
- Vorbereitung einer RNAi-gesättigten Kultur
- Berechnen Sie die Menge der gesättigten Kultur, indem Sie die Anzahl der benötigten ausgesäten RNAi-Platten mit 300 μl bei Verwendung von 60 mm x 15 mm Platten oder mit 100 μl bei Verwendung von 35 mm x 10 mm Platten multiplizieren.
- Mit einer sterilen Technik werden die gewünschten RNAi-Bakterien in LB (beschrieben in den Schritten 1.2.1-1.2.3) mit 100 μg/ml Ampicillin über Nacht bei 37 °C gezüchtet, bis sie gesättigt sind. Nehmen Sie einen Bakterienstamm mit dem leeren Vektor L4440 als Negativkontrolle und einen Bakterienstamm mit einer RNAi-Positivkontrolle (z. B. Ei-5) auf, um zu überprüfen, ob die RNAi funktioniert. Fügen Sie zusätzlich eine "leere" Bedingung hinzu, die nur LB enthält, um sicherzustellen, dass keine Kontamination im Lager-LB vorhanden ist.
- Zur Induktion der dsRNA-Expression wird sterilfiltriertes IPTG in einer Endkonzentration von 1 mM in die gesättigte Übernachtkultur gegeben, um die dsRNA-Expression zu induzieren, und vor der Aussaat mindestens 2 h 30 min bei 37 °C inkubieren. Alternativ können Sie die gesättigte Übernachtkultur vor der Induktion mit IPTG aus der Log-Phase zurückverdünnen.
HINWEIS: IPTG ist sowohl in den Platten als auch in der gesättigten Kultur enthalten, um eine optimale dsRNA-Expression zu gewährleisten. - Mit einer sterilen Technik werden 300 μl der induzierten Kultur auf die Mitte einer 60 mm x 15 mm großen RNAi-Platte (verfestigt) übertragen. Alternativ können Sie 100 μl gesättigte Kultur für eine 35 mm x 10 mm große Platte verwenden.
- Lassen Sie die Platten 1-3 Tage bei RT trocknen, bis der RNAi-Bakterienfleck nicht mehr flüssig ist. Stellen Sie sicher, dass die Platten trocken und frei von Verunreinigungen sind, bevor Sie sie verwenden. Lagern Sie die Platten bei 4 °C auf dem Kopf stehend für die zukünftige Verwendung.
HINWEIS: Verwenden Sie RNAi-Platten so schnell wie möglich, um einen optimalen Knockdown zu erzielen, da die RNAi-Wirksamkeit mit der Zeit abnimmt. Entsorgen, wenn >1 Monat alt.
- Wie in Abschnitt 2 beschrieben, synchronisieren Sie die Würmer auf RNAi-Platten.
- Überprüfen Sie die Würmer 1-2 Tage nach der Ei-Legen-Synchronisation auf Entwicklungsverzögerungen.
- Einige RNAi können Entwicklungsstörungen verursachen, wenn der Knockdown bei der Eiablage beginnt. Platzieren Sie bei Bedarf zu einem späteren Zeitpunkt Würmer auf den RNAi-Platten, z. B. L3.
- Wie in den Abschnitten 3 und 4 beschrieben, montieren Sie die Würmer und bereiten Sie die Bildgebung mit einem beliebigen Mikroskopsystem vor, das für den Nachweis von GFP-Fluoreszenz ausgerüstet ist.
7. Bildgebung von Calcium während der frühen Embryogenese bei C. elegans
- Synchronisieren Sie die Würmer, wie in Abschnitt 2 beschrieben. Verwenden Sie den EAG28-Stamm, der den CaFE-Reporter und einen pleckstrin-homologischen Domänen-mCherry-Marker kombiniert, der die Plasmamembrangrenze hervorhebt. Um befruchtete Embryonen ex utero sichtbar zu machen, schneiden Sie den Wurm entlang der Gebärmutter vorsichtig mit einer sterilen Spritzennadel auf, um die Embryonen freizusetzen.
- Bereiten Sie Würmer oder Embryonen für die Bildgebung vor, wie in Abschnitt 3 beschrieben, und befolgen Sie die in Abschnitt 4 beschriebenen Anweisungen zum Einstellen der Bildgebungsparameter.
- Zentrieren Sie den interessierenden Bereich auf die Gebärmutter des Wurms, um Embryonen oder ein gelegtes Ei zu visualisieren.
- Achten Sie bei Embryonen in utero darauf, dass sich die Gebärmutter nicht hinter dem Darm befindet, die Gebärmutter nicht überfüllt ist und dass die einzelnen Zellen im Embryo in der Fokalebene klar sind.
- Bei Eiern ex utero ist darauf zu achten, dass die Stadien der Zellen das Kommastadium des Wurmlebenszyklus nicht überschreiten.
- Wählen Sie Zeitraffer- oder Zeitreihenprotokoll und passen Sie die gewünschten Einstellungen an.
- Für eine optimale Visualisierung der Kalziumtransienten des Embryos erhöhen Sie die Zeiteinstellungen für längere Filme. Denken Sie daran, dass die ständige Exposition gegenüber dem Laser den Embryo über lange Zeiträume schädigt.
- Nehmen Sie Videos oder Bilder auf, indem Sie sich auf Zellen in Embryonen im gewünschten Zellstadium konzentrieren.
- Verwenden Sie für die Bildgebung ein 40-fach-Immersionsobjektiv oder mehr, obwohl die Transienten mit einem 20-fach-Luftobjektiv leicht erkannt werden können.
Ergebnisse
Unter Verwendung der in diesem Manuskript beschriebenen Protokolle wurden die dynamischen Muster der Kalziumsignalgebung bei der Befruchtung und Embryogenese bei C. elegans beobachtet.
Eine typische Befruchtungssequenz bei Würmern, die den CaFE-Reporter enthalten, ist in Abbildung 2 dargestellt. Um die Analyse zu erleichtern, wurde der EAG28-Stamm verwendet, der den CaFE-Reporter mit einem Transgen13 der Pleckstrin-Homologiedomäne (PH-mCherry) kombiniert. Der PH-mCherry-Marker lokalisiert an der Plasmamembran und ermöglicht eine einfachere Visualisierung von Zellgrenzen, insbesondere im Gonadenarm. Der EAG28-Stamm entstand durch Kreuzung des EAG16-Stammes, der den CaFE-Reporter enthält, mit dem OD70 PH-mCherry-Reporterstamm13. Bei C. elegans finden Eisprung und Befruchtung gleichzeitig statt. Der Zeitpunkt 0 der Befruchtung gilt als der erste Rahmen, in dem ein Fluoreszenzsignal in der ovulierenden Eizelle nachgewiesen wird (Abbildung 2). Ein heller Fluoreszenzausbruch tritt an der Stelle der Spermienfusion auf, sobald die Vorderkante der Eizelle in die Spermatheca, das Speicherorgan für Spermien7, eintritt. Das Signal erscheint, bevor der Eisprung abgeschlossen ist. Die Kalziumwelle ist zweiphasig, mit einem schnellen anfänglichen Ausbruch, gefolgt von einer Fluoreszenzwelle vom Eintrittspunkt zum gegenüberliegenden Pol. Die gesamte Eizelle fluoresziert in <30 s.
Beachten Sie, dass der CaFE-Reporter in einer einzigen Kopie vorliegt und das Signal daher nicht so hell ist wie bei anderen Transgenen. Ein Z-Stack-Zeitraffer wird während des Eisprungs nicht empfohlen, es sei denn, eine konfokale Spinning-Scheibe ist verfügbar und optimiert. Z-Stapel-Bilder können vor oder nach einem Film aufgenommen werden, um 3D-Veränderungen in der Gonadenmorphologie zu erfassen. Obwohl die Bilder hier mit einem konfokalen Mikroskop aufgenommen wurden, wurden die Transienten auch mit der Weitfeld-Fluoreszenzmikroskopie beobachtet, die häufiger und kostengünstiger ist als konfokal. Bei Verwendung eines Laserscanning-Konfoals beträgt die Bildrate ca. 1 fps. Bei Verwendung einer konfokalen rotierenden Scheibe beträgt die Bildrate 10 fps oder schneller. Das Hintergrundsignal des CaFE-Reporters reicht aus, um die reifenden Eizellen im Gonadenarm zu beleuchten. Bei der Befruchtung wird typischerweise ein 1,5- bis 2-facher Anstieg der GFP-Signalintensität beobachtet.
Für die Quantifizierung gibt es mehrere alternative Methoden. Zum Beispiel ist die Bildanalysesoftware Imaris, die von Oxford Instruments (der gleichen Muttergesellschaft wie das konfokale Spinning-Disk-Mikroskop Andor Dragonfly) entwickelt wurde, in der Lage, das Signal in jedem Bild nur von der transitierenden Eizelle zu quantifizieren, anstatt einen größeren ROI zu erzielen. Die Imaris-Software ist jedoch nicht kostenlos. Darüber hinaus wurde eine detaillierte Bildanalysestrategie zur Messung von Kalziumwellen in der Eizelle von Takayama, Fujita und Onami sehr detailliert beschrieben und verwendet ImageJ, das frei verfügbar ist14. Alternativ erfordern Kymographen zur Veranschaulichung der Welle des Fluoreszenzsignals über eine einzelne Eizelle eine umfangreichere Bildmanipulation als hier beschrieben, werden aber in Takayama und Onamibeschrieben 3.
Ein deutlicher Vorteil von C. elegans ist seine Fähigkeit, die Expression fast jedes Gens zu unterbinden, indem die Würmer mit Bakterien gefüttert werden, die dsRNA exprimieren, um endogene RNAiauszulösen 15,16. Für eine optimale dsRNA-Expression enthalten sowohl die RNAi-Platten als auch die gesättigte RNAi-Kultur IPTG. Die dsRNA wird hergestellt, indem das interessierende Gen mit flankierenden Promotoren exprimiert wird. Hier haben wir die Ahringer-Bibliothek verwendet, die auf den größten Teil des Wurmgenoms abzielt, indem wir 16.256 Bakterienstämmeverwenden 17. Alternativ wurde ein ORFeome von ~11.000 RNAi-Klonen vom Vidal-Labor unter Verwendung des Gateway-Systems erstellt und kann über Horizon Discovery18 erworben werden.
Um den Nutzen des CaFE-Reporters in Kombination mit RNAi zu demonstrieren, untersuchten wir den vorzeitigen Eisprung. Eine partielle Deletion in ipp-5 induziert ein doppeltes Ovulationsereignis, bei dem sowohl die -1-Eizelle als auch die -2-Eizelle während desselben Eisprungs in die Spermatheca gelangen19. IPP-5 ist eine Phosphatase, die auf IP3 wirkt und den Spiegel des IP3-Botenstoffs effektiv senkt. Wir fanden heraus, dass der RNAi-Knockdown von ipp-5 einen doppelten Ovulationsphänotyp auslöst, der dem der Mutante ähnelt.
Die Analyse des CaFE-Reporters während des doppelten Eisprungs, der durch den ipp-5-RNAi-Knockdown induziert wurde, enthüllte neue Aspekte der OET. Zunächst wurde ein Kalziumsignal unmittelbar nach dem Eintritt in die Spermathek in der führenden -1-Eizelle beobachtet, nicht jedoch in der nachfolgenden -2-Eizelle (Abbildung 3). Obwohl 2 Eizellen ovuliert werden, ist die -1-Eizelle immer noch die einzige Eizelle, die Anzeichen einer ordnungsgemäßen Reifung zeigt, insbesondere den Abbau der Kernhülle (NEBD). Diese Daten deuten darauf hin, dass die -2-Eizelle, obwohl sie sich in Gegenwart von Spermien befindet, nicht befruchtet werden kann, da sie noch nicht richtig gereift ist. Zweitens weist die -2-Eizelle eine verzögerte Kalziumwelle auf, typischerweise wenn die Eizelle die Spermatheca verlässt und in die Gebärmutter gelangt. Obwohl die Eizellreifung normalerweise eine Voraussetzung für den Eisprung ist, deuten diese Daten darauf hin, dass die Reifung der Eizelle und die Befruchtungsfähigkeit auch nach dem Eisprung noch stattfinden können. Die Visualisierung der verzögerten Kalziumwelle während des ipp-5-Knockdowns unterstreicht die Vorteile des CaFE-Reporters zur Abfrage der Befruchtungskompetenz und des Timings.
Darüber hinaus haben wir entdeckt, dass der CaFE-Reporter in gelegten Eiern nachweisbar ist und einzellige Kalziumtransienten während der frühen Embryogenese in C. elegans aufdeckt. Embryonen aus synchronisierten adulten EAG28 (CaFE; PH-mCherry)-Würmer wurden unter Verwendung der Protokolle aus den Abschnitten 1-5 mit der konfokalen Spinning-Scheibe der Andor Dragonfly unter Verwendung der Zyla-Kamera abgebildet (Abbildung 4). Die Kalziumtransienten sind auf einzelne Zellen beschränkt und benötigen weniger Zeit als die Befruchtungskalziumwelle (~9 s, n=11). Die Kalziumwelle während der Embryogenese scheint jedoch nicht zweiphasig zu sein. Calciumtransienten wurden vor dem 8-Zellen-Stadium nicht beobachtet. Bemerkenswert ist, dass Calcium bei C. elegans nicht in der Spaltfurche lokalisiert ist, wie es bei vielen anderen Organismen, einschließlich des Menschen und Xenopus 20,21,22, der Fall ist. Einzellige Calciumtransienten wurden gut nach der Gastrulation (~200 min nach der Befruchtung) und in gelegten Eiern beobachtet. Ein einzelner Embryo zeigt im Laufe der Zeit mehrere Kalziumtransienten, jedoch in verschiedenen Zellen und typischerweise in einer Zelle nach der anderen. Beachten Sie, dass eine kontinuierliche Exposition gegenüber der Laserstimulation die Embryonen schädigt. Es wird entweder eine geringere LED-/Laserleistung oder eine diskontinuierliche Belichtung empfohlen. Da die Kalziumwellen jedoch mit ~9 s relativ kurz sind, empfehlen wir nicht, langsamer als 2 s zwischen LED/Laser-Stimulationsereignissen zu sein.
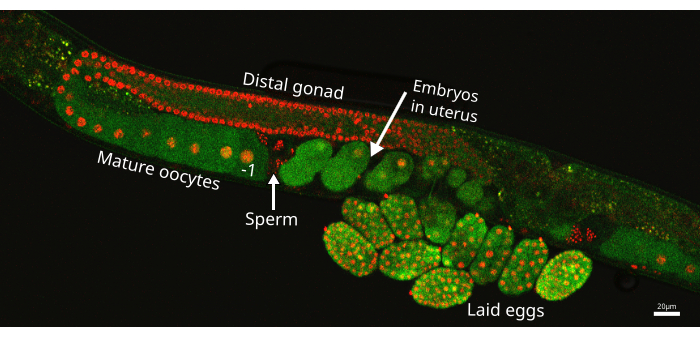
Abbildung 1: Gonadenarm von C. elegans . Bild eines Gonadenarms aus dem Wurmstamm EAG25, der den CaFE-Reporter GFP (grün) und den Histon-2B-mCherry-Marker (rot) zeigt, der die Zellkerne in der Keimbahn hervorhebt. Die proximale Eizelle wird mit der -1 direkt vor der Spermathek bezeichnet, in der die Spermien gelagert werden (Pfeil). Der Embryo, der sich am nächsten an der Spermatheca, aber in der Gebärmutter befindet, ist der jüngste Embryo. Das Bild wurde mit dem Nikon ECLIPSE Ti2 Laserscanning-Konfokal aufgenommen. Maßstabsleiste = 20 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
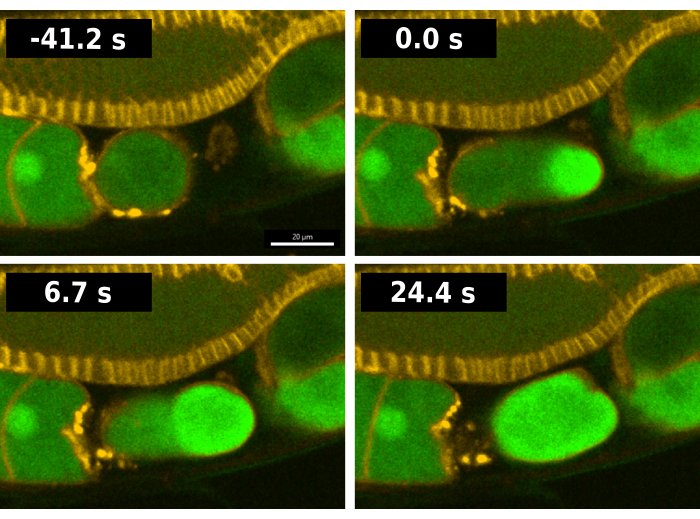
Abbildung 2: Visualisierung der Kalziumwelle während der Befruchtung. Zeitreihenbilder der Fluoreszenz, die intrazelluläres Kalzium während der Befruchtung im Wurmstamm EAG28 reflektiert. Der EAG28-Stamm enthält sowohl den CaFE-Reporter (grün) als auch eine Pleckstrin-Homologie-Domäne-mCherry-Fusion (gelb), die Plasmamembranen hervorhebt. Die Zeit 0 zeigt das erste Bild an, das einen deutlichen Anstieg des Fluoreszenzsignals in der Eizelle anzeigt. Die Zeit 24,4 s spiegelt das erste Bild wider, das die Fluoreszenz der gesamten Eizelle zeigt. Zeitreihenbilder wurden mit der andor Dragonfly rotierenden Scheibe konfokal mit einer Zyla-Kamera aufgenommen. Maßstabsleiste = 20 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
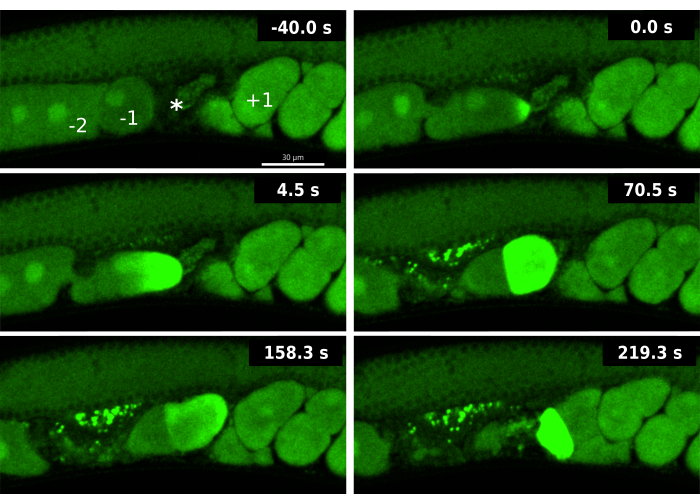
Abbildung 3: Kalziumwellen während eines ipp-5-RNAi-induzierten doppelten Ovulationsereignisses. Zeitrafferaufnahmen von sequentiellen Kalziumwellen der -1 Eizelle und der -2 Eizelle in einem EAG16-Stamm (CaFE in GFP grün), die ipp-5 RNAi ausgesetzt waren. Der -40,0 s-Rahmen enthält Markierungen für die -1 Eizelle (-1), die -2 Eizelle (-2), die Spermatheka (*) und die +1 Embryo (+1). Die Zeit 0 s zeigt die anfängliche Zunahme der Fluoreszenz der Kalziumwelle der -1-Eizelle. Die Zeit 4,5 s zeigt die Kalziumwelle, die sich in der -1-Eizelle zum entgegengesetzten Pol ausbreitet. Die Zeit 70,5 s zeigt die Ganzzellfluoreszenz in der -1-Eizelle, begleitet von der -2-Eizelle zu Studienbeginn des GFP-Signals; Beide Eizellen befinden sich in der Spermatheca. Die befruchtete -1-Eizelle und die unbefruchtete -2-Eizelle gelangen zum Zeitpunkt 158,3 s in die Gebärmutter. Bei 219,3 s zeigt die -2-Eizelle eine spätfluoreszierende Kalziumwelle. Die Bilder wurden mit der andor Dragonfly rotierenden Scheibe konfokal mit einer Zyla-Kamera aufgenommen. Maßstabsleiste = 30 μM Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
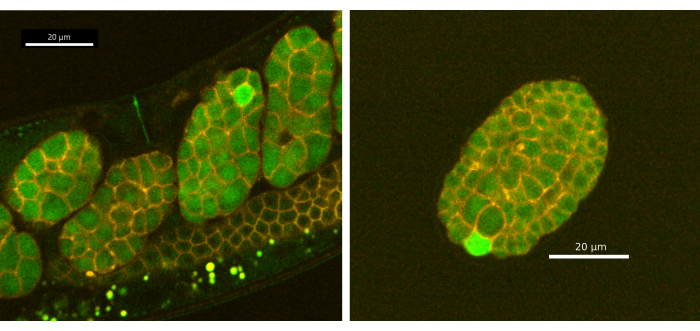
Abbildung 4: Einzellige Kalziumtransienten in der frühen Embryogenese. (A) Embryonen in utero und (B) ex utero (gelegte Eizelle) zeigten während der Embryogenese ein Fluoreszenzsignal in einer einzelnen Zelle. Die Bilder wurden mit der andor Dragonfly rotierenden Scheibe konfokal mit einer Zyla-Kamera aufgenommen. Maßstabsleiste = 20 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Ein einfaches Tool mit einem robusten Protokoll ist eine wirksame Kombination, um schwierige wissenschaftliche Fragen zu lösen. In dieser Arbeit werden Methoden zur Visualisierung von Calcium als leicht detektierbarem Proxy für die Befruchtung mit Hilfe des CaFE-Reporters vorgestellt. Derselbe Reporter bleibt bis in die frühe Embryogenese bestehen und ermöglicht auch die Visualisierung von Kalziumtransienten im weiteren Verlauf der Entwicklung. Die Kalzium-Signalübertragung dient als kritischer zweiter Botenstoff, der große Verschiebungen in der zellulären Funktion kennzeichnet, insbesondere für die Entwicklungsbiologie. In der Eizelle markiert die erste Burst-Phase der Kalziumwelle nicht nur den Zeitpunkt der Befruchtung, sondern auch den Ort der Spermienfusion3. Bei C. elegans wird die A-P-Achse auch durch den Ort der Spermienfusion bestimmt23. Die Möglichkeit, Kalzium in Eizellen und Embryonen sichtbar zu machen, ermöglicht daher die Untersuchung komplexer Fragestellungen, die für die Zell- und Entwicklungsbiologie von zentraler Bedeutung sind.
Die hier beschriebene Methode mit dem CaFE-Reporter sollte für Nematoden-Neulinge einfach genug sein. Bisherige Methoden zum Nachweis von Kalziumwellen in Oozyten von C. elegans beruhten auf Farbstoffinjektion 3,8,14. Obwohl diese Studien wichtig und aufschlussreich waren, ist die Farbstoffinjektion arbeitsintensiv und Injektionsgeräte sind nicht in jedem Labor verfügbar.
Die hier vorgestellten Protokolle wurden für gesunde Würmer optimiert. Um die Erfolgschancen zu maximieren, stellen Sie sicher, dass sich keine Kontamination auf dem Wurmmedium oder im Bakterienrasen befindet, auf dem sich die Würmer ernähren. Setzen Sie die Würmer keinen Stressbedingungen wie Temperaturen aus, da diese den Eisprung und die Befruchtung beeinträchtigen. Darüber hinaus sollten Kontrollen für die RNAi-Wirksamkeit in jedes Experiment einbezogen werden, da die Wirksamkeit mit der Zeit abnimmt. Verwenden Sie eine Positivkontrolle für die RNAi-Wirksamkeit wie Ei-5, die Embryonen, aber keine lebensfähigen Nachkommen erzeugt, da die Eierschalenbildung beeinträchtigt ist24. Darüber hinaus müssen die Bilderfassungsparameter für jedes Mikroskopiesystem optimiert werden. Unsere Spezifikationen sind hier als Referenz enthalten, es ist jedoch mit Abweichungen zu rechnen. Obwohl dieses System zytoplasmatisches Kalzium als Proxy für die Befruchtung leicht nachweist, stellt es nicht unbedingt ein echtes Spermien-Eizell-Fusionsereignis dar.
Die breite Palette an Werkzeugen und Mutanten, die innerhalb der Wurm-Community frei verteilt werden, erhöht den Nutzen des CaFE-Reporters. Der Reporter ist in das Wurmgenom integriert und kann leicht mit anderen mutierten oder Reporterstämmen von C. elegans gekreuzt werden25. Hier wird über die Erstellung von EAG25 berichtet, das den CaFE-Reporter exprimiert, mit einem Histon-H2B-mCherry-Marker, um Zellkerne sichtbar zu machen (Abbildung 1) und von EAG28 sowohl mit dem CaFE-Reporter als auch mit einem Pleckstrin-Homologie-Domain-mCherry-Marker, der die Zellperipherie hervorhebt (Abbildung 2)13,26. Beide Stämme helfen bei der Visualisierung von Zellen in der Keimbahn und während der Embryogenese. Darüber hinaus ist die Einrichtung von RNAi in
C. elegans haben in Verbindung mit dem CaFE-Reporter neue Erkenntnisse über die Befruchtungskompetenz gewonnen. Wie in Abbildung 3 der durch den ipp-5-RNAi-Knockdown induzierte doppelte Eisprung gezeigt wird, reicht das Vorhandensein von Spermien und einer ovulierten Eizelle nicht aus, um ein Befruchtungsereignis zu stimulieren.
Diese Ergebnisse deuten darauf hin, dass ein anderes Signal oder das Fehlen eines Inhibitors vorhanden sein muss, das die Befruchtung der Eizelle ermöglicht. Die vorzeitig ovulierte Eizelle zeigt eine verzögerte Kalzium-Transiente, wenn die Eizelle in die Gebärmutter wandert. Diese späte Kalziumwelle deutet darauf hin, dass die vorzeitig ovulierte -2-Eizelle mit der Zeit Befruchtungskompetenz entwickeln kann. Wir gehen davon aus, dass Studien zum Zeitpunkt der Befruchtung, insbesondere im Hinblick auf die Zell-Zell-Signalübertragung und -Regulation, durch den Einsatz des CaFE-Reporters unterstützt werden. Darüber hinaus persistiert der CaFE-Reporter bis in die Embryogenese und zeigt einzellige Kalziumtransienten. Dieser embryonale Kalzium-Signalweg wurde auch innerhalb der ersten 24 Stunden nach der Entwicklung des Zebrafisches berichtet27. Die Rolle der Kalziumtransienten ist unbekannt, aber das Vorhandensein deutet auf ein zelluläres Signalereignis während der Entwicklung hin, das noch nicht erforscht ist. Bemerkenswert ist, dass die Kalziumtransienten bei neu befruchteten Zygoten nicht beobachtet wurden. Daher lokalisiert sich Calcium nicht in der Spaltfurche, wie es bei mehreren anderen Organismen, einschließlich Xenopus und Menschen, dokumentiert wurde 20,21,22.
Wichtig ist, dass der CaFE-Reporter einfach genug ist, um von Studenten mit minimaler Schulung verwendet zu werden. Wir haben ein 1-Credit-CURE-Labor (course-based undergraduate research experience) für Biologiestudenten mit den hier beschriebenen Stämmen und Protokollen konzipiert und durchgeführt. Im Laufe eines Semesters traf sich die Klasse einmal pro Woche für 3 h oder zweimal pro Woche für jeweils 1 h 30 min. Die Schülerinnen und Schüler hatten die Wahl, alleine oder in 2er-Gruppen zu arbeiten. Jeder Student/jedes Paar wählte aus einer kuratierten Liste ein anderes Gen aus, das er untersuchen wollte. Sie führten RNAi gegen ihr ausgewähltes Gen im EAG28-Stamm durch und untersuchten Würmer auf Auswirkungen auf Fruchtbarkeit, Befruchtung und/oder Gonadenmorphologie. Basierend auf ihren Ergebnissen und ihrer Hintergrundlektüre anhand von Primärliteratur entwickelten die Schülerinnen und Schüler Hypothesen, die sie in nachfolgenden Experimenten überprüfen konnten. Dieses iterative Design war entscheidend für die Steigerung des Engagements der Studierenden28. Die Studierenden sammelten authentische Forschungserfahrungen und erwarben Fähigkeiten in Modellorganismen, genetischen Screenings und Fluoreszenzmikroskopie. Dank der einfachen Bedienung des CaFE-Reporters konnten auch Studenten ohne Forschungserfahrung erfolgreich sein. Danach äußerten die Studierenden mit überwältigender Mehrheit ihre Präferenz für das CURE-Format gegenüber traditionellen Laborkursen, wobei viele Studierende den Wunsch äußerten, mit der Forschung fortzufahren. Zusammengenommen helfen diese Werkzeuge und Protokolle sowohl bei der Bildung als auch bei der Erforschung früher Entwicklungsprozesse.
Offenlegungen
Die Autoren erklären keine konkurrierenden oder finanziellen Interessen.
Danksagungen
Das KSKG wurde durch ein Stipendium des Eunice Kennedy Shriver National Institute of Child Health and Human Development (R15HD111986) finanziert. Einige Stämme wurden vom CGC zur Verfügung gestellt, das vom NIH Office of Research Infrastructure Programs (P40 OD4010440) finanziert wird. Wir danken WormBase.
Materialien
| Name | Company | Catalog Number | Comments |
| Agar | Fisher Scientific | DF0140-07-4 | 2 kg; Powder dissolves easier than flakes |
| Agarose | MidSci | BE-A125 | 500 g |
| Alcohol lamp | Fisher Scientific | S13475 | Use with 95% ethanol |
| Ammonium chloride (NH4Cl) | Fisher Scientific | AAA1500030 | 250 g |
| Ampicillin | Fisher Scientific | BP1760-5 | 5 g |
| AMSCO 400 Series Small Steam Sterilizer | Steris Healthcare | N/A | |
| Bacto-peptone | Fisher Scientific | BP1420-500 | 500 g |
| Bacto-tryptone | Fisher Scientific | DF0123-17-3 | 500 g |
| Calcium chloride (CaCl2) | Fisher Scientific | C69-500 | 500 g |
| Cholesterol | Thermo Scientific | A11470.18 | 50 g |
| Dragonfly 200 spinning disk confocal | Oxford Instruments Andor | N/A | Used with Leica microscope |
| Fisherbrand Superfrost Cytogenics Microscope Slides | Fisher Scientific | 22-035-900 | 144 slides per pack |
| Flat Nose Pliers, Smooth Jaw | Home Depot | 305530604 | Ensure pliers are smooth jaw |
| Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Fisher Scientific | BP1755-10 | 10 g; dioxane-free |
| Laboratory tape | Fisher Scientific | 15-901-10R | 0.5 inch tape is used to tape microscope slides |
| Levamisole | Fisher Scientific | AC187870100 | 10 g |
| Magnesium sulfate (MgSO4) | Fisher Scientific | M63-500 | 500 g |
| Microscope cover glass | Fisher Scientific | 12541016 | 1 oz pack |
| Nikon ECLIPSE Ti2 laser scanning confocal | Nikon | N/A | |
| Nikon NIS Elements software | Nikon | N/A | Confocal |
| OP50 Escherichia coli | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Platinum Wire | TriTech | PT-9010 | |
| Potassium phosphate dibasic (K2HPO4) | Fisher Scientific | P288-500 | 500 g |
| Potassium phosphate monobasic (KH2PO4) | Fisher Scientific | AA1159436 | 500 g |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | 500 g |
| Sodium phosphate dibasic heptahydrate (Na2HPO4) | Fisher Scientific | S471-3 | 3 kg |
| Stereo microscope | Leica | KL300 LED | |
| Sterile Petri dish (35 mm x 10 mm) | CellTreat | 229638 | 960 Petri dishes per case |
| Sterile Petri dish (60 mm x 15 mm) | CellTreat | 229665 | 500 Petri dishes per case |
| Strain EAG16 spn-4p::jGCaMP7s::pie-1u | Caenorhabditis Genetics Center (CGC) | EAG16 | Created by Kim Guisbert Lab |
| Strain EAG25 spn-4p::jGCaMP7s::pie-1u; ujIs113 II. | Caenorhabditis Genetics Center (CGC) | EAG25 | Created by Kim Guisbert Lab |
| Strain EAG28 spn-4p::jGCaMP7s::pie-1u; unc-119(ed3) III; ltIs44 V. | Caenorhabditis Genetics Center (CGC) | EAG28 | Created by Kim Guisbert Lab |
| Strain JIM113 ujIs113 II [pie-1p::mCherry::H2B::pie-1 3'UTR + nhr-2p::his-24::mCherry::let-858 3'UTR + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | JIM113 | Created by E. Preston - Murray Lab |
| Strain OD70 unc-119(ed3) III; ltIs44 V [pie-1p::mCherry::PH(PLC1delta1) + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | OD70 | Created by Audhya/Oegema - Greenstein Lab |
| Tritech Worm Pick Handle | TriTech | TWPH1 | |
| Yeast extract | IBI Scientific | IB49160 | 500 g |
Referenzen
- Stein, P., Savy, V., Williams, A. M., Williams, C. J. Modulators of calcium signalling at fertilization. Open Biol. 10 (7), 200118(2020).
- McAvey, B. A., Wortzman, G. B., Williams, C. J., Evans, J. P. Involvement of calcium signaling and the actin cytoskeleton in the membrane block to polyspermy in mouse eggs. Biolo Reprod. 67 (4), 1342-1352 (2002).
- Takayama, J., Onami, S. The sperm TRP-3 channel mediates the onset of a Ca 2+ wave in the fertilized C. elegans oocyte. Cell Rep. 15 (3), 625-637 (2016).
- Yamamoto, I., Kosinski, M. E., Greenstein, D. Start me up: Cell signaling and the journey from oocyte to embryo in C. elegans. Dev Dyn. 235 (3), 571-585 (2006).
- Hubbard, E. J. A., Greenstein, D. The Caenorhabditis elegans gonad: A test tube for cell and developmental biology. Dev Dyn. 218 (1), 2-22 (2000).
- Korta, D. Z., Hubbard, E. J. A. Soma-germline interactions that influence germline proliferation in Caenorhabditis elegans. Dev Dyn. 239 (5), 1449-1459 (2010).
- Toperzer, K. M., Brennan, S. J., Carroll, D. J., Guisbert, E. A., Kim Guisbert,, S, K. Visualization of the biphasic calcium wave during fertilization in Caenorhabditis elegans using a genetically encoded calcium indicator. Biol Open. 12 (9), 059832(2023).
- Samuel, A. D., Murthy, V. N., Hengartner, M. O. Calcium dynamics during fertilization in C. elegans. BMC Dev Biol. 1 (1), 8(2001).
- Dana, H., et al. High-performance calcium sensors for imaging activity in neuronal populations and microcompartments. Nat Methods. 16 (7), 649-657 (2019).
- Golden, N. L., Plagens, R. N., Kim Guisbert, K. S., Guisbert, E. Standardized methods for measuring induction of the heat shock response in Caenorhabditis elegans. J Vis Exp. (161), e61030(2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamath, R., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Kachur, T. M., Audhya, A., Pilgrim, D. B. UNC-45 is required for NMY-2 contractile function in early embryonic polarity establishment and germline cellularization in C. elegans. Dev Biol. 314 (2), 287-299 (2008).
- Takayama, J., Fujita, M., Onami, S. In vivo live imaging of calcium waves and other cellular processes during fertilization in Caenorhabditis elegans. Bio Protoc. 7 (7), e2205(2017).
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391 (6669), 806-811 (1998).
- Conte, D., MacNeil, L. T., Walhout, A. J. M., Mello, C. C. RNA Interference in Caenorhabditis elegans. Curr Protoc Mol Biol. 109, 1-30 (2015).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421 (6920), 231-237 (2003).
- Reboul, J., et al. elegans ORFeome version 1.1: experimental verification of the genome annotation and resource for proteome-scale protein expression. Nat Genet. 34 (1), 35-41 (2003).
- Bui, Y. K., Sternberg, P. W. Caenorhabditis elegans inositol 5-phosphatase homolog negatively regulates inositol 1,4,5-triphosphate signaling in ovulation. Mol Biol Cell. 13 (5), 1641-1651 (2002).
- Muto, A., Kume, S., Inoue, T., Okano, H., Mikoshiba, K. Calcium waves along the cleavage furrows in cleavage-stage Xenopus embryos and its inhibition by heparin. J Cell Biol. 135 (1), 181-190 (1996).
- Paudel, S., Sindelar, R., Saha, M. Calcium signaling in vertebrate development and its role in disease. Int J Mol Sci. 19 (11), 3390(2018).
- Paudel, S., Yue, M., Nalamalapu, R., Saha, M. S. Deciphering the calcium code: A review of calcium activity analysis methods employed to identify meaningful activity in early neural development. Biomolecules. 14 (1), 138(2024).
- Goldstein, B., Hird, S. N. Specification of the anteroposterior axis in Caenorhabditis elegans. Development. 122 (5), 1467-1474 (1996).
- Johnston, W. L., Dennis, J. W. The eggshell in the C. elegans oocyte-to-embryo transition. Genesis. 50 (4), 333-349 (2012).
- Stevenson, Z. C., Moerdyk-Schauwecker, M. J., Jamison, B., Phillips, P. C. Rapid self-selecting and clone-free integration of transgenes into engineered CRISPR safe harbor locations in Caenorhabditis elegans. G3. 10 (10), Bethesda. 3775-3782 (2020).
- Zacharias, A. L., Walton, T., Preston, E., Murray, J. I. Quantitative differences in nuclear β-catenin and TCF pattern embryonic cells in C. elegans. PLoS Genet. 11 (10), e1005585(2015).
- Webb, S. E., Miller, A. L. Calcium signalling during zebrafish embryonic development. BioEssays. 22 (2), 113-123 (2000).
- Wiseman, E., Carroll, D. J., Fowler, S. R., Guisbert, E. Iteration in an inquiry-based undergraduate laboratory strengthens student engagement and incorporation of scientific skills. J Scholarsh Teach Learn. 20 (2), 99-112 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten