Method Article
C. elegansの受精および初期発生中のカルシウム過渡現象のin vivoでの可視化
要約
ここでは、モデル線虫 C.エレガンスの生殖系列で発現する遺伝的にコードされたカルシウムレポーターを使用して、受精および初期胚形成中のカルシウムを視覚化するためのプロトコルとツールを紹介します。
要約
カルシウムは、卵子から胚への移行(OET)および初期胚形成における重要なシグナル伝達分子です。雌雄同体の線虫 Caenorhabditis elegans は、透明で、20°Cで~23分ごとに1つの成熟卵子を生成する秩序立った生殖腺を持つため、OETの研究にいくつかのユニークな利点を提供します。 私たちは、遺伝子にコードされたカルシウムインジケーターjGCaMP7sを、生体内の受精の瞬間を蛍光で示すように改変しました。このレポーターを、 C.エレガンスの受精中のカルシウムの「CaFE」と名付けました。CaFEレポーターは、シングルコピーでセーフハーバー遺伝子座に組み込まれており、生理機能や繁殖力に大きな影響を与えず、受精時に強力なシグナルを発します。ここでは、CaFEレポーターをOETおよび胚発生を解剖するための in vivo ツールとして利用するための一連のプロトコルを示します。寄生虫の同期方法、RNAiノックダウンの影響を調べる方法、イメージング用の寄生虫のマウント方法、卵子や胚のカルシウムの可視化などの方法が含まれています。さらに、このタイプの分析を支援するための追加のワーム株の生成についても紹介します。CaFEレポーターによる受精のタイミングの可視化の有用性を実証し、 ipp-5 がRNAiによって標的とされると二重排卵が起こり、最初の卵子のみが即時受精を受けることを報告します。さらに、初期胚形成における単一細胞カルシウム過渡現象の発見がここで報告されており、CaFEレポーターが初期発生まで持続することを示しています。重要なことに、ワームのCaFEレポーターは、コースベースの学部研究(CURE)の研究室のクラスに組み込むために使用できるほどシンプルです。CaFEレポーターは、生殖腺の秩序化と線虫のRNAiの容易さと相まって、内部受精と初期胚形成の調節に必要な細胞間ダイナミクスの調査を促進します。
概要
受精は新しいライフサイクルの始まりを示していますが、受精の正確な瞬間を定義することは困難です。受精の保存された特徴は、精子融合の直後に卵子全体にカルシウムの波が押し寄せることです1。カルシウム波の性質は、頻度と速度の点で種によって異なりますが、ほぼすべての生物が受精後に細胞内カルシウムの一時的な増加を示します。カルシウム波は、多精子、卵子の活性化、およびその他の重要な細胞イベント2のブロックに重要な役割を果たします。カルシウム波は精子融合の部位から始まるため、カルシウムは受精のマーカーとして機能します3。
Caenorhabditis elegansは、初期発生を研究するための理想的なモデル生物です。ワームは透明な雌雄同体で、遺伝的に扱いやすい4。受精の研究にとって最も重要なことは、C.エレガンスの成虫は、厳密に順序付けられた生殖腺5を使用して卵子を継続的に産生することです。実際、卵母細胞は、体細胞が成人期に有糸分裂後であるため、生成される唯一の新しい細胞です6。図1は、発達中の卵母細胞の2つの対称的なU字型のチューブで構成される生殖腺を強調しています。精子(精子の貯蔵器官)に最も近い卵子は、-1卵子と呼ばれます。この線虫生殖腺の組み立てライン設計は、若年成人では20°Cで~23分ごとに成熟した卵子を精子に排卵します7。その後、新たに受精した胚は、単一の外陰部に横たわる前に共有子宮に移動します。
C.エレガンスの受精中のカルシウムを視覚化する以前の技術は、カルシウム感受性色素のマイクロインジェクションに依存していました3,8。精子-卵子融合時のカルシウム波をより簡単に見るために、jGCaMP7sに基づく遺伝的にコードされたカルシウム指示薬を、クラスター化された規則的に間隔を空けた短い回文反復(CRISPR)7,9を使用して、セーフハーバー遺伝子座にシングルコピーで挿入した。このレポーターは、C.エレガンスの受精中のカルシウムのCaFEと呼ばれていました。レポーターは、生理機能や繁殖力に重大な欠陥を示していません。
ここでは、CaFEレポーターを使用して C.エレガン スの卵子と胚のカルシウム波を可視化するためのプロトコルを示します。CaFEレポーターは、RNA干渉(RNAi)やmCherry生殖腺マーカーなど、線虫コミュニティで利用可能な無数のツールと組み合わせることで、体内受精イベントの制御、特に受精能力と受精のタイミングに関する調査を容易にします。さらに、CaFEレポーターは初期発生まで持続し、胚形成を調査するためのユニークなツールです。
プロトコル
1.C.エレガンスのメンテナンス
- 線虫増殖培地(NGM)プレート調製
- 3 gの塩化ナトリウム(NaCl)、2.5 gのバクトペプトン、20 gの寒天、および1 Lの脱イオン水を4 Lフラスコに入れます( 材料の表を参照)。オートクレーブする前に、フラスコの上部をアルミホイルで覆います。
- 液体滅菌サイクル設定を使用して滅菌するオートクレーブ。滅菌サイクルを121°Cで30分間に設定することをお勧めします。
- 混合物を~50°Cまで、室温(RT)で約20分間冷却します。
- 滅菌済み 1 M KH2PO4 25 mL(水に溶解、pH = 6)、滅菌済み 1 M CaCl2 1 mL(水に溶解)、滅菌済み 1 mL の 1 M MgSO4 (水に溶解)、および 5 mg/mL コレステロール 1 mL(100% エタノールに溶解)を添加し、添加するたびに穏やかに渦巻かせます( 材料の表を参照)。
- 滅菌技術を使用して、混合物の一部を滅菌300mLビーカーに移し、注ぎやすくします。温かいメディアを注ぎ、プレートの底を覆います。
注:60 mm x 15 mmの滅菌ペトリプレート( 材料の表を参照)のプレートあたり約10 mLは、100枚のプレートを生成します。或いは、35 mm x 10 mmの滅菌ペトリ皿を使用してもよい、この場合、プレート当たり~4 mLは~250プレートを生成する。 - NGMプレートをRTで少なくとも1日間乾燥させてから、バクテリアを播種します。プレートは逆さまにして4°Cで保管し、後で使用してください。
- OP50細菌芝生の準備
- 2 Lフラスコに5 gの酵母抽出物、10 gのバクトトリプトン、5 gのNaCl、および1 Lの脱イオン水を組み合わせて、ルリアブロス(LB)を作ります( 材料の表を参照)。攪拌板と攪拌子を使用して、溶解するまで混合します。
- LBをボトルとオートクレーブに分注し、液体滅菌サイクル設定を使用します。滅菌サイクルを121°Cで30分間に設定することをお勧めします。スクリューキャップ付きの容器を使用する場合は、オートクレーブサイクル中に適切に換気するために、スクリューキャップを4分の1回転緩めます。
- ブロスをRTまで冷ましてから使用してください。
- 滅菌技術を使用して、250 mLの三角フラスコに50 mLのLBにOP50細菌を接種し、振とうしながら37°Cで一晩増殖させます。
- 滅菌技術を使用して、300 μLの飽和培養物を60 mm x 15 mmのNGMプレートの中心に移します。35 mm x 10 mmプレートの場合、100 μLの飽和培養が推奨されます。
- OP50細菌スポットが液体でなくなるまで、プレートをRTで1〜3日間乾燥させます。プレートが乾燥し、汚染されていないことを確認してから使用してください。プレートは、将来の使用のために4°Cで保管してください。プレートを逆さまに収納すると、プレート表面の結露が減少します。
- ウォームピック構造
- シルバーカラーをワームピックハンドル( 材料表を参照)のヘッドに向かって緩め、4つのセクションがバラバラになるようにゆっくりと緩めます。
- はさみを使用して1.5インチ(親指の長さ)のプラチナワイヤー( 材料の表を参照)を切り取り、4つのセクションの中央に置きます。ハンドルから約1インチ突き出ます。
- 襟を締めながら、ワイヤーを中央に保持します。4つのセクションが中央で合わさり、プラチナ線を所定の位置に保持します。
- フラットノーズペンチを使用してワイヤーを慎重に形作ります。虹色または円弧の形は、ピッキングに最適な傾向があります。
- フラットノーズプライヤーで繰り返し締め付けて、ワイヤーの端に約2〜3mmの「足」を作成します。この「足」を使用してワームを拾い上げ、「足」をプレートの表面に対して平らに保ちます。
注意: clamp 強すぎると、プラチナ線が切断されます。フラットノーズプライヤー(または同等のもの)を使用して鋭いエッジを作成しないようにして、ワームやプレートに穴を開けないようにします。
- ウォームハンドリング
- 鉛筆のようにワームピックを持ち、95%エタノールランプの炎にワイヤーを置いて「足」を明るいオレンジ色になるまで滅菌します。
- OP50播種プレートを解剖スコープのステージに置き、蓋を取り外します。ピックの「足」をバクテリアの芝生の領域にそっと触れ、穏やかにスワイプしてバクテリアを底に集めます。ピックに付着したバクテリア(「ベトベト」と呼ばれます)は、ワームを拾うための接着剤として機能します。
- ワームのプレートを解剖スコープステージに移動し、ピックが他の表面に触れないようにしながら蓋を取り外します。ピックの「足」をワームにすばやくそっと触れて、ピックを拾います。この技術により、複数のウォームをプレート間で同時に移動させることができます。
- ピックの「足」を新しいプレートの種がまかれていない部分にそっと触れて、ワームを移します。通常、これはセクション1.4.2でバクテリアが採取されたのと同じプレートです。
- 汚染を避けるために、各プレートおよび/またはワーム株の間のピックを滅菌し、滅菌後にピックを冷まします。
注意: ワームのホットピックに触れると、動物にストレスを与えたり殺したりします。ワームピックは、バクテリアの芝生に触れると冷えて「グー」を拾い上げ、ワームを移します。
- OP50 Escherichia coli 細菌を播種したNGMプレート(ステップ1.1)で線虫を20°Cに維持します(ステップ1.2)。生理機能を変える可能性のある飢餓を避けるために、ワームピック(ステップ1.3-1.4)を使用して数人の成虫を新鮮なプレート(60 mm x 15 mm)に移すことにより、週に2〜3回ワームを通過させます。
注: C.エレガンス 株とOP50細菌は、Caenorhabditis Genetics Center(CGC)を通じて入手できます。
2. C.エレガンス の発生同期
- 時限産卵を使用して線虫の年齢同期を行います(ここで説明)。教室やその他の目的で多数のワームが必要な場合は、Golden et al.10で説明されているように漂白剤の同期を実行します。
- 成虫をプラチナワイヤーピック(セクション1で説明)を使用して、新しい播種OP50プレートに移します。ワームの数は、実験の必要性によって決まります。1日目のワームは1時間あたり約3個の卵を産みます。したがって、10匹のワームは1時間で約30個の卵を生成します。できれば、成虫の初日(1日目)にワームを使用してください。
- 約1時間後、成虫をプレートから取り出し、卵だけを残します。
注:誤ってワームを置き去りにすることは、よくあるエラーです。残ったワームは卵を産み続け、同期を乱します。線虫は、20°Cでインキュベートすると、産卵から約72時間後に1日目の成虫に成熟します。
3. イメージング用ウォームの取り付け
- 受精イベントをイメージングするときは、1日目の成人を使用して視覚化の機会を最大化します。
注: 1 日目の成人では、卵子は ~23 分ごとに受精します。しかし、排卵は年齢とともに遅くなり、典型的な雌雄同体は4日目以降に産卵を停止します。 - 3 gのアガロースを100 mLの水に混合することにより、3%アガロースの水溶液を調製します。アガロース溶液が十分に溶解するまで、マイクロ波でアガロース溶液を加熱します。固化した溶液を再利用するには、完全に溶解するまで加熱します。数回使用した後、新しい溶液を生成します。
- スライドの長軸を横切るように1層のラボテープを持つ他の2つの顕微鏡スライドの間に、イメージング用のブランク顕微鏡スライドを置きます( 材料の表を参照)。テープはアガロースパッドのスペーサーを作成し、均一な分布と均一な幅を可能にします。3枚のスライドが長辺が接触するように一列に並んでいることを確認します(空白の顕微鏡スライドに隣接するテープ付きの2枚のスライド)。
- 加熱した3%アガロース溶液(~100-150 μL)を、1 mLピペットを使用して、テープで留めたスライドの間にある顕微鏡スライドの中央に滴下します。
- 新しい顕微鏡スライドをアガロース液滴にすばやく置き、新しいスライドがアガロース液滴のあるスライドに対して垂直になり、隣接するテープスライドを横切って静止することを確認します。
- スライドをそっと分離します。パッドを引き裂かないでください。
- アガロースパッドは、イメージングの毎日新鮮に準備します。撮像準備の際はパッドを複数枚作ることをお勧めしますが、同時に複数のパッドを露出させることはお控えください。露出したパッドは急速に乾きます。
- 20 μLのピペットを使用して、M9バッファー中の1 mMレバミゾールをアガロースパッドの中心に少量(~7 μL)加え、線虫を麻痺させます。レバミゾールのフィルター滅菌済み100 mMストック溶液を使用して、1 mMの作業溶液を調製します。
- M9緩衝液を作製するには、6 gのNa2HPO4、3 gのKH2PO4、0.5 gのNaCl、1 gのNH4Cl、および1 Lの脱イオン水を適切な容器に混ぜ合わせます( 材料の表を参照)。
- 液体滅菌サイクル設定を使用して溶液をオートクレーブし、溶液をRTまで冷ましてから使用してください。スクリューキャップ付きの容器を使用する場合は、適切な換気のためにスクリューキャップを約4分の1回転緩めます。滅菌サイクルを121°Cで30分間に設定することをお勧めします。
- 少なくとも5つの同期ワームを選び、「足」をレバミゾール液滴にそっと触れて、すべてのワームを移します。体の向きによって腸が生殖細胞系を不明瞭にし、卵子のカルシウム波の視覚化を妨げる可能性があるため、スライドごとに少なくとも5つの線虫を使用してください。
注意: アガロースパッドに5分以上放置するとレバミゾールが蒸発するため、ワームが迅速に移動していることを確認してください。 - アガロースパッドをカバースリップで慎重に覆います。カバースリップが初期配置からワームの分散を引き起こす可能性があるため、ワームの位置を確認してください。顕微鏡スライドに、線虫の名前など、適切な詳細をラベル付けします( 材料の表を参照)。
注:レバミゾールへの長時間の曝露は有毒であるため、ワームをマウントした後はできるだけ早くイメージングが行われることを確認してください。.
4.C.エレガンスの受精中のカルシウムのイメージング
- 選択した顕微鏡で、4倍などの低倍率の対物レンズを使用して線虫を見つけます。明視野照明を使用します。
- 卵子のカルシウム波をはっきりと視覚化するために、最低20倍の空気対物レンズを使用してください。40倍以上の没入型対物レンズを使用してください。
- 使用するシステムにもよりますが、通常、加振強度は固定値です。蛍光レポーターを最適に視覚化するには、ゲインまたは露光時間を調整します。
注:連続した高強度の光はサンプルを損傷し、ワームの健康に有害です。必要に応じて、受精イベントを待つ間はレーザーをオフにして、継続的なGFP照明を避けます。セクション4.8〜4.9の受精イベントの特徴を参照してください。 - 488 nm GFPチャンネルが、CaFEレポーター信号が見やすいレーザー出力に設定されていることを確認します。
- Nikon ECLIPSE Ti2 レーザー走査型共焦点の場合、GFP チャネルのレーザー出力 30% とゲイン 75 で CaFE レポーターを検出します。Nikon ECLIPSE Ti2レーザー走査型共焦点のピンホールサイズは30μmです。
- Andor Dragonfly スピニングディスク共焦点レンズの場合、Zyla カメラを使用して、GFP チャネルのレーザー出力 30% で CaFE レポーターを検出します。Andor Dragonflyスピニングディスク共焦点のピンホールサイズは40μmです。
- 検査するすべてのサンプルのレーザー設定と露光時間を調整します。
- EAG28(CaFE;PH-mCherry)またはEAG25(CaFE;H2B-mCherry)ひずみの場合、GFPチャンネルに加えてmCherry信号が簡単に見えるレーザー出力に赤チャンネルが設定されていることを確認します。
- Nikon ECLIPSE Ti2 レーザースキャニング共焦点の場合、RFP チャネルのレーザー出力 15 とゲイン 130 で mCherry レポーターを検出します。
- Andor Dragonfly スピニングディスク共焦点レンズを Zyla カメラで、RFP チャンネルのレーザー出力 30% で mCherry レポーターを検出します。
- 検査するすべてのサンプルのレーザー設定と露光時間を調整します。
- フレームレートまたはフレーム/秒(fps)をシステムが許可する速度で設定します。
注:CaFEレポーターは通常、Zylaカメラを使用したNikon ECLIPSE Ti2レーザースキャニング共焦点レンズで約1 fps、Andor Dragonfly回転ディスク共焦点で約10 fpsで観察されます。 - タイムラプスまたは時系列プロトコルを選択し、必要な設定に調整します。タイムラプス画像を30分以内に撮影し、repeatsオプションを「1」に設定します。
注:時間間隔が完了する前に受精イベントが発生した場合は、ビデオを停止してファイルサイズを保持します。 - スライド上の線虫の位置を確認し、近位生殖腺が見える線虫を選択します。腸と生殖腺は、ワーム内で互いにねじれています。そのため、生殖腺の腕は腸によって隠されることが多く、受精の視覚化が妨げられます。-1 卵子が隠れている場合は、視覚化するために別の線虫を選択します。選択したワームが目に見える生殖腺アームを持つ確率を高めるには、スライドごとに最低5つのワームをマウントします。1日目の早い成体を使用して、受精を視覚化します。
- 線虫の生殖腺腕の各-1卵子を調べて、受精イベントが差し迫っているかどうかを判断します。
注:受精前、減数分裂成熟中に、-1卵子の核は卵母細胞の後ろに移動し、核膜は溶解します。これらのイベントは、CaFEレポーターを使用してNomarksiチャネルとGFPチャネルの両方で確認でき、このようなイベントは、排卵が次の~10分以内に発生することを示しています。 - 核膜が破壊された後、最も近位の-1卵子の細胞骨格が再配置され、丸みを帯びた外観になり、-2卵子から分離します。この時点で、排卵が差し迫って起こります。これらのイベントが観察された場合は、すぐにビデオの録画を開始してください。広視野で動画を撮影する。
注:20分後に核の動きや卵子の丸みが見えない場合、排卵イベントが発生する可能性は低いです。別のワームに焦点を当てるか、新しいスライドを準備します。
5. C. elegans の受精におけるカルシウム過渡現象の定量
- Nikon NIS Elements(ここで説明)などの顕微鏡システムソフトウェアを使用して、受精ビデオからカルシウム過渡現象を定量します。ImageJ/FIJIは、画像解析にも適しています11。
- 受精ビデオの蛍光の変化を定量化するには、時間 0 の信号、対象のフレームからの信号、およびベースライン信号の信号値を使用します。
- -1卵子で発生する蛍光の最初のバーストを捕捉する時間とフレームを特定します。このフレームを時間 0 として定義します。
- 対象フレームからシグナルを取得するには、受精前の-1卵子、精子、および子宮内で新たに受精した卵子の領域を含む関心領域(ROI)を描画します。ROIに表示される信号はF1と呼ばれます。
注:ROIが卵子によって輸送される全領域をカバーしていることを確認してください。ROIは、分析対象のタイムラプス記録のすべてのフレームで同一である必要があります。 - 各フレームの背景信号を計算するために、手順 5.4 で定義した ROI と同じ寸法を使用して、蛍光のない画像の空白領域を定義します。すべてのフレームから背景信号を減算します。
- ベースライン信号 (F0) を取得するには、時間 0 の少なくとも 15 フレーム前にフレームを配置します。手順 5.4 と同じ ROI を使用して、時間 0 までの各フレームの信号に注意してください。開始フレームから時間 0 までの信号を平均化 (バックグラウンド減算あり) して、ベースライン信号を計算します。
- 次の式を使用して蛍光の変化(ΔF)を計算します:
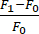 。F1 は、バックグラウンド信号を含まない ROI の信号として各フレームで定義されます。F0 は、セクション 5.6 で計算した、時間 0 の ≥15 フレーム前の平均ベースライン蛍光シグナルです。
。F1 は、バックグラウンド信号を含まない ROI の信号として各フレームで定義されます。F0 は、セクション 5.6 で計算した、時間 0 の ≥15 フレーム前の平均ベースライン蛍光シグナルです。 - 2 つのフレーム間の蛍光の変化を差し引き、時間で割ることにより、フレーム間の変化率を決定します。
6. C.エレガンスにおけるRNAi
- RNAiプレートの調製
- ステップ1.1に従ってプレートを調製し、ステップ6.1.4でRNAiによる遺伝子ノックダウンを可能にするために、以下の試薬を追加します。
- 1 mLの1 M滅菌ろ過イソプロピルβ-d-1-チオガラクトピラノシド(IPTG)(脱イオン水に溶解)を加えて、培地中の最終濃度を1 mM IPTGにします( 材料の表を参照)。
- 1 mLの滅菌ろ過アンピシリン(脱イオン水に溶解)を1 mL加えて、培地中の最終濃度を100 μg/mLにします( 材料の表を参照)。あるいは、40 μg/mL のカルベニシリン12 を使用します。
- 手順1.1.4〜1.1.5で説明されているように、プレートを混合して注ぐために、穏やかに、しかし完全に渦を巻いて注ぎます。
- RNAi飽和培養液の調製
- 必要な播種RNAiプレートの数に、60 mm x 15 mmプレートを使用する場合は300 μL、35 mm x 10 mmプレートを使用する場合は100 μLを掛けて、飽和培養量を計算します。
- 滅菌技術を使用して、100 μg/mL のアンピシリンを含む LB (ステップ 1.2.1-1.2.3 で説明) で目的の RNAi 細菌を 37 °C で飽和するまで一晩増殖します。L4440エンプティベクターをネガティブコントロールとして含む細菌株と、RNAiポジティブコントロールを含む細菌株( 卵-5など)を含めて、RNAiが機能していることを確認します。さらに、LBのみを含む「空白」条件を含めて、ストックLBに汚染がないことを確認します。
- 滅菌ろ過したIPTGを最終濃度1 mMまで飽和オーバーナイト培養液に添加してdsRNA発現を誘導し、播種前に37°Cで少なくとも2時間30分間インキュベートします。あるいは、IPTGで誘導する前に、飽和した一晩の培養物を対数期から希釈します。
注:IPTGは、最適なdsRNA発現のためにプレートと飽和培養物の両方に含まれています。 - 滅菌技術を使用して、誘導した培養物300μLを60 mm x 15 mmのRNAiプレート(固化)の中心に移します。あるいは、35 mm x 10 mmプレートに100 μLの飽和培養物を使用します。
- プレートをRTで1〜3日間乾燥させ、RNAi細菌の斑点が液体でなくなるまで待ちます。プレートが乾燥し、汚染されていないことを確認してからご使用ください。プレートは4°Cを逆さまにして保管し、将来の使用に備えてください。
注:RNAiの有効性は時間の経過とともに低下するため、最適なノックダウンのためにできるだけ早くRNAiプレートを使用してください。生後>1か月の場合は廃棄します。
- セクション2で説明したように、線虫をRNAiプレートに同期させます。
- 産卵同期の1〜2日後に、発生遅延がないかワームを確認します。
- 一部のRNAiは、ノックダウンが産卵から始まるときに発生障害を引き起こす可能性があります。必要に応じて、L3などの後の段階で線虫をRNAiプレートに置きます。
- セクション3および4で説明したように、線虫をマウントし、GFP蛍光を検出するために装備された任意の顕微鏡システムを使用してイメージングの準備をします。
7. C.エレガンスの初期胚形成期カルシウムのイメージング
- セクション 2 の説明に従ってワームを同期します。CaFEレポーターと、原形質膜境界を強調するpleckstrin-homology domain-mCherryマーカーを組み合わせたEAG28株を使用します。あるいは、 子宮外で受精した胚を視覚化するには、滅菌注射針を使用して子宮に沿って線虫をそっと切り開き、胚を放出します。
- セクション 3 で説明されているように、イメージング用の線虫または胚を準備し、イメージング パラメーターの設定については、セクション 4 で説明されているのと同じ手順に従います。
- 対象領域を線虫の子宮の中央に配置して、胚または産卵を視覚化します。
- 子宮内の胚の場合、子宮が腸の後ろにないこと、子宮が密集していないこと、および胚の個々の細胞が焦点面で明確であることを確認してください。
- 子宮外卵子の場合、細胞の段階が線虫のライフサイクルのコンマ段階を超えていないことを確認してください。
- タイムラプスまたは時系列プロトコルを選択し、必要な設定に調整します。
- 胚のカルシウム過渡現象を最適に視覚化するには、長時間の動画の時間設定を増やします。レーザーに絶えずさらされると、長期間にわたって胚に害を及ぼすことに注意してください。
- 目的の細胞段階の胚の細胞に焦点を当てて、ビデオや画像をキャプチャします。
- イメージングには40倍以上の液浸対物レンズを使用しますが、過渡現象は20倍の空気対物レンズを使用して簡単に検出できます。
結果
この原稿で概説したプロトコルを使用して、受精および胚形成におけるカルシウムシグナル伝達の動的パターンが C.エレガンスで観察されました。
CaFEレポーターを含む線虫の典型的な受精配列を 図2に示します。解析を容易にするために、CaFEレポーターとプレクストリンホモロジードメイン(PH-mCherry)導入遺伝子13を組み合わせたEAG28株を用いた。PH-mCherryマーカーは原形質膜に局在し、特に生殖腺アームの細胞境界の可視化を容易にします。EAG28株は、CaFEレポーターを含むEAG16株とOD70 PH-mCherryレポーター13株を交配することによって作成されました。 C.エレガンスでは、排卵と受精が同時に起こります。受精の時間0は、排卵細胞で蛍光シグナルが検出される最初のフレームと見なされます(図2)。精子融合部位では、卵母細胞の前縁が精子の貯蔵器官である精子7に入るとすぐに、明るい蛍光のバーストが起こります。排卵が完了する前に信号が表示されます。カルシウム波は二相性で、最初のバーストが急速に発生し、その後、入口から反対側の極に向かって蛍光の波が続きます。卵子全体が<30秒で蛍光になります。
CaFEレポーターは1つのコピーにあるため、シグナルは他の導入遺伝子ほど明るくないことに注意してください。排卵期のZスタックタイムラプスは、スピニングディスク共焦点が利用可能で最適化されていない限り、推奨されません。Zスタック画像は、生殖腺の形態の3D変化をキャプチャするために、ムービーの前または後に取得できます。ここでの画像は共焦点顕微鏡で撮影されましたが、トランジェントは、共焦点よりも一般的で手頃な価格の広視野蛍光顕微鏡を使用しても観察されています。レーザースキャンコンフォカルを使用すると、フレームレートは約1fpsになります。回転するディスク共焦点を使用すると、フレームレートは10fps以上になります。CaFEレポーターのバックグラウンドシグナルは、生殖腺腕の成熟した卵子を照らすのに十分です。受精時には、通常、GFPシグナル強度の1.5〜2倍の増加が観察されます。
定量にはいくつかの代替方法があります。例えば、Oxford Instruments社(Andor Dragonflyスピニングディスク共焦点顕微鏡と同じ親会社)が開発した画像解析ソフトウェア「Imaris」は、各フレームのシグナルをトランジットする卵子のみから定量化する機能を備えており、ROIは大きくなりません。ただし、Imarisソフトウェアは無料ではありません。また、卵子のカルシウム波を測定するための詳細な画像解析戦略は、高山、藤田、大波によって非常に詳細に説明されており、自由に利用可能なImageJを使用しています14。あるいは、単一の卵子を横切る蛍光シグナルの波を示すためのキモグラフは、ここで述べるよりも広範な画像操作を必要とするが、高山と大波3で説明されている。
C.エレガンスの明確な利点は、dsRNAを発現する細菌を内因性RNAi15,16をトリガーする細菌をワームに供給することにより、ほぼすべての遺伝子の発現をノックダウンする能力です。最適なdsRNA発現のために、RNAiプレートと飽和RNAi培養物の両方にIPTGが含まれています。dsRNAは、目的の遺伝子を隣接するプロモーターで発現させることによって産生されます。ここでは、16,256の細菌株17を使用して線虫ゲノムの大部分を標的とするAhringerライブラリを使用しました。また、~11,000個のRNAiクローンからなるORFeomeは、Gatewayシステムを使用してVidalラボによって作成され、Horizon Discovery18を通じて購入することができます。
RNAiと組み合わせたCaFEレポーターの有用性を実証するために、早漏を調べました。 ipp-5 の部分的な欠失は、-1卵子と-2卵子の両方が同じ排卵中に精子に入る二重排卵イベントを誘発します19。IPP-5は、IP3に作用し、IP3セカンドメッセンジャーのレベルを効果的に低下させるホスファターゼです。 その結果、ipp-5 のRNAiノックダウンは、変異体と同様の二重排卵表現型を誘発することを発見しました。
ipp-5 RNAiノックダウンによって誘発された二重排卵中のCaFEレポーターの分析により、OETの新たな側面が明らかになりました。まず、カルシウムシグナルは、先行する-1卵子の精子に入るとすぐに観察されましたが、末尾の-2卵子には観察されませんでした(図3)。2つの卵子が排卵されますが、-1卵子は依然として適切な成熟の兆候を示す唯一の卵子であり、特に核膜(NEBD)の崩壊を示します。これらのデータは、-2卵子が精子の存在下にあるにもかかわらず、まだ適切に成熟していないため、受精する能力がないことを示唆しています。第二に、-2卵子は、通常、卵母細胞が子宮の精子から出るときに、遅延したカルシウム波を示します。卵子の成熟は通常、排卵の前提条件ですが、これらのデータは、卵子の成熟と受精能力が排卵後も起こり得ることを示唆しています。ipp-5ノックダウン中の遅延カルシウム波の視覚化は、受精能力とタイミングを調査するCaFEレポーターの利点を強調しています。
さらに、CaFEレポーターは産卵中に検出可能であり、C.エレガンスでは初期胚形成における単一細胞カルシウム過渡現象を明らかにすることを発見しました。同期した1日目の成人EAG28(CaFE;PH-mCherry)ワームは、セクション1〜5のプロトコルを使用して、Zylaカメラを使用したAndor Dragonfly回転ディスク共焦点でイメージングしました(図4)。カルシウムの過渡現象は単一細胞に限定され、受精カルシウム波よりも完了までの時間が短くなります(~9秒、n = 11)。しかし、胚形成中のカルシウム波は二相性ではないようです。カルシウムの過渡性は、8細胞期の前には観察されませんでした。注目すべきは、カルシウムは、ヒトやXenopus 20,21,22を含む他の多くの生物に見られるように、C.エレガンスの卵割溝に局在しないことです。単一細胞カルシウムの過渡現象は、原腸陥入後(受精後~200分)および産卵卵で観察されました。1つの胚は、時間の経過とともに複数のカルシウム過渡現象を示しますが、異なる細胞で、通常は一度に1つの細胞で発生します。レーザー刺激に継続的にさらされると、胚が損傷することに注意してください。LED/レーザー出力を低くするか、不連続露光をお勧めします。ただし、カルシウム波は~9秒と比較的短いため、LED/レーザー刺激イベント間の2秒より遅いことはお勧めしません。
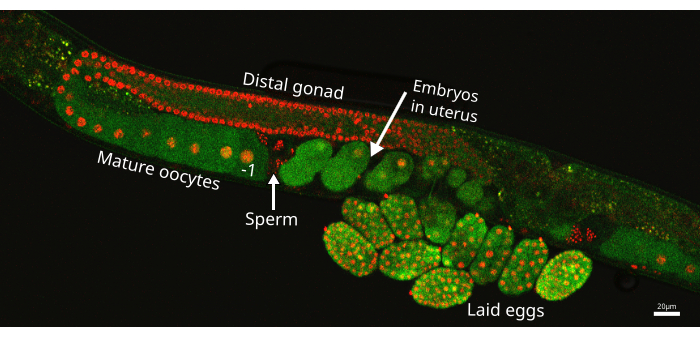
図1: C.エレガンス 生殖腺腕。 EAG25線虫株の生殖腺アームの1本の画像で、CaFEレポーターGFP(緑)と生殖細胞系の核を強調するヒストン2B-mCherryマーカー(赤)を示しています。近位卵子は、精子が保存されている精子の直前に-1で表されます(矢印)。精子に最も近いが子宮内にある胚が最新の胚です。画像は、Nikon ECLIPSE Ti2レーザースキャニングコンフォカルを使用して撮影されました。スケールバー = 20 μm。 この図の拡大版を表示するには、ここをクリックしてください。
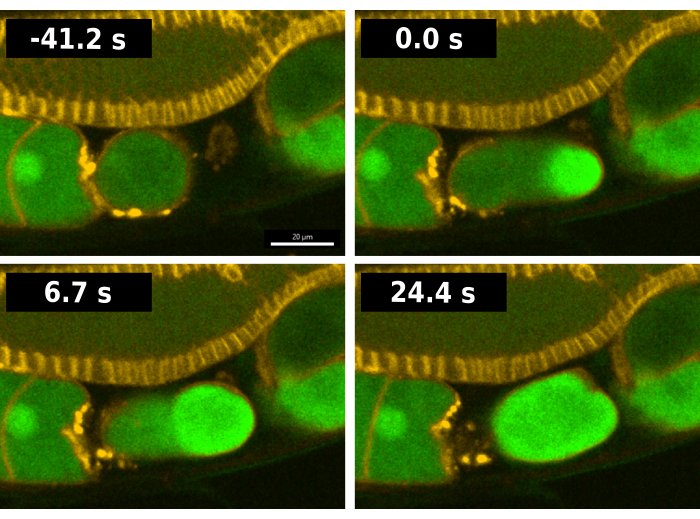
図2:受精中のカルシウム波の可視化。 EAG28線虫株の受精中の細胞内カルシウムを反映した蛍光の時系列画像。EAG28株には、CaFEレポーター(緑)と、原形質膜を強調表示するプレックストリンホモロジードメイン-mCherry融合(黄色)の両方が含まれています。時間0は、卵子の蛍光シグナルの明らかな増加を示す最初のフレームを示します。時間 24.4 秒は、卵子全体の蛍光を示す最初のフレームを反映しています。時系列画像は、Andor Dragonfly回転円盤共焦点とZylaカメラを使用して撮影されました。スケールバー = 20 μm。 この図の拡大版を表示するには、ここをクリックしてください。
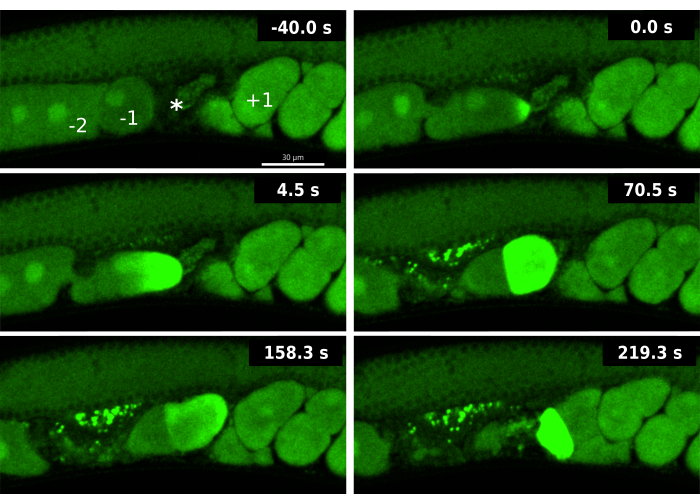
図3:ipp-5 RNAiによる二重排卵イベント中のカルシウム波。 ipp-5 RNAiに曝露したEAG16株(GFPグリーンのCaFE)における-1卵子と-2卵子の連続カルシウム波のタイムラプス画像。-40.0 s フレームには、-1 個の卵子 (-1)、-2 個の卵子 (-2)、精子 (*)、+1 個の胚 (+1) のラベルが含まれています。時間0秒は、-1卵子のカルシウム波の蛍光の初期増加を示します。時間4.5秒は、カルシウム波が-1卵子の反対側の極に向かって広がっていることを示しています。時間 70.5 秒は、ベースライン GFP シグナルで -1 卵子と -2 卵子の全細胞蛍光を示します。両方の卵子は精子にあります。受精した-1卵子と未受精の-2卵子は、158.3秒の時間に子宮に入り、219.3秒で、-2卵子は遅い蛍光カルシウム波を示します。画像は、Andor Dragonfly回転円盤共焦点とZylaカメラを使用して撮影されました。スケールバー= 30 μM この図の拡大版を表示するには、ここをクリックしてください。
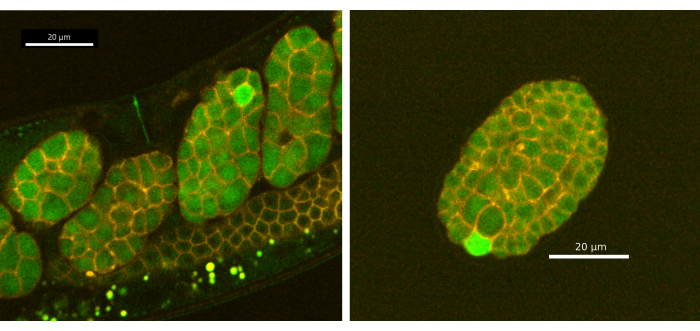
図4:初期胚形成における単一細胞カルシウム過渡現象。 (A) 子宮内の 胚と(B) 子宮外 (産卵)は、胚形成中に単一細胞で蛍光シグナルを示しました。画像は、Andor Dragonfly回転円盤共焦点とZylaカメラを使用して撮影されました。スケールバー = 20 μm。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
堅牢なプロトコルを備えたシンプルなツールは、困難な科学的問題に取り組むための強力な組み合わせです。ここでは、CaFEレポーターを使用して、受精の容易に検出可能なプロキシとしてカルシウムを視覚化する方法を紹介します。この同じレポーターは、初期胚形成まで持続し、カルシウムの過渡現象をさらに発生にまで可視化することも可能にします。カルシウムシグナル伝達は、特に発生生物学において、細胞機能の大きな変化を区切る重要なセカンドメッセンジャーとして機能します。卵子では、カルシウム波の最初の破裂期は、受精のタイミングだけでなく、精子融合の部位も示しています3。 C.エレガンスでは、A-P軸は精子融合の部位によっても決定される23。したがって、卵母細胞や胚のカルシウムを可視化する能力は、細胞生物学や発生生物学の中心となる複雑な問題を調査することを可能にします。
ここでCaFEレポーターを使用して説明する方法は、線虫の初心者にとって十分に簡単なはずです。C.エレガンスの卵子のカルシウム波を検出する以前の方法は、色素注入に依存していました3,8,14。これらの研究は重要で啓発的なものでしたが、色素注入は労働集約的であり、注入装置はすべてのラボで利用できるわけではありません。
ここで紹介するプロトコルは、健康な線虫向けに最適化されています。成功の可能性を最大限に高めるには、ワームの培地やワームが餌を与えるバクテリアの芝生に汚染がないことを確認してください。ワームを温度などのストレス条件にさらさないでください、これらは排卵と受精に影響を与えるからです。さらに、RNAiの有効性のコントロールは、時間の経過とともに有効性が低下するため、すべての実験に含める必要があります。胚を生成する 卵-5のようなRNAiの有効性のポジティブコントロールを使用しますが、卵殻の形成が損なわれるため、生存可能な子孫はいません24。さらに、画像キャプチャパラメータは、各顕微鏡システムに対して最適化する必要があります。ここでは参考として弊社仕様を記載しておりますが、乖離が予想されます。このシステムは、受精の代理として細胞質カルシウムを容易に検出しますが、必ずしも真正な精子-卵子融合イベントを表すわけではありません。
線虫コミュニティ内で自由に配布されるさまざまなツールと変異体は、CaFEレポーターの有用性を高めます。レポーターは線虫ゲノムに統合され、他の変異体またはレポーターC.エレガンス株25に容易に交配される。ここで報告されているのは、CaFEレポーターをヒストンH2B-mCherryマーカーで発現させて核を可視化するEAG25(図1)と、CaFEレポーターと細胞末梢を強調表示するプレックストリンホモロジードメイン-mCherryマーカー(図2)の両方を持つEAG28の作成です13,26。どちらの系統も、生殖細胞系および胚形成中の細胞の可視化を支援します。さらに、RNAiの施設は
C.エレガンスをCaFEレポーターと一緒に使用すると、受精能力に関する新たな洞察が明らかになりました。図3のipp-5 RNAiノックダウンによって誘発される二重排卵に示されているように、精子と排卵細胞の存在は、受精イベントを刺激するには不十分です。
これらの結果は、卵子が受精することを可能にする別のシグナル、または阻害剤の不在が存在する必要があることを示しています。早産卵した卵子は、卵子が子宮に移動するときに一過性の遅延カルシウムを示します。この後期カルシウム波は、早期に排卵した-2卵子が時間とともに受精能力を発達させることを示唆しています。受精のタイミング、特に細胞間シグナル伝達と制御に関する研究は、CaFEレポーターの使用によって支援されると期待しています。さらに、CaFEレポーターは胚発生まで持続し、単一細胞のカルシウム過渡現象を示します。この胚性カルシウムシグナル伝達は、ゼブラフィッシュの発生後24時間以内にも報告されました27。カルシウム過渡現象の役割は不明ですが、その存在は、まだ調査されていない発生中の細胞シグナル伝達イベントを示しています。特に、カルシウムの過渡性は、新たに受精した接合子では観察されませんでした。したがって、カルシウムは、Xenopusやヒト20,21,22を含む他のいくつかの生物で文書化されているように、卵割溝に局在しません。
重要なのは、CaFEレポーターは、最小限のトレーニングで学部生が使用できるほど簡単であるということです。私たちは、ここに記載されている系統とプロトコルを使用して、生物学の学生向けに1単位のCURE(コースベースの学部研究経験)ラボを設計し、実行しました。学期中、クラスは週に1回3時間、または週に2回、それぞれ1時間30分で集まりました。生徒たちは、一人で作業するか、2人のグループで作業するかの選択肢を与えられました。各学生/ペアは、厳選されたリストから研究する異なる遺伝子を選択しました。彼らは、EAG28株で選択した遺伝子に対してRNAiを実行し、生殖能力、受精、および/または生殖腺の形態に対する線虫の影響を調べました。その結果と一次文献を使用した背景の読み物に基づいて、学生はその後の実験で検証できる仮説を立てました。この反復的なデザインは、学生のエンゲージメントを高めるために重要でした28。学生たちは、モデル生物、遺伝子スクリーニング、蛍光顕微鏡法などの本格的な研究経験を積み、スキルを習得しました。CaFEレポーターの使い勝手の良さもあって、研究経験のない学生でも成功することができました。その後、従来の研究室の授業よりもCURE形式を好む学生が圧倒的に多く、研究を続けたいという意欲を示す学生も多くいました。これらのツールとプロトコルを組み合わせることで、初期の発達過程に関する教育と研究の両方を支援します。
開示事項
著者は、競合する利益や金銭的な利益を宣言しません。
謝辞
KSKGは、ユーニス・ケネディ・シュライバー国立小児保健発達研究所(R15HD111986)からの助成金によって資金提供されました。一部の株は、NIH研究インフラストラクチャープログラム局(P40 OD4010440)が資金提供するCGCによって提供されました。WormBase に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Agar | Fisher Scientific | DF0140-07-4 | 2 kg; Powder dissolves easier than flakes |
| Agarose | MidSci | BE-A125 | 500 g |
| Alcohol lamp | Fisher Scientific | S13475 | Use with 95% ethanol |
| Ammonium chloride (NH4Cl) | Fisher Scientific | AAA1500030 | 250 g |
| Ampicillin | Fisher Scientific | BP1760-5 | 5 g |
| AMSCO 400 Series Small Steam Sterilizer | Steris Healthcare | N/A | |
| Bacto-peptone | Fisher Scientific | BP1420-500 | 500 g |
| Bacto-tryptone | Fisher Scientific | DF0123-17-3 | 500 g |
| Calcium chloride (CaCl2) | Fisher Scientific | C69-500 | 500 g |
| Cholesterol | Thermo Scientific | A11470.18 | 50 g |
| Dragonfly 200 spinning disk confocal | Oxford Instruments Andor | N/A | Used with Leica microscope |
| Fisherbrand Superfrost Cytogenics Microscope Slides | Fisher Scientific | 22-035-900 | 144 slides per pack |
| Flat Nose Pliers, Smooth Jaw | Home Depot | 305530604 | Ensure pliers are smooth jaw |
| Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Fisher Scientific | BP1755-10 | 10 g; dioxane-free |
| Laboratory tape | Fisher Scientific | 15-901-10R | 0.5 inch tape is used to tape microscope slides |
| Levamisole | Fisher Scientific | AC187870100 | 10 g |
| Magnesium sulfate (MgSO4) | Fisher Scientific | M63-500 | 500 g |
| Microscope cover glass | Fisher Scientific | 12541016 | 1 oz pack |
| Nikon ECLIPSE Ti2 laser scanning confocal | Nikon | N/A | |
| Nikon NIS Elements software | Nikon | N/A | Confocal |
| OP50 Escherichia coli | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Platinum Wire | TriTech | PT-9010 | |
| Potassium phosphate dibasic (K2HPO4) | Fisher Scientific | P288-500 | 500 g |
| Potassium phosphate monobasic (KH2PO4) | Fisher Scientific | AA1159436 | 500 g |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | 500 g |
| Sodium phosphate dibasic heptahydrate (Na2HPO4) | Fisher Scientific | S471-3 | 3 kg |
| Stereo microscope | Leica | KL300 LED | |
| Sterile Petri dish (35 mm x 10 mm) | CellTreat | 229638 | 960 Petri dishes per case |
| Sterile Petri dish (60 mm x 15 mm) | CellTreat | 229665 | 500 Petri dishes per case |
| Strain EAG16 spn-4p::jGCaMP7s::pie-1u | Caenorhabditis Genetics Center (CGC) | EAG16 | Created by Kim Guisbert Lab |
| Strain EAG25 spn-4p::jGCaMP7s::pie-1u; ujIs113 II. | Caenorhabditis Genetics Center (CGC) | EAG25 | Created by Kim Guisbert Lab |
| Strain EAG28 spn-4p::jGCaMP7s::pie-1u; unc-119(ed3) III; ltIs44 V. | Caenorhabditis Genetics Center (CGC) | EAG28 | Created by Kim Guisbert Lab |
| Strain JIM113 ujIs113 II [pie-1p::mCherry::H2B::pie-1 3'UTR + nhr-2p::his-24::mCherry::let-858 3'UTR + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | JIM113 | Created by E. Preston - Murray Lab |
| Strain OD70 unc-119(ed3) III; ltIs44 V [pie-1p::mCherry::PH(PLC1delta1) + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | OD70 | Created by Audhya/Oegema - Greenstein Lab |
| Tritech Worm Pick Handle | TriTech | TWPH1 | |
| Yeast extract | IBI Scientific | IB49160 | 500 g |
参考文献
- Stein, P., Savy, V., Williams, A. M., Williams, C. J. Modulators of calcium signalling at fertilization. Open Biol. 10 (7), 200118 (2020).
- McAvey, B. A., Wortzman, G. B., Williams, C. J., Evans, J. P. Involvement of calcium signaling and the actin cytoskeleton in the membrane block to polyspermy in mouse eggs. Biolo Reprod. 67 (4), 1342-1352 (2002).
- Takayama, J., Onami, S. The sperm TRP-3 channel mediates the onset of a Ca 2+ wave in the fertilized C. elegans oocyte. Cell Rep. 15 (3), 625-637 (2016).
- Yamamoto, I., Kosinski, M. E., Greenstein, D. Start me up: Cell signaling and the journey from oocyte to embryo in C. elegans. Dev Dyn. 235 (3), 571-585 (2006).
- Hubbard, E. J. A., Greenstein, D. The Caenorhabditis elegans gonad: A test tube for cell and developmental biology. Dev Dyn. 218 (1), 2-22 (2000).
- Korta, D. Z., Hubbard, E. J. A. Soma-germline interactions that influence germline proliferation in Caenorhabditis elegans. Dev Dyn. 239 (5), 1449-1459 (2010).
- Toperzer, K. M., Brennan, S. J., Carroll, D. J., Guisbert, E. A., Kim Guisbert, ., S, K. Visualization of the biphasic calcium wave during fertilization in Caenorhabditis elegans using a genetically encoded calcium indicator. Biol Open. 12 (9), 059832 (2023).
- Samuel, A. D., Murthy, V. N., Hengartner, M. O. Calcium dynamics during fertilization in C. elegans. BMC Dev Biol. 1 (1), 8 (2001).
- Dana, H., et al. High-performance calcium sensors for imaging activity in neuronal populations and microcompartments. Nat Methods. 16 (7), 649-657 (2019).
- Golden, N. L., Plagens, R. N., Kim Guisbert, K. S., Guisbert, E. Standardized methods for measuring induction of the heat shock response in Caenorhabditis elegans. J Vis Exp. (161), e61030 (2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamath, R., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Kachur, T. M., Audhya, A., Pilgrim, D. B. UNC-45 is required for NMY-2 contractile function in early embryonic polarity establishment and germline cellularization in C. elegans. Dev Biol. 314 (2), 287-299 (2008).
- Takayama, J., Fujita, M., Onami, S. In vivo live imaging of calcium waves and other cellular processes during fertilization in Caenorhabditis elegans. Bio Protoc. 7 (7), e2205 (2017).
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391 (6669), 806-811 (1998).
- Conte, D., MacNeil, L. T., Walhout, A. J. M., Mello, C. C. RNA Interference in Caenorhabditis elegans. Curr Protoc Mol Biol. 109, 1-30 (2015).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421 (6920), 231-237 (2003).
- Reboul, J., et al. elegans ORFeome version 1.1: experimental verification of the genome annotation and resource for proteome-scale protein expression. Nat Genet. 34 (1), 35-41 (2003).
- Bui, Y. K., Sternberg, P. W. Caenorhabditis elegans inositol 5-phosphatase homolog negatively regulates inositol 1,4,5-triphosphate signaling in ovulation. Mol Biol Cell. 13 (5), 1641-1651 (2002).
- Muto, A., Kume, S., Inoue, T., Okano, H., Mikoshiba, K. Calcium waves along the cleavage furrows in cleavage-stage Xenopus embryos and its inhibition by heparin. J Cell Biol. 135 (1), 181-190 (1996).
- Paudel, S., Sindelar, R., Saha, M. Calcium signaling in vertebrate development and its role in disease. Int J Mol Sci. 19 (11), 3390 (2018).
- Paudel, S., Yue, M., Nalamalapu, R., Saha, M. S. Deciphering the calcium code: A review of calcium activity analysis methods employed to identify meaningful activity in early neural development. Biomolecules. 14 (1), 138 (2024).
- Goldstein, B., Hird, S. N. Specification of the anteroposterior axis in Caenorhabditis elegans. Development. 122 (5), 1467-1474 (1996).
- Johnston, W. L., Dennis, J. W. The eggshell in the C. elegans oocyte-to-embryo transition. Genesis. 50 (4), 333-349 (2012).
- Stevenson, Z. C., Moerdyk-Schauwecker, M. J., Jamison, B., Phillips, P. C. Rapid self-selecting and clone-free integration of transgenes into engineered CRISPR safe harbor locations in Caenorhabditis elegans. G3. 10 (10), 3775-3782 (2020).
- Zacharias, A. L., Walton, T., Preston, E., Murray, J. I. Quantitative differences in nuclear β-catenin and TCF pattern embryonic cells in C. elegans. PLoS Genet. 11 (10), e1005585 (2015).
- Webb, S. E., Miller, A. L. Calcium signalling during zebrafish embryonic development. BioEssays. 22 (2), 113-123 (2000).
- Wiseman, E., Carroll, D. J., Fowler, S. R., Guisbert, E. Iteration in an inquiry-based undergraduate laboratory strengthens student engagement and incorporation of scientific skills. J Scholarsh Teach Learn. 20 (2), 99-112 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved