Method Article
Visualizzazione in vivo dei transitori di calcio durante la fecondazione e lo sviluppo precoce in C. elegans
In questo articolo
Riepilogo
Qui, presentiamo protocolli e strumenti per visualizzare il calcio durante la fecondazione e l'embriogenesi precoce utilizzando un reporter di calcio geneticamente codificato espresso nella linea germinale del nematode modello C. elegans.
Abstract
Il calcio è un'importante molecola di segnalazione durante la transizione da ovocita a embrione (OET) e l'embriogenesi precoce. Il nematode ermafrodita Caenorhabditis elegans offre diversi vantaggi unici per lo studio dell'OET in quanto è trasparente e ha una gonade ordinata che produce un ovocita maturo ogni ~23 minuti a 20 °C. Abbiamo modificato l'indicatore di calcio geneticamente codificato jGCaMP7s per indicare in modo fluorescente il momento della fecondazione all'interno di un organismo vivente. Abbiamo chiamato questo reporter "CaFE" per il calcio durante la fecondazione in C. elegans. Il reporter CaFE è stato progettato in un locus di approdo sicuro in un'unica copia, non ha alcun impatto significativo sulla fisiologia o sulla fecondità e produce un segnale robusto dopo la fecondazione. Qui, viene presentata una serie di protocolli per l'utilizzo del reporter CaFE come strumento in vivo per sezionare l'OET e l'embriogenesi. Includiamo metodi per sincronizzare i vermi, esaminare gli effetti del knockdown dell'RNAi, montare vermi per l'imaging e visualizzare il calcio negli ovociti e negli embrioni. Inoltre, presentiamo la generazione di ulteriori ceppi di vermi per aiutare in questo tipo di analisi. Dimostrando l'utilità del reporter CaFE per visualizzare i tempi della fecondazione, riportiamo che la doppia ovulazione si verifica quando ipp-5 è bersaglio dell'RNAi e che solo il primo ovocita subisce una fecondazione immediata. Inoltre, qui viene riportata la scoperta di transienti di calcio a singola cellula durante l'embriogenesi precoce, dimostrando che il reporter CaFE persiste nello sviluppo precoce. È importante sottolineare che il reporter CaFE nei vermi è abbastanza semplice da utilizzare per l'incorporazione nelle lezioni di laboratorio di ricerca universitaria (CURE). Il reporter CaFE, insieme alla gonade ordinata e alla facilità dell'RNAi nei vermi, facilita l'indagine sulla dinamica cellula-cellula necessaria per regolare la fecondazione interna e l'embriogenesi precoce.
Introduzione
La fecondazione segna l'inizio di un nuovo ciclo vitale, ma definire il momento preciso della fecondazione è impegnativo. Una caratteristica conservata della fecondazione è un'ondata di calcio attraverso l'ovocita subito dopo la fusione degli spermatozoi1. Sebbene la natura dell'onda di calcio, in termini di frequenza e velocità, differisca tra le specie, quasi tutti gli organismi mostrano un aumento transitorio del calcio intracellulare dopo la fecondazione. L'onda di calcio svolge un ruolo fondamentale nel blocco della polispermia, nell'attivazione dell'uovo e in altri importanti eventi cellulari2. Poiché l'onda di calcio inizia nel sito di fusione degli spermatozoi, il calcio funge da marcatore per la fecondazione3.
Caenorhabditis elegans è un organismo modello ideale per lo studio dello sviluppo precoce. I vermi sono ermafroditi trasparenti che sono geneticamente trattabili4. Ancora più importante per lo studio della fecondazione, gli adulti di C. elegans producono continuamente ovociti utilizzando una gonade5 rigorosamente ordinata. Infatti, gli ovociti sono le uniche nuove cellule prodotte poiché il soma è post-mitotico in età adulta6. La Figura 1 evidenzia la gonade, che consiste in 2 tubi simmetrici a forma di U di ovociti in via di sviluppo. L'ovocita più vicino alla spermateca (organo di stoccaggio degli spermatozoi) è chiamato ovocita -1. Questo design a catena di montaggio della gonade del verme ovula un ovocita maturo nella spermateca ogni ~23 minuti a 20 °C nei giovani adulti7. L'embrione appena fecondato si sposta quindi in un utero condiviso prima di essere deposto attraverso una singola vulva.
Le tecniche precedenti per visualizzare il calcio durante la fecondazione in C. elegans si basavano sulla microiniezione di coloranti sensibili al calcio 3,8. Per visualizzare più facilmente l'onda di calcio dopo la fusione spermatozoo-uovo, un indicatore di calcio geneticamente codificato basato su jGCaMP7s è stato inserito in copia singola in un locus di approdo sicuro utilizzando brevi ripetizioni palindromiche raggruppate regolarmente intervallate (CRISPR)7,9. Il giornalista è stato chiamato CaFE per il calcio durante la fecondazione in C. elegans. Il reporter non mostra difetti significativi nella fisiologia o nella fecondità.
Qui vengono presentati i protocolli per la visualizzazione dell'onda di calcio negli ovociti e negli embrioni di C. elegans utilizzando il reporter CaFE. In combinazione con la miriade di strumenti disponibili nella comunità dei vermi, come l'interferenza dell'RNA (RNAi) e i marcatori delle gonadi mCherry, il reporter CaFE facilita l'indagine sulla regolazione degli eventi di fecondazione interna, in particolare la competenza in materia di fecondazione e i tempi di fecondazione. Inoltre, il reporter CaFE persiste nello sviluppo precoce ed è uno strumento unico per sondare l'embriogenesi.
Protocollo
1. Manutenzione di C. elegans
- Preparazione della piastra con terreno di crescita dei nematodi (NGM)
- Unire 3 g di cloruro di sodio (NaCl), 2,5 g di Bacto-Peptone, 20 g di agar e 1 litro di acqua deionizzata in un pallone da 4 litri (vedi Tabella dei materiali). Coprire la parte superiore del pallone con un foglio di alluminio prima della sterilizzazione in autoclave.
- Autoclave per sterilizzare utilizzando le impostazioni del ciclo di sterilizzazione a liquido. Si consiglia un ciclo di sterilizzazione impostato a 121 °C per 30 minuti.
- Lasciare raffreddare la miscela a ~50 °C, circa 20 minuti a temperatura ambiente (RT).
- Aggiungere 25 mL di 1 M KH2PO4 sterile (disciolto in acqua, pH = 6), 1 mL di 1 M CaCl2 sterile (disciolto in acqua), 1 mL di 1 M MgSO4 sterile (disciolto in acqua) e 1 mL di colesterolo 5 mg/mL (disciolto in etanolo al 100%), agitando delicatamente dopo ogni aggiunta (vedere la Tabella dei materiali).
- Utilizzando una tecnica sterile, trasferire una parte della miscela in un becher sterile da 300 ml per facilitare il versamento. Versare il supporto caldo fino a coprire il fondo del piatto.
NOTA: Circa 10 ml per piastra in piastre Petri sterili da 60 mm x 15 mm (vedere la Tabella dei materiali) produrranno 100 piastre. In alternativa, è possibile utilizzare piastre di Petri sterili da 35 mm x 10 mm, dove ~4 mL per piastra produrranno ~250 piastre. - Lasciare asciugare le piastre NGM a RT per almeno 1 giorno prima di seminare con i batteri. Conservare le piastre capovolte a 4 °C per un uso futuro.
- Preparazione del prato batterico OP50
- Prepara il brodo di Luria (LB) unendo 5 g di estratto di lievito, 10 g di Bacto-Triptone, 5 g di NaCl e 1 L di acqua deionizzata in un pallone da 2 L (vedi Tabella dei materiali). Usando una piastra di agitazione e una barra di agitazione, mescolare fino a quando non si sarà sciolto.
- Aliquotare LB in bottiglie e autoclave utilizzando le impostazioni del ciclo di sterilizzazione liquida. Si consiglia un ciclo di sterilizzazione impostato a 121 °C per 30 minuti. Se si utilizza un contenitore con tappo a vite, allentare il tappo a vite di un quarto di giro per una corretta ventilazione durante il ciclo in autoclave.
- Lasciare raffreddare il brodo fino a RT prima dell'uso.
- Utilizzando una tecnica sterile, inoculare 50 mL di LB in un pallone di Erlenmeyer da 250 mL con batteri OP50 e far crescere per una notte a 37 °C con agitazione.
- Utilizzando una tecnica sterile, trasferire 300 μL di coltura satura al centro di una piastra NGM da 60 mm x 15 mm. Per piastre da 35 mm x 10 mm, si consigliano 100 μl di coltura satura.
- Lasciare asciugare le piastre a RT per 1-3 giorni fino a quando la macchia batterica OP50 non è più liquida. Assicurarsi che le piastre siano asciutte e prive di contaminazioni prima dell'uso. Conservare le piastre a 4 °C per un uso futuro. La conservazione delle piastre capovolte riduce la formazione di condensa sulla superficie della piastra.
- Costruzione a vite senza fine
- Allentare il collare d'argento verso la testa dell'impugnatura del plettro a vite senza fine (vedere la Tabella dei materiali) svitandolo delicatamente in modo che le quattro sezioni si separino.
- Tagliare 1,5 pollici (circa la lunghezza del pollice) di filo di platino (vedi Tabella dei materiali) usando le forbici e posizionarlo al centro delle quattro sezioni; Circa un pollice sporgerà dall'impugnatura.
- Tenere il filo al centro mentre si stringe il collare; Le quattro sezioni si uniranno al centro e manterranno il filo di platino in posizione.
- Modellare con cura il filo utilizzando un paio di pinze a becchi piatti. Una forma arcobaleno o ad arco tende a funzionare meglio per la raccolta.
- Creare un "piede" di circa 2-3 mm all'estremità del filo bloccando ripetutamente con le pinze a becchi piatti. Usa questo "piedino" per raccogliere i vermi e mantenere il "piede" piatto sulla superficie della piastra.
NOTA: Clamp troppo forte taglierà il filo di platino. Evitare di creare spigoli vivi utilizzando le pinze a becchi piatti (o equivalenti) per evitare di forare i vermi o le piastre.
- Movimentazione di viti senza fine
- Tieni il plettro simile a una matita e sterilizza il "piede" mettendo il filo nella fiamma di una lampada a etanolo al 95% fino a quando non diventa arancione brillante.
- Posizionare una piastra con semi OP50 sul tavolino di un cannocchiale da dissezione e rimuovere il coperchio. Tocca delicatamente il "piede" del piccone su un'area del prato batterico e usa un leggero movimento di scorrimento per raccogliere i batteri sul fondo. I batteri che sono attaccati al plettro (indicati come "appiccicosi") agiranno come una colla per raccogliere i vermi.
- Spostare una piastra di vermi nella fase di dissezione e rimuovere il coperchio evitando che il plettro tocchi altre superfici. Tocca rapidamente e delicatamente il "piede" del piccone su un verme per raccoglierlo. Questa tecnica consente di spostare contemporaneamente più vermi tra le placche.
- Toccare delicatamente il "piede" del piccone sulla parte non seminata di una nuova piastra per trasferire i vermi; Tipicamente, questa è la stessa piastra da cui sono stati prelevati i batteri nella sezione 1.4.2.
- Sterilizzare il plettro tra ogni piastra e/o ceppo di verme per evitare la contaminazione e lasciare raffreddare il plettro dopo la sterilizzazione.
NOTA: Toccare un piccone caldo su un verme stresserà o ucciderà l'animale. Il verme si raffredderà dopo aver toccato il prato batterico per raccogliere la "melma" per il trasferimento del verme.
- Mantenere i vermi a 20 °C su piastre NGM (fase 1.1) seminate con batteri OP50 Escherichia coli (fase 1.2). Passa i vermi 2 - 3 volte a settimana trasferendo diversi adulti su piastre fresche (60 mm x 15 mm) utilizzando un piccone (passaggi 1.3-1.4) per evitare la fame, che può alterare la fisiologia.
NOTA: I ceppi di C. elegans e i batteri OP50 sono disponibili attraverso il Caenorhabditis Genetics Center (CGC).
2. Sincronizzazione dello sviluppo di C. elegans
- Sincronizzare l'età dei vermi utilizzando una deposizione delle uova a tempo (descritta qui). Se è necessario un gran numero di vermi per una classe o per altri scopi, eseguire la sincronizzazione della candeggina come descritto in Golden et al.10.
- Trasferire i vermi adulti in una nuova piastra OP50 seminata utilizzando un plettro di platino (descritto nella sezione 1). Il numero di vermi è dettato dalla necessità sperimentale. I vermi del giorno 1 depongono circa 3 uova all'ora. Pertanto, 10 vermi genereranno circa 30 uova in 1 ora. Preferibilmente, usa i vermi il primo giorno dell'età adulta (Giorno 1).
- Dopo circa 1 ora, rimuovere i vermi adulti dal piatto, lasciando solo le uova.
NOTA: Lasciare inavvertitamente un worm è un errore comune. I worm che rimangono continueranno a deporre le uova e a interrompere la sincronizzazione. I vermi maturano in adulti del giorno 1 circa 72 ore dopo essere stati deposti quando incubati a 20°C.
3. Montaggio di viti senza fine per l'imaging
- Quando si esegue l'imaging degli eventi di fecondazione, utilizzare gli adulti del giorno 1 per massimizzare le opportunità di visualizzazione.
NOTA: Nel giorno 1 degli adulti, un ovocita viene fecondato ogni ~23 minuti. Tuttavia, l'ovulazione rallenta con l'età e un tipico ermafrodito smetterà di deporre le uova dopo il quarto giorno. - Preparare una soluzione di agarosio al 3% in acqua mescolando 3 g di agarosio in 100 ml di acqua. Riscaldare la soluzione di agarosio usando un forno a microonde fino a quando l'agarosio non è completamente sciolto. Per riutilizzare una soluzione solidificata, riscaldare fino a completa dissoluzione. Dopo diversi utilizzi, generare una nuova soluzione.
- Posizionare un vetrino da microscopio vuoto per l'imaging tra altri due vetrini da microscopio che hanno un singolo strato di nastro da laboratorio lungo l'asse lungo del vetrino (vedere Tabella dei materiali). Il nastro crea un distanziatore per il tampone di agarosio, consentendo una distribuzione uniforme e una larghezza uniforme. Assicurarsi che i tre vetrini siano in fila con i lati lunghi che si toccano: due vetrini con nastro adesivo che fiancheggiano un vetrino da microscopio vuoto.
- Far cadere la soluzione riscaldata di agarosio al 3% (~100-150 μL) al centro del vetrino del microscopio che si trova tra i vetrini nastrati utilizzando una pipetta da 1 mL.
- Posizionare rapidamente un nuovo vetrino da microscopio sulla gocciolina di agarosio, assicurandosi che il nuovo vetrino sia perpendicolare al vetrino con la gocciolina di agarosio e appoggiato sui vetrini nastrati adiacenti.
- Separare delicatamente le diapositive. Evitare di strappare il tampone.
- Prepara i tamponi di agarosio freschi ogni giorno di imaging. Si consiglia di realizzare più tamponi durante la preparazione per l'imaging, ma di astenersi dall'esporre più di un tampone contemporaneamente. I cuscinetti esposti si asciugheranno rapidamente.
- Utilizzando una pipetta da 20 μl, aggiungere una piccola goccia (~7 μl) di 1 mM di levamisolo in tampone M9 al centro del tampone di agarosio per paralizzare i vermi. Utilizzare una soluzione madre sterilizzata con filtro da 100 mM di levamisolo per ottenere la soluzione di lavoro da 1 mM.
- Per preparare il tampone M9, unire 6 g di Na2HPO4, 3 g di KH2PO4, 0,5 g di NaCl, 1 g di NH4Cl e 1 L di acqua deionizzata in un contenitore adatto (vedi Tabella dei materiali).
- Autoclavare la soluzione utilizzando le impostazioni del ciclo di sterilizzazione liquida e lasciare raffreddare la soluzione a RT prima dell'uso. Se si utilizza un contenitore con tappo a vite, allentare il tappo a vite di circa un quarto di giro per una corretta ventilazione. Si consiglia un ciclo di sterilizzazione impostato a 121 °C per 30 minuti.
- Raccogli almeno 5 vermi sincronizzati e tocca delicatamente il "piede" sulla gocciolina di levamisolo per trasferire tutti i vermi. Utilizzare almeno 5 vermi per vetrino, poiché l'orientamento del corpo può far sì che l'intestino oscuri la linea germinale, bloccando la visualizzazione dell'onda di calcio nell'ovocita.
NOTA: Assicurarsi che i vermi vengano trasferiti rapidamente poiché il levamisolo evaporerà se lasciato sul tampone di agarosio per più di 5 minuti. - Coprire accuratamente il tampone di agarosio con un vetrino coprioggetti. Controllare la posizione dei vermi, poiché il vetrino coprioggetti può causare la dispersione dei vermi dal posizionamento iniziale. Etichettare il vetrino del microscopio con i dettagli appropriati, come il nome del ceppo di verme (vedere la Tabella dei materiali).
NOTA: Assicurarsi che l'imaging venga eseguito il prima possibile dopo il montaggio dei vermi, poiché l'esposizione prolungata al levamisolo è tossica.
4. Imaging del calcio durante la fecondazione in C. elegans
- Localizza i vermi utilizzando un obiettivo a bassa potenza, come 4x, sul microscopio preferito. Utilizzare l'illuminazione a campo chiaro.
- Utilizzare un obiettivo ad aria minimo 20x per visualizzare chiaramente l'onda di calcio negli ovociti. Utilizzare un obiettivo a immersione di 40x o superiore.
- A seconda del sistema utilizzato, l'intensità di eccitazione è tipicamente un valore fisso. Per una visualizzazione ottimale dei reporter fluorescenti, regolare il guadagno o il tempo di esposizione.
NOTA: La luce continua ad alta intensità danneggerà il campione ed è dannosa per la salute dei vermi. Se lo si desidera, spegnere il laser in attesa di un evento di fecondazione per evitare l'illuminazione GFP continua. Vedere i segni distintivi di un evento di fecondazione nelle sezioni 4.8 - 4.9. - Assicurarsi che il canale GFP a 488 nm sia impostato su una potenza laser in cui il segnale del reporter CaFE sia facilmente visibile.
- Per una scansione laser confocale Nikon ECLIPSE Ti2, rilevare il reporter CaFE a una potenza laser del 30% per il canale GFP e un guadagno di 75. La dimensione del foro stenopeico per il confocale a scansione laser Nikon ECLIPSE Ti2 è di 30 μm.
- Per il confocale a disco rotante Andor Dragonfly utilizzando la fotocamera Zyla, rilevare il reporter CaFE a una potenza laser del 30% per il canale GFP. La dimensione del foro stenopeico per il confocale a disco rotante Andor Dragonfly è di 40 μm.
- Regolare le impostazioni del laser e il tempo di esposizione per ogni campione esaminato.
- Se si utilizza il sistema EAG28 (CaFE; PH-mCherry) o EAG25 (CaFE; H2B-mCherry), assicurarsi che il canale rosso sia impostato su una potenza laser in cui il segnale mCherry sia facilmente visibile oltre al canale GFP.
- Per la scansione laser confocale Nikon ECLIPSE Ti2, rilevare il reporter mCherry a una potenza laser di 15 per il canale RFP e un guadagno di 130.
- Per il confocale a disco rotante Andor Dragonfly che utilizza la fotocamera Zyla, rileva il reporter mCherry a una potenza laser del 30% per il canale RFP.
- Regolare le impostazioni del laser e il tempo di esposizione per ogni campione esaminato.
- Imposta la frequenza dei fotogrammi o i fotogrammi al secondo (fps) alla velocità consentita dal sistema.
NOTA: Il reporter CaFE è tipicamente osservato a circa 1 fps per il confocale a scansione laser Nikon ECLIPSE Ti2 e a 10 fps per il confocale a disco rotante Andor Dragonfly con la fotocamera Zyla. - Selezionare il protocollo time lapse o time series e regolare le impostazioni desiderate. Acquisisci immagini time-lapse per 30 minuti o meno, con l'opzione di ripetizione impostata su '1'.
NOTA: Se l'evento di fecondazione si verifica prima del completamento dell'intervallo di tempo, interrompere il video per preservare le dimensioni del file. - Individua i vermi sul vetrino e seleziona un verme in cui è visibile la gonade prossimale. L'intestino e la gonade si attorcigliano l'uno intorno all'altro all'interno del verme. In quanto tale, un braccio della gonade è spesso oscurato dall'intestino, impedendo la visualizzazione della fecondazione. Se l'ovocita -1 è oscurato, selezionare un altro worm per la visualizzazione. Per aumentare la probabilità che il verme selezionato abbia un braccio gonadico visibile, montare un minimo di 5 vermi per vetrino. Usa gli adulti del primo giorno per visualizzare la fecondazione.
- Esamina ogni ovocita -1 nei bracci delle gonadi del verme per determinare se un evento di fecondazione è imminente.
NOTA: Prima della fecondazione, durante la maturazione meiotica, il nucleo dell'ovocita -1 migrerà verso la parte posteriore dell'ovocita e l'involucro nucleare si dissolverà. Questi eventi sono visibili sia nei canali Nomarksi che GFP utilizzando il reporter CaFE e tali eventi indicano che l'ovulazione avverrà entro i prossimi ~10 minuti. - Dopo la rottura dell'involucro nucleare, il citoscheletro dell'ovocita -1 più prossimale si riorganizzerà, assumerà un aspetto arrotondato e si separerà dall'ovocita -2. A questo punto, l'ovulazione avverrà imminentemente. Iniziare immediatamente a registrare il video se si osservano questi eventi. Registra video in un ambiente widefield.
NOTA: Se il movimento del nucleo o l'arrotondamento dell'ovocita non sono visibili dopo 20 minuti, è improbabile che si verifichi un evento di ovulazione. Concentrati su un verme diverso e/o prepara un nuovo vetrino.
5. Quantificazione dei transitori del calcio nella fecondazione con C. elegans
- Quantificare i transitori di calcio dai video di fecondazione utilizzando il software del sistema di microscopio come Nikon NIS Elements (descritto qui). ImageJ/FIJI sono adatti anche per l'analisi delle immagini11.
- Per quantificare la variazione della fluorescenza dei video di fecondazione, utilizzare i seguenti valori di segnale: il segnale al tempo 0, il segnale dal fotogramma di interesse e il segnale di base.
- Identificare il tempo e l'intervallo che cattura il primo lampo di fluorescenza che si verifica nell'ovocita -1. Definisci questo fotogramma come tempo 0.
- Per ottenere il segnale dal frame di interesse, disegnare la regione di interesse (ROI) per comprendere le seguenti aree: l'ovocita -1 prima della fecondazione, la spermateca e l'ovocita appena fecondato nell'utero. Il segnale visualizzato nella ROI è denominato F1.
NOTA: Assicurarsi che il ROI copra l'intera area trafficata dall'ovocita. Il ROI dovrebbe essere identico in ogni fotogramma della registrazione time-lapse analizzata. - Definisci un'area vuota dell'immagine senza fluorescenza utilizzando le stesse dimensioni della ROI definita nel passaggio 5.4 per calcolare il segnale di fondo in ogni fotogramma. Sottrarre il segnale di fondo da ogni fotogramma.
- Per ottenere il segnale di base (F0), posizionare il fotogramma in un minimo di 15 fotogrammi prima del tempo 0. Utilizzando lo stesso ROI del passaggio 5.4, annotare il segnale ad ogni fotogramma fino al tempo 0. Fare la media dei segnali (con la sottrazione di fondo) dal fotogramma iniziale fino al tempo 0 per calcolare il segnale di base.
- Calcolare la variazione della fluorescenza (ΔF) utilizzando la seguente formula:
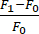 . F1 è definito in ogni fotogramma come il segnale nella ROI senza il segnale di fondo. F0 è il segnale fluorescente medio di base di ≥15 fotogrammi prima del tempo 0, come calcolato nella sezione 5.6.
. F1 è definito in ogni fotogramma come il segnale nella ROI senza il segnale di fondo. F0 è il segnale fluorescente medio di base di ≥15 fotogrammi prima del tempo 0, come calcolato nella sezione 5.6. - Determinare la velocità di variazione tra i fotogrammi sottraendo la variazione di fluorescenza tra 2 fotogrammi e dividendo per il tempo.
6. RNAi in C. elegans
- Preparare le piastre RNAi
- Preparare le piastre secondo il passaggio 1.1 e aggiungere i seguenti reagenti aggiuntivi per consentire il knockdown genico tramite RNAi al passaggio 6.1.4.
- Aggiungere 1 mL di 1 mL di β-d-1-tiogalattopiranoside isopropilico filtrato sterile 1 M (disciolto in acqua deionizzata) per una concentrazione finale di 1 mM di IPTG nel terreno (vedere la Tabella dei materiali).
- Aggiungere 1 mL di 100 mg/mL di ampicillina filtrata sterile (disciolta in acqua deionizzata) per una concentrazione finale di 100 μg/mL di ampicillina nel terreno (vedere la Tabella dei materiali). In alternativa, utilizzare 40 μg/mL di carbenicillina12.
- Agitare delicatamente ma accuratamente per mescolare e versare le piastre come descritto nei passaggi 1.1.4 - 1.1.5.
- Preparazione della coltura satura di RNAi
- Calcolare la quantità di coltura satura moltiplicando il numero di piastre RNAi seminate necessarie per 300 μl quando si utilizzano piastre da 60 mm x 15 mm o per 100 μl quando si utilizzano piastre da 35 mm x 10 mm.
- Utilizzando una tecnica sterile, far crescere i batteri RNAi desiderati in LB (descritto nei passaggi 1.2.1-1.2.3) contenenti 100 μg/mL di ampicillina per una notte a 37 °C fino a saturazione. Includere un ceppo batterico contenente il vettore vuoto L4440 come controllo negativo e un ceppo batterico contenente un controllo positivo all'RNAi (ad es. uovo-5) per verificare che l'RNAi funzioni. Inoltre, includi una condizione "in bianco" contenente solo LB per verificare che non vi siano contaminazioni nel LB di serie.
- Aggiungere IPTG filtrato sterile a una concentrazione finale di 1 mM alla coltura notturna satura per l'induzione dell'espressione del dsRNA e incubare per almeno 2 ore e 30 minuti a 37 °C prima della semina. In alternativa, diluire nuovamente la coltura notturna satura fuori dalla fase logaritmica prima di indurre con IPTG.
NOTA: L'IPTG è incluso sia nelle piastre che nella coltura satura per un'espressione ottimale del dsRNA. - Utilizzando una tecnica sterile, trasferire 300 μl della coltura indotta al centro di una piastra RNAi di 60 mm x 15 mm (solidificata). In alternativa, utilizzare 100 μl di coltura satura per una piastra da 35 mm x 10 mm.
- Lasciare asciugare le piastre a RT per 1-3 giorni fino a quando la macchia batterica RNAi non è più liquida. Assicurarsi che le piastre siano asciutte e prive di contaminazioni prima dell'uso. Conservare le piastre a 4 °C capovolte per un uso futuro.
NOTA: Utilizzare le piastre RNAi il prima possibile per un knockdown ottimale, poiché l'efficacia dell'RNAi diminuisce nel tempo. Scartare se >1 mese.
- Come descritto nella sezione 2, sincronizzare i vermi su piastre RNAi.
- Controllare i vermi 1-2 giorni dopo la sincronizzazione della deposizione delle uova per verificare la presenza di ritardi nello sviluppo.
- Alcuni RNAi possono causare difetti di sviluppo quando il knockdown inizia alla deposizione delle uova. Se necessario, posizionare i vermi sulle piastre RNAi in una fase successiva, come L3.
- Come descritto nelle sezioni 3 e 4, montare i vermi e prepararsi per l'imaging utilizzando qualsiasi sistema di microscopio attrezzato per rilevare la fluorescenza GFP.
7. Imaging del calcio durante l'embriogenesi precoce in C. elegans
- Sincronizzare i worm come descritto nella sezione 2. Utilizzare il ceppo EAG28, che combina il reporter CaFE e un marcatore pleckstrin-homology domain-mCherry che evidenzia il confine della membrana plasmatica. In alternativa, per visualizzare gli embrioni fecondati ex utero, tagliare delicatamente il verme lungo l'utero usando un ago da siringa sterile per rilasciare gli embrioni.
- Preparare i vermi o gli embrioni per l'imaging come descritto nella sezione 3 e seguire le stesse istruzioni descritte nella sezione 4 per l'impostazione dei parametri di imaging.
- Centrare l'area di interesse sull'utero del verme per visualizzare gli embrioni o un uovo deposto.
- Per gli embrioni in utero, assicurarsi che l'utero non sia dietro l'intestino, che l'utero non sia affollato e che le singole cellule dell'embrione siano libere sul piano focale.
- Per gli ovuli ex utero, assicurarsi che gli stadi delle cellule non abbiano superato lo stadio virgola del ciclo di vita del verme.
- Selezionare il protocollo time lapse o time series e regolare le impostazioni desiderate.
- Per una visualizzazione ottimale dei transitori di calcio dell'embrione, aumentare le impostazioni temporali per i filmati prolungati. Tieni presente che l'esposizione costante al laser danneggerà l'embrione per lunghi periodi di tempo.
- Cattura video o immagini concentrandoti sulle cellule negli embrioni nello stadio cellulare desiderato.
- Utilizzare un obiettivo a immersione 40x o superiore per l'imaging, anche se i transitori vengono facilmente rilevati utilizzando un obiettivo ad aria 20x.
Risultati
Utilizzando i protocolli delineati in questo manoscritto, i modelli dinamici di segnalazione del calcio nella fecondazione e nell'embriogenesi sono stati osservati in C. elegans.
Una tipica sequenza di fecondazione nei vermi contenenti il reporter CaFE è mostrata nella Figura 2. Per facilitare l'analisi, è stato utilizzato il ceppo EAG28 che combina il reporter CaFE con un transgene13 del dominio di omologia della pleckstrin (PH-mCherry). Il marcatore PH-mCherry si localizza sulla membrana plasmatica e consente una più facile visualizzazione dei confini cellulari, in particolare nel braccio delle gonadi. Il ceppo EAG28 è stato creato incrociando il ceppo EAG16 contenente il reporter CaFE con il ceppo reporter OD70 PH-mCherry13. In C. elegans, l'ovulazione e la fecondazione avvengono contemporaneamente. Il tempo 0 della fecondazione è considerato il primo fotogramma in cui viene rilevato un segnale di fluorescenza nell'ovocita in ovulazione (Figura 2). Un'esplosione luminosa di fluorescenza si verifica nel sito di fusione degli spermatozoi non appena il bordo anteriore dell'ovocita entra nella spermateca, l'organo di stoccaggio degli spermatozoi7. Il segnale appare prima che l'ovulazione sia completa. L'onda di calcio è bifasica, con una rapida esplosione iniziale seguita da un'onda di fluorescenza dal punto di ingresso verso il polo opposto. L'intero ovocita diventa fluorescente in <30 s.
Si noti che il reporter CaFE è in una singola copia e, quindi, il segnale non è così luminoso come altri transgeni. Il time-lapse Z-stack non è raccomandato durante l'ovulazione a meno che non sia disponibile e ottimizzato un confocale a disco rotante. Le immagini Z stack possono essere ottenute prima o dopo un filmato per catturare i cambiamenti 3D nella morfologia delle gonadi. Sebbene le immagini qui siano state scattate con un microscopio confocale, i transienti sono stati osservati anche utilizzando la microscopia a fluorescenza a campo largo, che è più comune ed economica di quella confocale. Utilizzando una scansione confocale laser, la frequenza dei fotogrammi è di circa 1 fps. Utilizzando un confocale a disco rotante, la frequenza dei fotogrammi è di 10 fps o superiore. Il segnale di fondo del reporter CaFE è sufficiente per illuminare gli ovociti in maturazione nel braccio delle gonadi. Dopo la fecondazione, si osserva tipicamente un aumento di 1,5-2 volte dell'intensità del segnale GFP.
Esistono diversi metodi alternativi per la quantificazione. Ad esempio, il software di analisi delle immagini Imaris, creato da Oxford Instruments (la stessa società madre del microscopio confocale a disco rotante Andor Dragonfly), ha la capacità di quantificare il segnale in ogni fotogramma proveniente solo dall'ovocita in transito invece di un ROI maggiore. Tuttavia, il software Imaris non è gratuito. Inoltre, una strategia dettagliata di analisi delle immagini per misurare le onde di calcio nell'ovocita è stata descritta in modo estremamente dettagliato da Takayama, Fujita e Onami e utilizza ImageJ, che è disponibile gratuitamente14. In alternativa, i chimografi per illustrare l'onda del segnale fluorescente attraverso un singolo ovocita richiedono una manipolazione dell'immagine più estesa di quella descritta qui, ma sono descritti in Takayama e Onami3.
Un netto vantaggio di C. elegans è la sua capacità di abbattere l'espressione di quasi tutti i geni alimentando i vermi con batteri che esprimono dsRNA per innescare l'RNAi endogeno15,16. Per un'espressione ottimale del dsRNA, sia le piastre RNAi che la coltura di RNAi saturi contengono IPTG. Il dsRNA è prodotto esprimendo il gene di interesse con i promotori fiancheggiatori. Qui, abbiamo utilizzato la libreria Ahringer che prende di mira la maggior parte del genoma del verme attraverso l'uso di 16.256 ceppi batterici17. In alternativa, un ORFeome di ~11.000 cloni di RNAi è stato creato dal laboratorio Vidal utilizzando il sistema Gateway ed è disponibile per l'acquisto tramite Horizon Discovery18.
Per dimostrare l'utilità del reporter CaFE in combinazione con l'RNAi, abbiamo esaminato l'ovulazione prematura. Una delezione parziale in ipp-5 induce un doppio evento di ovulazione in cui sia l'ovocita -1 che l'ovocita -2 entrano nella spermateca durante la stessa ovulazione19. IPP-5 è una fosfatasi che agisce su IP3 e diminuisce efficacemente i livelli del secondo messaggero IP3. Abbiamo scoperto che il knockdown RNAi di ipp-5 suscita un fenotipo di doppia ovulazione simile al mutante.
L'analisi del reporter CaFE durante la doppia ovulazione indotta dal knockdown dell'RNAi di ipp-5 ha rivelato nuovi aspetti dell'OET. In primo luogo, un segnale di calcio è stato osservato immediatamente dopo l'ingresso nella spermateca nell'ovocita -1 principale ma non nell'ovocita -2 finale (Figura 3). Sebbene 2 ovociti siano ovulati, l'ovocita -1 è ancora l'unico ovocita che mostra segni di corretta maturazione, in particolare la rottura dell'involucro nucleare (NEBD). Questi dati suggeriscono che anche se l'ovocita -2 è in presenza di spermatozoi, non è competente per essere fecondato in quanto non è ancora maturato correttamente. In secondo luogo, l'ovocita -2 mostra un'onda di calcio ritardata, in genere quando l'ovocita esce dalla spermateca per l'utero. Sebbene la maturazione degli ovociti sia normalmente un prerequisito per l'ovulazione, questi dati suggeriscono che la maturazione dell'ovocita e la competenza per la fecondazione possono ancora avvenire dopo l'ovulazione. La visualizzazione dell'onda di calcio ritardata durante il knockdown di ipp-5 evidenzia i vantaggi del reporter CaFE per interrogare la competenza e la tempistica della fertilizzazione.
Inoltre, abbiamo scoperto che il reporter CaFE è rilevabile nelle uova deposte e rivela transitori di calcio a singola cellula durante l'embriogenesi precoce in C. elegans. Gli embrioni provenienti da EAG28 adulto sincronizzato del giorno 1 (CaFE; PH-mCherry) sono stati ripresi utilizzando i protocolli delle sezioni 1-5 con il disco rotante confocale Andor Dragonfly utilizzando la fotocamera Zyla (Figura 4). I transitori di calcio sono confinati a singole cellule e impiegano meno tempo rispetto all'onda di calcio della fecondazione per essere completati (~9 s, n=11). Tuttavia, l'onda di calcio durante l'embriogenesi non sembra essere bifasica. I transitori di calcio non sono stati osservati prima dello stadio a 8 cellule. In particolare, il calcio non si localizza nel solco di scissione in C. elegans, come si vede in molti altri organismi, inclusi gli esseri umani e Xenopus 20,21,22. I transienti di calcio unicellulari sono stati osservati ben dopo la gastrulazione (~200 minuti dopo la fecondazione) e nelle uova deposte. Un singolo embrione mostra più transitori di calcio nel tempo, ma in cellule diverse e in genere una cellula alla volta. Si noti che l'esposizione continua alla stimolazione laser danneggerà gli embrioni. Si consiglia una potenza LED/laser inferiore o un'esposizione discontinua. Tuttavia, poiché le onde di calcio sono relativamente brevi a ~9 s, non consigliamo di rallentare più di 2 s tra gli eventi di stimolazione LED/laser.
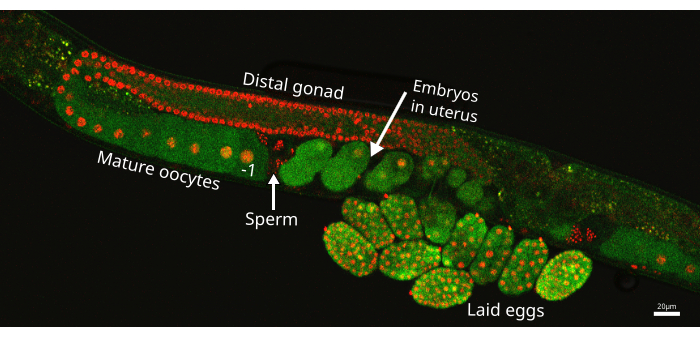
Figura 1: Braccio della gonade di C. elegans . Immagine di un braccio di gonade del ceppo di verme EAG25, che mostra il reporter CaFE GFP (verde) e l'istone 2B-mCherry marker (rosso), che evidenzia i nuclei nella linea germinale. L'ovocita prossimale è indicato con il -1 direttamente prima della spermateca, dove sono conservati gli spermatozoi (freccia). L'embrione situato più vicino alla spermateca ma all'interno dell'utero è l'embrione più recente. L'immagine è stata scattata utilizzando la scansione laser confocale Nikon ECLIPSE Ti2. Barra della scala = 20 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
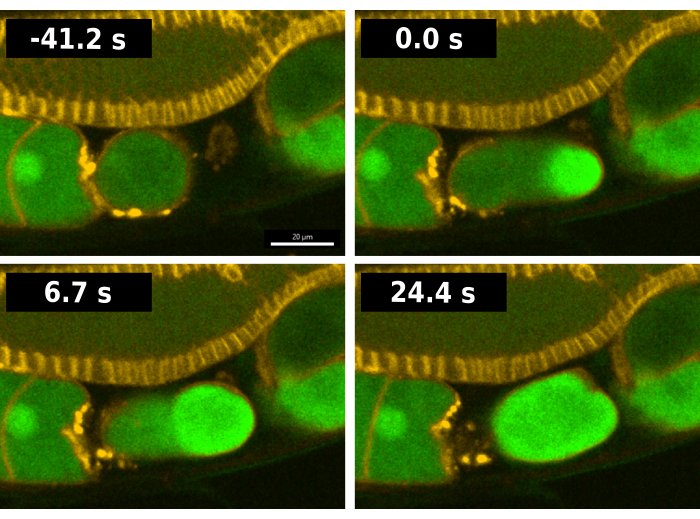
Figura 2: Visualizzazione dell'onda di calcio durante la fecondazione. Immagini di serie temporali della fluorescenza che riflette il calcio intracellulare durante la fecondazione nel ceppo di vermi EAG28. Il ceppo EAG28 contiene sia il reporter CaFE (verde) che una fusione pleckstrin-homology domain-mCherry (giallo), che evidenzia le membrane plasmatiche. Il tempo 0 indica il primo fotogramma che ritrae un chiaro aumento del segnale fluorescente nell'ovocita. Il tempo di 24,4 s riflette il primo fotogramma che mostra la fluorescenza dell'intero ovocita. Le immagini delle serie temporali sono state scattate utilizzando il confocale a disco rotante Andor Dragonfly con una fotocamera Zyla. Barra della scala = 20 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
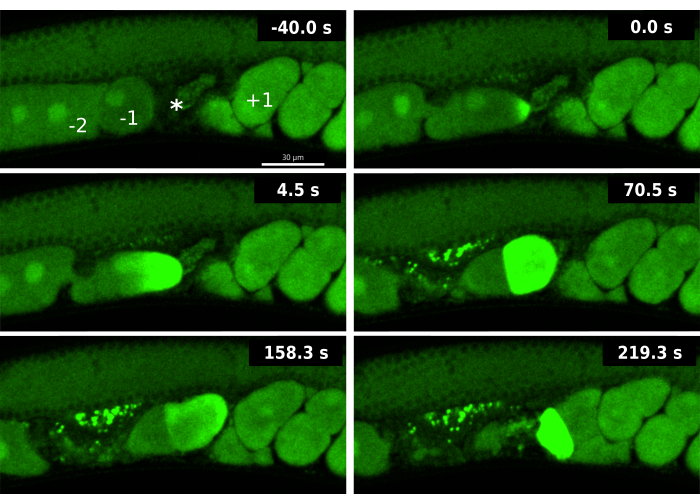
Figura 3: Onde di calcio durante un evento di doppia ovulazione indotta da ipp-5 RNAi. Immagini time-lapse di onde sequenziali di calcio dell'ovocita -1 e dell'ovocita -2 in un ceppo EAG16 (CaFE in verde GFP) esposto all'RNAi ipp-5 . Il frame -40.0 s include etichette per l'ovocita -1 (-1), -2 ovocita (-2), spermateca (*) e +1 embrione (+1). Il tempo 0 s mostra l'aumento iniziale della fluorescenza dell'onda di calcio dell'ovocita -1. Il tempo di 4,5 s mostra l'onda di calcio che si diffonde verso il polo opposto nell'ovocita -1. Il tempo 70,5 s mostra la fluorescenza dell'intera cellula nell'ovocita -1 accompagnata dall'ovocita -2 al segnale GFP basale; Entrambi gli ovociti si trovano nella spermateca. L'ovocita -1 fecondato e l'ovocita -2 non fecondato entrano nell'utero al tempo 158,3 s. A 219,3 s, l'ovocita -2 mostra un'onda di calcio fluorescente tardiva. Le immagini sono state scattate utilizzando il confocale a disco rotante Andor Dragonfly con una fotocamera Zyla. Barra di scala = 30 μM Fare clic qui per visualizzare una versione più grande di questa figura.
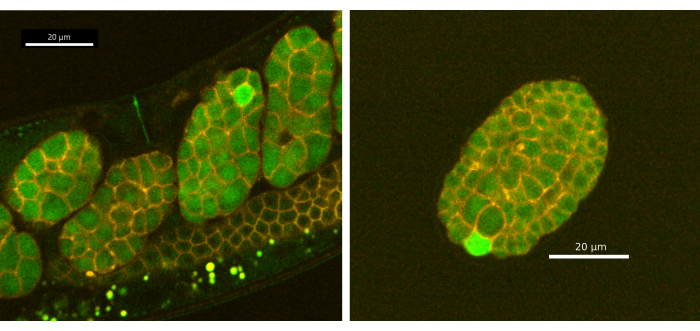
Figura 4: Transitori di calcio a cellula singola nell'embriogenesi precoce. (A) Gli embrioni in utero e (B) ex utero (uovo deposto) hanno mostrato un segnale fluorescente in una singola cellula durante l'embriogenesi. Le immagini sono state scattate utilizzando il confocale a disco rotante Andor Dragonfly con una fotocamera Zyla. Barra della scala = 20 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Uno strumento semplice con un protocollo robusto è una potente combinazione per affrontare difficili questioni scientifiche. Qui, i metodi per la visualizzazione del calcio come proxy facilmente rilevabile per la fecondazione sono presentati utilizzando il reporter CaFE. Questo stesso reporter persiste nell'embriogenesi precoce e consente anche la visualizzazione dei transitori di calcio più avanti nello sviluppo. La segnalazione del calcio funge da secondo messaggero critico che demarca i principali cambiamenti nella funzione cellulare, in particolare per la biologia dello sviluppo. Nell'ovocita, la prima fase di scoppio dell'onda di calcio segna non solo il momento della fecondazione, ma anche il sito di fusione degli spermatozoi3. In C. elegans, l'asse A-P è determinato anche dal sito di fusione degli spermatozoi23. Pertanto, la capacità di visualizzare il calcio negli ovociti e negli embrioni consente di indagare su questioni complesse che sono centrali per la biologia cellulare e dello sviluppo.
Il metodo qui descritto con il reporter CaFE dovrebbe essere abbastanza semplice per i principianti dei nematodi. I metodi precedenti per rilevare le onde di calcio negli ovociti di C. elegans si basavano sull'iniezione di colorante 3,8,14. Sebbene questi studi siano stati importanti e illuminanti, l'iniezione di colorante è laboriosa e le apparecchiature per l'iniezione non sono disponibili in tutti i laboratori.
I protocolli qui presentati sono stati ottimizzati per i vermi sani. Per massimizzare le possibilità di successo, assicurarsi che non vi siano contaminazioni sul mezzo di verme o nel prato batterico su cui si nutrono i vermi. Non esporre i vermi a condizioni di stress, come la temperatura, poiché queste influenzeranno l'ovulazione e la fecondazione. Inoltre, i controlli per l'efficacia dell'RNAi dovrebbero essere inclusi in ogni esperimento poiché l'efficacia diminuisce nel tempo. Utilizzare un controllo positivo per l'efficacia dell'RNAi come l'uovo-5, che genera embrioni, ma nessuna progenie vitale poiché la formazione del guscio d'uovo è compromessa24. Inoltre, i parametri di acquisizione delle immagini devono essere ottimizzati per ogni sistema di microscopia. Le nostre specifiche sono incluse qui come riferimento, ma ci si possono aspettare delle deviazioni. Sebbene questo sistema rilevi prontamente il calcio citoplasmatico come proxy per la fecondazione, non rappresenta necessariamente un vero e proprio evento di fusione spermatozoo-ovocita.
L'ampia gamma di strumenti e mutanti distribuiti gratuitamente all'interno della comunità dei worm migliora l'utilità del reporter CaFE. Il reporter è integrato nel genoma del verme ed è facilmente incrociato con altri ceppi mutanti o reporter di C. elegans 25. Qui è riportata la creazione di EAG25 che esprime il reporter CaFE con un marcatore istone H2B-mCherry per visualizzare i nuclei (Figura 1) e EAG28 sia con il reporter CaFE che con un marcatore pleckstrin-homology domain-mCherry, che evidenzia la periferia cellulare (Figura 2)13,26. Entrambi i ceppi aiutano nella visualizzazione delle cellule nella linea germinale e durante l'embriogenesi. Inoltre, la facility di RNAi in
C. elegans, quando utilizzato con il reporter CaFE, ha rivelato nuove intuizioni sulla competenza in fertilizzazione. Come mostrato nella doppia ovulazione indotta dal knockdown dell'RNAi ipp-5 nella Figura 3, la presenza di spermatozoi e di un ovocita ovulato è insufficiente per stimolare un evento di fecondazione.
Questi risultati indicano che deve esistere un altro segnale, o l'assenza di un inibitore, che permetta all'ovocita di fecondarsi. L'ovocita ovulato prematuramente mostra un transitorio di calcio ritardato quando l'ovocita si sposta nell'utero. Questa ondata tardiva di calcio suggerisce che l'ovocita -2 ovulato prematuramente può sviluppare competenza di fecondazione con il tempo. Prevediamo che gli studi sui tempi della fecondazione, in particolare per quanto riguarda la segnalazione e la regolazione cellula-cellula, saranno aiutati dall'uso del reporter CaFE. Inoltre, il reporter CaFE persiste nell'embriogenesi e mostra transitori di calcio a singola cellula. Questa segnalazione embrionale di calcio è stata segnalata anche entro le prime 24 ore dallo sviluppo del zebrafish27. Il ruolo dei transitori del calcio è sconosciuto, ma la presenza suggerisce un evento di segnalazione cellulare durante lo sviluppo che non è stato ancora esplorato. In particolare, i transitori del calcio non sono stati osservati negli zigoti appena fecondati. Pertanto, il calcio non si localizza nel solco di scissione come è stato documentato in molti altri organismi, tra cui Xenopus e l'uomo 20,21,22.
È importante sottolineare che il reporter CaFE è abbastanza facile da essere utilizzato da studenti universitari con una formazione minima. Abbiamo progettato ed eseguito un laboratorio CURE (course-based undergraduate research experience) da 1 credito per studenti di biologia con i ceppi e i protocolli qui descritti. Nel corso di un semestre, la classe si è incontrata una volta alla settimana per 3 ore o due volte a settimana per 1 ora e 30 minuti ciascuna. Agli studenti è stata data la possibilità di lavorare da soli o in gruppi di 2. Ogni studente/coppia ha selezionato un gene diverso da studiare da un elenco curato. Hanno eseguito l'RNAi contro il gene scelto nel ceppo EAG28 e hanno esaminato i vermi per gli effetti sulla fecondità, sulla fecondazione e/o sulla morfologia delle gonadi. Sulla base dei loro risultati e delle loro letture di base utilizzando la letteratura primaria, gli studenti hanno sviluppato ipotesi che avrebbero potuto testare in esperimenti successivi. Questo design iterativo è stato fondamentale per aumentare il coinvolgimento degli studenti28. Gli studenti hanno acquisito un'autentica esperienza di ricerca e competenze in organismi modello, screening genetici e microscopia a fluorescenza. Data la facilità d'uso del reporter CaFE, gli studenti senza esperienza di ricerca sono stati in grado di avere successo. In seguito, gli studenti hanno espresso in modo schiacciante una preferenza per il formato CURE rispetto alle tradizionali lezioni di laboratorio, con molti studenti che hanno espresso il desiderio di continuare con la ricerca. Nel loro insieme, questi strumenti e protocolli aiutano sia nell'istruzione che nella ricerca sui processi di sviluppo precoce.
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti o finanziari.
Riconoscimenti
KSKG è stato finanziato da una sovvenzione dell'Eunice Kennedy Shriver National Institute of Child Health and Human Development (R15HD111986). Alcuni ceppi sono stati forniti dal CGC, che è finanziato dall'Ufficio NIH dei programmi di infrastruttura di ricerca (P40 OD4010440). Ringraziamo WormBase.
Materiali
| Name | Company | Catalog Number | Comments |
| Agar | Fisher Scientific | DF0140-07-4 | 2 kg; Powder dissolves easier than flakes |
| Agarose | MidSci | BE-A125 | 500 g |
| Alcohol lamp | Fisher Scientific | S13475 | Use with 95% ethanol |
| Ammonium chloride (NH4Cl) | Fisher Scientific | AAA1500030 | 250 g |
| Ampicillin | Fisher Scientific | BP1760-5 | 5 g |
| AMSCO 400 Series Small Steam Sterilizer | Steris Healthcare | N/A | |
| Bacto-peptone | Fisher Scientific | BP1420-500 | 500 g |
| Bacto-tryptone | Fisher Scientific | DF0123-17-3 | 500 g |
| Calcium chloride (CaCl2) | Fisher Scientific | C69-500 | 500 g |
| Cholesterol | Thermo Scientific | A11470.18 | 50 g |
| Dragonfly 200 spinning disk confocal | Oxford Instruments Andor | N/A | Used with Leica microscope |
| Fisherbrand Superfrost Cytogenics Microscope Slides | Fisher Scientific | 22-035-900 | 144 slides per pack |
| Flat Nose Pliers, Smooth Jaw | Home Depot | 305530604 | Ensure pliers are smooth jaw |
| Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Fisher Scientific | BP1755-10 | 10 g; dioxane-free |
| Laboratory tape | Fisher Scientific | 15-901-10R | 0.5 inch tape is used to tape microscope slides |
| Levamisole | Fisher Scientific | AC187870100 | 10 g |
| Magnesium sulfate (MgSO4) | Fisher Scientific | M63-500 | 500 g |
| Microscope cover glass | Fisher Scientific | 12541016 | 1 oz pack |
| Nikon ECLIPSE Ti2 laser scanning confocal | Nikon | N/A | |
| Nikon NIS Elements software | Nikon | N/A | Confocal |
| OP50 Escherichia coli | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Platinum Wire | TriTech | PT-9010 | |
| Potassium phosphate dibasic (K2HPO4) | Fisher Scientific | P288-500 | 500 g |
| Potassium phosphate monobasic (KH2PO4) | Fisher Scientific | AA1159436 | 500 g |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | 500 g |
| Sodium phosphate dibasic heptahydrate (Na2HPO4) | Fisher Scientific | S471-3 | 3 kg |
| Stereo microscope | Leica | KL300 LED | |
| Sterile Petri dish (35 mm x 10 mm) | CellTreat | 229638 | 960 Petri dishes per case |
| Sterile Petri dish (60 mm x 15 mm) | CellTreat | 229665 | 500 Petri dishes per case |
| Strain EAG16 spn-4p::jGCaMP7s::pie-1u | Caenorhabditis Genetics Center (CGC) | EAG16 | Created by Kim Guisbert Lab |
| Strain EAG25 spn-4p::jGCaMP7s::pie-1u; ujIs113 II. | Caenorhabditis Genetics Center (CGC) | EAG25 | Created by Kim Guisbert Lab |
| Strain EAG28 spn-4p::jGCaMP7s::pie-1u; unc-119(ed3) III; ltIs44 V. | Caenorhabditis Genetics Center (CGC) | EAG28 | Created by Kim Guisbert Lab |
| Strain JIM113 ujIs113 II [pie-1p::mCherry::H2B::pie-1 3'UTR + nhr-2p::his-24::mCherry::let-858 3'UTR + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | JIM113 | Created by E. Preston - Murray Lab |
| Strain OD70 unc-119(ed3) III; ltIs44 V [pie-1p::mCherry::PH(PLC1delta1) + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | OD70 | Created by Audhya/Oegema - Greenstein Lab |
| Tritech Worm Pick Handle | TriTech | TWPH1 | |
| Yeast extract | IBI Scientific | IB49160 | 500 g |
Riferimenti
- Stein, P., Savy, V., Williams, A. M., Williams, C. J. Modulators of calcium signalling at fertilization. Open Biol. 10 (7), 200118 (2020).
- McAvey, B. A., Wortzman, G. B., Williams, C. J., Evans, J. P. Involvement of calcium signaling and the actin cytoskeleton in the membrane block to polyspermy in mouse eggs. Biolo Reprod. 67 (4), 1342-1352 (2002).
- Takayama, J., Onami, S. The sperm TRP-3 channel mediates the onset of a Ca 2+ wave in the fertilized C. elegans oocyte. Cell Rep. 15 (3), 625-637 (2016).
- Yamamoto, I., Kosinski, M. E., Greenstein, D. Start me up: Cell signaling and the journey from oocyte to embryo in C. elegans. Dev Dyn. 235 (3), 571-585 (2006).
- Hubbard, E. J. A., Greenstein, D. The Caenorhabditis elegans gonad: A test tube for cell and developmental biology. Dev Dyn. 218 (1), 2-22 (2000).
- Korta, D. Z., Hubbard, E. J. A. Soma-germline interactions that influence germline proliferation in Caenorhabditis elegans. Dev Dyn. 239 (5), 1449-1459 (2010).
- Toperzer, K. M., Brennan, S. J., Carroll, D. J., Guisbert, E. A., Kim Guisbert, ., S, K. Visualization of the biphasic calcium wave during fertilization in Caenorhabditis elegans using a genetically encoded calcium indicator. Biol Open. 12 (9), 059832 (2023).
- Samuel, A. D., Murthy, V. N., Hengartner, M. O. Calcium dynamics during fertilization in C. elegans. BMC Dev Biol. 1 (1), 8 (2001).
- Dana, H., et al. High-performance calcium sensors for imaging activity in neuronal populations and microcompartments. Nat Methods. 16 (7), 649-657 (2019).
- Golden, N. L., Plagens, R. N., Kim Guisbert, K. S., Guisbert, E. Standardized methods for measuring induction of the heat shock response in Caenorhabditis elegans. J Vis Exp. (161), e61030 (2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamath, R., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Kachur, T. M., Audhya, A., Pilgrim, D. B. UNC-45 is required for NMY-2 contractile function in early embryonic polarity establishment and germline cellularization in C. elegans. Dev Biol. 314 (2), 287-299 (2008).
- Takayama, J., Fujita, M., Onami, S. In vivo live imaging of calcium waves and other cellular processes during fertilization in Caenorhabditis elegans. Bio Protoc. 7 (7), e2205 (2017).
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391 (6669), 806-811 (1998).
- Conte, D., MacNeil, L. T., Walhout, A. J. M., Mello, C. C. RNA Interference in Caenorhabditis elegans. Curr Protoc Mol Biol. 109, 1-30 (2015).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421 (6920), 231-237 (2003).
- Reboul, J., et al. elegans ORFeome version 1.1: experimental verification of the genome annotation and resource for proteome-scale protein expression. Nat Genet. 34 (1), 35-41 (2003).
- Bui, Y. K., Sternberg, P. W. Caenorhabditis elegans inositol 5-phosphatase homolog negatively regulates inositol 1,4,5-triphosphate signaling in ovulation. Mol Biol Cell. 13 (5), 1641-1651 (2002).
- Muto, A., Kume, S., Inoue, T., Okano, H., Mikoshiba, K. Calcium waves along the cleavage furrows in cleavage-stage Xenopus embryos and its inhibition by heparin. J Cell Biol. 135 (1), 181-190 (1996).
- Paudel, S., Sindelar, R., Saha, M. Calcium signaling in vertebrate development and its role in disease. Int J Mol Sci. 19 (11), 3390 (2018).
- Paudel, S., Yue, M., Nalamalapu, R., Saha, M. S. Deciphering the calcium code: A review of calcium activity analysis methods employed to identify meaningful activity in early neural development. Biomolecules. 14 (1), 138 (2024).
- Goldstein, B., Hird, S. N. Specification of the anteroposterior axis in Caenorhabditis elegans. Development. 122 (5), 1467-1474 (1996).
- Johnston, W. L., Dennis, J. W. The eggshell in the C. elegans oocyte-to-embryo transition. Genesis. 50 (4), 333-349 (2012).
- Stevenson, Z. C., Moerdyk-Schauwecker, M. J., Jamison, B., Phillips, P. C. Rapid self-selecting and clone-free integration of transgenes into engineered CRISPR safe harbor locations in Caenorhabditis elegans. G3. 10 (10), 3775-3782 (2020).
- Zacharias, A. L., Walton, T., Preston, E., Murray, J. I. Quantitative differences in nuclear β-catenin and TCF pattern embryonic cells in C. elegans. PLoS Genet. 11 (10), e1005585 (2015).
- Webb, S. E., Miller, A. L. Calcium signalling during zebrafish embryonic development. BioEssays. 22 (2), 113-123 (2000).
- Wiseman, E., Carroll, D. J., Fowler, S. R., Guisbert, E. Iteration in an inquiry-based undergraduate laboratory strengthens student engagement and incorporation of scientific skills. J Scholarsh Teach Learn. 20 (2), 99-112 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon