Method Article
Visualisation in vivo des transitoires calciques pendant la fécondation et le développement précoce chez C. elegans
Dans cet article
Résumé
Ici, nous présentons des protocoles et des outils pour visualiser le calcium pendant la fécondation et l’embryogenèse précoce à l’aide d’un rapporteur de calcium génétiquement codé exprimé dans la lignée germinale du nématode modèle C. elegans.
Résumé
Le calcium est une molécule de signalisation importante lors de la transition ovocyte-embryon (OET) et de l’embryogenèse précoce. Le nématode hermaphrodite Caenorhabditis elegans offre plusieurs avantages uniques pour l’étude de l’OET car il est transparent et possède une gonade ordonnée qui produit un ovocyte mature toutes les ~23 min à 20 °C. Nous avons modifié l’indicateur calcique génétiquement codé jGCaMP7s pour indiquer par fluorescence le moment de la fécondation au sein d’un organisme vivant. Nous avons appelé ce rapporteur « CaFE » pour Calcium pendant la fécondation chez C. elegans. Le rapporteur CaFE a été conçu dans un locus de refuge en une seule copie, n’a pas d’impact significatif sur la physiologie ou la fécondité, et produit un signal robuste lors de la fécondation. Ici, une série de protocoles est présentée pour l’utilisation du rapporteur CaFE comme outil in vivo pour disséquer l’OET et l’embryogenèse. Nous incluons des méthodes pour synchroniser les vers, examiner les effets de l’inactivation de l’ARNi, monter des vers pour l’imagerie et pour visualiser le calcium dans les ovocytes et les embryons. De plus, nous présentons la génération de souches de vers supplémentaires pour aider à ce type d’analyse. Démontrant l’utilité du rapporteur CaFE pour visualiser le moment de la fécondation, nous rapportons que la double ovulation se produit lorsque ipp-5 est ciblé par l’ARNi et que seul le premier ovocyte subit une fécondation immédiate. De plus, la découverte de transitoires calciques unicellulaires au cours de l’embryogenèse précoce est rapportée ici, démontrant que le rapporteur CaFE persiste dans le développement précoce. Il est important de noter que le rapporteur CaFE dans les vers est suffisamment simple pour être incorporé dans les cours de laboratoire de recherche de premier cycle (CURE). Le rapporteur CaFE, associé à la gonade ordonnée et à la facilité de l’ARNi chez les vers, facilite l’étude de la dynamique cellule-cellule nécessaire pour réguler la fécondation interne et l’embryogenèse précoce.
Introduction
La fertilisation marque le début d’un nouveau cycle de vie, mais il est difficile de définir le moment précis de la fécondation. Une caractéristique conservée de la fécondation est une vague de calcium à travers l’ovocyte immédiatement après la fusion des spermatozoïdes1. Bien que la nature de l’onde calcique, en termes de fréquence et de vitesse, diffère d’une espèce à l’autre, presque tous les organismes présentent une augmentation transitoire du calcium intracellulaire après la fécondation. L’onde calcique joue un rôle essentiel dans le blocage de la polyspermie, l’activation des ovules et d’autres événements cellulaires importants2. Étant donné que l’onde calcique commence au site de fusion des spermatozoïdes, le calcium sert de marqueur pour la fécondation3.
Caenorhabditis elegans est un organisme modèle idéal pour l’étude du développement précoce. Les vers sont des hermaphrodites transparents qui sont génétiquement traitables4. Plus important encore pour l’étude de la fécondation, les adultes de C. elegans produisent continuellement des ovocytes en utilisant une gonade5 strictement ordonnée. En fait, les ovocytes sont les seules nouvelles cellules produites car le soma est post-mitotique à l’âge adulte6. La figure 1 met en évidence la gonade, qui se compose de 2 tubes symétriques en forme de U d’ovocytes en développement. L’ovocyte le plus proche de la spermathèque (organe de stockage des spermatozoïdes) est appelé ovocyte -1. Cette conception à la chaîne de montage de la gonade du ver ovule : un ovocyte mature dans la spermathèque toutes les ~23 min à 20 °C chez les jeunesadultes7. L’embryon nouvellement fécondé se déplace ensuite dans un utérus partagé avant d’être pondu à travers une seule vulve.
Les techniques précédentes pour visualiser le calcium pendant la fécondation chez C. elegans reposaient sur la micro-injection de colorants sensibles au calcium 3,8. Pour visualiser plus facilement l’onde calcique lors de la fusion spermatozoïde-ovule, un indicateur calcique génétiquement codé basé sur jGCaMP7s a été inséré en copie unique au niveau d’un locus refuge à l’aide de courtes répétitions palindromiques groupées et régulièrement espacées (CRISPR)7,9. Le rapporteur a été appelé CaFE pour Calcium pendant la fécondation chez C. elegans. Le rapporteur ne montre aucun défaut significatif de physiologie ou de fécondité.
Ici, des protocoles sont présentés pour la visualisation de l’onde calcique dans les ovocytes et les embryons de C. elegans à l’aide du rapporteur CaFE. Combiné à la myriade d’outils disponibles dans la communauté des vers, tels que l’interférence ARN (ARNi) et les marqueurs de gonades mCherry, le rapporteur CaFE facilite l’étude de la régulation des événements de fécondation interne, en particulier la compétence de fécondation et le moment de la fécondation. De plus, le rapporteur CaFE persiste dans le développement précoce et constitue un outil unique pour sonder l’embryogenèse.
Protocole
1. L’entretien de C. elegans
- Préparation de la plaque de milieu de croissance des nématodes (NGM)
- Mélanger 3 g de chlorure de sodium (NaCl), 2,5 g de Bacto-Peptone, 20 g de gélose et 1 L d’eau déminéralisée dans une fiole de 4 L (voir le tableau des matières). Couvrir le haut du ballon d’une feuille d’aluminium avant l’autoclavage.
- Autoclave pour stériliser à l’aide des paramètres du cycle de stérilisation liquide. Il est recommandé d’effectuer un cycle de stérilisation à 121 °C pendant 30 minutes.
- Laisser refroidir le mélange à ~50 °C, environ 20 min à température ambiante (RT).
- Ajouter 25 mL de 1 M KH2PO4 stérile (dissous dans l’eau, pH = 6), 1 mL de 1 M CaCl2 stérile (dissous dans l’eau), 1 mL de 1 M MgSO4 stérile (dissous dans l’eau) et 1 mL de cholestérol 5 mg/mL (dissous dans de l’éthanol à 100 %), en remuant doucement après chaque ajout (voir le tableau des matières).
- À l’aide d’une technique stérile, transférez une partie du mélange dans un bécher stérile de 300 ml pour faciliter le versement. Versez le média chaud pour couvrir le fond de l’assiette.
REMARQUE : Environ 10 ml par plaque dans des plaques de Pétri stériles de 60 mm x 15 mm (voir le tableau des matériaux) produiront 100 plaques. Alternativement, des boîtes de Pétri stériles de 35 mm x 10 mm peuvent être utilisées, où ~4 ml par plaque produiront ~250 plaques. - Laissez les plaques NGM sécher à RT pendant au moins 1 jour avant de semer avec des bactéries. Conservez les assiettes à l’envers à 4 °C pour une utilisation future.
- Préparation bactérienne de la pelouse OP50
- Préparez le bouillon Luria (LB) en combinant 5 g d’extrait de levure, 10 g de Bacto-Tryptone, 5 g de NaCl et 1 L d’eau déminéralisée dans une fiole de 2 L (voir le tableau des matériaux). À l’aide d’une plaque d’agitation et d’un barreau, mélanger jusqu’à dissolution.
- Aliquote LB dans des bouteilles et l’autoclave en utilisant les paramètres du cycle de stérilisation liquide. Il est recommandé d’effectuer un cycle de stérilisation à 121 °C pendant 30 minutes. Si vous utilisez un récipient avec un bouchon à vis, desserrez le bouchon à vis d’un quart de tour pour une bonne ventilation pendant le cycle d’autoclave.
- Laisser refroidir le bouillon à RT avant de l’utiliser.
- À l’aide d’une technique stérile, inoculer 50 mL de LB dans un erlenmeyer de 250 mL avec la bactérie OP50 et faire pousser pendant la nuit à 37 °C en agitant.
- À l’aide d’une technique stérile, transférez 300 μL de culture saturée au centre d’une plaque NGM de 60 mm x 15 mm. Pour les plaques de 35 mm x 10 mm, 100 μL de culture saturée sont recommandés.
- Laissez les plaques sécher à RT pendant 1 à 3 jours jusqu’à ce que la tache bactérienne OP50 ne soit plus liquide. Assurez-vous que les plaques sont sèches et exemptes de contamination avant utilisation. Conservez les plaques à 4 °C pour une utilisation future. Le stockage des plaques à l’envers réduit la condensation à la surface de la plaque.
- Construction à vis sans fin
- Desserrez le collier argenté vers la tête de la poignée du pic à vis sans fin (voir tableau des matériaux) en dévissant doucement pour que les quatre sections se séparent.
- Coupez 1,5 pouce (environ la longueur du pouce) de fil de platine (voir le tableau des matériaux) à l’aide de ciseaux et placez-le au milieu des quatre sections ; environ un pouce dépassera de la poignée.
- Tenez le fil au centre tout en serrant le collier ; Les quatre sections se rejoindront au milieu et maintiendront le fil de platine en place.
- Façonnez soigneusement le fil à l’aide d’une paire de pinces à bec plat. Une forme d’arc-en-ciel ou d’arc a tendance à mieux fonctionner pour la cueillette.
- Créez un « pied » d’environ 2-3 mm à l’extrémité du fil en serrant à plusieurs reprises avec la pince à bec plat. Utilisez ce « pied » pour ramasser les vers et gardez le « pied » à plat sur la surface de la plaque.
REMARQUE : Un serrage trop fort coupera le fil de platine. Évitez de créer des arêtes vives à l’aide de la pince à bec plat (ou équivalent) pour éviter de percer les vers ou les plaques.
- Manipulation des vers sans fin
- Tenez le cure-vis comme un crayon et stérilisez le « pied » en plaçant le fil dans la flamme d’une lampe à 95 % d’éthanol jusqu’à ce qu’il devienne orange vif.
- Placez une plaque ensemencée OP50 sur la platine d’une lunette de dissection et retirez le couvercle. Touchez doucement le « pied » de la pioche à une zone de la pelouse bactérienne et utilisez un léger mouvement de balayage pour rassembler les bactéries sur le fond. Les bactéries qui sont collées à la pioche (appelées « gluantes ») agiront comme une colle pour ramasser les vers.
- Déplacez une plaque de vers vers vers l’étape de la lunette de dissection et retirez le couvercle tout en empêchant le pic de toucher d’autres surfaces. Touchez rapidement et doucement le « pied » de la pioche sur une vis sans fin pour la ramasser. Cette technique permet de déplacer simultanément plusieurs vers entre les plaques.
- Touchez doucement le « pied » de la pioche sur la partie non ensemencée d’une nouvelle plaque pour transférer les vers ; En règle générale, il s’agit de la même plaque sur laquelle les bactéries ont été prélevées à la section 1.4.2.
- Stérilisez le pic entre chaque plaque et/ou souche de ver pour éviter la contamination et laissez le pic refroidir après la stérilisation.
REMARQUE : Toucher une pioche chaude sur un ver stressera ou tuera l’animal. Le pic à vers refroidira après avoir touché la pelouse bactérienne pour ramasser la « glu » pour le transfert des vers.
- Maintenir les vers à 20 °C sur des plaques NGM (étape 1.1) ensemencées avec la bactérie OP50 Escherichia coli (étape 1.2). Passage des vers 2 à 3 fois par semaine en transférant plusieurs adultes dans des assiettes fraîches (60 mm x 15 mm) à l’aide d’un cure-vers (étapes 1.3-1.4) pour éviter la famine, ce qui peut altérer la physiologie.
REMARQUE : Les souches de C. elegans et la bactérie OP50 sont disponibles auprès du Centre de génétique de Caenorhabditis (CGC).
2. Synchronisation développementale de C. elegans
- Synchronisez l’âge des vers à l’aide d’une ponte d’œuf chronométrée (décrite ici). Si un grand nombre de vers sont nécessaires pour une salle de classe ou à d’autres fins, effectuez la synchronisation de l’eau de Javel comme décrit dans Golden et al.10.
- Transférez les vers adultes dans une nouvelle plaque OP50 ensemencée à l’aide d’un pic en fil de platine (décrit à la section 1). Le nombre de vers est dicté par le besoin expérimental. Les vers du jour 1 pondent environ 3 œufs par heure. Par conséquent, 10 vers généreront environ 30 œufs en 1 h. De préférence, utilisez des vers le premier jour de l’âge adulte (jour 1).
- Après environ 1 h, retirez les vers adultes de la plaque, ne laissant que les œufs.
REMARQUE : Laisser par inadvertance un ver derrière soi est une erreur courante. Les vers qui restent continueront à pondre des œufs et perturberont la synchronisation. Les vers atteignent l’âge adulte du jour 1 environ 72 heures après leur ponte lorsqu’ils sont incubés à 20 °C.
3. Montage des vis sans fin pour l’imagerie
- Lors de l’imagerie des événements de fécondation, utilisez les adultes du jour 1 pour maximiser les possibilités de visualisation.
REMARQUE : Chez les adultes du jour 1, un ovocyte est fécondé toutes les ~23 min. Cependant, l’ovulation ralentit avec l’âge, et un hermaphrodite typique cessera de pondre des œufs après le jour 4. - Préparez une solution d’agarose à 3 % dans l’eau en mélangeant 3 g d’agarose dans 100 mL d’eau. Chauffez la solution d’agarose à l’aide d’un micro-ondes jusqu’à ce que l’agarose soit complètement dissoute. Pour réutiliser une solution solidifiée, chauffer jusqu’à dissolution complète. Après plusieurs utilisations, générez une solution fraîche.
- Placez une lame de microscope vierge pour l’imagerie entre deux autres lames de microscope qui ont une seule couche de ruban adhésif de laboratoire sur l’axe long de la lame (voir le tableau des matériaux). Le ruban crée une entretoise pour le tampon d’agarose, permettant une répartition uniforme et une largeur uniforme. Assurez-vous que les trois lames sont alignées avec les côtés longs qui se touchent : deux lames avec du ruban adhésif qui flanquent une lame de microscope vierge.
- À l’aide d’une pipette de 1 ml, déposer la solution d’agarose chauffée à 3 % (~100-150 μL) au centre de la lame de microscope, entre les lames collées.
- Placez rapidement une nouvelle lame de microscope sur la gouttelette d’agarose, en vous assurant que la nouvelle lame est perpendiculaire à la lame avec la gouttelette d’agarose et repose sur les lames adjacentes collées.
- Séparez délicatement les lames. Évitez de déchirer le tampon.
- Préparez les coussinets d’agarose frais chaque jour d’imagerie. Il est recommandé de fabriquer plusieurs pastilles lors de la préparation de l’imagerie, mais évitez d’exposer plus d’une pastille en même temps. Les tampons exposés se dessèchent rapidement.
- À l’aide d’une pipette de 20 μL, ajoutez une petite goutte (~7 μL) de 1 mM de lévamisole dans un tampon M9 au centre du tampon d’agarose pour paralyser les vers. Utilisez une solution mère de 100 mM de lévamisole stérilisée par filtre pour obtenir la solution de travail de 1 mM.
- Pour faire un tampon M9, combinez 6 g de Na2HPO4, 3 g de KH2PO4, 0,5 g de NaCl, 1 g de NH4Cl et 1 L d’eau déminéralisée dans un récipient approprié (voir le tableau des matériaux).
- Autoclavez la solution en utilisant les paramètres du cycle de stérilisation liquide et laissez la solution refroidir à RT avant utilisation. Si vous utilisez un récipient avec un bouchon à vis, desserrez le bouchon à vis d’environ un quart de tour pour une bonne ventilation. Il est recommandé d’effectuer un cycle de stérilisation à 121 °C pendant 30 minutes.
- Choisissez au moins 5 vers synchronisés et touchez doucement le « pied » à la gouttelette de lévamisole pour transférer tous les vers. Utilisez au moins 5 vers par lame, car l’orientation du corps peut amener l’intestin à obscurcir la lignée germinale, bloquant la visualisation de l’onde calcique dans l’ovocyte.
REMARQUE : Assurez-vous que les vers sont transférés rapidement car le lévamisole s’évaporera s’il est laissé sur le tampon d’agarose pendant plus de 5 minutes. - Couvrez soigneusement le tampon d’agarose avec une lamelle. Vérifiez l’emplacement des vers, car la lamelle peut provoquer la dispersion des vers dès leur placement initial. Étiquetez la lame de microscope avec les détails appropriés, tels que le nom de la souche de ver (voir le tableau des matériaux).
REMARQUE : Assurez-vous que l’imagerie est effectuée dès que possible après l’installation des vers, car une exposition prolongée au lévamisole est toxique.
4. Imagerie du calcium pendant la fécondation chez C. elegans
- Localisez les vers à l’aide d’un objectif de faible puissance, tel que 4x, sur le microscope de votre choix. Utilisez un éclairage en fond clair.
- Utilisez un objectif aérien d’au moins 20x pour visualiser clairement l’onde calcique dans les ovocytes. Utilisez un objectif d’immersion de 40x ou plus.
- Selon le système utilisé, l’intensité d’excitation est généralement une valeur fixe. Pour une visualisation optimale des rapporteurs fluorescents, ajustez le gain ou le temps d’exposition.
REMARQUE : Une lumière continue de haute intensité endommagera l’échantillon et est délétère pour la santé des vers. Si vous le souhaitez, éteignez le laser en attendant un événement de fécondation pour éviter un éclairage GFP continu. Voir les caractéristiques d’un événement de fécondation aux sections 4.8 et 4.9. - Assurez-vous que le canal GFP de 488 nm est réglé sur une puissance laser où le signal rapporteur CaFE est facilement visible.
- Pour un confocal à balayage laser Nikon ECLIPSE Ti2, détectez le rapporteur CaFE à une puissance laser de 30 % pour le canal GFP et un gain de 75. La taille du sténopé pour le balayage laser confocal Nikon ECLIPSE Ti2 est de 30 μm.
- Pour le disque rotatif Andor Dragonfly confocal à l’aide de la caméra Zyla, détectez le rapporteur CaFE à une puissance laser de 30 % pour le canal GFP. La taille du sténopé pour le disque rotatif Andor Dragonfly confocal est de 40 μm.
- Ajustez les paramètres laser et le temps d’exposition pour chaque échantillon examiné.
- Si vous utilisez l’EAG28 (CaFE ; PH-mCherry) ou EAG25 (CaFE ; H2B-mCherry), assurez-vous que le canal rouge est réglé sur une puissance laser où le signal mCherry est facilement visible en plus du canal GFP.
- Pour le confocal à balayage laser Nikon ECLIPSE Ti2, détectez le rapporteur mCherry à une puissance laser de 15 pour le canal RFP et un gain de 130.
- Pour le disque rotatif Andor Dragonfly confocal à l’aide de la caméra Zyla, détectez le rapporteur mCherry à une puissance laser de 30 % pour le canal RFP.
- Ajustez les paramètres laser et le temps d’exposition pour chaque échantillon examiné.
- Réglez la fréquence d’images ou le nombre d’images par seconde (ips) aussi rapidement que le système le permet.
REMARQUE : Le rapporteur CaFE est généralement observé à environ 1 ips pour le confocal à balayage laser Nikon ECLIPSE Ti2 et 10 ips pour le disque rotatif Andor Dragonfly confocal avec l’appareil photo Zyla. - Sélectionnez le protocole time lapse ou time series et ajustez les paramètres souhaités. Acquérez des images en accéléré pendant 30 minutes ou moins, avec l’option de répétitions réglée sur « 1 ».
REMARQUE : si l’événement de fertilisation se produit avant la fin de l’intervalle de temps, arrêtez la vidéo pour préserver la taille du fichier. - Localisez les vers sur la lame et sélectionnez un ver où la gonade proximale est visible. L’intestin et la gonade se tordent l’un autour de l’autre à l’intérieur du ver. En tant que tel, un bras gonadique est souvent obscurci par l’intestin, empêchant la visualisation de la fécondation. Si l’ovocyte -1 est masqué, sélectionnez un autre ver pour la visualisation. Pour augmenter la probabilité que le ver sélectionné ait un bras gonadique visible, montez un minimum de 5 vers par lame. Utilisez les adultes du jour 1 précoce pour visualiser la fécondation.
- Examinez chaque ovocyte -1 dans les bras gonades du ver pour déterminer si un événement de fécondation est imminent.
REMARQUE : Avant la fécondation, pendant la maturation méiotique, le noyau de l’ovocyte -1 migrera vers l’arrière de l’ovocyte et l’enveloppe nucléaire se dissoudra. Ces événements sont visibles à la fois dans les canaux Nomarksi et GFP à l’aide du rapporteur CaFE, et ces événements indiquent que l’ovulation se produira dans les ~10 prochaines minutes. - Après la rupture de l’enveloppe nucléaire, le cytosquelette de l’ovocyte -1 le plus proximal se réarrange, prend un aspect arrondi et se sépare de l’ovocyte -2. À ce stade, l’ovulation se produira de manière imminente. Lancez immédiatement l’enregistrement de la vidéo si ces événements sont observés. Prenez des vidéos dans un cadre large.
REMARQUE : Si le mouvement du noyau ou l’arrondi de l’ovocyte n’est pas visible après 20 min, il est peu probable qu’un événement d’ovulation se produise. Concentrez-vous sur un autre ver et/ou préparez une nouvelle lame.
5. Quantification des transitoires calciques dans la fécondation de C. elegans
- Quantifiez les transitoires calciques à partir de vidéos de fertilisation à l’aide du logiciel du système de microscope comme Nikon NIS Elements (décrit ici). ImageJ/FIJI conviennent également à l’analyse d’images11.
- Pour quantifier la variation de fluorescence des vidéos de fécondation, utilisez les valeurs de signal suivantes : le signal au temps 0, le signal de l’image d’intérêt et le signal de base.
- Identifiez le moment et l’image qui capturent la première poussée de fluorescence dans l’ovocyte -1. Définissez cette image comme le temps 0.
- Pour obtenir le signal du cadre d’intérêt, dessinez la région d’intérêt (ROI) pour englober les zones suivantes : l’ovocyte -1 avant la fécondation, la spermathèque et l’ovocyte nouvellement fécondé dans l’utérus. Le signal affiché dans le ROI est appelé F1.
REMARQUE : Assurez-vous que le retour d’intérêt couvre toute la zone fréquentée par l’ovocyte. Le retour sur investissement doit être identique dans chaque image de l’enregistrement time-lapse analysé. - Définissez une zone vide de l’image sans fluorescence en utilisant les mêmes dimensions que le ROI défini à l’étape 5.4 afin de calculer le signal d’arrière-plan dans chaque image. Soustrayez le signal d’arrière-plan de chaque image.
- Pour obtenir le signal de base (F0), localisez l’image dans un minimum de 15 images avant le temps 0. En utilisant le même retour sur investissement qu’à l’étape 5.4, notez le signal à chaque image jusqu’à l’instant 0. Faites la moyenne des signaux (avec soustraction de l’arrière-plan) de l’image de départ jusqu’au temps 0 pour calculer le signal de base.
- Calculez la variation de fluorescence (ΔF) à l’aide de la formule suivante :
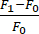 . F1 est défini dans chaque trame comme le signal dans le ROI sans le signal de fond. e F0 est le signal fluorescent de référence moyen de ≥15 images avant le temps 0, tel que calculé à la section 5.6.
. F1 est défini dans chaque trame comme le signal dans le ROI sans le signal de fond. e F0 est le signal fluorescent de référence moyen de ≥15 images avant le temps 0, tel que calculé à la section 5.6. - Déterminez le taux de variation entre les images en soustrayant la variation de fluorescence entre 2 images et en divisant par le temps.
6. ARNi chez C. elegans
- Préparer les plaques d’ARNi
- Préparez les plaques conformément à l’étape 1.1 et ajoutez les réactifs supplémentaires suivants pour permettre l’inactivation du gène par l’ARNi à l’étape 6.1.4.
- Ajouter 1 mL d’isopropyle β-d-1-thiogalactopyranoside (IPTG) filtré stérilement (IPTG) (dissous dans de l’eau désionisée) pour une concentration finale de 1 mM d’IPTG dans le milieu (voir le tableau des matériaux).
- Ajouter 1 mL d’ampicilline filtrée stérile à 100 mg/mL (dissoute dans de l’eau désionisée) pour obtenir une concentration finale de 100 μg/mL d’ampicilline dans le milieu (voir le Tableau des matières). Vous pouvez également utiliser 40 μg/mL de carbénicilline12.
- Agitez doucement mais soigneusement pour mélanger et versez les assiettes comme décrit aux étapes 1.1.4 à 1.1.5.
- Préparation d’une culture saturée en ARNi
- Calculez la quantité de culture saturée en multipliant le nombre de plaques d’ARNi ensemencées nécessaires par 300 μL lorsque vous utilisez des plaques de 60 mm x 15 mm ou par 100 μL lorsque vous utilisez des plaques de 35 mm x 10 mm.
- À l’aide d’une technique stérile, faire pousser les bactéries ARNi souhaitées dans des bactéries LB (décrites aux étapes 1.2.1 à 1.2.3) contenant 100 μg/mL d’ampicilline pendant une nuit à 37 °C jusqu’à saturation. Incluez une souche bactérienne contenant le vecteur vide L4440 comme témoin négatif et une souche bactérienne contenant un témoin positif à l’ARNi (p. ex., œuf-5) pour vérifier que l’ARNi fonctionne. De plus, incluez une condition « vierge » contenant uniquement LB pour vérifier qu’il n’y a pas de contamination dans le LB d’origine.
- Ajouter de l’IPTG filtré stérile à une concentration finale de 1 mM à la culture de nuit saturée pour l’induction de l’expression de l’ARNdb et incuber pendant au moins 2 h 30 min à 37 °C avant l’ensemencement. Alternativement, diluer la culture de nuit saturée hors de la phase logarithmique avant de l’induire avec IPTG.
REMARQUE : L’IPTG est inclus à la fois dans les plaques et dans la culture saturée pour une expression optimale de l’ARNdb. - À l’aide d’une technique stérile, transférez 300 μL de la culture induite au centre d’une plaque d’ARNi de 60 mm x 15 mm (solidifiée). Vous pouvez également utiliser 100 μL de culture saturée pour une plaque de 35 mm x 10 mm.
- Laissez les plaques sécher à RT pendant 1 à 3 jours jusqu’à ce que la tache bactérienne ARNi ne soit plus liquide. Assurez-vous que les plaques sont sèches et exemptes de contamination avant utilisation. Conservez les assiettes à 4 °C à l’envers pour une utilisation future.
REMARQUE : Utilisez les plaques d’ARNi dès que possible pour un renversement optimal, car l’efficacité de l’ARNi diminue avec le temps. Jeter si >1 mois.
- Comme décrit dans la section 2, synchronisez les vers sur les plaques ARNi.
- Vérifiez les vers 1 à 2 jours après la synchronisation de la ponte pour détecter des retards de développement.
- Certains ARNi peuvent provoquer des anomalies du développement lorsque l’inactivation commence à la ponte. Si nécessaire, placez les vers sur les plaques d’ARNi à un stade ultérieur, comme L3.
- Comme décrit dans les sections 3 et 4, montez les vers et préparez-les pour l’imagerie à l’aide de tout système de microscope équipé pour détecter la fluorescence GFP.
7. Imagerie du calcium au cours de l’embryogenèse précoce chez C. elegans
- Synchronisez les vers comme décrit dans la section 2. Utilisez la souche EAG28, qui combine le rapporteur CaFE et un marqueur pleckstrin-homology domain-mCherry mettant en évidence la limite de la membrane plasmique. Alternativement, pour visualiser les embryons fécondés ex utero, coupez doucement le ver le long de l’utérus à l’aide d’une aiguille de seringue stérile pour libérer les embryons.
- Préparez les vers ou les embryons pour l’imagerie comme décrit dans la section 3 et suivez les mêmes instructions décrites dans la section 4 pour le réglage des paramètres d’imagerie.
- Centrez la zone d’intérêt sur l’utérus du ver pour visualiser des embryons ou un œuf pondu.
- Pour les embryons in utero, assurez-vous que l’utérus n’est pas derrière l’intestin, que l’utérus n’est pas surpeuplé et que les cellules individuelles de l’embryon sont claires dans le plan focal.
- Pour les œufs ex utero, assurez-vous que les étapes des cellules ne dépassent pas le stade de la virgule du cycle de vie du ver.
- Sélectionnez le protocole time lapse ou time series et ajustez les paramètres souhaités.
- Pour une visualisation optimale des transitoires calciques embryonnaires, augmentez les paramètres de temps pour les films prolongés. Gardez à l’esprit qu’une exposition constante au laser endommagera l’embryon sur de longues périodes.
- Capturez des vidéos ou des images en vous concentrant sur les cellules des embryons au stade cellulaire souhaité.
- Utilisez un objectif à immersion 40x ou plus pour l’imagerie, bien que les transitoires soient facilement détectés à l’aide d’un objectif aérien 20x.
Résultats
En utilisant les protocoles décrits dans ce manuscrit, les modèles dynamiques de signalisation calcique dans la fécondation et l’embryogenèse ont été observés chez C. elegans.
La figure 2 montre une séquence de fécondation typique chez les vers contenant le rapporteur CaFE. Pour faciliter l’analyse, la souche EAG28 a été utilisée, qui combine le rapporteur CaFE avec un transgène13 du domaine d’homologie de la pleckstrine (PH-mCherry). Le marqueur PH-mCherry se localise dans la membrane plasmique et permet de visualiser plus facilement les limites cellulaires, en particulier dans le bras gonadique. La souche EAG28 a été créée en croisant la souche EAG16 contenant le rapporteur CaFE avec la souchedéclarante OD70 PH-mCherry 13. Chez C. elegans, l’ovulation et la fécondation se produisent simultanément. Le temps 0 de la fécondation est considéré comme la première image où un signal de fluorescence est détecté dans l’ovocyte en ovulation (Figure 2). Une brillante poussée de fluorescence se produit sur le site de fusion des spermatozoïdes dès que le bord d’attaque de l’ovocyte pénètre dans la spermathèque, l’organe de stockage des spermatozoïdes7. Le signal apparaît avant la fin de l’ovulation. L’onde calcique est biphasique, avec une poussée initiale rapide suivie d’une onde de fluorescence du point d’entrée vers le pôle opposé. L’ovocyte entier devient fluorescent en <30 s.
Notez que le rapporteur CaFE est en une seule copie, et que, par conséquent, le signal n’est pas aussi brillant que celui des autres transgènes. Le time-lapse Z-stack n’est pas recommandé pendant l’ovulation à moins qu’un disque confocal rotatif ne soit disponible et optimisé. Des images de la pile Z peuvent être obtenues avant ou après un film pour capturer les changements 3D de la morphologie des gonades. Bien que les images ici aient été prises avec un microscope confocal, les transitoires ont également été observés à l’aide de la microscopie à fluorescence à grand champ, qui est plus courante et abordable que la microscopie confocale. En utilisant un confocal à balayage laser, la fréquence d’images est d’environ 1 ips. À l’aide d’un disque confocal rotatif, la fréquence d’images est de 10 ips ou plus. Le signal de fond du rapporteur CaFE est suffisant pour éclairer les ovocytes en maturation dans le bras gonadique. Lors de la fécondation, une augmentation de 1,5 à 2 fois de l’intensité du signal GFP est généralement observée.
Plusieurs méthodes alternatives de quantification existent. Par exemple, le logiciel d’analyse d’images Imaris, créé par Oxford Instruments (la même société mère que le microscope confocal à disque rotatif Andor Dragonfly), a la capacité de quantifier le signal dans chaque image à partir de l’ovocyte en transit uniquement au lieu d’un retour sur investissement plus important. Cependant, le logiciel Imaris n’est pas gratuit. De plus, une stratégie d’analyse d’images détaillée pour mesurer les ondes calciques dans l’ovocyte a été décrite en détail par Takayama, Fujita et Onami et utilise ImageJ, qui est disponible gratuitement14. Alternativement, les kymographes pour illustrer l’onde du signal fluorescent à travers un seul ovocyte nécessitent une manipulation d’image plus étendue que celle décrite ici, mais sont décrites dans Takayama et Onami3.
Un avantage distinct de C. elegans est sa capacité à inhiber l’expression de presque tous les gènes en nourrissant les bactéries du ver exprimant l’ARNdb pour déclencher l’ARNi endogène15,16. Pour une expression optimale de l’ARNdb, les plaques d’ARNi et la culture d’ARNi saturée contiennent de l’IPTG. L’ARNdb est produit en exprimant le gène d’intérêt avec des promoteurs flanquants. Ici, nous avons utilisé la bibliothèque Ahringer qui cible la majeure partie du génome du ver grâce à l’utilisation de 16 256 souches bactériennes17. Alternativement, un ORFeome de ~11 000 clones d’ARNi a été créé par le laboratoire Vidal à l’aide du système Gateway et est disponible à l’achat via Horizon Discovery18.
Pour démontrer l’utilité du rapporteur CaFE en combinaison avec l’ARNi, nous avons examiné l’ovulation prématurée. Une délétion partielle dans ipp-5 induit un double événement d’ovulation où l’ovocyte -1 et l’ovocyte -2 pénètrent dans la spermathèque au cours de la même ovulation19. L’IPP-5 est une phosphatase qui agit sur IP3 et diminue efficacement les niveaux du deuxième messager IP3. Nous avons constaté que l’inactivation de l’ARNi d’ipp-5 provoque un phénotype de double ovulation similaire à celui du mutant.
L’analyse du rapporteur CaFE pendant la double ovulation induite par l’inactivation de l’ARNi ipp-5 a révélé de nouveaux aspects de l’OET. Tout d’abord, un signal calcique a été observé immédiatement lors de l’entrée dans la spermathèque dans l’ovocyte de tête -1 mais pas dans l’ovocyte de queue -2 (Figure 3). Bien que 2 ovocytes soient ovulés, l’ovocyte -1 reste le seul ovocyte qui montre des signes de bonne maturation, en particulier la rupture de l’enveloppe nucléaire (NEBD). Ces données suggèrent que même si l’ovocyte -2 est en présence de spermatozoïdes, il n’est pas apte à être fécondé car il n’a pas encore atteint sa bonne maturité. Deuxièmement, l’ovocyte -2 présente une onde calcique retardée, généralement lorsque l’ovocyte sort de la spermathèque pour l’utérus. Bien que la maturation de l’ovocyte soit normalement une condition préalable à l’ovulation, ces données suggèrent que la maturation de l’ovocyte et la compétence pour la fécondation peuvent encore se produire après l’ovulation. La visualisation de l’onde calcique retardée lors de l’inactivation d’ipp-5 met en évidence les avantages du rapporteur CaFE pour interroger la compétence et le moment de la fécondation.
De plus, nous avons découvert que le rapporteur CaFE est détectable dans les œufs pondus et révèle des transitoires calciques unicellulaires au cours de l’embryogenèse précoce chez C. elegans. Embryons d’EAG28 adultes synchronisés du jour 1 (CaFE ; PH-mCherry) ont été imagés à l’aide des protocoles des sections 1 à 5 avec le disque rotatif Andor Dragonfly confocal à l’aide de la caméra Zyla (Figure 4). Les transitoires calciques sont confinés dans des cellules uniques et prennent moins de temps que l’onde calcique de fécondation (~9 s, n = 11). Cependant, l’onde calcique au cours de l’embryogenèse ne semble pas être biphasique. Les transitoires calciques n’ont pas été observés avant le stade 8 cellules. Notamment, le calcium ne se localise pas dans le sillon de clivage chez C. elegans, comme on le voit dans de nombreux autres organismes, y compris les humains et le xénope 20,21,22. Des transitoires calciques unicellulaires ont été observés bien après la gastrulation (~200 min après la fécondation) et dans les œufs pondus. Un seul embryon présente plusieurs transitoires calciques au fil du temps, mais dans différentes cellules et généralement une cellule à la fois. Notez qu’une exposition continue à la stimulation laser endommagera les embryons. Une puissance LED/laser plus faible ou une exposition discontinue est recommandée. Cependant, comme les ondes calciques sont relativement courtes à ~9 s, nous ne recommandons pas une stimulation plus lente de 2 s entre les événements de stimulation LED/laser.
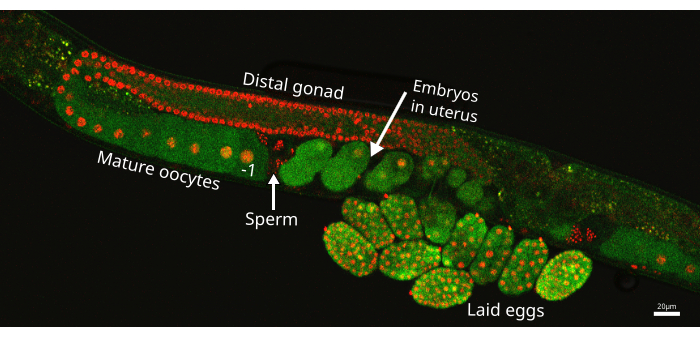
Figure 1 : Bras de la gonade de C. elegans. Image d’un bras gonadique de la souche de ver EAG25, montrant le rapporteur CaFE GFP (vert) et le marqueur d’histone 2B-mCherry (rouge), qui met en évidence les noyaux dans la lignée germinale. L’ovocyte proximal est désigné par le -1 directement avant les spermathèques, où les spermatozoïdes sont stockés (flèche). L’embryon situé le plus près de la spermathèque mais à l’intérieur de l’utérus est l’embryon le plus récent. L’image a été prise à l’aide du confocal à balayage laser Nikon ECLIPSE Ti2. Barre d’échelle = 20 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
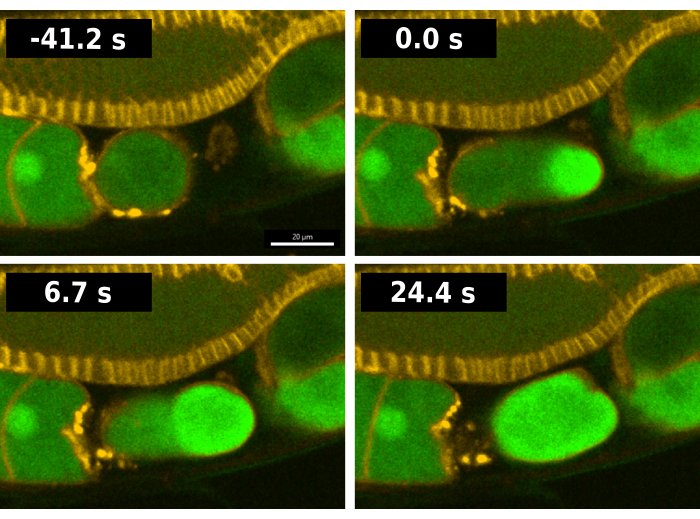
Figure 2 : Visualisation de l’onde calcique pendant la fécondation. Images chronologiques de la fluorescence reflétant le calcium intracellulaire pendant la fécondation dans la souche de ver EAG28. La souche EAG28 contient à la fois le rapporteur CaFE (vert) et une fusion mCherry (jaune) dans le domaine d’homologie de la pleckstrine, qui met en évidence les membranes plasmiques. Le temps 0 indique la première image représentant une nette augmentation du signal fluorescent dans l’ovocyte. Le temps 24,4 s reflète la première image montrant la fluorescence de l’ovocyte entier. Des images de séries temporelles ont été prises à l’aide du disque rotatif Andor Dragonfly confocal avec une caméra Zyla. Barre d’échelle = 20 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
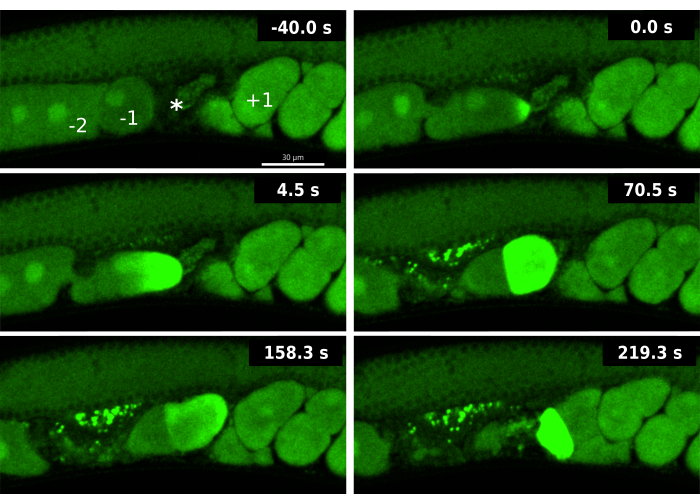
Figure 3 : Ondes calciques lors d’une double ovulation induite par l’ARNi ipp-5. Images en accéléré d’ondes calciques séquentielles de l’ovocyte -1 et de l’ovocyte -2 dans une souche EAG16 (CaFE en vert GFP) exposée à l’ARNi ipp-5. Le cadre -40,0 s comprend des étiquettes pour l’ovocyte -1 (-1), l’ovocyte -2 (-2), la spermathèque (*) et l’embryon +1 (+1). Le temps 0 s montre l’augmentation initiale de la fluorescence de l’onde calcique de l’ovocyte -1. Le temps 4,5 s montre que l’onde calcique se propage vers le pôle opposé dans l’ovocyte -1. Le temps de 70,5 s affiche la fluorescence de la cellule entière dans l’ovocyte -1 accompagné de l’ovocyte -2 au signal GFP de base ; Les deux ovocytes se trouvent dans la spermathèque. L’ovocyte fécondé -1 et l’ovocyte -2 non fécondé pénètrent dans l’utérus à l’heure 158,3 s. À 219,3 s, l’ovocyte -2 présente une onde calcique fluorescente tardive. Les images ont été prises à l’aide du disque rotatif Andor Dragonfly confocal avec une caméra Zyla. Barre d’échelle = 30 μM Veuillez cliquer ici pour voir une version plus grande de cette figure.
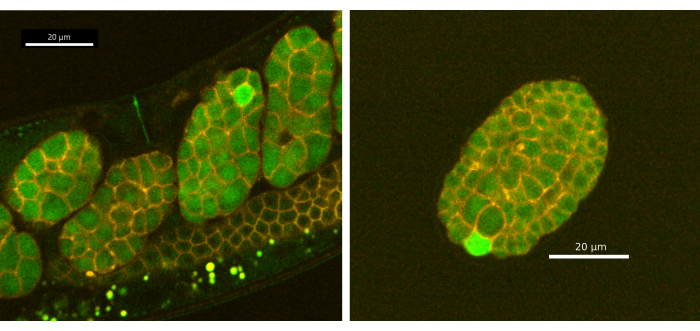
Figure 4 : Transitoires calciques unicellulaires au début de l’embryogenèse. (A) Les embryons in utero et (B) ex utero (œuf pondu) ont présenté un signal fluorescent dans une seule cellule pendant l’embryogenèse. Les images ont été prises à l’aide du disque rotatif Andor Dragonfly confocal avec une caméra Zyla. Barre d’échelle = 20 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Un outil simple avec un protocole robuste est une combinaison puissante pour s’attaquer à des questions scientifiques difficiles. Ici, des méthodes de visualisation du calcium en tant qu’indicateur facilement détectable de la fertilisation sont présentées à l’aide du rapporteur CaFE. Ce même rapporteur persiste jusqu’au début de l’embryogenèse et permet également de visualiser les transitoires calciques plus tard dans le développement. La signalisation calcique sert de deuxième messager critique qui délimite les changements majeurs dans la fonction cellulaire, en particulier pour la biologie du développement. Dans l’ovocyte, la première phase d’explosion de l’onde calcique marque non seulement le moment de la fécondation, mais aussi le site de fusion des spermatozoïdes3. Chez C. elegans, l’axe A-P est également déterminé par le site de fusion des spermatozoïdes23. Par conséquent, la capacité de visualiser le calcium dans les ovocytes et les embryons permet d’étudier des questions complexes qui sont au cœur de la biologie cellulaire et du développement.
La méthode décrite ici avec le rapporteur CaFE devrait être assez simple pour les novices en nématodes. Les méthodes précédentes pour détecter les ondes calciques dans les ovocytes de C. elegans reposaient sur l’injection de colorant 3,8,14. Bien que ces études aient été importantes et éclairantes, l’injection de colorant demande beaucoup de main-d’œuvre et le matériel d’injection n’est pas disponible dans tous les laboratoires.
Les protocoles présentés ici ont été optimisés pour les vers sains. Pour maximiser les chances de succès, assurez-vous qu’il n’y a pas de contamination sur le milieu de vers ou dans la pelouse bactérienne sur laquelle les vers se nourrissent. N’exposez pas les vers à des conditions de stress, telles que la température, car celles-ci affecteront l’ovulation et la fécondation. De plus, des contrôles de l’efficacité de l’ARNi doivent être inclus dans chaque expérience, car l’efficacité diminue avec le temps. Utilisez un contrôle positif pour l’efficacité de l’ARNi comme l’œuf 5, qui génère des embryons, mais pas de descendance viable car la formation de la coquille de l’œuf est compromise24. De plus, les paramètres de capture d’image doivent être optimisés pour chaque système de microscopie. Nos spécifications sont incluses ici à titre de référence, mais des écarts sont à prévoir. Bien que ce système détecte facilement le calcium cytoplasmique comme indicateur de la fécondation, il ne représente pas nécessairement un véritable événement de fusion spermatozoïdes-ovocytes.
Le large éventail d’outils et de mutants librement distribués au sein de la communauté des vers renforce l’utilité du rapporteur CaFE. Le rapporteur est intégré dans le génome du ver et est facilement croisé avec d’autres souches mutantes ou rapporteures de C. elegans 25. On rapporte ici la création d’EAG25 exprimant le rapporteur CaFE avec un marqueur d’histone H2B-mCherry pour visualiser les noyaux (Figure 1) et EAG28 avec le rapporteur CaFE et un marqueur pleckstrin-homology domain-mCherry, qui met en évidence la périphérie cellulaire (Figure 2)13,26. Les deux souches aident à la visualisation des cellules dans la lignée germinale et pendant l’embryogenèse. De plus, la facilité de l’ARNi dans
C. elegans, lorsqu’il est utilisé avec le rapporteur CaFE, a révélé de nouvelles connaissances sur la compétence en matière de fécondation. Comme le montre la double ovulation induite par l’inactivation de l’ARNi ipp-5 sur la figure 3, la présence de spermatozoïdes et d’un ovocyte ovulé est insuffisante pour stimuler un événement de fécondation.
Ces résultats indiquent qu’il doit exister un autre signal, ou l’absence d’inhibiteur, qui permet à l’ovocyte de devenir fécondé. L’ovocyte ovulé prématurément présente un transitoire calcique retardé lorsque l’ovocyte se déplace dans l’utérus. Cette onde calcique tardive suggère que l’ovocyte -2 ovulé prématurément peut développer une compétence de fécondation avec le temps. Nous prévoyons que les études sur le moment de la fécondation, en particulier en ce qui concerne la signalisation et la régulation cellule-cellule, seront facilitées par l’utilisation du rapporteur CaFE. De plus, le rapporteur CaFE persiste dans l’embryogenèse et présente des transitoires calciques unicellulaires. Cette signalisation calcique embryonnaire a également été signalée dans les premières 24 heures suivant le développement du poisson-zèbre27. Le rôle des transitoires calciques est inconnu, mais leur présence suggère un événement de signalisation cellulaire au cours du développement qui n’a pas encore été exploré. Notamment, les transitoires calciques n’ont pas été observés chez les zygotes nouvellement fécondés. Par conséquent, le calcium ne se localise pas dans le sillon de clivage comme cela a été documenté chez plusieurs autres organismes, y compris le xénope et les humains 20,21,22.
Il est important de noter que le CaFE reporter est suffisamment facile à utiliser pour être utilisé par des étudiants de premier cycle ayant une formation minimale. Nous avons conçu et exécuté un laboratoire CURE (expérience de recherche de premier cycle basé sur un cours) de 1 crédit pour les étudiants en biologie avec les souches et les protocoles décrits ici. Au cours d’un semestre, la classe s’est réunie une fois par semaine pendant 3 h ou deux fois par semaine pendant 1 h 30 min chacune. Les élèves ont eu le choix de travailler seuls ou en groupes de 2. Chaque élève/paire a sélectionné un gène différent à étudier à partir d’une liste organisée. Ils ont effectué l’ARNi contre le gène de leur choix dans la souche EAG28 et ont examiné les vers pour détecter les effets sur la fécondité, la fécondation et/ou la morphologie des gonades. Sur la base de leurs résultats et de leurs lectures de base à l’aide de la littérature primaire, les étudiants ont développé des hypothèses qu’ils ont pu tester dans des expériences ultérieures. Cette conception itérative était essentielle pour accroître l’engagement des étudiants28. Les étudiants ont acquis une expérience de recherche authentique et des compétences dans les organismes modèles, les cribles génétiques et la microscopie à fluorescence. Compte tenu de la facilité d’utilisation du rapporteur CaFE, les étudiants sans expérience de recherche ont pu réussir. Par la suite, la grande majorité des étudiants ont exprimé une préférence pour le format CURE par rapport aux cours de laboratoire traditionnels, de nombreux étudiants exprimant le désir de poursuivre leurs recherches. Ensemble, ces outils et protocoles contribuent à la fois à l’éducation et à la recherche sur les processus de développement précoce.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent ou financier.
Remerciements
KSKG a été financé par une subvention de l’Institut national Eunice Kennedy Shriver de la santé infantile et du développement humain (R15HD111986). Certaines souches ont été fournies par la CGC, qui est financée par le Bureau des programmes d’infrastructure de recherche des NIH (P40 OD4010440). Nous remercions WormBase.
matériels
| Name | Company | Catalog Number | Comments |
| Agar | Fisher Scientific | DF0140-07-4 | 2 kg; Powder dissolves easier than flakes |
| Agarose | MidSci | BE-A125 | 500 g |
| Alcohol lamp | Fisher Scientific | S13475 | Use with 95% ethanol |
| Ammonium chloride (NH4Cl) | Fisher Scientific | AAA1500030 | 250 g |
| Ampicillin | Fisher Scientific | BP1760-5 | 5 g |
| AMSCO 400 Series Small Steam Sterilizer | Steris Healthcare | N/A | |
| Bacto-peptone | Fisher Scientific | BP1420-500 | 500 g |
| Bacto-tryptone | Fisher Scientific | DF0123-17-3 | 500 g |
| Calcium chloride (CaCl2) | Fisher Scientific | C69-500 | 500 g |
| Cholesterol | Thermo Scientific | A11470.18 | 50 g |
| Dragonfly 200 spinning disk confocal | Oxford Instruments Andor | N/A | Used with Leica microscope |
| Fisherbrand Superfrost Cytogenics Microscope Slides | Fisher Scientific | 22-035-900 | 144 slides per pack |
| Flat Nose Pliers, Smooth Jaw | Home Depot | 305530604 | Ensure pliers are smooth jaw |
| Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Fisher Scientific | BP1755-10 | 10 g; dioxane-free |
| Laboratory tape | Fisher Scientific | 15-901-10R | 0.5 inch tape is used to tape microscope slides |
| Levamisole | Fisher Scientific | AC187870100 | 10 g |
| Magnesium sulfate (MgSO4) | Fisher Scientific | M63-500 | 500 g |
| Microscope cover glass | Fisher Scientific | 12541016 | 1 oz pack |
| Nikon ECLIPSE Ti2 laser scanning confocal | Nikon | N/A | |
| Nikon NIS Elements software | Nikon | N/A | Confocal |
| OP50 Escherichia coli | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Platinum Wire | TriTech | PT-9010 | |
| Potassium phosphate dibasic (K2HPO4) | Fisher Scientific | P288-500 | 500 g |
| Potassium phosphate monobasic (KH2PO4) | Fisher Scientific | AA1159436 | 500 g |
| Sodium chloride (NaCl) | Fisher Scientific | S271-500 | 500 g |
| Sodium phosphate dibasic heptahydrate (Na2HPO4) | Fisher Scientific | S471-3 | 3 kg |
| Stereo microscope | Leica | KL300 LED | |
| Sterile Petri dish (35 mm x 10 mm) | CellTreat | 229638 | 960 Petri dishes per case |
| Sterile Petri dish (60 mm x 15 mm) | CellTreat | 229665 | 500 Petri dishes per case |
| Strain EAG16 spn-4p::jGCaMP7s::pie-1u | Caenorhabditis Genetics Center (CGC) | EAG16 | Created by Kim Guisbert Lab |
| Strain EAG25 spn-4p::jGCaMP7s::pie-1u; ujIs113 II. | Caenorhabditis Genetics Center (CGC) | EAG25 | Created by Kim Guisbert Lab |
| Strain EAG28 spn-4p::jGCaMP7s::pie-1u; unc-119(ed3) III; ltIs44 V. | Caenorhabditis Genetics Center (CGC) | EAG28 | Created by Kim Guisbert Lab |
| Strain JIM113 ujIs113 II [pie-1p::mCherry::H2B::pie-1 3'UTR + nhr-2p::his-24::mCherry::let-858 3'UTR + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | JIM113 | Created by E. Preston - Murray Lab |
| Strain OD70 unc-119(ed3) III; ltIs44 V [pie-1p::mCherry::PH(PLC1delta1) + unc-119(+)] | Caenorhabditis Genetics Center (CGC) | OD70 | Created by Audhya/Oegema - Greenstein Lab |
| Tritech Worm Pick Handle | TriTech | TWPH1 | |
| Yeast extract | IBI Scientific | IB49160 | 500 g |
Références
- Stein, P., Savy, V., Williams, A. M., Williams, C. J. Modulators of calcium signalling at fertilization. Open Biol. 10 (7), 200118(2020).
- McAvey, B. A., Wortzman, G. B., Williams, C. J., Evans, J. P. Involvement of calcium signaling and the actin cytoskeleton in the membrane block to polyspermy in mouse eggs. Biolo Reprod. 67 (4), 1342-1352 (2002).
- Takayama, J., Onami, S. The sperm TRP-3 channel mediates the onset of a Ca 2+ wave in the fertilized C. elegans oocyte. Cell Rep. 15 (3), 625-637 (2016).
- Yamamoto, I., Kosinski, M. E., Greenstein, D. Start me up: Cell signaling and the journey from oocyte to embryo in C. elegans. Dev Dyn. 235 (3), 571-585 (2006).
- Hubbard, E. J. A., Greenstein, D. The Caenorhabditis elegans gonad: A test tube for cell and developmental biology. Dev Dyn. 218 (1), 2-22 (2000).
- Korta, D. Z., Hubbard, E. J. A. Soma-germline interactions that influence germline proliferation in Caenorhabditis elegans. Dev Dyn. 239 (5), 1449-1459 (2010).
- Toperzer, K. M., Brennan, S. J., Carroll, D. J., Guisbert, E. A., Kim Guisbert,, S, K. Visualization of the biphasic calcium wave during fertilization in Caenorhabditis elegans using a genetically encoded calcium indicator. Biol Open. 12 (9), 059832(2023).
- Samuel, A. D., Murthy, V. N., Hengartner, M. O. Calcium dynamics during fertilization in C. elegans. BMC Dev Biol. 1 (1), 8(2001).
- Dana, H., et al. High-performance calcium sensors for imaging activity in neuronal populations and microcompartments. Nat Methods. 16 (7), 649-657 (2019).
- Golden, N. L., Plagens, R. N., Kim Guisbert, K. S., Guisbert, E. Standardized methods for measuring induction of the heat shock response in Caenorhabditis elegans. J Vis Exp. (161), e61030(2020).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamath, R., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Kachur, T. M., Audhya, A., Pilgrim, D. B. UNC-45 is required for NMY-2 contractile function in early embryonic polarity establishment and germline cellularization in C. elegans. Dev Biol. 314 (2), 287-299 (2008).
- Takayama, J., Fujita, M., Onami, S. In vivo live imaging of calcium waves and other cellular processes during fertilization in Caenorhabditis elegans. Bio Protoc. 7 (7), e2205(2017).
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391 (6669), 806-811 (1998).
- Conte, D., MacNeil, L. T., Walhout, A. J. M., Mello, C. C. RNA Interference in Caenorhabditis elegans. Curr Protoc Mol Biol. 109, 1-30 (2015).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421 (6920), 231-237 (2003).
- Reboul, J., et al. elegans ORFeome version 1.1: experimental verification of the genome annotation and resource for proteome-scale protein expression. Nat Genet. 34 (1), 35-41 (2003).
- Bui, Y. K., Sternberg, P. W. Caenorhabditis elegans inositol 5-phosphatase homolog negatively regulates inositol 1,4,5-triphosphate signaling in ovulation. Mol Biol Cell. 13 (5), 1641-1651 (2002).
- Muto, A., Kume, S., Inoue, T., Okano, H., Mikoshiba, K. Calcium waves along the cleavage furrows in cleavage-stage Xenopus embryos and its inhibition by heparin. J Cell Biol. 135 (1), 181-190 (1996).
- Paudel, S., Sindelar, R., Saha, M. Calcium signaling in vertebrate development and its role in disease. Int J Mol Sci. 19 (11), 3390(2018).
- Paudel, S., Yue, M., Nalamalapu, R., Saha, M. S. Deciphering the calcium code: A review of calcium activity analysis methods employed to identify meaningful activity in early neural development. Biomolecules. 14 (1), 138(2024).
- Goldstein, B., Hird, S. N. Specification of the anteroposterior axis in Caenorhabditis elegans. Development. 122 (5), 1467-1474 (1996).
- Johnston, W. L., Dennis, J. W. The eggshell in the C. elegans oocyte-to-embryo transition. Genesis. 50 (4), 333-349 (2012).
- Stevenson, Z. C., Moerdyk-Schauwecker, M. J., Jamison, B., Phillips, P. C. Rapid self-selecting and clone-free integration of transgenes into engineered CRISPR safe harbor locations in Caenorhabditis elegans. G3. 10 (10), Bethesda. 3775-3782 (2020).
- Zacharias, A. L., Walton, T., Preston, E., Murray, J. I. Quantitative differences in nuclear β-catenin and TCF pattern embryonic cells in C. elegans. PLoS Genet. 11 (10), e1005585(2015).
- Webb, S. E., Miller, A. L. Calcium signalling during zebrafish embryonic development. BioEssays. 22 (2), 113-123 (2000).
- Wiseman, E., Carroll, D. J., Fowler, S. R., Guisbert, E. Iteration in an inquiry-based undergraduate laboratory strengthens student engagement and incorporation of scientific skills. J Scholarsh Teach Learn. 20 (2), 99-112 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon