Method Article
Cuantificación microscópica en tiempo real y de alto rendimiento de la liberación de trampas extracelulares de neutrófilos humanos y evaluación de la farmacología de los antagonistas
En este artículo
Resumen
Este protocolo definido describe un enfoque de microscopía de alto rendimiento en tiempo real para visualizar y cuantificar la liberación de trampas extracelulares (NET) de neutrófilos humanos in vitro. El método reproducible permite la investigación de las características y la cinética de la liberación de NET tras la estimulación con distintos inductores de NETosis y permite la evaluación de la farmacología de los antagonistas de NETosis.
Resumen
Los neutrófilos desempeñan un papel importante en la defensa inmunitaria innata mediante el uso de varias estrategias, incluida la liberación de trampas extracelulares de neutrófilos (NET) en un proceso conocido como NETosis. Sin embargo, en las últimas dos décadas, ha quedado claro que la acumulación de TNE en los tejidos contribuye a la fisiopatología de múltiples enfermedades inflamatorias y autoinmunes. Por lo tanto, el interés en el desarrollo de antagonistas de NETosis ha aumentado. Se desarrollaron métodos variables y no estandarizados para detectar y analizar la NETosis de forma concomitante, cada uno con sus propias ventajas y limitaciones. Aquí, describimos un método de microscopía en tiempo real para la cuantificación de la liberación humana de NET, que permite estudiar la NETosis y la inhibición de NET de una manera de alto rendimiento. El análisis semiautomatizado basado en el área de superficie reconoce los NET y los distingue de los neutrófilos activados sin red. Demostramos que los inductores no fisiológicos de NETosis, el ionóforo de calcio y el forbol-12-miristato-13-acetato (PMA), desencadenan la liberación de NETs con diferentes características y cinéticas. Además, demostramos que este enfoque permite estudiar la liberación de NET en respuesta a estímulos relevantes para la enfermedad, incluidos los complejos inmunes, la N-formilmetionina-leucil-fenilalanina (fMLF), los cristales de urato monosódico y los cristales de pirofosfato de calcio. Para ejemplificar la utilidad de este método en el estudio de los antagonistas de NETosis, utilizamos CIT-013, un inhibidor de la liberación de NET de anticuerpos monoclonales de primera clase. CIT-013 se dirige a las histonas citrulinadas H2A y H4 e inhibe eficazmente la liberación de NET con un IC50 de 4,6 nM. Otros anticuerpos antihistonas probados carecían de esta capacidad inhibidora de NETosis. En conjunto, demostramos que este protocolo permite la cuantificación específica, fiable y reproducible de alto rendimiento de los NETs, mejorando el estudio de las características de liberación de NET, la cinética y la farmacología de los antagonistas de NETosis.
Introducción
Los neutrófilos están abundantemente presentes en la sangre y migran a los tejidos tras una infección o inflamación. Desempeñan un papel importante en la defensa inmunitaria innata mediante el uso de un amplio armamento para proteger al huésped contra los microbios. Los neutrófilos matan a los patógenos a través de la fagocitosis, la degranulación, la generación de especies reactivas de oxígeno (ROS) y la liberación de cromatina descondensada denominadas trampas extracelulares de neutrófilos (NET) mediante un proceso llamado NETosis1. Los NET son estructuras extracelulares de cromatina decoradas con, entre otras, proteínas granulares y calprotectina 2,3, y se liberan tras la estimulación con un amplio espectro de moléculas4. A grandes rasgos, la NETosis puede clasificarse en dos vías principales: dependiente de la NADPH oxidasa o independiente 5,6,7. Además, la citrulinación de arginina de las colas de histonas N-terminales por el peptidil arginina deiminasa 4 (PAD4) se ha relacionado estrechamente con la NETosis y promueve la descondensación de la cromatina que en última instancia conduce a la expulsión de la cromatina descondensada al entorno extracelular.
Aunque la liberación de NET está involucrada en la eliminación de patógenos, numerosos estudios han demostrado que la liberación anormal y prolongada de NET está relacionada con el desarrollo de diversos trastornos inflamatorios,incluyendo lesión pulmonar aguda 8, artritis reumatoide (AR)9, vasculitis10 e hidradenitis supurativa11. El papel perjudicial de los TNE en la enfermedad es multifacético, ya que los TNE son proinflamatorios, son fuente de autoantígenos, son citotóxicos para los tejidos circundantes, desencadenan inmunotrombosis y promueven la diferenciación de los osteoclastos y la erosión ósea 9,12,13. La inhibición farmacológica de la vía NETosis por inhibidores de PAD4 de moléculas pequeñas demuestra que las terapias dirigidas a NETosis tienen potencial como tratamientos para enfermedades en las que la acumulación de NET es un impulsor importante de la patogénesis14. En lugar de dirigirnos a la enzima PAD4, utilizamos un anticuerpo monoclonal anti-histonas humanizado anti-citrulinado inhibidor de NETosis, CIT-013, que se une específicamente a las histonas citrulinadas H2A y H415. CIT-013 tiene un mecanismo de acción dual único al inhibir la liberación de NET y mejorar la fagocitosis NET mediada por macrófagos16. CIT-013 y las moléculas precursoras han demostrado eficacia terapéutica en múltiples modelos murinos de inflamación asociada a NET17.
Para estudiar la liberación de NET, se han desarrollado diferentes métodos a lo largo de los años, tales como, entre otros, 1) la detección de ADN utilizando un trazador de ADN impermeable a la membrana plasmática en combinación con un lector de placas inmunofluorescentes, 2) la detección basada en el ensayo de inmunoabsorción enzimática (ELISA) de ADN y ADN complejado con proteínas específicas de NET en sobrenadantes, 3) colocalización de moléculas asociadas a NET con ADN extracelular a través de inmunohistoquímica, y 4) enfoques de citometría de flujo para detectar neutrófilos en red. Todos estos métodos tienen sus propias ventajas y limitaciones. Desarrollamos un enfoque de alto rendimiento en tiempo real para la cuantificación microscópica de la liberación humana de NET, que utiliza un colorante de ADN impermeable a la membrana plasmática16,18. El método descrito permite investigar la cinética y las características de NETosis de una manera fácil, fiable y reproducible y permite evaluar la farmacología de los antagonistas de NETosis como CIT-013.
Protocolo
Todos los donantes de sangre dieron su consentimiento informado de acuerdo con la Declaración de Helsinki, y el estudio se realizó de acuerdo con las directrices éticas de Citryll para la investigación en humanos.
NOTA: Todas las actividades con sangre humana y neutrófilos aislados deben realizarse en condiciones estériles en una cabina de flujo laminar. Cuando los ajustes de freno y aceleración para la centrifugación no se mencionan en el protocolo, se pueden considerar al máximo.
1. Aislamiento de neutrófilos de la sangre
- Recolectar sangre periférica de voluntarios sanos en tubos de heparina de litio y transferir sangre a un tubo nuevo de 50 ml.
- Enjuague los tubos de heparina de litio con 1x DPBS y transfiéralos al mismo tubo de 50 ml al que se transfirió la sangre. Asegúrese de que la proporción final de sangre a DPBS sea de 1:1. Mezcle la sangre y el DPBS mediante pipeteo para obtener una solución homogénea.
- Añada 13 mL de solución de gradiente de densidad a otro tubo nuevo de 50 mL y añada lentamente (con una pipeta de 25 mL) la sangre diluida 1:1 sobre la solución de gradiente de densidad hasta un máximo de 50 mL.
- Centrífuga a 400 x g y temperatura ambiente (RT) durante 40 min con aceleración y frenado mínimos.
NOTA: Se forman capas. Las capas de arriba a abajo son las siguientes: 1. plasma; 2. células mononucleares de sangre periférica (PBMC); 3. Solución de gradiente de densidad; 4. Eritrocitos/neutrófilos. - En primer lugar, deseche el plasma con una pipeta de 10 ml y, a continuación, deseche las PBMC y la capa de solución de gradiente de densidad con una pipeta Pasteur de plástico tanto como sea posible sin alterar la capa de eritrocitos/neutrófilos.
- Vuelva a suspender suavemente la capa de eritrocitos/neutrófilos agitando y vuelva a suspender con una pipeta de 25 ml en 15 ml de 1x DPBS.
- Añadir 25 mL de solución de Dextrano al 6%/NaCl al 0,9%, mezclar diez veces invirtiendo el tubo y colocar el tubo en posición vertical durante 25 min a RT.
NOTA: Se forman capas. Las capas de arriba a abajo son las siguientes: 1. Neutrófilos; 2 eritrocitos. - Transfiera la capa de neutrófilos a un tubo nuevo de 50 mL con una pipeta de 10 mL y centrifugue durante 10 min a 500 x g y RT.
NOTA: Tenga cuidado ya que la pastilla de neutrófilos todavía contiene algunos eritrocitos y no está firmemente unida al tubo. - Deseche el sobrenadante por decantación y vuelva a suspender el pellet de la celda con una pipeta de 10 mL en 10 mL de tampón de lisis de cloruro de amonio y potasio (ACK) (155 mM NH4Cl, 10 mM KHCO3 y 0,1 mM Na2EDTA; pH = 7,2) y luego agregue inmediatamente 40 mL de tampón de lisis ACK.
- Incubar en RT mientras se invierte continuamente el tubo hasta que la solución se vuelva translúcida (esto tarda de 1 a 5 minutos y puede variar para cada donante) y centrifugar durante 10 min a 350 x g y RT.
- Retire el sobrenadante y agregue lentamente y gota a gota 5 mL de medio de cultivo que contenga L-glutamina suplementado con suero fetal bovino (FBS) inactivado por calor al 10% (v/v), 50 U/mL de penicilina y 50 μg/mL de estreptomicina (denominado medio de cultivo al 10% en adelante) sobre la pastilla de neutrófilos sin poner los neutrófilos en suspensión. Esto eliminará eficientemente la mayoría de los eritrocitos de la pelleta de neutrófilos.
- Agitar suavemente el tubo de 50 ml hasta que la mayoría de los eritrocitos, presentes en la parte superior de la gránula de neutrófilos, se resuspendan en el medio de cultivo al 10%. Posteriormente, retire el sobrenadante decantando el tubo.
- Vuelva a suspender el pellet de neutrófilos en 10 mL de medio de cultivo al 10% y, cuando esté completamente resuspendido, agregue hasta 50 mL de medio de cultivo al 10%. Centrífuga durante 10 min a 350 x g y RT.
- Retirar el sobrenadante y resuspender el pellet celular en 10 mL de medio de cultivo al 10%.
2. Tinción de neutrófilos para comprobar la pureza mediante citometría de flujo
- Para determinar la concentración celular, diluya la suspensión de neutrófilos en una solución de azul de tripano al 0,4% (proporción 1:1) y cuente los neutrófilos con un contador de células de campo claro.
NOTA: También se pueden utilizar otros métodos de conteo. - Transfiera 1 x 105 neutrófilos en medio de cultivo al 10% a los pocillos de una placa de 96 pocillos con fondo en V y centrifugar durante 3 min a 400 x g y RT.
- Deseche el sobrenadante y vuelva a suspender las células en 50 μL de DPBS 1x suplementado con albúmina sérica bovina (BSA) al 1% (p/v) y NaN3 al 0,1% (v/v) (en adelante, tampón de clasificación celular activada por fluorescencia (FACS)) que contenga bloqueo del receptor Fc (diluido 50x).
- Incubar durante 15 min en RT y añadir 50 μL de tampón FACS que contiene 50 veces el anticuerpo anti-CD45 humano conjugado con APC-Cy7 diluido, el anticuerpo anti-CD16 humano PerCP-Cy5.5 conjugado con PerCP-Cy5.5 diluido, el anticuerpo anti-CD66b humano de ratón conjugado con FITC diluido 133 veces y el colorante de viabilidad fijable eFluor 506 diluido 500x.
NOTA: CD45 se expresa en los leucocitos. CD66b se expresa exclusivamente en granulocitos. El CD16 se expresa en gran medida en los neutrófilos, se expresa en los eosinófilos y no se expresa en los basófilos. - Incubar durante 30 min en RT en la oscuridad y centrifugar durante 3 min a 400 x g y RT.
- Deseche el sobrenadante, vuelva a suspender los neutrófilos en 175 μL de tampón FACS y centrifugue durante 3 min a 400 x g y RT. Repita este paso una vez.
- Deseche el sobrenadante y vuelva a suspender la pellet de neutrófilos en 175 μL de tampón FACS. Analice muestras utilizando un sistema de citometría de flujo y software asociado.
3. Análisis de pureza de neutrófilos mediante software de análisis de citometría de flujo
NOTA: El análisis de los datos de citometría de flujo se realizó utilizando el software de análisis de citometría de flujo como se indica en la Tabla de Materiales.
- Realice la compuerta de acuerdo con el siguiente procedimiento paso a paso.
- Establezca una puerta de tiempo en el gráfico FSC-A vs. Tiempo para seleccionar un área con el flujo de celda adecuado.
- Establezca una puerta de celda en el trazado SSC-A frente a FSC-A para seleccionar celdas y excluir residuos.
- Establezca una puerta de celda viable en la parcela FSC-A vs. AmCyan-A para seleccionar celdas viables.
- Establezca una puerta de leucocitos en la gráfica FSC-A vs. APC-Cy7 para seleccionar células CD45+.
- Establezca una puerta de neutrófilos en la gráfica FITC vs. PerCP-Cy5.5 para distinguir entre CD66b+CD16+ (neutrófilos), CD66b-CD16+ (eosinófilos) y CD66b-CD16- (basófilos, monocitos y linfocitos).
- Establezca una puerta de una sola célula en el gráfico FSC-H vs. FSC-A para seleccionar neutrófilos individuales.
- Determine la pureza de los neutrófilos presentando neutrófilos individuales como la frecuencia de las células (en la puerta de la célula).
NOTA: Para continuar, la pureza de los neutrófilos debe superar el 85%.
4. Microscopía de imagen en vivo
NOTA: Este ensayo está optimizado para múltiples placas de imágenes de 96 pocillos y varios estímulos y antagonistas de NETosis. El protocolo a continuación describe una vista general del enfoque, que se puede especificar utilizando las tablas incluidas.
- Prepare una solución de poli-L-lisina al 0,001% diluyendo poli-L-lisina al 0,01% en H2O estéril con una proporción de 1:10.
- Añadir una solución de poli-L-lisina al 0,001% a cada pocillo e incubar al menos 1 h a 37 °C.
NOTA: El volumen por pocillo depende de la placa de imagen de 96 pocillos (Tabla 1). - Lave los pocillos 3 veces con 200 μL de DPBS para eliminar el exceso de poli-L-lisina. Realice todos los pasos en la cabina de flujo laminar. Retire la tapa de la placa y seque al aire los pocillos abriendo la placa en la cabina de flujo laminar hasta que se seque (aproximadamente 1 h).
- Calcule el número de neutrófilos necesarios para el experimento y transfiera un excedente a un tubo de 15 mL. Centrífuga durante 10 min a 350 x g y RT.
NOTA: La cantidad de neutrófilos en cada pocillo varía según el tipo de placa de imagen de 96 pocillos (Tabla 1). La densidad de células está optimizada para permitir una buena separación de las células individuales, lo que es necesario para un análisis adecuado. Si la densidad de neutrófilos es demasiado alta, las células y los NET se superpondrán con las células y los NET adyacentes, lo que afecta a la calidad del análisis. - Deseche el sobrenadante y vuelva a suspender los neutrófilos en un medio de cultivo sin rojo de fenol suplementado con FBS al 2% (v/v), 50 U/mL de penicilina y 50 μg/mL de estreptomicina, 10 mM de HEPES y 1 mM de CaCl2 (denominado en adelante tampón de ensayo NET).
- Prepare las siguientes soluciones de trabajo.
- Prepare un tinte de ADN de 80 nM en tampón de ensayo NET.
- Prepare los estímulos NETosis en el tampón de ensayo NET.
- Preparación de antagonistas de NETosis en tampón de ensayo NET.
NOTA: Tenga en cuenta que las concentraciones de las soluciones de trabajo descritas anteriormente son 4 veces la concentración final necesaria en el pozo. Las concentraciones recomendadas de estímulos y antagonistas de NETosis varían (Tabla 2 y Tabla 3).
- Agregue 4 veces el tinte de ADN concentrado en el tampón de ensayo NET a cada pocillo.
- Agregue 4 veces los estímulos concentrados de NETosis en el tampón de ensayo NET a los pocillos correspondientes, y agregue el tampón de ensayo NET solo a los pocillos sin estímulo o a los pocillos que contengan inmunocomplejos recubiertos (cIC; protocolo en la tabla 2).
- Agregue 4 veces antagonistas concentrados de NETosis en el tampón de ensayo NET a los pocillos correspondientes. Agregue tampón de ensayo NET solo a los pocillos sin un antagonista.
- Agregue suspensión de neutrófilos a cada pocillo.
NOTA: El volumen de 4 veces el colorante de ADN concentrado, 4 veces los estímulos concentrados de NETosis en el tampón de ensayo NET, 4 veces el antagonista concentrado de NETosis en el tampón de ensayo NET, el número de neutrófilos y el volumen de suspensión de neutrófilos por pocillo dependen de la placa de imágenes de 96 pocillos (Tabla 1). Evite la formación de burbujas al pipetear. - Centrifugar la placa durante 2 min a 100 x g y RT e insertar la placa de imagen de 96 pocillos en el sistema de análisis de microscopía de células vivas colocado en una incubadora a 37 °C y 5% de CO2.
NOTA: La condensación puede desarrollarse en la parte inferior de la placa de imágenes de 96 pocillos durante el primer minuto de incubación. Esto debe retirarse con un pañuelo de papel.
5. Configuración del software del sistema de análisis de microscopía de células vivas para la adquisición
NOTA: Las imágenes de contraste de fase e inmunofluorescencia se adquirieron mediante un sistema de análisis de microscopía de células vivas controlado por su software de análisis.
- Abra el software del sistema de análisis de microscopía de células vivas y haga clic en Conectar al dispositivo. Ingrese el nombre de usuario y la contraseña. Haga clic en Programar para adquirir y haga clic en el botón más (Lanzar, agregar embarcación). A continuación, seleccione Escanear según programación y haga clic en Siguiente.
- Para crear una nueva embarcación desde cero, seleccione Nuevo en la sección Crear embarcación y haga clic en Siguiente. Seleccione Estándar en la sección Tipo de escaneo y haga clic en Siguiente.
- Seleccione la siguiente configuración de escaneo y haga clic en Siguiente: Celda por celda: Ninguna; Canales de imagen: Contraste de fase y verde (tiempo de adquisición 100 ms); Objetivo: 20x.
- Seleccione el tipo de embarcación adecuado para escanear en la sección Selección de embarcación y haga clic en Siguiente.
- Si utiliza una placa de 96 pocillos #1, seleccione Corning, placa, 96, N/A, 3603, 96 pocillos Corning (Blk/Wht), Microplacas.
- Si utiliza una placa de 96 pocillos #2, seleccione Nunc, Placa, 96, N/A, 152028, Nunc de 96 pocillos opt bottom (Blk/Wht), Microplacas.
- Especifique la ubicación del recipiente en el cajón y haga clic en Siguiente. En la sección Patrón de exploración , seleccione los pocillos que se deben escanear, seleccione el número de imágenes por pocillo (escanee 4 imágenes por pocillo para obtener una descripción general representativa) y haga clic en Siguiente.
NOTA: No seleccione pozos vacíos porque afectará el enfoque automático. - Proporcione información sobre la embarcación insertando, entre otros, el nombre de la placa en la sección del cuaderno de la embarcación y haga clic en Siguiente. Seleccione Aplazar análisis hasta más adelante en la sección Configuración de análisis y haga clic en Siguiente.
- Defina el programa de escaneo para el buque en la sección Programa de escaneo . Seleccione Crear nueva programación con exámenes a intervalos de y seleccione 1 hora. Seleccione Detener análisis y 00:05 horas después del primer análisis. Haga clic en Siguiente y haga clic en Agregar a la programación cuando la información de escaneo sea correcta.
- Cuando sea necesario editar el programa de escaneo, haga doble clic en el Programa de escaneo en la parte superior de la pantalla. Haga clic con el botón derecho en el Programa de escaneo y edite y ajuste como desee. Haga clic en el icono del disquete para guardar el programa de escaneo.
NOTA: Al obtener imágenes de varios recipientes (placas) en el mismo experimento, se pueden agregar recipientes adicionales al programa de escaneo existente haciendo clic en el botón más (Lanzar agregar recipiente), seleccionando Escanear en programa y haciendo clic en Siguiente. Cree un nuevo recipiente desde cero (consulte el paso 5.2 de este protocolo), copie un recipiente existente o utilice un recipiente escaneado anteriormente. Esto da como resultado un escaneo secuencial a la misma frecuencia y configuración que la placa anterior.
- Cuando sea necesario editar el programa de escaneo, haga doble clic en el Programa de escaneo en la parte superior de la pantalla. Haga clic con el botón derecho en el Programa de escaneo y edite y ajuste como desee. Haga clic en el icono del disquete para guardar el programa de escaneo.
6. Microscopía de imagen en vivo: análisis de ensayo NET utilizando el software de análisis de microscopía de células vivas
NOTA: Se utilizó un software de análisis de microscopía de células vivas para analizar el contraste de fase y las imágenes de inmunofluorescencia (Tabla de materiales). Cuando no se utiliza este sistema, se pueden realizar análisis NET similares utilizando un paquete de software de dominio público 4,18,19,20.
- Abra el software de análisis de microscopía de células vivas y haga clic en Conectar al dispositivo. Ingrese el nombre de usuario y la contraseña y haga clic en Ver escaneos recientes. Abra el experimento que desee analizar haciendo doble clic en el nombre del recipiente del experimento.
- Haga clic en Iniciar análisis y seleccione Crear nueva definición de análisis. Haga clic en Siguiente.
- Seleccione Basic Analyzer en la sección de tipo de análisis y haga clic en Siguiente. En la sección Canal de imagen , seleccione Verde, anule la selección de Fase y haga clic en Siguiente.
- Abra la ventana Capas de imagen y seleccione Verde y anule la selección de Fase. Desactive el escalado automático en la sección verde y establezca manualmente el mínimo y el máximo del canal verde para distinguir las redes del fondo.
NOTA: El contraste de fase se puede activar y desactivar cuando sea necesario. Por ejemplo, para determinar si los TNE se expulsan en el entorno extracelular. - Abra la ventana Tiempos de exploración de embarcaciones y seleccione la hora de exploración de embarcaciones adecuada en la que deben estar presentes las NET. Seleccione un conjunto de imágenes que representen la variabilidad dentro del experimento (imágenes de pocillos de control positivo con NET e imágenes de pocillos de control negativo sin NET). Estas imágenes se utilizarán para obtener una vista previa y refinar el análisis. Haga clic en Siguiente.
- Abra la ventana Configuración de definición de análisis y comience a ajustar la configuración de análisis para el canal verde de tal manera que las estructuras NET se delineen sin crear NET falsos positivos o falsos negativos. Utilice la siguiente configuración y haga clic en Vista previa actual o Vista previa de todo para aplicar la configuración a la imagen actual o a todas las imágenes, respectivamente.
- En Nombre de objeto, seleccione NET.
- Para la segmentación, seleccione Sin sustracción de fondo - Adaptable.
- Para threshold GCU, seleccione entre 3 y 5 (en función del experimento).
- Para la división de bordes, seleccione ON (entre -10 y 0).
- Para la limpieza, seleccione Relleno de agujero: 100 μm2 y Ajustar tamaño: -1 píxeles.
- Para los filtros, seleccione en función del experimento, pero los valores de corte recomendados son: Área: >200 μm2, Intensidad media: <24,6, Intensidad integrada: >7000.
NOTA: Las estructuras que se seleccionan en función de la configuración utilizada se delinean con magenta. Al pasar el ratón por encima de estas estructuras, se obtendrá más información sobre los parámetros de los objetos (es decir, área, excentricidad, intensidad integrada e intensidad media), lo que ayuda a optimizar el análisis relacionado con los criterios de inclusión o exclusión de los parámetros NET. Compruebe varias imágenes (de pozos de control positivos y negativos) para comprobar si la configuración del análisis es correcta.
- Cuando la configuración del análisis NET sea correcta, haga clic en Siguiente. Seleccione los puntos de tiempo y los pozos que desea analizar en la sección Horas y pozos de exploración y haga clic en Siguiente. Inserte el nombre de la definición en la sección Guardar y aplicar definición de análisis y haga clic en Siguiente. Haga clic en Finalizar cuando la información del análisis esté verificada y sea correcta.
NOTA: Al analizar varias placas o utilizar análisis de experimentos anteriores, inicie el análisis y seleccione Copiar definición de análisis existente o Usar definición de análisis existente y haga clic en Siguiente. Seleccione la definición de análisis existente adecuada y haga clic en Siguiente. Para el procedimiento de análisis posterior, vaya al paso 6.4 si se ha elegido copiar definición de análisis existente , o al paso 6.7 cuando se ha elegido usar definición de análisis existente . - Abra el análisis del experimento haciendo clic en el análisis dentro del recipiente de interés. Abra la ventana Métricas de gráfico .
- Haga clic en el botón más (Crear métrica). Los datos se pueden presentar como porcentaje de confluencia NET (el porcentaje del área de la imagen marcada como NET) o porcentaje de neutrófilos netos (número de NET dividido por el número de celdas).
- Al presentar los datos como un porcentaje de confluencia neta, seleccione Área en la sección Métrica y seleccione Confluencia en la sección Valor . Al presentar datos como neutrofilos netos porcentuales, seleccione Recuento de objetos en la sección Métrica y seleccione Por imagen en la sección Valor .
- Haga clic en Aceptar. Seleccione Confluencia de área de NETs (%) o Recuento de objetos NETs por imagen (por imagen) en la sección Métricas definidas por el usuario . Seleccione todos los puntos de tiempo que deben analizarse en la sección Seleccionar escaneos . Seleccione todos los pozos que deben analizarse en la sección Seleccionar pozos y haga clic en Exportar datos.
- Seleccione los siguientes ajustes en la sección Exportación de gráficos .
- Seleccione Mostrar cada escaneo como su propia tabla (columnas: 1, 2... filas: A, B...).
- Seleccione Mostrar etiquetas de fila y columna.
- Seleccione Todos los análisis de un archivo y haga clic en Examinar para ajustar la ubicación y el nombre del archivo.
- Seleccione Incluir detalles del experimento en el encabezado.
- Seleccione Desglosar datos en imágenes individuales.
- Haga clic en Exportar para exportar los datos como archivo ".txt", que se puede importar y analizar más a fondo en una hoja de cálculo.
NOTA: Para determinar el porcentaje de neutrófilos en red, es necesario determinar el número de neutrófilos en t = 0. Para ello, continúe con los siguientes pasos del protocolo. - Vaya al paso 6.1 para iniciar el análisis adicional necesario para contar el número de neutrófilos para las imágenes en t = 0.
- En el paso 6.3, seleccione Fase y anule la selección de Verde en la sección del canal de imagen.
- En el paso 6.4, seleccione t = 0 en la sección de tiempos de exploración de vasos.
- En el paso 6.5, anule la selección de Verde, seleccione Fase y ajuste la Configuración del canal de fase cuando sea necesario.
- En el paso 6.6, agregue Nombre de objeto, seleccione Confluencia clásica en la sección Segmentación y establezca el ajuste de segmentación en 0 (Fondo), ajuste el Relleno de agujero a 100 μm2 y ajuste el Área a 50 μm2.
- En el paso 6.7, seleccione t = 0 en la sección seleccionar escaneos.
- En el paso 6.8, abra Análisis de recuento de neutrófilos.
- En el paso 6.9, seleccione Recuento de objetos por imagen (por imagen) en la sección Métricas definidas por el usuario .
- Por último, utilice el siguiente cálculo para generar el porcentaje de neutrófilos en red: (número de NETs / número de neutrófilos en t = 0) x 100%.
Resultados
La microscopía de alto rendimiento en tiempo real, en combinación con un tinte de ADN impermeable, permite estudiar la cinética, las características y las vías subyacentes de NETosis y permite la evaluación de posibles inhibidores de la liberación de NET. Con este enfoque, los NET se definieron como estructuras positivas para el colorante de ADN con un área superficial significativamente mayor en comparación con el área superficial de los neutrófilos sanos (Figura 1A), lo que indica que la cromatina ha sido expulsada al ambiente extracelular18. El análisis basado en el área de superficie nos permitió distinguir entre NET y neutrófilos activados con integridad de la membrana plasmática comprometida, mostrando una tinción brillante de ADN intracelular (Figura 1A).
El ionóforo de calcio (A23187) y el PMA se utilizan habitualmente para inducir NETosis in vitro. A pesar de ser estímulos no fisiológicos, son valiosos porque activan distintas vías de NETosis y aseguran una inducción consistente de NETosis con baja variabilidad entre donantes. A23187 desencadena la entrada de calcio, lo que conduce a la activación de PAD4 y la liberación de NETs ricos en histonas citrulinadas, mientras que PMA activa el complejo NADPH oxidasa, lo que resulta en la producción de especies reactivas de oxígeno (ROS) y la posterior liberación de NETs con bajos niveles de histonas citrulinadas 5,16,21. La velocidad y la magnitud de la respuesta de NETosis dependen de la concentración de cada estímulo (Figura 1B,C), con A23187 induciendo NETosis más rápida y PMA dando como resultado una mayor proporción de neutrófilos liberando NET (Figura 1D-F). Los NET resultantes de la estimulación de A23187 (Figura 1D; flecha roja) se distinguieron de los NET inducidos por PMA (Figura 1D; flecha amarilla) por ser más difusos más allá de la membrana plasmática de los neutrófilos, mientras que los NET inducidos por PMA permanecieron más adyacentes a la membrana plasmática de los neutrófilos. Además, la estimulación de A23187 dio lugar a neutrófilos permeables y no reticulantes con colorante de ADN (Figura 1D; flecha azul), que no expulsaron su ADN al espacio extracelular. La detección de neutrófilos permeables no neting con colorante de ADN positivo dependió de la concentración de A23187 (Figura 1G) y estuvo casi ausente independientemente de la concentración de PMA utilizada para estimular la NETosis (Figura 1D, H). Además de utilizar los estímulos no fisiológicos de NETosis A23187 y PMA, este ensayo también es adecuado para estudiar los desencadenantes de NETosis relevantes para la enfermedad. Por ejemplo, los neutrófilos activados con inmunocomplejos solubles (sIC) o cristales presentes en la enfermedad por disposición de pirofosfato de calcio (CPPD) mostraron un aumento significativo de la liberación de NET en comparación con ningún estímulo. Se observó una tendencia hacia niveles elevados de NET cuando los neutrófilos se activaron con complejos inmunes recubiertos (cIC), fMLP y cristales de urato monosódico (MSU) (Figura 1I); Sin embargo, para estos estímulos, observamos una variación considerable de un donante a otro.
La inhibición farmacológica de la vía NETosis y de las enzimas asociadas a NETosis ha demostrado que las terapias dirigidas a NETosis podrían ser tratamientos eficaces para enfermedades en las que la acumulación de NET impulsa significativamente la patología 14,17,22,23,24. Este ensayo NET de microscopía en tiempo real es un enfoque fácil, fiable y reproducible para estudiar los antagonistas de NETosis de forma eficaz. Para ejemplificar esto, hemos utilizado un anticuerpo monoclonal humanizado de primera clase, CIT-013, que se dirige a las histonas citrulinadas H2A y H4 con alta afinidad15,16. A23187 activa la vía NETosis, lo que da lugar a NETs que contienen histonas citrulinadas que posteriormente son objetivo de CIT-013 para realizar su función inhibidora de NETosis16. De hecho, la liberación de NET en respuesta a A23187 fue completamente inhibida por CIT-013 (Figura 2A y Video Suplementario 1), con un IC50 de 4,6 nM (Figura 2B). La capacidad inhibidora de NETosis de CIT-013 es única, ya que otros anticuerpos comerciales dirigidos a diferentes histonas (no) citrulinadas no fueron capaces de inhibir la liberación de NET (Figura 2C).
Anteriormente, hemos demostrado que una molécula precursora muy similar de CIT-013 (que difiere dos aminoácidos pero se une a epítopos similares con igual afinidad) bloquea la NETosis en respuesta a estímulos fisiológicos como las plaquetas activadas, el líquido sinovial de la gota y el líquido sinovial de la AR17. Aquí, mostramos que la liberación de NET inducida por sIC puede ser inhibida por CIT-013 (Figura 2D). La relevancia terapéutica de la inhibición de la NETosis inducida por este estímulo es resaltada por el LES, la AR y otras enfermedades autoinmunes, donde los autoanticuerpos en el suero o en el líquido sinovial apoyan la formación de CI, que desencadena la NETosis25,26.
En conjunto, estos datos demuestran que este enfoque de microscopía de alto rendimiento en tiempo real es adecuado para estudiar la cinética y las características de la liberación de NET y permite el estudio de los inhibidores de NETosis. Aunque este método ha sido optimizado para el uso de neutrófilos humanos, con modificaciones también podría ser adecuado para estudiar neutrófilos de otras especies. Los datos generados con este ensayo son una piedra angular de la justificación de CIT-013 como una terapia potente y eficaz para las enfermedades impulsadas por NET.
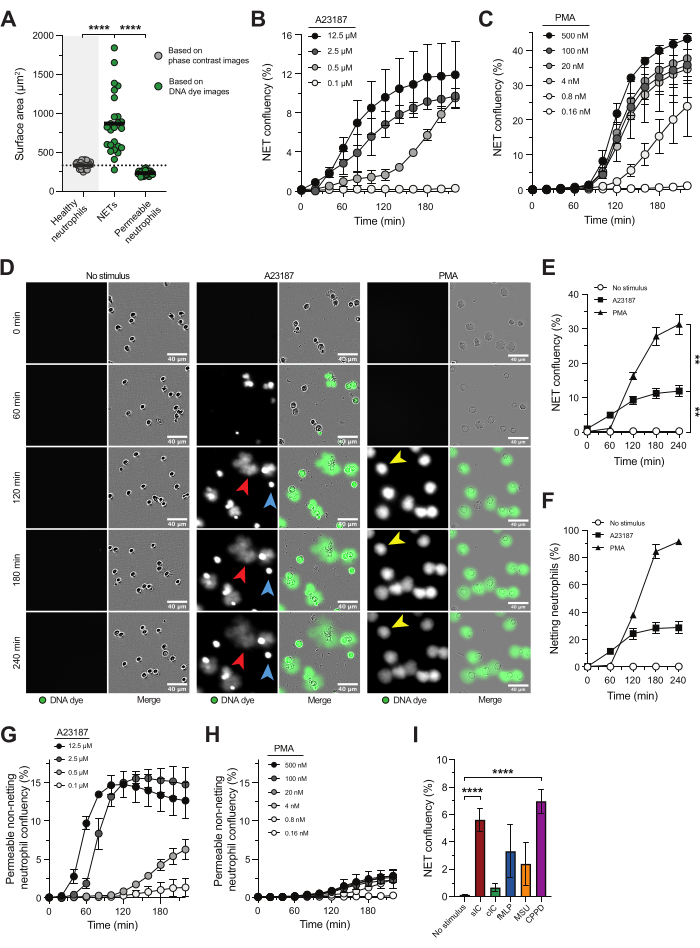
Figura 1: Microscopía de alto rendimiento en tiempo real para el estudio de la liberación de NET. Los neutrófilos aislados de la sangre de voluntarios sanos se estimularon con A23187 o PMA para desencadenar la vía NETosis. La liberación de NET se visualizó con microscopía de alto rendimiento en tiempo real utilizando un colorante de ADN impermeable a la membrana plasmática y se cuantificó en función del área de superficie con el software del sistema de análisis de microscopía de células vivas. (A) Análisis de la superficie de neutrófilos sanos no estimulados (gris), NET extracelulares y neutrófilos permeables no rederos con tinción de ADN intracelular (verde). (B,C) Cuantificación de la liberación de NET a lo largo del tiempo de neutrófilos estimulados con las concentraciones indicadas de A23187 o PMA (n = 2). (D) Imágenes representativas de la liberación de NET en respuesta a A23187 (flechas rojas) y PMA (flechas amarillas) en diferentes puntos de tiempo. Los ejemplos de neutrófilos permeables que no forman redes se indican con flechas azules. (E) Cuantificación de la liberación neta a lo largo del tiempo presentada como un porcentaje de la confluencia neta (n = 5). Las estadísticas se realizaron en t = 240 min. (F) Cuantificación de la liberación neta a lo largo del tiempo presentada como un porcentaje de neutrófilos en red (n = 2). (G, H) Cuantificación de neutrófilos permeables no rederos a lo largo del tiempo a partir de neutrófilos que han sido estimulados con las concentraciones indicadas de A23187 o PMA (n = 2). (I) Cuantificación de la liberación neta a t = 240 min inducida por inmunocomplejos solubles (sIC), IC recubiertos (cIC), fMLP, cristales de urato monosódico (MSU) y cristales presentes en la enfermedad por disposición al pirofosfato de calcio (CPPD) (n = 8-28). Los resultados se presentan como media ± error estándar de la media. **P < 0.01 y ****P < 0.0001, ANOVA de un factor de medidas repetidas con la prueba de comparaciones múltiples de Dunn (B), prueba de Kruskal-Wallis con la prueba de comparaciones múltiples de Dunn (I). Los paneles A-F han sido modificados con permiso de van der Linden et al.16. Haga clic aquí para ver una versión más grande de esta figura.
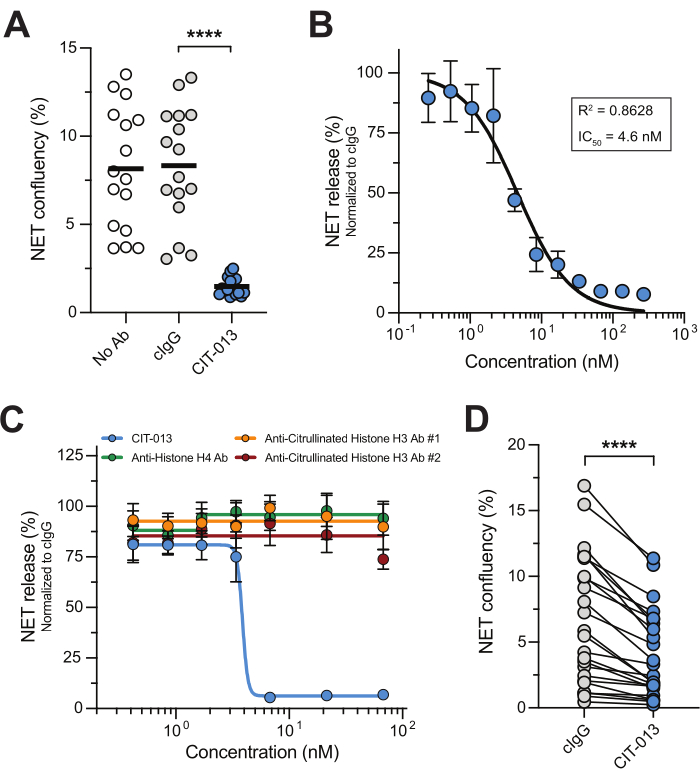
Figura 2: CIT-013 inhibe la liberación de NET. (A) Cuantificación de la liberación NET inducida por A23187 a t = 240 min en ausencia (No Ab) o presencia de CIT-013 o anticuerpo de control de isotipo (cIgG). (B) Inhibición dependiente de la dosis de la liberación de NET inducida por A23187 con CIT-013 a t = 240 min (n = 3). Los datos se normalizaron a cIgG (establecido como 100% de liberación neta). (C) Cuantificación de la liberación neta inducida por A23187 a t = 240 min en presencia de concentraciones indicadas de CIT-013, anticuerpo antihistona H4, anticuerpo antihistona H3 anticitrulinado #1 o anticuerpo antihistona H3 citrulinado #2 (n = 6). (D) Cuantificación de la liberación neta a t = 240 min inducida por inmunocomplejos solubles (ICs) en presencia de CIT-013 o cIgG. Los resultados se presentan como media ± error estándar de la media. P < 0.0001, ANOVA de un factor de medidas repetidas con comparaciones múltiples de Tukey (A) o prueba de rango con signo de pares emparejados de Wilcoxon de dos colas (D). Los paneles A, B y D han sido modificados con permiso de van der Linden et al.16. Haga clic aquí para ver una versión más grande de esta figura.
| Placa de 96 pocillos #1 | Placa de 96 pocillos #2 | |
| Volumen 0,001% de solución de poli-L-lisina por pocillo | 50 μL | 100 μL |
| Volumen de suspensión de neutrófilos por pocillo | 50 μL | 87,5 μL |
| Número de neutrófilos por pocillo | 2 x 104 celdas | 3.5 x 104 celdas |
| Volumen 4x concentrado (= 80 nM) de colorante de ADN en tampón de ensayo NET por pocillo | 50 μL | 87,5 μL |
| Volumen 4 veces concentrado de estímulos NETosis en tampón de ensayo NET por pocillo | 50 μL | 87,5 μL |
| Volumen 4 veces concentrado de antagonistas de NETosis en tampón de ensayo NET por pocillo | 50 μL | 87,5 μL |
Tabla 1: Volúmenes y números de celdas optimizados para diferentes placas de imágenes de 96 pocillos.
| Solución de trabajo de concentración (4x concentrado) | Concentración final | |
| Ionóforo de calcio (A23187) | 50 μM | 12,5 μM |
| PMA | 16 nM | 4 nM |
| fMLP | 4 μM | 1 μM |
| Cristales de urato monosódico (MSU) | 400 μg/mL | 100 μg/mL |
| Cristales de enfermedad por disposición de pirofosfato de calcio (CPPD) | 400 μg/mL | 100 μg/mL |
| Complejos inmunes solubles (sIC) | 1. Añadir 5 μg/mL de albúmina sérica humana (HSA) en DPBS a 282,5 μg/mL de anticuerpo policlonal anti-HSA de conejo en DPBS. | |
| 2. Incubar durante al menos 90 min a 37 °C. | ||
| 3. Homogeneizar por vórtice y agregar 50 μL de solución de sIC a los pozos correspondientes. | ||
| Complejos inmunitarios recubiertos (cIC) | 1. Agregue 10 μg/mL de HSA en DPBS en los pocillos correspondientes de la placa de 96 pocillos. | |
| 2. Incubar durante la noche a 4 °C. | ||
| 3. Lave los pocillos 3 veces con 200 μL 0.05% Tween-20 en DPBS (denominado PBS/0.05%Tween en adelante). | ||
| 4. Bloquear los pocillos con 200 μL 1% (p/v) de albúmina sérica bovina en PBS/0,05% Tween (en adelante, tampón de bloqueo). | ||
| 5. Incubar durante 120 min a temperatura ambiente y agitar suavemente (400 rpm). | ||
| 6. Lavar los pocillos 3 veces con 200 μL PBS/0,05% Tween. | ||
| 7. Añadir 50 μL de anticuerpo policlonal anti-HSA de conejo en tampón de bloqueo a los pocillos correspondientes. | ||
| 8. Incubar durante 60 min a temperatura ambiente y agitación suave (400 rpm). | ||
| 9. Lave los pocillos 3 veces con 200 μL PBS/0.05% Tween. | ||
| 10. Finalmente, lave los pocillos 3 veces con 200 μL DPBS. Los pozos ya están listos para el paso 4.7 del protocolo. | ||
Tabla 2: Concentraciones recomendadas para los estímulos NETosis.
| Solución de trabajo de concentración (4x concentrado) | Concentración final | |
| Anticuerpo lisozima de huevo de gallina (anticuerpo de control; cIgG) | 80 nM | 20 nM |
| CIT-013 | 80 nM | 20 nM |
Tabla 3: Concentraciones recomendadas para el antagonista de NETosis.
Video complementario 1. Los neutrófilos se estimularon con A23187 en presencia de cIgG (izquierda) o CIT-013 (derecha), y la liberación de NET se visualizó a lo largo del tiempo utilizando el colorante de ADN impermeable a la membrana plasmática. La liberación de NET se inhibe en presencia de CIT-013. La película es una superposición de tinte de ADN (verde) y contraste de fase. Este video fue obtenido con permiso de van der Linden et al.16. Haga clic aquí para descargar este video.
Discusión
Desde el descubrimiento de los NETs en 2004, se han desarrollado muchas estrategias para investigar experimentalmente la liberación de NET in vitro, siendo la microscopía de inmunofluorescencia la técnica más común para cuantificar la NETosis27,28. Aunque la microscopía es útil para visualizar la liberación de NET, tiene limitaciones, ya que la cuantificación microscópica no automatizada de imágenes de puntos de tiempo fijos es bastante inexacta y puede sufrir un sesgo del observador. Otra técnica utilizada para estudiar la liberación de NET es la citometría de flujo de imágenes multiespectrales29,30, que mide un gran número de neutrófilos en red y adopta un análisis imparcial, pero se centra en los neutrófilos en las primeras etapas de la NETosis y no cuantifica los neutrófilos que han liberado sus NET. Muchos estudios sobre la cinética de NETosis utilizan un colorante de ADN para cuantificar la liberación de NET en combinación con un lector de placas de fluorescencia31. Esta técnica es incapaz de distinguir los NET de los neutrófilos activados o moribundos con tinción de ADN intracelular y, por lo tanto, no es adecuada para la cuantificación de la liberación de NET y el estudio de los antagonistas de NETosis. Lo anterior enfatiza que los enfoques actualmente en uso para estudiar la NETosis son valiosos, pero tienen sus limitaciones.
El método de microscopía en tiempo real descrito en este estudio aborda muchos problemas encontrados en las técnicas reportadas anteriormente. Ofrece una cuantificación NET imparcial, semiautomatizada, de alto rendimiento, reproducible y precisa. De hecho, este enfoque minimiza los artefactos de pipeteo, como la morfología estirada de los NET18, y es capaz de distinguir la NETosis de la necrosis y la apoptosis a través de la morfología y la cinética distintivas de la liberación de ADN18,32. Además, este enfoque brinda la oportunidad de estudiar diferentes vías bioquímicas para la muerte celular regulada, como la formación de NET en neutrófilos necroptóticos, que generan estructuras que cumplen con los criterios funcionales y fenotípicos de los NETs33.
Hay varios pasos críticos en este protocolo que deben seguirse para una cuantificación exitosa de los NET. En primer lugar, el número correcto de celdas por pocillo es importante para una cuantificación precisa de NET. Cuando la densidad de neutrófilos es demasiado alta, las células y los NET se superpondrán con las células y los NET adyacentes, lo que dificulta su distinción y, como resultado, la cuantificación será inexacta. En segundo lugar, es un requisito previo utilizar una baja concentración de colorante impermeable de unión al ADN de membrana plasmática no tóxico para teñir los NET cuando se secretan en el entorno extracelular. Los tintes de unión al ADN permeables a la membrana plasmática pueden inducir fácilmente la activación de neutrófilos o la muerte celular. En tercer lugar, se deben escanear múltiples imágenes por pocillo para obtener una visión general representativa de la liberación de NET entre la población heterogénea de neutrófilos. En cuarto lugar, la imagen de contraste de fase de los neutrófilos a t = 0 es necesaria para corregir el número de neutrófilos por imagen y calcular el porcentaje de neutrófilos en red.
Aunque este enfoque de microscopía de alto rendimiento en tiempo real tiene muchas ventajas sobre otros ensayos de detección de NET, este método tiene limitaciones ya que, hasta donde sabemos, no hay colorantes fluorescentes disponibles para detectar componentes adicionales de NET para confirmar la liberación de NET. Se pueden utilizar anticuerpos marcados con fluorescencia para detectar componentes adicionales de NET, pero podrían tener efectos no deseados, ya que los complejos anticuerpo-inmunitario influyen en la activación de los neutrófilos y, posiblemente, en la NETosis. Por lo tanto, preferimos no utilizar anticuerpos adicionales en esta configuración de ensayo y recomendamos utilizar en su lugar estímulos inductores de NETosis conocidos. Cuando se utilizan estímulos NETosis aún no establecidos, recomendamos el uso de un ELISA para detectar ADN complejado con proteínas específicas de NET, como histonas citrulinadas, antes del ensayo de imagen en vivo. En segundo lugar, la variación intra e interensayo puede ser el resultado de la variabilidad de un donante a otro, ya que los neutrófilos de donantes sanos responden de manera diferente a varios estímulos de NETosis debido a la heterogeneidad dentro de la población sana. Para minimizar la variación del ensayo, los neutrófilos deben aislarse en 1 h de la sangre recién extraída y utilizarse inmediatamente en el experimento, ya que los neutrófilos tienen una vida corta y no se pueden criopreservar. Además, es importante que se valide y adopte un protocolo de aislamiento de neutrófilos para minimizar la activación de los neutrófilos. Los neutrófilos son células sensibles y pueden cambiar su capacidad de respuesta a los estímulos durante el proceso de purificación debido al estrés mecánico y otros tipos de estrés. El estado de activación de los neutrófilos y la liberación de NET pueden verse influenciados por el tipo de aguja, los tubos de extracción de sangre utilizados, la temperatura de incubación, la velocidad de centrifugación y el tiempo desde la recolección de sangre hasta el aislamiento de los neutrófilos 34,35,36,37. Krémer et al.36 describen un método adicional de aislamiento de neutrófilos que puede ser considerado, utilizando un método de selección inmunomagnética negativo sin lisis de glóbulos rojos. Este método se asemeja a los neutrófilos intactos en la sangre total y podría ser adecuado para prevenir cualquier activación de neutrófilos durante el proceso de purificación. Todo lo anterior debe servir para advertir al campo que los datos de diferentes grupos de investigación necesitan ser comparados con mucho cuidado.
En general, el método de microscopía de alto rendimiento en tiempo real descrito permite la cuantificación precisa de los NET de una manera reproducible y eficiente y se puede utilizar para estudiar las características, la magnitud y la cinética de la liberación de NET y permite la investigación de la actividad de los antagonistas de NETosis. Como ejemplo de esto último, utilizamos el anticuerpo monoclonal humanizado anti-citrulinado H2A y H4 CIT-013, que actualmente se encuentra en fase de desarrollo clínico.
Divulgaciones
Los autores son empleados de Citryll y tienen intereses financieros.
Agradecimientos
Los autores quieren agradecer a Paul Vink la gestión de parte del proyecto relacionado con el método de microscopía de imagen en vivo.
Materiales
| Name | Company | Catalog Number | Comments |
| A-23187 Free Acid (Calcimycin) | Thermo Fisher | A1493 | |
| Ammonium chloride (NH4Cl) | Sigma Aldrich | A9434 | |
| Anti-Hen egg lysozyme (control antibody; cIgG) | CrownBio | C0001 | |
| APC-Cy7-conjugated mouse anti-human CD45 antibody (Clone 2D1) | Biolegend | 368515 | Use at 1 µg/mL final concentration |
| BD FACSCantoTM II system + FACSDiva software (version 8.0.1) | BD | n/a | Flow cytometry system |
| Bovine Serum Albumin (BSA) Fraction V | Roche | 10735108001 | |
| CaCl2 (1 M) | VWR | E506 | |
| Calcium pyrophosphate disposition disease (CPPD) crystals | InvivoGen | lrl-cppd | |
| Cellometer Auto T4 Bright Field Cell Counter + analysis software (version 3.3.9.5) | Nexcelom Bioscience | n/a | Bright field cell counter |
| CIT-013 | Citryll B.V. | n/a | |
| Costar black 96-well plate, clear bottom | Corning | 3603 | 96-well plate #1 |
| Dextran T500 | Pharmacosmos | 551005009006 | |
| DPBS (1x) | Gibco | 14190-144 | |
| Fetal Bovine Serum (FBS) | Serena | S-FBS-SA-015 | |
| Ficoll | GE Healthcare | 17-1440-02 | Density gradient solution |
| FITC-conjugated mouse anti-human CD66b antibody (Clone G10F5) | eBioscience | 17-0666-42 | Use at 1.5 µg/mL final concentration |
| Fixable viability dye eFluor 506 | eBioscience | 65-0866-14 | |
| Flow cytometry analysis software | FLowJo | Version 10.8.0 | |
| fMLP | Sigma Aldrich | 47729-10MG-F | |
| HEPES (1M) | Gibco | 15630-080 | |
| Human serum albumin (HSA) | VWR | 31020 | |
| Human TruStain FcX | Biolegend | 422302 | Fc receptor block |
| IBIDI 96-well plate, clear bottom | IBIDI | 89626 | 96-well plate #2 |
| IncuCyte SX3 + analysis software (version 2022B Rev1) | Satorius | n/a | Live cell microscopic analysis system |
| Monosodium urate crystals | InvivoGen | tlrl-msu | |
| Mouse anti-histone H3 (citrulline R2 + R8 + R17) antibody | Cayman | 17939 | Clone 11D3; referred to as #1 |
| Mouse anti-histone H4 (K8Ac + K12Ac + K16Ac) antibody (Clone KM-2) | Absolute Antibodies | Ab01681-2.0 | |
| Na2EDTA | Sigma Aldrich | E5134 | |
| NaCl (0.9%) | B. Braun | 25900 | |
| Penicillin (5000 U/mL) - Streptomycin (5000 µg/mL) | Gibco | 15070-063 | |
| PerCP-Cy5.5-conjugated mouse anti-human CD16 antibody (Clone 3G8) | Biolegend | 302027 | Use at 0.33 µg/mL final concentration |
| PMA | Sigma Aldrich | P1585 | |
| Polyclonal rabbit anti-HSA antibody | Sigma Aldrich | A0433-2ml | |
| Poly-L-Lysine (0.01%) | Sigma Aldrich | P4832 | |
| Potassium bicarbonate (KHCO3) | Sigma Aldrich | 237205 | |
| Rabbit anti-histone H3 (citrulline R2 + R8 + R17) antibody | Abcam | ab281584 | Multiclonal; referred to as #2 |
| RPMI 1640 with GlutaMAXTM supplement | Gibco | 61870-010 | Culture medium containing L-glutamine |
| RPMI 1640 without phenol red | Gibco | 11835-030 | Culture medium without phenol red |
| Sodium azide (NaN3) | Sigma Aldrich | S2002 | |
| Sterile H2O | Gibco | 15230204 | |
| Sytox Green Nucleid Acid Stain | Thermo Fisher | S7020 | DNA dye |
| Trypan blue solution (0.4%) | Gibco | 15250-061 | |
| Tween-20 | Sigma Aldrich | P1379 | |
| Vacutainer blood tubes Li-Heparin (17 IU/mL) | BD | 367526 |
Referencias
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- De Bont, C., Pruijn, G. J. M. Citrulline is not a major determinant of autoantibody reactivity to neutrophil extracellular traps. Philos Trans R Soc Lond B Biol Sci. 378 (1890), 20220249 (2023).
- Urban, C. F., et al. Neutrophil extracellular traps contain calprotectin, a cytosolic protein complex involved in host defense against Candida albicans. PLoS Pathog. 5 (10), e1000639 (2009).
- Hoppenbrouwers, T., et al. In vitro induction of NETosis: Comprehensive live imaging comparison and systematic review. PLoS One. 12 (5), e0176472 (2017).
- Kenny, E. F., et al. Diverse stimuli engage different neutrophil extracellular trap pathways. ELife. 6, e24437 (2017).
- Parker, H., Dragunow, M., Hampton, M. B., Kettle, A. J., Winterbourn, C. C. Requirements for NADPH oxidase and myeloperoxidase in neutrophil extracellular trap formation differ depending on the stimulus. J Leukoc Biol. 92 (4), 841-849 (2012).
- Neeli, I., Radic, M. Opposition between PKC isoforms regulates histone deimination and neutrophil extracellular chromatin release. Front Immunol. 4, 38 (2013).
- Bosmann, M., Ward, P. A. Protein-based therapies for acute lung injury: targeting neutrophil extracellular traps. Expert Opin Ther Targets. 18 (6), 703-714 (2014).
- Papayannopoulos, V. Neutrophil extracellular traps in immunity and disease. Nat Rev Immunol. 18 (2), 134-147 (2018).
- Shiratori-Aso, S., Nakazawa, D. The involvement of NETs in ANCA-associated vasculitis. Front Immunol. 14, 1261151 (2023).
- Byrd, A. S., et al. Neutrophil extracellular traps, B cells, and type I interferons contribute to immune dysregulation in hidradenitis suppurativa. Sci Transl Med. 11 (508), eaav5908 (2019).
- Kimball, A. S., Obi, A. T., Diaz, J. A., Henke, P. K. The emerging role of NETs in venous thrombosis and immunothrombosis. Front Immunol. 7, 236 (2016).
- O'Neil, L. J., et al. Neutrophil extracellular trap-associated carbamylation and histones trigger osteoclast formation in rheumatoid arthritis. Ann Rheum Dis. 82 (5), 630-638 (2023).
- Li, M., et al. A novel peptidylarginine deiminase 4 (PAD4) inhibitor BMS-P5 blocks formation of neutrophil extracellular traps and delays progression of multiple myeloma. Mol Cancer Ther. 19 (7), 1530-1538 (2020).
- Chirivi, R. G. S., et al. Anti-citrullinated protein antibodies as novel therapeutic drugs in rheumatoid arthritis. J Clin Cell Immunol. S6, 006 (2013).
- Van der Linden, M., et al. Anti-citrullinated histone monoclonal antibody CIT-013, a dual action therapeutic for neutrophil extracellular trap-associated autoimmune diseases. MAbs. 15 (1), 2281763 (2023).
- Chirivi, R. G. S., et al. Therapeutic ACPA inhibits NET formation: a potential therapy for neutrophil-mediated inflammatory diseases. Cell Mol Immunol. 18 (6), 1528-1544 (2021).
- Van der Linden, M., Westerlaken, G. H. A., Van Der Vlist, M., Van Montfrans, J., Meyaard, L. Differential signalling and kinetics of neutrophil extracellular trap release revealed by quantitative live imaging. Sci Rep. 7 (1), 6529 (2017).
- Hoffmann, J. H. O., Schaekel, K., Gaiser, M. R., Enk, A. H., Hadaschik, E. N. Interindividual variation of NETosis in healthy donors: introduction and application of a refined method for extracellular trap quantification. Exp Dermatol. 25 (11), 895-900 (2016).
- Silva, L. M., Moutsopoulos, N., Bugge, T. H., Doyle, A. Live imaging and quantification of neutrophil extracellular trap formation. Curr Protoc. 1 (7), e157 (2021).
- De Bont, C. M., Koopman, W. J. H., Boelens, W. C., Pruijn, G. J. M. Stimulus-dependent chromatin dynamics, citrullination, calcium signalling and ROS production during NET formation. Biochim Biophys Acta Mol Cell Res. 1865 (11 Pt A), 1621-1629 (2018).
- Gajendran, C., et al. Alleviation of arthritis through prevention of neutrophil extracellular traps by an orally available inhibitor of protein arginine deiminase 4. Sci Rep. 13 (1), 3189 (2023).
- Biron, B. M., Chung, C. -. S., O'Brien, X. M., Chen, Y., Reichner, J. S., Ayala, A. Cl-Amidine prevents histone 3 citrullination and neutrophil extracellular trap formation, and improves survival in a murine sepsis model. J Innate Immun. 9 (1), 22-32 (2017).
- Perdomo, J., et al. Neutrophil activation and NETosis are the major drivers of thrombosis in heparin-induced thrombocytopenia. Nat Commun. 10 (1), 1322 (2019).
- Lood, C., et al. Neutrophil extracellular traps enriched in oxidized mitochondrial DNA are interferogenic and contribute to lupus-like disease. Nat Med. 22 (2), 146-153 (2016).
- Kraaij, T., et al. A novel method for high-throughput detection and quantification of neutrophil extracellular traps reveals ROS-independent NET release with immune complexes. Autoimmun Rev. 15 (6), 577-584 (2016).
- De Buhr, N., Von Köckritz-Blickwede, M. How neutrophil extracellular traps become visible. J Immunol Res. 2016, 1-13 (2016).
- Brinkmann, V., Laube, B., Abu Abed, U., Goosmann, C., Zychlinsky, A. Neutrophil extracellular traps: How to generate and visualize them. J Vis Exp. (36), e1724 (2010).
- Zhao, W., Fogg, D. K., Kaplan, M. J. A novel image-based quantitative method for the characterization of NETosis. J Immunol Methods. 423, 104-110 (2015).
- Gavillet, M., et al. Flow cytometric assay for direct quantification of neutrophil extracellular traps in blood samples. Am J Hematol. 90 (12), 1155-1158 (2015).
- Sil, P., Yoo, D., Floyd, M., Gingerich, A., Rada, B. High throughput measurement of extracellular DNA release and quantitative NET formation in human neutrophils in vitro. J Vis Exp. (112), e52779 (2016).
- Gupta, S., Chan, D. W., Zaal, K. J., Kaplan, M. J. A high-throughput real-time imaging technique to quantify NETosis and distinguish mechanisms of cell death in human neutrophils. J Immunol. 200 (2), 869-879 (2018).
- D'Cruz, A. A., et al. The pseudokinase MLKL activates PAD4-dependent NET formation in necroptotic neutrophils. Sci Signal. 11 (546), eaao1716 (2018).
- Nash, G., Abbitt, K., Tate, K., Jetha, K., Egginton, S. Changes in the mechanical and adhesive behaviour of human neutrophils on cooling in vitro. Pflugers Arch. 442 (5), 762-770 (2001).
- Hundhammer, T., Gruber, M., Wittmann, S. Paralytic impact of centrifugation on human neutrophils. Biomedicines. 10 (11), 2896 (2022).
- Krémer, V., Godon, O., Bruhns, P., Jönsson, F., De Chaisemartin, L. Isolation methods determine human neutrophil responses after stimulation. Front Immunol. 14, 1301183 (2023).
- Freitas, M., Porto, G., Lima, J. L. F. C., Fernandes, E. Isolation and activation of human neutrophils in vitro. The importance of the anticoagulant used during blood collection. Clin Biochem. 41 (7-8), 570-575 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados