Method Article
Echtzeit-Hochdurchsatz-Mikroskopische Quantifizierung der Freisetzung extrazellulärer Fallen bei humanen Neutrophilen und Bewertung der Pharmakologie von Antagonisten
In diesem Artikel
Zusammenfassung
Dieses definierte Protokoll beschreibt einen Echtzeit-Hochdurchsatz-Mikroskopie-Ansatz zur Visualisierung und Quantifizierung der Freisetzung von extrazellulären Traps (NET) bei humanen Neutrophilen in vitro. Die reproduzierbare Methode ermöglicht die Untersuchung der Eigenschaften und Kinetik der NET-Freisetzung nach Stimulation mit unterschiedlichen NETosis-Induktoren und ermöglicht die Beurteilung der Pharmakologie von NETosis-Antagonisten.
Zusammenfassung
Neutrophile spielen eine wichtige Rolle bei der angeborenen Immunabwehr, indem sie verschiedene Strategien anwenden, einschließlich der Freisetzung von extrazellulären Fallen (NETs) für Neutrophile in einem Prozess, der als NETose bezeichnet wird. In den letzten zwei Jahrzehnten hat sich jedoch gezeigt, dass die Akkumulation von NETs in Geweben zur Pathophysiologie mehrerer Entzündungs- und Autoimmunerkrankungen beiträgt. Daher ist das Interesse an der Entwicklung von NETosis-Antagonisten gestiegen. Gleichzeitig wurden variable und nicht-standardisierte Methoden zur Detektion und Analyse von NETosis entwickelt, jede mit ihren eigenen Vorteilen und Einschränkungen. In dieser Arbeit beschreiben wir eine Echtzeit-Mikroskopie-Methode zur Quantifizierung der menschlichen NET-Freisetzung, die es ermöglicht, sowohl die NETose als auch die NET-Inhibition im Hochdurchsatz zu untersuchen. Die oberflächenbasierte halbautomatische Analyse erkennt NETs und unterscheidet sie von nicht Netting-aktivierten Neutrophilen. Wir zeigen, dass die nicht-physiologischen NETosis-Induktoren, Calciumionophor und Phorbol-12-Myristat-13-acetat (PMA), die Freisetzung von NETs mit unterschiedlichen Eigenschaften und Kinetiken auslösen. Darüber hinaus zeigen wir, dass dieser Ansatz die Untersuchung der NET-Freisetzung als Reaktion auf krankheitsrelevante Stimuli ermöglicht, einschließlich Immunkomplexen, N-Formylmethionin-leucyl-phenylalanin (fMLF), Mononatriumuratkristallen und Calciumpyrophosphatkristallen. Um die Nützlichkeit dieser Methode zur Untersuchung von NETosis-Antagonisten zu veranschaulichen, verwendeten wir CIT-013, einen First-in-Class-monoklonalen Antikörper-Inhibitor der NET-Freisetzung. CIT-013 zielt auf citrulliniertes Histon H2A und H4 ab und hemmt die NET-Freisetzung effizient mit einem IC50 von 4,6 nM. Anderen getesteten Anti-Histon-Antikörpern fehlte diese NETose-hemmende Fähigkeit. Insgesamt zeigen wir, dass dieses Protokoll eine spezifische, zuverlässige und reproduzierbare Hochdurchsatz-Quantifizierung von NETs ermöglicht und die Untersuchung der NET-Freisetzungseigenschaften, der Kinetik und der Pharmakologie von NETosis-Antagonisten verbessert.
Einleitung
Neutrophile sind reichlich im Blut vorhanden und wandern bei einer Infektion oder Entzündung ins Gewebe. Sie spielen eine wichtige Rolle bei der angeborenen Immunabwehr, indem sie den Wirt mit umfangreichen Waffen vor Mikroben schützen. Neutrophile töten Krankheitserreger durch Phagozytose, Degranulation, Erzeugung reaktiver Sauerstoffspezies (ROS) und die Freisetzung von dekondensiertem Chromatin, die als neutrophile extrazelluläre Fallen (NETs) bezeichnet werden, durch einen Prozess namens NETosis1 ab. NETs sind extrazelluläre Strukturen des Chromatins, die u.a. mit granulären Proteinen und Calprotectin 2,3 dekoriert sind und bei Stimulation mit einem breiten Spektrum von Molekülen freigesetztwerden 4. NETose kann grob in zwei Hauptwege eingeteilt werden: NADPH-Oxidase-abhängig oder unabhängig 5,6,7. Darüber hinaus ist die Arginin-Citrullinierung von N-terminalen Histonschwänzen durch Peptidylarginin-Deiminase 4 (PAD4) eng mit NETosis verbunden und fördert die Chromatin-Dekondensation, was letztendlich zur Ausstoßung von dekondensiertem Chromatin in die extrazelluläre Umgebung führt.
Obwohl die NET-Freisetzung an der Eliminierung von Krankheitserregern beteiligt ist, haben zahlreiche Studien gezeigt, dass eine abnormale und verlängerte NET-Freisetzung mit der Entwicklung verschiedener entzündlicher Erkrankungen verbunden ist, darunter akute Lungenschädigung8, rheumatoide Arthritis (RA)9, Vaskulitis10 und Hidradenitis suppurativa11. Die nachteilige Rolle von NETs bei Krankheiten ist vielfältig, da NETs proinflammatorisch sind, eine Quelle von Autoantigenen sind, zytotoxisch für das umgebende Gewebe sind, Immunthrombosen auslösen und die Osteoklastendifferenzierung und Knochenerosion fördern 9,12,13. Die pharmakologische Hemmung des NETosis-Signalwegs durch niedermolekulare PAD4-Inhibitoren zeigt, dass NETosis-targetende Therapeutika das Potenzial haben, Krankheiten zu behandeln, bei denen die NET-Akkumulation ein wichtiger Treiber der Pathogenese ist14. Anstatt auf das PAD4-Enzym abzuzielen, verwendeten wir einen First-in-Class-NETose-hemmenden humanisierten anti-citrullinierten monoklonalen Histon-Antikörper, CIT-013, der spezifisch an die citrullinierten Histone H2A und H4bindet 15. CIT-013 hat einen einzigartigen dualen Wirkmechanismus, indem es die NET-Freisetzung hemmt und die Makrophagen-vermittelte NET-Phagozytose verstärkt16. CIT-013 und Vorläufermoleküle haben in mehreren Mausmodellen der NET-assoziierten Entzündung therapeutische Wirksamkeit gezeigt17.
Um die NET-Freisetzung zu untersuchen, wurden im Laufe der Jahre verschiedene Methoden entwickelt, wie z. B. 1) DNA-Nachweis mit einem plasmamembranundurchlässigen DNA-Tracer in Kombination mit einem Immunfluoreszenzplatten-Reader, 2) ELISA-basierter Nachweis von DNA und DNA-komplexierten mit NET-spezifischen Proteinen in Überständen, 3) Co-Lokalisierung von NET-assoziierten Molekülen mit extrazellulärer DNA durch Immunhistochemie, und 4) durchflusszytometrische Ansätze zum Nachweis von Netting-Neutrophilen. Alle diese Methoden haben ihre eigenen Vorteile und Grenzen. Wir haben einen Echtzeit-Hochdurchsatzansatz zur mikroskopischen Quantifizierung der humanen NET-Freisetzung entwickelt, der einen plasmamembranundurchlässigen DNA-Farbstoff16,18 verwendet. Das beschriebene Verfahren ermöglicht die Untersuchung der NETosis-Kinetik und -Eigenschaften auf einfache, zuverlässige und reproduzierbare Weise und ermöglicht die Beurteilung der Pharmakologie von NETosis-Antagonisten wie CIT-013.
Protokoll
Alle Blutspender gaben eine Einverständniserklärung in Übereinstimmung mit der Deklaration von Helsinki, und die Studie wurde gemäß den ethischen Richtlinien von Citryll für die Humanforschung durchgeführt.
HINWEIS: Alle Aktivitäten mit menschlichem Blut und isolierten Neutrophilen müssen unter sterilen Bedingungen in einem Laminar-Flow-Schrank durchgeführt werden. Wenn Brems- und Beschleunigungseinstellungen für die Zentrifugation nicht im Protokoll erwähnt werden, können sie maximal berücksichtigt werden.
1. Isolierung von Neutrophilen aus dem Blut
- Sammeln Sie peripheres Blut von gesunden Probanden in Lithium-Heparin-Röhrchen und übertragen Sie das Blut in ein frisches 50-ml-Röhrchen.
- Spülen Sie Lithium-Heparin-Röhrchen mit 1x DPBS und geben Sie es in dasselbe 50-ml-Röhrchen, in das das Blut überführt wurde. Stellen Sie sicher, dass das endgültige Blut-DPBS-Verhältnis 1:1 beträgt. Mischen Sie Blut und DPBS durch Pipettieren, um eine homogene Lösung zu erhalten.
- Geben Sie 13 ml Dichtegradientenlösung in ein weiteres frisches 50-ml-Röhrchen und fügen Sie langsam (mit einer 25-ml-Pipette) das 1:1 verdünnte Blut auf die Dichtegradientenlösung bis zu einem Maximum von 50 ml hinzu.
- Zentrifugieren Sie bei 400 x g und Raumtemperatur (RT) für 40 min mit minimaler Beschleunigung und Bremse.
HINWEIS: Es werden Schichten gebildet. Die Schichten von oben nach unten sind wie folgt: 1. Plasma; 2. mononukleäre Zellen des peripheren Blutes (PBMCs); 3. Lösung des Dichtegradienten; 4. Erythrozyten/Neutrophile. - Zuerst wird das Plasma mit einer 10-ml-Pipette verworfen und dann die PBMCs und die Dichtegradientenlösungsschicht mit einer Pasteurpipette aus Kunststoff so weit wie möglich verworfen, ohne die Erythrozyten-/Neutrophilenschicht zu stören.
- Die Erythrozyten-/Neutrophilenschicht durch Schütteln vorsichtig resuspendieren und mit einer 25-ml-Pipette in 15 mL 1x DPBS resuspendieren.
- Fügen Sie 25 ml 6%ige Dextran/0,9%ige NaCl-Lösung hinzu, mischen Sie das Röhrchen zehnmal, indem Sie es umdrehen, und stellen Sie das Röhrchen 25 Minuten lang bei RT aufrecht.
HINWEIS: Es werden Schichten gebildet. Die Schichten von oben nach unten sind wie folgt: 1. Neutrophile; 2 Erythrozyten. - Die Neutrophilenschicht mit einer 10 mL Pipette in ein frisches 50 mL Röhrchen überführen und 10 min bei 500 x g und RT zentrifugieren.
HINWEIS: Seien Sie vorsichtig, da das neutrophile Pellet noch einige Erythrozyten enthält und nicht fest mit dem Röhrchen verbunden ist. - Verwerfen Sie den Überstand durch Dekantieren und resuspendieren Sie das Zellpellet mit einer 10-ml-Pipette in 10 mL Ammoniumchlorid-Kalium (ACK)-Lysepuffer (155 mM NH4Cl, 10 mM KHCO3 und 0,1 mM Na2EDTA; pH = 7,2) und fügen Sie dann sofort 40 mL ACK-Lysepuffer hinzu.
- Inkubieren Sie bei RT, während Sie das Röhrchen kontinuierlich umdrehen, bis die Lösung durchscheinend wird (dies dauert 1-5 Minuten und kann je nach Spender variieren) und zentrifugieren Sie 10 Minuten lang bei 350 x g und RT.
- Entfernen Sie den Überstand und geben Sie langsam und tropfenweise 5 ml Kulturmedium mit L-Glutamin, ergänzt mit 10 % (v/v) hitzeinaktiviertem fötalem Rinderserum (FBS), 50 μl/ml Penicillin und 50 μg/ml Streptomycin (im Folgenden als Kulturmedium 10 % bezeichnet) auf das Neutrophilenpellet, ohne die Neutrophilen in Suspension zu bringen. Dadurch werden die meisten Erythrozyten effizient aus dem neutrophilen Pellet entfernt.
- Schwenken Sie das 50-ml-Röhrchen vorsichtig, bis die meisten Erythrozyten, die sich auf dem neutrophilen Pellet befinden, im Kulturmedium resuspendiert sind 10%. Entfernen Sie anschließend den Überstand, indem Sie das Röhrchen umfüllen.
- Resuspendieren Sie das Neutrophilenpellet in 10 ml Kulturmedium 10 % und fügen Sie nach vollständiger Resuspendierung bis zu 50 ml Kulturmedium 10 % hinzu. 10 min bei 350 x g und RT zentrifugieren.
- Entfernen Sie den Überstand und resuspendieren Sie das Zellpellet in 10 ml Kulturmedium 10 %.
2. Neutrophilenfärbung zur Überprüfung der Reinheit durch Durchflusszytometrie
- Um die Zellkonzentration zu bestimmen, verdünnen Sie die Neutrophilensuspension in 0,4%iger Trypanblaulösung (Verhältnis 1:1) und zählen Sie die Neutrophilen mit einem Hellfeld-Zellzähler.
HINWEIS: Es können auch andere Zählmethoden verwendet werden. - 1 x 105 Neutrophile in Kulturmedium 10 % in die Vertiefungen einer 96-Well-Platte mit V-Boden überführen und 3 Minuten lang bei 400 x g und RT zentrifugieren.
- Der Überstand wird verworfen und die Zellen in 50 μl 1x DPBS, ergänzt mit 1 % (w/v) Rinderserumalbumin (BSA) und 0,1 % (v/v) NaN3 (im Folgenden als Fluoreszenz-aktivierter Zellsortierungspuffer (FACS) bezeichnet), der einen Fc-Rezeptorblock (50x verdünnt) enthält, resuspendiert.
- Inkubieren Sie 15 Minuten lang bei RT und fügen Sie 50 μl FACS-Puffer hinzu, der 50x verdünnten APC-Cy7-konjugierten Maus-Anti-Human-CD45-Antikörper, 600x verdünnten PerCP-Cy5.5-konjugierten Maus-Anti-Human-CD16-Antikörper, 133x verdünnten FITC-konjugierten Maus-Anti-Human-CD66b-Antikörper und 500x verdünnten fixierbaren Viabilitätsfarbstoff eFluor 506 enthält.
HINWEIS: CD45 wird auf Leukozyten exprimiert. CD66b wird ausschließlich in Granulozyten exprimiert. CD16 wird in Neutrophilen stark exprimiert, in Eosinophilen niedrig exprimiert und in Basophilen nicht exprimiert. - 30 min bei RT im Dunkeln inkubieren und 3 min bei 400 x g und RT zentrifugieren.
- Der Überstand wird verworfen, die Neutrophilen in 175 μl FACS-Puffer resuspendiert und 3 Minuten lang bei 400 x g und RT zentrifugiert. Dieser Schritt wird einmal wiederholt.
- Der Überstand wird verworfen und das neutrophile Pellet in 175 μl FACS-Puffer resuspendiert. Analysieren Sie Proben mit einem Durchflusszytometriesystem und der zugehörigen Software.
3. Reinheitsanalyse von Neutrophilen mit einer Durchflusszytometrie-Analysesoftware
HINWEIS: Die Analyse der Durchflusszytometriedaten wurde mit der Durchflusszytometrie-Analysesoftware durchgeführt, wie in der Materialtabelle angegeben.
- Führen Sie das Gating gemäß der folgenden Schritt-für-Schritt-Anleitung durch.
- Legen Sie ein Zeitfenster im Diagramm FSC-A über Zeit fest, um einen Bereich mit dem richtigen Zellfluss auszuwählen.
- Legen Sie im Diagramm SSC-A vs. FSC-A ein Zellentor fest, um Zellen auszuwählen und Ablagerungen auszuschließen.
- Legen Sie ein Viable Cell Gate im Diagramm FSC-A vs. AmCyan-A fest, um lebensfähige Zellen auszuwählen.
- Setzen Sie ein Leukozyten-Gate im Diagramm FSC-A vs. APC-Cy7 , um CD45+-Zellen auszuwählen.
- Legen Sie ein Neutrophilen-Gate im Diagramm FITC vs. PerCP-Cy5.5 fest, um zwischen CD66b+CD16+ (Neutrophile), CD66b-CD16+ (Eosinophile) und CD66b-CD16- (Basophile, Monozyten und Lymphozyten) zu unterscheiden.
- Legen Sie ein einzelnes Zellgatter im Diagramm FSC-H vs. FSC-A fest, um einzelne Neutrophile auszuwählen.
- Bestimmen Sie die Reinheit der Neutrophilen, indem Sie einzelne Neutrophile als Häufigkeit der Zellen (im Zelltor) darstellen.
HINWEIS: Um fortzufahren, muss die Reinheit der Neutrophilen 85% überschreiten.
4. Live-Imaging-Mikroskopie
HINWEIS: Dieser Assay ist für mehrere 96-Well-Speicherfolien und verschiedene NETosis-Stimuli und Antagonisten optimiert. Das folgende Protokoll beschreibt eine allgemeine Sicht auf den Ansatz, die anhand der beigefügten Tabellen spezifiziert werden kann.
- Eine 0,001%ige Poly-L-Lysin-Lösung wird durch Verdünnung von 0,01 % Poly-L-Lysin in sterilem H2O im Verhältnis 1:10 hergestellt.
- 0,001 %ige Poly-L-Lysin-Lösung in jede Vertiefung geben und mindestens 1 h bei 37 °C inkubieren.
HINWEIS: Das Volumen pro Vertiefung hängt von der 96-Well-Speicherfolie ab (Tabelle 1). - Waschen Sie die Vertiefungen 3 Mal mit 200 μl DPBS, um überschüssiges Poly-L-Lysin zu entfernen. Führen Sie alle Schritte im Laminar-Flow-Schrank aus. Entfernen Sie den Deckel der Platte und trocknen Sie die Vertiefungen an der Luft, indem Sie die Platte im Laminar-Flow-Schrank öffnen, bis sie trocken ist (ca. 1 h).
- Berechnen Sie die Anzahl der für das Experiment benötigten Neutrophilen und übertragen Sie einen Überschuss in ein 15-ml-Röhrchen. 10 min bei 350 x g und RT zentrifugieren.
HINWEIS: Die Menge der Neutrophilen in jeder Vertiefung variiert je nach Art der 96-Well-Speicherfolie (Tabelle 1). Die Zelldichte ist optimiert, um eine gute Trennung der einzelnen Zellen zu ermöglichen, was für eine ordnungsgemäße Analyse erforderlich ist. Wenn die Neutrophilendichte zu hoch ist, überlappen sich Zellen und NETs mit benachbarten Zellen und NETs, was sich auf die Qualität der Analyse auswirkt. - Verwerfen Sie den Überstand und resuspendieren Sie die Neutrophilen in einem Kulturmedium ohne Phenolrot, ergänzt mit 2 % (v/v) FBS, 50 U/ml Penicillin und 50 μg/ml Streptomycin, 10 mM HEPES und 1 mM CaCl2 (im Folgenden als NET-Assay-Puffer bezeichnet).
- Bereiten Sie die folgenden Arbeitslösungen vor.
- Bereiten Sie 80 nM DNA-Farbstoff in einem NET-Assay-Puffer vor.
- Bereiten Sie NETosis-Stimuli in einem NET-Assay-Puffer vor.
- Bereiten Sie NETosis-Antagonisten in NET-Assay-Puffer vor.
HINWEIS: Beachten Sie, dass die Konzentrationen der oben beschriebenen Arbeitslösungen das 4-fache der im Bohrloch benötigten Endkonzentration betragen. Die empfohlenen Konzentrationen von NETosis-Stimuli und NETosis-Antagonisten variieren (Tabelle 2 und Tabelle 3).
- Geben Sie 4x konzentrierten DNA-Farbstoff in NET-Assay-Puffer in jede Vertiefung.
- Geben Sie 4x konzentrierte NETosis-Stimuli in NET-Assay-Puffer in die entsprechenden Vertiefungen und fügen Sie NET-Assay-Puffer nur zu Vertiefungen ohne Stimulus oder zu Vertiefungen mit beschichteten Immunkomplexen (cIC; Protokoll in Tabelle 2) hinzu.
- Geben Sie 4x konzentrierte NETosis-Antagonisten in NET-Assay-Puffer in die entsprechenden Wells. Geben Sie NET-Assay-Puffer nur in Vertiefungen ohne Antagonisten.
- Geben Sie Neutrophilensuspension in jede Vertiefung.
HINWEIS: Das Volumen von 4x konzentriertem DNA-Farbstoff, 4x konzentrierten NETosis-Stimuli im NET-Assay-Puffer, 4x konzentriertem NETosis-Antagonisten im NET-Assay-Puffer, der Anzahl der Neutrophilen und dem Volumen der Neutrophilensuspension pro Well hängt von der 96-Well-Speicherplatte ab (Tabelle 1). Vermeiden Sie die Bildung von Blasen beim Pipettieren. - Zentrifugieren Sie die Platte 2 Minuten lang bei 100 x g und RT und setzen Sie die 96-Well-Speicherfolie in das Lebendzellmikroskopie-Analysesystem ein, das in einem Inkubator bei 37 °C und 5 % CO2 platziert wird.
HINWEIS: Während der ersten Minute der Inkubation kann sich Kondenswasser auf dem Boden der 96-Well-Speicherfolie bilden. Diese sollte mit einem Taschentuch entfernt werden.
5. Einstellen der Software für das Lebendzellmikroskopie-Analysesystem für die Erfassung
HINWEIS: Phasenkontrast- und Immunfluoreszenzbilder wurden von einem Lebendzellmikroskopie-Analysesystem aufgenommen, das von seiner Analysesoftware gesteuert wurde.
- Öffnen Sie die Software für das Lebendzellmikroskopie-Analysesystem und klicken Sie auf Mit Gerät verbinden. Geben Sie den Benutzernamen und das Passwort ein. Klicken Sie auf Schedule to acquire und klicken Sie auf die Plus-Schaltfläche (Launch add vessel (Schiff starten). Wählen Sie anschließend Nach Zeitplan scannen und klicken Sie auf Weiter.
- Um ein neues Gefäß von Grund auf neu zu erstellen, wählen Sie im Abschnitt "Gefäß erstellen" die Option "Neu" aus und klicken Sie auf "Weiter". Wählen Sie im Abschnitt Scantyp die Option Standard aus, und klicken Sie auf Weiter.
- Wählen Sie die folgenden Scan-Einstellungen aus und klicken Sie auf Weiter: Zelle für Zelle: Keine; Bildkanäle: Phasenkontrast und Grün (Erfassungszeit 100 ms); Ziel: 20x.
- Wählen Sie im Abschnitt "Gefäßauswahl " den entsprechenden Behältertyp aus und klicken Sie auf "Weiter".
- Wenn Sie eine 96-Well-Platte #1 verwenden, wählen Sie Corning, Platte, 96, N/A, 3603, 96-Well-Corning (Blk/Wht), Mikrotiterplatten.
- Wenn Sie eine 96-Well-Platte #2 verwenden, wählen Sie Nunc, Platte, 96, N/A, 152028, 96-Well-Nunc opt bottom (Blk/Wht), Mikrotiterplatten.
- Geben Sie den Standort des Gefäßes in der Schublade an und klicken Sie auf Weiter. Wählen Sie im Abschnitt Scanmuster die Vertiefungen aus, die gescannt werden müssen, wählen Sie die Anzahl der Bilder pro Vertiefung aus (scannen Sie 4 Bilder pro Vertiefung, um einen repräsentativen Überblick zu erhalten) und klicken Sie auf Weiter.
HINWEIS: Wählen Sie keine leeren Vertiefungen aus, da dies den Autofokus beeinträchtigt. - Geben Sie Informationen über das Schiff an, indem Sie unter anderem den Namen des Kennzeichens in den Abschnitt des Schiffsnotizbuchs einfügen und auf Weiter klicken. Wählen Sie im Abschnitt Analyse-Setup die Option Analyse auf einen späteren Zeitpunkt verschieben aus, und klicken Sie auf Weiter.
- Definieren Sie den Scan-Zeitplan für das Schiff im Abschnitt Scan-Zeitplan . Wählen Sie Neuen Zeitplan mit Scans in Abständen von erstellen und dann 1 Stunde aus. Wählen Sie Scan stoppen und 00:05 Stunden nach dem ersten Scan aus. Klicken Sie auf Weiter und dann auf Zum Zeitplan hinzufügen , wenn die Scaninformationen korrekt sind.
- Wenn der Scanzeitplan bearbeitet werden muss, doppelklicken Sie auf den Scanzeitplan oben auf dem Bildschirm. Klicken Sie mit der rechten Maustaste auf den Scan-Zeitplan und bearbeiten und passen Sie ihn wie gewünscht an. Klicken Sie auf das Diskettensymbol , um den Scanzeitplan zu speichern.
HINWEIS: Wenn Sie mehrere Gefäße (Platten) im selben Experiment abbilden, können dem vorhandenen Scan-Zeitplan zusätzliche Gefäße hinzugefügt werden, indem Sie auf die Plus-Schaltfläche (Gefäß hinzufügen starten) klicken, Scan nach Zeitplan auswählen und auf Weiter klicken. Erstellen Sie entweder ein neues Gefäß von Grund auf neu (siehe Schritt 5.2 in diesem Protokoll), kopieren Sie ein vorhandenes Gefäß oder verwenden Sie ein zuvor gescanntes Gefäß. Dies führt zu sequentiellem Scannen mit der gleichen Häufigkeit und den gleichen Einstellungen wie bei der vorherigen Platte.
- Wenn der Scanzeitplan bearbeitet werden muss, doppelklicken Sie auf den Scanzeitplan oben auf dem Bildschirm. Klicken Sie mit der rechten Maustaste auf den Scan-Zeitplan und bearbeiten und passen Sie ihn wie gewünscht an. Klicken Sie auf das Diskettensymbol , um den Scanzeitplan zu speichern.
6. Live-Imaging-Mikroskopie-NET-Assay-Analyse mit der Lebendzellmikroskopie-Analysesoftware
HINWEIS: Zur Analyse von Phasenkontrast- und Immunfluoreszenzbildern wurde eine Software zur Analyse der Lebendzellmikroskopie verwendet (Materialtabelle). Wenn dieses System nicht verwendet wird, kann eine ähnliche NET-Analyse mit einem gemeinfreien Softwarepaket 4,18,19,20 durchgeführt werden.
- Öffnen Sie die Software zur Analyse der Lebendzellmikroskopie und klicken Sie auf Mit Gerät verbinden. Geben Sie den Benutzernamen und das Passwort ein und klicken Sie auf Letzte Scans anzeigen. Öffnen Sie das Experiment Ihrer Wahl, um es zu analysieren, indem Sie auf den Gefäßnamen des Experiments doppelklicken.
- Klicken Sie auf Analyse starten , und wählen Sie Neue Analysedefinition erstellen aus. Klicken Sie auf Weiter.
- Wählen Sie Basic Analyzer aus dem Abschnitt Analysetyp aus, und klicken Sie auf Weiter. Wählen Sie im Abschnitt Bildkanal die Option Grün aus, deaktivieren Sie Phase und klicken Sie auf Weiter.
- Öffnen Sie das Fenster Bildebenen, wählen Sie Grün und deaktivieren Sie Phase. Deaktivieren Sie die automatische Skalierung im grünen Bereich, und legen Sie manuell min und max für den grünen Kanal fest, um NETs vom Hintergrund zu unterscheiden.
HINWEIS: Der Phasenkontrast kann bei Bedarf ein- und ausgeschaltet werden. Zum Beispiel, um festzustellen, ob NETs in der extrazellulären Umgebung ausgestoßen werden. - Öffnen Sie das Fenster "Vessel Scan Times" und wählen Sie die entsprechende Vessel Scan-Zeit aus, in der NETs vorhanden sein müssen. Wählen Sie eine Reihe von Bildern aus, die die Variabilität innerhalb des Experiments darstellen (Bilder aus positiven Kontrollvertiefungen mit NETs und Bilder aus negativen Kontrollvertiefungen ohne NETs). Diese Bilder werden verwendet, um die Analyse in der Vorschau anzuzeigen und zu verfeinern. Klicken Sie auf Weiter.
- Öffnen Sie das Fenster Einstellungen für die Analysedefinition und beginnen Sie, die Analyseeinstellungen für den grünen Kanal so anzupassen, dass die NET-Strukturen umrissen werden, ohne falsch positive oder falsch negative NETs zu erzeugen. Verwenden Sie die folgenden Einstellungen und klicken Sie auf "Vorschau aktuell " oder "Vorschau aller Bilder", um die Einstellungen auf das aktuelle bzw. alle Bilder anzuwenden.
- Wählen Sie als Objektname die Option NETs aus.
- Wählen Sie für die Segmentierung Keine Hintergrundsubtraktion - Adaptiv aus.
- Wählen Sie für den Schwellenwert GCU zwischen 3 und 5 (je nach Experiment).
- Wählen Sie für die Kantenteilung die Option EIN (zwischen -10 und 0) aus.
- Wählen Sie für die Bereinigung Lochfüllung: 100 μm2 und Größe anpassen: -1 Pixel.
- Bei den Filtern wählen Sie je nach Experiment, aber die empfohlenen Grenzwerte sind: Fläche: >200 μm2, Mittlere Intensität: <24,6, Integrierte Intensität: >7000.
HINWEIS: Strukturen, die auf der Grundlage der verwendeten Einstellungen ausgewählt werden, werden magenta umrandet. Wenn Sie mit der Maus über diese Strukturen fahren, erhalten Sie weitere Informationen über die Parameter der Objekte (d. h. Fläche, Exzentrizität, integrierte Intensität und mittlere Intensität), was dazu beiträgt, die Analyse in Bezug auf Ein- oder Ausschlusskriterien für NET-Parameter zu optimieren. Überprüfen Sie mehrere Bilder (aus positiven und negativen Kontrollvertiefungen), um zu testen, ob die Analyseeinstellungen korrekt sind.
- Wenn die Einstellungen für die NET-Analyse korrekt sind, klicken Sie auf Weiter. Wählen Sie die zu analysierenden Zeitpunkte und Wells im Abschnitt Scanzeiten und Wells aus und klicken Sie auf Weiter. Geben Sie den Definitionsnamen in den Abschnitt Analysedefinition speichern und anwenden ein, und klicken Sie auf Weiter. Klicken Sie auf Fertig stellen , wenn die Analyseinformationen überprüft und korrekt sind.
HINWEIS: Wenn Sie mehrere Platten analysieren oder Analysen aus früheren Experimenten verwenden, starten Sie die Analyse, wählen Sie Vorhandene Analysedefinition kopieren oder Vorhandene Analysedefinition verwenden und klicken Sie auf Weiter. Wählen Sie die entsprechende vorhandene Analysedefinition aus, und klicken Sie auf Weiter. Für die anschließende Analyseprozedur gehen Sie zu Schritt 6.4, wenn Sie die Option Vorhandene Analysedefinition kopieren gewählt haben, bzw. mit Schritt 6.7, wenn Sie die Option Vorhandene Analysedefinition verwenden gewählt haben. - Öffnen Sie die Experimentanalyse, indem Sie auf die Analyse innerhalb des gewünschten Gefäßes klicken. Öffnen Sie das Fenster Diagrammmetriken .
- Klicken Sie auf die Plus-Schaltfläche (Metrik erstellen). Die Daten können als prozentuale NET-Konfluenz (der Prozentsatz des Bildbereichs, der als NET markiert ist) oder als prozentuale Netting-Neutrophile (Anzahl der NETs geteilt durch die Anzahl der Zellen) dargestellt werden.
- Wenn Sie Daten als prozentuale NET-Konfluenz darstellen, wählen Sie im Abschnitt Metrik die Option Bereich und dann im Abschnitt Wert die Option Konfluenz aus. Wenn Sie Daten als prozentuale Netting-Neutrophile darstellen, wählen Sie im Abschnitt "Metrik" die Option "Objektanzahl" und im Abschnitt "Wert" die Option "Pro Bild" aus.
- Klicken Sie auf OK. Wählen Sie NETs Area Confluence (%) oder NETs Object Count Per Image (Pro Image) im Abschnitt Benutzerdefinierte Metriken aus. Wählen Sie im Abschnitt "Scans auswählen " alle Zeitpunkte aus, die analysiert werden sollen. Wählen Sie im Abschnitt Wells auswählen alle Wells aus, die analysiert werden müssen, und klicken Sie auf Daten exportieren.
- Wählen Sie die folgenden Einstellungen im Abschnitt Grafischer Export aus.
- Wählen Sie Jeden Scan als eigene Tabelle anzeigen (Spalten: 1, 2... Reihen: A, B...).
- Wählen Sie Zeilen- und Spaltenbeschriftungen anzeigen aus.
- Wählen Sie Alle Scans in einer Datei aus und klicken Sie auf Durchsuchen , um den Speicherort und den Namen der Datei anzupassen.
- Wählen Sie Experimentdetails in Kopfzeile einschließen aus.
- Wählen Sie Daten in einzelne Bilder aufschlüsseln aus.
- Klicken Sie auf Exportieren , um die Daten als ".txt"-Datei zu exportieren, die importiert und in einer Tabelle weiter analysiert werden kann.
HINWEIS: Um den Prozentsatz der Netting-Neutrophilen zu bestimmen, muss die Anzahl der Neutrophilen bei t = 0 bestimmt werden. Fahren Sie dazu mit den folgenden Schritten des Protokolls fort. - Fahren Sie mit Schritt 6.1 fort, um die zusätzliche Analyse zu starten, die erforderlich ist, um die Anzahl der Neutrophilen für Bilder bei t = 0 zu zählen.
- Wählen Sie in Schritt 6.3 Phase aus und deaktivieren Sie Grün im Bildkanalbereich.
- Wählen Sie in Schritt 6.4 im Abschnitt "Scanzeiten des Schiffes" die Option t = 0 aus.
- Deaktivieren Sie in Schritt 6.5 Grün, wählen Sie Phase und passen Sie die Phasenkanaleinstellungen bei Bedarf an.
- Fügen Sie in Schritt 6.6 den Objektnamen hinzu, wählen Sie im Abschnitt Segmentierung die Option Klassische Konfluenz aus und setzen Sie die Segmentierungsanpassung auf 0 (Hintergrund), stellen Sie die Lochfüllung auf 100 μm2 und die Fläche auf 50 μm2 ein.
- Wählen Sie in Schritt 6.7 im Abschnitt "Scans auswählen" die Option t = 0 aus.
- Öffnen Sie in Schritt 6.8 die Neutrophilenzahlanalyse.
- Wählen Sie in Schritt 6.9 im Abschnitt Benutzerdefinierte Metriken die Option Objektanzahl pro Bild (pro Bild) aus.
- Verwenden Sie abschließend die folgende Berechnung, um den Prozentsatz der Netting-Neutrophilen zu generieren: (Anzahl der NETs / Anzahl der Neutrophilen bei t = 0) x 100%.
Ergebnisse
Die Echtzeit-Hochdurchsatzmikroskopie in Kombination mit einem undurchlässigen DNA-Farbstoff ermöglicht die Untersuchung der Kinetik, der Eigenschaften und der zugrundeliegenden Signalwege von NETose und ermöglicht die Bewertung potenzieller Inhibitoren der NETose-Freisetzung. Mit diesem Ansatz wurden NETs als Strukturen definiert, die positiv für DNA-Farbstoffe sind und im Vergleich zur Oberfläche gesunder Neutrophilen eine signifikant größere Oberfläche aufweisen (Abbildung 1A), was darauf hindeutet, dass Chromatin in die extrazelluläre Umgebung ausgestoßen wurde18. Die oberflächenbasierte Analyse ermöglichte es uns, zwischen NETs und aktivierten Neutrophilen mit beeinträchtigter Plasmamembranintegrität zu unterscheiden, was eine helle intrazelluläre DNA-Färbung zeigte (Abbildung 1A).
Calciumionophor (A23187) und PMA werden häufig verwendet, um NETose in vitro zu induzieren. Obwohl es sich um nicht-physiologische Stimuli handelt, sind sie wertvoll, da sie unterschiedliche NETosis-Signalwege aktivieren und eine konsistente NETosis-Induktion mit geringer Variabilität zwischen den Spendern gewährleisten. A23187 löst den Kalziumeinstrom aus, was zur PAD4-Aktivierung und Freisetzung von NETs führt, die reich an citrullinierten Histonen sind, während PMA den NADPH-Oxidase-Komplex aktiviert, was zu einer Produktion reaktiver Sauerstoffspezies (ROS) und der anschließenden Freisetzung von NETs mit geringen Gehalten an citrullinierten Histonen führt 5,16,21. Die Geschwindigkeit und das Ausmaß der NETosis-Antwort hängen von der Konzentration jedes Stimulus ab (Abbildung 1B, C), wobei A23187 eine schnellere NETose induziert und PMA zu einem höheren Anteil von Neutrophilen führt, die NETs freisetzen (Abbildung 1D-F). NETs, die aus der Stimulation von A23187 resultierten (Abbildung 1D; roter Pfeil), unterschieden sich von PMA-induzierten NETs (Abbildung 1D; gelber Pfeil) dadurch, dass sie jenseits der neutrophilen Plasmamembran diffuser waren, während PMA-induzierte NETs weiter in der Nähe der neutrophilen Plasmamembran blieben. Darüber hinaus führte die Stimulation mit A23187 zu permeablen nicht-netzenden DNA-Farbstoff-positiven Neutrophilen (Abbildung 1D; blauer Pfeil), die ihre DNA nicht in den extrazellulären Raum ausstießen. Der Nachweis von permeablen nicht-netting-DNA-Farbstoff-positiven Neutrophilen hing von der Konzentration von A23187 ab (Abbildung 1G) und war unabhängig von der Konzentration von PMA, die zur Stimulierung der NETose verwendet wurde, fast nicht vorhanden (Abbildung 1D,H). Neben der Verwendung der nicht-physiologischen NETose-Stimuli A23187 und PMA eignet sich dieser Assay auch zur Untersuchung krankheitsrelevanter Auslöser der NETosis. Zum Beispiel zeigten Neutrophile, die mit löslichen Immunkomplexen (sIC) oder Kristallen aktiviert wurden, die bei der Calciumpyrophosphat-Dispositionskrankheit (CPPD) vorhanden sind, eine signifikant erhöhte NET-Freisetzung im Vergleich zu keinem Stimulus. Ein Trend zu erhöhten NET-Spiegeln wurde beobachtet, wenn Neutrophile mit beschichteten Immunkomplexen (cIC), fMLP und Mononatriumurat (MSU)-Kristallen aktiviert wurden (Abbildung 1I); Bei diesen Stimuli beobachteten wir jedoch eine beträchtliche Variation von Spender zu Spender.
Die pharmakologische Hemmung des NETosis-Signalwegs und der NETosis-assoziierten Enzyme hat gezeigt, dass NETosis-targeting-Therapeutika wirksame Behandlungen für Krankheiten sein könnten, bei denen die NET-Akkumulation die Pathologie signifikant beeinflusst 14,17,22,23,24. Dieser Echtzeit-Mikroskopie-NET-Assay ist ein einfacher, zuverlässiger und reproduzierbarer Ansatz zur Untersuchung von NETosis-Antagonisten mit hohem Durchsatz. Um dies zu veranschaulichen, haben wir einen First-in-Class-humanisierten monoklonalen Antikörper, CIT-013, verwendet, der auf citrulliniertes Histon H2A und H4 mit hoher Affinität abzielt15,16. A23187 aktiviert den NETosis-Signalweg, was zu NETs führt, die citrullinierte Histone enthalten, die anschließend von CIT-013 angegriffen werden, um seine NETosis-inhibitorische Funktion auszuführen16. Tatsächlich wurde die NET-Freisetzung als Reaktion auf A23187 durch CIT-013 vollständig gehemmt (Abbildung 2A und ergänzendes Video 1) mit einem IC50 von 4,6 nM (Abbildung 2B). Die NETose-hemmende Kapazität von CIT-013 ist einzigartig, da andere kommerzielle Antikörper, die auf andere (nicht-) citrullinierte Histone abzielen, nicht in der Lage waren, die NET-Freisetzung zu hemmen (Abbildung 2C).
Zuvor haben wir gezeigt, dass ein sehr ähnliches Vorläufermolekül von CIT-013 (das zwei Aminosäuren unterscheidet, aber ähnliche Epitope mit gleicher Affinität bindet) die NETose als Reaktion auf physiologische Reize wie aktivierte Blutplättchen, Gicht-Synovialflüssigkeit sowie RA-Synovialflüssigkeit blockiert17. Hier zeigen wir, dass die durch sIC induzierte NET-Freisetzung durch CIT-013 gehemmt werden kann (Abbildung 2D). Die therapeutische Relevanz der Hemmung der durch diesen Stimulus induzierten NETose wird durch SLE, RA und andere Autoimmunerkrankungen unterstrichen, bei denen Autoantikörper im Serum oder in der Synovialflüssigkeit die Bildung von IC unterstützen, die NETosis auslöst25,26.
Zusammengenommen zeigen diese Daten, dass dieser Echtzeit-Hochdurchsatz-Mikroskopie-Ansatz für die Untersuchung der Kinetik und Eigenschaften der NET-Freisetzung geeignet ist und die Untersuchung von Inhibitoren der NETose ermöglicht. Obwohl diese Methode für die Verwendung von menschlichen Neutrophilen optimiert wurde, könnte sie mit Modifikationen auch geeignet sein, Neutrophile anderer Spezies zu untersuchen. Die mit diesem Assay generierten Daten sind ein Eckpfeiler der Begründung für CIT-013 als wirksame und wirksame Therapie für NET-bedingte Krankheiten.
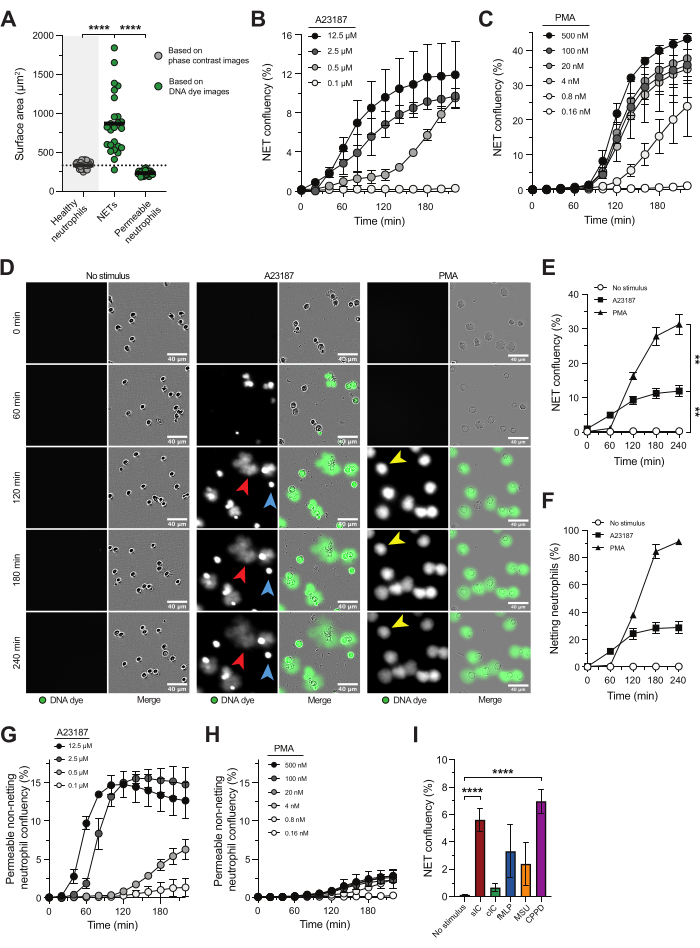
Abbildung 1: Echtzeit-Hochdurchsatzmikroskopie zur Untersuchung der NET-Version. Neutrophile, die aus dem Blut gesunder Probanden isoliert wurden, wurden mit A23187 oder PMA stimuliert, um den NETosis-Signalweg auszulösen. NET-Freisetzung wurde mit Echtzeit-Hochdurchsatzmikroskopie unter Verwendung eines plasmamembranundurchlässigen DNA-Farbstoffs visualisiert und auf der Grundlage der Oberfläche mit der Software des Lebendzellmikroskopie-Analysesystems quantifiziert. (A) Analyse der Oberfläche von nicht stimulierten gesunden Neutrophilen (grau), extrazellulären NETs und permeablen Nicht-Netting-Neutrophilen mit intrazellulärer DNA-Färbung (grün). (B,C) Quantifizierung der NET-Freisetzung über die Zeit von Neutrophilen, die mit den angegebenen A23187- oder PMA-Konzentrationen (n = 2) stimuliert wurden. (D) Repräsentative Bilder der NET-Freisetzung als Reaktion auf A23187 (rote Pfeile) und PMA (gelbe Pfeile) zu unterschiedlichen Zeitpunkten. Beispiele für permeable, nicht netzende Neutrophile sind mit blauen Pfeilen gekennzeichnet. (E) Quantifizierung der NET-Freisetzung im Zeitverlauf, dargestellt als Prozentsatz der NET-Konfluenz (n = 5). Die Statistik wurde durchgeführt für t = 240 min. (F) Quantifizierung der NET-Freisetzung über die Zeit, dargestellt als Prozentsatz der Netting-Neutrophilen (n = 2). (G, H) Quantifizierung permeabler nicht-nettierender Neutrophilen im Zeitverlauf von Neutrophilen, die mit den angegebenen A23187- oder PMA-Konzentrationen (n = 2) stimuliert wurden. (I) Quantifizierung der NET-Freisetzung bei t = 240 min, induziert durch lösliche Immunkomplexe (sIC), beschichtete IC (cIC), fMLP, Mononatriumurat (MSU)-Kristalle und Kristalle, die bei der Calciumpyrophosphat-Dispositionskrankheit (CPPD) vorhanden sind (n = 8-28). Die Ergebnisse werden als Mittelwert ± Standardfehler des Mittelwerts angegeben. **P < 0,01 und ****P < 0,0001, Wiederholte Messungen der unidirektionalen ANOVA mit dem Dunnett-Mehrfachvergleichstest (B), Kruskal-Wallis-Test mit dem Dunn-Mehrfachvergleichstest (I). Die Tafeln A-F wurden mit Genehmigung von van der Linden et al. geändert.16. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
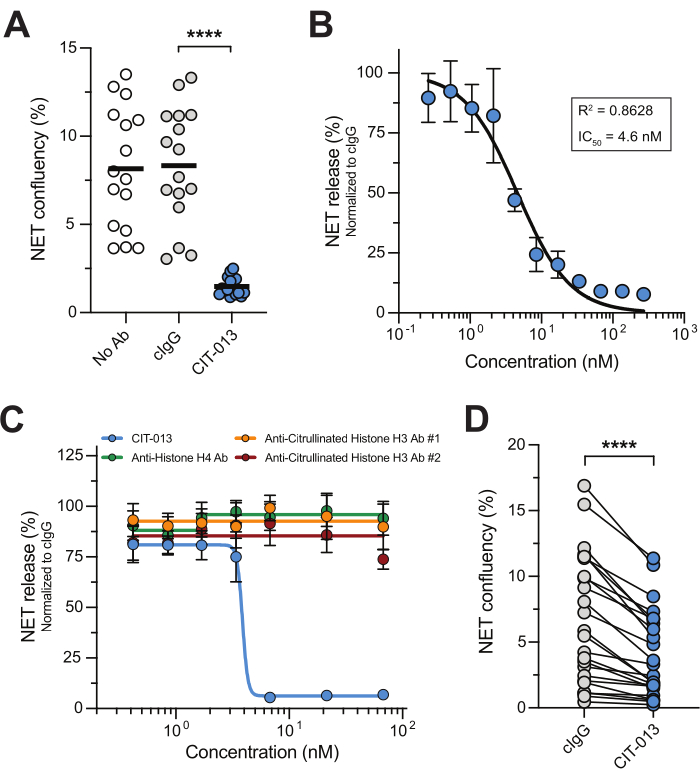
Abbildung 2: CIT-013 verhindert die NET-Freigabe. (A) Quantifizierung der A23187-induzierten NET-Freisetzung bei t = 240 min in Abwesenheit (kein Ab) oder Vorhandensein von CIT-013 oder Isotyp-Kontroll-Antikörper (cIgG). (B) Dosisabhängige Hemmung der A23187-induzierten NET-Freisetzung mit CIT-013 bei t = 240 min (n = 3). Die Daten wurden auf cIgG normalisiert (festgelegt als 100% NET-Release). (C) Quantifizierung der A23187-induzierten NET-Freisetzung bei t = 240 min in Gegenwart der angegebenen Konzentrationen von CIT-013, Anti-Histon-H4-Antikörper, anti-citrulliniertem Histon-H3-Antikörper #1 oder anti-citrulliniertem Histon-H3-Antikörper #2 (n = 6). (D) Quantifizierung der NET-Freisetzung bei t = 240 min, induziert durch lösliche Immunkomplexe (ICs) in Gegenwart von CIT-013 oder cIgG. Die Ergebnisse werden als Mittelwert ± Standardfehler des Mittelwerts angegeben. P < 0,0001, Wiederholte Messungen der unidirektionalen ANOVA mit Tukey-Mehrfachvergleichen (A) oder zweiseitigem Wilcoxon-Matched-Pairs-Rangtest mit Vorzeichen (D). Die Tafeln A, B und D wurden mit Genehmigung von van der Linden et al. modifiziert.16. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| 96-Well-Platte #1 | 96-Well-Platte #2 | |
| Volumen 0,001 % Poly-L-Lysin-Lösung pro Vertiefung | 50 μL | 100 μL |
| Volumen der Neutrophilensuspension pro Vertiefung | 50 μL | 87,5 μl |
| Anzahl der Neutrophilen pro Vertiefung | 2 x 104 Zellen | 3,5 x 104 Zellen |
| Volumen 4x konzentrierter (= 80 nM) DNA-Farbstoff im NET-Assay-Puffer pro Well | 50 μL | 87,5 μl |
| Volumen 4x konzentrierte NETosis-Stimuli in NET-Assay-Puffer pro Vertiefung | 50 μL | 87,5 μl |
| Volumen 4x konzentrierte NETosis-Antagonisten in NET-Assay-Puffer pro Well | 50 μL | 87,5 μl |
Tabelle 1: Volumina und Zellzahlen, optimiert für verschiedene 96-Well-Bildgebungsplatten.
| Konzentrationslösung (4x konzentriert) | Endkonzentration | |
| Calcium-Ionophor (A23187) | 50 μM | 12,5 μM |
| PMA | 16 nM | 4 nM |
| fMLP | 4 μM | 1 μM |
| Mononatriumurat (MSU)-Kristalle | 400 μg/mL | 100 μg/ml |
| Kristalle der Calciumpyrophosphat-Dispositionskrankheit (CPPD) | 400 μg/mL | 100 μg/ml |
| Lösliche Immunkomplexe (sIC) | 1. Fügen Sie 5 μg/ml humanes Serumalbumin (HSA) in DPBS zu 282,5 μg/ml polyklonalem Kaninchen-Anti-HSA-Antikörper in DPBS hinzu. | |
| 2. Mindestens 90 Minuten bei 37 °C inkubieren. | ||
| 3. Homogenisieren Sie durch Vortexen und geben Sie 50 μl sIC-Lösung in die entsprechenden Wells. | ||
| Beschichtete Immunkomplexe (cIC) | 1. Geben Sie 10 μg/ml HSA in DPBS in die entsprechenden Vertiefungen der 96-Well-Platte. | |
| 2. Über Nacht bei 4 °C inkubieren. | ||
| 3. Waschen Sie die Vertiefungen 3 Mal mit 200 μl 0,05 % Tween-20 in DPBS (im Folgenden als PBS/0,05 % Tween bezeichnet). | ||
| 4. Blockieren Sie die Vertiefungen mit 200 μl 1 % (w/v) Rinderserumalbumin in PBS/0,05 % Tween (im Folgenden als Blockierungspuffer bezeichnet). | ||
| 5. 120 min bei Raumtemperatur und leichtem Rühren (400 U/min) inkubieren. | ||
| 6. Waschen Sie die Vertiefungen 3 Mal mit 200 μl PBS/0,05 % Tween. | ||
| 7. Geben Sie 50 μl polyklonalen Kaninchen-Anti-HSA-Antikörper in Blockpuffer in die entsprechenden Vertiefungen. | ||
| 8. 60 Minuten bei Raumtemperatur und leichtem Rühren (400 U/min) inkubieren. | ||
| 9. Waschen Sie die Vertiefungen 3 Mal mit 200 μl PBS/0,05 % Tween. | ||
| 10. Zum Schluss waschen Sie die Vertiefungen 3 Mal mit 200 μL DPBS. Die Bohrlöcher sind nun bereit für Schritt 4.7 im Protokoll. | ||
Tabelle 2: Empfohlene Konzentrationen für NETosis-Stimuli.
| Konzentrationslösung (4x konzentriert) | Endkonzentration | |
| Anti-Hühnerei-Lysozym-Antikörper (Kontroll-Antikörper; cIgG) | 80 nm | 20 nM |
| CIT-013 | 80 nm | 20 nM |
Tabelle 3: Empfohlene Konzentrationen für NETosis-Antagonisten.
Ergänzendes Video 1. Neutrophile wurden mit A23187 in Gegenwart von cIgG (links) oder CIT-013 (rechts) stimuliert, und die NET-Freisetzung wurde im Laufe der Zeit mit dem plasmamembranundurchlässigen DNA-Farbstoff visualisiert. NET-Freisetzung wird in Gegenwart von CIT-013 blockiert. Der Film ist eine Überlagerung von DNA-Farbstoff (grün) und Phasenkontrast. Dieses Video wurde mit Genehmigung von van der Linden et al.16 erstellt. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Diskussion
Seit der Entdeckung von NETs im Jahr 2004 wurden viele Strategien entwickelt, um die Freisetzung von NETs in vitro experimentell zu untersuchen, wobei die Immunfluoreszenzmikroskopie die gebräuchlichste Technik zur Quantifizierung von NETosisist 27,28. Obwohl die Mikroskopie nützlich ist, um die NET-Veröffentlichung zu visualisieren, hat sie Einschränkungen, da die nicht-automatisierte mikroskopische Quantifizierung von Bildern mit festem Zeitpunkt ziemlich ungenau ist und unter Beobachterverzerrungen leiden kann. Eine weitere Technik, die zur Untersuchung der NET-Freisetzung verwendet wird, ist die multispektrale bildgebende Durchflusszytometrie29,30, die eine große Anzahl von Netting-Neutrophilen misst und eine unvoreingenommene Analyse anwendet, sich jedoch auf Neutrophile in den frühen Stadien der NETose konzentriert und Neutrophile, die ihre NETs freigesetzt haben, nicht quantifiziert. Viele Studien zur NETosis-Kinetik verwenden einen DNA-Farbstoff zur Quantifizierung der NET-Freisetzung in Kombination mit einem Fluoreszenzplatten-Reader31. Diese Technik ist nicht in der Lage, NETs von aktivierten oder sterbenden Neutrophilen mit intrazellulärer DNA-Färbung zu unterscheiden und ist daher für die Quantifizierung der NET-Freisetzung und die Untersuchung von NETosis-Antagonisten ungeeignet. Das Obige unterstreicht, dass die derzeit verwendeten Ansätze zur Untersuchung von NETosis wertvoll sind, aber ihre Grenzen haben.
Die in dieser Studie beschriebene Echtzeitmikroskopie-Methode befasst sich mit vielen Problemen, die bei zuvor berichteten Techniken gefunden wurden. Es bietet eine unvoreingenommene, halbautomatische, reproduzierbare und genaue NET-Quantifizierung mit hohem Durchsatz. Tatsächlich minimiert dieser Ansatz Pipettierartefakte, wie die gestreckte Morphologie der NETs18, und ist in der Lage, NETose von Nekrose und Apoptose durch die unterschiedliche Morphologie und Kinetik der DNA-Freisetzungzu unterscheiden 18,32. Darüber hinaus bietet dieser Ansatz die Möglichkeit, verschiedene biochemische Wege für den regulierten Zelltod zu untersuchen, wie z.B. die NET-Bildung in nekrotischen Neutrophilen, die Strukturen erzeugen, die die funktionellen und phänotypischen Kriterien von NETs erfüllen33.
Es gibt mehrere kritische Schritte in diesem Protokoll, die für eine erfolgreiche Quantifizierung von NETs befolgt werden müssen. Erstens ist die richtige Anzahl von Zellen pro Well für eine genaue NET-Quantifizierung wichtig. Wenn die Neutrophilendichte zu hoch ist, überlappen sich Zellen und NETs mit benachbarten Zellen und NETs, was ihre Unterscheidung erschwert und die Quantifizierung infolgedessen ungenau wird. Zweitens ist es eine Voraussetzung, eine niedrige Konzentration des ungiftigen Plasmamembran-undurchlässigen DNA-Bindungsfarbstoffs zu verwenden, um NETs zu färben, wenn sie in die extrazelluläre Umgebung sezerniert werden. Plasmamembran-permeable DNA-Bindungsfarbstoffe können leicht die Aktivierung von Neutrophilen oder den Zelltod induzieren. Drittens müssen mehrere Bilder pro Vertiefung gescannt werden, um einen repräsentativen Überblick über die NET-Freisetzung in der heterogenen Neutrophilenpopulation zu erhalten. Viertens ist das Phasenkontrastbild der Neutrophilen bei t = 0 notwendig, um die Anzahl der Neutrophilen pro Bild zu korrigieren und den Prozentsatz der Netto-Neutrophilen zu berechnen.
Obwohl dieser Echtzeit-Hochdurchsatz-Mikroskopieansatz viele Vorteile gegenüber anderen NET-Detektionsassays hat, gibt es Einschränkungen bei dieser Methode, da unseres Wissens nach keine Fluoreszenzfarbstoffe zur Verfügung stehen, um zusätzliche NET-Komponenten zu detektieren und die Freisetzung von NETs zu bestätigen. Fluoreszenzmarkierte Antikörper zum Nachweis zusätzlicher NET-Komponenten können verwendet werden, könnten aber unerwünschte Wirkungen haben, da Antikörper-Immun-Komplexe die Aktivierung von Neutrophilen und möglicherweise die NETose beeinflussen. Daher ziehen wir es vor, keine zusätzlichen Antikörper in diesem Assay-Setup zu verwenden und empfehlen stattdessen, bekannte NETosis-induzierende Stimuli zu verwenden. Wenn noch nicht etablierte NETosis-Stimuli verwendet werden, befürworten wir die Verwendung eines ELISAs zum Nachweis von DNA, die mit NET-spezifischen Proteinen wie citrullinisierten Histonen komplexiert ist, bevor der Live-Imaging-Assay durchgeführt wird. Zweitens kann die Variation innerhalb und zwischen den Assays aus der Variabilität von Spender zu Spender resultieren, wobei Neutrophile von gesunden Spendern aufgrund der Heterogenität innerhalb der gesunden Population unterschiedlich auf verschiedene NETosis-Stimuli reagieren. Um die Variation des Assays zu minimieren, müssen Neutrophile innerhalb von 1 h aus frisch entnommenem Blut isoliert und sofort im Experiment verwendet werden, da Neutrophile kurzlebig sind und nicht kryokonserviert werden können. Darüber hinaus ist es wichtig, dass ein Protokoll zur Isolierung von Neutrophilen validiert und übernommen wird, um die Aktivierung von Neutrophilen zu minimieren. Neutrophile Zellen sind empfindliche Zellen und können ihre Reaktionsfähigkeit auf Reize während des Reinigungsprozesses aufgrund von mechanischem und anderen Stress verändern. Der Aktivierungsstatus der Neutrophilen und die NET-Freisetzung können durch die Art der Nadel, die verwendeten Blutentnahmeröhrchen, die Inkubationstemperatur, die Zentrifugationsgeschwindigkeit und die Zeit von der Blutentnahme bis zur Isolierung der Neutrophilen beeinflusst werden 34,35,36,37. Eine zusätzliche in Betracht kommende Methode zur Isolierung von Neutrophilen wird von Krémer et al.36 unter Verwendung einer negativen immunomagnetischen Selektionsmethode ohne Erythrozytenlyse beschrieben. Diese Methode ähnelt unberührten Neutrophilen im Vollblut und könnte geeignet sein, eine Aktivierung von Neutrophilen während des Reinigungsprozesses zu verhindern. All dies sollte dazu dienen, das Feld darauf hinzuweisen, dass Daten aus verschiedenen Forschungsgruppen mit großer Sorgfalt verglichen werden müssen.
Insgesamt ermöglicht die beschriebene Echtzeit-Hochdurchsatz-Mikroskopie-Methode eine genaue Quantifizierung von NETs auf reproduzierbare und effiziente Weise und kann zur Untersuchung der Eigenschaften, des Ausmaßes und der Kinetik der NET-Freisetzung verwendet werden und ermöglicht die Untersuchung der Aktivität von NETosis-Antagonisten. Als Beispiel für Letzteres haben wir den humanisierten anti-citrullinierten Histon H2A und H4 monoklonalen Antikörper CIT-013 verwendet, der sich derzeit in der klinischen Entwicklung befindet.
Offenlegungen
Die Autoren sind Mitarbeiter von Citryll und haben finanzielle Interessen.
Danksagungen
Die Autoren danken Paul Vink für die Leitung eines Teils des Projekts zur Live-Imaging-Mikroskopie-Methode.
Materialien
| Name | Company | Catalog Number | Comments |
| A-23187 Free Acid (Calcimycin) | Thermo Fisher | A1493 | |
| Ammonium chloride (NH4Cl) | Sigma Aldrich | A9434 | |
| Anti-Hen egg lysozyme (control antibody; cIgG) | CrownBio | C0001 | |
| APC-Cy7-conjugated mouse anti-human CD45 antibody (Clone 2D1) | Biolegend | 368515 | Use at 1 µg/mL final concentration |
| BD FACSCantoTM II system + FACSDiva software (version 8.0.1) | BD | n/a | Flow cytometry system |
| Bovine Serum Albumin (BSA) Fraction V | Roche | 10735108001 | |
| CaCl2 (1 M) | VWR | E506 | |
| Calcium pyrophosphate disposition disease (CPPD) crystals | InvivoGen | lrl-cppd | |
| Cellometer Auto T4 Bright Field Cell Counter + analysis software (version 3.3.9.5) | Nexcelom Bioscience | n/a | Bright field cell counter |
| CIT-013 | Citryll B.V. | n/a | |
| Costar black 96-well plate, clear bottom | Corning | 3603 | 96-well plate #1 |
| Dextran T500 | Pharmacosmos | 551005009006 | |
| DPBS (1x) | Gibco | 14190-144 | |
| Fetal Bovine Serum (FBS) | Serena | S-FBS-SA-015 | |
| Ficoll | GE Healthcare | 17-1440-02 | Density gradient solution |
| FITC-conjugated mouse anti-human CD66b antibody (Clone G10F5) | eBioscience | 17-0666-42 | Use at 1.5 µg/mL final concentration |
| Fixable viability dye eFluor 506 | eBioscience | 65-0866-14 | |
| Flow cytometry analysis software | FLowJo | Version 10.8.0 | |
| fMLP | Sigma Aldrich | 47729-10MG-F | |
| HEPES (1M) | Gibco | 15630-080 | |
| Human serum albumin (HSA) | VWR | 31020 | |
| Human TruStain FcX | Biolegend | 422302 | Fc receptor block |
| IBIDI 96-well plate, clear bottom | IBIDI | 89626 | 96-well plate #2 |
| IncuCyte SX3 + analysis software (version 2022B Rev1) | Satorius | n/a | Live cell microscopic analysis system |
| Monosodium urate crystals | InvivoGen | tlrl-msu | |
| Mouse anti-histone H3 (citrulline R2 + R8 + R17) antibody | Cayman | 17939 | Clone 11D3; referred to as #1 |
| Mouse anti-histone H4 (K8Ac + K12Ac + K16Ac) antibody (Clone KM-2) | Absolute Antibodies | Ab01681-2.0 | |
| Na2EDTA | Sigma Aldrich | E5134 | |
| NaCl (0.9%) | B. Braun | 25900 | |
| Penicillin (5000 U/mL) - Streptomycin (5000 µg/mL) | Gibco | 15070-063 | |
| PerCP-Cy5.5-conjugated mouse anti-human CD16 antibody (Clone 3G8) | Biolegend | 302027 | Use at 0.33 µg/mL final concentration |
| PMA | Sigma Aldrich | P1585 | |
| Polyclonal rabbit anti-HSA antibody | Sigma Aldrich | A0433-2ml | |
| Poly-L-Lysine (0.01%) | Sigma Aldrich | P4832 | |
| Potassium bicarbonate (KHCO3) | Sigma Aldrich | 237205 | |
| Rabbit anti-histone H3 (citrulline R2 + R8 + R17) antibody | Abcam | ab281584 | Multiclonal; referred to as #2 |
| RPMI 1640 with GlutaMAXTM supplement | Gibco | 61870-010 | Culture medium containing L-glutamine |
| RPMI 1640 without phenol red | Gibco | 11835-030 | Culture medium without phenol red |
| Sodium azide (NaN3) | Sigma Aldrich | S2002 | |
| Sterile H2O | Gibco | 15230204 | |
| Sytox Green Nucleid Acid Stain | Thermo Fisher | S7020 | DNA dye |
| Trypan blue solution (0.4%) | Gibco | 15250-061 | |
| Tween-20 | Sigma Aldrich | P1379 | |
| Vacutainer blood tubes Li-Heparin (17 IU/mL) | BD | 367526 |
Referenzen
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- De Bont, C., Pruijn, G. J. M. Citrulline is not a major determinant of autoantibody reactivity to neutrophil extracellular traps. Philos Trans R Soc Lond B Biol Sci. 378 (1890), 20220249 (2023).
- Urban, C. F., et al. Neutrophil extracellular traps contain calprotectin, a cytosolic protein complex involved in host defense against Candida albicans. PLoS Pathog. 5 (10), e1000639 (2009).
- Hoppenbrouwers, T., et al. In vitro induction of NETosis: Comprehensive live imaging comparison and systematic review. PLoS One. 12 (5), e0176472 (2017).
- Kenny, E. F., et al. Diverse stimuli engage different neutrophil extracellular trap pathways. ELife. 6, e24437 (2017).
- Parker, H., Dragunow, M., Hampton, M. B., Kettle, A. J., Winterbourn, C. C. Requirements for NADPH oxidase and myeloperoxidase in neutrophil extracellular trap formation differ depending on the stimulus. J Leukoc Biol. 92 (4), 841-849 (2012).
- Neeli, I., Radic, M. Opposition between PKC isoforms regulates histone deimination and neutrophil extracellular chromatin release. Front Immunol. 4, 38 (2013).
- Bosmann, M., Ward, P. A. Protein-based therapies for acute lung injury: targeting neutrophil extracellular traps. Expert Opin Ther Targets. 18 (6), 703-714 (2014).
- Papayannopoulos, V. Neutrophil extracellular traps in immunity and disease. Nat Rev Immunol. 18 (2), 134-147 (2018).
- Shiratori-Aso, S., Nakazawa, D. The involvement of NETs in ANCA-associated vasculitis. Front Immunol. 14, 1261151 (2023).
- Byrd, A. S., et al. Neutrophil extracellular traps, B cells, and type I interferons contribute to immune dysregulation in hidradenitis suppurativa. Sci Transl Med. 11 (508), eaav5908 (2019).
- Kimball, A. S., Obi, A. T., Diaz, J. A., Henke, P. K. The emerging role of NETs in venous thrombosis and immunothrombosis. Front Immunol. 7, 236 (2016).
- O'Neil, L. J., et al. Neutrophil extracellular trap-associated carbamylation and histones trigger osteoclast formation in rheumatoid arthritis. Ann Rheum Dis. 82 (5), 630-638 (2023).
- Li, M., et al. A novel peptidylarginine deiminase 4 (PAD4) inhibitor BMS-P5 blocks formation of neutrophil extracellular traps and delays progression of multiple myeloma. Mol Cancer Ther. 19 (7), 1530-1538 (2020).
- Chirivi, R. G. S., et al. Anti-citrullinated protein antibodies as novel therapeutic drugs in rheumatoid arthritis. J Clin Cell Immunol. S6, 006 (2013).
- Van der Linden, M., et al. Anti-citrullinated histone monoclonal antibody CIT-013, a dual action therapeutic for neutrophil extracellular trap-associated autoimmune diseases. MAbs. 15 (1), 2281763 (2023).
- Chirivi, R. G. S., et al. Therapeutic ACPA inhibits NET formation: a potential therapy for neutrophil-mediated inflammatory diseases. Cell Mol Immunol. 18 (6), 1528-1544 (2021).
- Van der Linden, M., Westerlaken, G. H. A., Van Der Vlist, M., Van Montfrans, J., Meyaard, L. Differential signalling and kinetics of neutrophil extracellular trap release revealed by quantitative live imaging. Sci Rep. 7 (1), 6529 (2017).
- Hoffmann, J. H. O., Schaekel, K., Gaiser, M. R., Enk, A. H., Hadaschik, E. N. Interindividual variation of NETosis in healthy donors: introduction and application of a refined method for extracellular trap quantification. Exp Dermatol. 25 (11), 895-900 (2016).
- Silva, L. M., Moutsopoulos, N., Bugge, T. H., Doyle, A. Live imaging and quantification of neutrophil extracellular trap formation. Curr Protoc. 1 (7), e157 (2021).
- De Bont, C. M., Koopman, W. J. H., Boelens, W. C., Pruijn, G. J. M. Stimulus-dependent chromatin dynamics, citrullination, calcium signalling and ROS production during NET formation. Biochim Biophys Acta Mol Cell Res. 1865 (11 Pt A), 1621-1629 (2018).
- Gajendran, C., et al. Alleviation of arthritis through prevention of neutrophil extracellular traps by an orally available inhibitor of protein arginine deiminase 4. Sci Rep. 13 (1), 3189 (2023).
- Biron, B. M., Chung, C. -. S., O'Brien, X. M., Chen, Y., Reichner, J. S., Ayala, A. Cl-Amidine prevents histone 3 citrullination and neutrophil extracellular trap formation, and improves survival in a murine sepsis model. J Innate Immun. 9 (1), 22-32 (2017).
- Perdomo, J., et al. Neutrophil activation and NETosis are the major drivers of thrombosis in heparin-induced thrombocytopenia. Nat Commun. 10 (1), 1322 (2019).
- Lood, C., et al. Neutrophil extracellular traps enriched in oxidized mitochondrial DNA are interferogenic and contribute to lupus-like disease. Nat Med. 22 (2), 146-153 (2016).
- Kraaij, T., et al. A novel method for high-throughput detection and quantification of neutrophil extracellular traps reveals ROS-independent NET release with immune complexes. Autoimmun Rev. 15 (6), 577-584 (2016).
- De Buhr, N., Von Köckritz-Blickwede, M. How neutrophil extracellular traps become visible. J Immunol Res. 2016, 1-13 (2016).
- Brinkmann, V., Laube, B., Abu Abed, U., Goosmann, C., Zychlinsky, A. Neutrophil extracellular traps: How to generate and visualize them. J Vis Exp. (36), e1724 (2010).
- Zhao, W., Fogg, D. K., Kaplan, M. J. A novel image-based quantitative method for the characterization of NETosis. J Immunol Methods. 423, 104-110 (2015).
- Gavillet, M., et al. Flow cytometric assay for direct quantification of neutrophil extracellular traps in blood samples. Am J Hematol. 90 (12), 1155-1158 (2015).
- Sil, P., Yoo, D., Floyd, M., Gingerich, A., Rada, B. High throughput measurement of extracellular DNA release and quantitative NET formation in human neutrophils in vitro. J Vis Exp. (112), e52779 (2016).
- Gupta, S., Chan, D. W., Zaal, K. J., Kaplan, M. J. A high-throughput real-time imaging technique to quantify NETosis and distinguish mechanisms of cell death in human neutrophils. J Immunol. 200 (2), 869-879 (2018).
- D'Cruz, A. A., et al. The pseudokinase MLKL activates PAD4-dependent NET formation in necroptotic neutrophils. Sci Signal. 11 (546), eaao1716 (2018).
- Nash, G., Abbitt, K., Tate, K., Jetha, K., Egginton, S. Changes in the mechanical and adhesive behaviour of human neutrophils on cooling in vitro. Pflugers Arch. 442 (5), 762-770 (2001).
- Hundhammer, T., Gruber, M., Wittmann, S. Paralytic impact of centrifugation on human neutrophils. Biomedicines. 10 (11), 2896 (2022).
- Krémer, V., Godon, O., Bruhns, P., Jönsson, F., De Chaisemartin, L. Isolation methods determine human neutrophil responses after stimulation. Front Immunol. 14, 1301183 (2023).
- Freitas, M., Porto, G., Lima, J. L. F. C., Fernandes, E. Isolation and activation of human neutrophils in vitro. The importance of the anticoagulant used during blood collection. Clin Biochem. 41 (7-8), 570-575 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten