Method Article
Quantification microscopique en temps réel et à haut débit de la libération de pièges extracellulaires de neutrophiles humains et évaluation de la pharmacologie des antagonistes
Dans cet article
Résumé
Ce protocole définit une approche de microscopie à haut débit en temps réel pour visualiser et quantifier la libération de pièges extracellulaires (TNE) de neutrophiles humains in vitro. La méthode reproductible permet d’étudier les caractéristiques et la cinétique de la libération de TNE lors de la stimulation avec des inducteurs de NETosis distincts et permet d’évaluer la pharmacologie des antagonistes de NETosis.
Résumé
Les neutrophiles jouent un rôle important dans la défense immunitaire innée en utilisant plusieurs stratégies, notamment la libération de pièges extracellulaires (TNE) de neutrophiles dans un processus appelé NETosis. Cependant, au cours des deux dernières décennies, il est devenu clair que l’accumulation de TNE dans les tissus contribue à la physiopathologie de multiples maladies inflammatoires et auto-immunes. Par conséquent, l’intérêt pour le développement d’antagonistes de NETosis a augmenté. Des méthodes variables et non standardisées de détection et d’analyse de NETosis ont été développées en même temps, chacune avec ses propres avantages et limites. Ici, nous décrivons une méthode de microscopie en temps réel pour la quantification de la libération de TNE humaines, permettant d’étudier la NETosis ainsi que l’inhibition des TNE de manière à haut débit. L’analyse semi-automatisée basée sur la surface reconnaît les TNE et les distingue des neutrophiles activés sans filet. Nous démontrons que les inducteurs non physiologiques de NETosis, l’ionophore de calcium et le phorbol-12-myristate-13-acétate (PMA), déclenchent la libération de TNE ayant des caractéristiques et des cinétiques différentes. De plus, nous montrons que cette approche permet d’étudier la libération de TNE en réponse à des stimuli pertinents pour la maladie, notamment les complexes immunitaires, la N-formylméthionine-leucyl-phénylalanine (fMLF), les cristaux d’urate monosodique et les cristaux de pyrophosphate de calcium. Pour illustrer l’utilité de cette méthode pour étudier les antagonistes de NETosis, nous avons utilisé CIT-013, un inhibiteur d’anticorps monoclonal de première classe de la libération de NET. CIT-013 cible les histones H2A et H4 citrullinées et inhibe efficacement la libération de NET avec une IC50 de 4,6 nM. D’autres anticorps anti-histones testés n’avaient pas cette capacité inhibitrice de la NETose. Dans l’ensemble, nous démontrons que ce protocole permet une quantification spécifique, fiable et reproductible à haut débit des TNE, améliorant ainsi l’étude des caractéristiques de libération des TNE, de la cinétique et de la pharmacologie des antagonistes de la NETosis.
Introduction
Les neutrophiles sont abondamment présents dans le sang et migrent vers les tissus lors d’une infection ou d’une inflammation. Ils jouent un rôle important dans la défense immunitaire innée en utilisant un armement étendu pour protéger l’hôte contre les microbes. Les neutrophiles tuent les agents pathogènes par phagocytose, dégranulation, génération d’espèces réactives de l’oxygène (ROS) et libération de chromatine décondensée appelée pièges extracellulaires de neutrophiles (TNE) par un processus appelé NETosis1. Les TNE sont des structures extracellulaires de la chromatine décorées, entre autres, de protéines granulaires et de calprotectine 2,3, et sont libérées lors de la stimulation avec un large spectre de molécules4. La NETosis peut être largement classée en deux voies principales : NADPH oxydase dépendante ou indépendante 5,6,7. De plus, la citrullination de l’arginine des queues d’histones N-terminales par la peptidyl arginine déiminase 4 (PAD4) a été étroitement liée à la NETosis et favorise la décondensation de la chromatine, ce qui conduit finalement à l’expulsion de la chromatine décondensée dans l’environnement extracellulaire.
Bien que la libération de TNE soit impliquée dans l’élimination des agents pathogènes, de nombreuses études ont montré que la libération anormale et prolongée de TNE est liée au développement de divers troubles inflammatoires, notamment les lésions pulmonaires aiguës8, la polyarthrite rhumatoïde (PR)9, la vascularite10 et l’hidradénite suppurée11. Le rôle néfaste des TNE dans la maladie est multiforme car les TNE sont pro-inflammatoires, sont une source d’auto-antigènes, sont cytotoxiques pour les tissus environnants, déclenchent l’immunothrombose et favorisent la différenciation des ostéoclastes et l’érosion osseuse 9,12,13. L’inhibition pharmacologique de la voie NETosis par de petites molécules inhibitrices de PAD4 démontre que les thérapies ciblant la NETose ont un potentiel en tant que traitements pour les maladies dans lesquelles l’accumulation de TNE est un moteur important de la pathogenèse14. Au lieu de cibler l’enzyme PAD4, nous avons utilisé un anticorps monoclonal humanisé inhibiteur de NETosis inhibiteur d’histone humanisé, CIT-013, qui se lie spécifiquement aux histones citrullinées H2A et H415. Le CIT-013 a un double mécanisme d’action unique en inhibant la libération de TNE et en renforçant la phagocytose des TNE médiée par les macrophages16. Le CIT-013 et les molécules précurseurs ont montré une efficacité thérapeutique dans plusieurs modèles murins d’inflammation associée aux TNE17.
Pour étudier la libération de NET, différentes méthodes ont été développées au fil des ans, telles que, mais sans s’y limiter, 1) la détection de l’ADN à l’aide d’un traceur d’ADN imperméable à la membrane plasmique en combinaison avec un lecteur de plaque immunofluorescente, 2) la détection basée sur l’essai immuno-enzymatique (ELISA) de l’ADN et de l’ADN complexé avec des protéines spécifiques de NET chez les surnageants, 3) la co-localisation des molécules associées aux TNE avec l’ADN extracellulaire via l’immunohistochimie, et 4) approches de cytométrie en flux pour détecter les neutrophiles en filet. Toutes ces méthodes ont leurs propres avantages et limites. Nous avons développé une approche à haut débit en temps réel pour la quantification microscopique de la libération de TNE humaines, qui utilise un colorant d’ADN imperméable à la membrane plasmique16,18. La méthode décrite permet d’étudier la cinétique et les caractéristiques de NETosis de manière simple, fiable et reproductible et permet d’évaluer la pharmacologie des antagonistes de NETosis tels que CIT-013.
Protocole
Tous les donneurs de sang ont donné leur consentement éclairé conformément à la Déclaration d’Helsinki, et l’étude a été réalisée conformément aux directives éthiques de Citryll pour la recherche sur l’homme.
REMARQUE : Toutes les activités avec du sang humain et des neutrophiles isolés doivent être effectuées dans des conditions stériles dans une armoire à flux laminaire. Lorsque les paramètres de freinage et d’accélération pour la centrifugation ne sont pas mentionnés dans le protocole, ils peuvent être pris en compte au maximum.
1. Isolement des neutrophiles dans le sang
- Prélevez le sang périphérique de volontaires sains dans des tubes lithium-héparine et transférez le sang dans un tube frais de 50 ml.
- Rincer les tubes au lithium-héparine avec 1 DPBS et les transférer dans le même tube de 50 ml où le sang a été transféré. Assurez-vous que le rapport sanguin/DPBS final est de 1:1. Mélangez le sang et le DPBS par pipetage pour obtenir une solution homogène.
- Ajouter 13 mL de solution à gradient de densité dans un autre tube frais de 50 mL et ajouter lentement (avec une pipette de 25 mL) le sang dilué 1:1 sur la solution à gradient de densité jusqu’à un maximum de 50 mL.
- Centrifugeuse à 400 x g et température ambiante (RT) pendant 40 min avec une accélération et un freinage minimaux.
REMARQUE : Des couches se forment. Les couches de haut en bas sont les suivantes : 1. plasma ; 2. les cellules mononucléées du sang périphérique (PBMC) ; 3. solution de gradient de densité ; 4. Érythrocytes/neutrophiles. - Tout d’abord, jetez le plasma à l’aide d’une pipette de 10 ml, puis jetez les PBMC et la couche de solution à gradient de densité à l’aide d’une pipette Pasteur en plastique autant que possible sans perturber la couche érythrocytaire ou neutrophile.
- Remettez doucement la couche érythrocytaire/neutrophile en suspension en agitant et en suspendant à nouveau avec une pipette de 25 ml dans 15 mL de 1x DPBS.
- Ajouter 25 ml de solution de Dextran à 6 % et de NaCl à 0,9 %, mélanger dix fois en inversant le tube et placer le tube à la verticale pendant 25 minutes à RT.
REMARQUE : Des couches se forment. Les couches de haut en bas sont les suivantes : 1. Neutrophiles ; 2 érythrocytes. - Transférez la couche de neutrophiles dans un tube frais de 50 mL à l’aide d’une pipette de 10 mL et centrifugez pendant 10 min à 500 x g et RT.
REMARQUE : Soyez prudent car la pastille de neutrophiles contient encore des érythrocytes et n’est pas fermement attachée au tube. - Jeter le surnageant par décantation et remettre en suspension la pastille cellulaire à l’aide d’une pipette de 10 mL dans 10 mL de tampon de lyse d’ammonium-chlorure-potassium (ACK) (155 mM de NH4Cl, 10 mM de KHCO3 et 0,1 mM de Na2EDTA ; pH = 7,2), puis ajouter immédiatement 40 mL de tampon de lyse ACK.
- Incuber à RT tout en inversant continuellement le tube jusqu’à ce que la solution devienne translucide (cela prend 1 à 5 minutes et peut varier pour chaque donneur) et centrifuger pendant 10 min à 350 x g et RT.
- Retirer le surnageant et ajouter lentement et goutte à goutte 5 mL de milieu de culture contenant de la L-glutamine complétée par 10 % (v/v) de sérum fœtal bovin inactivé par la chaleur, 50 U/mL de pénicilline et 50 μg/mL de streptomycine (ci-après appelé milieu de culture à 10 %) sur la pastille de neutrophiles sans mettre les neutrophiles en suspension. Cela éliminera efficacement la plupart des érythrocytes de la pastille de neutrophiles.
- Agiter doucement le tube de 50 mL jusqu’à ce que la plupart des érythrocytes, présents sur la pastille de neutrophiles, soient remis en suspension dans le milieu de culture à 10 %. Par la suite, retirez le surnageant en transvasant le tube.
- Remettre la pastille de neutrophiles en suspension dans 10 mL de milieu de culture à 10 % et, lorsqu’elle est complètement remise en suspension, ajouter jusqu’à 50 mL de milieu de culture à 10 %. Centrifugeuse pendant 10 min à 350 x g et RT.
- Retirer le surnageant et remettre en suspension la pastille cellulaire dans 10 mL de milieu de culture à 10 %.
2. Coloration des neutrophiles pour vérifier la pureté par cytométrie en flux
- Pour déterminer la concentration cellulaire, diluez la suspension de neutrophiles dans une solution de bleu de trypan à 0,4 % (rapport 1:1) et comptez les neutrophiles à l’aide d’un compteur de cellules à champ clair.
REMARQUE : D’autres méthodes de comptage peuvent également être utilisées. - Transvaser 1 x 105 neutrophiles dans un milieu de culture à 10 % dans les puits d’une plaque à fond en V à 96 puits et centrifuger pendant 3 min à 400 x g et RT.
- Jeter le surnageant et remettre les cellules en suspension dans 50 μL de 1x DPBS complété par 1 % (p/v) d’albumine sérique bovine (BSA) et 0,1 % (v/v) de NaN3 (appelé tampon de tri cellulaire activé par fluorescence (FACS) ci-après) contenant un bloc récepteur Fc (50x dilué).
- Incuber pendant 15 min à RT et ajouter 50 μL de tampon FACS contenant 50 anticorps anti-CD45 humains de souris conjugués APC-Cy7 dilués, 600 anticorps anti-CD16 humains de souris conjugués PerCP-Cy5.5 dilués, 133 anticorps anti-CD66b humains de souris conjugués FITC dilués et 500 fois le colorant de viabilité fixable dilué eFluor 506.
REMARQUE : Le CD45 est exprimé sur les leucocytes. CD66b est exprimé exclusivement dans les granulocytes. CD16 est fortement exprimé chez les neutrophiles, faiblement exprimé chez les éosinophiles et non chez les basophiles. - Incuber pendant 30 min à RT dans l’obscurité et centrifuger pendant 3 min à 400 x g et RT.
- Jeter le surnageant, remettre les neutrophiles en suspension dans 175 μL de tampon FACS et centrifuger pendant 3 min à 400 x g et RT. Répétez cette étape une fois.
- Jeter le surnageant et remettre en suspension la pastille de neutrophiles dans 175 μL de tampon FACS. Analysez des échantillons à l’aide d’un système de cytométrie en flux et des logiciels associés.
3. Analyse de la pureté des neutrophiles à l’aide d’un logiciel d’analyse par cytométrie en flux
REMARQUE : L’analyse des données de cytométrie en flux a été effectuée à l’aide du logiciel d’analyse de cytométrie en flux, comme indiqué dans la table des matériaux.
- Effectuez le contrôle selon la procédure étape par étape suivante.
- Définissez une porte temporelle dans le graphique FSC-A en fonction du temps pour sélectionner une zone avec un flux de cellules approprié.
- Définissez une porte de cellule dans le tracé SSC-A vs. FSC-A pour sélectionner les cellules et exclure les débris.
- Définissez une porte de cellule viable dans le graphique FSC-A par rapport à AmCyan-A pour sélectionner les cellules viables.
- Définissez une porte leucocytaire dans le graphique FSC-A par rapport à APC-Cy7 pour sélectionner les cellules CD45+.
- Définissez une porte neutrophile dans le graphique FITC vs. PerCP-Cy5.5 pour distinguer CD66b+CD16+ (neutrophiles), CD66b-CD16+ (éosinophiles) et CD66b-CD16- (basophiles, monocytes et lymphocytes).
- Définissez une porte de cellule unique dans le graphique FSC-H vs. FSC-A pour sélectionner des neutrophiles uniques.
- Déterminez la pureté des neutrophiles en présentant les neutrophiles uniques comme la fréquence des cellules (dans la porte cellulaire).
REMARQUE : Pour continuer, la pureté des neutrophiles doit dépasser 85 %.
4. Microscopie à imagerie en direct
REMARQUE : Ce test est optimisé pour plusieurs plaques d’imagerie à 96 puits et divers stimuli et antagonistes de NETosis. Le protocole ci-dessous décrit une vue générale de l’approche, qui peut être spécifiée à l’aide des tableaux inclus.
- Préparez une solution de poly-L-lysine à 0,001 % en diluant la poly-L-lysine à 0,01 % dans duH2O stérile dans un rapport de 1:10.
- Ajouter une solution de poly-L-lysine à 0,001 % dans chaque puits et incuber au moins 1 h à 37 °C.
REMARQUE : Le volume par puits dépend de la plaque d’imagerie à 96 puits (Tableau 1). - Lavez les puits 3 fois avec 200 μL de DPBS pour éliminer l’excès de poly-L-lysine. Effectuez toutes les étapes dans l’armoire à flux laminaire. Retirez le couvercle de la plaque et faites sécher les puits à l’air libre en ouvrant la plaque dans l’armoire à flux laminaire jusqu’à ce qu’elle soit sèche (environ 1 h).
- Calculez le nombre de neutrophiles requis pour l’expérience et transférez le surplus dans un tube de 15 ml. Centrifugeuse pendant 10 min à 350 x g et RT.
REMARQUE : La quantité de neutrophiles dans chaque puits varie en fonction du type de plaque d’imagerie à 96 puits (tableau 1). La densité cellulaire est optimisée pour permettre une bonne séparation des cellules individuelles, ce qui est nécessaire à une analyse correcte. Si la densité de neutrophiles est trop élevée, les cellules et les TNE se chevaucheront avec les cellules et les TNE adjacentes, ce qui affecte la qualité de l’analyse. - Jeter le surnageant et remettre les neutrophiles en suspension dans un milieu de culture sans rouge de phénol complété par 2 % (v/v) de FBS, 50 U/mL de pénicilline et 50 μg/mL de streptomycine, 10 mM d’HEPES et 1 mM de CaCl2 (ci-après dénommé tampon d’essai NET).
- Préparez les solutions de travail suivantes.
- Préparez un colorant d’ADN de 80 nM dans un tampon de test NET.
- Préparez les stimuli NETosis dans le tampon d’essai NET.
- Préparer les antagonistes de NETosis dans le tampon de test NET.
REMARQUE : Sachez que les concentrations des solutions de travail décrites ci-dessus sont 4 fois supérieures à la concentration finale nécessaire dans le puits. Les concentrations recommandées de stimuli et d’antagonistes de NETosis varient (tableau 2 et tableau 3).
- Ajouter 4 fois plus de colorant d’ADN concentré dans un tampon de test NET dans chaque puits.
- Ajouter 4 stimuli concentrés de NETosis dans le tampon de test NET aux puits correspondants, et ajouter le tampon de test NET uniquement aux puits sans stimulus ou aux puits contenant des complexes immuns enrobés (cIC ; protocole dans le tableau 2).
- Ajouter 4 antagonistes concentrés de NETosis dans le tampon de test NET aux puits correspondants. Ajoutez le tampon de test NET uniquement aux puits sans antagoniste.
- Ajouter une suspension de neutrophiles dans chaque puits.
REMARQUE : Le volume de 4 colorants d’ADN concentrés, de 4 stimuli NETosis concentrés dans le tampon de test de TNE, de 4 x antagonistes de NETosis concentrés dans le tampon de test de TNE, le nombre de neutrophiles et le volume de suspension de neutrophiles par puits dépendent de la plaque d’imagerie à 96 puits (Tableau 1). Évitez la formation de bulles lors du pipetage. - Centrifuger la plaque pendant 2 min à 100 x g et RT et insérer la plaque d’imagerie à 96 puits dans le système d’analyse par microscopie à cellules vivantes placé dans un incubateur à 37 °C et 5 % de CO2.
REMARQUE : De la condensation peut se développer au bas de la plaque d’imagerie à 96 puits pendant la première minute d’incubation. Celui-ci doit être enlevé avec un mouchoir.
5. Réglage du logiciel du système d’analyse de microscopie à cellules vivantes pour l’acquisition
REMARQUE : Les images de contraste de phase et d’immunofluorescence ont été acquises par un système d’analyse de microscopie de cellules vivantes contrôlé par son logiciel d’analyse.
- Ouvrez le logiciel du système d’analyse de microscopie à cellules vivantes et cliquez sur Connecter à l’appareil. Entrez le nom d’utilisateur et le mot de passe. Cliquez sur Planifier l’acquisition et cliquez sur le bouton plus (Lancer ajouter un navire). Ensuite, sélectionnez Analyser selon un calendrier et cliquez sur Suivant.
- Pour créer un nouveau navire à partir de zéro, sélectionnez Nouveau dans la section Créer un navire et cliquez sur Suivant. Sélectionnez Standard dans la section Type de balayage et cliquez sur Suivant.
- Sélectionnez les paramètres d’analyse suivants et cliquez sur Suivant : Cellule par cellule : Aucune ; Canaux d’image : Contraste de phase et Vert (temps d’acquisition 100 ms) ; Objectif : 20x.
- Sélectionnez le type de navire approprié à scanner dans la section Sélection du navire et cliquez sur Suivant.
- Si vous utilisez une plaque #1 à 96 puits, sélectionnez Corning, plaque, 96, N/A, 3603, Corning 96 puits (Blk/Wht), Microplaques.
- Si vous utilisez une plaque à 96 puits #2, sélectionnez Nunc, Plaque, 96, N/A, 152028, Nunc opt bottom à 96 puits (Blk/Wht), Microplaques.
- Spécifiez l’emplacement du récipient dans le tiroir et cliquez sur Suivant. Dans la section Modèle de balayage , sélectionnez les puits qui doivent être numérisés, sélectionnez le nombre d’images par puits (numérisez 4 images par puits pour obtenir une vue d’ensemble représentative) et cliquez sur Suivant.
REMARQUE : Ne sélectionnez pas de puits vides car cela affecterait l’autofocus. - Fournissez des informations sur le navire en insérant, entre autres, le nom de la plaque dans la section du carnet du navire et cliquez sur Suivant. Sélectionnez Différer l’analyse à plus tard dans la section Configuration de l’analyse et cliquez sur Suivant.
- Définissez le calendrier de balayage du navire dans la section Calendrier de balayage . Sélectionnez Créer un nouveau calendrier avec des analyses à intervalles de et sélectionnez 1 heure. Sélectionnez Arrêter l’analyse et 00:05 heures après la première analyse. Cliquez sur Suivant , puis sur Ajouter à la planification lorsque les informations de numérisation sont correctes.
- Lorsque le calendrier d’analyse doit être modifié, double-cliquez sur le calendrier d’analyse en haut de l’écran. Faites un clic droit sur le calendrier de numérisation et modifiez-le et ajustez-le comme vous le souhaitez. Cliquez sur l’icône de la disquette pour enregistrer le calendrier d’analyse.
REMARQUE : Lors de l’imagerie de plusieurs récipients (plaques) dans la même expérience, des récipients supplémentaires peuvent être ajoutés au programme de balayage existant en cliquant sur le bouton plus (Lancer l’ajout d’un récipient), en sélectionnant Scanner selon le calendrier, puis en cliquant sur Suivant. Créez un nouveau navire à partir de zéro (voir l’étape 5.2 de ce protocole), copiez un navire existant ou utilisez un navire précédemment scanné. Il en résulte un balayage séquentiel à la même fréquence et aux mêmes paramètres que la plaque précédente.
- Lorsque le calendrier d’analyse doit être modifié, double-cliquez sur le calendrier d’analyse en haut de l’écran. Faites un clic droit sur le calendrier de numérisation et modifiez-le et ajustez-le comme vous le souhaitez. Cliquez sur l’icône de la disquette pour enregistrer le calendrier d’analyse.
6. Analyse du test NET de microscopie à imagerie en direct à l’aide du logiciel d’analyse de microscopie à cellules vivantes
REMARQUE : Un logiciel d’analyse par microscopie à cellules vivantes a été utilisé pour analyser les images de contraste de phase et d’immunofluorescence (tableau des matériaux). Lorsque vous n’utilisez pas ce système, une analyse NET similaire peut être effectuée à l’aide d’un progiciel du domaine public 4,18,19,20.
- Ouvrez le logiciel d’analyse de microscopie à cellules vivantes et cliquez sur Connecter à l’appareil. Entrez le nom d’utilisateur et le mot de passe , puis cliquez sur Afficher les analyses récentes. Ouvrez l’expérience de votre choix à analyser en double-cliquant sur le nom du récipient de l’expérience.
- Cliquez sur Lancer l’analyse et sélectionnez Créer une définition d’analyse. Cliquez sur Suivant.
- Sélectionnez Analyseur de base dans la section du type d’analyse et cliquez sur Suivant. Dans la section Couche d’image , sélectionnez Vert, désélectionnez Phase, puis cliquez sur Suivant.
- Ouvrez la fenêtre Calques d’image, sélectionnez Vert et désélectionnez Phase. Désactivez la mise à l’échelle automatique dans la section Vert et définissez manuellement min et max pour le canal vert afin de distinguer les NET de l’arrière-plan.
REMARQUE : Le contraste de phase peut être activé et désactivé si nécessaire. Par exemple, pour déterminer si les TNE sont expulsées dans l’environnement extracellulaire. - Ouvrez la fenêtre Heures de balayage du navire et sélectionnez l’heure de balayage appropriée pendant laquelle les TNE doivent être présentes. Sélectionnez un ensemble d’images représentant la variabilité au sein de l’expérience (images de puits de contrôle positifs avec TNE et images de puits de contrôle négatifs sans TNE). Ces images seront utilisées pour prévisualiser et affiner l’analyse. Cliquez sur Suivant.
- Ouvrez la fenêtre Paramètres de définition d’analyse et commencez à ajuster les paramètres d’analyse du canal vert de manière à ce que les structures NET soient décrites sans créer de NET faussement positifs ou faussement négatifs. Utilisez les paramètres suivants et cliquez sur Aperçu actuel ou Aperçu tout pour appliquer les paramètres à l’image actuelle ou à toutes les images, respectivement.
- Pour Nom de l’objet, sélectionnez TNE.
- Pour la segmentation, sélectionnez Aucune soustraction d’arrière-plan - Adaptative.
- Pour la GCU de seuil, sélectionnez entre 3 et 5 (selon l’expérience).
- Pour la division des bords, sélectionnez ON (entre -10 et 0).
- Pour le nettoyage, sélectionnez Remplissage du trou : 100 μm2 et Ajuster la taille : -1 pixels.
- Pour les filtres, sélectionnez en fonction de l’expérience, mais les valeurs seuils recommandées sont les suivantes : Aire : >200 μm2, Intensité moyenne : <24,6, Intensité intégrée : >7000.
REMARQUE : Les structures sélectionnées en fonction des paramètres utilisés sont encadrées de magenta. En survolant ces structures, vous obtiendrez plus d’informations sur les paramètres des objets (c’est-à-dire l’aire, l’excentricité, l’intensité intégrée et l’intensité moyenne), ce qui permet d’optimiser l’analyse liée aux critères d’inclusion ou d’exclusion des paramètres NET. Vérifiez plusieurs images (à partir de puits de contrôle positifs et négatifs) pour vérifier si les paramètres d’analyse sont corrects.
- Lorsque les paramètres de l’analyse NET sont corrects, cliquez sur Suivant. Sélectionnez les points temporels et les puits à analyser dans la section Temps et puits de balayage, puis cliquez sur Suivant. Insérez le nom de la définition dans la section Enregistrer et appliquer la définition de l’analyse, puis cliquez sur Suivant. Cliquez sur Terminer lorsque les informations d’analyse sont vérifiées et correctes.
REMARQUE : Lors de l’analyse de plusieurs plaques ou de l’utilisation d’analyses d’expériences précédentes, lancez l’analyse et sélectionnez Copier la définition d’analyse existante ou Utiliser la définition d’analyse existante et cliquez sur Suivant. Sélectionnez la définition d’analyse existante appropriée et cliquez sur Suivant. Pour la procédure d’analyse suivante, passez à l’étape 6.4 si l’option Copier la définition d’analyse existante a été sélectionnée, ou à l’étape 6.7 lorsque l’option Utiliser la définition d’analyse existante a été sélectionnée. - Ouvrez l’analyse de l’expérience en cliquant sur l’analyse dans le récipient qui vous intéresse. Ouvrez la fenêtre Métriques du graphique .
- Cliquez sur le bouton plus (Créer une mesure). Les données peuvent être présentées en pourcentage de confluence des TNE (le pourcentage de la zone de l’image marquée comme NET) ou en pourcentage de neutrophiles de la MNE (nombre de TNE divisé par le nombre de cellules).
- Lorsque vous présentez des données sous forme de pourcentage de confluence NET, sélectionnez Zone dans la section Métrique et sélectionnez Confluence dans la section Valeur . Lorsque vous présentez des données sous forme de pourcentage de neutrophiles nets, sélectionnez Nombre d’objets dans la section Métrique et Par image dans la section Valeur .
- Cliquez sur OK. Sélectionnez Confluence de zone NETs ( %) ou Nombre d’objets NETs par image (par image) dans la section Métriques définies par l’utilisateur . Sélectionnez tous les points temporels qui doivent être analysés dans la section Sélectionner les numérisations . Sélectionnez tous les puits qui doivent être analysés dans la section Sélectionner des puits et cliquez sur Exporter les données.
- Sélectionnez les paramètres suivants dans la section Exportation graphique .
- Sélectionnez Afficher chaque scan comme son propre tableau (colonnes : 1, 2... rangées : A, B...).
- Sélectionnez Afficher les libellés de ligne et de colonne.
- Sélectionnez Toutes les numérisations dans un seul fichier et cliquez sur Parcourir pour ajuster l’emplacement et le nom du fichier.
- Sélectionnez Inclure les détails du test dans l’en-tête.
- Sélectionnez Décomposer les données en images individuelles.
- Cliquez sur Exporter pour exporter les données sous forme de fichier « .txt », qui peut être importé et analysé plus en détail dans une feuille de calcul.
REMARQUE : Pour déterminer le pourcentage de neutrophiles en filet, il faut déterminer le nombre de neutrophiles à t = 0. Pour ce faire, passez aux étapes suivantes du protocole. - Passez à l’étape 6.1 pour lancer l’analyse supplémentaire nécessaire pour compter le nombre de neutrophiles pour les images à t = 0.
- À l’étape 6.3, sélectionnez Phase et désélectionnez Vert dans la section du canal de l’image.
- À l’étape 6.4, sélectionnez t = 0 dans la section Temps de balayage du navire.
- À l’étape 6.5, désélectionnez Vert, sélectionnez Phase et ajustez les paramètres du canal de phase si nécessaire.
- À l’étape 6.6, ajoutez le nom de l’objet, sélectionnez Confluence classique dans la section Segmentation et réglez le réglage de la segmentation sur 0 (arrière-plan), ajustez le remplissage des trous à 100 μm2 et ajustez la zone à 50 μm2.
- À l’étape 6.7, sélectionnez t = 0 dans la section de sélection des analyses.
- À l’étape 6.8, ouvrez l’analyse de la numération des neutrophiles.
- À l’étape 6.9, sélectionnez Nombre d’objets par image (par image) dans la section Mesures définies par l’utilisateur .
- Enfin, utilisez le calcul suivant pour générer le pourcentage de neutrophiles nets : (nombre de TNE / nombre de neutrophiles à t = 0) x 100 %.
Résultats
La microscopie à haut débit en temps réel, associée à un colorant d’ADN imperméable, permet d’étudier la cinétique, les caractéristiques et les voies sous-jacentes de NETosis et permet d’évaluer les inhibiteurs potentiels de la libération de NET. Avec cette approche, les TNE ont été définies comme des structures positives pour le colorant d’ADN avec une surface significativement plus grande par rapport à la surface des neutrophiles sains (Figure 1A), indiquant que la chromatine a été expulsée dans l’environnement extracellulaire18. L’analyse basée sur la surface nous a permis de distinguer les TNE des neutrophiles activés dont l’intégrité de la membrane plasmique est compromise, montrant une coloration brillante de l’ADN intracellulaire (Figure 1A).
L’ionophore calcique (A23187) et le PMA sont couramment utilisés pour induire la NETosis in vitro. Bien qu’il s’agisse de stimuli non physiologiques, ils sont précieux car ils activent des voies distinctes de NETosis et assurent une induction cohérente de NETosis avec une faible variabilité entre les donneurs. A23187 déclenche un afflux de calcium, entraînant l’activation de PAD4 et la libération de TNE riches en histones citrullinées, tandis que la PMA active le complexe NADPH oxydase, entraînant la production d’espèces réactives de l’oxygène (ROS) et la libération ultérieure de TNE avec de faibles niveaux d’histones citrullinées 5,16,21. La vitesse et l’amplitude de la réponse NETosis dépendent de la concentration de chaque stimulus (Figure 1B,C), A23187 induisant une NETosis plus rapide, et PMA entraînant une proportion plus élevée de neutrophiles libérant des TNE (Figure 1D-F). Les TNE résultant de la stimulation A23187 (figure 1D ; flèche rouge) se distinguaient des TNE induites par la PMA (figure 1D ; flèche jaune) en ce qu’elles étaient plus diffuses au-delà de la membrane plasmique des neutrophiles, tandis que les TNE induites par la PMA restaient plus adjacentes à la membrane plasmique des neutrophiles. De plus, la stimulation A23187 a donné des neutrophiles perméables non nettoyants à l’ADN positif (Figure 1D ; flèche bleue), qui n’ont pas expulsé leur ADN dans l’espace extracellulaire. La détection de neutrophiles à ADN non nettoyants perméables et positifs à un colorant était dépendante de la concentration de A23187 (figure 1G) et presque absente quelle que soit la concentration de PMA utilisée pour stimuler la NETosis (figures 1D, H). En plus d’utiliser les stimuli non physiologiques NETosis A23187 et PMA, ce test convient également à l’étude des déclencheurs de NETosis pertinents pour la maladie. À titre d’exemple, les neutrophiles activés par des complexes immunitaires solubles (sIC) ou des cristaux présents dans la maladie de disposition du pyrophosphate de calcium (CPPD) ont montré une augmentation significative de la libération de TNE par rapport à l’absence de stimulus. Une tendance à des taux élevés de TNE a été observée lorsque les neutrophiles étaient activés avec des complexes immuns enrobés (cIC), du fMLP et des cristaux d’urate monosodique (MSU) (figure 1I) ; Cependant, pour ces stimuli, nous avons observé une variation considérable d’un donneur à l’autre.
L’inhibition pharmacologique de la voie NETosis et des enzymes associées à la NETosis a montré que les thérapies ciblant la NETosis pourraient être des traitements efficaces pour les maladies où l’accumulation de TNE entraîne de manière significative la pathologie 14,17,22,23,24. Ce test NET de microscopie en temps réel est une approche simple, fiable et reproductible pour étudier les antagonistes de NETosis à haut débit. Pour illustrer cela, nous avons utilisé un anticorps monoclonal humanisé de première classe, CIT-013, qui cible les histones citrullinées H2A et H4 avec une grande affinité15,16. A23187 active la voie NETosis, ce qui permet aux TNE de contenir des histones citrullinées qui sont ensuite ciblées par CIT-013 pour remplir leur fonction inhibitrice de NETosis16. En effet, la libération de TNE en réponse à A23187 a été complètement inhibée par CIT-013 (Figure 2A et Vidéo supplémentaire 1), avec une CI50 de 4,6 nM (Figure 2B). La capacité inhibitrice de la NETose de CIT-013 est unique car d’autres anticorps commerciaux ciblant différentes histones (non-)citrullinées n’ont pas été en mesure d’inhiber la libération de TNE (Figure 2C).
Auparavant, nous avons montré qu’une molécule précurseur très similaire de CIT-013 (deux acides aminés différents mais liant des épitopes similaires avec une affinité égale) bloque NETosis en réponse à des stimuli physiologiques tels que les plaquettes activées, le liquide synovial de la goutte ainsi que le liquide synovial de la RA17. Ici, nous montrons que la libération de TNE induite par le sIC peut être inhibée par CIT-013 (Figure 2D). La pertinence thérapeutique de l’inhibition de la NETosis induite par ce stimulus est mise en évidence par le LED, la PR et d’autres maladies auto-immunes, où les auto-anticorps dans le sérum ou le liquide synovial soutiennent la formation de la CI, qui déclenche la NETosis25,26.
L’ensemble de ces données démontre que cette approche de microscopie à haut débit en temps réel est adaptée à l’étude de la cinétique et des caractéristiques de la libération de TNE et permet l’étude des inhibiteurs de la NETosis. Bien que cette méthode ait été optimisée pour l’utilisation de neutrophiles humains, avec des modifications, elle pourrait également convenir pour étudier les neutrophiles d’autres espèces. Les données générées par ce test sont la pierre angulaire de la raison d’être du CIT-013 en tant que traitement puissant et efficace pour les maladies induites par les TNE.
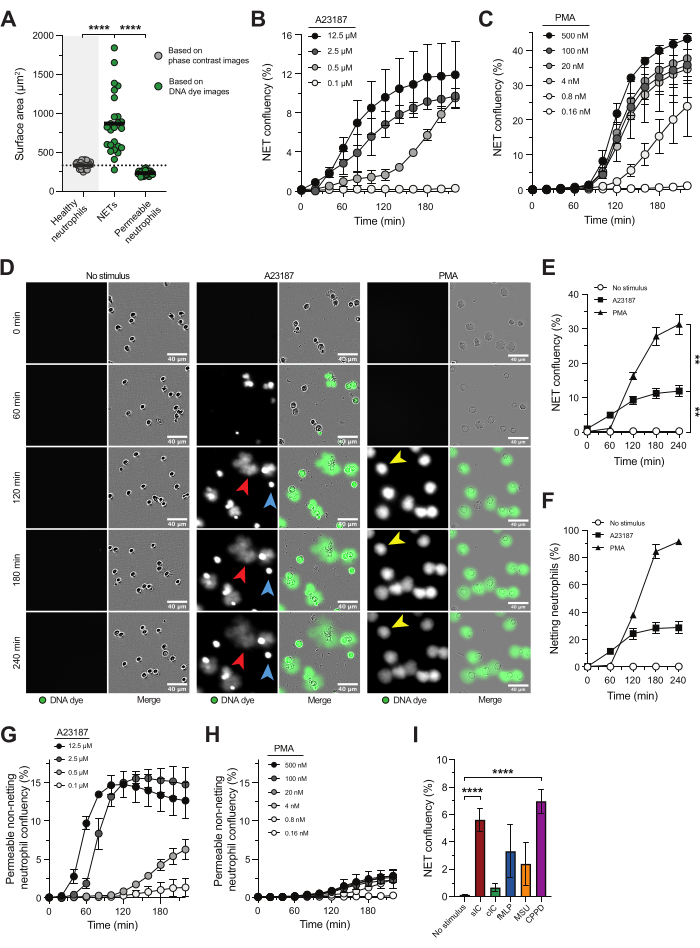
Figure 1 : Microscopie à haut débit en temps réel pour l’étude de la libération de TNE. Les neutrophiles isolés du sang de volontaires sains ont été stimulés avec A23187 ou PMA pour déclencher la voie NETosis. La libération de TNE a été visualisée par microscopie à haut débit en temps réel à l’aide d’un colorant d’ADN imperméable à la membrane plasmique et quantifiée en fonction de la surface avec le logiciel du système d’analyse de microscopie à cellules vivantes. (A) Analyse de la surface des neutrophiles sains non stimulés (gris), des TNE extracellulaires et des neutrophiles perméables non netsants avec coloration intracellulaire de l’ADN (vert). (B, C) Quantification de la libération de TNE au fil du temps par les neutrophiles stimulés par les concentrations indiquées A23187 ou PMA (n = 2). (D) Images représentatives du rejet de TNE en réponse à A23187 (flèches rouges) et PMA (flèches jaunes) à différents moments. Des exemples de neutrophiles perméables non réticulaires sont indiqués par des flèches bleues. (E) Quantification de la libération de TNE au fil du temps présentée en pourcentage de la confluence des TNE (n = 5). Les statistiques ont été effectuées sur t = 240 min. (F) Quantification de la libération de TNE au fil du temps présentée en pourcentage des neutrophiles nets (n = 2). (G, H) Quantification des neutrophiles non résilles perméables au fil du temps à partir de neutrophiles qui ont été stimulés avec les concentrations indiquées de A23187 ou de PMA (n = 2). (I) Quantification de la libération de TNE à t = 240 min induite par des complexes immuns solubles (sIC), des CI enrobés (cIC), des fMLP, des cristaux d’urate monosodique (MSU) et des cristaux présents dans la maladie de disposition du pyrophosphate de calcium (CPPD) (n = 8-28). Les résultats sont présentés sous forme de moyenne ±'erreur type de la moyenne. **P < 0,01 et ****P < 0,0001, ANOVA à mesures répétées à un facteur avec le test de comparaisons multiples de Dunnett (B), test de Kruskal-Wallis avec le test de comparaisons multiples de Dunn (I). Les panneaux A-F ont été modifiés avec l’autorisation de van der Linden et al.16. Veuillez cliquer ici pour voir une version agrandie de cette figure.
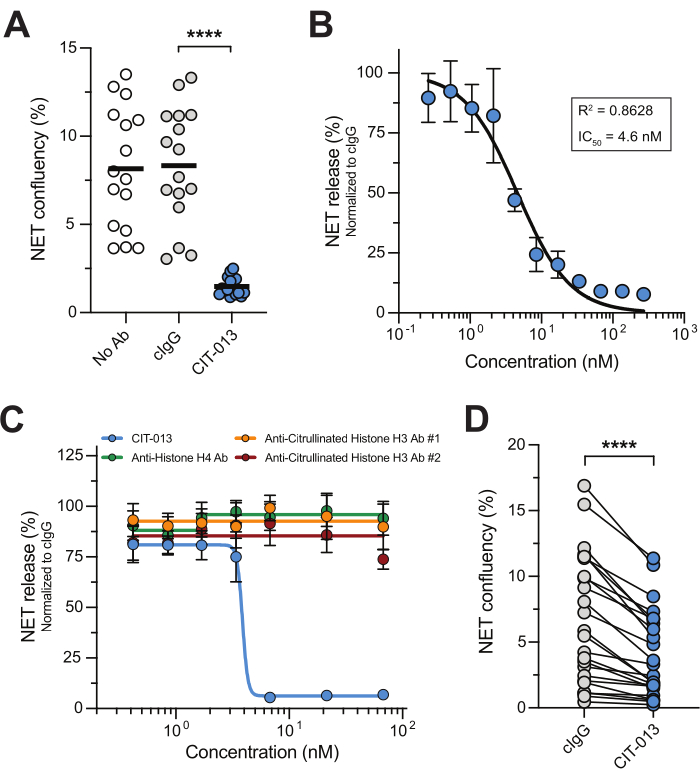
Figure 2 : CIT-013 inhibe la libération de NET. (A) Quantification de la libération de TNE induite par A23187 à t = 240 min en l’absence (pas d’Ab) ou la présence de CIT-013 ou d’anticorps de contrôle d’isotype (cIgG). (B) Inhibition dose-dépendante de la libération de TNE induite par A23187 avec CIT-013 à t = 240 min (n = 3). Les données ont été normalisées en cIgG (définie à 100 % de libération de TNE). (C) Quantification de la libération de TNE induite par A23187 à t = 240 min en présence de concentrations indiquées de CIT-013, d’anticorps anti-histone H4, d’anticorps anti-histone H3 #1 ou d’anticorps #2 contre l’histone H3 #2 (n = 6). (D) Quantification de la libération de TNE à t = 240 min induite par des complexes immuns solubles (CI) en présence de CIT-013 ou de cIgG. Les résultats sont présentés sous forme de moyenne ±'erreur type de la moyenne. P < 0,0001, ANOVA unidirectionnelle à mesures répétées avec comparaisons multiples de Tukey (A) ou test de rang signé des paires appariées de Wilcoxon bilatérales (D). Les panneaux A, B et D ont été modifiés avec la permission de van der Linden et al.16. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Plaque à 96 puits #1 | Plaque à 96 puits #2 | |
| Volume 0,001 % solution de poly-L-lysine par puits | 50 μL | 100 μL |
| Volume de suspension de neutrophiles par puits | 50 μL | 87,5 μL |
| Nombre de neutrophiles par puits | 2 x 104 cellules | 3,5 x 104 cellules |
| Volume 4x colorant d’ADN concentré (= 80 nM) dans le tampon de test NET par puits | 50 μL | 87,5 μL |
| Volume 4x stimuli NETosis concentrés dans le tampon d’essai NET par puits | 50 μL | 87,5 μL |
| Volume 4x antagonistes concentrés de NETosis dans le tampon de test NET par puits | 50 μL | 87,5 μL |
Tableau 1 : Volumes et numéros de cellules optimisés pour différentes plaques d’imagerie à 96 puits.
| Solution de travail de concentration (4x concentrée) | Concentration finale | |
| Ionophore de calcium (A23187) | 50 μM | 12,5 μM |
| L' | 16 nM | 4 nM |
| fMLP | 4 μM | 1 μM |
| Cristaux d’urate monosodique (MSU) | 400 μg/mL | 100 μg/mL |
| Cristaux de maladie de disposition du pyrophosphate de calcium (CPPD) | 400 μg/mL | 100 μg/mL |
| Complexes immuns solubles (sIC) | 1. Ajouter 5 μg/mL d’albumine sérique humaine (HSA) dans le DPBS à 282,5 μg/mL d’anticorps polyclonaux anti-HSA de lapin dans le DPBS. | |
| 2. Incuber pendant au moins 90 min à 37 °C. | ||
| 3. Homogénéiser par vortex et ajouter une solution de sIC de 50 μL dans les puits correspondants. | ||
| Complexes immuns enrobés (cIC) | 1. Ajouter 10 μg/mL de HSA dans le DPBS dans les puits correspondants de la plaque à 96 puits. | |
| 2. Incuber toute la nuit à 4 °C. | ||
| 3. Laver les puits 3 fois avec 200 μL 0,05 % Tween-20 dans DPBS (ci-après dénommé PBS/0,05 % Tween). | ||
| 4. Bloquer les puits avec 200 μL 1 % (p/v) d’albumine sérique bovine dans PBS/0,05 % Tween (ci-après appelé tampon de blocage). | ||
| 5. Incuber pendant 120 min à température ambiante et en agitant doucement (400 tr/min). | ||
| 6. Lavez les puits 3 fois avec 200 μL de PBS/0,05 % de préadolescent. | ||
| 7. Ajouter 50 μL d’anticorps polyclonaux de lapin anti-HSA dans le tampon de blocage dans les puits correspondants. | ||
| 8. Incuber pendant 60 min à température ambiante et en agitant doucement (400 tr/min). | ||
| 9. Lavez les puits 3 fois avec 200 μL de PBS/0,05 % de Tween. | ||
| 10. Enfin, lavez les puits 3 fois avec 200 μL DPBS. Les puits sont maintenant prêts pour l’étape 4.7 du protocole. | ||
Tableau 2 : Concentrations recommandées pour les stimuli NETosis.
| Solution de travail de concentration (4x concentrée) | Concentration finale | |
| Anticorps anti-lysozyme d’œuf de poule (anticorps de contrôle ; cIgG) | 80 nM | 20 nM |
| CIT-013 (en anglais) | 80 nM | 20 nM |
Tableau 3 : Concentrations recommandées pour l’antagoniste de NETosis.
Vidéo supplémentaire 1. Les neutrophiles ont été stimulés avec A23187 en présence de cIgG (à gauche) ou de CIT-013 (à droite), et la libération de TNE a été visualisée au fil du temps à l’aide du colorant d’ADN imperméable à la membrane plasmique. La libération de TNE est inhibée en présence de CIT-013. Le film est une superposition de colorant d’ADN (vert) et de contraste de phase. Cette vidéo a été obtenue avec la permission de van der Linden et al.16. Veuillez cliquer ici pour télécharger cette vidéo.
Discussion
Depuis la découverte des TNE en 2004, de nombreuses stratégies ont été développées pour étudier expérimentalement la libération de TNE in vitro, la microcopie par immunofluorescence étant la technique la plus courante pour quantifier la NETosis27,28. Bien que la microscopie soit utile pour visualiser la libération de TNE, elle présente des limites car la quantification microscopique non automatisée d’images de points temporels fixes est plutôt imprécise et peut souffrir d’un biais d’observateur. Une autre technique utilisée pour étudier la libération de TNE est la cytométrie en flux d’imagerie multispectrale29,30, qui mesure un grand nombre de neutrophiles en réseau et adopte une analyse impartiale, mais se concentre sur les neutrophiles aux premiers stades de la NETosis et ne quantifie pas les neutrophiles qui ont libéré leurs TNE. De nombreuses études sur la cinétique NETosis utilisent un colorant d’ADN pour quantifier la libération de NET en combinaison avec un lecteur de plaque de fluorescence31. Cette technique est incapable de distinguer les TNE des neutrophiles activés ou mourants avec coloration intracellulaire de l’ADN et, par conséquent, ne convient pas à la quantification de la libération de TNE et à l’étude des antagonistes de la NETosis. Ce qui précède souligne que les approches actuellement utilisées pour étudier NETosis sont précieuses mais ont leurs limites.
La méthode de microscopie en temps réel décrite dans cette étude résout de nombreux problèmes rencontrés dans les techniques précédemment rapportées. Il offre une quantification NET impartiale, semi-automatisée, à haut débit, reproductible et précise. En effet, cette approche minimise les artefacts de pipetage, comme la morphologie étirée des TNE18, et elle est capable de distinguer la NETosis de la nécrose et de l’apoptose grâce à la morphologie et à la cinétique distinctes de la libération d’ADN18,32. De plus, cette approche offre l’opportunité d’étudier différentes voies biochimiques pour la mort cellulaire régulée, comme la formation de TNE dans les neutrophiles nécroptotiques, qui génèrent des structures répondant aux critères fonctionnels et phénotypiques des TNE33.
Ce protocole comporte plusieurs étapes critiques qui doivent être suivies pour une quantification réussie des TNE. Tout d’abord, le nombre correct de cellules par puits est important pour une quantification précise des TNE. Lorsque la densité de neutrophiles est trop élevée, les cellules et les TNE se chevauchent avec les cellules et les TNE adjacentes, ce qui les rend difficiles à distinguer et, par conséquent, la quantification devient imprécise. Deuxièmement, il est indispensable d’utiliser une faible concentration de colorant de liaison à l’ADN imperméable à la membrane plasmique non toxique pour colorer les TNE lorsqu’elles sont sécrétées dans l’environnement extracellulaire. Les colorants de liaison à l’ADN perméables à la membrane plasmique peuvent facilement induire l’activation des neutrophiles ou la mort cellulaire. Troisièmement, plusieurs images par puits doivent être analysées pour obtenir une vue d’ensemble représentative de la libération de TNE parmi la population hétérogène de neutrophiles. Quatrièmement, l’image en contraste de phase des neutrophiles à t = 0 est nécessaire pour corriger le nombre de neutrophiles par image et calculer le pourcentage de neutrophiles nettoyants.
Bien que cette approche de microscopie en temps réel à haut débit présente de nombreux avantages par rapport aux autres tests de détection des TNE, cette méthode présente des limites car, pour autant que nous le sachions, il n’existe pas de colorants fluorescents disponibles pour détecter des composants supplémentaires de TNE afin de confirmer la libération de TNE. Des anticorps marqués par fluorescence pour détecter d’autres composants de TNE peuvent être utilisés, mais pourraient avoir des effets indésirables, car les complexes anticorps-immunité influencent l’activation des neutrophiles et éventuellement la NETosis. Par conséquent, nous préférons ne pas utiliser d’anticorps supplémentaires dans cette configuration de test et recommandons d’utiliser à la place des stimuli bien connus induisant la NETose. Lorsque des stimuli NETosis non encore établis sont utilisés, nous préconisons l’utilisation d’un ELISA pour détecter l’ADN complexé avec des protéines spécifiques de NET, telles que les histones citrullinées, avant le test d’imagerie en direct. Deuxièmement, la variation intra et inter-test peut résulter de la variabilité d’un donneur à l’autre, les neutrophiles de donneurs sains répondant différemment à divers stimuli de NETosis en raison de l’hétérogénéité au sein de la population saine. Pour minimiser les variations du dosage, les neutrophiles doivent être isolés dans les 1 h à partir de sang fraîchement prélevé et utilisés immédiatement dans l’expérience, car les neutrophiles ont une courte durée de vie et ne peuvent pas être cryoconservés. De plus, il est important qu’un protocole d’isolement des neutrophiles soit validé et adopté pour minimiser l’activation des neutrophiles. Les neutrophiles sont des cellules sensibles et peuvent modifier leur réactivité aux stimuli pendant le processus de purification en raison de contraintes mécaniques et autres. L’état d’activation des neutrophiles et la libération de TNE peuvent être influencés par le type d’aiguille, les tubes de prélèvement sanguin utilisés, la température d’incubation, la vitesse de centrifugation et le temps écoulé entre le prélèvement sanguin et l’isolement des neutrophiles 34,35,36,37. Une méthode supplémentaire d’isolement des neutrophiles qui peut être envisagée est décrite par Krémer et al.36 à l’aide d’une méthode de sélection immunomagnétique négative sans lyse des globules rouges. Cette méthode ressemble aux neutrophiles intacts dans le sang total et pourrait convenir pour empêcher toute activation des neutrophiles pendant le processus de purification. Tout ce qui précède devrait servir à avertir le domaine que les données de différents groupes de recherche doivent être comparées avec le plus grand soin.
Dans l’ensemble, la méthode de microscopie à haut débit en temps réel décrite permet une quantification précise des TNE de manière reproductible et efficace et peut être utilisée pour étudier les caractéristiques, l’amplitude et la cinétique de la libération de TNE et permet d’étudier l’activité des antagonistes de NETosis. À titre d’exemple de ce dernier, nous avons utilisé l’anticorps monoclonal CIT-013, un anticorps monoclonal anti-citrulliné H2A et H4, qui est actuellement en développement clinique.
Déclarations de divulgation
Les auteurs sont des employés de Citryll et ont des intérêts financiers.
Remerciements
Les auteurs tiennent à remercier Paul Vink pour la gestion d’une partie du projet concernant la méthode de microscopie à imagerie en direct.
matériels
| Name | Company | Catalog Number | Comments |
| A-23187 Free Acid (Calcimycin) | Thermo Fisher | A1493 | |
| Ammonium chloride (NH4Cl) | Sigma Aldrich | A9434 | |
| Anti-Hen egg lysozyme (control antibody; cIgG) | CrownBio | C0001 | |
| APC-Cy7-conjugated mouse anti-human CD45 antibody (Clone 2D1) | Biolegend | 368515 | Use at 1 µg/mL final concentration |
| BD FACSCantoTM II system + FACSDiva software (version 8.0.1) | BD | n/a | Flow cytometry system |
| Bovine Serum Albumin (BSA) Fraction V | Roche | 10735108001 | |
| CaCl2 (1 M) | VWR | E506 | |
| Calcium pyrophosphate disposition disease (CPPD) crystals | InvivoGen | lrl-cppd | |
| Cellometer Auto T4 Bright Field Cell Counter + analysis software (version 3.3.9.5) | Nexcelom Bioscience | n/a | Bright field cell counter |
| CIT-013 | Citryll B.V. | n/a | |
| Costar black 96-well plate, clear bottom | Corning | 3603 | 96-well plate #1 |
| Dextran T500 | Pharmacosmos | 551005009006 | |
| DPBS (1x) | Gibco | 14190-144 | |
| Fetal Bovine Serum (FBS) | Serena | S-FBS-SA-015 | |
| Ficoll | GE Healthcare | 17-1440-02 | Density gradient solution |
| FITC-conjugated mouse anti-human CD66b antibody (Clone G10F5) | eBioscience | 17-0666-42 | Use at 1.5 µg/mL final concentration |
| Fixable viability dye eFluor 506 | eBioscience | 65-0866-14 | |
| Flow cytometry analysis software | FLowJo | Version 10.8.0 | |
| fMLP | Sigma Aldrich | 47729-10MG-F | |
| HEPES (1M) | Gibco | 15630-080 | |
| Human serum albumin (HSA) | VWR | 31020 | |
| Human TruStain FcX | Biolegend | 422302 | Fc receptor block |
| IBIDI 96-well plate, clear bottom | IBIDI | 89626 | 96-well plate #2 |
| IncuCyte SX3 + analysis software (version 2022B Rev1) | Satorius | n/a | Live cell microscopic analysis system |
| Monosodium urate crystals | InvivoGen | tlrl-msu | |
| Mouse anti-histone H3 (citrulline R2 + R8 + R17) antibody | Cayman | 17939 | Clone 11D3; referred to as #1 |
| Mouse anti-histone H4 (K8Ac + K12Ac + K16Ac) antibody (Clone KM-2) | Absolute Antibodies | Ab01681-2.0 | |
| Na2EDTA | Sigma Aldrich | E5134 | |
| NaCl (0.9%) | B. Braun | 25900 | |
| Penicillin (5000 U/mL) - Streptomycin (5000 µg/mL) | Gibco | 15070-063 | |
| PerCP-Cy5.5-conjugated mouse anti-human CD16 antibody (Clone 3G8) | Biolegend | 302027 | Use at 0.33 µg/mL final concentration |
| PMA | Sigma Aldrich | P1585 | |
| Polyclonal rabbit anti-HSA antibody | Sigma Aldrich | A0433-2ml | |
| Poly-L-Lysine (0.01%) | Sigma Aldrich | P4832 | |
| Potassium bicarbonate (KHCO3) | Sigma Aldrich | 237205 | |
| Rabbit anti-histone H3 (citrulline R2 + R8 + R17) antibody | Abcam | ab281584 | Multiclonal; referred to as #2 |
| RPMI 1640 with GlutaMAXTM supplement | Gibco | 61870-010 | Culture medium containing L-glutamine |
| RPMI 1640 without phenol red | Gibco | 11835-030 | Culture medium without phenol red |
| Sodium azide (NaN3) | Sigma Aldrich | S2002 | |
| Sterile H2O | Gibco | 15230204 | |
| Sytox Green Nucleid Acid Stain | Thermo Fisher | S7020 | DNA dye |
| Trypan blue solution (0.4%) | Gibco | 15250-061 | |
| Tween-20 | Sigma Aldrich | P1379 | |
| Vacutainer blood tubes Li-Heparin (17 IU/mL) | BD | 367526 |
Références
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- De Bont, C., Pruijn, G. J. M. Citrulline is not a major determinant of autoantibody reactivity to neutrophil extracellular traps. Philos Trans R Soc Lond B Biol Sci. 378 (1890), 20220249 (2023).
- Urban, C. F., et al. Neutrophil extracellular traps contain calprotectin, a cytosolic protein complex involved in host defense against Candida albicans. PLoS Pathog. 5 (10), e1000639 (2009).
- Hoppenbrouwers, T., et al. In vitro induction of NETosis: Comprehensive live imaging comparison and systematic review. PLoS One. 12 (5), e0176472 (2017).
- Kenny, E. F., et al. Diverse stimuli engage different neutrophil extracellular trap pathways. ELife. 6, e24437 (2017).
- Parker, H., Dragunow, M., Hampton, M. B., Kettle, A. J., Winterbourn, C. C. Requirements for NADPH oxidase and myeloperoxidase in neutrophil extracellular trap formation differ depending on the stimulus. J Leukoc Biol. 92 (4), 841-849 (2012).
- Neeli, I., Radic, M. Opposition between PKC isoforms regulates histone deimination and neutrophil extracellular chromatin release. Front Immunol. 4, 38 (2013).
- Bosmann, M., Ward, P. A. Protein-based therapies for acute lung injury: targeting neutrophil extracellular traps. Expert Opin Ther Targets. 18 (6), 703-714 (2014).
- Papayannopoulos, V. Neutrophil extracellular traps in immunity and disease. Nat Rev Immunol. 18 (2), 134-147 (2018).
- Shiratori-Aso, S., Nakazawa, D. The involvement of NETs in ANCA-associated vasculitis. Front Immunol. 14, 1261151 (2023).
- Byrd, A. S., et al. Neutrophil extracellular traps, B cells, and type I interferons contribute to immune dysregulation in hidradenitis suppurativa. Sci Transl Med. 11 (508), eaav5908 (2019).
- Kimball, A. S., Obi, A. T., Diaz, J. A., Henke, P. K. The emerging role of NETs in venous thrombosis and immunothrombosis. Front Immunol. 7, 236 (2016).
- O'Neil, L. J., et al. Neutrophil extracellular trap-associated carbamylation and histones trigger osteoclast formation in rheumatoid arthritis. Ann Rheum Dis. 82 (5), 630-638 (2023).
- Li, M., et al. A novel peptidylarginine deiminase 4 (PAD4) inhibitor BMS-P5 blocks formation of neutrophil extracellular traps and delays progression of multiple myeloma. Mol Cancer Ther. 19 (7), 1530-1538 (2020).
- Chirivi, R. G. S., et al. Anti-citrullinated protein antibodies as novel therapeutic drugs in rheumatoid arthritis. J Clin Cell Immunol. S6, 006 (2013).
- Van der Linden, M., et al. Anti-citrullinated histone monoclonal antibody CIT-013, a dual action therapeutic for neutrophil extracellular trap-associated autoimmune diseases. MAbs. 15 (1), 2281763 (2023).
- Chirivi, R. G. S., et al. Therapeutic ACPA inhibits NET formation: a potential therapy for neutrophil-mediated inflammatory diseases. Cell Mol Immunol. 18 (6), 1528-1544 (2021).
- Van der Linden, M., Westerlaken, G. H. A., Van Der Vlist, M., Van Montfrans, J., Meyaard, L. Differential signalling and kinetics of neutrophil extracellular trap release revealed by quantitative live imaging. Sci Rep. 7 (1), 6529 (2017).
- Hoffmann, J. H. O., Schaekel, K., Gaiser, M. R., Enk, A. H., Hadaschik, E. N. Interindividual variation of NETosis in healthy donors: introduction and application of a refined method for extracellular trap quantification. Exp Dermatol. 25 (11), 895-900 (2016).
- Silva, L. M., Moutsopoulos, N., Bugge, T. H., Doyle, A. Live imaging and quantification of neutrophil extracellular trap formation. Curr Protoc. 1 (7), e157 (2021).
- De Bont, C. M., Koopman, W. J. H., Boelens, W. C., Pruijn, G. J. M. Stimulus-dependent chromatin dynamics, citrullination, calcium signalling and ROS production during NET formation. Biochim Biophys Acta Mol Cell Res. 1865 (11 Pt A), 1621-1629 (2018).
- Gajendran, C., et al. Alleviation of arthritis through prevention of neutrophil extracellular traps by an orally available inhibitor of protein arginine deiminase 4. Sci Rep. 13 (1), 3189 (2023).
- Biron, B. M., Chung, C. -. S., O'Brien, X. M., Chen, Y., Reichner, J. S., Ayala, A. Cl-Amidine prevents histone 3 citrullination and neutrophil extracellular trap formation, and improves survival in a murine sepsis model. J Innate Immun. 9 (1), 22-32 (2017).
- Perdomo, J., et al. Neutrophil activation and NETosis are the major drivers of thrombosis in heparin-induced thrombocytopenia. Nat Commun. 10 (1), 1322 (2019).
- Lood, C., et al. Neutrophil extracellular traps enriched in oxidized mitochondrial DNA are interferogenic and contribute to lupus-like disease. Nat Med. 22 (2), 146-153 (2016).
- Kraaij, T., et al. A novel method for high-throughput detection and quantification of neutrophil extracellular traps reveals ROS-independent NET release with immune complexes. Autoimmun Rev. 15 (6), 577-584 (2016).
- De Buhr, N., Von Köckritz-Blickwede, M. How neutrophil extracellular traps become visible. J Immunol Res. 2016, 1-13 (2016).
- Brinkmann, V., Laube, B., Abu Abed, U., Goosmann, C., Zychlinsky, A. Neutrophil extracellular traps: How to generate and visualize them. J Vis Exp. (36), e1724 (2010).
- Zhao, W., Fogg, D. K., Kaplan, M. J. A novel image-based quantitative method for the characterization of NETosis. J Immunol Methods. 423, 104-110 (2015).
- Gavillet, M., et al. Flow cytometric assay for direct quantification of neutrophil extracellular traps in blood samples. Am J Hematol. 90 (12), 1155-1158 (2015).
- Sil, P., Yoo, D., Floyd, M., Gingerich, A., Rada, B. High throughput measurement of extracellular DNA release and quantitative NET formation in human neutrophils in vitro. J Vis Exp. (112), e52779 (2016).
- Gupta, S., Chan, D. W., Zaal, K. J., Kaplan, M. J. A high-throughput real-time imaging technique to quantify NETosis and distinguish mechanisms of cell death in human neutrophils. J Immunol. 200 (2), 869-879 (2018).
- D'Cruz, A. A., et al. The pseudokinase MLKL activates PAD4-dependent NET formation in necroptotic neutrophils. Sci Signal. 11 (546), eaao1716 (2018).
- Nash, G., Abbitt, K., Tate, K., Jetha, K., Egginton, S. Changes in the mechanical and adhesive behaviour of human neutrophils on cooling in vitro. Pflugers Arch. 442 (5), 762-770 (2001).
- Hundhammer, T., Gruber, M., Wittmann, S. Paralytic impact of centrifugation on human neutrophils. Biomedicines. 10 (11), 2896 (2022).
- Krémer, V., Godon, O., Bruhns, P., Jönsson, F., De Chaisemartin, L. Isolation methods determine human neutrophil responses after stimulation. Front Immunol. 14, 1301183 (2023).
- Freitas, M., Porto, G., Lima, J. L. F. C., Fernandes, E. Isolation and activation of human neutrophils in vitro. The importance of the anticoagulant used during blood collection. Clin Biochem. 41 (7-8), 570-575 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon