Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Microbiota de los hormigueros de Atine: visualización de un paisaje microbiano mediante microscopía electrónica de barrido
En este artículo
Resumen
Proponemos un protocolo optimizado de Microscopía Electrónica de Barrido para la visualización de muestras altamente heterogéneas y delicadas que contienen biomasa vegetal y fúngica, junto con microbiota y biofilm. Este protocolo permite describir las dimensiones espaciales de la organización de la microbiota.
Resumen
En los ecosistemas a macroescala, como las selvas tropicales o los arrecifes de coral, la localización espacial de los organismos es la base de nuestra comprensión de la ecología de comunidades. En el mundo microbiano, asimismo, los ecosistemas a microescala están lejos de ser una mezcla aleatoria y homogénea de organismos y hábitats. Acceder a la distribución espacial de los microbios es fundamental para comprender el funcionamiento y la ecología de la microbiota, ya que las especies que cohabitan tienen más probabilidades de interactuar e influir en la fisiología de las demás.
Un ecosistema microbiano entre reinos es el núcleo de las colonias de hormigas productoras de hongos, que cultivan hongos basidiomicetos como recurso nutricional. Las hormigas attine buscan diversos sustratos (en su mayoría de origen vegetal), metabolizados por el hongo cultivado mientras forman una estructura esponjosa, un "jardín microbiano" que actúa como un intestino externo. El jardín es una malla entrelazada de hifas fúngicas que crecen metabolizando el sustrato, abriendo nichos para que se establezca una microbiota característica y adaptada. Se cree que la microbiota contribuye a la degradación del sustrato y al crecimiento de hongos, aunque su organización espacial aún no se ha determinado.
Aquí, describimos cómo empleamos la microscopía electrónica de barrido (SEM) para investigar, con un detalle sin precedentes, la organización espacial de la microbiota y la biopelícula en diferentes sistemas de cultivo de hongos de hormigas cultivadoras. Las imágenes SEM han proporcionado una descripción de la estructura y organización espacial de la microbiota. El SEM reveló que la microbiota suele agruparse en biopelículas, una estructura muy extendida de los paisajes microbianos de la fungicultura. Presentamos los protocolos empleados para fijar, deshidratar, secar, pulverizar el recubrimiento y obtener imágenes de una comunidad tan compleja. Estos protocolos se optimizaron para tratar muestras delicadas y heterogéneas, que comprenden biomasa vegetal y fúngica, así como la microbiota y el biofilm.
Introducción
Los ecosistemas están compuestos por organismos interconectados por procesos en una ubicación geográfica específica (es decir, el medio ambiente). Los organismos interactúan con su entorno a lo largo del tiempo, a partir del cual emergen patrones espaciales complejos y heterogéneos. Los patrones espaciales determinan la diversidad ecológica y la estabilidad y, en última instancia, el funcionamiento de los ecosistemas 1,2,3,4. En los ecosistemas a macroescala, como los humedales, las sabanas, los arrecifes de coral y los ecosistemas áridos, los patrones espaciales están correlacionados con el flujo y la concentración de recursos. Al permitir la optimización de los recursos, la heterogeneidad espacial y el modelado dan como resultado ecosistemas más resilientes que homogéneos2. La localización espacial de los organismos, que está en la base de la ecología de las comunidades, también se traslada al mundo microbiano.
Los ecosistemas microbianos, lejos de los organismos mezclados aleatoria y homogéneamente a través de los microhábitats, exhiben patrones espaciales que definen gran parte de su funcionamiento 5,6,7. Desde las columnas de Winogradsky hasta la microbiota asociada al medio ambiente y al huésped, estos ecosistemas están organizados de forma heterogénea en el espacio, con disposiciones espaciales que provocan diferentes respuestas fenotípicas. Las especies que cohabitan son más propensas a interactuar e influir en la fisiología de las demás. Así, la organización espacial comunitaria, más que su composición per se, delimita propiedades ecosistémicas y nichos ecológicos 5,7,8. Para ilustrar estos conceptos, los cambios en el patrón espacial parecen estar correlacionados con la progresión patológica de las placas dentales, caries, enfermedades de las encías 9,10, enfermedad inflamatoria intestinal11, fibrosis quística, infecciones pulmonares, infecciones crónicas de heridas12,13, cáncer colorrectal y adenomas14.
Bajo el alcance de la biogeografía microbiana (el estudio de la distribución y el patrón de la biodiversidad a través del espacio y el tiempo a microescala), el conocimiento de los ecosistemas microbianos se beneficia enormemente al comprender sus patrones espaciales 6,13,15,16,17. Hemos analizado los patrones espaciales de un ecosistema microbiano construido por insectos, que se encuentra en el núcleo de las carismáticas colonias de hormigas attina (Hymenoptera: Formicidae: Myrmicinae: Attini: Attina). Allí reside un "jardín microbiano", centrado en torno a un hongo basidiomiceto de la tribu Leucocoprinae (Basidiomycota: Agaricaceae) o de la familia Pterulaceae (Basidiomycota: Agaricales)18,19,20,21,22. El jardín es una estructura esponjosa que emerge de una malla entrelazada de hifas que crece metabolizando el sustrato mayoritariamente vegetal incorporado por las hormigas (Figura 1). Estos pueden incluir, de acuerdo con los géneros de átines: partes secas de plantas, excrementos y cadáveres de insectos, hojas recién cortadas, semillas y partes florales 23,24. Análogo a un intestino herbívoro externo, el jardín convierte enzimática y químicamente los polímeros recalcitrantes en recursos nutricionales lábiles, proporcionando a las hormigas aminoácidos esenciales, lípidos y azúcares solubles 21,25,26,27,28.
Los análisis ultraestructurales, enzimáticos y transcriptómicos realizados para jardines de los géneros Atta y Acromyrmex sugieren que estos ambientes estructuran un continuo de degradación del sustrato y parches nutricionales 26,29,30,31,32. Las partes jóvenes del jardín tienden a ser más oscuras debido al sustrato recién incorporado después de ser fragmentado. Estos sustratos recién añadidos a menudo se colonizan desde los bordes, que fueron cortados por hormigas obreras e inoculados con grupos miceliales. Irradiando desde los bordes cortados, las hifas fúngicas se extienden sobre el sustrato 29,32,33. La abundancia de hifos aumenta a medida que avanza la degradación del sustrato, dando lugar a regiones blanquecinas y metabólicamente activas 30,31,32. Las regiones más antiguas, con sustrato más degradado y una microbiota abundante29,32, tienden a presentar tonos parduzcos y mayor humedad. Los trabajadores retiran fragmentos de esta región, separándolos en pilas de desechos, donde también toman sustratos que dañan al hongosimbionte 34,35,36. Las pilas de residuos, aunque físicamente separadas del jardín, son un punto de degradación continua del sustrato y del ciclo de nutrientes por parte de la abundante microbiota habitante 29,32,37,38,39.
Una microbiota compuesta principalmente por Enterobacter, Klebsiella, Pantoea, Pseudomonas y Serratia, también habita en el jardín, al parecer compartida por diversos sistemas de cultivo de hongos. La microbiota, que codifica vías metabólicas que podrían complementar el metabolismo fúngico, participa potencialmente en las respuestas fisiológicas del jardín 40,41,42,43,44. Los datos metagenómicos no solo indicaron que la microbiota estaba allí41,42, sino que también el análisis de microscopía electrónica de barrido (SEM) del cultivo de hormigas cortadoras de hojas mostró principalmente bacterias en forma de bastoncillos sobre el sustrato de la planta32. A pesar de que las bacterias (incluyendo cepas celulolíticas) fueron aisladas de todo el jardín, se visualizaron solo en las partes más antiguas del jardín y en las pilas de desechos, así como en la pelletización inicial que llevaban las reinas fundadoras29,32. Tampoco estaba claro si la microbiota podía formar biopelículas in vivo (es decir, en el jardín y en los residuos), como sugería su capacidad metabólica42 y se observaba in vitro44.
Aquí, empleamos SEM para comprender mejor la organización espacial de la microbiota en las regiones del jardín, detallando las interacciones físicas microbiota-sustrato y microbiota-hifas. Al proporcionar imágenes con mayor profundidad focal, SEM permite observar estructuras microscópicas tridimensionales en alta resolución, lo que permite un análisis exhaustivo de los patrones espaciales de la microbiota del jardín. Detallamos los pasos para fijar, deshidratar, secar, rociar y obtener imágenes de muestras tan heterogéneas y delicadas a base de hongos. Al eliminar la etapa de postfijación con tetróxido de osmio (OsO4) y reducir el tiempo de deshidratación, simplificamos los protocolos 32,33,45 para la preparación de muestras de jardín y residuos para el análisis SEM. Este protocolo adaptado preserva los patrones estructurales de las hifas, así como la organización espacial de la microbiota y el biofilm, y podría aplicarse a otros ecosistemas microbianos y biofilms delicados.
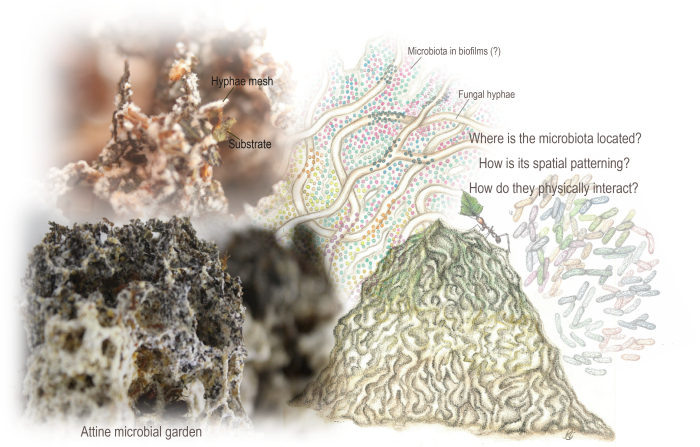
Figura 1: Jardines microbianos de Attine. El jardín es una estructura esponjosa resultante de una malla entrelazada de hifas que crece metabolizando el sustrato mayoritariamente vegetal incorporado por las hormigas. También habita en el jardín la microbiota, que codifica vías metabólicas que podrían complementar el metabolismo de los hongos. Los datos metagenómicos y los análisis previos de microscopía electrónica de barrido indicaron su presencia, aunque teníamos escaso conocimiento de su organización espacial e interacciones físicas con el sustrato y las hifas fúngicas. Empleamos SEM para desvelar la organización y el patrón espacial de la microbiota y la biopelícula. Ilustraciones de Mariana Barcoto (jardín y microbiota adaptados de Barcoto y Rodrigues 94), y fotos de Mariana Barcoto y Enzo Sorrentino. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Muestreo de colonias de campo
NOTA: Al recolectar colonias de hormigas, certifique que se obtienen todos los permisos requeridos por la legislación local antes de recolectar. En nuestro caso, el permiso de recolección #74585 fue emitido por el Instituto Chico Mendes de Conservação e Biodiversidade (ICMBio). Cuando las muestras provengan de una colonia de laboratorio, vaya a la sección 2.
- Localiza y marca la colonia. Excave una zanja que rodee el área del nido hasta que la cámara del jardín quede expuesta (Figura 2A).
NOTA: Algunas especies de áttina pueden construir sus colonias debajo de la hojarasca o dentro de troncos podridos. En tales casos, gire con cuidado la cama o rompa con cuidado los troncos para recoger las muestras. Para obtener información detallada sobre la localización, recolección y mantenimiento de colonias vivas de diversas especies de hormigas atinadas, véase Sosa-Calvo et al.46. - Abra la cámara de jardín lateralmente para evitar que la tierra caiga sobre la superficie del jardín. Recoja cuidadosamente las muestras de jardín con pinzas entomológicas, una cuchara o una espumadera de cocina, según el tamaño del jardín.
NOTA: Asegúrese de esterilizar las herramientas antes de recolectarlas. Cuando recoja los jardines de las hormigas cortadoras de hojas, use guantes de tela gruesa para evitar (o al menos atenuar) las picaduras de las obreras. Para otras especies de atina, los guantes de tela son opcionales. - Transfiera las muestras de jardín a un recipiente de plástico limpio que contenga una capa de yeso para equilibrar la humedad del jardín. Después de transferir el jardín y las hormigas, cierre herméticamente el recipiente para evitar que la muestra se seque. Almacene las muestras de jardín a 23-25 °C hasta su procesamiento.
- Cierre la zanja con tierra previamente removida.
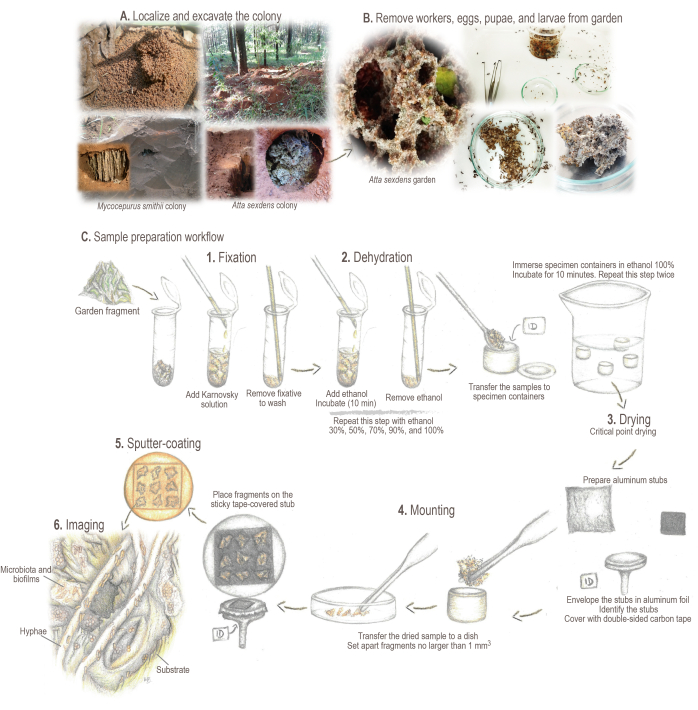
Figura 2: Protocolo de preparación de la muestra. A) Muestreo de las colonias de campo. (B) Procesamiento de muestras. (C) Fundamentos breves y flujo de trabajo para la preparación de muestras: 1. Fijación: para fortalecer y preservar la estructura de la muestra. 2. Deshidratación: el contenido de agua de las muestras se intercambia por etanol. 3. Secado de puntos críticos: el CO2 líquido sustituye al etanol y se evapora. 4. Montaje: muestra mostrada para su análisis. 5. Recubrimiento por pulverización catódica con oro: evite la carga de la muestra. 6. Imagenología. Ilustraciones y fotos de Mariana Barcoto. Haga clic aquí para ver una versión más grande de esta figura.
2. Reactivos
NOTA: Tenga en cuenta que las siguientes soluciones deben prepararse de antemano.
- Prepare tampón de cacodilato de sodio 0,2 M. Para ello, disuelva 42,8 g de cacodilato de sodio en 800 mL de agua destilada, agitando hasta la disolución y la estabilización del pH a 7,2 (si es necesario, ajuste el pH con ácido clorhídrico). Aumente el volumen de la solución a 1 L añadiendo agua destilada. Almacene la solución a 4 °C (durante ~1 mes).
PRECAUCIÓN: El glutaraldehído y el paraformaldehído son tóxicos y deben manipularse dentro de una campana extractora. Use guantes de nitrilo y gafas protectoras todo el tiempo mientras manipula dichos reactivos. - Preparar el fijador de Karnovsky (modificado de Karnovsky)47. Para ello, añadir 10 mL de solución acuosa de glutaraldehído al 25% y 10 mL de solución acuosa de paraformaldehído al 20% a 25 mL de tampón de cacodilato de sodio 0,2 M y mezclar. Añadir 1 mL de cloruro cálcico 0,1 M (CaCl2) y aumentar el volumen a 100 mL añadiendo agua destilada. Almacene la solución a 4 °C (hasta 1 mes).
PRECAUCIÓN: El fijador de Karnovsky es dañino cuando se inhala y puede causar irritación de la piel y los ojos. Por lo tanto, evite respirar vapores, usándolo solo al aire libre o en un área bien ventilada. Use guantes de nitrilo y gafas protectoras todo el tiempo cuando manipule la solución. - Usando etanol absoluto (grado analítico), prepare soluciones de etanol al 30%, 50%, 70% y 90% en agua destilada; También se requiere etanol al 100%.
3. Fijación de la muestra
NOTA: Los fijadores endurecen y preservan las muestras, manteniendo las características morfológicas. Los aldehídos (como el paraformaldehído y el glutaraldehído) son fijadores no coagulantes de tipo reticulante, que inducen enlaces cruzados dentro y entre las proteínas y los ácidos nucleicos48.
- Retire las obreras, los huevos, las pupas y las larvas de las muestras del jardín con pinzas entomológicas (Figura 2B). Separe los fragmentos de jardín que no superen los 5 mm3. Agregue los fragmentos a un tubo de 2 mL (Figura 2C).
- A los tubos que contienen las muestras, utilice una pipeta de vidrio Pasteur para añadir ~1 mL de solución fijadora Karnovsky (asegúrese de que la muestra esté completamente cubierta). Mezclar agitando suavemente para ayudar a que la muestra se remoje e incube a 4 °C durante al menos 24 h antes de continuar con el procesamiento de la muestra (Figura 2C.1).
NOTA: Cuando los siguientes pasos de deshidratación no se llevarán a cabo inmediatamente después de la fijación, el protocolo puede detenerse en este paso y las muestras pueden almacenarse durante ~ 1 año a 4 °C.
Sugerimos utilizar una pipeta de vidrio Pasteur, ya que está compuesta por un material inerte y es más fácil de limpiar para su posterior reutilización. El material de jardín es altamente hidrofóbico y tiende a flotar sobre la superficie de la solución fijadora. Por lo general, tarda hasta 5 minutos en empaparse completamente en el fijador. Asegúrese de que el fijador cubra los fragmentos del jardín, ya que su volumen tiende a reducirse a medida que ingresa a los poros de la muestra. Verificamos empíricamente que después de que la muestra se empapó y se humedeció, sus componentes (en particular, el delicado micelio fúngico) se volvieron susceptibles de romperse cuando se mezclaron más. Por lo tanto, recomendamos evitar agitar las muestras tanto como sea posible.
4. Deshidratación de la muestra
NOTA: La serie de lavado de etanol cambia gradualmente el agua de las muestras por etanol. Es importante comenzar con una solución de etanol de baja concentración (ver más abajo) para evitar dañar excesivamente o colapsar muestras tan delicadas49.
- Retire completamente la solución fijadora de Karnovsky con una pipeta de vidrio, teniendo cuidado de no alterar la muestra (Figura 2C.1).
NOTA: Deseche el fijador de Karnovsky en un recipiente debidamente etiquetado para el manejo de residuos químicos tóxicos. - Inmediatamente después de retirar el fijador, añadir 1 mL de etanol al 30%, teniendo cuidado de no alterar la muestra, e incubar durante 10 min a temperatura ambiente (Figura 2C.2).
NOTA: Las muestras siempre deben estar empapadas en solución. Asegúrese de reemplazar rápidamente las soluciones durante la serie de lavado gradual de etanol. A medida que el jardín pierde su aspecto poroso y se agrega en el fondo del tubo, 1 mL de etanol tiende a ser suficiente para cubrir la muestra (cuando la muestra no supera los 5mm3). Sin embargo, si las muestras no están completamente cubiertas, agregue etanol hasta que la muestra esté completamente cubierta. - Elimine completamente el etanol al 30% con una pipeta de vidrio, teniendo cuidado de no interrumpir la muestra. Deseche el etanol al 30% correctamente.
NOTA: Para toda la serie de lavado gradual de etanol, deseche el etanol en un recipiente debidamente etiquetado para el manejo de residuos químicos tóxicos. - Añadir 1 mL de etanol al 50% e incubar durante 10 min a temperatura ambiente. Elimine completamente el etanol al 50% con una pipeta de vidrio, teniendo cuidado de no alterar la muestra. Deseche el etanol al 50% correctamente.
- Añadir 1 mL de etanol al 70% e incubar durante 10 min a temperatura ambiente. Elimine completamente el etanol al 70% con una pipeta de vidrio, teniendo cuidado de no alterar la muestra. Deseche el etanol al 70% correctamente.
NOTA: En la etapa de lavado con etanol al 70%, el usuario puede pausar el protocolo si es necesario, ya que los tubos de muestra pueden almacenarse durante la noche a 4 °C, cuando el material no se procesará inmediatamente. - Agregue 1 mL de etanol al 90% e incube durante 10 min a temperatura ambiente. Elimine completamente el etanol al 90% con una pipeta de vidrio, teniendo cuidado de no alterar la muestra. Deseche el etanol al 90% correctamente.
- Agregue 1 mL de etanol 100% e incube durante 10 min a temperatura ambiente. Retire completamente el etanol 100% con una pipeta de vidrio, teniendo cuidado de no interrumpir la muestra. Deseche el etanol 100% correctamente.
- Con una pinza y/o una espátula, transfiera cuidadosamente las muestras a recipientes de muestras para el secador de puntos críticos (CPD), que contengan etiquetas de identificación de la muestra (hechas previamente en papel y lápiz). Para evitar el secado de las muestras, este trasvase se realiza con el recipiente colocado en una placa de Petri cubierta con etanol 100%.
- Coloque las tapas en los recipientes y sumérjalos en un vaso de precipitados de vidrio graduado que contenga 100% de etanol suficiente para sumergir los recipientes. Cubra el vaso de precipitados e incube durante 10 minutos a temperatura ambiente; A continuación, deseche adecuadamente el etanol al 100%.
- Transfiera los recipientes de muestras a otro vaso de precipitados de vidrio graduado que contenga suficiente etanol al 100% para sumergir los recipientes. Cubra el vaso de precipitados e incube durante 10 minutos a temperatura ambiente; A continuación, transfiera los recipientes de muestras al secador de puntos críticos.
NOTA: Después de la serie de lavado, las pipetas y vasos de precipitados de vidrio deben enjuagarse abundantemente con agua destilada, y esta agua residual debe desecharse en un recipiente debidamente etiquetado para el manejo de residuos químicos tóxicos. Después del enjuague, los artículos de vidrio se pueden lavar con detergente neutro, enjuagar con agua del grifo y secar al aire.
5. Secado de puntos críticos
NOTA: Un secador de punto crítico intercambia el etanol en las muestras por dióxido de carbono líquido (CO2), que se evapora de la muestra a mayor temperatura y presión. Siga las instrucciones del fabricante para dichos procedimientos.
- Encienda el equipo.
- Abra la cámara, coloque los recipientes de muestra dentro y agregue etanol 100% hasta que cubra los contenedores. Cierre la cámara.
- Active la opción Frío y espere hasta que la temperatura alcance los 10 °C.
- Abra la válvula del cilindro deCO2 y active la opción Agitador .
- Activa la opción de CO2 in, siempre revisando la cámara para verificar cuánto CO2 ya la ha llenado. Cuando la cámara esté casi llena, desactive la opción de entrada de CO2 y active la opción de intercambio, manteniéndola activada hasta que haya suficiente CO2 solo para cubrir los recipientes. Asegúrese de que los recipientes estén siempre cubiertos con CO2 (es decir, desactive la opción de intercambio antes de que todo el CO2 haya salido de la cámara). Repita el paso 5.5 6x.
- Active el CO2 por última vez y llene la cámara hasta que los recipientes estén cubiertos.
- Active la opción Calor y desactive la opción Agitador . Cierre la válvula del cilindro deCO2.
- Espere hasta que la temperatura suba a 35 °C; luego, active la opción Salida de gas .
NOTA: A unos 30 °C, la cámara alcanza presiones de 70-80 bar, alcanzando el punto crítico, donde desaparece el líquido. - Cuando la presión de la cámara alcanza 1 bar, se ha eliminado todo el contenido de gas. Abra la cámara y retire los recipientes.
- Apague el equipo.
6. Montaje
- Prepare portamuestras SEM (es decir, talones de aluminio; Figura 2C.4).
- Envuelva los talones con un trozo de papel de aluminio, cubriendo solo la parte superior, para facilitar la limpieza de los talones después del análisis.
- Identifique los talones escribiendo el código/número de muestra en la parte inferior del soporte, garantizando la identificación de lo que se coloca en la parte superior.
- Cubre la parte superior con cinta de carbono de doble cara. Coloque los talones en un portamuestras.
- Abra la tapa del recipiente de muestras y transfiera con cuidado la muestra seca a una placa de Petri de vidrio con pinzas y espátula.
NOTA: Los jardines de punto crítico de secado tienden a aglomerarse formando muestras muy compactas que deben separarse cuidadosamente como fragmentos de no más de 1 mm3de tamaño. - Coloque con cuidado los fragmentos en la superficie pegajosa del trozo cubierto con cinta adhesiva. Una vez que el fragmento de jardín toca la cinta, es muy difícil (re)moverlo, así que tenga cuidado de no colocarlo en lugares o posiciones no deseadas. Agregue hasta nueve fragmentos por stub.
- Repita los pasos 6.2 y 6.3 para cada muestra.
7. Recubrimiento por pulverización catódica con oro
NOTA: Es necesario recubrir la muestra para evitar que se cargue. Siga las instrucciones del fabricante para ajustar configuraciones como la presión del gas de operación (0,5 × 10-1 mm Hg de presión de gas en este protocolo), el tiempo de pulverización catódica (220 s), el grosor de la capa de oro (~120 Å), la corriente (50 mA) y el suministro de voltaje. La pulverización catódica tiende a seguir un flujo de trabajo común, aunque los equipos de diferentes fabricantes pueden funcionar de manera ligeramente diferente.
- Abra el brazo objetivo con bisagras y coloque los talones en la mesa de muestras.
- Cierre el brazo de destino con bisagras y compruebe si el escudo contra astillas de la cámara de vacío de vidrio está incrustado correctamente.
- Abra la válvula del cilindro de argón y encienda el interruptor de alimentación principal.
- Siga el aumento del vacío en la pantalla del equipo hasta que alcance la marca de 0,5 × 10-1 mm Hg en la pantalla; a continuación, active el enjuague. Repita la operación 5 veces.
- Encienda el sistema de circulación de agua, active la opción HV On y abra la tapa de la película de oro. Confirme que el color del plasma es rosado. Para seguir este protocolo, configure 220 s de pulverización catódica con un voltaje de 50 mA, que depositará una capa de oro de ~ 120 Å (12 nm).
- HV On se apaga automáticamente. Apague el sistema de circulación de agua y cierre la tapa de la película de oro.
- Apague el interruptor de alimentación principal, permitiendo que el aire ingrese a la cámara de vacío. Para muestras de jardín y desechos, repita los pasos 7.1-7.7 3 veces.
NOTA: Si las imágenes no se realizan inmediatamente después de la preparación de la muestra, guarde los talones en un recipiente hermético lleno de una capa de sílice para evitar rehidratar las muestras.
8. Imágenes
NOTA: Siga las instrucciones del fabricante para ajustar la configuración de SEM, como el diámetro de apertura del objetivo, el voltaje de funcionamiento, la alineación del sistema de haz de electrones, la alineación axial y los estigmas.
- Coloque los talones en el portamuestras, tomando notas sobre la posición de cada muestra.
NOTA: Use guantes cuando inserte o retire el soporte de muestras y mantenga el soporte lo más limpio posible. - Inicie el software de operación desde el escritorio.
- Seleccione la configuración del instrumento. Visualice muestras de jardín con un diámetro de apertura objetivo de 30 μm (es decir, en la segunda etapa), operando en alto vacío, detectando señales de electrones secundarios (SED), voltaje de aceleración de 20 kV, distancia de trabajo de 15 a 20 mm, corriente de sonda de 40.0 (en modo de alta corriente) y variando el aumento de acuerdo con la muestra.
- Siguiendo las instrucciones de navegación, presione el icono de ventilación y espere a que se ventile la cámara de muestras. Una barra de progreso indica el estado del vacío.
- Cuando se haya alcanzado la presión atmosférica, abra la cámara de muestras e inserte con cuidado el portamuestras.
- Cierre suavemente la puerta de la cámara y presione el icono Evac para evacuar el instrumento, siguiendo el estado de vacío a través de la barra de progreso. El sistema de navegación mostrará la posición de la etapa móvil y proporcionará un gráfico de soporte cuando se complete el movimiento. Presione el icono de la cámara para grabar una foto del soporte para obtener una vista superior que ayudará a navegar entre las muestras mientras se toman imágenes.
- Presione el icono de observación para encender el cañón de electrones y espere a que se forme la imagen. Utilice la interfaz de usuario manual (control manual) para mover manualmente el eje Z a la altura adecuada (en función de la altura de la muestra y la distancia de trabajo determinada).
NOTA: Para prolongar la vida útil del filamento de tungsteno, detenga la función de observación siempre que no esté tomando imágenes activamente. - Utilice las opciones que se muestran en la pantalla o el control manual para cambiar la velocidad de ráster y la posición de la imagen. Mueva la platina para obtener una vista completa de la muestra, empleando la función RDC para centrarse en áreas específicas. Al observar una estructura objetivo (o interesante), ajuste la ampliación, el enfoque, el brillo, el contraste y la estigmatización en consecuencia. Corrija la estigmatización mediante el uso de la interfaz de usuario manual para mover el escenario a las direcciones X e Y.
- Ajuste la ampliación entre 100x y 700x para visualizar aspectos generales del jardín (como la densidad de hifas, el sustrato y los patrones de colonización).
- Ajuste el aumento entre 700x y 1.500x para visualizar los patrones espaciales de la microbiota.
- Ajuste el aumento entre 1.500x y 3.000x para observar las interacciones físicas de la microbiota y la biopelícula.
- Ajuste el aumento entre 3.000x y 4.000x para centrarse en grupos microbianos específicos.
- Para guardar una imagen, utilice la función Congelar , haga clic en el icono de la foto y configure la ruta del archivo. Como estándar, analice al menos tres fragmentos de jardín, tomando imágenes de cada uno en todos los rangos de aumento mencionados en el paso 8.8. Esto da como resultado al menos 12 imágenes por muestra, aunque más imágenes pueden admitir descripciones más detalladas. Sugerimos que de 15 a 25 imágenes por muestra, con todos los aumentos en el paso 8.8, tienden a proporcionar detalles finos para la descripción de la muestra.
NOTA: Rasterizar y guardar una imagen lleva unos segundos durante los cuales se debe evitar cualquier vibración en la mesa flotante SEM. - Después de terminar la toma de imágenes, presione la opción Vent para ventilar la cámara, siguiendo el estado del vacío a través de la barra de progreso. Retire con cuidado el soporte, cierre suavemente la puerta de la cámara y presione Evac para evacuar la cámara.
- Tan pronto como se complete la evacuación, salga del software de operación.
- Ajuste el brillo y el contraste para mejorar la visualización con un editor de imágenes.
Access restricted. Please log in or start a trial to view this content.
Resultados
Aquí, presentamos un protocolo simplificado para visualizar los componentes de las muestras de jardín y desechos, como hifas fúngicas, sustrato, microbiota y biopelículas. El SEM ha mejorado nuestra comprensión de cómo el jardín y los residuos moldean los patrones estructurales de la microbiota (Figura 3). En los jardines de ática, las hifas fúngicas son estructuras en forma de rama que cubren partes de la superficie del sustrato. Dado que las hifas...
Access restricted. Please log in or start a trial to view this content.
Discusión
SEM utiliza un haz de electrones para escanear la muestra, generando una imagen ampliada de la misma de modo que se pueden visualizar microestructuras tridimensionales en alta resolución. Dado que el SEM funciona en alto vacío, es necesario eliminar hasta más del 99% del agua de las muestras. Dentro de la cámara de vacío SEM, las muestras parcialmente hidratadas pueden deshidratarse y colapsar, además de dispersar electrones. Para la obtención de imágenes de alta resolución en S...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores desean agradecer a la Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP) por su apoyo financiero (Subvención #2019/03746-0). MOB agradece la beca de doctorado recibida de la FAPESP (proceso 2021/08013-0) y la Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - Brasil (CAPES) - Código de Finanzas 001. AR también agradece al Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) por una beca de investigación (#305269/2018). Los autores desean agradecer a Marcia Regina de Moura Aouada y Antonio Teruyoshi Yabuki por ayudar con las pruebas piloto para la preparación de muestras, a Renato Barbosa Salaroli por su asistencia técnica y a Enzo Sorrentino por ayudar en la sesión fotográfica. Este estudio se realizó bajo la autorización de acceso al patrimonio genético #SISGen AA39A6D.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 2 mL tube | Axygen | MCT-200-C-BRA | To fix and dehydrate samples |
| Calcium chloride anhydrous | Merck | C4901 | CaCl2 anhydrous to prepare Karnovsky’s fixative |
| Critical point dryer | Leica | EM CPD 300 | For critical point drying |
| Double Sided Carbon Conductive Tape, 12 mm (W) X 5 M (L) | Electron Microscopy Sciences | 77819-12 | For mounting samples |
| Entomological forceps | No specific supplier | To manipulate garden samples | |
| Ethyl alcohol (=ethanol), pure (≥99.5%) | Sigma-Aldrich | 459836 | For dehydration |
| Forceps | No specific supplier | To manipulate garden samples | |
| Glass beaker | No specific supplier | For dehydration | |
| Glass Petri dish | No specific supplier | To manipulate garden samples | |
| Glass pipette | No specific supplier | To fix and dehydrate samples | |
| Glutaraldehyde (Aqueous Glutaraldehyde EM Grade 25%) | Electron Microscopy Sciences | 16220 | To prepare Karnovsky’s fixative |
| Gold target | Ted Pella, Inc. | 8071 | To sputter coat with gold |
| Hydrochloric acid | Sigma-Aldrich | 320331 | For adjusting solutions pH |
| Image editor | Photoshop | any version | To adjust images |
| Paraformaldehyde (Paraformaldehyde 20% Aqueous Solution EM Grade) | Electron Microscopy Sciences | 15713 | To prepare Karnovsky’s fixative |
| Propilene recipient | No specific supplier | For maintaining alive ant colonies | |
| Scanning Electron Microscope | JEOL | IT300 SEM | For sample imaging |
| Sodium cacodylate trihydrate | Sigma-Aldrich | C0250 | For preparing sodium cacodylate buffer |
| Spatula | No specific supplier | To manipulate garden samples | |
| Specimen containers with 15 mm dia. x 10 mm high | Ted Pella, Inc. | 4591 | For critical point drying |
| Sputter coater | Baltec | SCD 050 | To coat with gold |
| Stub (Aluminium mount, flat end pin) 12.7 mm x 8 mm | Electron Microscopy Sciences | 75520 | For mounting samples |
Referencias
- Turner, M. G. Landscape ecology: the effect of pattern on process. Annu Rev Ecol Evol Syst. 20 (1), 171-197 (1989).
- Rietkerk, M., Van de Koppel, J. Regular pattern formation in real ecosystems. Trends Ecol Evol. 23 (3), 169-175 (2008).
- Schmitz, O. J. Spatial dynamics and ecosystem functioning. PLOS Biol. 8 (5), e1000378(2010).
- Pringle, R. M., Doak, D. F., Brody, A. K., Jocqué, R., Palmer, T. M. Spatial pattern enhances ecosystem functioning in an African savanna. PLOS Biol. 8 (5), e1000377(2010).
- Wimpenny, J. W. Spatial order in microbial ecosystems. Biol Rev. 56 (3), 295-342 (1981).
- Martiny, J. B. H., et al. Microbial biogeography: putting microorganisms on the map. Nat Rev. Microbiol. 4 (2), 102-112 (2006).
- McCallum, G., Tropini, C. The gut microbiota and its biogeography. Nat Rev Microbiol. 22 (2), 105-118 (2024).
- Lamont, R. J., Hajishengallis, G., Koo, H. Social networking at the microbiome-host interface. Infec Immun. 91 (9), e00124-e00223 (2023).
- Welch, J. L. M., Rossetti, B. J., Rieken, C. W., Dewhirst, F. E., Borisy, G. G. Biogeography of a human oral microbiome at the micron scale. Proc Natl Acad Sci USA. 113 (6), E791-E800 (2016).
- Kim, D., et al. Spatial mapping of polymicrobial communities reveals a precise biogeography associated with human dental caries. Proc Natl Acad Sci USA. 117 (22), 12375-12386 (2020).
- Swidsinski, A., Weber, J., Loening-Baucke, V., Hale, L. P., Lochs, H. Spatial organization and composition of the mucosal flora in patients with inflammatory bowel disease. J Clin Microbiol. 43 (7), 3380-3389 (2005).
- Ibberson, C. B., Barraza, J. P., Holmes, A. L., Cao, P., Whiteley, M. Precise spatial structure impacts antimicrobial susceptibility of S. aureus in polymicrobial wound infections. Proc Natl Acad Sci USA. 119 (51), e2212340119(2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Dejea, C. M., et al. Microbiota organization is a distinct feature of proximal colorectal cancers. Proc Natl Acad Sci USA. 111 (51), 18321-18326 (2014).
- Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., Martiny, J. B. Beyond biogeographic patterns: processes shaping the microbial landscape. Nat Rev Microbiol. 10 (7), 497-506 (2012).
- Adade, E. E., Al Lakhen, K., Lemus, A. A., Valm, A. M. Recent progress in analyzing the spatial structure of the human microbiome: Distinguishing biogeography and architecture in the oral and gut communities. Curr Opin Endocr. 18, 275-283 (2021).
- Mony, C., Bohannan, B. J., Leibold, M. A., Peay, K., Vandenkoornhuyse, P. Microbial landscape ecology: Highlights on the invisible corridors. Front Ecol Evol. 9, 753213(2021).
- Hölldobler, B., Wilson, E. O. The Ants. , Harvard University Press. (1990).
- Chapela, I. H., Rehner, S. A., Schultz, T. R., Mueller, U. G. Evolutionary history of the symbiosis between fungus-growing ants and their fungi. Science. 266 (5191), 1691-1694 (1994).
- Mueller, U. G., Rehner, S. A., Schultz, T. R. The evolution of agriculture in ants. Science. 281 (5385), 2034-2038 (1998).
- Mueller, U. G., Gerardo, N. M., Aanen, D. K., Six, D. L., Schultz, T. R. The evolution of agriculture in insects. Annu Rev Ecol Evol Syst. 36, 563-595 (2005).
- Dentinger, B. T., Lodge, D. J., Munkacsi, A. B., Desjardin, D. E., McLaughlin, D. J. Phylogenetic placement of an unusual coral mushroom challenges the classic hypothesis of strict coevolution in the Apterostigma pilosum group ant-fungus mutualism. Evolution. 63 (8), 2172-2178 (2009).
- Schultz, T. R., Brady, S. G. Major evolutionary transitions in ant agriculture. Proc Natl Acad Sci USA. 105, 5435-5440 (2008).
- de Fine Licht, H. H., Boomsma, J. J. Forage collection, substrate preparation, and diet composition in fungus-growing ants. Ecol Entomol. 35 (3), 259-269 (2010).
- Martin, M. M. The biochemical basis of the fungus-attine ant symbiosis: A complex symbiosis is based upon integration of the carbon and nitrogen metabolisms of the two organisms. Science. 169 (3940), 16-20 (1970).
- Grell, M. N., et al. The fungal symbiont of Acromyrmex leaf-cutting ants expresses the full spectrum of genes to degrade cellulose and other plant cell wall polysaccharides. BMC Genomics. 14, 928(2013).
- Lange, L., Grell, M. N. The prominent role of fungi and fungal enzymes in the ant-fungus biomass conversion symbiosis. Appl Microbiol Biotechnol. 98, 4839-4851 (2014).
- Huang, E. L., et al. The fungus gardens of leaf-cutter ants undergo a distinct physiological transition during biomass degradation. Environ Microbiol Rep. 6 (4), 389-395 (2014).
- Craven, S. E., Dix, M. W., Michaels, G. E. Attine fungus gardens contain yeasts. Science. 169 (3941), 184-186 (1970).
- De Fine Licht, H. H., Boomsma, J. J., Tunlid, A. Symbiotic adaptations in the fungal cultivar of leaf-cutting ants. Nat Commun. 5 (1), 5675(2014).
- Aylward, F. O., et al. Enrichment and broad representation of plant biomass-degrading enzymes in the specialized hyphal swellings of Leucoagaricus gongylophorus, the fungal symbiont of leaf-cutter ants. PLoS One. 10 (8), e0134752(2015).
- Moreira-Soto, R. D., Sanchez, E., Currie, C. R., Pinto-Tomás, A. A. Ultrastructural and microbial analyses of cellulose degradation in leaf-cutter ant colonies. Microbiology. 163 (11), 1578-1589 (2017).
- Erthal Jr, M., Silva, C. P., Cooper, R. M., Samuels, R. I. Hydrolytic enzymes of leaf-cutting ant fungi. Comp Biochem. 152 (1), 54-59 (2009).
- North, R. D., Jackson, C. W., Howse, P. E. Communication between the fungus garden and workers of the leaf-cutting ant, Atta sexdens rubropilosa, regarding choice of substrate for the fungus. Physiol Entomol. 24 (2), 127-133 (1999).
- Herz, H., Hölldobler, B., Roces, F. Delayed rejection in a leaf-cutting ant after foraging on plants unsuitable for the symbiotic fungus. Behav Ecol. 19 (3), 575-582 (2008).
- Schiøtt, M., De Fine Licht, H. H., Lange, L., Boomsma, J. J. Towards a molecular understanding of symbiont function: identification of a fungal gene for the degradation of xylan in the fungus gardens of leaf-cutting ants. BMC Microbiol. 8, 40(2008).
- Bot, A. N., Currie, C. R., Hart, A. G., Boomsma, J. J. Waste management in leaf-cutting ants. Ethol Ecol Evol. 13 (3), 225-237 (2001).
- Scott, J. J., et al. Microbial community structure of leaf-cutter ant fungus gardens and refuse dumps. PLOS One. 5 (3), e9922(2010).
- Lewin, G. R., et al. Cellulose-enriched microbial communities from leaf-cutter ant (Atta colombica) refuse dumps vary in taxonomic composition and degradation ability. PLOS One. 11 (3), e0151840(2016).
- Suen, G., et al. An insect herbivore microbiome with high plant biomass-degrading capacity. PLOS Genet. 6 (9), e1001129(2010).
- Aylward, F. O., et al. Convergent bacterial microbiotas in the fungal agricultural systems of insects. MBio. 5 (6), e02077(2014).
- Barcoto, M. O., et al. Fungus-growing insects host a distinctive microbiota apparently adapted to the fungiculture environment. Sci Rep. 10 (1), 12384(2020).
- Francoeur, C. B., et al. Bacteria contribute to plant secondary compound degradation in a generalist herbivore system. mBio. 11, e02146-e02220 (2020).
- Martiarena, M. J. S., Deveau, A., Montoya, Q. V., Flórez, L. V., Rodrigues, A. The hyphosphere of leaf-cutting ant cultivars is enriched with helper bacteria. Microb Ecol. 86 (3), 1773-1788 (2023).
- Leal-Dutra, C. A., et al. Evidence that the domesticated fungus Leucoagaricus gongylophorus recycles its cytoplasmic contents as nutritional rewards to feed its leafcutter ant farmers. IMA Fungus. 14 (1), 19(2023).
- Sosa-Calvo, J., Jesovnik, A., Okonski, E., Schultz, T. R. Locating, collecting, and maintaining colonies of fungus-farming ants (Hymenoptera: Myrmicinae: Attini). Sociobiology. 62 (2), 300-320 (2015).
- Karnovsky, M. J. A formaldehyde glutaraldehyde fixative of high osmolality for use in electron microscopy. J Cell Biol. 27, 137-138 (1965).
- Eltoum, I., Fredenburgh, J., Myers, R. B., Grizzle, W. E. Introduction to the theory and practice of fixation of tissues. J Histotechnol. 24 (3), 173-190 (2001).
- Gusnard, D., Kirschner, R. H. Cell and organelle shrinkage during preparation for scanning electron microscopy: effects of fixation, dehydration and critical point drying. J Microsc. 110 (1), 51-57 (1977).
- Surman, S. B., et al. Comparison of microscope techniques for the examination of biofilms. J Microbiol Methods. 25 (1), 57-70 (1996).
- Augustin, J. O., et al. Yet more "weeds" in the garden: fungal novelties from nests of leaf-cutting ants. PLOS One. 8 (12), e82265(2013).
- Montoya, Q. V., Martiarena, M. J. S., Polezel, D. A., Rodrigues, A. More pieces to a huge puzzle: Two new Escovopsis species from fungus gardens of attine ants. MycoKeys. 46, 97(2019).
- Varanda-Haifig, S. S., et al. Nature of the interactions between hypocrealean fungi and the mutualistic fungus of leaf-cutter ants. Antonie van Leeuwenhoek. 110, 593-605 (2017).
- Custodio, B. C., Rodrigues, A. Escovopsis kreiselii specialization to its native hosts in the fungiculture of the lower attine ant Mycetophylax morschi. Antonie van Leeuwenhoek. 112, 305-317 (2019).
- Schröttner, H., Schmied, M., Scherer, S. Comparison of 3D surface reconstruction data from certified depth standards obtained by SEM and an Infinite Focus Measurement Machine (IFM). Microchim Acta. 155, 279-284 (2006).
- Zhou, W., Apkarian, R., Wang, Z. L., Joy, D. Fundamentals of scanning electron microscopy (SEM). Scanning microscopy for nanotechnology: Techniques and applications. Zhou, W., Wang, Z. L. , Springer. New York, NY. 1-40 (2007).
- Kannan, M. Scanning electron microscopy: Principle, components and applications. A Textbook on Fundamentals and Applications of Nanotechnology. Raja, K., Subramanian, K. S., Kannan, M. , Daya Publishing House. 81-92 (2018).
- Ruffolo, J. J. Jr Critical point drying of protozoan cells and other biological specimens for scanning electron microscopy: apparatus and methods of specimen preparation. Trans Am Microsc. 93 (1), 124-131 (1974).
- Echlin, P. Recent advances in specimen coating techniques for electron microscopy. Scanning Electron Microscopy 1981/1. , IITRI. Chicago. 79-90 (1981).
- Ris, H. The cytoplasmic filament system in critical point-dried whole mounts and plastic-embedded sections. J Cell Biol. 100 (5), 1474-1487 (1985).
- Bray, D. F., Bagu, J., Koegler, P. Comparison of hexamethyldisilazane (HMDS), Peldri II, and critical-point drying methods for scanning electron microscopy of biological specimens. Microsc Res Tech. 26 (6), 489-495 (1993).
- Bergmans, L., Moisiadis, P., Van Meerbeek, B., Quirynen, M., Lambrechts, P. Microscopic observation of bacteria: review highlighting the use of environmental SEM. Int Endod J. 38 (11), 775-788 (2005).
- Kirk, S. E., Skepper, J. N., Donald, A. M. Application of environmental scanning electron microscopy to determine biological surface structure. J Microsc. 233 (2), 205-224 (2009).
- Kemmenoe, B. H., Bullock, G. R. Structure analysis of sputter-coated and ion-beam sputter-coated films: a comparative study. J Microsc. 132 (2), 153-163 (1983).
- Kinden, D. A., Brown, M. F. Technique for scanning electron microscopy of fungal structures within plant cells. Phytopathology. 65, 74-76 (1975).
- Masaphy, S., Levanon, D., Tchelet, R., Henis, Y. Scanning electron microscope studies of interactions between Agaricus bisporus (Lang) Sing hyphae and bacteria in casing soil. Appl Environ Microbiol. 53 (5), 1132-1137 (1987).
- Massicotte, H. B., Melville, L. H., Peterson, R. L. Scanning electron microscopy of ectomycorrhizae potential and limitations. Scanning Microsc. 1 (3), 58(1987).
- Visen, A., Singh, P. N., Chakraborty, B., Singh, A., Bisht, T. S. Scanning electron microscopy indicates Pseudomonad strains facilitate AMF mycorrhization in litchi (Litchi chinensis Sonn.) air-layers and improving survivability, growth and leaf nutrient status. Curr Res Microb Sci. 2, 100063(2021).
- Fleeman, R. M., Mikesh, M., Davies, B. W. Investigating Klebsiella pneumoniae biofilm preservation for scanning electron microscopy. Access Microbiol. 5 (2), 000470-000473 (2023).
- Wells, M., Mikesh, M., Gordon, V. Structure-preserving fixation allows scanning electron microscopy to reveal biofilm microstructure and interactions with immune cells. J Microsc. 293 (1), 59-68 (2024).
- Nadell, C. D., Drescher, K., Foster, K. R. Spatial structure, cooperation and competition in biofilms. Nat Rev Microbiol. 14 (9), 589-600 (2016).
- Madsen, J. S., et al. Coexistence facilitates interspecific biofilm formation in complex microbial communities. Environ Microbiol. 18 (8), 2565-2574 (2016).
- Madsen, J. S., Sørensen, S. J., Burmølle, M. Bacterial social interactions and the emergence of community-intrinsic properties. Curr Opin Microbiol. 42, 104-109 (2018).
- Flemming, H. C., Wuertz, S. Bacteria and archaea on Earth and their abundance in biofilms. Nat Rev Microbiol. 17 (4), 247-260 (2019).
- Bringhurst, B., Allert, M., Greenwold, M., Kellner, K., Seal, J. N. Environments and hosts structure the bacterial microbiomes of fungus-gardening ants and their symbiotic fungus gardens. Microb Ecol. 86 (2), 1374-1392 (2023).
- Bringhurst, B., Greenwold, M., Kellner, K., Seal, J. N. Symbiosis, dysbiosis and the impact of horizontal exchange on bacterial microbiomes in higher fungus-gardening ants. Sci Rep. 14 (1), 3231(2024).
- Vargas, S., et al. Body-plan reorganization in a sponge correlates with microbiome change. Mol Biol Evol. 40 (6), msad138(2023).
- Apprill, A., et al. Humpback whale populations share a core skin bacterial community: towards a health index for marine mammals. PLOS One. 9 (3), e90785(2014).
- Fraune, S., et al. Bacteria-bacteria interactions within the microbiota of the ancestral metazoan Hydra contribute to fungal resistance. ISME J. 9 (7), 1543-1556 (2015).
- Gowen, R., Gamal, A., Di Martino, L., McCormick, T. S., Ghannoum, M. A. Modulating the microbiome for Crohn's disease treatment. Gastroenterology. 164 (5), 828-840 (2023).
- Moya, P., et al. Molecular phylogeny and ultrastructure of the lichen microalga Asterochloris mediterranea sp. nov. from Mediterranean and Canary Islands ecosystems. Int J Syst Evol Microbiol. 65 (6), 1838-1854 (2015).
- Zettler, E. R., Mincer, T. J., Amaral-Zettler, L. A. Life in the "plastisphere": microbial communities on plastic marine debris. Environ Sci Technol. 47 (13), 7137-7146 (2013).
- Porter, K. R., Kallman, F. The properties and effects of osmium tetroxide as a tissue fixative with special reference to its use for electron microscopy. Exp Cell Res. 4 (1), 127-141 (1953).
- Ligon, J. J., Abraham, J. L., Boyd, A. S. Traumatic osmium tetroxide inoculation. J Am Acad Dermatol. 45 (6), 949-952 (2001).
- Friedova, N., et al. Osmium absorption after osmium tetroxide skin and eye exposure. Basic Clin Pharmacol Toxicol. 127 (5), 429-433 (2020).
- Fratesi, S. E., Lynch, F. L., Kirkland, B. L., Brown, L. R. Effects of SEM preparation techniques on the appearance of bacteria and biofilms in the Carter Sandstone. J Sediment. 74 (6), 858-867 (2004).
- Dassanayake, R. P., et al. Identification of a reliable fixative solution to preserve the complex architecture of bacterial biofilms for scanning electron microscopy evaluation. PLOS One. 15 (5), e0233973(2020).
- Desiro, A., et al. Detection of a novel intracellular microbiome hosted in arbuscular mycorrhizal fungi. ISME J. 8 (2), 257-270 (2014).
- Morales, D. P., et al. Advances and challenges in fluorescence in situ hybridization for visualizing fungal endobacteria. Front Microbiol. 13, 892227(2022).
- Shi, H., et al. Highly multiplexed spatial mapping of microbial communities. Nature. 588 (7839), 676-681 (2020).
- Valm, A. M., Welch, J. L. M., Borisy, G. G. CLASI-FISH: principles of combinatorial labeling and spectral imaging. Syst Appl Microbiol. 35 (8), 496-502 (2012).
- Fernandez-Brime, S., Muggia, L., Maier, S., Grube, M., Wedin, M. Bacterial communities in an optional lichen symbiosis are determined by substrate, not algal photobionts. FEMS Microbiol Ecol. 95 (3), fiz012(2019).
- Schluter, S., Eickhorst, T., Mueller, C. W. Correlative imaging reveals holistic view of soil microenvironments. Environ Sci Technol. 53 (2), 829-837 (2018).
- Barcoto, M. O., Rodrigues, A. Lessons from insect fungiculture: from microbial ecology to plastics degradation. Front Microbiol. 13, 812143(2022).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados