Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Microbiote des jardins de fourmis attines : visualisation d’un paysage microbien par microscopie électronique à balayage
Dans cet article
Résumé
Nous proposons un protocole optimisé de microscopie électronique à balayage pour visualiser des échantillons hautement hétérogènes et délicats contenant de la biomasse végétale et fongique, ainsi que du microbiote et du biofilm. Ce protocole permet de décrire les dimensions spatiales de l’organisation du microbiote.
Résumé
Dans les écosystèmes à grande échelle, tels que les forêts tropicales ou les récifs coralliens, la localisation spatiale des organismes est à la base de notre compréhension de l’écologie des communautés. Dans le monde microbien, de même, les écosystèmes à l’échelle microbienne sont loin d’être un mélange aléatoire et homogène d’organismes et d’habitats. L’accès à la distribution spatiale des microbes est fondamental pour comprendre le fonctionnement et l’écologie du microbiote, car les espèces cohabitantes sont plus susceptibles d’interagir et d’influencer la physiologie de l’autre.
Un écosystème microbien interrègne est au cœur des colonies de fourmis fongiques, qui cultivent des champignons basidiomycètes comme ressource nutritionnelle. Les fourmis attines se nourrissent de substrats variés (principalement d’origine végétale), métabolisés par le champignon cultivé tout en formant une structure spongieuse, un « jardin microbien » qui agit comme un intestin externe. Le jardin est un maillage entrelacé d’hyphes fongiques qui poussent en métabolisant le substrat, ouvrant des niches pour qu’un microbiote caractéristique et adapté s’établisse. On pense que le microbiote contribue à la dégradation du substrat et à la croissance fongique, bien que son organisation spatiale reste à déterminer.
Ici, nous décrivons comment nous utilisons la microscopie électronique à balayage (MEB) pour étudier, avec des détails sans précédent, l’organisation spatiale du microbiote et du biofilm à travers différents systèmes de culture fongique des fourmis en croissance. L’imagerie MEB a fourni une description de la structure spatiale et de l’organisation du microbiote. Le MEB a révélé que le microbiote s’assemble couramment dans des biofilms, une structure répandue des paysages microbiens en fungiculture. Nous présentons les protocoles utilisés pour fixer, déshydrater, sécher, pulvériser le revêtement et imager une communauté aussi complexe. Ces protocoles ont été optimisés pour traiter des échantillons délicats et hétérogènes, comprenant de la biomasse végétale et fongique, ainsi que le microbiote et le biofilm.
Introduction
Les écosystèmes sont composés d’organismes interconnectés par des processus dans un lieu géographique précis (c’est-à-dire l’environnement). Les organismes interagissent avec leur environnement au fil du temps, d’où émergent des motifs spatiaux complexes et hétérogènes. La structure spatiale détermine la diversité et la stabilité écologiques et, en fin de compte, le fonctionnement de l’écosystème 1,2,3,4. Dans les écosystèmes à grande échelle, tels que les zones humides, les savanes, les récifs coralliens et les écosystèmes arides, les modèles spatiaux sont corrélés avec le flux et la concentration des ressources. L’optimisation des ressources, l’hétérogénéité spatiale et la structuration permettent d’obtenir des écosystèmes plus résilients que des écosystèmes homogènes2. La localisation spatiale des organismes qui est à la base de l’écologie des communautés se traduit également dans le monde microbien.
Les écosystèmes microbiens, loin des organismes mélangés de manière aléatoire et homogène dans les microhabitats, présentent des modèles spatiaux définissant une grande partie de leur fonctionnement 5,6,7. Des colonnes de Winogradsky au microbiote associé à l’environnement et à l’hôte, ces écosystèmes sont organisés de manière hétérogène dans l’espace, avec des arrangements spatiaux suscitant différentes réponses phénotypiques. Les espèces qui cohabitent sont plus susceptibles d’interagir et d’influencer la physiologie de l’autre. Ainsi, l’organisation spatiale communautaire, plus que sa composition en soi, délimite les propriétés de l’écosystème et les niches écologiques 5,7,8. À l’appui de ces concepts, les changements dans la structure spatiale semblent corrélés à la progression pathologique des plaques dentaires, des caries, des maladies des gencives 9,10, des maladies inflammatoires de l’intestin11, des infections pulmonaires de la fibrose kystique, des infections chroniques des plaies12,13, du cancer colorectal et des adénomes14.
Dans le cadre de la biogéographie microbienne (l’étude de la distribution et de la structuration de la biodiversité à travers l’espace et le temps à une échelle microbienne), la connaissance des écosystèmes microbiens est largement enrichie par la compréhension de leurs modèles spatiaux 6,13,15,16,17. Nous nous sommes penchés sur les modèles spatiaux d’un écosystème microbien construit par des insectes, trouvé au cœur des colonies charismatiques de fourmis attines (Hymenoptera : Formicidae : Myrmicinae : Attini : Attina). Il existe un « jardin microbien », centré autour d’un champignon basidiomycète dans la tribu des Leucocoprinae (Basidiomycètes : Agaricaceae) ou dans la famille des Ptérulaceae (Basidiomycètes : Agaricales)18,19,20,21,22. Le jardin est une structure spongieuse émergeant d’un maillage entrelacé d’hyphes qui se développe en métabolisant le substrat principalement végétal incorporé par les fourmis (Figure 1). Il peut s’agir, selon les genres d’attines : de parties sèches de plantes, d’excréments et de carcasses d’insectes, de feuilles fraîchement coupées, de graines et de parties florales23,24. Analogue à un intestin herbivore externe, le jardin convertit enzymatiquement et chimiquement les polymères récalcitrants en ressources nutritionnelles labiles, fournissant aux fourmis des acides aminés essentiels, des lipides et des sucres solubles 21,25,26,27,28.
Les analyses ultrastructurales, enzymatiques et transcriptomiques effectuées pour les jardins des genres coupeuses de feuilles Atta et Acromyrmex suggèrent que ces environnements structurent un continuum de dégradation du substrat et de taches nutritionnelles 26,29,30,31,32. Les jeunes parties du jardin ont tendance à être plus foncées en raison du substrat fraîchement incorporé après avoir été fragmenté. Ces substrats récemment ajoutés sont souvent colonisés par les bords, qui ont été coupés par les ouvrières des fourmis et inoculés avec des touffes de mycélium. Rayonnant à partir des bords coupés, des hyphes fongiques se répandent sur le substrat 29,32,33. L’abondance des hyphes augmente à mesure que la dégradation du substrat progresse, ce qui entraîne des régions blanchâtres et métaboliquement actives 30,31,32. Les régions plus anciennes, avec un substrat plus dégradé et un microbiote abondant 29,32, ont tendance à présenter des tons brunâtres et une humidité plus élevée. Les ouvrières enlèvent des fragments de cette région, les séparant en tas de déchets, où elles prennent également des substrats qui nuisent au symbiote fongique 34,35,36. Les tas de déchets, bien que physiquement détachés du jardin, sont un point de dégradation continue du substrat et de cycle des nutriments par le microbiote abondant des habitants 29,32,37,38,39.
Un microbiote principalement composé d’Enterobacter, Klebsiella, Pantoea, Pseudomonas et Serratia, habite également le jardin, apparemment partagé par divers systèmes de fongiculture de l’attine. Codant pour des voies métaboliques qui pourraient compléter le métabolisme fongique, le microbiote participe potentiellement aux réponses physiologiques du jardin 40,41,42,43,44. Non seulement les données métagénomiques indiquaient que le microbiote était là41,42, mais l’analyse par microscopie électronique à balayage (MEB) de la fungiculture des fourmis coupeuses de feuilles a montré principalement des bactéries en forme de bâtonnet sur le substrat végétal32. Bien que les bactéries (y compris les souches cellulolytiques) aient été isolées de l’ensemble du jardin, elles n’ont été visualisées que dans les parties les plus anciennes du jardin et dans les tas de déchets, ainsi que dans la pastille initiale transportée par les reines fondatrices29,32. Il n’était pas non plus certain que le microbiote puisse former des biofilms in vivo (c’est-à-dire dans le jardin et les déchets), comme le suggère leur capacité métabolique42 et observée in vitro44.
Ici, nous avons utilisé le SEM pour mieux comprendre l’organisation spatiale du microbiote dans les régions du jardin, en détaillant les interactions physiques microbiote-substrat et microbiote-hyphes. En fournissant des images avec une plus grande profondeur focale, le MEB permet d’observer des structures microscopiques tridimensionnelles à haute résolution, ce qui permet une analyse approfondie des modèles spatiaux du microbiote du jardin. Nous détaillons les étapes de fixation, de déshydratation, de séchage, de pulvérisation et d’imagerie de ces échantillons hétérogènes et délicats à base de champignons. En supprimant l’étape de postfixation à l’aide de tétroxyde d’osmium (OsO4) et en réduisant le temps de déshydratation, nous avons simplifié les protocoles 32,33,45 de préparation des échantillons de jardin et de déchets pour l’analyse MEB. Ce protocole adapté préserve les motifs structurels des hyphes, ainsi que l’organisation spatiale du microbiote et du biofilm, et pourrait être appliqué à d’autres écosystèmes microbiens délicats et aux biofilms.
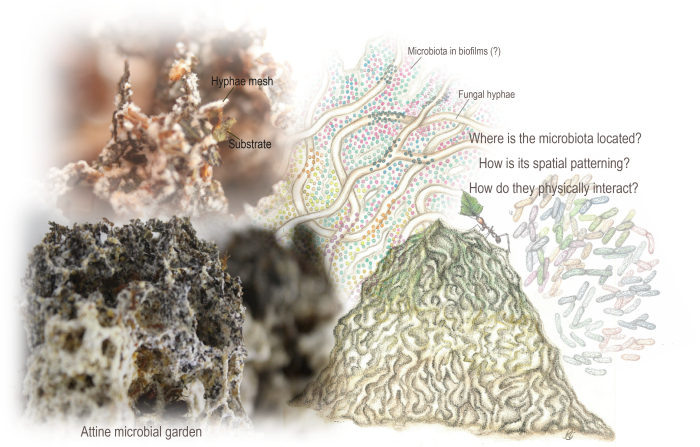
Figure 1 : Jardins microbiens d’Attine. Le jardin est une structure semblable à une éponge résultant d’un maillage entrelacé d’hyphes qui se développe en métabolisant le substrat principalement végétal incorporé par les fourmis. Le jardin est également habité par le microbiote, qui code pour des voies métaboliques qui pourraient compléter le métabolisme fongique. Les données métagénomiques et les analyses antérieures en microscopie électronique à balayage ont indiqué sa présence, bien que nous ayons une connaissance limitée de son organisation spatiale et de ses interactions physiques avec le substrat et les hyphes fongiques. Nous avons utilisé le SEM pour dévoiler l’organisation spatiale et la structuration du microbiote et du biofilm. Illustrations de Mariana Barcoto (jardin et microbiote adaptés de Barcoto et Rodrigues 94), et photos de Mariana Barcoto et Enzo Sorrentino. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Échantillonnage des colonies de terrain
REMARQUE : Lorsque vous collectez des colonies de fourmis, certifiez que toutes les autorisations requises par la législation locale sont obtenues avant la collecte. Dans notre cas, le permis de collection #74585 a été délivré par l’Instituto Chico Mendes de Conservação e Biodiversidade (ICMBio). Lorsque les échantillons proviennent d’une colonie de laboratoire, passez à la section 2.
- Localisez et marquez la colonie. Creuser une tranchée autour de la zone du nid jusqu’à ce que la chambre de jardin soit exposée (figure 2A).
REMARQUE : Certaines espèces d’attines peuvent construire leurs colonies sous la litière de feuilles ou à l’intérieur de bûches pourries. Dans de tels cas, tournez soigneusement la litière ou cassez soigneusement les bûches pour recueillir les échantillons. Pour des informations détaillées sur la localisation, la collecte et le maintien de colonies vivantes de diverses espèces de fourmis attines, voir Sosa-Calvo et al.46. - Ouvrez la chambre de jardin latéralement pour éviter que la terre ne tombe sur la surface du jardin. Prélevez soigneusement des échantillons de jardin à l’aide d’une pince entomologique, d’une cuillère ou d’une écumoire de cuisine, selon la taille du jardin.
REMARQUE : Assurez-vous de stériliser les outils avant de les collecter. Lorsque vous ramassez des fourmis coupeuses de feuilles, portez des gants en tissu épais pour prévenir (ou au moins atténuer) les morsures des ouvrières. Pour les autres espèces d’attines, les gants en tissu sont facultatifs. - Transférez les échantillons de jardin dans un récipient en plastique propre contenant une couche de plâtre pour équilibrer l’humidité du jardin. Après avoir transféré les ouvrières du jardin et des fourmis, fermez hermétiquement le récipient pour éviter le séchage de l’échantillon. Conservez les échantillons de jardin à 23-25 °C jusqu’au traitement.
- Fermez la tranchée avec de la terre préalablement enlevée.
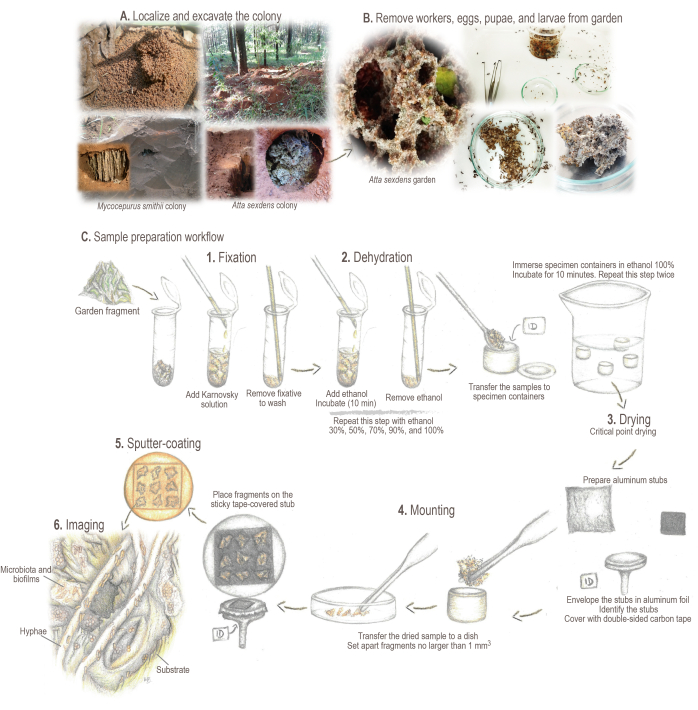
Figure 2 : Protocole de préparation des échantillons. (A) Échantillonnage des colonies de terrain. (B) Traitement des échantillons. (C) Principes fondamentaux et flux de travail pour la préparation des échantillons : 1. Fixation : pour renforcer et préserver la structure de l’échantillon. 2. Déshydratation : la teneur en eau des échantillons est remplacée par de l’éthanol. 3. Séchage au point critique : leCO2 liquide remplace l’éthanol et s’évapore. 4. Montage : échantillon affiché pour analyse. 5. Revêtement par pulvérisation cathodique avec de l’or : empêcher le chargement de l’échantillon. 6. L’imagerie. Illustrations et photos de Mariana Barcoto. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Réactifs
REMARQUE : Gardez à l’esprit que les solutions suivantes doivent être préparées à l’avance.
- Préparez un tampon de cacodylate de sodium de 0,2 M. Pour cela, dissoudre 42,8 g de cacodylate de sodium dans 800 mL d’eau distillée, en remuant jusqu’à dissolution et stabilisation du pH à 7,2 (si nécessaire, ajuster le pH à l’aide d’acide chlorhydrique). Augmentez le volume de la solution à 1 L en ajoutant de l’eau distillée. Conservez la solution à 4 °C (pendant ~1 mois).
ATTENTION : Le glutaraldéhyde et le paraformaldéhyde sont toxiques et doivent être manipulés dans une hotte. Portez des gants en nitrile et des lunettes de protection tout le temps lorsque vous manipulez de tels réactifs. - Préparez le fixateur de Karnovsky (modifié de Karnovsky)47. Pour cela, ajouter 10 ml de solution aqueuse de glutaraldéhyde à 25 % et 10 ml de solution aqueuse de paraformaldéhyde à 20 % à 25 ml de tampon de cacodylate de sodium à 0,2 M et mélanger. Ajouter 1 mL de chlorure de calcium 0,1 M (CaCl2) et augmenter le volume à 100 mL en ajoutant de l’eau distillée. Conservez la solution à 4 °C (jusqu’à 1 mois).
ATTENTION : Le fixateur de Karnovsky est nocif lorsqu’il est inhalé et peut provoquer une irritation de la peau et des yeux. Ainsi, évitez de respirer des vapeurs, en utilisant uniquement à l’extérieur ou dans un endroit bien ventilé. Portez des gants en nitrile et des lunettes de protection tout le temps lorsque vous manipulez la solution. - À l’aide d’éthanol absolu (qualité analytique), préparer des solutions d’éthanol à 30 %, 50 %, 70 % et 90 % dans de l’eau distillée ; 100 % d’éthanol est également requis.
3. Fixation de l’échantillon
REMARQUE : Les fixateurs durcissent et préservent les échantillons, en maintenant les caractéristiques morphologiques. Les aldéhydes (tels que le paraformaldéhyde et le glutaraldéhyde) sont des fixateurs non coagulants de type réticulation, induisant des réticulations à l’intérieur et entre les protéines et les acides nucléiques48.
- Prélever les ouvrières, les œufs, les pupes et les larves des échantillons de jardin à l’aide d’une pince entomologique (figure 2B). Écartez les fragments de jardin ne dépassant pas 5 mm3. Ajouter les fragments dans un tube de 2 mL (figure 2C).
- Dans les tubes contenant les échantillons, utilisez une pipette en verre Pasteur pour ajouter ~1 mL de solution fixatrice Karnovsky (assurez-vous que l’échantillon est complètement couvert). Mélanger en agitant doucement pour aider l’échantillon à tremper et incuber à 4 °C pendant au moins 24 h avant de poursuivre le traitement de l’échantillon (figure 2C.1).
REMARQUE : Lorsque les étapes de déshydratation suivantes ne sont pas effectuées immédiatement après la fixation, le protocole peut être interrompu à cette étape et les échantillons peuvent être stockés pendant ~1 an à 4 °C.
Nous vous suggérons d’utiliser une pipette en verre Pasteur, car elle est composée d’un matériau inerte et est plus facile à nettoyer pour être réutilisée par la suite. Le matériau du jardin est très hydrophobe, ayant tendance à flotter à la surface de la solution fixatrice. Il faut généralement jusqu’à 5 minutes pour être complètement trempé dans le fixateur. Assurez-vous que le fixateur recouvre les fragments de jardin, car son volume a tendance à être réduit lorsqu’il pénètre dans les pores de l’échantillon. Nous avons vérifié empiriquement qu’une fois que l’échantillon a été trempé et est devenu humide, ses composants (en particulier, le délicat mycélium fongique) sont devenus susceptibles de se briser lorsqu’ils sont mélangés davantage. Ainsi, nous vous recommandons d’éviter autant que possible de secouer les échantillons.
4. Déshydratation de l’échantillon
REMARQUE : La série de lavage à l’éthanol remplace progressivement l’eau des échantillons par de l’éthanol. Il est important de commencer avec une solution d’éthanol à faible concentration (voir ci-dessous) pour éviter d’endommager excessivement ou de faire s’effondrer des échantillons aussi délicats49.
- Retirer complètement la solution fixatrice de Karnovsky à l’aide d’une pipette en verre, en prenant soin de ne pas perturber l’échantillon (Figure 2C.1).
REMARQUE : Jetez le fixateur de Karnovsky dans un récipient correctement étiqueté pour la gestion des résidus chimiques toxiques. - Immédiatement après avoir retiré le fixateur, ajoutez 1 mL d’éthanol à 30 %, en prenant soin de ne pas perturber l’échantillon, et incubez pendant 10 min à température ambiante (figure 2C.2).
REMARQUE : Les échantillons doivent toujours être trempés dans la solution. Assurez-vous de remplacer rapidement les solutions pendant la série de lavage progressif à l’éthanol. Comme le jardin perd son aspect poreux et s’agrège au fond du tube, 1 mL d’éthanol a tendance à suffire à recouvrir l’échantillon (lorsque l’échantillon ne dépasse pas 5mm3). Cependant, si les échantillons ne sont pas complètement recouverts, ajoutez de l’éthanol jusqu’à ce que l’échantillon soit complètement recouvert. - Retirez complètement l’éthanol à 30 % à l’aide d’une pipette en verre, en faisant attention de ne pas perturber l’échantillon. Jetez correctement l’éthanol à 30 %.
REMARQUE : Pour toute la série de lavage progressif à l’éthanol, jetez l’éthanol dans un récipient correctement étiqueté pour la gestion des résidus chimiques toxiques. - Ajouter 1 mL d’éthanol à 50 % et incuber pendant 10 min à température ambiante. Retirez complètement l’éthanol à 50 % à l’aide d’une pipette en verre, en faisant attention de ne pas perturber l’échantillon. Jetez correctement l’éthanol à 50 %.
- Ajouter 1 mL d’éthanol à 70 % et incuber pendant 10 minutes à température ambiante. Retirez complètement l’éthanol à 70 % à l’aide d’une pipette en verre, en faisant attention de ne pas perturber l’échantillon. Jetez correctement l’éthanol à 70 %.
REMARQUE : À l’étape de lavage à 70 % d’éthanol, l’utilisateur peut interrompre le protocole si nécessaire, car les tubes d’échantillon peuvent être stockés pendant la nuit à 4 °C, lorsque le matériau ne sera pas immédiatement traité. - Ajouter 1 mL d’éthanol à 90 % et incuber pendant 10 minutes à température ambiante. Retirez complètement l’éthanol à 90 % à l’aide d’une pipette en verre, en faisant attention de ne pas perturber l’échantillon. Jetez correctement l’éthanol à 90 %.
- Ajouter 1 mL d’éthanol à 100 % et incuber pendant 10 min à température ambiante. Retirez complètement l’éthanol à 100 % à l’aide d’une pipette en verre, en faisant attention de ne pas perturber l’échantillon. Jetez correctement l’éthanol à 100 %.
- À l’aide d’une pince et/ou d’une spatule, transférez soigneusement les échantillons dans des récipients pour le sécheur à point critique (DPC), contenant des étiquettes d’identification d’échantillon (préalablement faites en papier et au crayon). Pour éviter le séchage des échantillons, ce transfert s’effectue avec le récipient placé dans une boîte de Pétri recouverte à 100 % d’éthanol.
- Mettez les couvercles sur les récipients et plongez-les dans un bécher en verre gradué contenant suffisamment d’éthanol pour immerger les récipients. Couvrez le bécher en verre et laissez incuber 10 min à température ambiante ; Ensuite, jetez correctement l’éthanol à 100 %.
- Transférez les récipients d’échantillons dans un autre bécher en verre gradué contenant suffisamment d’éthanol à 100 % pour immerger les récipients. Couvrez le bécher en verre et laissez incuber 10 min à température ambiante ; Ensuite, transférez les récipients d’échantillons dans le séchoir à point critique.
REMARQUE : Après la série de lavage, les pipettes et les béchers en verre doivent être abondamment rincés à l’eau distillée, et cette eau résiduelle doit être jetée dans un récipient correctement étiqueté pour la gestion des résidus chimiques toxiques. Après le rinçage, les articles en verre peuvent être lavés avec un détergent neutre, rincés à l’eau du robinet et séchés à l’air.
5. Séchage du point critique
REMARQUE : Un sécheur à point critique remplace l’éthanol dans les échantillons par du dioxyde de carbone liquide (CO2), qui s’évapore de l’échantillon à une température et une pression plus élevées. Veuillez suivre les instructions du fabricant pour de telles procédures.
- Allumez l’équipement.
- Ouvrez la chambre, placez les récipients d’échantillons à l’intérieur et ajoutez de l’éthanol à 100 % jusqu’à ce qu’il recouvre les contenants. Fermez la chambre.
- Activez l’option Refroidir et attendez que la température atteigne 10 °C.
- Ouvrez le robinet de la bouteille de CO2 et activez l’option Agitateur .
- Activez l’option CO2 in , en vérifiant toujours la chambre pour vérifier la quantité de CO2 qui l’a déjà remplie. Lorsque la chambre est presque remplie, désactivez l’option CO2 in et activez l’option d’échange , en la gardant activée jusqu’à ce qu’il y ait suffisamment de CO2 pour couvrir les conteneurs. Assurez-vous que les conteneurs sont toujours couverts de CO2 (c’est-à-dire désactiver l’option d’échange avant que tout le CO2 n’ait quitté la chambre). Répétez l’étape 5.5 6x.
- Activez une dernière fois le CO2 et remplissez la chambre jusqu’à ce que les récipients soient recouverts.
- Activez l’option Chaleur et désactivez l’option Agitateur . Fermez le robinet de la bouteille de CO2.
- Attendez que la température monte à 35 °C ; ensuite, activez l’option Sortie de gaz .
REMARQUE : À environ 30 °C, la chambre atteint des pressions de 70-80 bars, atteignant le point critique, où le liquide disparaît. - Lorsque la pression de la chambre atteint 1 bar, tout le contenu en gaz a été éliminé. Ouvrez la chambre et retirez les contenants.
- Éteignez l’équipement.
6. Montage
- Préparer les supports d’échantillons MEB (c.-à-d. talons d’aluminium ; Figure 2C.4).
- Enveloppez les talons avec un morceau de papier d’aluminium, ne couvrant que le dessus, pour faciliter le nettoyage des talons après l’analyse.
- Identifiez les talons en inscrivant le code/numéro d’échantillon sur le fond du support, garantissant l’identification de ce qui est placé sur le dessus.
- Couvrez la partie supérieure avec du ruban carbone double face. Placez les talons dans un porte-échantillons.
- Ouvrez le couvercle du récipient de l’échantillon et transférez soigneusement l’échantillon séché dans une boîte de Pétri en verre à l’aide d’une pince et d’une spatule.
REMARQUE : Les jardins séchés aux points critiques ont tendance à s’agglomérer en formant des échantillons très compacts qui doivent être soigneusement séparés en fragments ne dépassant pas 1 mm3. - Placez soigneusement les fragments sur la surface collante du talon recouvert de ruban adhésif. Une fois que le fragment de jardin touche le ruban, il est très difficile de le (re)déplacer, alors attention à ne pas le placer dans des endroits ou des positions indésirables. Ajoutez jusqu’à neuf fragments par talon.
- Répétez les étapes 6.2 et 6.3 pour chaque échantillon.
7. Revêtement par pulvérisation cathodique avec de l’or
REMARQUE : L’enrobage de l’échantillon est nécessaire pour éviter sa charge. Suivez les instructions du fabricant pour régler les paramètres tels que la pression de gaz de fonctionnement (0,5 × 10-1 mm Hg de pression de gaz dans ce protocole), le temps de pulvérisation (220 s), l’épaisseur de la couche d’or (~120 Å), le courant (50 mA) et la tension d’alimentation. La pulvérisation cathodique a tendance à suivre un flux de travail commun, bien que les équipements de différents fabricants puissent fonctionner légèrement différemment.
- Ouvrez le bras cible articulé et placez les embouts sur la table d’échantillons.
- Fermez le bras cible articulé et vérifiez si le pare-éclats de la chambre à vide en verre est correctement intégré.
- Ouvrez le robinet de la bouteille d’argon et allumez l’interrupteur d’alimentation principal.
- Suivez la montée du vide sur l’écran de l’équipement jusqu’à ce qu’il atteigne la marque 0,5 × 10-1 mm Hg sur l’écran ; ensuite, activez le rinçage. Répétez l’opération 5 fois.
- Allumez le système de circulation d’eau, activez l’option HV On et ouvrez le couvercle en film doré. Vérifiez que la couleur du plasma est rosâtre. Pour suivre ce protocole, mettez en place 220 s de pulvérisation avec une tension de 50 mA, qui déposera une couche d’or de ~120 Å (12 nm).
- HV On est automatiquement désactivé. Éteignez le système de circulation d’eau et fermez le couvercle en film doré.
- Éteignez l’interrupteur d’alimentation principal, permettant à l’air de pénétrer dans la chambre à vide. Pour les échantillons de jardin et de déchets, répétez les étapes 7.1 à 7.7 3 fois.
REMARQUE : Si l’imagerie n’a pas lieu immédiatement après la préparation de l’échantillon, conservez les talons dans un récipient hermétique rempli d’une couche de silice pour éviter de réhydrater les échantillons.
8. Imagerie
REMARQUE : Suivez les instructions du fabricant pour régler les paramètres SEM, tels que le diamètre de l’ouverture de l’objectif, la tension de fonctionnement, l’alignement du système de faisceau d’électrons, l’alignement axial et les stigmateurs.
- Placez les talons dans le porte-échantillon, en prenant des notes sur la position de chaque échantillon.
REMARQUE : Portez des gants lors de l’insertion ou du retrait du porte-échantillon et gardez le support aussi propre que possible. - Lancez le logiciel d’exploitation à partir du poste de travail.
- Sélectionnez les paramètres de l’instrument. Visualisez des échantillons de jardin avec un diamètre d’ouverture d’objectif de 30 μm (c’est-à-dire dans la deuxième étape), fonctionnant dans le vide poussé, détectant des signaux d’électrons secondaires (SED), accélérant la tension de 20 kV, distance de travail de 15 à 20 mm, courant de sonde de 40,0 (en mode courant élevé) et faisant varier le grossissement en fonction de l’échantillon.
- En suivant les instructions de navigation, appuyez sur l’icône Vent et attendez que la chambre de l’échantillon soit ventilée. Une barre de progression indique l’état du vide.
- Lorsque la pression atmosphérique est atteinte, ouvrez la chambre de l’échantillon et insérez soigneusement le porte-échantillon.
- Fermez doucement la porte de la chambre et appuyez sur l’icône Evac pour évacuer l’instrument, en suivant l’état du vide via la barre de progression. Le système de navigation affichera la position de la platine en mouvement et fournira un graphique du support lorsque le mouvement est terminé. Appuyez sur l’icône de l’appareil photo pour enregistrer une photo du support afin d’obtenir une vue de dessus qui vous aidera à naviguer entre les échantillons pendant l’imagerie.
- Appuyez sur l’icône d’observation pour allumer le canon à électrons et attendez que l’image se forme. Utilisez l’interface utilisateur manuelle (commande manuelle) pour déplacer manuellement l’axe Z à la bonne hauteur (en fonction de la hauteur de l’échantillon et de la distance de travail déterminée).
REMARQUE : Pour prolonger la durée de vie du filament de tungstène, mettez en pause la fonction d’observation chaque fois que vous n’imagez pas activement. - Utilisez les options affichées à l’écran ou la commande manuelle pour modifier la fréquence raster et la position de l’image. Déplacez la platine pour obtenir une vue complète de l’échantillon, en utilisant la fonction RDC pour vous concentrer sur des zones spécifiques. Lorsque vous observez une cible (ou une structure intéressante), ajustez le grossissement, la mise au point, la luminosité, le contraste et la stigmatisation en conséquence. Corrigez la stigmatisation en utilisant l’interface utilisateur manuelle pour déplacer la scène dans les directions X et Y.
- Ajustez le grossissement entre 100x et 700x pour visualiser les aspects généraux du jardin (tels que la densité des hyphes, le substrat et les modèles de colonisation).
- Ajustez le grossissement entre 700x et 1 500x pour visualiser les modèles spatiaux du microbiote.
- Ajustez le grossissement entre 1 500x et 3 000x pour observer les interactions physiques du microbiote et du biofilm.
- Ajustez le grossissement entre 3 000x et 4 000x pour vous concentrer sur des amas microbiens spécifiques.
- Pour enregistrer une image, utilisez la fonction Figer , cliquez sur l’icône Photo et configurez le chemin d’accès au fichier. En standard, analysez au moins trois fragments de jardin, en imageant chacun d’eux dans toutes les plages de grossissement mentionnées à l’étape 8.8. Cela se traduit par au moins 12 images par échantillon, bien que plus d’images puissent prendre en charge des descriptions plus détaillées. Nous suggérons que 15 à 25 images par échantillon, couvrant tous les grossissements de l’étape 8.8, tendent à fournir des détails fins pour la description de l’échantillon.
REMARQUE : La mise en tramage et l’enregistrement d’une image prennent quelques secondes pendant lesquelles toute vibration sur la table flottante SEM doit être évitée. - Une fois l’imagerie terminée, appuyez sur l’option Vent pour ventiler la chambre, en suivant l’état du vide à travers la barre de progression. Retirez délicatement le support, fermez doucement la porte de la chambre et appuyez sur Evac pour évacuer la chambre.
- Dès que l’évacuation est terminée, quittez le logiciel d’exploitation.
- Ajustez la luminosité et le contraste pour améliorer la visualisation à l’aide d’un éditeur d’images.
Access restricted. Please log in or start a trial to view this content.
Résultats
Ici, nous avons présenté un protocole simplifié pour visualiser les composants du jardin d’attine et des échantillons de déchets, tels que les hyphes fongiques, le substrat, le microbiote et les biofilms. La MEB a amélioré notre compréhension de la façon dont le jardin et les déchets échafaudent les modèles structurels du microbiote (Figure 3). Dans les jardins d’attines, les hyphes fongiques sont des structures en forme de branches couvrant ...
Access restricted. Please log in or start a trial to view this content.
Discussion
Le MEB utilise un faisceau d’électrons pour balayer l’échantillon, générant une image agrandie de celui-ci de sorte que l’on peut visualiser des microstructures tridimensionnelles en haute résolution. Comme le MEB fonctionne sous vide poussé, il est nécessaire d’éliminer jusqu’à 99 % de l’eau des échantillons. À l’intérieur de la chambre à vide du MEB, les échantillons partiellement hydratés peuvent se déshydrater et s’effondrer, en plus de disperser les ?...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs tiennent à remercier la Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP) pour son soutien financier (Subvention #2019/03746-0). Remerciements du MOB pour la bourse de doctorat reçue de la FAPESP (processus 2021/08013-0) et du Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - Brésil (CAPES) - Code des finances 001. AR remercie également le Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) pour une bourse de recherche (#305269/2018). Les auteurs tiennent à remercier Marcia Regina de Moura Aouada et Antonio Teruyoshi Yabuki pour leur aide lors des essais pilotes de préparation des échantillons, Renato Barbosa Salaroli pour son assistance technique et Enzo Sorrentino pour son aide lors de la prise de photos. Cette étude a été réalisée dans le cadre de l’autorisation d’accès au patrimoine génétique #SISGen AA39A6D.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 2 mL tube | Axygen | MCT-200-C-BRA | To fix and dehydrate samples |
| Calcium chloride anhydrous | Merck | C4901 | CaCl2 anhydrous to prepare Karnovsky’s fixative |
| Critical point dryer | Leica | EM CPD 300 | For critical point drying |
| Double Sided Carbon Conductive Tape, 12 mm (W) X 5 M (L) | Electron Microscopy Sciences | 77819-12 | For mounting samples |
| Entomological forceps | No specific supplier | To manipulate garden samples | |
| Ethyl alcohol (=ethanol), pure (≥99.5%) | Sigma-Aldrich | 459836 | For dehydration |
| Forceps | No specific supplier | To manipulate garden samples | |
| Glass beaker | No specific supplier | For dehydration | |
| Glass Petri dish | No specific supplier | To manipulate garden samples | |
| Glass pipette | No specific supplier | To fix and dehydrate samples | |
| Glutaraldehyde (Aqueous Glutaraldehyde EM Grade 25%) | Electron Microscopy Sciences | 16220 | To prepare Karnovsky’s fixative |
| Gold target | Ted Pella, Inc. | 8071 | To sputter coat with gold |
| Hydrochloric acid | Sigma-Aldrich | 320331 | For adjusting solutions pH |
| Image editor | Photoshop | any version | To adjust images |
| Paraformaldehyde (Paraformaldehyde 20% Aqueous Solution EM Grade) | Electron Microscopy Sciences | 15713 | To prepare Karnovsky’s fixative |
| Propilene recipient | No specific supplier | For maintaining alive ant colonies | |
| Scanning Electron Microscope | JEOL | IT300 SEM | For sample imaging |
| Sodium cacodylate trihydrate | Sigma-Aldrich | C0250 | For preparing sodium cacodylate buffer |
| Spatula | No specific supplier | To manipulate garden samples | |
| Specimen containers with 15 mm dia. x 10 mm high | Ted Pella, Inc. | 4591 | For critical point drying |
| Sputter coater | Baltec | SCD 050 | To coat with gold |
| Stub (Aluminium mount, flat end pin) 12.7 mm x 8 mm | Electron Microscopy Sciences | 75520 | For mounting samples |
Références
- Turner, M. G. Landscape ecology: the effect of pattern on process. Annu Rev Ecol Evol Syst. 20 (1), 171-197 (1989).
- Rietkerk, M., Van de Koppel, J. Regular pattern formation in real ecosystems. Trends Ecol Evol. 23 (3), 169-175 (2008).
- Schmitz, O. J. Spatial dynamics and ecosystem functioning. PLOS Biol. 8 (5), e1000378(2010).
- Pringle, R. M., Doak, D. F., Brody, A. K., Jocqué, R., Palmer, T. M. Spatial pattern enhances ecosystem functioning in an African savanna. PLOS Biol. 8 (5), e1000377(2010).
- Wimpenny, J. W. Spatial order in microbial ecosystems. Biol Rev. 56 (3), 295-342 (1981).
- Martiny, J. B. H., et al. Microbial biogeography: putting microorganisms on the map. Nat Rev. Microbiol. 4 (2), 102-112 (2006).
- McCallum, G., Tropini, C. The gut microbiota and its biogeography. Nat Rev Microbiol. 22 (2), 105-118 (2024).
- Lamont, R. J., Hajishengallis, G., Koo, H. Social networking at the microbiome-host interface. Infec Immun. 91 (9), e00124-e00223 (2023).
- Welch, J. L. M., Rossetti, B. J., Rieken, C. W., Dewhirst, F. E., Borisy, G. G. Biogeography of a human oral microbiome at the micron scale. Proc Natl Acad Sci USA. 113 (6), E791-E800 (2016).
- Kim, D., et al. Spatial mapping of polymicrobial communities reveals a precise biogeography associated with human dental caries. Proc Natl Acad Sci USA. 117 (22), 12375-12386 (2020).
- Swidsinski, A., Weber, J., Loening-Baucke, V., Hale, L. P., Lochs, H. Spatial organization and composition of the mucosal flora in patients with inflammatory bowel disease. J Clin Microbiol. 43 (7), 3380-3389 (2005).
- Ibberson, C. B., Barraza, J. P., Holmes, A. L., Cao, P., Whiteley, M. Precise spatial structure impacts antimicrobial susceptibility of S. aureus in polymicrobial wound infections. Proc Natl Acad Sci USA. 119 (51), e2212340119(2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Dejea, C. M., et al. Microbiota organization is a distinct feature of proximal colorectal cancers. Proc Natl Acad Sci USA. 111 (51), 18321-18326 (2014).
- Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., Martiny, J. B. Beyond biogeographic patterns: processes shaping the microbial landscape. Nat Rev Microbiol. 10 (7), 497-506 (2012).
- Adade, E. E., Al Lakhen, K., Lemus, A. A., Valm, A. M. Recent progress in analyzing the spatial structure of the human microbiome: Distinguishing biogeography and architecture in the oral and gut communities. Curr Opin Endocr. 18, 275-283 (2021).
- Mony, C., Bohannan, B. J., Leibold, M. A., Peay, K., Vandenkoornhuyse, P. Microbial landscape ecology: Highlights on the invisible corridors. Front Ecol Evol. 9, 753213(2021).
- Hölldobler, B., Wilson, E. O. The Ants. , Harvard University Press. (1990).
- Chapela, I. H., Rehner, S. A., Schultz, T. R., Mueller, U. G. Evolutionary history of the symbiosis between fungus-growing ants and their fungi. Science. 266 (5191), 1691-1694 (1994).
- Mueller, U. G., Rehner, S. A., Schultz, T. R. The evolution of agriculture in ants. Science. 281 (5385), 2034-2038 (1998).
- Mueller, U. G., Gerardo, N. M., Aanen, D. K., Six, D. L., Schultz, T. R. The evolution of agriculture in insects. Annu Rev Ecol Evol Syst. 36, 563-595 (2005).
- Dentinger, B. T., Lodge, D. J., Munkacsi, A. B., Desjardin, D. E., McLaughlin, D. J. Phylogenetic placement of an unusual coral mushroom challenges the classic hypothesis of strict coevolution in the Apterostigma pilosum group ant-fungus mutualism. Evolution. 63 (8), 2172-2178 (2009).
- Schultz, T. R., Brady, S. G. Major evolutionary transitions in ant agriculture. Proc Natl Acad Sci USA. 105, 5435-5440 (2008).
- de Fine Licht, H. H., Boomsma, J. J. Forage collection, substrate preparation, and diet composition in fungus-growing ants. Ecol Entomol. 35 (3), 259-269 (2010).
- Martin, M. M. The biochemical basis of the fungus-attine ant symbiosis: A complex symbiosis is based upon integration of the carbon and nitrogen metabolisms of the two organisms. Science. 169 (3940), 16-20 (1970).
- Grell, M. N., et al. The fungal symbiont of Acromyrmex leaf-cutting ants expresses the full spectrum of genes to degrade cellulose and other plant cell wall polysaccharides. BMC Genomics. 14, 928(2013).
- Lange, L., Grell, M. N. The prominent role of fungi and fungal enzymes in the ant-fungus biomass conversion symbiosis. Appl Microbiol Biotechnol. 98, 4839-4851 (2014).
- Huang, E. L., et al. The fungus gardens of leaf-cutter ants undergo a distinct physiological transition during biomass degradation. Environ Microbiol Rep. 6 (4), 389-395 (2014).
- Craven, S. E., Dix, M. W., Michaels, G. E. Attine fungus gardens contain yeasts. Science. 169 (3941), 184-186 (1970).
- De Fine Licht, H. H., Boomsma, J. J., Tunlid, A. Symbiotic adaptations in the fungal cultivar of leaf-cutting ants. Nat Commun. 5 (1), 5675(2014).
- Aylward, F. O., et al. Enrichment and broad representation of plant biomass-degrading enzymes in the specialized hyphal swellings of Leucoagaricus gongylophorus, the fungal symbiont of leaf-cutter ants. PLoS One. 10 (8), e0134752(2015).
- Moreira-Soto, R. D., Sanchez, E., Currie, C. R., Pinto-Tomás, A. A. Ultrastructural and microbial analyses of cellulose degradation in leaf-cutter ant colonies. Microbiology. 163 (11), 1578-1589 (2017).
- Erthal Jr, M., Silva, C. P., Cooper, R. M., Samuels, R. I. Hydrolytic enzymes of leaf-cutting ant fungi. Comp Biochem. 152 (1), 54-59 (2009).
- North, R. D., Jackson, C. W., Howse, P. E. Communication between the fungus garden and workers of the leaf-cutting ant, Atta sexdens rubropilosa, regarding choice of substrate for the fungus. Physiol Entomol. 24 (2), 127-133 (1999).
- Herz, H., Hölldobler, B., Roces, F. Delayed rejection in a leaf-cutting ant after foraging on plants unsuitable for the symbiotic fungus. Behav Ecol. 19 (3), 575-582 (2008).
- Schiøtt, M., De Fine Licht, H. H., Lange, L., Boomsma, J. J. Towards a molecular understanding of symbiont function: identification of a fungal gene for the degradation of xylan in the fungus gardens of leaf-cutting ants. BMC Microbiol. 8, 40(2008).
- Bot, A. N., Currie, C. R., Hart, A. G., Boomsma, J. J. Waste management in leaf-cutting ants. Ethol Ecol Evol. 13 (3), 225-237 (2001).
- Scott, J. J., et al. Microbial community structure of leaf-cutter ant fungus gardens and refuse dumps. PLOS One. 5 (3), e9922(2010).
- Lewin, G. R., et al. Cellulose-enriched microbial communities from leaf-cutter ant (Atta colombica) refuse dumps vary in taxonomic composition and degradation ability. PLOS One. 11 (3), e0151840(2016).
- Suen, G., et al. An insect herbivore microbiome with high plant biomass-degrading capacity. PLOS Genet. 6 (9), e1001129(2010).
- Aylward, F. O., et al. Convergent bacterial microbiotas in the fungal agricultural systems of insects. MBio. 5 (6), e02077(2014).
- Barcoto, M. O., et al. Fungus-growing insects host a distinctive microbiota apparently adapted to the fungiculture environment. Sci Rep. 10 (1), 12384(2020).
- Francoeur, C. B., et al. Bacteria contribute to plant secondary compound degradation in a generalist herbivore system. mBio. 11, e02146-e02220 (2020).
- Martiarena, M. J. S., Deveau, A., Montoya, Q. V., Flórez, L. V., Rodrigues, A. The hyphosphere of leaf-cutting ant cultivars is enriched with helper bacteria. Microb Ecol. 86 (3), 1773-1788 (2023).
- Leal-Dutra, C. A., et al. Evidence that the domesticated fungus Leucoagaricus gongylophorus recycles its cytoplasmic contents as nutritional rewards to feed its leafcutter ant farmers. IMA Fungus. 14 (1), 19(2023).
- Sosa-Calvo, J., Jesovnik, A., Okonski, E., Schultz, T. R. Locating, collecting, and maintaining colonies of fungus-farming ants (Hymenoptera: Myrmicinae: Attini). Sociobiology. 62 (2), 300-320 (2015).
- Karnovsky, M. J. A formaldehyde glutaraldehyde fixative of high osmolality for use in electron microscopy. J Cell Biol. 27, 137-138 (1965).
- Eltoum, I., Fredenburgh, J., Myers, R. B., Grizzle, W. E. Introduction to the theory and practice of fixation of tissues. J Histotechnol. 24 (3), 173-190 (2001).
- Gusnard, D., Kirschner, R. H. Cell and organelle shrinkage during preparation for scanning electron microscopy: effects of fixation, dehydration and critical point drying. J Microsc. 110 (1), 51-57 (1977).
- Surman, S. B., et al. Comparison of microscope techniques for the examination of biofilms. J Microbiol Methods. 25 (1), 57-70 (1996).
- Augustin, J. O., et al. Yet more "weeds" in the garden: fungal novelties from nests of leaf-cutting ants. PLOS One. 8 (12), e82265(2013).
- Montoya, Q. V., Martiarena, M. J. S., Polezel, D. A., Rodrigues, A. More pieces to a huge puzzle: Two new Escovopsis species from fungus gardens of attine ants. MycoKeys. 46, 97(2019).
- Varanda-Haifig, S. S., et al. Nature of the interactions between hypocrealean fungi and the mutualistic fungus of leaf-cutter ants. Antonie van Leeuwenhoek. 110, 593-605 (2017).
- Custodio, B. C., Rodrigues, A. Escovopsis kreiselii specialization to its native hosts in the fungiculture of the lower attine ant Mycetophylax morschi. Antonie van Leeuwenhoek. 112, 305-317 (2019).
- Schröttner, H., Schmied, M., Scherer, S. Comparison of 3D surface reconstruction data from certified depth standards obtained by SEM and an Infinite Focus Measurement Machine (IFM). Microchim Acta. 155, 279-284 (2006).
- Zhou, W., Apkarian, R., Wang, Z. L., Joy, D. Fundamentals of scanning electron microscopy (SEM). Scanning microscopy for nanotechnology: Techniques and applications. Zhou, W., Wang, Z. L. , Springer. New York, NY. 1-40 (2007).
- Kannan, M. Scanning electron microscopy: Principle, components and applications. A Textbook on Fundamentals and Applications of Nanotechnology. Raja, K., Subramanian, K. S., Kannan, M. , Daya Publishing House. 81-92 (2018).
- Ruffolo, J. J. Jr Critical point drying of protozoan cells and other biological specimens for scanning electron microscopy: apparatus and methods of specimen preparation. Trans Am Microsc. 93 (1), 124-131 (1974).
- Echlin, P. Recent advances in specimen coating techniques for electron microscopy. Scanning Electron Microscopy 1981/1. , IITRI. Chicago. 79-90 (1981).
- Ris, H. The cytoplasmic filament system in critical point-dried whole mounts and plastic-embedded sections. J Cell Biol. 100 (5), 1474-1487 (1985).
- Bray, D. F., Bagu, J., Koegler, P. Comparison of hexamethyldisilazane (HMDS), Peldri II, and critical-point drying methods for scanning electron microscopy of biological specimens. Microsc Res Tech. 26 (6), 489-495 (1993).
- Bergmans, L., Moisiadis, P., Van Meerbeek, B., Quirynen, M., Lambrechts, P. Microscopic observation of bacteria: review highlighting the use of environmental SEM. Int Endod J. 38 (11), 775-788 (2005).
- Kirk, S. E., Skepper, J. N., Donald, A. M. Application of environmental scanning electron microscopy to determine biological surface structure. J Microsc. 233 (2), 205-224 (2009).
- Kemmenoe, B. H., Bullock, G. R. Structure analysis of sputter-coated and ion-beam sputter-coated films: a comparative study. J Microsc. 132 (2), 153-163 (1983).
- Kinden, D. A., Brown, M. F. Technique for scanning electron microscopy of fungal structures within plant cells. Phytopathology. 65, 74-76 (1975).
- Masaphy, S., Levanon, D., Tchelet, R., Henis, Y. Scanning electron microscope studies of interactions between Agaricus bisporus (Lang) Sing hyphae and bacteria in casing soil. Appl Environ Microbiol. 53 (5), 1132-1137 (1987).
- Massicotte, H. B., Melville, L. H., Peterson, R. L. Scanning electron microscopy of ectomycorrhizae potential and limitations. Scanning Microsc. 1 (3), 58(1987).
- Visen, A., Singh, P. N., Chakraborty, B., Singh, A., Bisht, T. S. Scanning electron microscopy indicates Pseudomonad strains facilitate AMF mycorrhization in litchi (Litchi chinensis Sonn.) air-layers and improving survivability, growth and leaf nutrient status. Curr Res Microb Sci. 2, 100063(2021).
- Fleeman, R. M., Mikesh, M., Davies, B. W. Investigating Klebsiella pneumoniae biofilm preservation for scanning electron microscopy. Access Microbiol. 5 (2), 000470-000473 (2023).
- Wells, M., Mikesh, M., Gordon, V. Structure-preserving fixation allows scanning electron microscopy to reveal biofilm microstructure and interactions with immune cells. J Microsc. 293 (1), 59-68 (2024).
- Nadell, C. D., Drescher, K., Foster, K. R. Spatial structure, cooperation and competition in biofilms. Nat Rev Microbiol. 14 (9), 589-600 (2016).
- Madsen, J. S., et al. Coexistence facilitates interspecific biofilm formation in complex microbial communities. Environ Microbiol. 18 (8), 2565-2574 (2016).
- Madsen, J. S., Sørensen, S. J., Burmølle, M. Bacterial social interactions and the emergence of community-intrinsic properties. Curr Opin Microbiol. 42, 104-109 (2018).
- Flemming, H. C., Wuertz, S. Bacteria and archaea on Earth and their abundance in biofilms. Nat Rev Microbiol. 17 (4), 247-260 (2019).
- Bringhurst, B., Allert, M., Greenwold, M., Kellner, K., Seal, J. N. Environments and hosts structure the bacterial microbiomes of fungus-gardening ants and their symbiotic fungus gardens. Microb Ecol. 86 (2), 1374-1392 (2023).
- Bringhurst, B., Greenwold, M., Kellner, K., Seal, J. N. Symbiosis, dysbiosis and the impact of horizontal exchange on bacterial microbiomes in higher fungus-gardening ants. Sci Rep. 14 (1), 3231(2024).
- Vargas, S., et al. Body-plan reorganization in a sponge correlates with microbiome change. Mol Biol Evol. 40 (6), msad138(2023).
- Apprill, A., et al. Humpback whale populations share a core skin bacterial community: towards a health index for marine mammals. PLOS One. 9 (3), e90785(2014).
- Fraune, S., et al. Bacteria-bacteria interactions within the microbiota of the ancestral metazoan Hydra contribute to fungal resistance. ISME J. 9 (7), 1543-1556 (2015).
- Gowen, R., Gamal, A., Di Martino, L., McCormick, T. S., Ghannoum, M. A. Modulating the microbiome for Crohn's disease treatment. Gastroenterology. 164 (5), 828-840 (2023).
- Moya, P., et al. Molecular phylogeny and ultrastructure of the lichen microalga Asterochloris mediterranea sp. nov. from Mediterranean and Canary Islands ecosystems. Int J Syst Evol Microbiol. 65 (6), 1838-1854 (2015).
- Zettler, E. R., Mincer, T. J., Amaral-Zettler, L. A. Life in the "plastisphere": microbial communities on plastic marine debris. Environ Sci Technol. 47 (13), 7137-7146 (2013).
- Porter, K. R., Kallman, F. The properties and effects of osmium tetroxide as a tissue fixative with special reference to its use for electron microscopy. Exp Cell Res. 4 (1), 127-141 (1953).
- Ligon, J. J., Abraham, J. L., Boyd, A. S. Traumatic osmium tetroxide inoculation. J Am Acad Dermatol. 45 (6), 949-952 (2001).
- Friedova, N., et al. Osmium absorption after osmium tetroxide skin and eye exposure. Basic Clin Pharmacol Toxicol. 127 (5), 429-433 (2020).
- Fratesi, S. E., Lynch, F. L., Kirkland, B. L., Brown, L. R. Effects of SEM preparation techniques on the appearance of bacteria and biofilms in the Carter Sandstone. J Sediment. 74 (6), 858-867 (2004).
- Dassanayake, R. P., et al. Identification of a reliable fixative solution to preserve the complex architecture of bacterial biofilms for scanning electron microscopy evaluation. PLOS One. 15 (5), e0233973(2020).
- Desiro, A., et al. Detection of a novel intracellular microbiome hosted in arbuscular mycorrhizal fungi. ISME J. 8 (2), 257-270 (2014).
- Morales, D. P., et al. Advances and challenges in fluorescence in situ hybridization for visualizing fungal endobacteria. Front Microbiol. 13, 892227(2022).
- Shi, H., et al. Highly multiplexed spatial mapping of microbial communities. Nature. 588 (7839), 676-681 (2020).
- Valm, A. M., Welch, J. L. M., Borisy, G. G. CLASI-FISH: principles of combinatorial labeling and spectral imaging. Syst Appl Microbiol. 35 (8), 496-502 (2012).
- Fernandez-Brime, S., Muggia, L., Maier, S., Grube, M., Wedin, M. Bacterial communities in an optional lichen symbiosis are determined by substrate, not algal photobionts. FEMS Microbiol Ecol. 95 (3), fiz012(2019).
- Schluter, S., Eickhorst, T., Mueller, C. W. Correlative imaging reveals holistic view of soil microenvironments. Environ Sci Technol. 53 (2), 829-837 (2018).
- Barcoto, M. O., Rodrigues, A. Lessons from insect fungiculture: from microbial ecology to plastics degradation. Front Microbiol. 13, 812143(2022).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon