Method Article
Optimización de la reacción de transcripción in vitro para la producción de ARNm mediante la monitorización cromatográfica en línea
* Estos autores han contribuido por igual
En este artículo
Resumen
La optimización de la transcripción in vitro (IVT) es fundamental para una producción rentable de ARNm. Este protocolo detalla un método cromatográfico analítico para el análisis en línea de la reacción IVT, el monitoreo del agotamiento de NTP y la producción de ARNm en modos por lotes o lotes alimentados, aplicable a varias modalidades de ARN, mejorando la productividad y reduciendo los costos.
Resumen
La reacción de transcripción in vitro (IVT) es una síntesis enzimática compleja y multicomponente de ARNm a partir de una plantilla de ADN lineal, catalizada por una ARN polimerasa, por ejemplo, T7. Debido al alto costo de los reactivos de IVT, IVT es un paso crítico en el proceso de producción de sustancias farmacológicas de ARNm y ha sido objeto de una intensa optimización en el campo. Para disminuir el costo de producción de ARNm, los reactivos deben utilizarse de manera óptima. La optimización efectiva requiere una comprensión integral del impacto de los reactivos individuales en la cinética de la reacción, es decir, el consumo de nucleósidos trifosfatos (NTP) y la producción de ARNm. Tradicionalmente, se han utilizado técnicas analíticas de punto final de bajo rendimiento para el análisis del ARNm. Aunque estos métodos brindan información valiosa sobre el contenido de ARNm, se necesitan análisis en línea casi en tiempo real para comprender completamente la reacción de IVT. Demostramos cómo un método analítico de cromatografía líquida que separa los NTP, así como el ADNp y el ARNm, se puede utilizar casi en tiempo real para estudiar la cinética de la reacción de IVT. Con el conocimiento de la influencia de los diferentes componentes de IVT en la cinética y el rendimiento, el análisis cromatográfico rápido se puede utilizar para convertir la reacción de IVT por lotes en un modo de lote alimentado que aumenta aún más la productividad y reduce el costo total de la reacción.
Introducción
La pandemia de COVID-19 ha catalizado una revolución sin precedentes en la biomedicina, que ha llevado al rápido desarrollo y autorización de vacunas basadas en ARNm, como Comirnaty de BioNTech/Pfizer y Spikevax de Moderna, tanto en Europa como en Estados Unidos 1,2,3. La impresionante eficacia y el rápido desarrollo de estas vacunas han puesto de manifiesto el inmenso potencial terapéutico de la tecnología de ARNm, no solo para las enfermedades infecciosas, sino también para las inmunoterapias contra el cáncer, las terapias de sustitución de proteínas, la medicina regenerativa y la reprogramación celular 4,5. Esta era, a menudo denominada la revolución del ARNm, subraya la necesidad de procesos de producción de ARNm eficientes y rentables.
La producción de ARNm implica operaciones unitarias múltiples, siendo la reacción de transcripción in vitro (IVT) una operación unitaria crítica y más costosa 6,7,8. La reacción IVT sintetiza ARNm a partir de un molde de ADN utilizando ARN polimerasa, típicamente ARN polimerasa T7 y nucleósidos trifosfatos (NTP) en presencia de iones de magnesio. El proceso es relativamente sencillo y permite la producción de grandes cantidades de ARNm en un corto período de tiempo, logrando rendimientos de reacción de 2-5 g/L en pocas horas y hasta 14 g/L en algunos informes 9,10,11. Sin embargo, la optimización del rendimiento de la reacción IVT es crucial para reducir los costos de producción y garantizar la escalabilidad de las vacunas y terapias de ARNm12 y, debido a las diferencias de secuencia que influyen en el consumo de NTP, puede ser necesario para cada constructo o familia de constructos.
La reacción generalmente se realiza como un proceso por lotes, pero los avances recientes en el procesamiento de lotes alimentados y el análisis en línea han abierto nuevas vías para optimizar la producción de ARNm 7,13. Las reacciones alimentadas-batch, que implican un bolo o la adición continua de reactivos, pueden potencialmente extender los tiempos de reacción y aumentar los rendimientos al prevenir la inhibición del sustrato y la degradación del producto dependiente del cofactor14,15.
El desarrollo de la analítica rápida en línea ha sido un avance significativo en el campo de la IVT, permitiendo la monitorización de los componentes clave de la reacción, como los NTP y el ARNm, con un retraso analítico mínimo13,16, añadiendo una dimensión adicional a la monitorización de la IVT, que normalmente sólo se centra en la concentración de ARNm17. Tradicionalmente, los ARNm se han analizado mediante técnicas como la electroforesis en gel de poliacrilamida (PAGE), la electroforesis en gel de agarosa o la electroforesis capilar. Estos son métodos de punto final y, por lo tanto, no se pueden utilizar para el monitoreo de IVT en tiempo real. Una alternativa a la analítica descrita en este protocolo son los métodos que utilizan pares de ARN aptámero y colorante de fluorescencia iluminados. En este método, el aptámero de ARN se marca con ARN y se incuba con un tinte de fluorescencia iluminado. La actividad de transcripción se puede visualizar entonces por la intensidad de fluorescencia17. Esto permite el análisis en tiempo real de la cantidad y la calidad del ARNm transcrito durante el IVT 21, pero no permite el seguimiento simultáneo de otros componentes del IVT, por ejemplo, NTP. Otra alternativa que potencialmente permite la cuantificación en tiempo real de NTP y ARNm es la espectroscopia Raman18, pero hasta la fecha, ningún informe ha demostrado su utilidad para el monitoreo de IVT, lo que sugiere que aún se requiere una mayor optimización del método para derivar la selectividad y sensibilidad necesarias.
El desarrollo continuo de análisis más rápidos y selectivos respalda la optimización de IVT para obtener un mayor rendimiento en los modos de lote y lote alimentado. La reacción sigue siendo un proceso complejo y multiparamétrico con múltiples factores que interactúan, para los cuales los óptimos pueden diferir entre los tipos de constructos (por ejemplo, ARNm frente a ARNsa). La concentración y la proporción de iones Mg2+ y NTP son particularmente influyentes, y sus niveles óptimos deben equilibrarse cuidadosamente para maximizar el rendimiento del ARNm y minimizar la formación de ARN bicatenario (dsRNA), un potente estimulante del sistema inmune innato19,20. Recientemente, se informó de la alimentación con UTP a niveles de estado estacionario como un enfoque para reducir la formación de dsRNA21. El monitoreo de los niveles de UTP casi en tiempo real agregaría un nivel adicional de control de procesos.
En este protocolo, demostramos cómo el monitoreo en línea de la reacción IVT puede aumentar el rendimiento de la producción de ARNm en modo por lotes y lotes alimentados.
Protocolo
1. Preparación del tampón
NOTA: Todos los tampones deben estar preparados sin RNasa, lo que significa que todos los productos químicos y cristalería deben usarse solo para trabajos libres de RNasa y manipularse con precaución. El agua utilizada para la preparación del tampón y la limpieza de la cristalería debe estar certificada como libre de nucleasas. Antes de realizar un experimento, todas las superficies de trabajo y cristalería deben rociarse con un reactivo de descontaminación que elimine las RNasas. El reactivo de descontaminación debe lavarse a fondo con agua libre de RNasa antes de utilizar la cristalería/área de trabajo. Si es posible, utilice consumibles estériles y de un solo uso.
- Preparación de tampones IVT: prepare 10x tampón IVT añadiendo 400 mM de Tris, 10 mM de DTT, 20 mM de espermidina, pH 7,9. Prepare un tampón de enfriamiento que contenga 100 mM de EDTA, pH 8. Filtre cada tampón a través de un filtro PES de 0,22 μm.
- Preparación de fases móviles: preparar la fase móvil A (MPA) añadiendo 50 mM de HEPES, pH 7,0, fase móvil B (MPB) añadiendo 50 mM de HEPES, 100 mM de Na4P2O7, pH 8,3, fase móvil C (MPC) añadiendo 0,1 M de NaOH, 1 M de NaCl, fase móvil D (MPD) añadiendo 0,5 M de HEPES, pH 7.0. Filtre cada tampón a través de un filtro PES de 0,22 μm.
2. Preparación de la reacción de transcripción in vitro (IVT)
NOTA 1: La reacción IVT puede ser una reacción por lotes, en la que se añaden todos los reactivos al principio, y la reacción se detiene después de que se agotan los NTP o la producción de ARNm alcanza una meseta, o puede ser una reacción por lotes alimentados, en la que los NTP agotados se reponen con NTP adicionales y, con ello, la producción de ARNm aumenta adicionalmente. La plantilla lineal de ADNp utilizada en la reacción IVT se puede obtener mediante el uso de una enzima de restricción que se escinde justo después de la secuencia poli(A) en el ADN plasmídico o mediante una reacción de PCR. En ambos casos, la reacción debe ir seguida de una purificación, ya sea cromatográfica o mediante el uso de kits comerciales de purificación de ADN.
- Reacción IVT por lotes
- Ajuste el termobloque a 37 °C y 300 rpm. Descongele todos los reactivos enumerados en la tabla 1 , excepto las enzimas, y precaliéntelos a 37 °C en el termobloque. Prepare la mezcla NTP de 25 mM utilizando una solución madre de 100 mM de ATP, CTP, UTP y GTP.
- Mientras se descongelan los reactivos, prepare las alícuotas de muestra para el experimento de curso de tiempo. Tome tubos estériles de 0,5 mL y pipetee 2 μL de 100 mM de EDTA en cada tubo. Marque cada tubo con un número IVT designado (p. ej., IVT001, IVT002, etc.) y un punto de tiempo (p. ej., 0 min, 15 min, 30 min, 60 min, etc.).
- Cuando los reactivos se descongelen y se mantengan a 37 °C durante aproximadamente 15 minutos, retire las enzimas (ARN polimerasa T7, pirofosfatasa, inhibidor de la RNASA) del congelador a -20 °C y manténgalo en una nevera hasta que se almacene nuevamente en el congelador.
- Para mezclar reactivos IVT individuales (excepto enzimas), utilice un vórtice. Mezcle las enzimas pipeteando suavemente hacia arriba y hacia abajo.
- Mezcle los reactivos IVT en las proporciones establecidas en el diseño experimental (por ejemplo, el protocolo IVT genérico se muestra en la Tabla 1) en un tubo de 1,5 mL. Al realizar la optimización de IVT, el volumen final de IVT puede oscilar entre 50 y 100 μL. Al diseñar el volumen total de IVT, considere el número de muestras de 2 μL que se tomarán de la reacción durante el experimento de curso de tiempo. Asegúrese de que el volumen total de las muestras no supere el 50% del volumen total de IVT (por ejemplo, si se toman muestras de 2 μL cada 15 minutos durante 3 h, el volumen total de alícuotas tomadas es de 30 μL, lo que significa que el volumen total de IVT debe ser de 60 μL como mínimo).
- Mezcle los IVT con cuidado, agregue la ARN polimerasa T7 como último reactivo. Cuando se agreguen todos los reactivos, mezcle bien IVT pipeteando hacia arriba y hacia abajo, no use vórtice.
- Inmediatamente después de añadir y homogeneizar todos los reactivos de IVT, extraiga 2 μL de la mezcla de IVT del tubo y pipetee en tubos de 0,5 mL previamente preparados que contengan 2 μL de EDTA 100 mM descrito en el paso 2.1.2 (punto de tiempo 0 min).
- Coloque los tubos de reacción que contengan mezclas de reacción IVT en un termobloque e incube a 37 °C.
- Ajuste el temporizador a los intervalos de tiempo deseados (por ejemplo, 15 min/30 min). En cada punto de tiempo, tome 2 μL de una muestra de IVT de cada mezcla de reacción de IVT preparada y pipetee en un tubo previamente preparado (consulte el paso 2.1.2) que contenga 2 μL de 100 mM de EDTA. Estas alícuotas se utilizarán para el análisis cromatográfico (ver paso 3).
- Después de que el análisis cromatográfico confirme el agotamiento completo de los NTP, enfríe las reacciones de IVT a granel con EDTA hasta una concentración final de 50 mM (por ejemplo, 30 μL de IVT inactivado con 30 μL de EDTA de 100 mM).
- Reacción IVT por lotes alimentados
NOTA: la producción de ARNm se puede aumentar adicionalmente agregando NTP e iones Mg2+ después de que se agoten los NTP.- Mezcle la reacción IVT por lotes y muestree como se describe en el paso 2.1. Prepare el alimento NTP + MgCl2 para el lote alimentado. Para preparar, por ejemplo, 500 μL de mezcla NTP+MgCl2 , mezcle 106 μL de cada 200 mM de NTP, 76,25 μL de 1 M de MgCl2 y 5,5 μL de ddH2O. Las concentraciones finales en la mezcla son 42,4 mM cada NTP y 152,5 mM MgCl2.
- Supervise el consumo de NTP con análisis en línea descritos en el paso 3. Cuando los análisis en línea confirmen que la concentración de NTP cae por debajo del 10% de la concentración inicial, agregue el alimento que contiene NTP + Mg2+ al IVT a granel.
- Mida/calcule el volumen preciso de reacción IVT a granel que queda en un tubo de 1,5 ml después de tomar todas las alícuotas para su análisis. Agregue un volumen apropiado de mezcla de NTP + MgCl2 a un IVT de modo que la concentración final sea de 4 mM de cada NTP y 12 mM de MgCl2. Por ejemplo, para 80 μL de IVT a granel que quedan en un tubo de 1,5 mL, agregue 16,8 μL de mezcla NTP+MgCl2 .
- Continúe con el muestreo como se describe en el paso 2.1. Después de que los NTP se agoten nuevamente, repita la sección 2.2.2. Continúe con la alimentación y el muestreo hasta que se logre la producción deseada de ARNm.
- Una vez alcanzada la producción deseada de ARNm, inactive toda la reacción como en el paso 2.1.10.
3. Preparación del análisis cromatográfico
- Preparación de pruebas de idoneidad estándar y del sistema (SST)
- Prepare la SST combinando el reactivo de taponamiento, los NTP, la plantilla de ADNp y el ARNm. Asegúrese de que las concentraciones finales sean de aproximadamente 3 μM para el reactivo de taponamiento, 5 μM para cada NTP, 4 ng/μL para la plantilla de ADNp y 10 ng/μL para el ARNm. Asegúrese de que la TSM final contenga 0,1 M de NaCl.
- Prepare una nueva curva de calibración antes de cada análisis. Cree el estándar de calibración diluyendo una muestra de ARNm purificada de concentración conocida con MPA. Los puntos de calibración deben ser 0,5 ng/μL, 2 ng/μL, 5 ng/μL, 10 ng/μL, 15 ng/μL y 20 ng/μL, cada uno con una concentración final de 0,1 M de NaCl.
- Si es necesario, prepare una curva de calibración para todos los NTP. Asegúrese de que las concentraciones finales de cada NTP en los estándares de calibración sean de 0,5 μM, 2 μM, 5 μM, 10 μM, 15 μM y 20 μM.
- Acondicionamiento de la columna y preparación para el análisis
- Equilibrar la columna analítica de 0,1 mL a temperatura ambiente durante 12 h antes del análisis. Fije la columna al sistema cromatográfico en la dirección indicada en la carcasa de la columna.
- Enjuague la columna con un caudal de 1 mL/min: primero con 50 volúmenes de columna (CV) de ddH2O y luego con 50 CV de MPA.
- Ejecute al menos 3 muestras en blanco (inyectando solo MPA) antes del análisis para establecer la línea de base. Utilice el método cromatográfico descrito en Skok et al.15.
- Una vez que la línea de base sea estable y dos mediciones consecutivas en blanco sean comparables (línea de base reproducible), proceda a ejecutar la muestra de SST y las muestras de la curva de calibración.
- Si se cumplen los criterios de SST y la separación del reactivo de taponamiento y los NTP es satisfactoria, se proceda con el análisis de las muestras de IVT. Después de analizar todas las muestras de IVT, inyecte nuevamente un estándar SST para confirmar que las condiciones del sistema permanecieron estables durante todo el análisis.
- Preparación de la muestra
NOTA: Las muestras para el análisis cromatográfico se preparan inmediatamente antes del análisis. De lo contrario, las muestras se conservan en un congelador a -20 °C (máximo 24 h) o <-65 °C (más de 24 h).- Antes de la dilución para el análisis cromatográfico, vórtice y centrifuga la muestra de IVT templada. Analice todas las muestras de IVT inactivadas con EDTA (ver paso 2.1.9) con análisis cromatográfico utilizando el método descrito en Skok et al.15.
- Antes del análisis, diluya las muestras de IVT para lograr una concentración dentro de la curva de calibración. Utilice el mismo factor de dilución durante todo el experimento de curso de tiempo para asegurarse de que las áreas de NTP se puedan comparar para determinar cuándo se agota un NTP.
- Para determinar la dilución mínima necesaria para las muestras de IVT, utilice la siguiente fórmula: Dilución necesaria = (concentración final esperada de ARNm) / (dilución durante la inactivación) / (estándar de calibración más alto)
Por ejemplo, si se espera una producción de ARNm de 8 mg/mL en IVT, la concentración en la muestra templada será de 4 mg/mL. Para lograr la concentración dentro de la curva de calibración, diluya aún más la muestra 200 veces.
Dilución necesaria = (8000 ng/μL]) / 2 / (20 ng/μL) = 200 - Pipetear el MPA y 4 M de NaCl en un vial de vidrio cónico. Asegúrese de que la concentración final de NaCl en la muestra sea de 100 mM. Por último, agregue la muestra de IVT templada.
- Vortex preparó la muestra y la insertó en el muestreador automático para su análisis. Ajuste la temperatura del muestreador automático a 4 °C.
- Inyecte 100 μL de muestras diluidas en la columna analítica. Mida la absorbancia A260 y A280 para cada muestra de IVT.
- Para analizar muestras con concentraciones conocidas, o si no es necesario realizar un seguimiento del consumo de NTP en función de las diferencias de área, diluya las muestras para alcanzar una concentración objetivo de 10 ng/μL.
4. Cuantificación de muestras y análisis de datos
- Antes de la integración de cada muestra de IVT, reste la muestra en blanco del cromatograma de muestra.
- Integre todos los picos de NTP, ADNp y ARNm en A260 y utilice las áreas de picos para calcular las concentraciones de NTP y ARNm en el IVT.
NOTA: Alternativamente, las áreas de pico se pueden usar para comparar el porcentaje de cada NTP restante en la reacción IVT, siempre que el factor de dilución sea consistente en todos los puntos de tiempo. - Sobre la base de las concentraciones de ARNm/NTP, determinadas por análisis cromatográfico en línea, decida si se puede inactivar IVT o si se deben introducir NTP adicionales en la reacción de IVT (consulte el paso 2).
Resultados
El análisis cromatográfico descrito en este protocolo se puede utilizar para la optimización de IVT o para convertir una reacción de IVT por lotes en una reacción de lotes alimentados (Figura 1).
Para probar el efecto de diferentes composiciones de tampones en la cinética de la reacción de IVT, se mezclaron tres reacciones diferentes de IVT de acuerdo con el protocolo escrito en la Tabla 2. El tampón que contenía Tris, donde el pH se ajustó con HCl, se comparó con el tampón Tris, donde el pH se ajustó con ácido acético. Además, ambos tampones Tris se compararon con el tampón HEPES, donde el pH se ajustó con NaOH. La composición de todos los tampones 1x IVT fue de 40 mM de Tris/HEPES, 10 mM de DTT, 2 mM de espermidina y pH de 7,9.
De 100 μL de mezcla de reacciones IVT, se extrajeron 2 μL de muestra IVT de la reacción IVT y se enfriaron con 2 μL de EDTA 100 mM cada 15 min en la primera hora de incubación y luego cada 30 min hasta 180 min de incubación. El rendimiento final esperado de ARNm para esta condición de reacción fue de 15 mg/mL, lo que significa que la dilución de 2 veces debido al enfriamiento fue seguida por una dilución de 400 veces en MPA + NaCl para el análisis. La muestra se diluyó 800 veces en total, lo que significa que incluso con una producción de 15 mg/mL, la concentración de ARNm cargada en la columna cromatográfica analítica todavía estaba dentro de las concentraciones de la curva de calibración.
Las áreas de NTP y ARNm en A260 se integraron para cada muestra individual en cada punto de tiempo individual (tx). A continuación, las áreas se convirtieron en concentraciones de ARNm, utilizando la curva de calibración de ARNm y el porcentaje de consumo para los NTP, utilizando el área A260 de NTP a 0 min (t0) como un 100% (véase la ecuación a continuación).
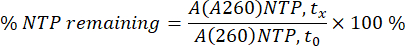
Los resultados se pueden mostrar como gráficos para cada reacción individual de IVT, donde el tiempo se muestra en el eje x y la concentración de ARNm y los NTP restantes se muestran en el eje y (Figura 2A). Todas las IVT, es decir, las concentraciones de ARNm en función del tiempo, también se pueden trazar juntas en un gráfico, y se puede estudiar la cinética de producción de ARNm y seleccionar las condiciones óptimas de IVT (Figura 2B).
Para el experimento de lotes alimentados, se mezcló IVT como se describe en la Tabla 3. De 300 μL de mezcla de reacción IVT, se extrajeron 2 μL de muestra IVT de la reacción IVT y se enfriaron con 2 μL de EDTA 100 mM cada 30 min. Además, la muestra se tomó inmediatamente después de cada adición masiva de NTP+MgCl2 . Se preparó previamente una solución de alimentación que contenía 42,4 mM de cada NTP y 152,5 mM de MgCl2 , y se mezclaron 106 μL de cada NTP individual de 200 mM con 76,2 μL de 1 M MgCl2. Se estableció un régimen de alimentación, donde se añadió bolo cada hora (a los 60 min, 120 min, 180 min y 240 min de incubación). El régimen de alimentación se describe en la Tabla 3 : Las muestras se analizaron casi en tiempo real con análisis como se describe para la reacción masiva de IVT. Después de 300 min de monitoreo, se extinguió la reacción de IVT.
Los resultados se pueden mostrar como la producción restante de NTPs/ARNm durante el tiempo de incubación. Dado que las adiciones de alimento en bolo también diluyen la reacción de IVT, la concentración de ARNm en IVT disminuye en cada adición de alimento (Figura 3A). El aumento de la masa de ARNm también puede medirse y presentarse mediante factores de transcripción (definidos comoARNm m/ADNp m), que muestran un aumento lineal de la producción de ARNm a lo largo del tiempo (Figura 3B).
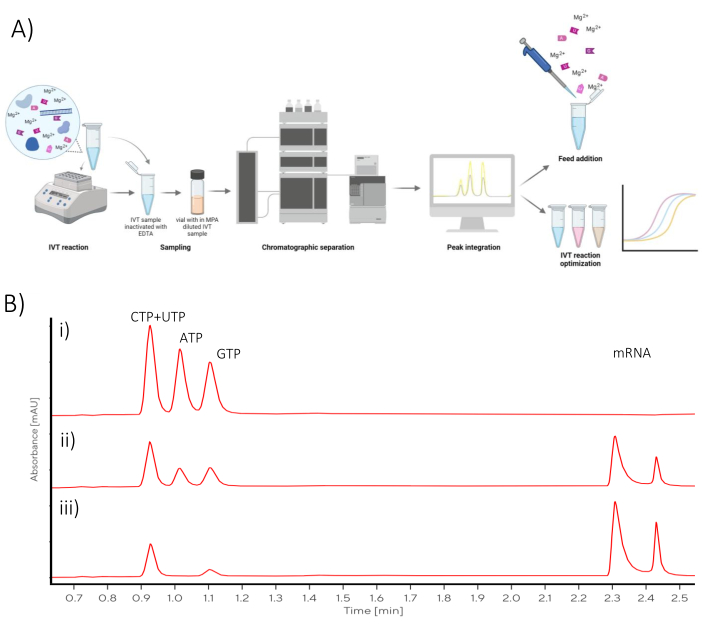
Figura 1: Resumen esquemático (A) Representación del flujo de trabajo de optimización de IVT con cuantificación de análisis cromatográfico en línea de NTP y ARNm. (B) Cromatogramas representativos de una reacción IVT por lotes muestreada en (i) t0, (ii) punto medio, (iii) puntos de tiempo de reacción final. Haga clic aquí para ver una versión más grande de esta figura.
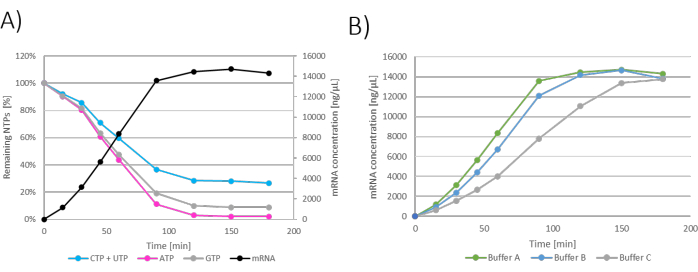
Figura 2: Gráficos de IVT por lotes. (A) Gráfico representativo de la reacción de IVT con la producción de ARNm y el consumo de NTP en el eje y. La meseta de concentración de ARNm visible a los 120 - 180 min de incubación que se correlaciona con el consumo de NTP como NTP limitante (ATP) se consume a los 120 min. (B) Gráfico de IVT que muestra la influencia del tampón IVT en la cinética de IVT. El tampón A (Tris + ácido acético) mostró la producción de ARNm más rápida, seguido del tampón B (Tris + HCl) y el tampón C (HEPES+NaOH) como los más lentos. Haga clic aquí para ver una versión más grande de esta figura.
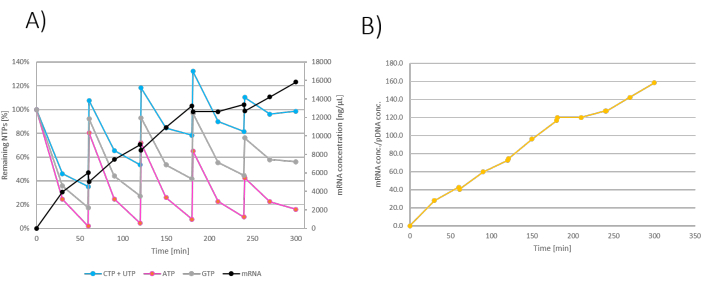
Figura 3: Gráfico IVT de lotes federales. (A) Gráfico de IVT en el que se añadieron alimentaciones NTP+MgCl2 a 60, 120, 180 y 240 min. de concentración de ARNm en la reacción IVT disminuye en cada adición de alimento, debido a la dilución con alimentación NTP+MgCl2 . (B) El aumento de la masa de ARNm mostrado por el factor de transcripción es lineal a lo largo de la incubación de la reacción. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Protocolo genérico IVT. Haga clic aquí para descargar esta tabla.
Tabla 2: Protocolo IVT por lotes. Haga clic aquí para descargar esta tabla.
Tabla 3: Protocolo IVT de lotes federales. Haga clic aquí para descargar esta tabla.
Discusión
En este método, las muestras de IVT se analizan en diferentes puntos temporales durante la incubación utilizando una columna cromatográfica analítica multimodal que separa los NTP, el ADNp y el ARNm, lo que permite un seguimiento estrecho del consumo de NTP y la producción de ARNm. Este método mide los NTP y la concentración de ARNm de manera cuantitativa en función de los cambios en las áreas A260 en puntos de tiempo designados. Dado que el método proporciona información sobre la concentración de NTP y ARNm, es muy adecuado para la optimización de IVT, donde el objetivo principal suele ser maximizar el rendimiento del ARNm y minimizar el tiempo de reacción; por lo tanto, es fundamental comprender el efecto de los diferentes reactivos de IVT en la cinética de la producción de ARNm22. Mostramos cómo se puede aplicar el monitoreo en línea para optimizar la reacción IVT en los modos de lote y lote alimentado.
La principal ventaja de este método es un análisis en línea que permite el monitoreo casi en tiempo real de IVT, ya que cada muestra se analiza en menos de 8 minutos. La preparación de las muestras para el análisis es sencilla, ya que solo se requiere la dilución en MPA sin ningún tratamiento previo de la muestra. El volumen de muestra requerido para el análisis es muy bajo, por ejemplo, 1 μL de IVT. Este pequeño volumen de pipeteo podría dar lugar a desviaciones analíticas debidas a errores de pipeteo. Sin embargo, dado que el alto rendimiento del enfoque analítico permite mediciones por triplicado, los valores atípicos son fácilmente identificables y las desviaciones de la curva cinética esperada no son difíciles de detectar.
Una de las limitaciones del método es la coelución cromatográfica de UTP y CTP. Por lo general, no es necesaria la cuantificación precisa de UTP y CTP, ya que el NTP limitante en una secuencia de ARNm suele ser ATP o GTP. Si se requiere una cuantificación separada de UTP y CTP, la diferencia en la absorbancia UV a 260 nm y 280 nm para UTP y CTP se puede aprovechar para derivar la abundancia relativa de cada NTP en el pico cromatográfico.
Este método analítico no separa las especies de ARN de más de 100 nucleótidos de longitud; por lo tanto, no se puede utilizar para detectar diferencias en la calidad del ARNm, por ejemplo, no diferencia entre ARNm no poliadenilado y poliadenilado, entre dsRNA o transcripciones abortivas y ssRNA y no es adecuado para estudios de estabilidad ya que no puede diferenciar entre ARNm degradado y no degradado. Sin embargo, el método se puede utilizar para la cuantificación de otras modalidades de ARN, como el circRNA, el ARNt y el ARNsa. Aunque estas moléculas varían en tamaño y estructura, se puede emplear el mismo método analítico para estudiar los rendimientos de producción de cada una en una reacción IVT.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer a Tomas Kostelec, Blaž Bakalar, Nejc Pavlin, Andreja Gramc Livk y Anže Martinčič Celjar por sus útiles discusiones.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
Referencias
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403(2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, Ö mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090(2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190(2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3(2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745(2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655(2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833(2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246(2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937(2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408(2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045(2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados