Method Article
אופטימיזציה של תגובת שעתוק במבחנה לייצור mRNA באמצעות ניטור כרומטוגרפי בקו
In This Article
Summary
אופטימיזציה של שעתוק במבחנה (IVT) היא קריטית לייצור mRNA חסכוני. פרוטוקול זה מפרט שיטה כרומטוגרפית אנליטית לניתוח מקוון של תגובת IVT, ניטור דלדול NTP וייצור mRNA במצבי אצווה או אצווה מוזנת, החלים על שיטות RNA שונות, שיפור הפרודוקטיביות והפחתת עלויות.
Abstract
תגובת שעתוק חוץ גופית (IVT) היא סינתזה אנזימטית מורכבת ורב-רכיבית של mRNA מתבנית DNA ליניארית, מזורזת על ידי RNA פולימראז, למשל T7. בשל העלות הגבוהה של ריאגנטים IVT, IVT הוא שלב קריטי בתהליך ייצור חומר תרופת mRNA והיה המוקד של אופטימיזציה אינטנסיבית בתחום. כדי להפחית את עלות ייצור ה-mRNA, יש לנצל ריאגנטים בצורה אופטימלית. אופטימיזציה יעילה מחייבת הבנה מקיפה של ההשפעה של ריאגנטים בודדים על קינטיקה של תגובה, כלומר צריכת נוקלאוזידים טריפוספטים (NTPs) וייצור mRNA. באופן מסורתי, נעשה שימוש בטכניקות אנליטיות בעלות תפוקה נמוכה ונקודת קצה לניתוח mRNA. למרות ששיטות כאלה נותנות מידע רב ערך על תכולת ה-mRNA, יש צורך בניתוח מקוון כמעט בזמן אמת כדי להבין את תגובת ה-IVT במלואה. אנו מדגימים כיצד ניתן להשתמש בשיטה אנליטית של כרומטוגרפיה נוזלית המפרידה בין NTPs, כמו גם pDNA ו-mRNA, כמעט בזמן אמת כדי לחקור קינטיקה של תגובת IVT. עם הידע על ההשפעה של רכיבי IVT שונים על קינטיקה ותפוקה, ניתן להשתמש בניתוח כרומטוגרפי מהיר כדי להמיר תגובת IVT אצווה למצב אצווה מוזנת המגדיל עוד יותר את הפרודוקטיביות ומפחית את העלות הכוללת של התגובה.
Introduction
מגיפת COVID-19 זירזה מהפכה חסרת תקדים בביו-רפואה, והובילה לפיתוח ואישור מהיר של חיסונים מבוססי mRNA, כגון Comirnaty של BioNTech/Pfizer ו-Spikevax של מודרנה, הן באירופה והן בארה"ב 1,2,3. היעילות המרשימה והפיתוח המהיר של חיסונים אלה הדגישו את הפוטנציאל הטיפולי העצום של טכנולוגיית mRNA, לא רק למחלות זיהומיות אלא גם לאימונותרפיה לסרטן, טיפולים בתחליפי חלבון, רפואה רגנרטיבית ותכנות מחדש של תאים 4,5. עידן זה, המכונה לעתים קרובות מהפכת ה-mRNA, מדגיש את הצורך בתהליכי ייצור mRNA יעילים וחסכוניים.
ייצור ה-mRNA כרוך בפעולות יחידות מרובות, כאשר תגובת השעתוק במבחנה (IVT) היא פעולת יחידה קריטית ועתירת עלות 6,7,8. תגובת IVT מסנתזת mRNA מתבנית DNA באמצעות RNA פולימראז, בדרך כלל T7 RNA פולימראז, ונוקלאוזידים טריפוספטים (NTPs) בנוכחות יוני מגנזיום. התהליך פשוט יחסית ומאפשר ייצור כמויות גדולות של mRNA בפרק זמן קצר, תוך השגת תפוקות תגובה של 2-5 גרם/ליטר תוך מספר שעות ועד 14 גרם/ליטר בחלק מהדיווחים 9,10,11. עם זאת, אופטימיזציה של תפוקת תגובת IVT היא חיונית להפחתת עלויות הייצור ולהבטחת יכולת ההרחבה של חיסוני mRNA וטיפולים12, ובשל הבדלי רצף המשפיעים על צריכת NTP, עשויה להידרש עבור כל מבנה או משפחת מבנים.
התגובה מבוצעת בדרך כלל כתהליך אצווה, אך ההתקדמות האחרונה בעיבוד אצווה מוזנת וניתוח מקוון פתחו אפיקים חדשים לאופטימיזציה של ייצור mRNA 7,13. תגובות אצווה של פד, הכוללות בולוס או תוספת מתמשכת של ריאגנטים, יכולות להאריך את זמני התגובה ולהגדיל את התשואות על ידי מניעת עיכוב סובסטרט ופירוק מוצר תלוי קו-פקטור14,15.
הפיתוח של אנליטיקה מהירה בקו היווה התקדמות משמעותית בתחום ה-IVT, המאפשר ניטור של רכיבי תגובה מרכזיים כגון NTPs ו-mRNA עם פיגור אנליטי מינימלי13,16, מה שמוסיף ממד נוסף לניטור IVT, שבדרך כלל מתמקד רק בריכוז mRNA17. באופן מסורתי, mRNAs נותחו באמצעות טכניקות כגון אלקטרופורזה של ג'ל פוליאקרילאמיד (PAGE), אלקטרופורזה של ג'ל אגרוז או אלקטרופורזה נימית. אלו הן שיטות נקודת קצה ולכן לא ניתן להשתמש בהן לניטור IVT בזמן אמת. אלטרנטיבה לניתוח המתוארת בפרוטוקול זה הן שיטות המשתמשות בזוגות צבע RNA מוארים וצבעים פלואורסצנטיים. בשיטה זו, אפטמר RNA מתויג ל-RNA ומודגר בצבע פלואורסצנטי מואר. לאחר מכן ניתן לדמיין את פעילות השעתוק על ידי עוצמת הקרינה17. זה מאפשר ניתוח בזמן אמת של כמות ואיכות ה-mRNA המתומלל במהלך IVT 21 אך אינו מאפשר ניטור סימולטני של רכיבי IVT אחרים, למשל, NTPs. חלופה נוספת שעשויה לאפשר כימות בזמן אמת של NTP ו-mRNA היא ספקטרוסקופיה Raman18, אך עד כה, אף דוח לא הראה את התועלת שלה לניטור IVT, מה שמצביע על כך שעדיין נדרשת אופטימיזציה נוספת של השיטה כדי להפיק את הסלקטיביות והרגישות הדרושות.
פיתוח מתמשך של ניתוח מהיר וסלקטיבי יותר תומך באופטימיזציה של IVT לתפוקה גבוהה יותר במצבי אצווה ואצווה מוזנת. התגובה נותרה תהליך מורכב ורב-פרמטרי עם גורמים אינטראקטיביים מרובים, שעבורם האופטימה עשויה להיות שונה בין סוגי המבנים (למשל, mRNA לעומת saRNA). הריכוז והיחס של יוני Mg2+ ו-NTPs משפיעים במיוחד, ויש לאזן בקפידה את הרמות האופטימליות שלהם כדי למקסם את תפוקת ה-mRNA תוך מזעור היווצרות RNA דו-גדילי (dsRNA), ממריץ חזק של מערכת החיסון המולדת19,20. לאחרונה, דווח על הזנת UTP ברמות מצב יציב כגישה להפחתת היווצרות dsRNA21. ניטור רמות UTP כמעט בזמן אמת יוסיף רמה נוספת של בקרת תהליכים.
בפרוטוקול זה, אנו מדגימים כיצד ניטור מקוון של תגובת IVT יכול להגדיל את התפוקה של ייצור mRNA במצב אצווה ואצווה מוזנת.
Protocol
1. הכנת חיץ
הערה: יש להכין את כל המאגרים ללא RNase, כלומר יש להשתמש בכל הכימיקלים וכלי הזכוכית רק לעבודה ללא RNase ולטפל בהם בזהירות. מים המשמשים להכנת חיץ וניקוי כלי זכוכית חייבים להיות מאושרים כנטולי נוקלאז. לפני ביצוע ניסוי, יש לרסס את כל משטחי העבודה וכלי הזכוכית במגיב טיהור המבטל RNases. יש לשטוף היטב את מגיב הטיהור במים נטולי RNase לפני השימוש בכלי זכוכית/אזור עבודה. במידת האפשר, השתמש בחומרים מתכלים סטריליים וחד פעמיים בלבד.
- הכנת מאגרי IVT: הכינו מאגר IVT 10x על ידי הוספת 400 מ"מ טריס, 10 מ"מ DTT, 20 מ"מ ספרמידין, pH 7.9. הכן מאגר מרווה המכיל 100 מ"מ EDTA, pH 8. מסננים כל מאגר דרך מסנן PES של 0.22 מיקרומטר.
- הכנת פאזות ניידות: הכנת שלב נייד A (MPA) על ידי הוספת 50 מ"מ HEPES, pH 7.0, שלב נייד B (MPB) על ידי הוספת 50 מ"מ HEPES, 100 מ"מ Na4P2O7, pH 8.3, שלב C נייד (MPC) על ידי הוספת 0.1 M NaOH, 1 M NaCl, שלב D נייד (MPD) על ידי הוספת 0.5 M HEPES, pH 7.0. מסננים כל מאגר דרך מסנן PES של 0.22 מיקרומטר.
2. הכנת תגובת שעתוק חוץ גופית (IVT)
הערה 1: תגובת IVT יכולה להיות תגובת אצווה, שבה כל הריאגנטים מתווספים בהתחלה, והתגובה נעצרת לאחר ש-NTPs מתרוקנים/ייצור ה-mRNA מגיע למישור, או יכולה להיות תגובת אצווה מוזנת, שבה NTPs מדוללים מתחדשים ב-NTPs נוספים ועם זה ייצור ה-mRNA גדל בנוסף. ניתן להשיג תבנית pDNA ליניארית המשמשת בתגובת IVT על ידי שימוש באנזים הגבלה שנבקע מיד לאחר רצף פולי (A) ב-DNA פלסמיד או על ידי תגובת PCR. בשני המקרים יש ללוות את התגובה בטיהור, כרומטוגרפי או באמצעות ערכות טיהור DNA מסחריות.
- תגובת IVT אצווה
- הגדר את התרמובלוק ל-37 מעלות צלזיוס ו-300 סל"ד. הפשירו את כל הריאגנטים המפורטים בטבלה 1 למעט אנזימים וחממו אותם מראש ב-37 מעלות צלזיוס בתרמו-בלוק. הכן תערובת NTP של 25 מ"מ באמצעות תמיסת מלאי של 100 מ"מ של ATP, CTP, UTP ו-GTP.
- בזמן שהריאגנטים מפשירים, הכינו דוגמאות לניסוי מהלך הזמן. קח צינורות סטריליים של 0.5 מ"ל ופיפטה 2 מיקרוליטר של 100 מ"מ EDTA לכל צינור. סמן כל צינור עם מספר IVT ייעודי (למשל, IVT001, IVT002 וכו') ונקודת זמן (למשל, 0 דקות, 15 דקות, 30 דקות, 60 דקות וכו').
- כאשר ריאגנטים מופשרים ונשמרים בטמפרטורה של 37 מעלות צלזיוס למשך כ-15 דקות, הוציאו אנזימים (T7 RNA פולימראז, פירופוספטאז, מעכב רנאז) מהמקפיא של -20 מעלות צלזיוס ושמרו אותו בצידנית עד לאחסון בחזרה במקפיא.
- לערבוב ריאגנטים IVT בודדים (למעט אנזימים), השתמש במערבולת. ערבבו אנזימים על ידי פיפטינג עדין למעלה ולמטה.
- ערבב ריאגנטים IVT ביחסים שנקבעו בתכנון הניסוי (למשל, פרוטוקול IVT גנרי מוצג בטבלה 1) בשפופרת של 1.5 מ"ל. בעת ביצוע אופטימיזציה של IVT, נפח ה-IVT הסופי יכול לנוע בין 50-100 מיקרוליטר. בעת תכנון נפח ה-IVT הכולל, קחו בחשבון את מספר דגימות 2 המיקרוליטר שיש להוציא מהתגובה במהלך ניסוי מהלך הזמן. ודא שהנפח הכולל של הדגימות אינו עולה על 50% מנפח ה-IVT הכולל (למשל, אם דגימה של 2 מיקרוליטר כל 15 דקות למשך 3 שעות, הנפח הכולל של הכמות שנלקחה הוא 30 מיקרוליטר, כלומר הנפח הכולל של IVT צריך להיות מינימום 60 מיקרוליטר).
- מערבבים IVT בזהירות, מוסיפים T7 RNA פולימראז כמגיב האחרון. כאשר מוסיפים את כל הריאגנטים, מערבבים IVT היטב על ידי פיפטינג למעלה ולמטה, אין להשתמש במערבולת.
- מיד לאחר הוספה והומוגניות של כל ריאגנטים IVT, הסר 2 מיקרוליטר של תערובת IVT מהצינור והכניס אותה לצינורות שהוכנו בעבר של 0.5 מ"ל המכילים 2 מיקרוליטר של 100 מ"מ EDTA המתואר בשלב 2.1.2 (נקודת זמן 0 דקות).
- שים צינורות תגובה המכילים תערובות תגובת IVT בתרמובלוק ודגר אותם בטמפרטורה של 37 מעלות צלזיוס.
- הגדר טיימר למרווחי הזמן הרצויים (למשל, 15 דקות/30 דקות). בכל נקודת זמן, קח 2 מיקרוליטר של דגימת IVT מכל תערובות תגובת IVT שהוכנו והכניס אותה לתוך שפופרת שהוכנה קודם לכן (ראה שלב 2.1.2) המכילה 2 מיקרוליטר של 100 מ"מ EDTA. ציטוטים אלה ישמשו לניתוח כרומטוגרפי (ראה שלב 3).
- לאחר שניתוח כרומטוגרפי מאשר דלדול מוחלט של NTPs, יש להרוות תגובות IVT בתפזורת עם EDTA לריכוז סופי של 50 מ"מ (למשל 30 מיקרוליטר של IVT מושבת עם 30 מיקרוליטר של 100 מ"מ EDTA).
- תגובת IVT באצווה הפדרלית
הערה: ניתן להגדיל את ייצור ה-mRNA בנוסף על ידי הוספת יוני NTPs ו-Mg2+ לאחר התרוקנות NTPs.- מערבבים תגובת IVT אצווה ודוגמים אותה כמתואר בשלב 2.1. הכן הזנת NTP+MgCl2 לאצווה מוזנת. להכנה, למשל 500 מיקרוליטר של תערובת NTP+MgCl2 , מערבבים 106 מיקרוליטר מכל 200 מ"מ NTP, 76.25 מיקרוליטר של 1 M MgCl2 ו-5.5 מיקרוליטר של ddH2O. הריכוזים הסופיים בתערובת הם 42.4 מ"מ כל NTP ו-152.5 מ"מ MgCl2.
- נטר את צריכת NTP באמצעות ניתוח מקוון המתואר בשלב 3. כאשר ניתוח מקוון מאשר שריכוז ה-NTP יורד מתחת ל-10% מהריכוז ההתחלתי, הוסף את ההזנה המכילה NTP+Mg2+ ל-IVT בתפזורת.
- מדוד/חשב את הנפח המדויק של תגובת IVT בתפזורת שנותרה בשפופרת של 1.5 מ"ל לאחר שכל המינונים נלקחים לניתוח. הוסף נפח מתאים של תערובת NTP+MgCl2 ל-IVT כך שהריכוז הסופי יהיה 4 מ"מ מכל NTP ו-12 מ"מ של MgCl2. לדוגמה, עבור 80 מיקרוליטר של IVT בתפזורת שנותרה בשפופרת של 1.5 מ"ל, הוסף 16.8 מיקרוליטר של תערובת NTP+MgCl2 .
- המשך עם הדגימה כפי שנכתב בשלב 2.1. לאחר ש-NTPs מתרוקנים שוב, חזור על סעיף 2.2.2. המשיכו בהאכלה ודגימה עד להשגת ייצור ה-mRNA הרצוי.
- לאחר השגת ייצור ה-mRNA הרצוי, השבת את כל התגובה כמו בשלב 2.1.10.
3. הכנת אנליזה כרומטוגרפית
- הכנת מבחן התאמה סטנדרטי ומערכת (SST)
- הכן את ה-SST על ידי שילוב של מגיב מכסה, NTPs, תבנית pDNA ו-mRNA. ודא שהריכוזים הסופיים הם כ-3 מיקרומטר עבור מגיב המכסה, 5 מיקרומטר עבור כל NTP, 4 ננוגרם/מיקרוליטר עבור תבנית ה-pDNA ו-10 ננוגרם/מיקרוליטר עבור ה-mRNA. ודא שה-SST הסופי מכיל 0.1 M NaCl.
- הכן עקומת כיול חדשה לפני כל ניתוח. צור את תקן הכיול על ידי דילול דגימת mRNA מטוהרת בריכוז ידוע עם MPA. נקודות הכיול צריכות להיות 0.5 ננוגרם/מיקרוליטר, 2 ננוגרם/מיקרוליטר, 5 ננוגרם/מיקרוליטר, 10 ננוגרם/מיקרוליטר, 15 ננוגרם/מיקרוליטר ו-20 ננוגרם/מיקרוליטר, כל אחת עם ריכוז סופי של 0.1 מ' NaCl.
- במידת הצורך, הכן עקומת כיול עבור כל NTPs. ודא שהריכוזים הסופיים של כל NTP בתקני הכיול הם 0.5 מיקרומטר, 2 מיקרומטר, 5 מיקרומטר, 10 מיקרומטר, 15 מיקרומטר ו-20 מיקרומטר.
- התניה של עמודים והכנה לניתוח
- שיווי משקל של עמודה אנליטית של 0.1 מ"ל בטמפרטורת החדר למשך 12 שעות לפני הניתוח. חבר את העמודה למערכת הכרומטוגרפית בכיוון המצוין על בית העמוד.
- שטוף את העמודה בקצב זרימה של 1 מ"ל/דקה: תחילה עם 50 נפחי עמודות (CV) של ddH2O ולאחר מכן עם 50 CV של MPA.
- הפעל לפחות 3 דגימות ריקות (הזרקת MPA בלבד) לפני הניתוח כדי להגדיר את קו הבסיס. השתמש בשיטה הכרומטוגרפית המתוארת ב-Skok et al.15.
- לאחר שקו הבסיס יציב ושתי מדידות ריקות רצופות ניתנות להשוואה (קו בסיס הניתן לשחזור), המשך להריץ את דגימת ה-SST ואת דגימות עקומת הכיול.
- אם הקריטריונים של SST מתקיימים וההפרדה בין מגיב המכסה ל-NTPs משביעת רצון, המשך בניתוח דגימות ה-IVT. לאחר ניתוח כל דגימות ה-IVT, יש להזריק שוב תקן SST כדי לאשר שתנאי המערכת נותרו יציבים לאורך כל הניתוח.
- הכנת מדגם
הערה: דגימות לניתוח כרומטוגרפי מוכנות מיד לפני הניתוח. אחרת, הדגימות נשמרות במקפיא בטמפרטורה של -20 מעלות צלזיוס (מקסימום 24 שעות) או <-65 מעלות צלזיוס (יותר מ-24 שעות).- לפני דילול לניתוח כרומטוגרפי, מערבולת וסובב את דגימת ה-IVT המרווה. נתח את כל דגימות ה-IVT המושבתות על ידי EDTA (ראה שלב 2.1.9) עם ניתוח כרומטוגרפי בשיטה המתוארת ב-Skok et al.15.
- לפני הניתוח, יש לדלל את דגימות ה-IVT כדי להשיג ריכוז בתוך עקומת הכיול. השתמש באותו גורם דילול לאורך כל ניסוי מהלך הזמן כדי להבטיח שניתן להשוות אזורי NTP כדי לקבוע מתי NTP מתרוקן.
- כדי לקבוע את הדילול המינימלי הדרוש לדגימות IVT, השתמש בנוסחה הבאה: דרוש דילול = (ריכוז mRNA סופי צפוי) / (דילול במהלך השבתה) / (תקן הכיול הגבוה ביותר)
לדוגמה, אם צפוי ייצור mRNA של 8 מ"ג/מ"ל ב-IVT, הריכוז בדגימה מרווה יהיה 4 מ"ג/מ"ל. כדי להשיג ריכוז בתוך עקומת הכיול, יש לדלל עוד יותר את הדגימה פי 200.
דרוש דילול = (8000 ננוגרם/מיקרוליטר]) / 2 / (20 ננוגרם/מיקרוליטר) = 200 - הכניסו את ה-MPA ו-4 M NaCl לבקבוקון זכוכית חרוטי. ודא שהריכוז הסופי של NaCl בדגימה הוא 100 מ"מ. לבסוף, הוסף את דגימת ה-IVT המרווה.
- וורטקס הכינה את הדגימה והכניסה אותה לדגימה האוטומטית לניתוח. הגדר את טמפרטורת הדגימה האוטומטית ל-4 מעלות צלזיוס.
- הזרקת 100 מיקרוליטר של דגימות מדוללות על העמודה האנליטית. מדוד את הספיגה A260 ו-A280 עבור כל דגימת IVT.
- לניתוח דגימות עם ריכוזים ידועים, או אם אין צורך במעקב אחר צריכת NTP על סמך הבדלי שטח, יש לדלל את הדגימות כדי להשיג ריכוז יעד של 10 ננוגרם/מיקרוליטר.
4. כימות מדגם וניתוח נתונים
- לפני השילוב של כל דגימת IVT, הפחיתו את הדגימה הריקה מהכרומטוגרמה של הדגימה.
- שלב את כל פסגות ה-NTP, pDNA וה-mRNA ב-A260 והשתמש באזורי השיא כדי לחשב את הריכוזים של NTPs ו-mRNA ב-IVT.
הערה: לחלופין, ניתן להשתמש באזורי השיא כדי להשוות את האחוז של כל NTP שנותר בתגובת IVT, בתנאי שמקדם הדילול היה עקבי בכל נקודות הזמן. - בהתבסס על ריכוזי mRNA/NTP, שנקבעו על ידי ניתוח כרומטוגרפי מקוון, החליטו אם ניתן להשבית את ה-IVT או אם יש להזין NTPs נוספים לתגובת ה-IVT (ראה שלב 2).
תוצאות
הניתוח הכרומטוגרפי המתואר בפרוטוקול זה יכול לשמש לאופטימיזציה של IVT או להמרת תגובת IVT אצווה לתגובת אצווה מוזנת (איור 1).
כדי לבחון את ההשפעה של הרכבי חיץ שונים על הקינטיקה של תגובת IVT, עורבבו שלוש תגובות IVT שונות על פי הפרוטוקול הכתוב בטבלה 2. בופר המכיל Tris, שבו ה-pH הותאם עם HCl, הושווה למאגר Tris, שבו ה-pH הותאם עם חומצה אצטית. יתר על כן, שני מאגרי Tris הושוו למאגר HEPES, שם ה-pH הותאם עם NaOH. כל ההרכב של מאגר IVT 1x היה 40 מ"מ Tris/HEPES, 10 מ"מ DTT, 2 מ"מ ספרמידין ו-pH 7.9.
מ-100 מיקרוליטר של תערובת תגובות IVT, 2 מיקרוליטר של דגימת IVT הוצאו מתגובת IVT והורו ב-2 מיקרוליטר של 100 מ"מ EDTA כל 15 דקות בשעה הראשונה של הדגירה ולאחר מכן כל 30 דקות עד 180 דקות של הדגירה. תפוקת ה-mRNA הסופית הצפויה למצב תגובה זה הייתה 15 מ"ג/מ"ל, כלומר דילול פי 2 עקב מרווה לווה בדילול פי 400 ב-MPA+NaCl לניתוח. הדגימה דוללה פי 800 בסך הכל, כלומר אפילו בייצור של 15 מ"ג/מ"ל, ריכוז ה-mRNA שהועמס על העמודה הכרומטוגרפית האנליטית עדיין היה בריכוזי עקומת הכיול.
אזורי NTP ו-mRNA ב-A260 שולבו עבור כל דגימה בודדת בכל נקודת זמן בודדת (tx). לאחר מכן הומרו אזורים לריכוזים עבור mRNA, תוך שימוש בעקומת כיול ה-mRNA ואחוז הצריכה עבור NTPs, תוך שימוש בשטח A260 של NTPs ב-0 דקות (t0) כ-100% (ראה משוואה למטה).
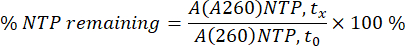
ניתן להציג את התוצאות כגרפים עבור כל תגובת IVT בודדת, כאשר הזמן מוצג על ציר ה-x וריכוז ה-mRNA ושאר ה-NTPs מוצגים על ציר ה-y (איור 2A). ניתן גם לשרטט את כל ה-IVT, כלומר ריכוזי mRNA בתלות בזמן, בגרף אחד, וניתן לחקור קינטיקה של ייצור mRNA ולבחור תנאי IVT אופטימליים (איור 2B).
עבור ניסוי האצווה הפדרלית, IVT היה מעורבב כפי שכתוב בטבלה 3. מ-300 מיקרוליטר של תערובת תגובת IVT, 2 מיקרוליטר של דגימת IVT הוסרו מתגובת IVT ורוו ב-2 מיקרוליטר של 100 מ"מ EDTA כל 30 דקות. בנוסף, הדגימה נלקחה מיד לאחר כל תוספת NTP+MgCl2 בתפזורת. תמיסת הזנה המכילה 42.4 מ"מ מכל NTP ו-152.5 מ"מ MgCl2 הוכנה מראש, ו-106 מיקרוליטר מכל 200 מ"מ NTP מעורבב עם 76.2 מיקרוליטר של 1 מ"מ MgCl2. הוקם משטר האכלה, שבו הוסיפו הזנת בולוס כל שעה (לאחר 60 דקות, 120 דקות, 180 דקות ו-240 דקות דגירה). משטר ההזנה מתואר בטבלה 3 הדגימות נותחו כמעט בזמן אמת עם ניתוח כמתואר עבור תגובת IVT בתפזורת. לאחר 300 דקות של ניטור תגובת ה-IVT דועכה.
ניתן להציג את התוצאות כייצור NTPs/mRNA שנותר לאורך זמן הדגירה. מכיוון שתוספות מזון בולוס גם מדללות את תגובת ה-IVT, ריכוז ה-mRNA ב-IVT יורד בכל תוספת הזנה (איור 3A). עלייה במסת ה-mRNA יכולה להימדד ולהיות מוצגת גם על ידי גורמי שעתוק (המוגדרים כ-mmRNA/mpDNA), המראים עלייה ליניארית בייצור mRNA לאורך זמן (איור 3B).
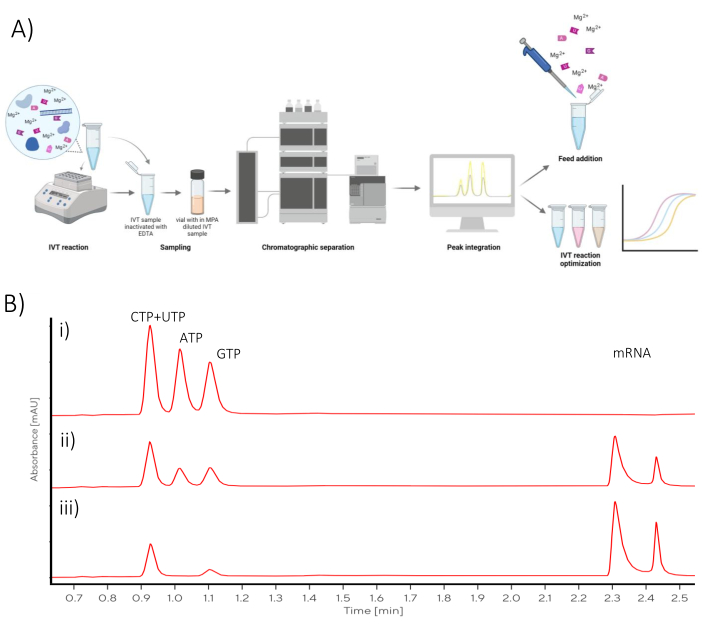
איור 1: סקירה סכמטית (A) ייצוג של זרימת עבודה של אופטימיזציה של IVT עם כימות ניתוח כרומטוגרפי מקוון של NTPs ו-mRNA. (B) כרומטוגרמות מייצגות של תגובת IVT אצווה שנדגמו ב-(i) t0, (ii) נקודת אמצע, (iii) נקודות זמן תגובה סופיות. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
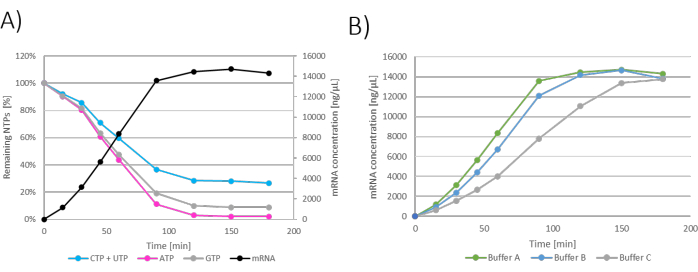
איור 2: גרפים IVT אצווה. (A) גרף מייצג של תגובת IVT עם ייצור mRNA וצריכת NTP על ציר ה-y. מישור ריכוז mRNA הנראה ב-120 - 180 דקות של דגירה, המתאם עם צריכת NTP כהגבלת NTP (ATP) נצרך ב-120 דקות. (B) גרף IVT המראה את ההשפעה של מאגר IVT על קינטיקה של IVT. IVT המכיל Buffer A (Tris+חומצה אצטית) הראה את ייצור ה-mRNA המהיר ביותר, ואחריו Buffer B (Tris+HCl) ו-Buffer C (HEPES+NaOH) כאיטי ביותר. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
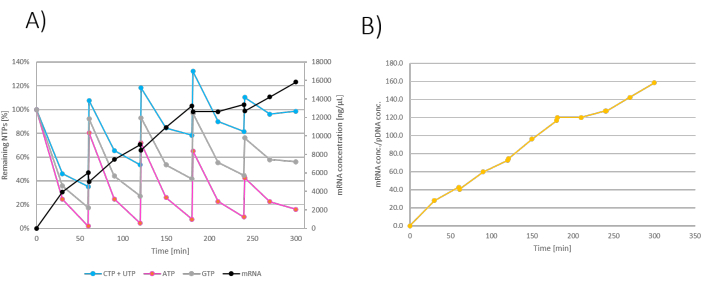
איור 3: גרף IVT של אצווה פד. (A) גרף IVT שבו הזנות NTP+MgCl2 נוספו ב-60, 120, 180 ו-240 דקות. ריכוז ה-mRNA בתגובת IVT יורד בכל תוספת הזנה עקב דילול עם הזנת NTP+MgCl2 . (B) עלייה במסת ה-mRNA שמוצגת על ידי גורם השעתוק היא ליניארית לאורך כל הדגירה של התגובה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
טבלה 1: פרוטוקול IVT גנרי. אנא לחץ כאן להורדת טבלה זו.
טבלה 2: פרוטוקול IVT אצווה. אנא לחץ כאן להורדת טבלה זו.
טבלה 3: פרוטוקול IVT של אצווה פד. אנא לחץ כאן להורדת טבלה זו.
Discussion
בשיטה זו מנותחות דגימות IVT בנקודות זמן שונות במהלך הדגירה באמצעות עמודה כרומטוגרפית אנליטית רב-מודאלית המפרידה בין NTPs, pDNA ו-mRNA, ובכך מאפשרת מעקב צמוד אחר צריכת NTP וייצור mRNA. שיטה זו מודדת NTPs וריכוז mRNA בצורה כמותית בהתבסס על שינויים באזורי A260 בנקודות זמן מוגדרות. מכיוון שהשיטה מספקת מידע על ריכוז NTP ו-mRNA, היא מתאימה מאוד לאופטימיזציה של IVT, כאשר המטרה העיקרית היא לרוב למקסם את תפוקת ה-mRNA ולמזער את זמן התגובה; לכן, הבנת ההשפעה של ריאגנטים שונים של IVT על הקינטיקה של ייצור mRNA היא קריטית22. אנו מראים כיצד ניתן ליישם ניטור מקוון כדי לייעל את תגובת ה-IVT במצבי אצווה ואצווה מוזנת.
היתרון העיקרי של שיטה זו הוא ניתוח מקוון המאפשר ניטור כמעט בזמן אמת של IVT כאשר כל דגימה מנותחת בפחות מ-8 דקות. הכנת דגימות לניתוח היא פשוטה, מכיוון שנדרש רק דילול ב-MPA ללא כל טיפול מקדים בדגימה. נפח הדגימה הנדרש לניתוח נמוך מאוד, למשל, 1 מיקרוליטר IVT. נפח פיפטינג קטן זה עלול לגרום לסטיות אנליטיות עקב שגיאות פיפטינג. עם זאת, מכיוון שהתפוקה הגבוהה של הגישה האנליטית מאפשרת מדידות משולשות, ניתן לזהות בקלות חריגים, ולא קשה לזהות סטיות מהעקומה הקינטית הצפויה.
אחת המגבלות של השיטה היא הפליטה הכרומטוגרפית של UTP ו-CTP. בדרך כלל אין צורך בכימות מדויק של UTP ו-CTP מכיוון שה-NTP המגביל ברצף mRNA הוא לרוב ATP או GTP. אם נדרש כימות נפרד של UTP ו-CTP, ניתן לרתום את ההבדל בספיגת UV ב-260 ננומטר ו-280 ננומטר עבור UTP ו-CTP כדי לגזור את השפע היחסי של כל NTP בשיא הכרומטוגרפי.
שיטה אנליטית זו אינה מפרידה בין מיני RNA מעל 100 נוקלאוטידים באורך; לכן, לא ניתן להשתמש בו כדי לזהות הבדלים באיכות ה-mRNA, למשל, הוא אינו מבדיל בין mRNA שאינו פוליאדניל לפוליאדניל, בין dsRNA או תעתיקים כושלים לבין ssRNA ואינו מתאים למחקרי יציבות מכיוון שהוא אינו יכול להבדיל בין mRNA מושפל ולא מתכלה. עם זאת, ניתן להשתמש בשיטה לכימות של שיטות RNA אחרות, כגון circRNA, tRNA ו-saRNA. למרות שמולקולות אלו משתנות בגודלן ובמבנהן, ניתן להשתמש באותה שיטה אנליטית כדי לחקור את תפוקות הייצור של כל אחת מהן בתגובת IVT.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
המחברים מבקשים להודות לתומאס קוסטלק, בלאז' בקאלאר, נייץ פבלין, אנדריאה גראמק ליבק ואנז'ה מרטינצ'יץ' צלאר על הדיונים המועילים.
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
References
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403(2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, Ö mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090(2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190(2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3(2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745(2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655(2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833(2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246(2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937(2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408(2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045(2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved