Method Article
크로마토그래피 앳라인(At-line) 모니터링을 사용한 mRNA 생산을 위한 체외 전사 반응 최적화(Optimization of in vitro transcription reaction for mRNA production using Chromatographic At-line monitoring)
요약
체외 전사(IVT)를 최적화하는 것은 비용 효율적인 mRNA 생산에 매우 중요합니다. 이 프로토콜은 IVT 반응의 앳라인 분석을 위한 분석 크로마토그래피 방법을 자세히 설명하고, 배치 또는 유가 배치 모드에서 NTP 고갈 및 mRNA 생산을 모니터링하며, 다양한 RNA 양식에 적용하여 생산성을 높이고 비용을 절감합니다.
초록
체외 전사 반응(IVT)은 RNA 중합효소(예: T7)에 의해 촉매되는 선형 DNA 주형에서 mRNA의 복잡한 다중 구성 요소 효소 합성입니다. IVT 시약의 높은 비용으로 인해 IVT는 mRNA 원료 의약품 생산 공정에서 중요한 단계이며 이 분야에서 집중적인 최적화의 초점이 되어 왔습니다. mRNA 생산 비용을 줄이려면 시약을 최적으로 활용해야 합니다. 효과적인 최적화를 위해서는 개별 시약이 반응 역학에 미치는 영향, 즉 뉴클레오시드 삼인산(NTP)의 소비 및 mRNA 생산에 대한 포괄적인 이해가 필요합니다. 전통적으로 mRNA 분석에는 저처리량, 종말점 분석 기법이 사용되었습니다. 이러한 방법은 mRNA 함량에 대한 귀중한 정보를 제공하지만, IVT 반응을 완전히 이해하려면 실시간에 가까운 실시간 분석이 필요합니다. NTP와 pDNA 및 mRNA를 분리하는 액체 크로마토그래피 분석 방법을 거의 실시간으로 사용하여 IVT 반응 역학을 연구하는 방법을 보여줍니다. 다양한 IVT 성분이 역학 및 수율에 미치는 영향에 대한 지식을 바탕으로 신속한 크로마토그래피 분석을 사용하여 배치 IVT 반응을 생산성을 더욱 높이고 반응의 전체 비용을 절감하는 유가식 모드 모드로 변환할 수 있습니다.
서문
COVID-19 팬데믹은 생물의학의 전례 없는 혁명을 촉발하여 유럽과 미국 모두에서 BioNTech/Pfizer의 Comirnaty 및 Moderna의 Spikevax와 같은 mRNA 기반 백신의 신속한 개발 및 승인으로 이어졌습니다 1,2,3. 이러한 백신의 놀라운 효능과 빠른 개발은 감염병뿐만 아니라 암 면역 요법, 단백질 대체 요법, 재생 의학 및 세포 재프로그래밍에 대한 mRNA 기술의 엄청난 치료 잠재력을 강조했습니다 4,5. 종종 mRNA 혁명이라고 불리는 이 시대는 효율적이고 비용 효율적인 mRNA 생산 공정의 필요성을 강조합니다.
mRNA의 생산에는 여러 단위 작업이 포함되며, 체외 전사(IVT) 반응은 중요하고 가장 비용 집약적인 단위 작업입니다 6,7,8. IVT 반응은 마그네슘 이온이 존재하는 상태에서 RNA 중합효소(일반적으로 T7 RNA 중합효소) 및 NTP(nucleoside triphosphates)를 사용하여 DNA 주형에서 mRNA를 합성합니다. 이 과정은 비교적 간단하며 짧은 시간 내에 많은 양의 mRNA를 생산할 수 있으며 몇 시간 내에 2-5g/L의 반응 수율을 달성하고 일부 보고서에서는 최대 14g/L의 반응 수율을 달성할 수 있습니다 9,10,11. 그러나 IVT 반응 수율의 최적화는 생산 비용을 절감하고 mRNA 백신 및 치료제의 확장성을 보장하는 데 중요하며12 NTP 소비에 영향을 미치는 서열 차이로 인해 각 구성물 또는 구성물 제품군에 대해 필요할 수 있습니다.
반응은 일반적으로 배치 공정으로 수행되지만 최근 유가식 처리 및 앳라인 분석의 발전으로 mRNA 생산을 최적화하기 위한 새로운 길이 열렸습니다 7,13. 볼루스 또는 시약의 연속 첨가를 포함하는 유가식 반응은 기질 억제 및 보조 인자 의존성 산물 분해를 방지하여 잠재적으로 반응 시간을 연장하고 수율을 증가시킬 수 있습니다14,15.
신속한 앳라인 분석의 개발은 IVT 분야에서 상당한 진전을 이루었으며, 최소한의 분석 지연으로 NTP 및 mRNA와 같은 주요 반응 성분을 모니터링할 수 있게 되었으며13,16 일반적으로 mRNA 농도17에만 초점을 맞추는 IVT 모니터링에 차원을 더했습니다. 전통적으로 mRNA는 폴리아크릴아미드 겔 전기영동(PAGE), 아가로스 겔 전기영동 또는 모세관 전기영동과 같은 기술을 사용하여 분석되었습니다. 이는 종말점 방법이므로 실시간 IVT 모니터링에 사용할 수 없습니다. 이 프로토콜에 설명된 분석의 대안은 light-up RNA, aptamer 및 형광 염료 쌍을 사용하는 방법입니다. 이 방법에서는 RNA 압타머를 RNA에 태그하고 발광 형광 염료로 배양합니다. 그런 다음 전사 활성을 형광 강도17로 시각화할 수 있습니다. 이를 통해 IVT 21 동안 전사된 mRNA의 양과 품질을 실시간으로 분석할 수 있지만 다른 IVT 구성 요소(예: NTP)를 동시에 모니터링할 수는 없습니다. NTP 및 mRNA의 실시간 정량화를 잠재적으로 제공할 수 있는 또 다른 대안은 Raman spectroscopy18이지만, 현재까지 IVT 모니터링에 대한 유용성을 입증한 보고서는 없으며, 이는 필요한 선택성과 감도를 도출하기 위해 방법의 추가 최적화가 여전히 필요함을 시사합니다.
더 빠르고 더 선택적인 분석의 지속적인 개발은 배치 및 유가식 배치 모드에서 더 높은 수율을 위해 IVT 최적화를 지원합니다. 반응은 여러 상호 작용 요인이 있는 복잡한 다중 파라미터 프로세스로 유지되며, 이에 대한 최적화는 구성 유형(예: mRNA 대 saRNA)에 따라 다를 수 있습니다. Mg2+ 이온 및 NTP의 농도 및 비율은 특히 영향력이 크며, 선천성 면역 체계의 강력한 자극제인 이중 가닥 RNA(dsRNA)의 형성을 최소화하면서 mRNA 수율을 극대화하기 위해 최적의 수준을 신중하게 균형을 맞춰야 합니다19,20. 최근에는 dsRNA 형성을 줄이기 위한 접근법으로 정상 상태 수준에서 UTP를 공급하는 것이 보고되었습니다21. 거의 실시간으로 UTP 수준을 모니터링하면 공정 제어 수준이 추가됩니다.
이 프로토콜에서는 IVT 반응의 앳라인 모니터링이 배치 및 유가식 배치 모드에서 mRNA 생산의 수율을 높일 수 있는 방법을 보여줍니다.
프로토콜
1. 완충액 준비
참고: 모든 버퍼는 RNase-free로 준비되어야 하며, 이는 모든 화학 물질 및 유리 제품이 RNase-free 작업에만 사용되어야 하며 주의해서 취급해야 함을 의미합니다. 완충액 준비 및 유리 기구 세척에 사용되는 물은 뉴클레아제가 없는 것으로 인증되어야 합니다. 실험을 수행하기 전에 모든 작업 표면과 유리 제품에 RNase를 제거하는 오염 제거 시약을 분무해야 합니다. 오염 제거 시약은 유리 제품/작업 영역을 사용하기 전에 RNase가 없는 물로 철저히 씻어내야 합니다. 가능하면 멸균 및 일회용 소모품을 사용하십시오.
- IVT 완충액 준비: 400mM Tris, 10mM DTT, 20mM 스페르미딘, pH 7.9를 첨가하여 10x IVT 완충액을 준비합니다. 100mM EDTA, pH 8을 함유하는 담금질 완충액을 준비합니다. 0.22μm PES 필터를 통해 각 버퍼를 필터링합니다.
- 이동상의 제조 : 50 mM HEPES, pH 7.0, 50 mM Hepes, 100 mM Na4P2O7, pH 8.3, 0.1 M NaOH, 1 M NaCl, 0.5 M HEPES를 첨가하여 이동상 D (MPD)를 첨가하여 이동상 A (MPA)를 제조하고, pH 7.0. 0.22μm PES 필터를 통해 각 버퍼를 필터링합니다.
2. 체외 전사 반응(IVT)의 준비
참고 1: IVT 반응은 처음에 모든 시약을 첨가하고 NTP가 고갈되거나 mRNA 생산이 안정기에 도달한 후 반응이 중지되는 배치 반응일 수 있으며, 또는 고갈된 NTP에 추가 NTP가 보충되고 해당 mRNA 생산이 추가로 증가하는 유가식 반응일 수 있습니다. IVT 반응에 사용되는 선형 pDNA 주형은 플라스미드 DNA에서 폴리(A) 서열 직후에 절단되는 제한 효소를 사용하거나 PCR 반응에 의해 얻을 수 있습니다. 두 경우 모두 반응 후 크로마토그래피 또는 상용 DNA 정제 키트를 사용한 정제가 이루어져야 합니다.
- 배치 IVT 반응
- Thermoblock을 37°C 및 300rpm으로 설정합니다. 효소를 제외한 표 1 에 나열된 모든 시약을 해동하고 써모블록에서 37°C로 예열합니다. ATP, CTP, UTP 및 GTP의 100mM 원액을 사용하여 25mM NTP 혼합물을 준비합니다.
- 시약이 해동되는 동안 시간 경과 실험을 위해 샘플 부분 표본을 준비합니다. 0.5mL 멸균 튜브와 2μL의 100mM EDTA를 각 튜브에 피펫팅합니다. 각 튜브에 지정된 IVT 번호(예: IVT001, IVT002 등)와 시점(예: 0분, 15분, 30분, 60분 등)을 표시합니다.
- 시약을 해동하고 37°C에서 약 15분 동안 보관한 경우 -20°C 냉동고에서 효소(T7 RNA 중합효소, 피로포스파타아제, Rnase 억제제)를 제거하고 냉동고에 다시 보관할 때까지 쿨러에 보관하십시오.
- 개별 IVT 시약(효소 제외)을 혼합하려면 와류를 사용하십시오. 위아래로 부드럽게 피펫팅하여 효소를 혼합합니다.
- 1.5mL 튜브에 실험 설계(예: 일반 IVT 프로토콜은 표 1에 나와 있음)에 설정된 비율로 IVT 시약을 혼합합니다. IVT 최적화를 수행할 때 최종 IVT 부피는 50-100μL 사이일 수 있습니다. 총 IVT 부피를 설계할 때 시간 경과 실험 중 반응에서 추출할 2μL 샘플의 수를 고려하십시오. 샘플의 총 부피가 총 IVT 부피의 50%를 초과하지 않는지 확인합니다(예: 3시간 동안 15분마다 2μL를 샘플링하는 경우 채취한 부분 표본의 총 부피는 30μL이며, 이는 IVT의 총 부피가 최소 60μL여야 함을 의미함).
- IVT를 조심스럽게 혼합하고 T7 RNA Polymerase를 마지막 시약으로 첨가합니다. 모든 시약이 추가되면 위아래로 피펫팅하여 IVT를 완전히 혼합하고 와류를 사용하지 마십시오.
- 모든 IVT 시약을 첨가하고 균질화한 직후, 튜브에서 2μL의 IVT 혼합물을 제거하고 2.1.2단계(시점 0분)에 설명된 2μL의 100mM EDTA를 포함하는 이전에 준비된 0.5mL 튜브에 피펫팅합니다.
- IVT 반응 혼합물이 포함된 반응 튜브를 열 블록에 넣고 37°C에서 배양합니다.
- 타이머를 원하는 시간 간격(예: 15분/30분)으로 설정합니다. 각 시점에서 준비된 각 IVT 반응 혼합물에서 2μL의 IVT 샘플을 채취하여 2μL의 100mM EDTA를 포함하는 이전에 준비된 튜브(단계 2.1.2 참조)에 피펫팅합니다. 이러한 분주물은 크로마토그래피 분석에 사용됩니다(3단계 참조).
- 크로마토그래피 분석에서 NTP의 완전한 고갈을 확인한 후 EDTA를 사용한 벌크 IVT 반응을 최종 농도 50mM까지 소멸합니다(예: 30μL의 IVT가 100mM EDTA 30μL로 비활성화됨).
- 유가식 IVT 반응
참고: NTP가 고갈된 후 NTP와 Mg2+ 이온을 첨가하여 mRNA 생산을 추가로 증가시킬 수 있습니다.- 배치 IVT 반응을 혼합하고 2.1단계에서 설명한 대로 샘플링합니다. 공급 배치에 대한 NTP+MgCl2 공급을 준비합니다. 예를 들어 500 μL의 NTP+MgCl2 혼합물을 준비하려면 각 200 mM NTP 106 μL, 1 M MgCl2 76.25 μL 및 ddH2O 5.5 μL를 혼합합니다. 혼합물의 최종 농도는 각 NTP 42.4 mM 및 152.5 mM MgCl2 입니다.
- 3단계에서 설명한 앳라인 분석을 통해 NTP 사용량을 모니터링합니다. 앳라인 분석에서 NTP 농도가 시작 농도의 10% 미만으로 떨어지는 것이 확인되면 NTP+Mg2+ 가 포함된 사료를 벌크 IVT에 추가합니다.
- 분석을 위해 모든 부분 표본을 채취한 후 1.5mL 튜브에 남아 있는 벌크 IVT 반응의 정확한 부피를 측정/계산합니다. 최종 농도가 각 NTP의 4mM과 MgCl2의 12mM가 되도록 적절한 부피의 NTP+MgCl2 혼합물을 IVT에 추가합니다. 예를 들어, 1.5mL 튜브에 80μL의 벌크 IVT가 남아 있는 경우 16.8μL의 NTP+MgCl2 혼합물을 추가합니다.
- 2.1단계에서 설명한 대로 샘플링을 계속합니다. NTP가 다시 고갈된 후 섹션 2.2.2를 반복합니다. 원하는 mRNA 생산이 이루어질 때까지 공급 및 샘플링을 계속합니다.
- 원하는 mRNA 생산이 달성된 후 2.1.10단계와 같이 전체 반응을 비활성화합니다.
3. 크로마토그래피 분석 준비
- 표준 및 시스템 적합성 테스트(SST) 준비
- 캡핑 시약, NTP, pDNA 템플릿 및 mRNA를 결합하여 SST를 준비합니다. 최종 농도가 캡핑 시약의 경우 약 3μM, 각 NTP의 경우 5μM, pDNA 템플릿의 경우 4ng/μL, mRNA의 경우 10ng/μL인지 확인합니다. 최종 SST에 0.1M NaCl이 포함되어 있는지 확인합니다.
- 각 해석 전에 새 검량선을 준비합니다. 알려진 농도의 정제된 mRNA 샘플을 MPA로 희석하여 검량 표준물질을 생성합니다. 검량점은 0.5ng/μL, 2ng/μL, 5ng/μL, 10ng/μL, 15ng/μL 및 20ng/μL이어야 하며, 각각 최종 농도는 0.1M NaCl입니다.
- 필요한 경우 모든 NTP에 대한 보정 곡선을 준비합니다. 보정 표준에서 각 NTP의 최종 농도가 0.5μM, 2μM, 5μM, 10μM, 15μM 및 20μM인지 확인합니다.
- 컬럼 컨디셔닝 및 분석 준비
- 분석 전 12시간 동안 실온에서 0.1mL 분석 컬럼을 평형화합니다. 컬럼 하우징에 표시된 방향으로 크로마토그래피 시스템에 컬럼을 부착합니다.
- 1mL/분 유속으로 컬럼을 플러시합니다: 먼저 50개의 ddH2O 컬럼 부피(CV)로, 그 후에는 50CV의 MPA로 플러시합니다.
- 기준선을 설정하기 위해 분석 전에 최소 3개의 빈 샘플(MPA만 주입)을 실행합니다. Skok et al.15에 기술된 크로마토그래피 방법을 사용합니다.
- 베이스라인이 안정되고 두 개의 연속 blank 측정이 유사하면(재현 가능한 베이스라인) SST 샘플과 검량선 샘플을 실행합니다.
- SST 기준이 충족되고 캡핑 시약과 NTP의 분리가 만족스러우면 IVT 샘플 분석을 진행합니다. 모든 IVT 샘플을 분석한 후 SST 표준물질을 다시 주입하여 분석 전반에 걸쳐 시스템 상태가 안정적으로 유지되었는지 확인합니다.
- 시료 전처리
참고: 크로마토그래피 분석을 위한 샘플은 분석 직전에 준비됩니다. 그렇지 않으면 샘플을 -20 ° C (최대 24 시간) 또는 <-65 ° C (24 시간 이상)의 냉동고에 보관합니다.- 크로마토그래피 분석을 위해 희석하기 전에 퀀칭된 IVT 샘플을 와류로 회전시키고 스핀다운합니다. Skok et al.15에 설명된 방법을 사용하여 크로마토그래피 분석으로 모든 EDTA 불활성화 IVT 샘플(2.1.9단계 참조)을 분석합니다.
- 분석하기 전에 IVT 샘플을 희석하여 검량선 내에서 농도를 달성합니다. 시간 과정 실험 전반에 걸쳐 동일한 희석 계수를 사용하여 NTP 영역을 비교하여 NTP가 고갈된 시기를 확인할 수 있는지 확인합니다.
- IVT 샘플에 필요한 최소 희석을 결정하려면 다음 공식을 사용하십시오: 필요한 희석 = (예상 말단 mRNA 농도) / (불활성화 중 희석) / (가장 높은 보정 표준)
예를 들어, IVT에서 8mg/mL의 mRNA 생산이 예상되는 경우 담금질된 샘플의 농도는 4mg/mL가 됩니다. 보정 곡선 내에서 농도를 달성하려면 샘플을 200배 더 희석하십시오.
필요한 희석 = (8000ng/μL]) / 2 / (20ng/μL) = 200 - MPA와 4M NaCl을 원뿔형 유리 바이알에 피펫팅합니다. 샘플에서 NaCl의 최종 농도가 100mM인지 확인합니다. 마지막으로 담금질된 IVT 샘플을 추가합니다.
- Vortex는 시료를 준비하고 분석을 위해 자동 시료 주입기에 삽입했습니다. 오토샘플러 온도를 4°C로 설정합니다.
- 100μL의 희석된 샘플을 분석 컬럼에 주입합니다. 각 IVT 시료에 대해 흡광도 A260 및 A280을 측정합니다.
- 농도가 알려진 시료를 분석하거나 면적 차이에 따른 NTP 소비량 추적이 필요하지 않은 경우, 시료를 희석하여 10ng/μL의 목표 농도를 달성합니다.
4. 시료 정량화 및 데이터 분석
- 각 IVT 샘플을 통합하기 전에 샘플 크로마토그램에서 빈 샘플을 뺍니다.
- A260에서 모든 NTP, pDNA 및 mRNA 피크를 통합하고 피크 영역을 사용하여 IVT의 NTP 및 mRNA 농도를 계산합니다.
참고: 또는 피크 면적을 사용하여 희석 계수가 모든 시점에서 일관된 경우 IVT 반응에 남아 있는 각 NTP의 백분율을 비교할 수 있습니다. - 앳라인(at-line) 크로마토그래피 분석으로 측정된 mRNA/NTP 농도를 기반으로 IVT를 비활성화할 수 있는지 또는 IVT 반응에 추가 NTP를 공급해야 하는지 여부를 결정합니다(2단계 참조).
결과
이 프로토콜에 설명된 크로마토그래피 분석은 IVT 최적화 또는 배치 IVT 반응을 유가식 반응으로 전환하는 데 사용할 수 있습니다(그림 1).
IVT 반응의 역학에 대한 상이한 완충액 조성의 효과를 테스트하기 위해, 표 2에 기재된 프로토콜에 따라 3개의 상이한 IVT 반응을 혼합하였다. pH가 HCl로 조정된 Tris를 포함하는 버퍼를 아세트산으로 pH를 조정한 Tris 버퍼와 비교했습니다. 또한, 두 Tris 완충액을 Hepes 완충액과 비교하고, 여기서 pH를 NaOH로 조정하였다. 모든 1x IVT 완충액의 조성은 40mM Tris/HEPES, 10mM DTT, 2mM 스페르미딘 및 pH 7.9였습니다.
100 μL의 IVT 반응 혼합물에서 2 μL의 IVT 샘플을 IVT 반응에서 꺼내 배양 첫 15 분마다 2 μL의 100 mM EDTA로 담금질한 다음 배양 180 분까지 30 분마다 담금질했습니다. 이 반응 조건에 대한 예상 최종 mRNA 수율은 15 mg/mL였으며, 이는 담금질로 인한 2배 희석 후 분석을 위해 MPA+NaCl을 400배 희석했음을 의미합니다. 샘플은 총 800배 희석되었는데, 이는 15mg/mL 생산에서도 분석 크로마토그래피 컬럼에 로드된 mRNA 농도가 여전히 검량선 농도 내에 있음을 의미합니다.
A260의 NTP 및 mRNA 영역은 각 개별 시점(tx)에서 각 개별 샘플에 대해 통합되었습니다. 그런 다음 mRNA 보정 곡선 및 NTP에 대한 소비율을 사용하여 0분(t0)에서 NTP의 A260 면적을 100%로 사용하여 영역을 mRNA의 농도로 변환했습니다(아래 방정식 참조).
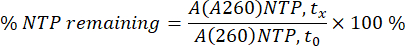
결과는 각 개별 IVT 반응에 대한 그래프로 표시할 수 있으며, 여기서 시간은 x축에, mRNA 농도는 y축에 표시되고 나머지 NTP는 y축에 표시됩니다(그림 2A). 시간에 의존하는 mRNA 농도를 의미하는 모든 IVT도 하나의 그래프에 함께 표시할 수 있으며, mRNA 생산 역학을 연구하고 최적의 IVT 조건을 선택할 수 있습니다(그림 2B).
유가식 배치 실험의 경우, IVT는 표 3과 같이 혼합되었습니다. 300 μL의 IVT 반응 혼합물에서 2 μL의 IVT 샘플을 IVT 반응에서 제거하고 30분마다 2 μL의 100 mM EDTA로 퀀칭했습니다. 또한, 각 벌크 NTP+MgCl2첨가 직 후 샘플을 채취했습니다. 각 NTP 42.4 mM 및 152.5 mMMgCl2 를 함유하는 공급 용액을 미리 제조하고, 각 개별 200 mM NTP 106 μL를 1 M MgCl2 76.2 μL와 혼합하였다. 수유 체제가 확립되어 매시간마다(60분, 120분, 180분, 240분 배양 시) 볼루스 사료를 추가했습니다. 공급 방식은 표 3 에 설명되어 있으며, 샘플은 벌크 IVT 반응에 대해 설명된 바와 같이 분석을 통해 거의 실시간으로 분석되었습니다. 300분 모니터링 후 IVT 반응을 소멸시켰습니다.
결과는 배양 시간 경과에 따른 잔여 NTP/mRNA 생산으로 표시할 수 있습니다. 볼루스 사료 첨가는 IVT 반응도 희석시키기 때문에 각 사료 첨가에서 IVT의 mRNA 농도가 떨어집니다 (그림 3A). mRNA 질량의 증가는 또한 전사 인자(mmRNA/mpDNA로 정의됨)에 의해 측정되고 제시될 수 있으며, 이는 시간 경과에 따른 mRNA 생산의 선형 증가를 보여줍니다 (그림 3B).
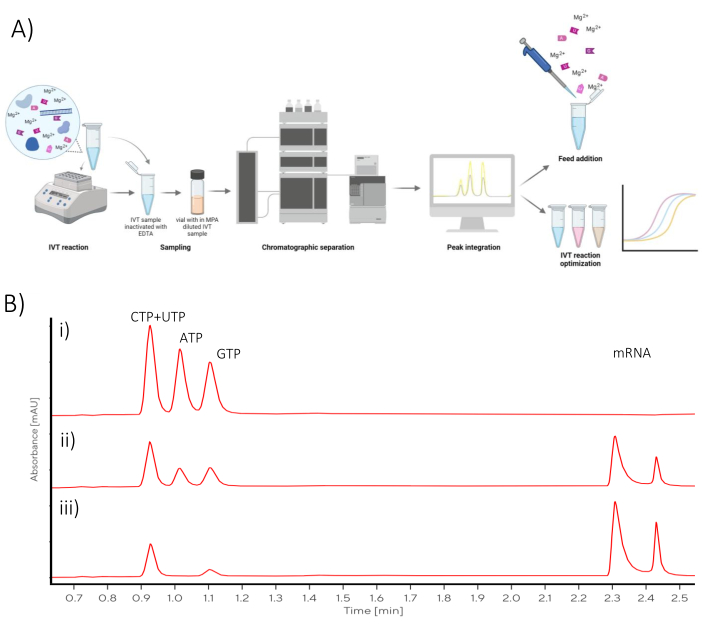
그림 1: 개략적 개요 (A) NTP 및 mRNA의 앳라인 크로마토그래피 분석 정량화를 통한 IVT 최적화 워크플로우 표현. (B) (i) t0, (ii) 중간점, (iii) 최종 반응 시간 지점에서 샘플링된 배치 IVT 반응의 대표 크로마토그램. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
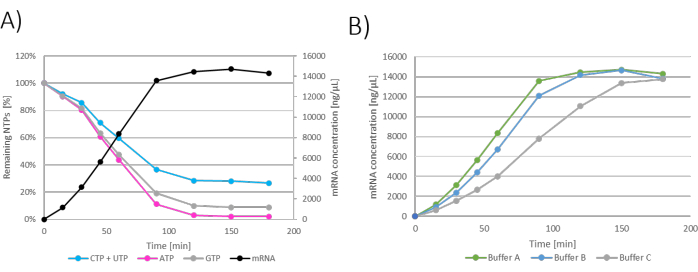
그림 2: 배치 IVT 그래프. (A) y축에서 mRNA 생산 및 NTP 소비에 따른 IVT 반응의 대표 그래프. 인큐베이션 120 - 180분에서 볼 수 있는 mRNA 농도 고원은 NTP(ATP)가 120분에 소비됨에 따라 NTP 소비와 상관관계가 있습니다. (B) IVT 버퍼가 IVT 동역학에 미치는 영향을 보여주는 IVT 그래프. 완충액 A(트리스+아세트산)를 함유한 IVT가 가장 빠른 mRNA 생산을 보였고, 완충액 B(트리스+HCl)와 완충액 C(HEPES+NaOH)가 가장 느렸다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
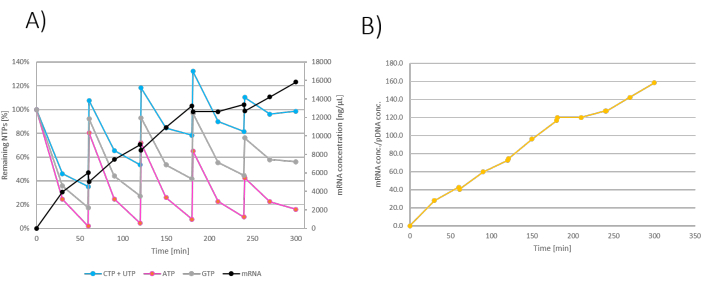
그림 3: 유가식 IVT 그래프. (A) NTP+MgCl2 피드가 60분, 120분, 180분 및 240분에 첨가된 IVT 그래프. NTP+MgCl2 피드로 희석되어 IVT 반응에서 mRNA 농도가 떨어집니다. (B) 전사 인자에 의해 나타난 mRNA 질량의 증가는 반응 배양 전반에 걸쳐 선형입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: 일반 IVT 프로토콜. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 2: 배치 IVT 프로토콜. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 3: Fed-batch IVT 프로토콜. 이 표를 다운로드하려면 여기를 클릭하십시오.
토론
이 방법에서는 NTP, pDNA 및 mRNA를 분리하는 분석 크로마토그래피 멀티모달 컬럼을 사용하여 배양 중 서로 다른 시점에서 IVT 샘플을 분석하므로 NTP 소비 및 mRNA 생산을 면밀히 모니터링할 수 있습니다. 이 방법은 지정된 시점에서 A260 영역의 변화를 기반으로 NTP 및 mRNA 농도를 정량적으로 측정합니다. 이 방법은 NTP 및 mRNA 농도에 대한 정보를 제공하기 때문에 mRNA 수율을 최대화하고 반응 시간을 최소화하는 것이 주요 목표인 IVT 최적화에 매우 적합합니다. 따라서 mRNA 생산의 동역학에 대한 다양한 IVT 시약의 효과를 이해하는 것이 중요합니다22. 배치 및 유가식 배치 모드에서 IVT 반응을 최적화하기 위해 앳라인 모니터링을 적용할 수 있는 방법을 보여줍니다.
이 방법의 주요 장점은 각 샘플이 8분 미만으로 분석될 때 IVT를 거의 실시간으로 모니터링할 수 있는 앳라인 분석입니다. 사전 시료 전처리 없이 MPA를 희석하기만 하면 되기 때문에 분석을 위한 시료 준비는 간단합니다. 분석에 필요한 시료 부피는 매우 적습니다(예: 1μL의 IVT). 이 작은 피펫팅 부피는 잠재적으로 피펫팅 오류로 인해 분석 편차를 초래할 수 있습니다. 그러나 분석 접근 방식의 높은 처리량으로 인해 3중 측정이 가능하기 때문에 이상값을 쉽게 식별할 수 있으며 예상 운동 곡선과의 편차를 감지하는 것이 어렵지 않습니다.
이 방법의 한계 중 하나는 UTP와 CTP의 크로마토그래피 공동 용리입니다. mRNA 염기서열에서 제한 NTP는 종종 ATP 또는 GTP이기 때문에 UTP 및 CTP의 정확한 정량화는 일반적으로 필요하지 않습니다. UTP와 CTP의 별도 정량화가 필요한 경우, UTP와 CTP에 대한 260nm 및 280nm에서의 UV 흡광도 차이를 활용하여 크로마토그래피 피크에서 각 NTP의 상대적 풍부도를 도출할 수 있습니다.
이 분석 방법은 길이가 100개 이상의 뉴클레오티드 RNA 종을 분리하지 않습니다. 그러므로, mRNA 품질의 차이를 검출하는 데 사용할 수 없으며, 예를 들어, 비폴리아데닐화 mRNA와 폴리아데닐화 mRNA, dsRNA 또는 유산 전사체와 ssRNA 사이를 구별하지 못하며, 분해된 mRNA와 비분해된 mRNA를 구별할 수 없기 때문에 안정성 연구에 적합하지 않습니다. 그러나 이 방법은 circRNA, tRNA 및 saRNA와 같은 다른 RNA 양식의 정량화에 활용될 수 있습니다. 이러한 분자는 크기와 구조가 다양하지만 동일한 분석 방법을 사용하여 IVT 반응에서 각각의 생산 수율을 연구할 수 있습니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
저자는 유용한 토론을 해준 Tomas Kostelec, Blaž Bakalar, Nejc Pavlin, Andreja Gramc Livk, Anže Martinčič Celjar에게 감사의 뜻을 전한다.
자료
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
참고문헌
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403 (2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, &. #. 2. 1. 4. ;. mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090 (2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190 (2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3 (2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745 (2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655 (2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833 (2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246 (2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937 (2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408 (2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045 (2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유