Method Article
クロマトグラフィーアットラインモニタリングを用いたmRNA産生のための in vitro 転写反応の最適化
要約
in vitro転写(IVT)の最適化は、費用対効果の高いmRNA産生にとって重要です。このプロトコルは、IVT反応のアットライン分析、バッチモードまたはフェドバッチモードでのNTP枯渇とmRNA産生のモニタリングのための分析クロマトグラフィー法を詳述しており、さまざまなRNAモダリティに適用でき、生産性を向上させ、コストを削減します。
要約
In vitro 転写反応(IVT)は、RNAポリメラーゼ(T7など)によって触媒される線状DNAテンプレートからのmRNAの複雑な多成分酵素合成です。IVT試薬のコストが高いため、IVTはmRNA原薬製造プロセスの重要なステップであり、この分野では集中的な最適化の焦点となっています。mRNA産生のコストを削減するには、試薬を最適に利用する必要があります。効果的な最適化には、個々の試薬が反応速度論に与える影響、すなわちヌクレオシド三リン酸(NTP)の消費とmRNAの産生を包括的に理解する必要があります。従来、mRNAの分析には、ロースループットのエンドポイント分析技術が使用されてきました。このような方法ではmRNA含有量に関する貴重な情報が得られますが、IVT反応を完全に理解するには、アットラインでほぼリアルタイムの分析が必要です。私たちは、NTP、pDNA、mRNAを分離する液体クロマトグラフィー分析法をほぼリアルタイムに使用して、IVT反応速度を研究する方法を示します。さまざまなIVT成分が速度論と収率に与える影響に関する知識があれば、迅速なクロマトグラフィー分析を使用して、バッチIVT反応をフェッドバッチモードに変換し、生産性をさらに向上させ、反応の全体的なコストを削減できます。
概要
COVID-19のパンデミックは、生物医学に前例のない革命をもたらし、BioNTech/PfizerのComirnatyやModernaのSpikevaxなどのmRNAベースのワクチンがヨーロッパと米国の両方で急速に開発され、承認されるようになりました1,2,3。これらのワクチンの優れた有効性と急速な開発は、感染症だけでなく、がん免疫療法、タンパク質補充療法、再生医療、細胞リプログラミングなど、mRNA技術の計り知れない治療の可能性を浮き彫りにしています4,5。この時代は、しばしばmRNA革命と呼ばれ、効率的で費用対効果の高いmRNA生産プロセスの必要性を強調しています。
mRNAの産生には複数の単位操作が含まれ、in vitro転写(IVT)反応は重要で最もコストのかかる単位操作である6,7,8。IVT反応は、マグネシウムイオンの存在下でRNAポリメラーゼ(通常はT7 RNAポリメラーゼ)とヌクレオシド三リン酸(NTP)を使用して、DNAテンプレートからmRNAを合成します。このプロセスは比較的単純で、短時間で大量のmRNAを産生することができ、数時間で2〜5 g/Lの反応収率を達成し、一部の報告では最大14 g/Lの反応収率を達成しています9,10,11。しかし、IVT反応収率の最適化は、mRNAワクチンおよび治療薬12の製造コストを削減し、スケーラビリティを確保するために重要であり、NTP消費に影響を与える配列の違いにより、各コンストラクトまたはコンストラクトファミリーに必要となる場合がある。
反応は通常、バッチプロセスとして行われるが、近年のフェッドバッチ処理とアットライン分析の進歩により、mRNA産生を最適化するための新たな道が開かれている7,13。ボーラスまたは試薬の連続的な添加を伴うフェドバッチ反応は、基質の阻害および補因子依存性の生成物の劣化を防ぐことにより、反応時間を延長し、収率を増加させることができる可能性がある14,15。
IVTの分野における高速アットライン分析の開発は大きな進歩であり、NTPやmRNAなどの主要な反応成分のモニタリングを最小限の分析遅延で可能にし13,16、通常はmRNA濃度のみに焦点を当てているIVTモニタリングに新たな次元を加えています17。従来、mRNAはポリアクリルアミドゲル電気泳動(PAGE)、アガロースゲル電気泳動、キャピラリー電気泳動などの技術を用いて解析されてきました。これらはエンドポイント方式であるため、リアルタイムのIVTモニタリングには使用できません。このプロトコルに記載されている分析の代替手段は、ライトアップRNAアプタマーと蛍光色素のペアを使用する方法です。この方法では、RNAアプタマーをRNAにタグ付けし、ライトアップ蛍光色素とインキュベートします。次いで、転写活性は蛍光強度17によって可視化することができる。これにより、IVT 21中に転写されたmRNAの量と質をリアルタイムで分析できますが、他のIVTコンポーネント(NTPなど)を同時にモニタリングすることはできません。NTPおよびmRNAのリアルタイム定量を可能にする可能性のある別の代替手段は、ラマン分光法18ですが、これまでのところ、IVTモニタリングへの有用性を示す報告はなく、必要な選択性と感度を引き出すためには、このメソッドのさらなる最適化が必要であることが示唆されています。
より迅速で選択的な分析の継続的な開発は、バッチモードとフェッドバッチモードでの高収率のためのIVTの最適化をサポートします。反応は、複数の相互作用因子を持つ複雑なマルチパラメータープロセスのままであり、最適性はコンストラクトタイプ(mRNAとsaRNAなど)によって異なる場合があります。Mg2+イオンとNTPの濃度と比率は特に影響力があり、それらの最適レベルは、自然免疫系の強力な刺激剤である二本鎖RNA(dsRNA)の形成を最小限に抑えながら、mRNA収量を最大化するために慎重にバランスをとる必要があります19,20。最近、定常状態でのUTPの供給は、dsRNA形成を減少させるアプローチとして報告された21。UTPレベルをほぼリアルタイムで監視することで、プロセス制御のレベルがさらに向上します。
このプロトコルでは、IVT反応のアットラインモニタリングにより、バッチモードおよびフェドバッチモードでのmRNA産生の収量がどのように増加するかを示します。
プロトコル
1. バッファーの調製
注:すべてのバッファーはRNaseフリーで調製する必要があり、すべての化学薬品とガラス器具はRNaseフリーの作業にのみ使用し、注意して取り扱う必要があります。緩衝液の調製とガラス器具の洗浄に使用される水は、ヌクレアーゼフリーであると認定されている必要があります。実験を行う前に、すべての作業面とガラス器具に、RNaseを除去する除染試薬を噴霧する必要があります。除染試薬は、ガラス器具/作業エリアを使用する前に、RNaseフリーの水で完全に洗い流す必要があります。可能であれば、滅菌済みで1回限りの消耗品を使用してください。
- IVTバッファーの調製:400 mM Tris、10 mM DTT、20 mMスペルミジン、pH 7.9を添加して、10x IVTバッファーを調製します。100 mM EDTA、pH 8を含むクエンチングバッファーを調製します。各バッファーを0.22 μm PESフィルターでろ過します。
- 移動相の調製:50 mM HEPES、pH 7.0 を添加して移動相 A(MPA)、50 mM HEPES を添加して移動相 B(MPB)、100 mM Na4P2O7、pH 8.3、0.1 M NaOH、1 M NaCl を添加して移動相 C(MPC)、0.5 M HEPES を添加して移動相 D(MPD)を調製、 pH 7.0です。各バッファーを0.22 μm PESフィルターでろ過します。
2. in vitro 転写反応(IVT)の調製
注1:IVT反応は、すべての試薬を最初に添加し、NTPが枯渇した/mRNA産生がプラトーに達した後に反応を停止するバッチ反応、または枯渇したNTPに追加のNTPを補充し、それに伴ってmRNA産生がさらに増加するフェドバッチ反応である場合があります。IVT反応で用いる直鎖状pDNAテンプレートは、プラスミドDNAのポリ(A)配列直後に切断する制限酵素を用いるか、PCR反応により取得することができます。いずれの場合も、反応の後には、クロマトグラフィーまたは市販のDNA精製キットを使用して精製する必要があります。
- バッチIVT反応
- サーモブロックを37°C、300rpmに設定します。 表1 に記載されている酵素を除くすべての試薬を解凍し、サーモブロック内で37°Cに予熱します。ATP、CTP、UTP、GTPの100 mM原液を使用して、25 mM NTP混合物を調製します。
- 試薬が解凍されている間に、タイムコース実験用のサンプルアリコートを準備します。0.5 mLの滅菌チューブを取り、各チューブに2 μLの100 mM EDTAをピペットで移します。各チューブに指定されたIVT番号(IVT001、IVT002など)と時点(0分、15分、30分、60分など)をマークします。
- 試薬を解凍し、37°Cで約15分間保存したら、-20°Cの冷凍庫から酵素(T7 RNAポリメラーゼ、ピロホスファターゼ、RNase阻害剤)を取り出し、冷凍庫に戻すまで冷蔵保存してください。
- 個々のIVT試薬(酵素を除く)を混合するには、ボルテックスを使用します。酵素を静かにピペッティングして混合します。
- 実験デザインで設定された比率でIVT試薬を混合します(例:ジェネリックIVTプロトコルを 表1に示します)1.5mLチューブに入れます。IVT最適化を実行する場合、最終的なIVT容量は50〜100μLの範囲になります。IVTの総容量を設計する際には、経時的な実験で反応から取り出す2 μLのサンプル数を考慮してください。サンプルの総量がIVTの総量の50%を超えないようにしてください(例:15分ごとに2μLを3時間サンプリングする場合、採取されたアリコートの総量は30μLであり、IVTの総量は最小60μLである必要があります)。
- IVTを慎重に混合し、最後の試薬としてT7 RNAポリメラーゼを添加します。すべての試薬を添加したら、ボルテックスを使用せずに、ピペッティングで上下させてIVTを完全に混合します。
- すべてのIVT試薬を添加して均質化した直後に、2 μLのIVT混合物をチューブから取り出し、ステップ2.1.2(時点0分)で説明した2 μLの100 mM EDTAを含む事前に調製した0.5 mLチューブにピペットで移します。
- IVT反応混合物を入れた反応チューブをサーモブロックに入れ、37°Cでインキュベートします。
- タイマーを希望の時間間隔(15分/ 30分など)に設定します。各時点で、調製した各IVT反応混合物から2 μLのIVTサンプルを取り出し、2 μLの100 mM EDTAを含む事前に調製したチューブ(ステップ2.1.2を参照)にピペットで移します。これらの分注は、クロマトグラフィー分析に使用されます(ステップ3を参照)。
- クロマトグラフィー分析によりNTPが完全に枯渇したことが確認された後、EDTAによるバルクIVT反応を最終濃度50 mMまでクエンチします(例:30 μLのIVT不活化と30 μLの100 mM EDTAによる不活化)。
- フェドバッチIVT反応
注:mRNAの産生は、NTPが枯渇した後にNTPとMg2+ イオンを添加することでさらに増やすことができます。- バッチIVT反応を混合し、ステップ2.1の説明に従ってサンプリングします。供給バッチ用のNTP + MgCl2 飼料を準備します。例えば、500 μLのNTP+MgCl2 混合物を調製するには、各200 mM NTPを106 μL、1 M MgCl2 を76.25 μL、および5.5 μLのddH2Oを混合します。混合物中の最終濃度は、NTPごとに42.4mM、およびMgCl2でそれぞれ152.5mMです。
- ステップ 3 で説明したアットライン分析を使用して NTP の消費を監視します。アットライン分析により、NTP濃度が開始濃度の10%未満に低下したことが確認された場合は、NTP+Mg2+ を含むフィードをバルクIVTに追加します。
- 分析のためにすべてのアリコートを採取した後、1.5 mLチューブに残っているバルクIVT反応の正確な量を測定/計算します。適切な量のNTP+MgCl2混合物をIVTに添加し、最終濃度が各NTPの4 mMとMgCl2の12 mMになるようにします。例えば、1.5 mLのチューブに80 μLのバルクIVTが残っている場合は、16.8 μLのNTP+MgCl2 混合物を加えます。
- 手順 2.1 で説明したように、サンプリングを続行します。NTP が再び枯渇したら、セクション 2.2.2 を繰り返します。目的のmRNA産生が達成されるまで、フィーディングとサンプリングを続けます。
- 所望のmRNA産生が達成されたら、ステップ2.1.10のように反応全体を不活性化します。
3. クロマトグラフィー分析の準備
- 標準およびシステム適合性試験(SST)の準備
- キャッピング試薬、NTP、pDNAテンプレート、およびmRNAを組み合わせてSSTを調製します。最終濃度がキャッピング試薬で約3 μM、各NTPで約5 μM、pDNAテンプレートで4 ng/μL、mRNAで10 ng/μLであることを確認してください。最終SSTに0.1 M NaClが含まれていることを確認します。
- 各分析の前に、新しい検量線を準備します。既知の濃度の精製mRNAサンプルをMPAで希釈することにより、キャリブレーションスタンダードを作成します。キャリブレーションポイントは、0.5 ng/μL、2 ng/μL、5 ng/μL、10 ng/μL、15 ng/μL、および20 ng/μLで、それぞれ最終濃度は0.1 M NaClです。
- 必要に応じて、すべてのNTPの検量線を準備します。校正標準試料中の各NTPの最終濃度が0.5 μM、2 μM、5 μM、10 μM、15 μM、20 μMであることを確認してください。
- カラムのコンディショニングと分析のための調製
- 0.1 mLの分析カラムを室温で12時間平衡化してから分析してください。カラムハウジングに示されている方向にカラムをクロマトグラフィーシステムに接続します。
- 1 mL/minの流速でカラムをフラッシュします:最初に50カラム容量(CV)のddH2Oで、その後50 CVのMPAで。
- 分析前に少なくとも 3 つのブランクサンプル(MPA のみを注入)を実行してベースラインを設定します。Skok et al.15に記載されているクロマトグラフィー法を使用してください。
- ベースラインが安定し、2 つの連続するブランク測定値が同等(再現性のあるベースライン)になったら、SST サンプルと検量線サンプルの実行に進みます。
- SST基準を満たし、キャッピング試薬とNTPの分離に問題がなければ、IVTサンプルの分析を進めます。すべてのIVTサンプルを分析した後、SST標準器を再度注入して、分析全体を通じてシステム条件が安定していることを確認します。
- サンプル調製
注:クロマトグラフィー分析用のサンプルは、分析の直前に調製されます。それ以外の場合、サンプルは-20°C(最大24時間)または<-65°C(24時間以上)の冷凍庫に保管されます。- クロマトグラフィー分析のために希釈する前に、急冷したIVTサンプルをボルテックスしてスピンダウンします。Skok et al.15 で説明されている方法を使用して、すべての EDTA 不活化 IVT サンプル(ステップ 2.1.9 を参照)をクロマトグラフィー分析で分析します。
- 分析の前に、IVTサンプルを希釈して、検量線内の濃度を達成します。時間経過実験全体で同じ希釈係数を使用して、NTP エリアを比較して NTP が枯渇するタイミングを判断できるようにします。
- IVTサンプルに必要な最小希釈率を決定するには、次の式を使用します:必要な希釈率 = (予想される終了mRNA濃度) / (不活化中の希釈) / (最高キャリブレーション標準)
例えば、IVTで8 mg/mLのmRNA産生が予想される場合、クエンチされたサンプルの濃度は4 mg/mLになります。検量線内で濃度を上げるには、サンプルをさらに200倍に希釈します。
必要希釈 = (8000 ng/μL]) / 2 / (20 ng/μL) = 200 - MPAと4 M NaClを円錐形のガラスバイアルにピペットで移します。サンプル中のNaClの最終濃度が100 mMであることを確認してください。最後に、焼入れしたIVTサンプルを追加します。
- Vortexはサンプルを調製し、分析のためにオートサンプラーに挿入しました。オートサンプラーの温度を4°Cに設定します。
- 希釈したサンプル100 μLを分析カラムに注入します。各IVTサンプルの吸光度A260およびA280を測定します。
- 濃度がわかっているサンプルを分析する場合、または面積差に基づいてNTP消費量を追跡する必要がない場合は、サンプルを希釈して目標濃度10 ng/μLを達成します。
4. サンプルの定量とデータ解析
- 各IVTサンプルを波形解析する前に、サンプルクロマトグラムからブランクサンプルを差し引きます。
- A260 ですべての NTP、pDNA、mRNA ピークを波形解析し、そのピーク面積を使用して IVT 中の NTP と mRNA の濃度を計算します。
注:あるいは、希釈係数がすべての時点で一貫していれば、ピーク面積を使用してIVT反応に残っている各NTPの割合を比較することもできます。 - アットラインクロマトグラフィー分析によって決定されたmRNA/NTP濃度に基づいて、IVTを不活性化できるかどうか、または追加のNTPをIVT反応に供給する必要があるかどうかを決定します(ステップ2を参照)。
結果
このプロトコールに記載されているクロマトグラフィー分析は、IVTの最適化、またはバッチIVT反応のフェドバッチ反応への変換に使用できます(図1)。
IVT反応の動力学に対する異なるバッファー組成の影響をテストするために、 表2に記述されたプロトコルに従って3つの異なるIVT反応を混合しました。pHがHClで調整されたTrisを含む緩衝液と、酢酸でpHが調整されたTris緩衝液を比較しました。さらに、両方のTris緩衝液をHEPES緩衝液と比較し、pHをNaOHで調整しました。すべての1x IVTバッファーの組成は、40 mM Tris/HEPES、10 mM DTT、2 mMスペルミジン、およびpH 7.9でした。
100 μL の IVT 反応混合物から 2 μL の IVT サンプルを IVT 反応液から取り出し、インキュベーションの最初の 1 時間で 15 分ごとに 2 μL の 100 mM EDTA でクエンチし、その後 180 分インキュベーションまで 30 分ごとにクエンチしました。この反応条件で予想される最終mRNA収量は15 mg/mLであり、クエンチングによる2倍希釈に続いてMPA+NaClで400倍希釈して分析したことを意味します。サンプルは合計で 800 倍に希釈されたため、15 mg/mL の調製時でも、分析用クロマトグラフィーカラムにロードされた mRNA 濃度は検量線の濃度内にとどまっていました。
A260 の NTP および mRNA 領域は、個々の時点 (tx) で個々のサンプルごとに統合されました。次に、mRNA 検量線と NTP の消費率を使用して、0 分(t0)の A260 NTP 面積を 100% として、面積を mRNA の濃度に変換しました(下記の式を参照)。
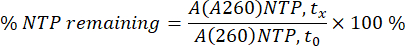
結果は、x軸に時間、y軸にmRNA濃度と残りのNTPを示すグラフとして、個々のIVT反応ごとにグラフで表示することができます(図2A)。すべてのIVT、つまり時間に依存するmRNA濃度も1つのグラフにまとめてプロットでき、mRNA産生速度論を研究し、最適なIVT条件を選択できます(図2B)。
フェドバッチ実験では、 表3に書かれているようにIVTを混合しました。300 μL の IVT 反応ミックスから 2 μL の IVT サンプルを IVT 反応から取り出し、2 μL の 100 mM EDTA で 30 分ごとにクエンチしました。さらに、サンプルは、各バルクNTP + MgCl2 添加の直後に採取しました。各NTPと152.5mMのMgCl2をそれぞれ42.4mMと152.5mMのMgCl2 を含有する供給溶液を予め調製し、各個々の200mMのNTPを106μLずつ1MのMgCl2と76.2μLを混合した。ボーラス飼料を1時間ごと(60分、120分、180分、および240分のインキュベーション)で追加する摂食体制が確立されました。フィーディングレジームは 表3 に説明されています:サンプルは、バルクIVT反応について説明した分析を使用してほぼリアルタイムで分析されました。300分間のモニタリング後、IVT反応は急冷されました。
結果は、インキュベーション期間にわたる残りのNTP/mRNA産生として示すことができます。ボーラス飼料添加はIVT反応も希釈するため、IVT中のmRNA濃度は添加するたびに低下します (図3A)。 mRNA質量の増加は、転写因子(mmRNA/mpDNAと定義)によっても測定および提示でき、時間の経過とともにmRNA産生が直線的に増加することを示しています (図3B)。
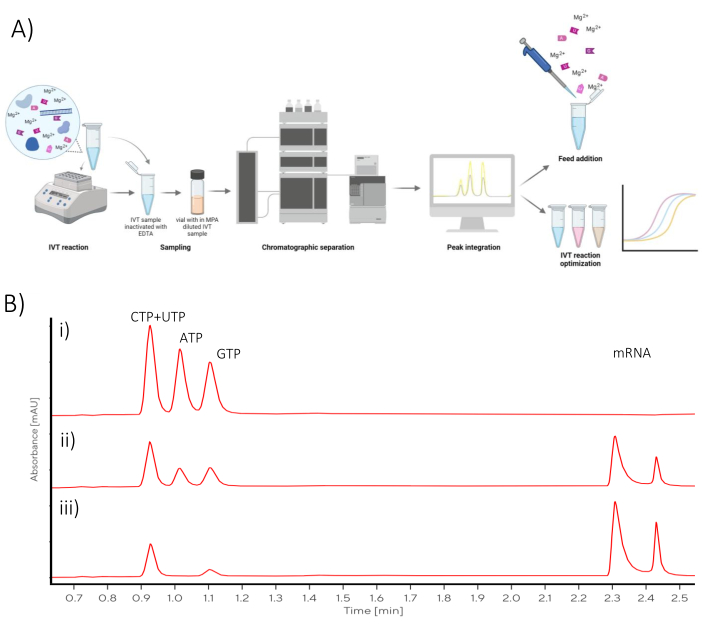
図1:概略図の概要 (A)NTPおよびmRNAのアットラインクロマトグラフィー定量によるIVT最適化ワークフローの表現。(B)(i)t0、(ii)中間点、(iii)最終反応時点にサンプリングされたバッチIVT反応の代表的なクロマトグラム。 この図の拡大版を表示するには、ここをクリックしてください。
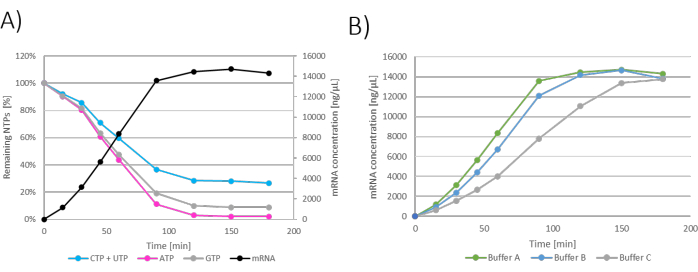
図2:バッチIVTグラフ。 (A)y軸上のmRNA産生とNTP消費によるIVT反応の代表的なグラフ。120分でNTP(ATP)が消費されるのを制限するため、NTP消費と相関するインキュベーションの120〜180分で見えるmRNA濃度のプラトー(B)IVTバッファーがIVT動態に及ぼす影響を示すIVTグラフ。バッファーA(Tris+酢酸)を含むIVTは、最も速いmRNA産生を示し、次いでバッファーB(Tris+HCl)とバッファーC(HEPES+NaOH)が最も遅いと示しました。 この図の拡大版を表示するには、ここをクリックしてください。
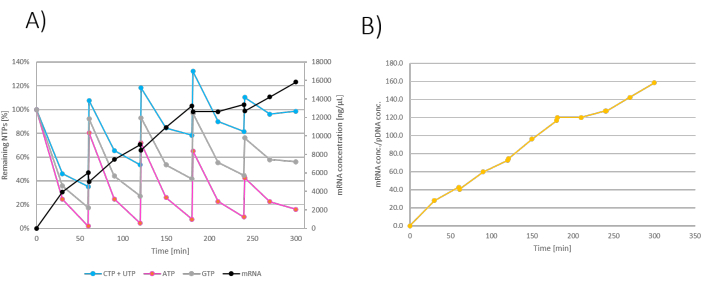
図3:フェデラルバッチIVTグラフ。 (A)NTP+MgCl2 飼料を60分、120分、180分、240分のmRNA濃度で添加したIVTグラフ NTP+MgCl2 飼料で希釈することにより、IVT反応液を添加するたびに低下します。(B)転写因子によって示されるmRNA質量の増加は、反応インキュベーション全体を通じて直線的である。 この図の拡大版を表示するには、ここをクリックしてください。
表1:汎用IVTプロトコル。この表をダウンロードするには、ここをクリックしてください。
表2:バッチIVTプロトコル。この表をダウンロードするには、ここをクリックしてください。
表3:フェドバッチIVTプロトコル。この表をダウンロードするには、ここをクリックしてください。
ディスカッション
この分析法では、NTP、pDNA、mRNAを分離する分析用クロマトグラフィーマルチモーダルカラムを使用して、インキュベーション中の異なる時点でIVTサンプルを分析し、NTP消費量とmRNA産生を綿密にモニタリングすることができます。この手法は、指定された時点におけるA260領域の変化に基づいて、NTPsとmRNAの濃度を定量的に測定するものです。この分析法はNTPおよびmRNA濃度に関する情報を提供するため、主な目標がmRNA収量の最大化と反応時間の最小化であることが多いIVT最適化に非常に適しています。したがって、mRNA産生の動態に対するさまざまなIVT試薬の影響を理解することは非常に重要です22。アットラインモニタリングを適用して、バッチモードとフェッドバッチモードでのIVT反応を最適化する方法を示します。
この方法の主な利点は、各サンプルが8分未満で分析されるため、IVTをほぼリアルタイムで監視できるアットライン分析です。分析用のサンプル調製は、事前のサンプル前処理なしでMPAでの希釈のみが必要なため、簡単です。分析に必要なサンプル量は非常に少なく、例えばIVTは1 μLです。この小さなピペッティング量は、ピペッティングエラーによる分析の逸脱を引き起こす可能性があります。しかし、分析アプローチのハイスループットにより3回の測定が可能になるため、外れ値を簡単に特定でき、予想される運動曲線からの逸脱を検出するのは難しくありません。
この分析法の限界の 1 つは、UTP と CTP のクロマトグラフィー共溶出です。mRNA配列の限定NTPはATPまたはGTPであることが多いため、UTPとCTPの正確な定量は、通常は必要ありません。UTP と CTP の定量を別々に行う必要がある場合は、UTP と CTP の 260 nm と 280 nm での UV 吸光度の差を利用して、クロマトグラフィーピーク中の各 NTP の相対存在量を導き出すことができます。
この分析法では、長さが100ヌクレオチドを超えるRNA種は分離されません。したがって、非ポリアデニル化mRNAとポリアデニル化mRNAを区別せず、dsRNAまたは流産性転写産物とssRNAを区別せず、分解されたmRNAと非分解されたmRNAを区別できないため、安定性研究には適していません。ただし、この方法は、circRNA、tRNA、saRNAなどの他のRNAモダリティの定量に利用できます。これらの分子のサイズと構造は異なりますが、IVT反応における各分子の産生収率を研究するために、同じ分析方法を採用することができます。
開示事項
著者は何も開示していません。
謝辞
著者は、有益な議論をしてくれたTomas Kostelec氏、Blaž Bakalar氏、Nejc Pavlin氏、Andreja Gramc Livk氏、Anže Martinčič Celjar氏に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
参考文献
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403 (2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, &. #. 2. 1. 4. ;. mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090 (2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190 (2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3 (2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745 (2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655 (2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833 (2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246 (2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937 (2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408 (2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045 (2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved