Method Article
Optimisation de la réaction de transcription in vitro pour la production d’ARNm à l’aide de la surveillance chromatographique en ligne
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
L’optimisation de la transcription in vitro (IVT) est essentielle pour une production rentable d’ARNm. Ce protocole détaille une méthode chromatographique analytique pour l’analyse en ligne de la réaction IVT, la surveillance de la déplétion en NTP et la production d’ARNm en mode batch ou fed-batch, applicable à diverses modalités d’ARN, améliorant la productivité et réduisant les coûts.
Résumé
La réaction de transcription in vitro (IVT) est une synthèse enzymatique complexe et multi-composants d’ARNm à partir d’une matrice d’ADN linéaire, catalysée par une ARN polymérase, par exemple T7. En raison du coût élevé des réactifs IVT, l’IVT est une étape critique dans le processus de production de substances médicamenteuses à ARNm et a fait l’objet d’une optimisation intense sur le terrain. Pour réduire le coût de production de l’ARNm, les réactifs doivent être utilisés de manière optimale. Une optimisation efficace nécessite une compréhension complète de l’impact des réactifs individuels sur la cinétique de réaction, c’est-à-dire la consommation de nucléosides triphosphates (NTP) et la production d’ARNm. Traditionnellement, des techniques analytiques à faible débit et à point final ont été utilisées pour l’analyse de l’ARNm. Bien que ces méthodes fournissent des informations précieuses sur le contenu de l’ARNm, une analyse en ligne et en temps quasi réel est nécessaire pour bien comprendre la réaction de l’IVT. Nous démontrons comment une méthode analytique de chromatographie liquide qui sépare les NTP, ainsi que l’ADNp et l’ARNm, peut être utilisée en temps quasi réel pour étudier la cinétique de réaction IVT. Grâce à la connaissance de l’influence des différents composants IVT sur la cinétique et le rendement, l’analyse chromatographique rapide peut être utilisée pour convertir la réaction IVT par lots en un mode fed-batch, ce qui augmente encore la productivité et réduit le coût global de la réaction.
Introduction
La pandémie de COVID-19 a catalysé une révolution sans précédent dans le domaine de la biomédecine, conduisant au développement et à l’autorisation rapides de vaccins à base d’ARNm, tels que le Comirnaty de BioNTech/Pfizer et le Spikevax de Moderna, en Europe et aux États-Unis 1,2,3. L’efficacité impressionnante et le développement rapide de ces vaccins ont mis en évidence l’immense potentiel thérapeutique de la technologie de l’ARNm, non seulement pour les maladies infectieuses, mais aussi pour les immunothérapies anticancéreuses, les thérapies de remplacement des protéines, la médecine régénérative et la reprogrammation cellulaire 4,5. Cette époque, souvent appelée la révolution de l’ARNm, souligne la nécessité de processus de production d’ARNm efficaces et rentables.
La production d’ARNm implique plusieurs opérations unitaires, la réaction de transcription in vitro (IVT) étant une opération unitaire critique et la plus coûteuse 6,7,8. La réaction IVT synthétise l’ARNm à partir d’une matrice d’ADN en utilisant l’ARN polymérase, généralement l’ARN polymérase T7 et les nucléosides triphosphates (NTP) en présence d’ions magnésium. Le processus est relativement simple et permet de produire de grandes quantités d’ARNm dans un court laps de temps, atteignant des rendements de réaction de 2 à 5 g/L en quelques heures et jusqu’à 14 g/L dans certains rapports 9,10,11. Cependant, l’optimisation du rendement de la réaction IVT est cruciale pour réduire les coûts de production et assurer l’évolutivité des vaccins à ARNm et des traitements12 et, en raison des différences de séquence qui influencent la consommation de NTP, peut être nécessaire pour chaque construction ou famille de construction.
La réaction est généralement réalisée sous forme de processus par lots, mais les progrès récents dans le traitement par lots et l’analyse en ligne ont ouvert de nouvelles voies pour optimiser la production d’ARNm 7,13. Les réactions par lots fédéraux, qui impliquent un bolus ou un ajout continu de réactifs, peuvent potentiellement prolonger les temps de réaction et augmenter les rendements en empêchant l’inhibition du substrat et la dégradation du produit dépendante des cofacteurs14,15.
Le développement de l’analyse rapide en ligne a constitué une avancée significative dans le domaine de l’IVT, permettant de surveiller les composants clés de la réaction tels que les NTP et l’ARNm avec un décalage analytique minimal13,16, ajoutant une dimension supplémentaire à la surveillance de l’IVT, qui est généralement axée uniquement sur la concentration d’ARNm17. Traditionnellement, les ARNm ont été analysés à l’aide de techniques telles que l’électrophorèse sur gel de polyacrylamide (PAGE), l’électrophorèse sur gel d’agarose ou l’électrophorèse capillaire. Il s’agit de méthodes finales et, par conséquent, ne peuvent pas être utilisées pour la surveillance IVT en temps réel. Une alternative à l’analyse décrite dans ce protocole sont les méthodes utilisant des paires d’aptamères d’ARN lumineux et de colorants de fluorescence. Dans cette méthode, l’aptamère d’ARN est marqué à l’ARN et incubé avec un colorant de fluorescence lumineux. L’activité de transcription peut alors être visualisée par l’intensité de fluorescence17. Cela permet d’analyser en temps réel la quantité et la qualité de l’ARNm transcrit au cours de l’IVT 21, mais ne permet pas la surveillance simultanée d’autres composants de l’IVT, par exemple les NTP. Une autre alternative qui permet potentiellement la quantification en temps réel du NTP et de l’ARNm est la spectroscopieRaman 18, mais à ce jour, aucun rapport n’a montré son utilité pour la surveillance IVT, ce qui suggère qu’une optimisation supplémentaire de la méthode est encore nécessaire pour obtenir la sélectivité et la sensibilité nécessaires.
Le développement continu d’analyses plus rapides et plus sélectives prend en charge l’optimisation de l’IVT pour un rendement plus élevé en modes batch et fed-batch. La réaction reste un processus complexe et multiparamétrique avec de multiples facteurs d’interaction, pour lesquels les optima peuvent différer entre les types de construction (par exemple, ARNm vs ARNsa). La concentration et le rapport des ions Mg2+ et des NTP sont particulièrement influents, et leurs niveaux optimaux doivent être soigneusement équilibrés pour maximiser le rendement en ARNm tout en minimisant la formation d’ARN double brin (ARNdb), un puissant stimulant du système immunitaire inné19,20. Récemment, l’alimentation des UTP à l’état d’équilibre a été signalée comme une approche pour réduire la formation d’ARNdb21. La surveillance des niveaux UTP en temps quasi réel ajouterait un niveau supplémentaire de contrôle du processus.
Dans ce protocole, nous démontrons comment la surveillance en ligne de la réaction IVT peut augmenter le rendement de la production d’ARNm en mode batch et fed-batch.
Protocole
1. Préparation du tampon
REMARQUE : Tous les tampons doivent être préparés sans RNase, ce qui signifie que tous les produits chimiques et la verrerie doivent être utilisés uniquement pour des travaux sans RNase et manipulés avec précaution. L’eau utilisée pour la préparation du tampon et le nettoyage de la verrerie doit être certifiée exempte de nucléases. Avant de réaliser une expérience, toutes les surfaces de travail et la verrerie doivent être vaporisées avec un réactif de décontamination qui élimine les RNases. Le réactif de décontamination doit être soigneusement lavé avec de l’eau exempte de RNase avant d’utiliser la verrerie/la zone de travail. Si possible, utilisez des consommables stériles et à usage unique.
- Préparation des tampons IVT : préparer 10x tampon IVT en ajoutant 400 mM de Tris, 10 mM de DTT, 20 mM de spermidine, pH 7,9. Préparez un tampon de trempe contenant 100 mM d’EDTA, pH 8. Filtrez chaque tampon à travers un filtre PES de 0,22 μm.
- Préparation des phases mobiles : préparer la phase mobile A (MPA) par l’ajout de 50 mM d’HEPES, pH 7,0, la phase mobile B (MPB) par l’ajout de 50 mM d’HEPES, 100 mM de Na4P2O7, pH 8,3, la phase mobile C (MPC) par l’ajout de 0,1 M de NaOH, 1 M de NaCl, la phase mobile D (MPD) par l’ajout de 0,5 M d’HEPES, pH 7,0. Filtrez chaque tampon à travers un filtre PES de 0,22 μm.
2. Préparation de la réaction de transcription in vitro (IVT)
NOTE 1 : La réaction IVT peut être une réaction par lots, où tous les réactifs sont ajoutés au début, et la réaction est arrêtée après que les NTP sont épuisés/la production d’ARNm atteint un plateau, ou peut être une réaction par lots fédéraux, où les NTP appauvris sont réapprovisionnés avec des NTP supplémentaires et avec cela la production d’ARNm augmente encore plus. La matrice linéaire d’ADNp utilisée dans la réaction IVT peut être obtenue en utilisant une enzyme de restriction qui se clive juste après la séquence poly(A) dans l’ADN plasmidique ou par réaction PCR. Dans les deux cas, la réaction doit être suivie d’une purification, soit chromatographique, soit à l’aide de kits de purification d’ADN commerciaux.
- Réaction IVT par lots
- Réglez le thermobloc à 37 °C et 300 tr/min. Décongeler tous les réactifs énumérés au tableau 1 , à l’exception des enzymes, et les préchauffer à 37 °C dans le thermobloc. Préparez un mélange NTP de 25 mM en utilisant une solution mère de 100 mM d’ATP, de CTP, d’UTP et de GTP.
- Pendant que les réactifs décongèlent, préparez des aliquotes d’échantillons pour l’expérience sur le cours du temps. Prélever des tubes stériles de 0,5 mL et pipeter 2 μL d’EDTA de 100 mM dans chaque tube. Marquez chaque tube avec un numéro IVT désigné (par exemple, IVT001, IVT002, etc.) et un point temporel (par exemple, 0 min, 15 min, 30 min, 60 min, etc.).
- Lorsque les réactifs sont décongelés et conservés à 37 °C pendant environ 15 minutes, retirez les enzymes (ARN polymérase T7, pyrophosphatase, inhibiteur de la RNASE) du congélateur à -20 °C et conservez-les dans une glacière jusqu’à ce qu’ils soient rangés dans le congélateur.
- Pour mélanger des réactifs IVT individuels (à l’exception des enzymes), utilisez un vortex. Mélangez les enzymes en pipetant doucement de haut en bas.
- Mélanger les réactifs IVT dans les proportions définies dans le plan d’expérience (p. ex., le protocole IVT générique est présenté dans le tableau 1) dans un tube de 1,5 ml. Lors de l’optimisation de l’IVT, le volume final de l’IVT peut varier entre 50 et 100 μL. Lors de la conception du volume total de l’IVT, tenez compte du nombre d’échantillons de 2 μL à prélever de la réaction pendant l’expérience sur le cours du temps. S’assurer que le volume total des échantillons ne dépasse pas 50 % du volume total de l’IVT (p. ex., si l’échantillonnage est de 2 μL toutes les 15 minutes pendant 3 h, le volume total des aliquotes prélevées est de 30 μL, ce qui signifie que le volume total de l’IVT doit être d’au moins 60 μL).
- Mélangez soigneusement les IVTs, ajoutez l’ARN polymérase T7 comme dernier réactif. Lorsque tous les réactifs sont ajoutés, mélangez soigneusement IVT en pipetant de haut en bas, n’utilisez pas de vortex.
- Immédiatement après l’ajout et l’homogénéisation de tous les réactifs IVT, prélever 2 μL de mélange IVT du tube et le pipeter dans des tubes de 0,5 mL préalablement préparés contenant 2 μL d’EDTA 100 mM décrits à l’étape 2.1.2 (point de temps 0 min).
- Placez les tubes réactionnels contenant des mélanges réactionnels IVT dans un thermobloc et incuberez-les à 37 °C.
- Réglez la minuterie sur les intervalles de temps souhaités (par exemple, 15 min/30 min). À chaque point temporel, prélever 2 μL d’un échantillon IVT de chaque mélange réactionnel IVT préparé et le pipeter dans un tube préalablement préparé (voir étape 2.1.2) contenant 2 μL de 100 mM d’EDTA. Ces aliquotes seront utilisées pour l’analyse chromatographique (voir étape 3).
- Après que l’analyse chromatographique a confirmé l’épuisement complet des NTP, tremper les réactions IVT en vrac avec l’EDTA jusqu’à une concentration finale de 50 mM (par exemple, 30 μL d’IVT inactivés avec 30 μL d’EDTA de 100 mM).
- Réaction IVT du Fed-batch
REMARQUE : la production d’ARNm peut être augmentée en ajoutant des ions NTP et Mg2+ après l’épuisement des NTP.- Mélangez la réaction IVT par lots et échantillonnez-la comme décrit à l’étape 2.1. Préparez l’alimentation NTP+MgCl2 pour le lot nourri. Pour préparer, par exemple 500 μL de mélange NTP+MgCl2 , mélanger 106 μL de chaque 200 mM NTP, 76,25 μL de 1 M MgCl2 et 5,5 μL de ddH2O. Les concentrations finales dans le mélange sont de 42,4 mM de NTP et de 152,5 mM de MgCl2.
- Surveillez la consommation NTP à l’aide de l’analyse en ligne décrite à l’étape 3. Lorsque l’analyse en ligne confirme que la concentration de NTP est inférieure à 10 % de la concentration initiale, ajoutez l’aliment contenant NTP+Mg2+ à l’IVT en vrac.
- Mesurez/calculez le volume précis de réaction IVT en vrac restant dans un tube de 1,5 mL après que toutes les aliquotes ont été prélevées pour l’analyse. Ajouter un volume approprié de mélange NTP+MgCl2 à un IVT de sorte que la concentration finale soit de 4 mM de chaque NTP et de 12 mM de MgCl2. Par exemple, pour 80 μL d’IVT en vrac laissés dans un tube de 1,5 mL, ajoutez 16,8 μL de mélange NTP+MgCl2 .
- Poursuivez avec l’échantillonnage comme indiqué à l’étape 2.1. Une fois que les NTP sont à nouveau épuisés, répétez la section 2.2.2. Poursuivre l’alimentation et l’échantillonnage jusqu’à ce que la production d’ARNm souhaitée soit atteinte.
- Une fois que la production d’ARNm souhaitée est atteinte, inactivez l’ensemble de la réaction comme à l’étape 2.1.10.
3. Préparation de l’analyse chromatographique
- Préparation des étalons et des essais d’adéquation du système (SST)
- Préparez le SST en combinant le réactif de coiffage, les NTP, la matrice d’ADNp et l’ARNm. Assurez-vous que les concentrations finales sont d’environ 3 μM pour le réactif de coiffage, 5 μM pour chaque NTP, 4 ng/μL pour la matrice d’ADNp et 10 ng/μL pour l’ARNm. Assurez-vous que le SST final contient 0,1 M de NaCl.
- Préparez une nouvelle courbe d’étalonnage avant chaque analyse. Créez l’étalon d’étalonnage en diluant un échantillon d’ARNm purifié de concentration connue avec de l’APM. Les points d’étalonnage doivent être de 0,5 ng/μL, 2 ng/μL, 5 ng/μL, 10 ng/μL, 15 ng/μL et 20 ng/μL, chacun avec une concentration finale de 0,1 M de NaCl.
- Au besoin, préparez une courbe d’étalonnage pour tous les NTP et assurez-vous que les concentrations finales de chaque NTP dans les étalons sont de 0,5 μM, 2 μM, 5 μM, 10 μM, 15 μM et 20 μM.
- Conditionnement de la colonne et préparation à l’analyse
- Équilibrer la colonne d’analyse de 0,1 mL à température ambiante pendant 12 h avant l’analyse. Fixez la colonne au système chromatographique dans la direction indiquée sur le boîtier de la colonne.
- Rincer la colonne avec un débit de 1 mL/min : d’abord avec 50 volumes de colonne (CV) de jjH2O et ensuite avec 50 CV de MPA.
- Exécutez au moins 3 échantillons à blanc (en injectant uniquement du MPA) avant l’analyse pour définir la ligne de base. Utiliser la méthode chromatographique décrite dans Skok et al.15.
- Une fois que la ligne de base est stable et que deux mesures consécutives à blanc sont comparables (ligne de base reproductible), procédez à l’analyse de l’échantillon SST et des échantillons de la courbe d’étalonnage.
- Si les critères SST sont remplis et que la séparation du réactif de bouchage et des NTP est satisfaisante, procéder à l’analyse des échantillons IVT. Après avoir analysé tous les échantillons IVT, injectez à nouveau un étalon SST pour confirmer que les conditions du système sont restées stables tout au long de l’analyse.
- Préparation des échantillons
REMARQUE : Les échantillons pour l’analyse chromatographique sont préparés immédiatement avant l’analyse. Sinon, les échantillons sont conservés dans un congélateur à -20 °C (maximum 24 h) ou <-65 °C (plus de 24 h).- Avant la dilution pour l’analyse chromatographique, tourbillonnez et essorez l’échantillon IVT trempé. Analyser tous les échantillons IVT inactivés par l’EDTA (voir l’étape 2.1.9) par analyse chromatographique à l’aide de la méthode décrite dans Skok et al.15.
- Avant l’analyse, diluez les échantillons IVT pour obtenir une concentration dans la courbe d’étalonnage. Utilisez le même facteur de dilution tout au long de l’expérience pour vous assurer que les zones NTP peuvent être comparées pour déterminer quand un NTP est épuisé.
- Pour déterminer la dilution minimale nécessaire pour les échantillons IVT, utilisez la formule suivante : Dilution nécessaire = (concentration finale attendue d’ARNm) / (dilution pendant l’inactivation) / (étalon d’étalonnage le plus élevé)
Par exemple, si une production d’ARNm de 8 mg/mL est attendue dans l’IVT, la concentration dans l’échantillon trempé sera de 4 mg/mL. Pour atteindre la concentration dans la courbe d’étalonnage, diluez davantage l’échantillon 200 fois.
Dilution nécessaire = (8000 ng/μL]) / 2 / (20 ng/μL) = 200 - Pipeter le MPA et le 4 M NaCl dans un flacon conique en verre. S’assurer que la concentration finale de NaCl dans l’échantillon est de 100 mM. Enfin, ajoutez l’échantillon IVT trempé.
- Vortex a préparé l’échantillon et l’a inséré dans l’échantillonneur automatique pour l’analyse. Réglez la température de l’échantillonneur automatique sur 4 °C.
- Injecter 100 μL d’échantillons dilués dans la colonne d’analyse. Mesurez l’absorbance A260 et A280 pour chaque échantillon IVT.
- Pour analyser des échantillons dont les concentrations sont connues, ou s’il n’est pas nécessaire de suivre la consommation de NTP en fonction des différences de surface, diluer les échantillons pour atteindre une concentration cible de 10 ng/μL.
4. Quantification de l’échantillon et analyse des données
- Avant l’intégration de chaque échantillon IVT, soustrayez l’échantillon blanc du chromatogramme de l’échantillon.
- Intégrez tous les pics de NTP, d’ADNp et d’ARNm à A260 et utilisez les zones de pics pour calculer les concentrations de NTP et d’ARNm dans l’IVT.
REMARQUE : Alternativement, les aires des pics peuvent être utilisées pour comparer le pourcentage de chaque NTP restant dans la réaction IVT, à condition que le facteur de dilution soit cohérent à tous les points temporels. - Sur la base des concentrations d’ARNm/NTP, déterminées par analyse chromatographique en ligne, décidez si l’IVT peut être inactivé ou si des NTP supplémentaires doivent être introduits dans la réaction IVT (voir étape 2).
Résultats
L’analyse chromatographique décrite dans ce protocole peut être utilisée soit pour l’optimisation de l’IVT, soit pour convertir une réaction IVT par lots en une réaction fed-batch (Figure 1).
Pour tester l’effet de différentes compositions de tampons sur la cinétique de la réaction IVT, trois réactions IVT différentes ont été mélangées selon le protocole écrit dans le tableau 2. Le tampon contenant du Tris, dont le pH a été ajusté avec du HCl, a été comparé au tampon Tris, où le pH a été ajusté avec de l’acide acétique. De plus, les deux tampons Tris ont été comparés au tampon HEPES, où le pH a été ajusté avec du NaOH. La composition de tous les tampons 1x IVT était de 40 mM de Tris/HEPES, 10 mM de DTT, 2 mM de spermidine et un pH de 7,9.
À partir de 100 μL de mélange de réactions IVT, 2 μL d’échantillon IVT ont été retirés de la réaction IVT et trempés avec 2 μL de 100 mM d’EDTA toutes les 15 minutes au cours de la première heure d’incubation, puis toutes les 30 minutes jusqu’à 180 minutes d’incubation. Le rendement final attendu de l’ARNm pour cette condition de réaction était de 15 mg/mL, ce qui signifie qu’une dilution de 2 fois due à l’extinction a été suivie d’une dilution de 400 fois dans le MPA+NaCl pour l’analyse. L’échantillon a été dilué 800 fois au total, ce qui signifie que même à une production de 15 mg/mL, la concentration d’ARNm chargée sur la colonne chromatographique analytique était toujours dans les concentrations de la courbe d’étalonnage.
Les zones NTP et ARNm à A260 ont été intégrées pour chaque échantillon individuel à chaque point temporel individuel (tx). Les aires ont ensuite été converties en concentrations pour l’ARNm, en utilisant la courbe d’étalonnage de l’ARNm et le pourcentage de consommation pour les NTP, en utilisant l’aire A260 des NTP à 0 min (t0) comme 100 % (voir l’équation ci-dessous).
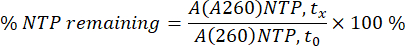
Les résultats peuvent être présentés sous forme de graphiques pour chaque réaction IVT individuelle, où le temps est indiqué sur l’axe des x et la concentration d’ARNm et les NTP restants sont indiqués sur l’axe des y (Figure 2A). Tous les IVT, c’est-à-dire les concentrations d’ARNm en fonction du temps, peuvent également être regroupés dans un graphique, et la cinétique de production d’ARNm peut être étudiée et les conditions optimales d’IVT sélectionnées (Figure 2B).
Pour l’expérience fed-batch, l’IVT a été mélangé comme indiqué dans le tableau 3. À partir de 300 μL de mélange réactionnel IVT, 2 μL d’échantillon IVT ont été retirés de la réaction IVT et trempés avec 2 μL de 100 mM d’EDTA toutes les 30 min. De plus, l’échantillon a été prélevé immédiatement après chaque ajout en vrac de NTP+MgCl2 . Une solution d’alimentation contenant 42,4 mM de chaque NTP et 152,5 mM de MgCl2 a été préparée à l’avance, et 106 μL de chaque 200 mM de NTP ont été mélangés avec 76,2 μL de 1 M de MgCl2. Un régime d’alimentation a été établi, où des bolus d’alimentation ont été ajoutés toutes les heures (à 60 min, 120 min, 180 min et 240 min d’incubation). Le régime d’alimentation est décrit dans le tableau 3 Les échantillons ont été analysés en temps quasi réel avec des analyses comme décrit pour la réaction IVT en vrac. Après 300 minutes de surveillance, la réaction IVT a été éteinte.
Les résultats peuvent être présentés comme la production restante de NTP/ARNm au cours de l’incubation. Étant donné que l’ajout d’un bolus dilue également la réaction IVT, la concentration d’ARNm dans l’IVT diminue à chaque ajout d’aliment (Figure 3A). L’augmentation de la masse d’ARNm peut également être mesurée et présentée par des facteurs de transcription (définis comme m: ARNm/md’ADNm), montrant une augmentation linéaire de la production d’ARNm au fil du temps (Figure 3B).
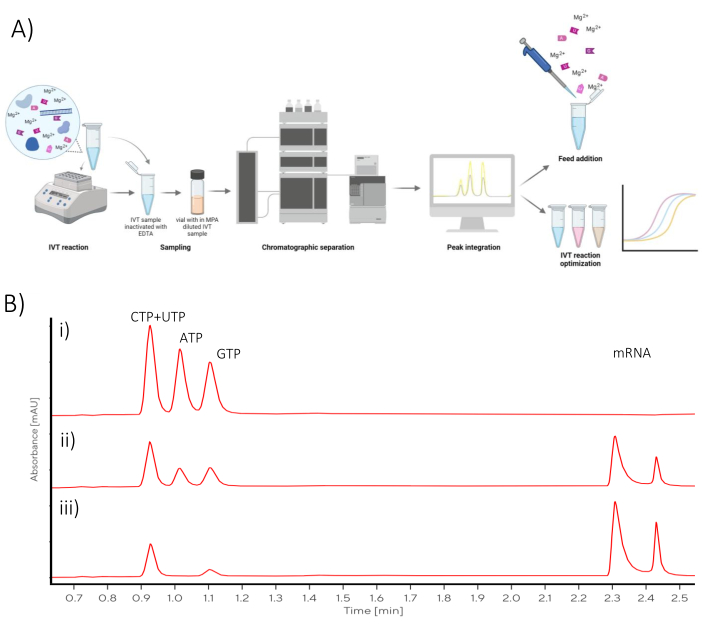
Figure 1 : Vue d’ensemble schématique (A) Représentation du flux de travail d’optimisation IVT avec analyse chromatographique en ligne, quantification des NTP et de l’ARNm. (B) Chromatogrammes représentatifs d’une réaction IVT par lots échantillonnés à (i) t0, (ii) point médian, (iii) points temporels de réaction finaux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
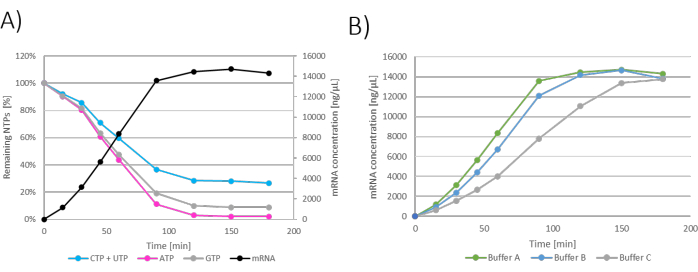
Figure 2 : Graphiques IVT par lots. (A) Graphique représentatif de la réaction IVT avec la production d’ARNm et la consommation de NTP sur l’axe des y. Plateau de concentration d’ARNm visible à 120 - 180 min d’incubation, en corrélation avec la consommation de NTP car le NTP limitant (ATP) est consommé à 120 min. (B) Graphique IVT montrant l’influence du tampon IVT sur la cinétique IVT. L’IVT contenant le tampon A (Tris + acide acétique) a montré la production d’ARNm la plus rapide, suivie du tampon B (Tris + HCl) et du tampon C (HEPES+NaOH) comme les plus lentes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
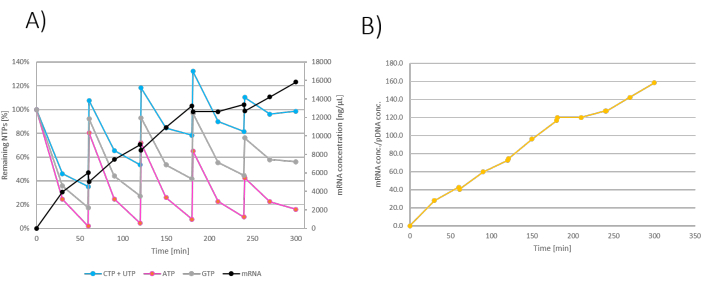
Figure 3 : Graphique IVT Fed-batch. (A) Graphique IVT où les alimentations NTP+MgCl2 ont été ajoutées à 60, 120, 180 et 240 min. La concentration d’ARNm dans la réaction IVT diminue à chaque ajout d’alimentation en raison de la dilution avec l’alimentation NTP+MgCl2. (B) L’augmentation de la masse d’ARNm montrée par le facteur de transcription est linéaire tout au long de l’incubation de la réaction. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Protocole IVT générique. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Protocole IVT par lots. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Protocole IVT Fed-batch. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Dans cette méthode, les échantillons IVT sont analysés à différents moments de l’incubation à l’aide d’une colonne chromatographique analytique multimodale qui sépare les NTP, l’ADNp et l’ARNm, permettant ainsi un suivi étroit de la consommation de NTP et de la production d’ARNm. Cette méthode mesure la concentration de NTP et d’ARNm de manière quantitative en fonction des changements dans les zones A260 à des points temporels désignés. Étant donné que la méthode fournit des informations sur la concentration de NTP et d’ARNm, elle convient parfaitement à l’optimisation IVT, où l’objectif principal est souvent de maximiser le rendement de l’ARNm et de minimiser le temps de réaction ; par conséquent, il est essentiel de comprendre l’effet des différents réactifs IVT sur la cinétique de production d’ARNm22. Nous montrons comment la surveillance en ligne peut être appliquée pour optimiser la réaction IVT en modes batch et fed-batch.
Le principal avantage de cette méthode est une analyse en ligne qui permet une surveillance en temps quasi réel de l’IVT, car chaque échantillon est analysé en moins de 8 minutes. La préparation des échantillons pour l’analyse est simple, car seule la dilution dans le MPA est nécessaire sans aucun prétraitement préalable de l’échantillon. Le volume d’échantillon requis pour l’analyse est très faible, par exemple, 1 μL d’IVT. Ce faible volume de pipetage pourrait entraîner des écarts analytiques dus à des erreurs de pipetage. Cependant, comme le débit élevé de l’approche analytique permet des mesures en trois exemplaires, les valeurs aberrantes sont facilement identifiables et les écarts par rapport à la courbe cinétique attendue ne sont pas difficiles à détecter.
L’une des limites de la méthode est la coélution chromatographique de l’UTP et de la CTP. Une quantification précise de l’UTP et du CTP n’est généralement pas nécessaire car le NTP limitant dans une séquence d’ARNm est souvent soit ATP, soit GTP. Si une quantification distincte de l’UTP et de la CTP est nécessaire, la différence d’absorbance UV à 260 nm et 280 nm pour l’UTP et la CTP peut être exploitée pour calculer l’abondance relative de chaque NTP dans le pic chromatographique.
Cette méthode analytique ne sépare pas les espèces d’ARN de plus de 100 nucléotides de longueur ; par conséquent, il ne peut pas être utilisé pour détecter les différences de qualité de l’ARNm, par exemple, il ne fait pas la différence entre l’ARNm non polyadénylé et polyadénylé, entre l’ARNdb ou les transcrits abortifs et l’ARNsb et ne convient pas aux études de stabilité car il ne peut pas différencier l’ARNm dégradé et non dégradé. Cependant, la méthode peut être utilisée pour la quantification d’autres modalités d’ARN, telles que le circARN, l’ARNt et l’ARNsa. Bien que ces molécules varient en taille et en structure, la même méthode analytique peut être utilisée pour étudier les rendements de production de chacune dans une réaction IVT.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs tiennent à remercier Tomas Kostelec, Blaž Bakalar, Nejc Pavlin, Andreja Gramc Livk et Anže Martinčič Celjar pour leurs discussions utiles.
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
Références
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403(2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, Ö mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090(2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190(2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3(2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745(2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655(2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833(2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246(2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937(2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408(2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045(2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon