Method Article
Optimierung der in vitro Transkriptionsreaktion für die mRNA-Produktion mittels chromatographischem At-Line-Monitoring
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Optimierung der In-vitro-Transkription (IVT) ist entscheidend für eine kostengünstige mRNA-Produktion. Dieses Protokoll beschreibt eine analytische chromatographische Methode zur At-Line-Analyse der IVT-Reaktion, zur Überwachung der NTP-Depletion und der mRNA-Produktion im Batch- oder Fed-Batch-Modus, die auf verschiedene RNA-Modalitäten anwendbar ist, um die Produktivität zu steigern und die Kosten zu senken.
Zusammenfassung
Die In-vitro-Transkriptionsreaktion (IVT) ist eine komplexe, mehrkomponentige enzymatische Synthese von mRNA aus einem linearen DNA-Template, katalysiert durch eine RNA-Polymerase, z.B. T7. Aufgrund der hohen Kosten für IVT-Reagenzien ist IVT ein kritischer Schritt im Produktionsprozess von mRNA-Wirkstoffen und stand im Mittelpunkt einer intensiven Optimierung in diesem Bereich. Um die Kosten für die mRNA-Produktion zu senken, müssen Reagenzien optimal genutzt werden. Eine effektive Optimierung erfordert ein umfassendes Verständnis des Einflusses einzelner Reagenzien auf die Reaktionskinetik, d.h. den Verbrauch von Nukleosidtriphosphaten (NTPs) und die Produktion von mRNA. Traditionell wurden Endpunkt-Analysetechniken mit niedrigem Durchsatz für die Analyse von mRNA verwendet. Obwohl solche Methoden wertvolle Informationen über den mRNA-Gehalt liefern, ist eine At-Line-Analyse nahezu in Echtzeit erforderlich, um die IVT-Reaktion vollständig zu verstehen. Wir zeigen, wie eine flüssigkeitschromatographische Analysemethode, die sowohl NTPs als auch pDNA und mRNA trennt, nahezu in Echtzeit zur Untersuchung der IVT-Reaktionskinetik eingesetzt werden kann. Mit dem Wissen um den Einfluss verschiedener IVT-Komponenten auf Kinetik und Ausbeute kann die schnelle chromatographische Analyse verwendet werden, um die Batch-IVT-Reaktion in einen Fed-Batch-Modus umzuwandeln, der die Produktivität weiter steigert und die Gesamtkosten der Reaktion senkt.
Einleitung
Die COVID-19-Pandemie hat eine beispiellose Revolution in der Biomedizin ausgelöst, die sowohl in Europa als auch in den USA zur raschen Entwicklung und Zulassung von mRNA-basierten Impfstoffen wie Comirnaty von BioNTech/Pfizer und Spikevax von Moderna geführt hat 1,2,3. Die beeindruckende Wirksamkeit und die schnelle Entwicklung dieser Impfstoffe haben das immense therapeutische Potenzial der mRNA-Technologie nicht nur für Infektionskrankheiten, sondern auch für Krebsimmuntherapien, Proteinersatztherapien, regenerative Medizin und zelluläre Reprogrammierung hervorgehoben 4,5. Diese Ära, die oft als mRNA-Revolution bezeichnet wird, unterstreicht die Notwendigkeit effizienter und kostengünstiger mRNA-Produktionsprozesse.
Die Herstellung von mRNA umfasst mehrere Einheitenoperationen, wobei die In-vitro-Transkriptionsreaktion (IVT) eine kritische und äußerst kostenintensive Einheitsoperation darstellt 6,7,8. Bei der IVT-Reaktion wird mRNA aus einem DNA-Template unter Verwendung von RNA-Polymerase, typischerweise T7-RNA-Polymerase, und Nukleosidtriphosphaten (NTPs) in Gegenwart von Magnesiumionen synthetisiert. Das Verfahren ist relativ einfach und ermöglicht die Herstellung großer Mengen an mRNA in einem kurzen Zeitrahmen, wodurch Reaktionsausbeuten von 2-5 g/l innerhalb weniger Stunden und bis zu 14 g/l in einigen Berichten erreichtwerden 9,10,11. Die Optimierung der IVT-Reaktionsausbeute ist jedoch entscheidend für die Senkung der Produktionskosten und die Sicherstellung der Skalierbarkeit von mRNA-Impfstoffen und -Therapeutika12 und kann aufgrund von Sequenzunterschieden, die den NTP-Verbrauch beeinflussen, für jedes Konstrukt oder jede Konstruktfamilie erforderlich sein.
Die Reaktion wird in der Regel als Batch-Prozess durchgeführt, aber die jüngsten Fortschritte in der Fed-Batch-Verarbeitung und der At-Line-Analytik haben neue Wege zur Optimierung der mRNA-Produktion eröffnet 7,13. Fed-Batch-Reaktionen, die einen Bolus oder eine kontinuierliche Zugabe von Reagenzien beinhalten, können möglicherweise die Reaktionszeiten verlängern und die Ausbeute erhöhen, indem sie eine Substrathemmung und einen co-faktorabhängigen Produktabbau verhindern14,15.
Die Entwicklung der schnellen At-Line-Analytik war ein bedeutender Fortschritt auf dem Gebiet der IVT, da sie die Überwachung wichtiger Reaktionskomponenten wie NTPs und mRNA mit minimaler analytischer Verzögerung ermöglicht13,16 und der IVT-Überwachung, die sich in der Regel nur auf die mRNA-Konzentration konzentriert, eine zusätzliche Dimension hinzufügt17. Traditionell werden mRNAs mit Techniken wie der Polyacrylamid-Gelelektrophorese (PAGE), der Agarose-Gelelektrophorese oder der Kapillarelektrophorese analysiert. Hierbei handelt es sich um Endpunktmethoden, die daher nicht für die Echtzeit-IVT-Überwachung verwendet werden können. Eine Alternative zur in diesem Protokoll beschriebenen Analytik sind Methoden, die Leucht-RNA-, Aptamer- und Fluoreszenzfarbstoffpaare verwenden. Bei dieser Methode wird RNA-Aptamer an RNA getaggt und mit einem Leuchtfluoreszenzfarbstoff inkubiert. Die Transkriptionsaktivität kann dann durch die Fluoreszenzintensität17 sichtbar gemacht werden. Dies ermöglicht eine Echtzeitanalyse der Menge und Qualität der transkribierten mRNA während der IVT 21, ermöglicht jedoch keine gleichzeitige Überwachung anderer IVT-Komponenten, z. B. NTPs. Eine weitere Alternative, die möglicherweise eine Echtzeit-Quantifizierung von NTP und mRNA ermöglicht, ist die Raman-Spektroskopie18, aber bisher hat kein Bericht ihren Nutzen für das IVT-Monitoring gezeigt, was darauf hindeutet, dass eine weitere Optimierung der Methode erforderlich ist, um die erforderliche Selektivität und Sensitivität abzuleiten.
Die kontinuierliche Entwicklung schnellerer und selektiverer Analysen unterstützt die Optimierung der IVT für eine höhere Ausbeute im Batch- und Fed-Batch-Modus. Die Reaktion bleibt ein komplexer Prozess mit mehreren mehreren interagierenden Faktoren, für die sich die Optima zwischen den Konstrukttypen (z. B. mRNA vs. saRNA) unterscheiden können. Die Konzentration und das Verhältnis von Mg2+-Ionen und NTPs sind besonders einflussreich, und ihre optimalen Spiegel müssen sorgfältig ausbalanciert werden, um die mRNA-Ausbeute zu maximieren und gleichzeitig die Bildung von doppelsträngiger RNA (dsRNA), einem starken Stimulans des angeborenen Immunsystems, zu minimieren19,20. Kürzlich wurde berichtet, dass die Fütterung von UTP im Steady-State-Level ein Ansatz zur Reduzierung der dsRNA-Bildung ist21. Die Überwachung der UTP-Werte nahezu in Echtzeit würde eine zusätzliche Ebene der Prozesskontrolle hinzufügen.
In diesem Protokoll zeigen wir, wie die At-Line-Überwachung der IVT-Reaktion die Ausbeute der mRNA-Produktion im Batch- und Fed-Batch-Modus erhöhen kann.
Protokoll
1. Vorbereitung des Puffers
HINWEIS: Alle Puffer müssen RNase-frei aufbereitet sein, d.h. alle Chemikalien und Glaswaren dürfen nur für RNase-freies Arbeiten verwendet und mit Vorsicht gehandhabt werden. Wasser, das zur Puffervorbereitung und Reinigung von Glaswaren verwendet wird, muss als nukleasefrei zertifiziert sein. Vor der Durchführung eines Experiments müssen alle Arbeitsflächen und Glaswaren mit einem Dekontaminationsreagenz besprüht werden, das RNasen eliminiert. Das Dekontaminationsreagenz muss gründlich mit RNase-freiem Wasser abgewaschen werden, bevor Glaswaren/Arbeitsbereich verwendet werden. Verwenden Sie nach Möglichkeit sterile und nur zum Einmalgebrauch bestimmte Verbrauchsmaterialien.
- Herstellung von IVT-Puffern: Bereiten Sie 10x IVT-Puffer vor, indem Sie 400 mM Tris, 10 mM DTT, 20 mM Spermidin, pH 7,9 hinzufügen. Bereiten Sie einen Quenching-Puffer mit 100 mM EDTA, pH 8 vor. Filtern Sie jeden Puffer durch einen 0,22 μm PES-Filter.
- Vorbereitung mobiler Phasen: Vorbereitung der mobilen Phase A (MPA) durch Zugabe von 50 mM HEPES, pH 7,0, mobile Phase B (MPB) durch Zugabe von 50 mM HEPES, 100 mM Na4P2O7, pH 8,3, mobile Phase C (MPC) durch Zugabe von 0,1 M NaOH, 1 M NaCl, mobile Phase D (MPD) durch Zugabe von 0,5 M HEPES, pH-Wert 7,0. Filtern Sie jeden Puffer durch einen 0,22 μm PES-Filter.
2. Vorbereitung der In-vitro-Transkriptionsreaktion (IVT)
ANMERKUNG 1: Die IVT-Reaktion kann eine Batch-Reaktion sein, bei der alle Reagenzien zu Beginn hinzugefügt werden und die Reaktion gestoppt wird, nachdem die NTPs erschöpft sind/die mRNA-Produktion ein Plateau erreicht hat, oder sie kann eine Fed-Batch-Reaktion sein, bei der depletierte NTPs mit zusätzlichen NTPs aufgefüllt werden und die mRNA-Produktion zusätzlich zunimmt. Lineare pDNA-Matrizen, die bei der IVT-Reaktion verwendet werden, können durch die Verwendung eines Restriktionsenzyms, das direkt nach der Poly(A)-Sequenz in der Plasmid-DNA spaltet, oder durch PCR-Reaktion erhalten werden. In beiden Fällen muss nach der Reaktion eine Aufreinigung durchgeführt werden, entweder chromatographisch oder mit kommerziellen DNA-Aufreinigungskits.
- Batch-IVT-Reaktion
- Stellen Sie den Thermoblock auf 37 °C und 300 U/min ein. Tauen Sie alle in Tabelle 1 aufgeführten Reagenzien mit Ausnahme der Enzyme auf und erhitzen Sie sie im Thermoblock auf 37 °C. Bereiten Sie ein 25 mM NTP-Gemisch mit 100 mM Stammlösung aus ATP, CTP, UTP und GTP vor.
- Während die Reagenzien auftauen, bereiten Sie Probenaliquots für das Zeitverlaufsexperiment vor. Nehmen Sie 0,5 ml sterile Röhrchen und pipettieren Sie 2 μl 100 mM EDTA in jedes Röhrchen. Markieren Sie jedes Röhrchen mit einer bestimmten IVT-Nummer (z. B. IVT001, IVT002 usw.) und einem Zeitpunkt (z. B. 0 min, 15 min, 30 min, 60 min usw.).
- Wenn die Reagenzien aufgetaut und etwa 15 Minuten lang bei 37 °C aufbewahrt werden, nehmen Sie die Enzyme (T7-RNA-Polymerase, Pyrophosphatase, RNASE-Hemmer) aus dem -20 °C-Gefrierschrank und bewahren Sie sie in einem Kühler auf, bis sie wieder im Gefrierschrank gelagert werden.
- Zum Mischen einzelner IVT-Reagenzien (außer Enzyme) verwenden Sie einen Vortex. Mischen Sie Enzyme, indem Sie vorsichtig auf und ab pipettieren.
- Mischen Sie IVT-Reagenzien in Verhältnissen, die im Versuchsdesign festgelegt sind (z. B. ist das generische IVT-Protokoll in Tabelle 1 dargestellt) in einem 1,5-ml-Röhrchen. Bei der IVT-Optimierung kann das endgültige IVT-Volumen zwischen 50 und 100 μl liegen. Bei der Gestaltung des IVT-Gesamtvolumens ist die Anzahl der 2 μl Proben zu berücksichtigen, die während des Zeitverlaufsexperiments aus der Reaktion entnommen werden sollen. Stellen Sie sicher, dass das Gesamtvolumen der Proben 50 % des gesamten IVT-Volumens nicht überschreitet (z. B. bei einer Probenahme von 2 μl alle 15 Minuten für 3 Stunden beträgt das Gesamtvolumen der entnommenen Aliquots 30 μl, was bedeutet, dass das Gesamtvolumen der IVT mindestens 60 μl betragen sollte).
- IVTs vorsichtig mischen, T7 RNA-Polymerase als letztes Reagenz hinzufügen. Wenn alle Reagenzien zugegeben sind, mischen Sie IVT gründlich, indem Sie auf und ab pipettieren, verwenden Sie keinen Vortex.
- Unmittelbar nachdem alle IVT-Reagenzien hinzugefügt und homogenisiert wurden, entfernen Sie 2 μl des IVT-Gemisches aus dem Röhrchen und pipettieren Sie es in zuvor vorbereitete 0,5-ml-Röhrchen mit 2 μl 100 mM EDTA, wie in Schritt 2.1.2 beschrieben (Zeitpunkt 0 min).
- Reaktionsgefäße mit IVT-Reaktionsgemischen in einen Thermoblock geben und bei 37 °C inkubieren.
- Stellen Sie den Timer auf die gewünschten Zeitintervalle ein (z. B. 15 min/30 min). Zu jedem Zeitpunkt werden 2 μl einer IVT-Probe aus jedem vorbereiteten IVT-Reaktionsgemisch entnommen und in ein zuvor vorbereitetes Röhrchen (siehe Schritt 2.1.2) pipettiert, das 2 μl 100 mM EDTA enthält. Diese Aliquote werden für die chromatographische Analyse verwendet (siehe Schritt 3).
- Nachdem die chromatographische Analyse die vollständige Depletion der NTPs bestätigt hat, werden IVT-Massenreaktionen mit EDTA auf eine Endkonzentration von 50 mM abgeschreckt (z. B. 30 μl IVT inaktiviert mit 30 μl 100 mM EDTA).
- Fed-Batch-IVT-Reaktion
HINWEIS: Die mRNA-Produktion kann zusätzlich durch Zugabe von NTPs und Mg2+ Ionen gesteigert werden, nachdem die NTPs aufgebraucht sind.- Mischen Sie die Batch-IVT-Reaktion und nehmen Sie die Probenahme wie in Schritt 2.1 beschrieben. Bereiten Sie das NTP+MgCl2-Futter für die gefütterte Charge vor. Zur Herstellung von z.B. 500 μl NTP+MgCl 2-Gemisch werden je 106 μl 200 mM NTP, 76,25 μl 1 M MgCl2 und 5,5 μl ddH2O gemischt. Die Endkonzentrationen in der Mischung betragen jeweils 42,4 mM NTP und 152,5 mM MgCl2.
- Überwachen Sie den NTP-Verbrauch mit der in Schritt 3 beschriebenen At-Line-Analyse. Wenn die At-Line-Analytik bestätigt, dass die NTP-Konzentration unter 10 % der Ausgangskonzentration fällt, fügen Sie das Futter, das NTP+Mg2+ enthält, zur Bulk-IVT hinzu.
- Messen/berechnen Sie das genaue Volumen der IVT-Bulk-Reaktion, das in einem 1,5-ml-Röhrchen verbleibt, nachdem alle Aliquote für die Analyse entnommen wurden. Geben Sie ein geeignetes Volumen des NTP+MgCl2-Gemisches zu einem IVT, so dass die Endkonzentration 4 mM jedes NTP und 12 mM MgCl2 beträgt. Fügen Sie beispielsweise für 80 μl IVT in einem 1,5-ml-Röhrchen 16,8 μl NTP+MgCl2-Mischung hinzu.
- Fahren Sie mit der Probenahme wie in Schritt 2.1 beschrieben fort. Nachdem die NTPs wieder aufgebraucht sind, wiederholen Sie Abschnitt 2.2.2. Fahren Sie mit der Fütterung und Probenahme fort, bis die gewünschte mRNA-Produktion erreicht ist.
- Nachdem die gewünschte mRNA-Produktion erreicht ist, inaktivieren Sie die gesamte Reaktion wie in Schritt 2.1.10.
3. Vorbereitung der chromatographischen Analyse
- Vorbereitung von Standard- und Systemeignungstests (SST)
- Bereiten Sie die SST vor, indem Sie das Deckelreagenz, NTPs, pDNA-Template und mRNA kombinieren. Stellen Sie sicher, dass die Endkonzentrationen ca. 3 μM für das Deckelreagenz, 5 μM für jedes NTP, 4 ng/μl für das pDNA-Template und 10 ng/μl für die mRNA betragen. Stellen Sie sicher, dass der endgültige SST 0,1 M NaCl enthält.
- Bereiten Sie vor jeder Analyse eine neue Kalibrierkurve vor. Erstellen Sie den Kalibrierstandard, indem Sie eine gereinigte mRNA-Probe mit bekannter Konzentration mit MPA verdünnen. Die Kalibrierpunkte sollten 0,5 ng/μL, 2 ng/μL, 5 ng/μL, 10 ng/μL, 15 ng/μL und 20 ng/μL betragen, jeweils mit einer Endkonzentration von 0,1 M NaCl.
- Bereiten Sie bei Bedarf eine Kalibrierkurve für alle NTPs vor. Stellen Sie sicher, dass die Endkonzentrationen jedes NTP in den Kalibrierstandards 0,5 μM, 2 μM, 5 μM, 10 μM, 15 μM und 20 μM betragen.
- Konditionierung der Säule und Vorbereitung für die Analyse
- Vor der Analyse wird die 0,1-ml-Analysesäule 12 Stunden lang bei Raumtemperatur äquilibriert. Befestigen Sie die Säule in der auf dem Säulengehäuse angegebenen Richtung an das chromatographische System.
- Spülen Sie die Säule mit einer Durchflussrate von 1 mL/min: zuerst mit 50 Säulenvolumina (CV) von ddH2O und danach mit 50 CV MPA.
- Führen Sie vor der Analyse mindestens 3 leere Proben (nur Injektion von MPA) durch, um die Baseline festzulegen. Verwenden Sie die chromatographische Methode, die in Skok et al.15 beschrieben ist.
- Sobald die Basislinie stabil ist und zwei aufeinanderfolgende Blindmessungen vergleichbar sind (reproduzierbare Basislinie), fahren Sie mit der Durchführung der SST-Probe und der Kalibrierkurvenproben fort.
- Wenn die SST-Kriterien erfüllt sind und die Trennung zwischen dem Verschließreagenz und den NTPs zufriedenstellend ist, fahren Sie mit der Analyse der IVT-Proben fort. Nachdem Sie alle IVT-Proben analysiert haben, injizieren Sie erneut einen SST-Standard, um zu bestätigen, dass die Systembedingungen während der gesamten Analyse stabil blieben.
- Probenvorbereitung
HINWEIS: Proben für die chromatographische Analyse werden unmittelbar vor der Analyse vorbereitet. Andernfalls werden die Proben im Gefrierschrank bei -20 °C (maximal 24 h) oder <-65 °C (mehr als 24 h) aufbewahrt.- Vor der Verdünnung für die chromatographische Analyse die abgeschreckte IVT-Probe vortexen und herunterschleudern. Analysieren Sie alle EDTA-inaktivierten IVT-Proben (siehe Schritt 2.1.9) mit chromatographischer Analyse nach der in Skok et al.15 beschriebenen Methode.
- Verdünnen Sie die IVT-Proben vor der Analyse, um eine Konzentration innerhalb der Kalibrierkurve zu erreichen. Verwenden Sie während des gesamten Zeitverlaufsexperiments denselben Verdünnungsfaktor, um sicherzustellen, dass NTP-Bereiche verglichen werden können, um zu bestimmen, wann ein NTP erschöpft ist.
- Um die minimale Verdünnung zu bestimmen, die für IVT-Proben erforderlich ist, verwenden Sie die folgende Formel: Erforderliche Verdünnung = (erwartete End-mRNA-Konzentration) / (Verdünnung während der Inaktivierung) / (höchster Kalibrierstandard)
Wenn beispielsweise bei der IVT eine mRNA-Produktion von 8 mg/ml erwartet wird, beträgt die Konzentration in der abgeschreckten Probe 4 mg/ml. Um eine Konzentration innerhalb der Kalibrierkurve zu erreichen, verdünnen Sie die Probe um das 200-fache weiter.
Erforderliche Verdünnung = (8000 ng/μL]) / 2 / (20 ng/μL) = 200 - Pipettieren Sie das MPA und 4 M NaCl in ein konisches Glasfläschchen. Stellen Sie sicher, dass die Endkonzentration von NaCl in der Probe 100 mM beträgt. Zum Schluss fügen Sie die abgeschreckte IVT-Probe hinzu.
- Vortex bereitete die Probe vor und führte sie zur Analyse in den Autosampler ein. Stellen Sie die Temperatur des Autosamplers auf 4 °C ein.
- Injizieren Sie 100 μl verdünnte Proben auf die analytische Säule. Messen Sie die Extinktion A260 und A280 für jede IVT-Probe.
- Zur Analyse von Proben mit bekannten Konzentrationen oder wenn eine Verfolgung des NTP-Verbrauchs auf der Grundlage von Flächenunterschieden nicht erforderlich ist, verdünnen Sie die Proben, um eine Zielkonzentration von 10 ng/μl zu erreichen.
4. Quantifizierung der Probe und Datenanalyse
- Vor der Integration jeder IVT-Probe subtrahieren Sie die Blindprobe vom Probenchromatogramm.
- Integrieren Sie alle NTP-, pDNA- und mRNA-Peaks an A260 und verwenden Sie die Peakbereiche, um die Konzentrationen von NTPs und mRNA in der IVT zu berechnen.
HINWEIS: Alternativ können die Peakbereiche verwendet werden, um den Prozentsatz jedes in der IVT-Reaktion verbleibenden NTP zu vergleichen, vorausgesetzt, der Verdünnungsfaktor war über alle Zeitpunkte hinweg konsistent. - Entscheiden Sie anhand der mRNA/NTP-Konzentrationen, die durch chromatographische At-line-Analyse bestimmt werden, ob IVT inaktiviert werden kann oder ob zusätzliche NTPs in die IVT-Reaktion eingespeist werden sollten (siehe Schritt 2).
Ergebnisse
Die in diesem Protokoll beschriebene chromatographische Analyse kann entweder zur IVT-Optimierung oder zur Umwandlung einer Batch-IVT-Reaktion in eine Fed-Batch-Reaktion verwendet werden (Abbildung 1).
Um die Wirkung verschiedener Pufferzusammensetzungen auf die Kinetik der IVT-Reaktion zu testen, wurden drei verschiedene IVT-Reaktionen gemäß dem in Tabelle 2 beschriebenen Protokoll gemischt. Tris-haltiger Puffer, bei dem der pH-Wert mit HCl eingestellt wurde, wurde mit Tris-Puffer verglichen, bei dem der pH-Wert mit Essigsäure eingestellt wurde. Des Weiteren wurden beide Tris-Puffer mit dem HEPES-Puffer verglichen, wobei der pH-Wert mit NaOH eingestellt wurde. Die Zusammensetzung aller 1x IVT-Puffer bestand aus 40 mM Tris/HEPES, 10 mM DTT, 2 mM Spermidin und pH 7,9.
Aus 100 μl IVT-Reaktionsmischung wurden 2 μl IVT-Probe aus der IVT-Reaktion entnommen und in der ersten Inkubationsstunde alle 15 Minuten und dann alle 30 Minuten bis 180 Minuten der Inkubation mit 2 μl 100 mM EDTA abgeschreckt. Die erwartete endgültige mRNA-Ausbeute für diese Reaktionsbedingung betrug 15 mg/ml, was bedeutet, dass auf die 2-fache Verdünnung durch Quenching eine 400-fache Verdünnung in MPA+NaCl für die Analyse folgte. Die Probe wurde insgesamt 800-fach verdünnt, was bedeutet, dass selbst bei einer Produktion von 15 mg/ml die auf die analytische chromatographische Säule geladene mRNA-Konzentration immer noch innerhalb der Konzentrationen der Kalibrierkurve lag.
NTP- und mRNA-Areale an A260 wurden für jede einzelne Probe zu jedem einzelnen Zeitpunkt (tx) integriert. Die Flächen wurden dann unter Verwendung der mRNA-Kalibrierungskurve und des prozentualen Verbrauchs für NTPs in Konzentrationen für mRNA umgerechnet, wobei die A260-Fläche von NTPs bei 0 min (t0) als 100 % verwendet wurde (siehe Gleichung unten).
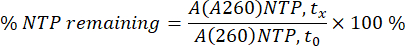
Die Ergebnisse können als Diagramme für jede einzelne IVT-Reaktion dargestellt werden, wobei die Zeit auf der x-Achse und die mRNA-Konzentration und die verbleibenden NTPs auf der y-Achse dargestellt sind (Abbildung 2A). Alle IVT, d. h. mRNA-Konzentrationen in Abhängigkeit von der Zeit, können ebenfalls zusammen in einem Diagramm dargestellt werden, und die Kinetik der mRNA-Produktion kann untersucht und die optimalen IVT-Bedingungen ausgewählt werden (Abbildung 2B).
Für das Fed-Batch-Experiment wurde IVT wie in Tabelle 3 beschrieben gemischt. Von 300 μl IVT-Reaktionsgemisch wurden 2 μl IVT-Probe aus der IVT-Reaktion entnommen und alle 30 min mit 2 μl 100 mM EDTA abgeschreckt. Zusätzlich wurde die Probe unmittelbar nach jeder Zugabe von NTP+MgCl2 in großen Mengen entnommen. Eine Zuführlösung, die 42,4 mM jedes NTP und 152,5 mM MgCl2 enthielt, wurde im Voraus hergestellt, und 106 μl jedes einzelnen 200 mM NTP wurden mit 76,2 μl 1 M MgCl2 gemischt. Es wurde ein Fütterungsschema eingeführt, bei dem stündlich Bolusnahrung hinzugefügt wurde (bei 60 Minuten, 120 Minuten, 180 Minuten und 240 Minuten Inkubation). Das Fütterungsschema ist in Tabelle 3 beschrieben. Die Proben wurden nahezu in Echtzeit analysiert, wobei die Analytik wie für die IVT-Reaktion in großen Mengen beschrieben wurde. Nach 300 min Überwachung wurde die IVT-Reaktion abgeschreckt.
Die Ergebnisse können als verbleibende NTPs/mRNA-Produktion über die Zeit der Inkubation dargestellt werden. Da die Zugabe von Bolusfutter auch die IVT-Reaktion verdünnt, sinkt die mRNA-Konzentration in IVT bei jeder Futterzugabe (Abbildung 3A). Die Zunahme der mRNA-Masse kann auch anhand von Transkriptionsfaktoren (definiert alsmRNA/mpDNA) gemessen und dargestellt werden, was einen linearen Anstieg der mRNA-Produktion im Laufe der Zeit zeigt (Abbildung 3B).
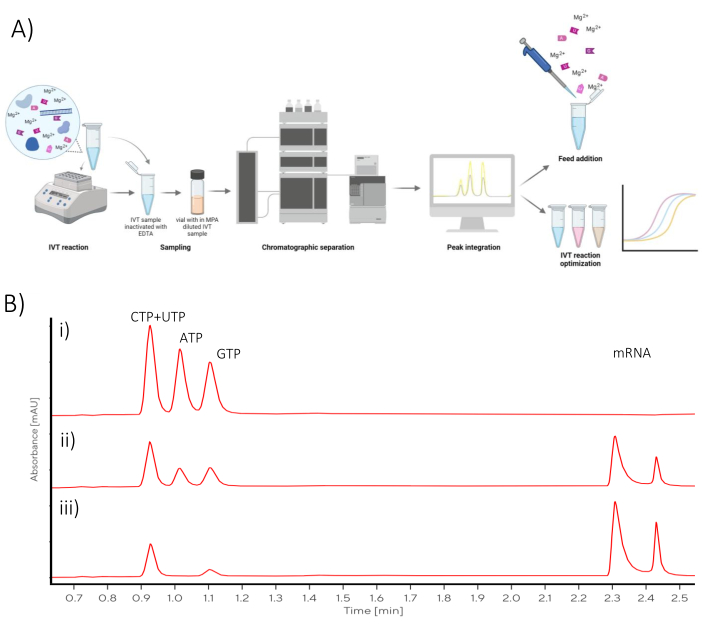
Abbildung 1: Schematische Übersicht (A) Darstellung des IVT-Optimierungsablaufs mit chromatographischer At-Line-Analyse, Quantifizierung von NTPs und mRNA. (B) Repräsentative Chromatogramme einer IVT-Batch-Reaktion, die zu (i) t0, (ii) Mittelpunkt und (iii) Endpunkt der Reaktion entnommen wurden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
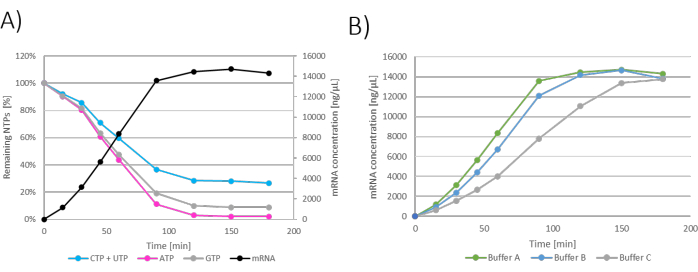
Abbildung 2: Batch-IVT-Diagramme. (A) Repräsentatives Diagramm der IVT-Reaktion mit mRNA-Produktion und NTP-Verbrauch auf der y-Achse. mRNA-Konzentrationsplateau, sichtbar bei 120 - 180 min Inkubation, das mit dem NTP-Verbrauch korreliert, da limitierendes NTP (ATP) nach 120 min verbraucht wird. (B) IVT-Diagramm, das den Einfluss von IVT-Puffer auf die IVT-Kinetik zeigt. IVT, das Puffer A (Tris+Essigsäure) enthält, zeigte die schnellste mRNA-Produktion, gefolgt von Puffer B (Tris+HCl) und Puffer C (HEPES+NaOH) als langsamste. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung 3: IVT-Diagramm der Fed-Charge. (A) IVT-Diagramm, in dem NTP+MgCl2-Futter bei 60, 120, 180 und 240 min zugegeben wurden. Die mRNA-Konzentration in der IVT-Reaktion sinkt bei jeder Futterzugabe aufgrund der Verdünnung mit NTP+MgCl2-Futter . (B) Die durch den Transkriptionsfaktor gezeigte Zunahme der mRNA-Masse ist während der gesamten Reaktionsinkubation linear. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Generisches IVT-Protokoll. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Batch-IVT-Protokoll. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: Fed-Batch-IVT-Protokoll. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Bei dieser Methode werden IVT-Proben zu verschiedenen Zeitpunkten während der Inkubation mit einer analytischen chromatographischen multimodalen Säule analysiert, die NTPs, pDNA und mRNA trennt und so eine genaue Überwachung des NTP-Verbrauchs und der mRNA-Produktion ermöglicht. Diese Methode misst die NTPs- und mRNA-Konzentration quantitativ auf der Grundlage von Veränderungen in A260-Bereichen zu bestimmten Zeitpunkten. Da die Methode Informationen über die NTP- und mRNA-Konzentration liefert, eignet sie sich sehr gut für die IVT-Optimierung, bei der das primäre Ziel oft darin besteht, die mRNA-Ausbeute zu maximieren und die Reaktionszeit zu minimieren. daher ist es von entscheidender Bedeutung, die Wirkung verschiedener IVT-Reagenzien auf die Kinetik der mRNA-Produktionzu verstehen 22. Wir zeigen, wie At-Line-Monitoring zur Optimierung der IVT-Reaktion im Batch- und Fed-Batch-Modus eingesetzt werden kann.
Der Hauptvorteil dieser Methode ist eine At-Line-Analyse, die eine nahezu Echtzeit-Überwachung der IVT ermöglicht, da jede Probe in weniger als 8 Minuten analysiert wird. Die Vorbereitung der Proben für die Analytik ist unkompliziert, da nur eine Verdünnung in MPA ohne vorherige Probenvorbehandlung erforderlich ist. Das für die Analyse erforderliche Probenvolumen ist sehr gering, z. B. 1 μl IVT. Dieses kleine Pipettiervolumen kann aufgrund von Pipettierfehlern zu analytischen Abweichungen führen. Da der hohe Durchsatz des analytischen Ansatzes jedoch dreifache Messungen ermöglicht, sind Ausreißer leicht erkennbar und Abweichungen von der erwarteten kinetischen Kurve nicht schwer zu erkennen.
Eine der Einschränkungen der Methode ist die chromatographische Co-Elution von UTP und CTP. Eine genaue Quantifizierung von UTP und CTP ist in der Regel nicht erforderlich, da das limitierende NTP in einer mRNA-Sequenz oft entweder ATP oder GTP ist. Wenn eine getrennte Quantifizierung von UTP und CTP erforderlich ist, kann die Differenz in der UV-Absorption bei 260 nm und 280 nm für UTP und CTP genutzt werden, um die relative Häufigkeit jedes NTP im chromatographischen Peak abzuleiten.
Bei dieser Analysemethode werden RNA-Spezies mit einer Länge von mehr als 100 Nukleotiden nicht getrennt. daher kann es nicht zum Nachweis von Unterschieden in der mRNA-Qualität verwendet werden, z. B. unterscheidet es nicht zwischen nicht-polyadenylierter und polyadenylierter mRNA, zwischen dsRNA oder abortiven Transkripten und ssRNA und ist nicht für Stabilitätsstudien geeignet, da es nicht zwischen degradierter und nicht degradierter mRNA unterscheiden kann. Die Methode kann jedoch auch für die Quantifizierung anderer RNA-Modalitäten wie circRNA, tRNA und saRNA verwendet werden. Obwohl diese Moleküle in Größe und Struktur variieren, kann die gleiche Analysemethode angewendet werden, um die Produktionsausbeute jedes einzelnen in einer IVT-Reaktion zu untersuchen.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Autoren danken Tomas Kostelec, Blaž Bakalar, Nejc Pavlin, Andreja Gramc Livk und Anže Martinčič Celjar für die hilfreichen Gespräche.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
Referenzen
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403(2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, Ö mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090(2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190(2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3(2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745(2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655(2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833(2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246(2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937(2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408(2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045(2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten