Method Article
Ottimizzazione della reazione di trascrizione in vitro per la produzione di mRNA mediante monitoraggio cromatografico in linea
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'ottimizzazione della trascrizione in vitro (IVT) è fondamentale per una produzione di mRNA economicamente vantaggiosa. Questo protocollo descrive in dettaglio un metodo cromatografico analitico per l'analisi in linea della reazione IVT, il monitoraggio della deplezione di NTP e della produzione di mRNA in modalità batch o fed-batch, applicabile a varie modalità di RNA, migliorando la produttività e riducendo i costi.
Abstract
La reazione di trascrizione in vitro (IVT) è una sintesi enzimatica complessa e multicomponente di mRNA da un modello di DNA lineare, catalizzata da una RNA polimerasi, ad esempio T7. A causa dell'alto costo dei reagenti IVT, l'IVT è una fase fondamentale nel processo di produzione di sostanze farmaceutiche a mRNA ed è stata al centro di un'intensa ottimizzazione sul campo. Per ridurre il costo della produzione di mRNA, i reagenti devono essere utilizzati in modo ottimale. Un'ottimizzazione efficace richiede una comprensione completa dell'impatto dei singoli reagenti sulla cinetica di reazione, ovvero il consumo di nucleosidi trifosfati (NTP) e la produzione di mRNA. Tradizionalmente, per l'analisi dell'mRNA sono state utilizzate tecniche analitiche end-point a basso rendimento. Sebbene tali metodi forniscano informazioni preziose sul contenuto di mRNA, è necessaria un'analisi online e quasi in tempo reale per comprendere appieno la reazione IVT. Dimostriamo come un metodo analitico di cromatografia liquida che separa gli NTP, così come il pDNA e l'mRNA, possa essere utilizzato quasi in tempo reale per studiare la cinetica di reazione IVT. Con la conoscenza dell'influenza dei diversi componenti IVT sulla cinetica e sulla resa, l'analisi cromatografica rapida può essere utilizzata per convertire la reazione batch IVT in una modalità fed-batch che aumenta ulteriormente la produttività e riduce il costo complessivo della reazione.
Introduzione
La pandemia di COVID-19 ha catalizzato una rivoluzione senza precedenti nel campo della biomedicina, portando al rapido sviluppo e all'autorizzazione di vaccini basati su mRNA, come Comirnaty di BioNTech/Pfizer e Spikevax di Moderna, sia in Europa che negli Stati Uniti 1,2,3. L'impressionante efficacia e il rapido sviluppo di questi vaccini hanno evidenziato l'immenso potenziale terapeutico della tecnologia mRNA, non solo per le malattie infettive, ma anche per le immunoterapie antitumorali, le terapie sostitutive delle proteine, la medicina rigenerativa e la riprogrammazione cellulare 4,5. Quest'epoca, spesso definita la rivoluzione dell'mRNA, sottolinea la necessità di processi di produzione di mRNA efficienti ed economici.
La produzione di mRNA comporta operazioni multiple di unità, con la reazione di trascrizione in vitro (IVT) che è un'operazione unitaria critica e più costosa 6,7,8. La reazione IVT sintetizza l'mRNA da un modello di DNA utilizzando l'RNA polimerasi, tipicamente l'RNA polimerasi T7, e i nucleosidi trifosfati (NTP) in presenza di ioni magnesio. Il processo è relativamente semplice e consente la produzione di grandi quantità di mRNA in un breve lasso di tempo, raggiungendo rese di reazione di 2-5 g/L in poche ore e fino a 14 g/L in alcuni rapporti 9,10,11. Tuttavia, l'ottimizzazione della resa della reazione IVT è fondamentale per ridurre i costi di produzione e garantire la scalabilità dei vaccini e delle terapie a mRNA12 e, a causa delle differenze di sequenza che influenzano il consumo di NTP, può essere necessaria per ogni costrutto o famiglia di costrutti.
La reazione viene solitamente eseguita come processo batch, ma i recenti progressi nell'elaborazione fed-batch e nell'analisi in linea hanno aperto nuove strade per ottimizzare la produzione di mRNA 7,13. Le reazioni Fed-batch, che comportano un bolo o l'aggiunta continua di reagenti, possono potenzialmente prolungare i tempi di reazione e aumentare le rese prevenendo l'inibizione del substrato e la degradazione del prodotto co-dipendente14,15.
Lo sviluppo di analisi veloci in linea ha rappresentato un progresso significativo nel campo dell'IVT, consentendo il monitoraggio di componenti chiave della reazione come NTP e mRNA con un ritardo analitico minimo13,16, aggiungendo un'ulteriore dimensione al monitoraggio dell'IVT, che in genere si concentra solo sulla concentrazione di mRNA17. Tradizionalmente, gli mRNA sono stati analizzati utilizzando tecniche come l'elettroforesi su gel di poliacrilammide (PAGE), l'elettroforesi su gel di agarosio o l'elettroforesi capillare. Si tratta di metodi end-point e, pertanto, non possono essere utilizzati per il monitoraggio IVT in tempo reale. Un'alternativa all'analisi descritta in questo protocollo sono i metodi che utilizzano l'aptamero dell'RNA light-up e le coppie di coloranti a fluorescenza. In questo metodo, l'aptamero dell'RNA viene marcato con l'RNA e incubato con un colorante a fluorescenza light-up. L'attività di trascrizione può quindi essere visualizzata con l'intensità di fluorescenza17. Ciò consente l'analisi in tempo reale della quantità e della qualità dell'mRNA trascritto durante l'IVT 21, ma non consente il monitoraggio simultaneo di altri componenti dell'IVT, ad esempio gli NTP. Un'altra alternativa che potenzialmente consente la quantificazione in tempo reale di NTP e mRNA è la spettroscopia Raman18, ma ad oggi nessun rapporto ha dimostrato la sua utilità per il monitoraggio IVT, suggerendo che è ancora necessaria un'ulteriore ottimizzazione del metodo per derivare la selettività e la sensibilità necessarie.
Lo sviluppo continuo di analisi più rapide e selettive supporta l'ottimizzazione dell'IVT per una maggiore resa in modalità batch e fed-batch. La reazione rimane un processo complesso e multiparametrico con più fattori interagenti, per i quali optima può differire tra i tipi di costrutto (ad esempio, mRNA vs saRNA). La concentrazione e il rapporto tra ioni Mg2+ e NTP sono particolarmente influenti e i loro livelli ottimali devono essere attentamente bilanciati per massimizzare la resa dell'mRNA riducendo al minimo la formazione di RNA a doppio filamento (dsRNA), un potente stimolante del sistema immunitario innato19,20. Recentemente, l'alimentazione di UTP a livelli stazionari è stata segnalata come approccio per ridurre la formazione di dsRNA21. Il monitoraggio dei livelli di UTP quasi in tempo reale aggiungerebbe un ulteriore livello di controllo del processo.
In questo protocollo, dimostriamo come il monitoraggio in linea della reazione IVT possa aumentare la resa della produzione di mRNA in modalità batch e fed-batch.
Protocollo
1. Preparazione del tampone
NOTA: Tutti i tamponi devono essere preparati senza RNasi, il che significa che tutti i prodotti chimici e la vetreria devono essere utilizzati solo per lavori privi di RNasi e maneggiati con precauzione. L'acqua utilizzata per la preparazione del tampone e la pulizia della vetreria deve essere certificata come priva di nucleasi. Prima di condurre un esperimento, tutte le superfici di lavoro e la vetreria devono essere spruzzate con un reagente di decontaminazione che elimina le RNasi. Il reagente di decontaminazione deve essere accuratamente lavato via con acqua priva di RNasi prima di utilizzare la vetreria/l'area di lavoro. Se possibile, utilizzare materiali di consumo sterili e monouso.
- Preparazione dei tamponi IVT: preparare 10 tamponi IVT aggiungendo 400 mM di Tris, 10 mM di DTT, 20 mM di spermidina, pH 7,9. Preparare un tampone di estinzione contenente 100 mM di EDTA, pH 8. Filtra ogni tampone attraverso un filtro PES da 0,22 μm.
- Preparazione delle fasi mobili: preparare la fase mobile A (MPA) aggiungendo 50 mM di HEPES, pH 7,0, la fase mobile B (MPB) aggiungendo 50 mM di HEPES, 100 mM di Na4P2O7, pH 8,3, la fase mobile C (MPC) aggiungendo 0,1 M di NaOH, 1 M di NaCl, la fase mobile D (MPD) aggiungendo 0,5 M di HEPES, pH 7,0. Filtra ogni tampone attraverso un filtro PES da 0,22 μm.
2. Preparazione della reazione di trascrizione in vitro (IVT)
NOTA 1: La reazione IVT può essere una reazione batch, in cui tutti i reagenti vengono aggiunti all'inizio e la reazione viene interrotta dopo che gli NTP sono esauriti/la produzione di mRNA raggiunge un plateau, o può essere una reazione fed-batch, in cui gli NTP impoveriti vengono reintegrati con NTP aggiuntivi e con ciò la produzione di mRNA aumenta ulteriormente. Il modello di pDNA lineare utilizzato nella reazione IVT può essere ottenuto utilizzando l'enzima di restrizione che si scinde subito dopo la sequenza di poli (A) nel DNA plasmidico o mediante reazione PCR. In entrambi i casi la reazione deve essere seguita da una purificazione, cromatografica o utilizzando kit di purificazione del DNA commerciali.
- Reazione IVT batch
- Impostare il blocco termico a 37 °C e 300 giri/min. Scongelare tutti i reagenti elencati nella tabella 1 , ad eccezione degli enzimi, e preriscaldarli a 37 °C nel termoblocco. Preparare una miscela NTP da 25 mM utilizzando una soluzione madre da 100 mM di ATP, CTP, UTP e GTP.
- Mentre i reagenti si scongelano, preparare le aliquote dei campioni per l'esperimento sull'andamento temporale. Prelevare provette sterili da 0,5 mL e pipettare 2 μL di EDTA da 100 mM in ciascuna provetta. Contrassegnare ogni provetta con un numero IVT designato (ad es. IVT001, IVT002, ecc.) e un punto temporale (ad es. 0 min, 15 min, 30 min, 60 min, ecc.).
- Quando i reagenti sono stati scongelati e mantenuti a 37 °C per circa 15 minuti, rimuovere gli enzimi (T7 RNA polimerasi, pirofosfatasi, inibitore della RNASI) dal congelatore a -20 °C e conservarlo in un frigorifero fino a quando non viene riposto nel congelatore.
- Per miscelare i singoli reagenti IVT (ad eccezione degli enzimi), utilizzare un vortice. Mescolare gli enzimi pipettando delicatamente su e giù.
- Miscelare i reagenti IVT nei rapporti stabiliti nel disegno sperimentale (ad esempio, il protocollo IVT generico è mostrato nella Tabella 1) in una provetta da 1,5 mL. Quando si esegue l'ottimizzazione IVT, il volume finale dell'IVT può variare tra 50 e 100 μl. Quando si progetta il volume totale dell'IVT, considerare il numero di campioni da 2 μL da estrarre dalla reazione durante l'esperimento nel corso del tempo. Assicurarsi che il volume totale dei campioni non superi il 50% del volume totale dell'IVT (ad esempio, se si campionano 2 μl ogni 15 minuti per 3 ore, il volume totale delle aliquote prelevate è di 30 μl, il che significa che il volume totale dell'IVT deve essere di almeno 60 μl).
- Mescolare attentamente gli IVT, aggiungere l'RNA polimerasi T7 come ultimo reagente. Quando tutti i reagenti sono stati aggiunti, mescolare accuratamente l'IVT pipettando su e giù, non usare il vortice.
- Immediatamente dopo l'aggiunta e l'omogeneizzazione di tutti i reagenti IVT, rimuovere 2 μL di miscela IVT dalla provetta e pipettarla in provette da 0,5 mL precedentemente preparate contenenti 2 μL di EDTA da 100 mM descritto al punto 2.1.2 (punto temporale 0 min).
- Mettere le provette di reazione contenenti miscele di reazione IVT in un termoblocco e incubarle a 37 °C.
- Impostare il timer sugli intervalli di tempo desiderati (ad es. 15 min/30 min). In ogni momento prelevare 2 μl di un campione IVT da ciascuna miscela di reazione IVT preparata e pipettarlo in una provetta precedentemente preparata (vedere il passaggio 2.1.2) contenente 2 μl di EDTA 100 mM. Queste aliquote verranno utilizzate per l'analisi cromatografica (vedi passaggio 3).
- Dopo che l'analisi cromatografica conferma la completa deplezione degli NTP, estinguere le reazioni IVT di massa con EDTA a una concentrazione finale di 50 mM (ad es. 30 μL di IVT inattivati con 30 μL di EDTA 100 mM).
- Reazione all'IVT in batch federale
NOTA: la produzione di mRNA può essere ulteriormente aumentata aggiungendo NTP e ioni Mg2+ dopo che gli NTP sono esauriti.- Miscelare la reazione IVT batch e campionarla come descritto al punto 2.1. Preparare il mangime NTP+MgCl2 per il lotto alimentato. Per preparare, ad esempio, 500 μL di miscela NTP+MgCl2 , miscelare 106 μL di 200 mM NTP, 76,25 μL di 1 M MgCl2 e 5,5 μL di ddH2O. Le concentrazioni finali nella miscela sono 42,4 mM per NTP e 152,5 mM per MgCl2.
- Monitorare il consumo di NTP con l'analisi in linea descritta nel passaggio 3. Quando l'analisi in linea conferma che la concentrazione di NTP scende al di sotto del 10% della concentrazione iniziale, aggiungere il mangime contenente NTP+Mg2+ all'IVT di massa.
- Misurare/calcolare il volume preciso della reazione IVT di massa rimanente in una provetta da 1,5 mL dopo che tutte le aliquote sono state prelevate per l'analisi. Aggiungere un volume appropriato di miscela NTP+MgCl2 a un IVT in modo che la concentrazione finale sia di 4 mM di ciascun NTP e 12 mM di MgCl2. Ad esempio, per 80 μL di IVT sfuso rimasto in una provetta da 1,5 mL, aggiungere 16,8 μL di miscela NTP+MgCl2 .
- Continuare con il campionamento come scritto nel passaggio 2.1. Dopo che gli NTP sono stati nuovamente esauriti, ripetere la sezione 2.2.2. Continuare con l'alimentazione e il campionamento fino a raggiungere la produzione di mRNA desiderata.
- Dopo aver ottenuto la produzione di mRNA desiderata, inattivare l'intera reazione come al punto 2.1.10.
3. Preparazione dell'analisi cromatografica
- Preparazione del test di idoneità standard e del sistema (SST)
- Preparare l'SST combinando il reagente di tappo, gli NTP, il modello di pDNA e l'mRNA. Assicurarsi che le concentrazioni finali siano di circa 3 μM per il reagente di tappatura, 5 μM per ogni NTP, 4 ng/μL per il modello di pDNA e 10 ng/μL per l'mRNA. Assicurarsi che l'SST finale contenga 0,1 M di NaCl.
- Preparare una nuova curva di calibrazione prima di ogni analisi. Creare lo standard di calibrazione diluendo un campione di mRNA purificato di concentrazione nota con MPA. I punti di calibrazione devono essere 0,5 ng/μL, 2 ng/μL, 5 ng/μL, 10 ng/μL, 15 ng/μL e 20 ng/μL, ciascuno con una concentrazione finale di 0,1 M NaCl.
- Se necessario, preparare una curva di calibrazione per tutti gli NTP. Assicurarsi che le concentrazioni finali di ciascun NTP negli standard di calibrazione siano 0,5 μM, 2 μM, 5 μM, 10 μM, 15 μM e 20 μM.
- Condizionamento della colonna e preparazione per l'analisi
- Equilibrare la colonna analitica da 0,1 mL a temperatura ambiente per 12 ore prima dell'analisi. Fissare la colonna al sistema cromatografico nella direzione indicata sull'alloggiamento della colonna.
- Lavare la colonna con una portata di 1 mL/min: prima con 50 volumi di colonna (CV) di ddH2O e poi con 50 CV di MPA.
- Eseguire almeno 3 campioni in bianco (iniettando solo MPA) prima dell'analisi per impostare la linea di base. Utilizzare il metodo cromatografico descritto in Skok et al.15.
- Una volta che la linea di base è stabile e due misurazioni consecutive in bianco sono comparabili (linea di base riproducibile), procedere con l'esecuzione del campione SST e dei campioni della curva di calibrazione.
- Se i criteri SST sono soddisfatti e la separazione del reagente di tappatura e degli NTP è soddisfacente, procedere con l'analisi dei campioni IVT. Dopo aver analizzato tutti i campioni IVT, iniettare nuovamente uno standard SST per confermare che le condizioni del sistema siano rimaste stabili durante l'analisi.
- Preparazione del campione
NOTA: I campioni per l'analisi cromatografica vengono preparati immediatamente prima dell'analisi. In caso contrario, i campioni vengono conservati in congelatore a -20 °C (massimo 24 ore) o <-65 °C (più di 24 ore).- Prima della diluizione per l'analisi cromatografica, agitare e centrifugare il campione IVT estinto. Analizzare tutti i campioni IVT inattivati con EDTA (vedere il passaggio 2.1.9) con l'analisi cromatografica utilizzando il metodo descritto in Skok et al.15.
- Prima dell'analisi, diluire i campioni IVT per ottenere una concentrazione all'interno della curva di calibrazione. Utilizzare lo stesso fattore di diluizione durante l'esperimento nel corso del tempo per garantire che le aree NTP possano essere confrontate per determinare quando un NTP è esaurito.
- Per determinare la diluizione minima necessaria per i campioni IVT, utilizzare la seguente formula: Diluizione necessaria = (concentrazione finale prevista di mRNA) / (diluizione durante l'inattivazione) / (standard di calibrazione più elevato)
Ad esempio, se si prevede una produzione di mRNA di 8 mg/mL nell'IVT, la concentrazione nel campione estinto sarà di 4 mg/mL. Per ottenere la concentrazione all'interno della curva di calibrazione, diluire ulteriormente il campione di 200 volte.
Diluizione necessaria = (8000 ng/μL]) / 2 / (20 ng/μL) = 200 - Pipettare l'MPA e 4 M di NaCl in un flaconcino di vetro. Assicurarsi che la concentrazione finale di NaCl nel campione sia di 100 mM. Infine, aggiungere il campione IVT estinto.
- Vortex ha preparato il campione e lo ha inserito nell'autocampionatore per l'analisi. Impostare la temperatura dell'autocampionatore a 4 °C.
- Iniettare 100 μl di campioni diluiti sulla colonna analitica. Misurare l'assorbanza A260 e A280 per ogni campione IVT.
- Per analizzare campioni con concentrazioni note, o se non è necessario monitorare il consumo di NTP in base alle differenze di area, diluire i campioni per raggiungere una concentrazione target di 10 ng/μL.
4. Quantificazione dei campioni e analisi dei dati
- Prima dell'integrazione di ciascun campione IVT, sottrarre il campione bianco dal cromatogramma del campione.
- Integra tutti i picchi di NTP, pDNA e mRNA in A260 e utilizza le aree dei picchi per calcolare le concentrazioni di NTP e mRNA nell'IVT.
NOTA: In alternativa, le aree di picco possono essere utilizzate per confrontare la percentuale di ciascun NTP rimanente nella reazione IVT, a condizione che il fattore di diluizione sia coerente in tutti i punti temporali. - Sulla base delle concentrazioni di mRNA/NTP, determinate mediante analisi cromatografica in linea, decidere se l'IVT può essere inattivato o se ulteriori NTP devono essere inseriti nella reazione IVT (vedere la fase 2).
Risultati
L'analisi cromatografica descritta in questo protocollo può essere utilizzata sia per l'ottimizzazione dell'IVT che per convertire una reazione IVT batch in una reazione fed-batch (Figura 1).
Per testare l'effetto di diverse composizioni di tampone sulla cinetica della reazione IVT, sono state miscelate tre diverse reazioni IVT secondo il protocollo scritto nella Tabella 2. Il tampone contenente Tris, in cui il pH è stato regolato con HCl, è stato confrontato con il tampone Tris, in cui il pH è stato regolato con acido acetico. Inoltre, entrambi i tamponi Tris sono stati confrontati con il tampone HEPES, dove il pH è stato regolato con NaOH. La composizione di tutti i tamponi IVT 1x era di 40 mM Tris/HEPES, 10 mM DTT, 2 mM di spermidina e pH 7,9.
Da 100 μL di miscela di reazioni IVT, 2 μL di campione IVT sono stati prelevati dalla reazione IVT e temprati con 2 μL di EDTA 100 mM ogni 15 minuti nella prima ora di incubazione e poi ogni 30 minuti fino a 180 minuti di incubazione. La resa finale di mRNA prevista per questa condizione di reazione era di 15 mg/mL, il che significa che la diluizione di 2 volte dovuta al quenching è stata seguita da una diluizione di 400 volte in MPA+NaCl per l'analisi. Il campione è stato diluito 800 volte in totale, il che significa che anche alla produzione di 15 mg/mL, la concentrazione di mRNA caricata sulla colonna cromatografica analitica era ancora all'interno delle concentrazioni della curva di calibrazione.
Le aree NTP e mRNA ad A260 sono state integrate per ogni singolo campione in ogni singolo punto temporale (tx). Le aree sono state quindi convertite in concentrazioni per l'mRNA, utilizzando la curva di calibrazione dell'mRNA e la percentuale di consumo per gli NTP, utilizzando l'area A260 degli NTP a 0 min (t0) come 100% (vedi equazione sotto).
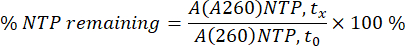
I risultati possono essere mostrati come grafici per ogni singola reazione IVT, dove il tempo è mostrato sull'asse x e la concentrazione di mRNA e gli NTP rimanenti sono mostrati sull'asse y (Figura 2A). Tutte le IVT, ovvero le concentrazioni di mRNA in funzione del tempo, possono anche essere tracciate insieme in un unico grafico e possono essere studiate le cinetiche di produzione dell'mRNA e selezionate le condizioni ottimali di IVT (Figura 2B).
Per l'esperimento fed-batch, l'IVT è stato miscelato come scritto nella Tabella 3. Da 300 μl di miscela di reazione IVT, 2 μl di campione IVT sono stati rimossi dalla reazione IVT e temprati con 2 μl di 100 mM EDTA ogni 30 minuti. Inoltre, il campione è stato prelevato immediatamente dopo ogni aggiunta di NTP+MgCl2 . Una soluzione di alimentazione contenente 42,4 mM di ciascun NTP e 152,5 mM di MgCl2 è stata preparata in anticipo e 106 μL di ogni singolo 200 mM NTP sono stati miscelati con 76,2 μL di 1 M MgCl2. È stato stabilito un regime di alimentazione, in cui il mangime in bolo è stato aggiunto ogni ora (a 60 minuti, 120 minuti, 180 minuti e 240 minuti di incubazione). Il regime di alimentazione è descritto nella Tabella 3 I campioni sono stati analizzati quasi in tempo reale con analisi come descritto per la reazione IVT di massa. Dopo 300 minuti di monitoraggio, la reazione IVT è stata estinta.
I risultati possono essere mostrati come produzione residua di NTP/mRNA nel tempo di incubazione. Poiché le aggiunte di mangime in bolo diluiscono anche la reazione IVT, la concentrazione di mRNA nell'IVT diminuisce ad ogni aggiunta di mangime (Figura 3A). L'aumento della massa dell'mRNA può anche essere misurato e presentato da fattori di trascrizione (definiti comem mRNA/m pDNA), mostrando un aumento lineare della produzione di mRNA nel tempo (Figura 3B).
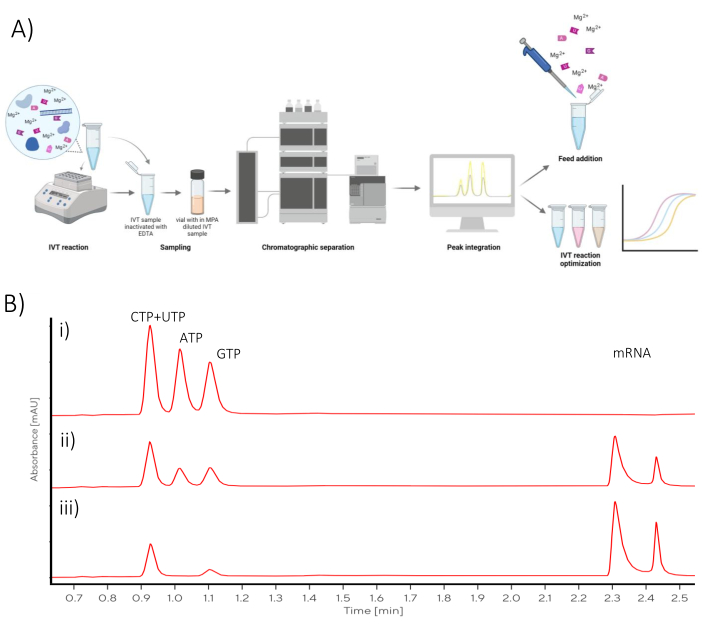
Figura 1: Panoramica schematica (A) Rappresentazione del flusso di lavoro di ottimizzazione IVT con quantificazione dell'analisi cromatografica in linea di NTP e mRNA. (B) Cromatogrammi rappresentativi di una reazione IVT batch campionata a (i) t0, (ii) punto medio, (iii) punti temporali di reazione finale. Clicca qui per visualizzare una versione più grande di questa figura.
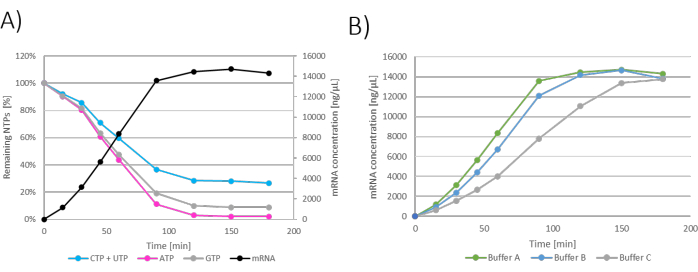
Figura 2: Grafici IVT in batch. (A) Grafico rappresentativo della reazione IVT con la produzione di mRNA e il consumo di NTP sull'asse y. Plateau di concentrazione di mRNA visibile a 120 - 180 minuti di incubazione, correlato al consumo di NTP poiché l'NTP limitante (ATP) viene consumato a 120 minuti. (B) Grafico IVT che mostra l'influenza del tampone IVT sulla cinetica IVT. L'IVT contenente il tampone A (Tris+acido acetico) ha mostrato la produzione di mRNA più veloce, seguita dal tampone B (Tris+HCl) e dal tampone C (HEPES+NaOH) come il più lento. Clicca qui per visualizzare una versione più grande di questa figura.
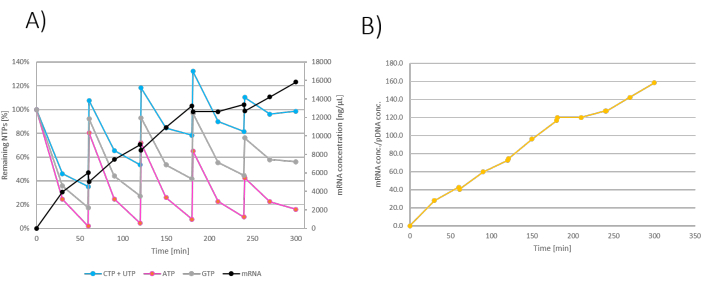
Figura 3: Grafico IVT Fed-batch. (A) Grafico IVT in cui sono stati aggiunti mangimi NTP+MgCl2 a 60, 120, 180 e 240 minuti. La concentrazione di mRNA nella reazione IVT diminuisce ad ogni aggiunta di mangime a causa della diluizione con il mangime NTP+MgCl2 . (B) L'aumento della massa dell'mRNA mostrato dal fattore di trascrizione è lineare durante l'incubazione della reazione. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1: Protocollo IVT generico. Clicca qui per scaricare questa tabella.
Tabella 2: Protocollo IVT batch. Clicca qui per scaricare questa tabella.
Tabella 3: Protocollo IVT Fed-batch. Clicca qui per scaricare questa tabella.
Discussione
In questo metodo, i campioni IVT vengono analizzati in diversi momenti durante l'incubazione utilizzando una colonna multimodale cromatografica analitica che separa NTP, pDNA e mRNA, consentendo così un attento monitoraggio del consumo di NTP e della produzione di mRNA. Questo metodo misura la concentrazione di NTP e mRNA in modo quantitativo sulla base delle variazioni nelle aree A260 in punti temporali designati. Poiché il metodo fornisce informazioni sulla concentrazione di NTP e mRNA, è molto adatto per l'ottimizzazione dell'IVT, dove l'obiettivo principale è spesso quello di massimizzare la resa dell'mRNA e ridurre al minimo il tempo di reazione; pertanto, comprendere l'effetto di diversi reagenti IVT sulla cinetica di produzione dell'mRNA è fondamentale22. Mostriamo come il monitoraggio in linea può essere applicato per ottimizzare la reazione IVT in modalità batch e fed-batch.
Il vantaggio principale di questo metodo è un'analisi in linea che consente il monitoraggio quasi in tempo reale dell'IVT poiché ogni campione viene analizzato in meno di 8 minuti. La preparazione dei campioni per l'analisi è semplice, poiché è necessaria solo la diluizione in MPA senza alcun pretrattamento preliminare del campione. Il volume del campione richiesto per l'analisi è molto basso, ad esempio 1 μL di IVT. Questo volume di pipettaggio ridotto potrebbe potenzialmente causare deviazioni analitiche dovute a errori di pipettaggio. Tuttavia, poiché l'elevata produttività dell'approccio analitico consente misurazioni triplicate, i valori anomali sono facilmente identificabili e le deviazioni dalla curva cinetica prevista non sono difficili da rilevare.
Uno dei limiti del metodo è la co-eluizione cromatografica di UTP e CTP. La quantificazione precisa di UTP e CTP di solito non è necessaria poiché l'NTP limitante in una sequenza di mRNA è spesso ATP o GTP. Se è necessaria una quantificazione separata di UTP e CTP, la differenza nell'assorbanza UV a 260 nm e a 280 nm per UTP e CTP può essere sfruttata per derivare l'abbondanza relativa di ciascun NTP nel picco cromatografico.
Questo metodo analitico non separa le specie di RNA di lunghezza superiore a 100 nucleotidi; pertanto, non può essere utilizzato per rilevare differenze nella qualità dell'mRNA, ad esempio, non distingue tra mRNA non poliadenilato e poliadenilato, tra dsRNA o trascritti abortivi e ssRNA e non è adatto per studi di stabilità in quanto non può distinguere tra mRNA degradato e non degradato. Tuttavia, il metodo può essere utilizzato per la quantificazione di altre modalità di RNA, come circRNA, tRNA e saRNA. Sebbene queste molecole varino in dimensioni e struttura, lo stesso metodo analitico può essere impiegato per studiare le rese di produzione di ciascuna in una reazione IVT.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori ringraziano Tomas Kostelec, Blaž Bakalar, Nejc Pavlin, Andreja Gramc Livk e Anže Martinčič Celjar per le utili discussioni.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
Riferimenti
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403 (2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, &. #. 2. 1. 4. ;. mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090 (2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190 (2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3 (2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745 (2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655 (2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833 (2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246 (2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937 (2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408 (2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045 (2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon