Method Article
Modificación enzimática y evaluación por citometría de flujo de proteínas mostradas en la superficie de la levadura
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, presentamos un protocolo para el uso de sustratos de levadura mostrados en la superficie para ensayos de modificación enzimática. La plataforma se demostró utilizando el análisis de la actividad de desfosforilación de la tirosina fosfatasa SHP-2 frente a uno de sus sustratos como ensayo representativo de modificación enzimática.
Resumen
La visualización de la superficie de la levadura es una estrategia de ligamiento genotipo-fenotipo que permite el cribado de alto rendimiento de la función de las proteínas. Tradicionalmente, la visualización de la superficie de la levadura se ha aplicado a la evolución de nuevas proteínas de unión, con citometría de flujo utilizada para evaluar y clasificar por niveles de fuerza de unión. Recientemente, ha habido un creciente interés en la aplicación de la visualización de la superficie de la levadura para la modificación enzimática del cribado de variantes de sustrato, con modificaciones aditivas (por ejemplo, fosforilación) o sustractivas (por ejemplo, proteólisis) que proporcionan un fenotipo legible por citometría de flujo. Tales modificaciones se aplican regularmente utilizando la colocalización intracelular, pero la capacidad de lograr la modificación enzimática extracelular de los sustratos mostrados podría abrir muchas más reacciones a la investigación. En este trabajo se describen las técnicas para diseñar y aplicar ensayos de cribado para la modificación enzimática extracelular de los sustratos candidatos que se muestran en la superficie de la levadura y su posterior evaluación mediante análisis de citometría de flujo. Proporcionamos estos protocolos en el contexto de las fosfatasas, la levadura desfosforilante, los sustratos mostrados que contienen residuos de tirosina fosforilada y comentamos cómo este marco aplicado se puede adaptar al desarrollo de ensayos de cribado para otros pares de enzimas-sustrato.
Introducción
La comprensión de las interacciones entre las enzimas y sus dianas se ha convertido en un área de investigación cada vez más interesante debido a su necesidad en la caracterización biológica de las vías que controlan la homeostasis celular y el desarrollo de enfermedades 1,2. Las enzimas son responsables de la catálisis de muchas de las reacciones que mantienen la vida biológica, controlando vías necesarias como el metabolismo celular 3,4, la señalización5, e incluso procesos fundamentales como la reparación del genoma 6,7. Debido a su papel en estos procesos, sus interacciones también juegan un papel en el desarrollo de muchas enfermedades, ya que las desviaciones en su actividad pueden causar una desregulación severa en la actividad celular, causando apoptosis o proliferación de células cancerosas dañinas2. El estudio de la actividad enzimática ha tenido importantes aplicaciones en el desarrollo de nuevas terapias 8,9, requiriendo ensayos que se adapten a cada interacción enzima-sustrato específica10. Se han establecido múltiples ensayos enzimáticos como protocolos estándar para la evaluación y caracterización de estas interacciones. Los ensayos desarrollados para analizar las interacciones enzimáticas se clasifican en ensayos de detección que monitorean la unión para la activación/inhibición11 o ensayos que monitorean la modificación del sustrato por enzimas12.
Una de las principales funciones de las enzimas es la regulación del comportamiento celular. La transducción de señales, la respuesta intracelular de una célula a un desencadenante extracelular13, es responsable de la supervivencia y funcionalidad celular. La proliferación celular, la diferenciación y muchos otros procesos funcionales involucran vías de señalización con interacciones enzimáticas que las gobiernan14,15. Las enzimas catalizan modificaciones postraduccionales, que a menudo modulan las redes masivas de señalización responsables de la correcta transmisión de mensajes extracelulares16. La fosforilación de proteínas es la modificación postraduccional más común, ubicua en la señalización celular y en muchas otras vías celulares. En consecuencia, las proteínas quinasas se han convertido en una proporción significativa de posibles dianas terapéuticas debido a su papel regulador crítico17. Las fosfatasas son las moléculas moduladoras naturales de los complejos de señalización celular basados en fosfato18,19, y tienen la capacidad de eliminar los residuos de fosfato de sus proteínas diana20. En la última década, las fosfatasas se han convertido en una diana terapéutica importante para el tratamiento del cáncer21 y las enfermedades inflamatorias22 debido a su participación en la regulación de las vías de señalización posteriores en múltiples tipos de células. Juntas, las proteínas quinasas y las fosfatasas proporcionan una amplia gama de interacciones, que pueden estudiarse mediante el desarrollo de protocolos de ensayos enzimáticos específicos.
La visualización de la superficie de la levadura se ha utilizado como herramienta para la caracterización y evaluación de la actividad enzimática23,24. Proporciona una plataforma de alto rendimiento para el cribado de procesos de modificación postraduccional cuando se combina con estrategias de secuestro del retículo endoplásmico25,26. Esto permite que los pares quinasa-sustrato se colocalicen y retengan en el retículo endoplásmico a través de la unión a los receptores KDEL27, donde la fosforilación del sustrato puede ocurrir a tasas mayores debido a la proximidad entre las quinasas y sus objetivos. La unión al receptor KDEL está mediada por una secuencia de retención del retículo endoplásmico FEHDEL C-terminal que ha demostrado tener una mayor capacidad de retención que otras secuencias HDEL25,28. A continuación, el sustrato fosforilado se ancla a la superficie de la levadura para su posterior evaluación mediante citometría de flujo29. Actualmente, no existen protocolos generalizables establecidos para la modificación enzimática de los sustratos mostrados en la superficie de la levadura. Ampliamos las capacidades de visualización de la superficie de la levadura aprovechando las variantes de sustrato fosforilado expresadas extracelularmente y modificándolas a través de la desfosforilación por su fosfatasa conocida. El análisis de citometría de flujo proporciona una plataforma para la evaluación fenotípica de los sustratos antes mencionados a través de la medición de alteraciones en la mediana de fosforilación como consecuencia de la incubación con la fosfatasa conocida. Esto proporciona un método adaptable para la modificación postraduccional de proteínas mostradas en la superficie, al tiempo que proporciona un método para el análisis de modificación enzimática de las interacciones cuando se utiliza la plataforma de visualización de la superficie de la levadura.
Presentamos técnicas para el desarrollo y aplicación de un ensayo de modificación enzimática que describe la introducción de una interacción quinasa-sustrato en la plataforma de visualización de la superficie de la levadura, la co-incubación del sustrato fosforilado expresado con una fosfatasa recombinante, y el posterior análisis de la actividad de desfosforilación mediante citometría de flujo. En este informe, esto se logra mediante la colocalización del dominio citoplasmático de CD28 con la quinasa de linfocitos (LCK) en el retículo endoplásmico de levadura, seguido de la visualización del CD28 fosforilado en la superficie de la levadura y la posterior desfosforilación por la región de homología Src que contiene fosfatasa-2 (SHP-2). Un anticuerpo antifosfotirosina pan (en este estudio, 4G10), que detecta residuos de tirosina fosforilada en una amplia variedad de secuencias peptídicas, se utiliza para cuantificar el nivel de fosforilación en función del tratamiento con fosfatasa. El proceso detallado proporciona un enfoque generalizable para investigar las interacciones enzima-sustrato; Una forma prospectiva de estudiar enzimas y sustratos de forma purificada.
Protocolo
1. Crecimiento celular de levaduras que albergan plásmidos e inducción de la expresión de proteínas
- Siguiendo la receta descrita en la Tabla 1, prepare los medios necesarios para el crecimiento de levaduras que no contienen plásmidos (YPD), el crecimiento de células de levadura que contienen plásmidos (SD-CAA) y la inducción de la expresión de proteínas (SRG-CAA), así como placas SD-CAA.
- El ADN plásmido de la muestra de levadura que contiene el par quinasa-sustrato en células de levadura EBY-100 a través del método basado en cationes de litio30,31, comúnmente adoptado para su uso en kits de transformación de levadura de una variedad de fabricantes.
NOTA: La electroporación32 u otras técnicas preferidas de transformación de plásmidos de levadura se pueden utilizar dependiendo de la construcción de plásmido que se esté transformando. - Prepare un tubo de cultivo de 14 mL con 10 mL de medio YPD. Inocular células EBY-100 y cultivarlas en una incubadora agitadora a 30 °C, 300 rpm hasta que el cultivo alcance una densidad óptica (OD600nm) de 0,8-1,0 (8 x 106- 1 x 107 células/mL).
NOTA: El diámetro exterior de600 nm se mide preparando cubetas de muestra de 3 ml que contienen diluciones 1:10 de cultivos de levadura en sus respectivos medios y cubetas en blanco de 3 ml que contienen los medios utilizados para las diluciones de muestras. El programa OD600nm en el espectrofotómetro se utiliza para medir primero la cubeta en blanco, luego cada cubeta de muestra configurando la dilución correspondiente preparada para cada muestra. 1 OD600 nm corresponde a 1 × 107 levaduras/mL. - Recolectar las células centrifugando el cultivo a 1.000 x g durante 3 min, y lavar con la1ª solución de lavado suministrada en el kit de transformación de levaduras, o TE (10 mM de Tris-HCl y 1,0 mM de EDTA)31.
- Vuelva a granular las células a 1.000 x g durante 3 min y vuelva a suspender en 1 mL del tampón de transformación proporcionado en el kit de transformación de levadura, o agua estéril. Las pilas deben alícuotas de 50 μL y pueden almacenarse a -80 °C durante un máximo de 6 meses.
- Para la transformación de plásmidos, se prepara una alícuota por plásmido y se descongela en hielo, luego, se agregan directamente a las células 0,5-1,5 μg de ADN plásmido que contiene la construcción de visualización de levadura. Se añaden 0,5 mL de la solución de transformación proporcionada en el kit de transformación de levadura, o 0,5 mL de una solución estéril de polietilenglicol al 50% y LiOAc0,1 M 31. Combine bien la mezcla de células, ADN plasmídico y solución de transformación mediante pipeteo.
- Incubar la mezcla de transformación estáticamente durante 30-60 min a 30 °C, la mezcla de vórtice a intervalos de 15 min. Recolectar las células centrifugando a 1.000 × g durante 3 min.
- Prepare un tubo de cultivo de 14 mL con 4,5 mL de medio SD-CAA. Vuelva a suspender las células que contienen el plásmido deseado en 500 μL de SD-CAA e inocule los 4,5 mL preparados.
- Teniendo cuidado de no perforar el agar, distribuya 50 μL de los 5 mL de cultivo inoculado en una placa SD-CAA e incube estáticamente a 30 °C durante 48 h para determinar la eficiencia de la transformación.
- Incubar los 5 mL de cultivo celular SD-CAA en una incubadora agitadora a 30 °C, 300 rpm durante al menos 18 h. Supervise la densidad óptica (OD600 nm) después de 16 h y 20 h. Una vez que la muestra haya crecido hasta una densidad óptica suficiente que no exceda de 6, centrifugar el cultivo durante 3 min a 2.500 × g. Deseche el sobrenadante sin alterar la pelletza de levadura.
- Vuelva a suspender el pellet de levadura en SRG-CAA a un diámetro exterior final de600 nm inferior a 1 (<1 × 107 levaduras/ml).
- Incubar el cultivo de levadura en una incubadora agitadora a 30 °C, 300 rpm durante al menos 8 h pero no más de 24 h.
NOTA: La inducción de la expresión de proteínas en las células de levadura puede variar entre 20 y 37 °C. 30 °C es adecuado para la síntesis de pares quinasa/sustrato29,33, pero puede ajustarse si se considera necesario para las proteínas específicas que se están estudiando. - Mida OD600 nm para determinar la densidad de celdas.
NOTA: El protocolo puede detenerse en este punto almacenando los cultivos de levadura a 4 °C.
2. Biotinilación del anticuerpo antifosfotirosina 4G10
- Vuelva a suspender un vial de 2 mg de PEG4-NHS-Biotina hasta una concentración final de 5 mM añadiendo 680 μL de PBS estéril.
NOTA: La resuspensión de PEG4-NHS-Biotina debe realizarse inmediatamente antes de que se lleve a cabo la reacción de biotinilación. NHS se hidroliza en solución acuosa. El uso de PBS y viales estériles es importante para la preparación de reactivos para el almacenamiento a largo plazo y para su uso en ensayos basados en células para mitigar cualquier efecto potencial de los contaminantes en la viabilidad de los reactivos o en los ensayos sensibles que se están realizando. - En función de la concentración de anticuerpos 4G10, agregue 100 μg de anticuerpo en un vial estéril de 1,7 ml.
- Añada 1 μL de los 5 mM de PEG4-NHS-Biotina preparados del paso 2.1 al vial que contiene los 100 μg de anticuerpo 4G10 para lograr una proporción molar de biotina a anticuerpo de 7,5:1. Pipetear la mezcla suavemente para homogeneizar la reacción.
- Incubar la reacción a temperatura ambiente con rotación constante durante al menos 2 h.
- Siga el protocolo del fabricante para las columnas de desalinización de centrifugación de 0,5 mL para intercambiar el tampón del 4G10 biotinilado (B-4G10) en PBS.
NOTA: Las columnas de desalinización con un corte de peso molecular (MWCO) de 7 kDa se usan comúnmente para la biotinilación de anticuerpos para permitir que la biotina y otras moléculas pequeñas sin reaccionar se eliminen mientras se retiene el anticuerpo más grande. - Diluir el anticuerpo B-4G10 hasta una concentración final de 1 μM en PBSA (PBS con 1 g/L de albúmina sérica bovina). Alícuota el anticuerpo B-4G10 en volúmenes más pequeños para evitar ciclos repetidos de congelación/descongelación.
NOTA: Los anticuerpos biotinilados pueden almacenarse a 4 °C para su uso diario durante un máximo de 3 meses sin perder una eficacia significativa. Almacene el resto de las alícuotas que no estén en uso a -20 °C durante un máximo de 2 años.
3. Desfosforilación de sustratos expresados en la superficie celular de la levadura
- Prepare la solución tampón de trabajo 2x como se describió anteriormente en la literatura34.
NOTA: Se recomienda 2x para facilitar la medición de los ingredientes necesarios. - Prepare el tampón de trabajo para las muestras en un vial de 1,7 ml diluyendo la solución tampón 2x preparada en el paso 3.1 1:2 en agua desionizada.
NOTA: El volumen total de reacción para cada muestra será de 20 μL, y cada muestra requerirá entre 10 μL y 18 μL de tampón de trabajo. Prepare suficiente tampón de trabajo para todas las muestras o controles. - Siguiendo la preparación de muestra recomendada descrita en la Tabla 2, etiquete los viales de 1,7 ml con el nombre de control o muestra correspondiente.
- Sobre la base del diámetro exterior de600 nm medido en el paso 1.9, calcule el volumen de cultivo necesario para recuperar dos millones (2 × 106) células de levadura del cultivo de levadura correspondiente para cada muestra.
- Añada el volumen de cultivo de levadura calculado en el paso anterior en el vial de 1,7 ml para cada muestra.
- Centrifugar el vial durante 1 min a 4.500 × g. Retire con cuidado el sobrenadante con una micropipeta y deséchelo como residuo biopeligroso.
- Vuelva a suspender las células peletizadas en 1 ml de PBSA y repita el paso 3.6.
NOTA: Es importante eliminar la mayor cantidad posible de sobrenadante sin alterar las células granuladas. - Sobre la base de su concentración de existencias, calcule el volumen de SHP-2 humano recombinante necesario para tener una concentración final de 1.000 nM en un volumen total de reacción de 20 μL.
NOTA: La SHP-2 recombinante debe alícuota en pequeños volúmenes para que cualquier proteína utilizada en cada ensayo no haya pasado por más de dos ciclos de congelación-descongelación. Cualquier SHP-2 que quede de una alícuota después de que todas las muestras se hayan preparado para un ensayo debe desecharse.
Las concentraciones de enzimas recombinantes pueden variar en función del número de lote. El SHP-2 recombinante se formula comúnmente a una concentración de 0,2-0,4 mg/mL. Para una concentración de stock de 0,324 mg/mL SHP-2, esto corresponde a una concentración de stock de 4,696 μM (SHP-2 tiene un peso molecular de 69 kDa). 4,26 μL de la cepa SHP-2 en una reacción de 20 μL dan como resultado una concentración de reacción final de 1.000 nM SHP-2. - Añadir 7,7 mg de DTT en 10 mL de agua desionizada preparada en un cónico de 15 mL para crear una solución de DTT de 5 mM. Si las limitaciones del equipo dificultan el peso de miligramos, agregue 0,77 g de DTT a 10 mL de agua desionizada, luego realice una dilución de 100x para crear la solución de DTT de 5 mM utilizada para el ensayo.
NOTA: La solución de TDT se puede preparar en soluciones madre de mayor concentración para diluir a 5 mM si no es posible medir cantidades a escala de miligramos utilizando el equipo disponible. La solución de DTT debe prepararse fresca antes de la resuspensión celular en un tampón de trabajo debido a su tendencia a hidrolizarse, lo que la hace inestable durante largos períodos cuando se diluye en agua. - Vuelva a suspender las células peletizadas en el tampón de trabajo preparado en el paso 3.2 de modo que el volumen final de reacción en cada muestra o control sea de 20 μl.
NOTA: La cantidad de tampón de trabajo añadido debe calcularse en función de la cantidad de DTT (2 μL) y SHP-2 (calculada en el paso 3.8) que habrá en cada muestra. - Añada 2 μL de la solución de DTT de 5 mM preparada en el paso 3.9 a cada muestra o control para obtener una concentración de reacción final de 0,5 mM de DTT.
- Añada el volumen de SHP-2 calculado en el paso 3.8 a cada muestra hasta obtener un volumen final de 20 μL y mezcle suavemente con una micropipeta.
- Envuelva las tapas de los viales de muestra en parafilm para evitar fugas o contaminación cruzada.
- Incubar las muestras a 37 °C durante 2 h en un rotor a velocidad constante.
- Retire las muestras del rotor y detenga la reacción agregando 1 mL de PBSA a cada muestra.
- Repita el paso 3.6.
4. Marcaje celular y análisis por citometría de flujo de sustratos desfosforilados
- Vuelva a suspender las muestras del paso 3.16 en una mezcla de 20 μL de sus reactivos primarios correspondientes, como se describe en la Tabla 2. Incubar las muestras durante 20 minutos a temperatura ambiente.
NOTA: Se ha calculado que todas las concentraciones de reactivos utilizadas son superiores al número de proteínas expresadas en la superficie de las células de levadura. El cálculo supone la expresión de 10.000 proteínas/célula de todas las levaduras 2 x 106 en una muestrade 35, cuando habitualmente solo ~50% de ellas lo hacen. La Tabla 3 muestra la proporción de reactivos de marcado en exceso para cada uno de los reactivos expresada en la Tabla 2, calculada de la siguiente manera:
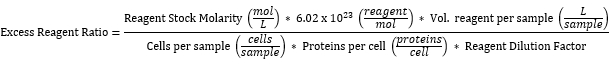
- Centrifugar las muestras a 4.500 × g durante 1 min y desechar el sobrenadante como residuo biopeligroso.
- Lave las células una vez resuspendiéndolas en 1 mL de PBSA. Repita el paso 4.2.
- Vuelva a suspender las muestras en una mezcla de 20 μL de sus reactivos secundarios correspondientes, como se describe en la Tabla 2. Incubar las muestras durante 15 min en ausencia de luz.
- Repita el paso 4.2.
- Repita el paso 4.3.
- Vuelva a suspender las muestras lavadas en 300-500 μL de PBSA y transfiéralas a tubos de poliestireno de 5 mL para analizarlas inmediatamente utilizando un citómetro de flujo adecuado.
NOTA: Si es necesario transportar muestras, manténgalas sobre hielo húmedo. No se recomienda, pero las muestras se pueden almacenar a 4 °C durante un máximo de 2 h como gránulos húmedos. - Después de realizar el inicio y la preparación necesarios del citómetro para un nuevo experimento, haga clic en el botón Nuevo experimento dentro del menú Archivo , asigne un nombre al experimento y haga clic en Guardar para asegurarse de que los datos adquiridos se guarden en la ruta de archivo deseada.
- Seleccione el icono de diagrama de puntos dentro de la barra de herramientas superior para crear dos o más diagramas de puntos para cada muestra que se va a ejecutar. Para uno de los diagramas de puntos, seleccione el nombre del eje X para mostrar el canal FSC-A y el nombre del eje Y para mostrar el canal SSC-A. Este gráfico muestra la dispersión lateral - área frente a la dispersión directa - área y se utiliza para controlar las células de levadura para su posterior análisis.
- En otro diagrama de puntos, seleccione el nombre del eje X para mostrar el canal en el que el reactivo secundario dirigido al anticuerpo primario de la etiqueta antiepítopo emite fluorescencia. Seleccione el nombre del eje Y para mostrar el canal en el que el reactivo secundario de estreptavidina emite fluorescencia. Este gráfico mostrará solo los eventos cerrados desde el diagrama de dispersión lateral frente al frontal como células de levadura y se utiliza para mostrar la fosforilación de tirosina en el eje Y y la expresión de la superficie del sustrato en el eje X.
NOTA: En este ejemplo, el reactivo secundario dirigido al anticuerpo primario anti-epítopo marca fluorescencia en el canal FITC (AF-488) y el reactivo secundario de estreptavidina emite fluorescencia en el canal AF-647. Los canales utilizados pueden variar en función de los reactivos primarios y secundarios utilizados durante el etiquetado. - Coloque cada tubo de muestra en el soporte del tubo del citómetro y seleccione Ejecutar para que el citómetro comience a cargar la muestra y adquirir datos. Ajuste los eventos que se van a mostrar, los eventos que se van a registrar, el tiempo que se va a registrar y el caudal de muestra según sea necesario.
- Defina una puerta que rodee las células de levadura sanas en la gráfica SSC-A frente a FSC-A creada en el paso 4.9. La figura 1 ilustra una representación descriptiva de la estrategia de compuerta que se va a aplicar.
NOTA: En el gráfico SSC-A frente a FSC-A, 100.000 eventos para mostrar y 50.000 eventos para registrar dentro de la puerta de levadura definida es una buena guía para visualizar y recopilar datos suficientes para un análisis posterior. Se pueden aplicar estrategias de compuerta más estrictas para seleccionar la levadura singlete en función de un gráfico de altura de dispersión directa frente a área de dispersión directa, como se informó recientemente36. La estrategia de compuerta que se muestra en la Figura 1 corresponde a un enfoque menos estricto, que incluye levadura singlete y algo de levadura doblete a través de la compuerta de dispersión. - Registre la fluorescencia de todas las muestras de control utilizando un analizador de citometría de flujo. Las muestras de control se recogen primero de forma rutinaria para ayudar a definir una estrategia de compuertas, como se describe a continuación en el paso 4.14.
- Defina una estrategia de compuerta para su parcela creada en el paso 4.10 antes de analizar las muestras tratadas. La figura 1 ilustra una representación descriptiva de la estrategia de compuerta que se va a aplicar.
- Registre la fluorescencia de las muestras desfosforiladas utilizando el citómetro de flujo y la estrategia de compuerta definida en los pasos 4.12 y 4.14.
- Analice los datos de citometría de flujo adquiridos mediante un software de análisis de citometría de flujo.
- Evalúe la desfosforilación midiendo y comparando la mediana del eje Y de las células que expresan proteínas en su superficie y la fosforilación de referencia proporcionada por las células que no se muestran entre las muestras y los controles. Calcule la diferencia porcentual de fosforilación mediana de la siguiente manera:

Resultados
El análisis de citometría de flujo a partir de una réplica individual de nuestro sistema modelo incubado durante 2 h sin (Figura 1A) y con (Figura 1B) 1.000 nM SHP-2 revela una diferencia de fosforilación mediana del 63,6%, que se define como la relación entre la mediana del eje Y (fosforilación) de todos los eventos mostrados en la superficie menos la señal de fosforilación de referencia definida como la mediana del eje Y de los eventos no mostrados entre la muestra tratada y el control no tratado, como se describe en la ecuación definido en el paso 4.12 del protocolo. Antes del análisis de las medianas, las muestras se comprimían en función de su tamaño (dispersión directa) y complejidad (dispersión lateral) para abarcar un grupo sano de células. En las condiciones definidas, la desfosforilación de la muestra debe distinguirse a simple vista (Figura 1C).
El ensayo se define mediante un procedimiento sencillo que consta de cuatro métodos principales (Figura 2A). El sistema de visualización de superficie de levadura que utilizamos y hemos publicado anteriormente se basa en un plásmido que contiene un promotor bidireccional con la capacidad de expresión inducible simultánea de un par quinasa-sustrato formado por la cola citoplasmática de CD28 y la tirosina quinasa LCK (Figura 2B)29. La colocalización de las proteínas traducidas está dirigida por un péptido señal dirigido al retículo endoplásmico y la modificación postraduccional forzada por el aumento del tiempo de residencia resultante de una secuencia de retención de ER C-terminal. La secreción del sustrato fosforilado fusionado con Aga2p conduce a la expresión superficial (Figura 2C). El sustrato está diseñado para estar flanqueado por dos etiquetas de epítopos, lo que permite la confirmación extracelular de la traducción exitosa y la posterior expresión de la superficie. La incubación del sustrato que muestra células de levadura con la fosfatasa de interés (en este caso, tirosina fosfatasa SHP-2) permite analizar la modificación enzimática a través de la disminución del fosfato unido al sustrato (Figura 2D).
A pesar de que se han definido las condiciones óptimas para el sistema modelo presentado, la generalizabilidad del ensayo permite la diversificación de las proteínas a analizar. Las condiciones óptimas para el ensayo se definieron a través de una serie de titulaciones donde se evaluaron diferentes combinaciones de tiempo y concentración de fosfatasa en cuadruplicado (Figura 3). Los datos analizados demostraron significación estadística a través de un ANOVA de dos vías con replicación (p < 0,05). Las condiciones elegidas de 2 h y 1.000 nM (48,8% ± 10%) ofrecieron una diferencia de fosforilación mediana aproximada del 50%, manteniendo la significación estadística cuando se comparó con su contraparte de 750 nM a las 2 h (p < 0,05) basada en un análisis de prueba t con varianza desigual. La prueba t tampoco reveló diferencias significativas con respecto al resultado de 2 h y 1.000 nM al aumentar el tiempo en 1 h bajo ninguna de las concentraciones que proporcionaron un porcentaje aproximado de diferencia de fosforilación mediana (p > 0,05 para 500 nM, 750 nM y 1.000 nM a las 3 h).
El análisis post hoc de Tukey HSD revela que todas las comparaciones medias entre los períodos de incubación en todas las concentraciones son significativamente diferentes, aparte de 1 h a 2 h. Al comparar las múltiples concentraciones ensayadas, solo observamos diferencias de medias estadísticamente significativas cuando se comparan 250 nM con todas las demás concentraciones, lo que indica que se esperan niveles comparables de actividad de fosfatasa dentro de los grupos, excepto para 250 nM. A pesar de observar una diferencia del 20% cuando las muestras se trataron a 4 h y 1.000 nM SHP-2 (22,1% ± 5,5%), en comparación con las condiciones óptimas (t-test, p < 0,05), decidimos no seguir esta combinación debido a la reducción de la expresión superficial y la disminución de la salud de la levadura por la larga incubación con DTT. Nuestra hipótesis es que esto es causado por las condiciones reductoras del tampón de trabajo, que es necesario para la función adecuada de la fosfatasa SHP-2.

Figura 1: Análisis de citometría de flujo del sistema modelo. Gráficos de densidad que muestran la dispersión directa (eje X) frente a la dispersión lateral (eje Y) (izquierda) y diagramas de puntos que muestran la expresión de la superficie a través del etiquetado de etiquetas de epítopo C-terminal del sustrato (eje X) frente a la fosforilación del sustrato (eje Y) (centro) del dominio citoplasmático CD28 incubado durante 2 h (A) sin SHP-2 y (B) con la concentración óptima de SHP-2 definida como 1.000 nM. La mediana Y se midió dentro de las puertas definidas que abarcan las células expresadas en la superficie (verde) solo como una medida de fosforilación relativa. Los eventos de señal no visualizables se comprimieron para definir la medición de la mediana Y de fondo (gris). (C) Superposición de diagrama de puntos de muestras que muestra una diferencia de vista simple en la mediana Y. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Modificación enzimática de las proteínas mostradas en la superficie de la levadura. (A) Esquema del ensayo que muestra los pasos críticos dentro de los cuatro métodos descritos: preparación de muestras con fosfatasa (tan) para el ensayo enzimático, incubación para la actividad enzimática deseada, lavado y etiquetado celular para la detección de actividad, y análisis de citometría de flujo y recopilación de datos. (B) Esquema genético que representa una estructura general del casete utilizado para el retículo endoplásmico, el secuestro de un par enzima-sustrato y la expresión superficial del sustrato. (C) Representación gráfica de la colocalización del retículo endoplásmico de un par quinasa-sustrato (izquierda) seguida de la secreción del sustrato modificado postraduccionalmente que se muestra en la superficie de la levadura y se marca con un anticuerpo anti-fosfotirosina (azul) y un anticuerpo anti-etiqueta epítopo (rosa) para confirmar la expresión en la superficie (derecha). (D) La incubación de células de levadura con fosfatasa (tan) elimina el grupo fosfato del sustrato mostrado, interrumpiendo el marcaje de anticuerpos antifosfotirosina, lo que facilita el análisis de modificación enzimática a través de la citometría de flujo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Actividad de la fosfatasa y titulación en el tiempo. Las células de levadura se expusieron a múltiples combinaciones de tiempo y concentración de fosfatasa, seguidas de un análisis de citometría de flujo. Todas las muestras tratadas se compararon con un control incubado durante el mismo período de tiempo y condiciones de tampón sin SHP-2. La diferencia porcentual de la mediana de fosforilación se definió como la relación entre la mediana Y de los eventos mostrados en la superficie menos la señal de ruido de referencia proporcionada por la mediana Y en los eventos no visualizables en las muestras que contienen SHP-2, dividida por la misma relación en su respectivo control. La hipótesis nula se rechaza cuando se observan diferencias significativas al comparar los cambios entre los grupos de tiempo y concentración utilizando ANOVA de dos vías (p < 0,05). El efecto de las variables descritas sobre la diferencia porcentual de fosforilación mediana es independiente entre sí (Interacción p > 0.05). La prueba HSD de Tukey se realizó para un análisis post hoc para obtener más información sobre la significación de la diferencia entre el tiempo total de incubación y los grupos de concentración, y se utilizaron una serie de pruebas t que asumían una varianza desigual para definir la significación estadística de los grupos individuales en un tiempo y concentración específicos. Los datos se presentan como media ± desviación estándar de cuatro repeticiones. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Pautas de preparación de medios de crecimiento de levaduras e inducción de proteínas. Descripción tabular de la masa requerida de cada componente químico para formular 1 L de Medio de Crecimiento de Levadura Selectiva, Medio de Inducción de Proteína de Levadura Selectiva y Medio de Extracto de Levadura Peptona Dextrosa. Una vez que el medio descrito se haya mezclado correctamente, filtrar-esterilizar antes de su uso. Se incluyen instrucciones adicionales para hacer placas de medios de crecimiento de levadura selectiva. Haga clic aquí para descargar esta tabla.
Tabla 2: Preparación recomendada de la muestra para la desfosforilación del sustrato y la estrategia de marcaje de anticuerpos. Descripción tabular de las muestras necesarias para la medición de las modificaciones enzimáticas de los sustratos fosforilados que se muestran en la superficie de la levadura. La preparación de la muestra y el seguimiento de la estrategia de marcaje de anticuerpos se especifica tanto para los controles requeridos como para cada muestra que se va a analizar, incluidas las diluciones correspondientes de los reactivos de marcado. Haga clic aquí para descargar esta tabla.
Tabla 3: Cálculos de la relación entre anticuerpos y proteínas en exceso de proteínas. Descripción tabular del número teórico de anticuerpos disponibles por proteína, expresados en la superficie de una célula de levadura mientras se marcan para citometría de flujo. El número teórico expresado supone que el 100% de las células de levadura expresan 10.000 proteínas en su superficie para garantizar el exceso de anticuerpos, y los cálculos de la relación de exceso se basan en la concentración de existencias de reactivos que se muestra en la tabla y se adquieren del proveedor. Haga clic aquí para descargar esta tabla.
Tabla suplementaria S1: Secuencia de aminoácidos de los casetes de construcción. Representación tabular de la secuencia de aminoácidos para los casetes de construcción ubicados a ambos lados del promotor Gal 1-10. Las secuencias resaltadas corresponden a sus descripciones codificadas por colores. El lado Gal-10 del plásmido se representa como una traducción inversa de la secuencia de aminoácidos para facilitar su comprensión. Todos los caracteres dejados en negro corresponden a la traducción de aminoácidos de los sitios de digestión de la enzima de restricción utilizada para proporcionar modularidad a la construcción. Haga clic aquí para descargar este archivo.
Discusión
El protocolo presentado permite el análisis de las interacciones enzimáticas utilizando la visualización extracelular de proteínas en la superficie de la levadura. La incorporación del secuestro del retículo endoplásmico en el plásmido de visualización de superficie utilizado introduce la capacidad de analizar extracelularmente interacciones específicas entre enzimas y sustratos modificados postraduccionalmente debido a las interacciones intracelulares que se pueden diseñar para que ocurran27,29. Los ensayos de interacción enzimática previamente establecidos que utilizan la visualización de la superficie de la levadura implican la expresión de las proteínas de interés por vía intracelular, siendo la visualización de la superficie únicamente una herramienta para la detección de las interacciones intracelulares que ocurren entre las proteínas diana 23,25,29.
Este protocolo se basa en esa plataforma al trasladar las interacciones enzimáticas específicas al entorno extracelular, lo que introduce flexibilidad adicional tanto en las enzimas que se pueden estudiar como en el entorno en el que se monitorea su actividad. Las interacciones estudiadas que ocurren extracelularmente brindan a los investigadores la oportunidad de adaptar el entorno de incubación para que sea más óptimo para la actividad enzimática, ampliando las enzimas que podrían estudiarse cuyas actividades se ven obstaculizadas en el retículo endoplásmico de la levadura, que es un entorno altamente oxidante37. Además, la capacidad de valorar la concentración de enzimas con respecto a un sustrato determinado permite realizar ensayos específicos de actividad enzimática que no se podrían realizar intracelularmente debido a la hipotética saturación de la velocidad de reacción durante el secuestro.
Dentro del protocolo, hay varios pasos críticos que deben tenerse en cuenta para garantizar que se observe el resultado deseado. La transformación exitosa de los constructos estudiados en levadura es esencial para obtener resultados óptimos a partir de los siguientes pasos. Se debe realizar un monitoreo preciso de la densidad óptica para rastrear el crecimiento saludable de los cultivos y garantizar que no crezcan demasiado antes de la inducción de proteínas o la preparación de la muestra. La fase logarítmica del crecimiento de las células de levadura abarca el período en el que la producción de proteínas es más alta, mientras que en la fase estacionaria, los mecanismos responsables de la producción de proteínas se detienen38. Teniendo esto en cuenta, las mediciones de densidad óptica proporcionan una medición precisa de las fases de crecimiento en las que se encuentran los cultivos de levadura, y pasos como la inducción de proteínas y la preparación para los ensayos deben realizarse fuera de la fase estacionaria o cuando los cultivos crecen demasiado (OD600 nm < 6).
Para el ensayo de modificación enzimática, el ambiente de incubación descrito fue específico para la enzima estudiada, SHP-2 y la actividad enzimática que se estaba realizando, la desfosforilación. Se utilizó DTT por el ambiente reductor que proporciona durante la incubación con SHP-234. Por lo tanto, es importante medir las concentraciones precisas de los productos químicos utilizados para modificar el entorno de incubación en los ensayos enzimáticos para garantizar una actividad enzimática constante entre las muestras y los experimentos. La SHP-2 se utilizó como proteína recombinante, y es fundamental regular la temperatura durante las diferentes etapas de la manipulación de la enzima. Para un ensayo exitoso, la enzima no debe haber pasado por más de dos ciclos de congelación-descongelación y debe estar en hielo durante la preparación de cada muestra. A continuación, es imperativo alícuota de la enzima recombinante en un volumen suficiente para satisfacer los requisitos del ensayo. Durante la incubación real, la temperatura debe controlarse estrictamente a la temperatura óptima para la enzima, 37 °C en este caso, con un movimiento constante de un rotor para garantizar la homogeneidad de la mezcla de incubación.
El método general para el análisis con enzimas recombinantes requirió modificaciones específicas para la interacción entre SHP-2 y el sustrato fosforilado mostrado en la superficie. La adaptación del protocolo a otras interacciones enzima-sustrato extracelular implica la modificación de las secuencias utilizadas, el entorno del tampón de actividad y el reactivo utilizado para la detección. Para ensayar otras interacciones quinasa-sustrato-fosfatasa, la adaptación implica la sustitución de las secuencias de proteínas de un par quinasa-sustrato en sus respectivas posiciones en el casete de construcción descrito en la Tabla Suplementaria S1. La secuencia proteica del sustrato, junto con al menos el dominio quinasa de la quinasa de interés, debe incluirse en el plásmido, y la fosfatasa dirigida al sustrato fosforilado producido debe tener la forma de una proteína recombinante. La interacción representativa entre LCK, CD28 y SHP-2 proporciona un ejemplo del uso del secuestro del retículo endoplásmico diseñado en el casete de construcción como herramienta para producir proteínas modificadas postraduccionalmente para ser investigadas extracelularmente con su enzima dirigida. Los sustratos de interés que no necesitan pasar por modificaciones postraduccionales (por ejemplo, sustratos que podrían fosforilarse extracelularmente utilizando quinasa añadida) se pueden expresar en la superficie de la levadura sin una enzima emparejada dentro del casete de construcción. En este caso, la secuencia de proteínas para la quinasa descrita en la Tabla Suplementaria S1 se eliminaría y solo se incluiría la secuencia del sustrato en el casete de construcción. Observamos a partir de nuestra experiencia previa que la colocalización de una serina-treonina quinasa con un sustrato conocido resultó en la exhibición de un sustrato que no estaba fosforilado (Ezagui y Stern, datos no publicados), por lo que se deben realizar pruebas rigurosas de modificación enzimática exitosa antes de aplicar fosfatasa extracelular. Hemos publicado previamente un protocolo para la colocalización de quinasa-sustrato que puede ser útil para este paso de calificación39.
Las quinasas y fosfatasas a menudo contienen residuos de cisteína desapareados que, cuando se oxidan, forman enlaces disulfuro dentro de la proteína o a través de proteínas, lo que puede interrumpir la actividad catalítica de la proteína debido al cambio conformacional40,41. La comprensión de la bioquímica de esta proteína es esencial para determinar el entorno de reacción adecuado para la modificación enzimática. Como resultado, es necesario complementar un agente reductor en el entorno de incubación para garantizar que la proteína recombinante utilizada permanezca activa. El DTT es un agente reductor común utilizado para estos fines, pero la concentración en el ensayo debe optimizarse. El uso de una concentración demasiado alta de DTT dificulta la visualización del sustrato en la superficie de la levadura, ya que los anclajes de Aga1p y Aga2p se mantienen unidos entre sí a través de enlaces disulfuro, que se reducen en presencia de DTT42. La concentración de DTT se ajustó a la concentración mínima que permitiera una actividad de fosfatasa máxima relativa sin tener efectos perjudiciales sobre la visualización superficial de los sustratos42. El entorno de incubación de la enzima que se esté ensayando debe optimizarse para garantizar la retención de la actividad enzimática cuando se utilice en este ensayo. Si una enzima requiere un entorno reductor considerablemente más fuerte que el DTT de 0,5 mM utilizado en este ensayo, la plataforma está limitada por la reducción en la visualización de la superficie y puede no ser óptima para el ensayo enzimático específico deseado. De manera similar, el tampón 2x utilizado durante la etapa de incubación en este protocolo se incluyó a partir de investigaciones previas sobre tampones aceptables que promueven la actividad de SHP-2, y se deben realizar investigaciones similares para formular un tampón de incubación para cualquier otra enzima que se utilice para este ensayo34. Los puntos de partida para la elaboración de estos tampones pueden incluir la búsqueda bibliográfica de aplicaciones in vitro exitosas de la enzima de interés o la recomendación del fabricante de la enzima para un tampón de actividad. La enzima recombinante que se utiliza debe valorarse específicamente para este ensayo a fin de identificar una concentración y un tiempo de incubación aceptables, lo que permite que se produzca la actividad enzimática específica antes de la recopilación de datos.
Para la adaptación de este protocolo a otros tipos de interacción enzima-sustrato, será necesario seleccionar nuevos reactivos de detección fluorescentes y valorarlos para determinar su sensibilidad. Otros estudios han demostrado ejemplos de esto, incluyendo el uso de anticuerpos dirigidos a epítopos para detectar la presencia o ausencia de sustratos peptídicos después del tratamiento con proteasas23,25 y anticuerpos sensibles a la acetilación para detectar modificaciones en las proteínas histonas43. Para calificar estos reactivos, se debe establecer un control positivo (uno que demuestre verificablemente la modificación del interés) y un control negativo (uno que demuestre verificablemente la falta de la modificación del interés). Esto podría hacerse a través de la visualización de la superficie de la levadura de un constructo que haya demostrado previamente la modificación de interés, o en algunos casos podría establecerse mediante la inmovilización de proteínas o péptidos recombinantes. Por ejemplo, en el caso de la fosforilación (y muchas otras modificaciones postraduccionales de interés), los péptidos de secuencia conocida podrían sintetizarse con fosfotirosina (control positivo) o tirosina no modificada (control negativo) y una biotina C-terminal que permitiría la inmovilización de los péptidos en perlas recubiertas de estreptavidina. Las perlas recubiertas de péptidos podrían marcarse con el anticuerpo específico de modificación y evaluarse para determinar la especificidad y sensibilidad de la detección mediante citometría de flujo con métodos similares a los descritos en la sección 4. Se deben utilizar diferentes diluciones de anticuerpo para encontrar una concentración que proporcione la máxima señal del control positivo y la mínima señal del control negativo, y que equilibre un exceso de pliegue suficiente de anticuerpo por proteína modificada (utilizando la ecuación de la nota del paso 4.1) con consideraciones económicas para el número de experimentos que se llevarán a cabo por alícuota de anticuerpo obtenido.
Describimos un protocolo para adaptar la facilidad de la plataforma de visualización de la superficie de la levadura para ensayos de actividad enzimática extracelular. El método se demuestra utilizando CD28 fosforilado que se muestra en la superficie de la levadura para ser desfosforilado por SHP-2 recombinante durante la incubación, pero es generalizable para muchos tipos de modificación enzimática a través de la modificación del tampón de trabajo y el par enzima-sustrato utilizado.
Divulgaciones
Los autores no tienen conflictos de intereses relacionados con este trabajo que divulgar.
Agradecimientos
Este trabajo contó con el apoyo de un premio NSF CAREER a L.A.S. (CBET - 2339172) y fondos de inicio de la Universidad del Sur de Florida.
En la Figura 2A, el icono de microtubo abierto translúcido de Servier https://smart.servier.com/ está licenciado bajo CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/. Las modificaciones incluyen la adición de un tampón y una célula de levadura (izquierda) y la adición de anticuerpos (centro-derecha).
El tubo de ensayo, la incubadora y el citómetro de flujo de la Figura 2A se proporcionaron a través de www.bioicons.com de acceso abierto.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
Referencias
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados