Method Article
Modificação enzimática e avaliação por citometria de fluxo de proteínas exibidas na superfície de leveduras
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para o uso de substratos exibidos na superfície da levedura para ensaios de modificação enzimática. A plataforma foi demonstrada usando a análise da atividade de desfosforilação da tirosina fosfatase SHP-2 contra um de seus substratos como um ensaio de modificação enzimática representativa.
Resumo
A exibição da superfície da levedura é uma estratégia de ligação genótipo-fenótipo que permite a triagem de alto rendimento da função da proteína. Tradicionalmente, a exibição da superfície da levedura tem sido aplicada à evolução de novas proteínas de ligação, com citometria de fluxo usada para avaliar e classificar por níveis de força de ligação. Recentemente, tem havido um interesse crescente na aplicação da exibição da superfície da levedura para triagem de modificação enzimática de variantes de substrato, com modificações aditivas (por exemplo, fosforilação) ou subtrativas (por exemplo, proteólise) fornecendo um fenótipo legível por citometria de fluxo. Tais modificações são aplicadas regularmente usando co-localização intracelular, mas a capacidade de alcançar a modificação enzimática extracelular dos substratos exibidos pode abrir muito mais reações à investigação. Aqui, descrevemos técnicas para projetar e aplicar ensaios de triagem para modificação enzimática extracelular em substratos candidatos exibidos na superfície da levedura e avaliação subsequente usando análise de citometria de fluxo. Fornecemos esses protocolos no contexto de fosfatases de levedura desfosforilante exibindo substratos contendo resíduos de tirosina fosforilada e comentamos como essa estrutura aplicada pode ser adaptada ao desenvolvimento de ensaios de triagem para outros pares enzima-substrato.
Introdução
A compreensão das interações entre enzimas e seus alvos tem se tornado uma área de pesquisa cada vez mais interessante devido à sua necessidade na caracterização biológica das vias que controlam a homeostase celular e o desenvolvimento da doença 1,2. As enzimas são responsáveis pela catálise de muitas das reações que mantêm a vida biológica, controlando vias necessárias, como o metabolismo celular 3,4, a sinalização5 e até processos fundamentais como o reparo do genoma 6,7. Devido ao seu papel nesses processos, suas interações também desempenham um papel no desenvolvimento de muitas doenças, pois desvios em sua atividade podem causar desregulação severa na atividade celular, causando apoptose ou proliferação de células cancerígenas nocivas2. O estudo da atividade enzimática tem tido aplicações importantes no desenvolvimento de novas terapêuticas 8,9, exigindo ensaios adaptados a cada interação enzima-substrato específico10. Múltiplos ensaios enzimáticos foram estabelecidos como protocolos padrão para a avaliação e caracterização dessas interações. Os ensaios desenvolvidos para analisar as interações enzimáticas são classificados em ensaios de detecção que monitoram a ligação para ativação/inibição11 ou ensaios que monitoram a modificação do substrato por enzimas12.
Um dos principais papéis das enzimas é regular o comportamento celular. A transdução de sinal, a resposta intracelular de uma célula a um gatilho extracelular13, é responsável pela sobrevivência e funcionalidade celular. A proliferação, diferenciação e muitos outros processos funcionais envolvem vias de sinalização com interações enzimáticas que as governam14,15. As enzimas catalisam modificações pós-traducionais, que muitas vezes modulam as redes de sinalização massivas responsáveis pela transmissão correta de mensagens extracelulares16. A fosforilação de proteínas é a modificação pós-traducional mais comum, onipresente na sinalização celular e em várias outras vias celulares. Consequentemente, as proteínas quinases surgiram como uma proporção significativa de potenciais alvos terapêuticos devido ao seu papel regulatório crítico17. As fosfatases são as moléculas modulatórias naturais para complexos de sinalização celular à base de fosfato18,19, tendo a capacidade de remover resíduos de fosfato de suas proteínas-alvo20. Na última década, as fosfatases tornaram-se um importante alvo terapêutico para o tratamento do câncer21 e doenças inflamatórias22 com base em seu envolvimento na regulação das vias de sinalização a jusante em vários tipos de células. Juntas, as proteínas quinases e fosfatases fornecem uma amplitude de interações, que podem ser estudadas por meio do desenvolvimento de protocolos de ensaio enzimático específicos.
A exibição da superfície da levedura tem sido utilizada como ferramenta para a caracterização e avaliação da atividade enzimática 23,24. Ele fornece uma plataforma de alto rendimento para a triagem de processos de modificação pós-traducional quando combinado com estratégias de sequestro de retículo endoplasmático25,26. Isso permite que os pares quinase-substrato sejam co-localizados e retidos no retículo endoplasmático por meio da ligação aos receptores KDEL27, onde a fosforilação do substrato pode ocorrer em taxas aumentadas devido à proximidade entre as quinases e seus alvos. A ligação ao receptor KDEL é mediada por uma sequência de retenção do retículo endoplasmático FEHDEL C-terminal que demonstrou ter uma capacidade de retenção mais forte do que outras sequências HDEL25,28. O substrato fosforilado é então ancorado à superfície da levedura para sua posterior avaliação por citometria de fluxo29. Atualmente, não existem protocolos generalizáveis estabelecidos para a modificação enzimática de substratos dispostos na superfície da levedura. Expandimos as capacidades de exibição da superfície da levedura aproveitando as variantes de substrato fosforilada expressas extracelularmente e modificando-as por meio da desfosforilação por sua fosfatase conhecida. A análise por citometria de fluxo fornece então uma plataforma para a avaliação fenotípica dos substratos acima mencionados através da medição de alterações na mediana da fosforilação como consequência da incubação com a fosfatase conhecida. Isso fornece um método adaptável para modificação pós-traducional de proteínas exibidas na superfície, ao mesmo tempo em que fornece um método para análise de modificação enzimática de interações ao usar a plataforma de exibição de superfície de levedura.
Apresentamos técnicas para o desenvolvimento e aplicação de um ensaio de modificação enzimática que descreve a introdução de uma interação quinase-substrato na plataforma de exibição da superfície da levedura, a co-incubação do substrato fosforilado expresso com uma fosfatase recombinante e a subsequente análise da atividade de desfosforilação por citometria de fluxo. Neste relatório, isso é feito co-localizando o domínio citoplasmático de CD28 com linfócitos quinase (LCK) no retículo endoplasmático de levedura, seguido pela exibição do CD28 fosforilado na superfície da levedura e subsequente desfosforilação pela fosfatase-2 contendo domínio da região de homologia Src 2 (SHP-2). Um anticorpo pan-antifosfotirosina (neste estudo, 4G10), que detecta resíduos de tirosina fosforilada em uma ampla variedade de sequências peptídicas, é usado para quantificação do nível de fosforilação em função do tratamento com fosfatase. O processo detalhado fornece uma abordagem generalizável para investigar as interações enzima-substrato; uma maneira prospectiva de estudar enzimas e substratos de forma purificada.
Protocolo
1. Crescimento celular de leveduras que abrigam plasmídeo e indução de expressão proteica
- Seguindo a receita descrita na Tabela 1, prepare o meio necessário para o crescimento de leveduras não contendo plasmídeo (YPD), crescimento de células de levedura contendo plasmídeo (SD-CAA) e indução da expressão de proteínas (SRG-CAA), bem como placas SD-CAA.
- Transforme o DNA do plasmídeo de exibição de levedura contendo o par quinase-substrato em células de levedura EBY-100 por meio do método baseado em cátion de lítio30,31, comumente adotado para uso em kits de transformação de levedura de vários fabricantes.
NOTA: A eletroporação32 ou outras técnicas preferidas de transformação de plasmídeo de levedura podem ser usadas dependendo da construção do plasmídeo que está sendo transformada. - Prepare um tubo de cultura de 14 mL com 10 mL de meio YPD. Inocular as células EBY-100 e crescer numa incubadora agitada a 30 °C, 300 rpm até que a cultura atinja uma densidade ótica (OD600nm) de 0,8-1,0 (8 x 106- 1 x 107 células/ml).
NOTA: OD600nm é medido preparando cubetas de amostra de 3 mL contendo diluições 1:10 de culturas de levedura em seus respectivos meios e cubetas em branco de 3 mL contendo o meio usado para diluições de amostra. O programa OD600nm no espectrofotômetro é usado para medir primeiro a cubeta em branco e, em seguida, cada cubeta de amostra, definindo a diluição correspondente preparada para cada amostra. 1 OD600nm corresponde a 1 × 107 levedura/mL. - Colha as células centrifugando a cultura a 1.000 x g por 3 min e lave com a1ª solução de lavagem fornecida no kit de transformação de levedura, ou TE (10 mM Tris-HCl e 1,0 mM EDTA) 31.
- Pellet as células novamente a 1.000 x g por 3 min e ressuspenda em 1 mL do tampão de transformação fornecido no kit de transformação de levedura ou água estéril. As células devem ser alíquotas em alíquotas de 50 μL e podem ser armazenadas a -80 °C por até 6 meses.
- Para a transformação do plasmídeo, uma alíquota por plasmídeo é preparada e descongelada em gelo e, em seguida, 0,5-1,5 μg de DNA de plasmídeo contendo a construção de exibição de levedura é adicionada diretamente às células. Adiciona-se 0,5 mL da solução de transformação fornecida no kit de transformação de levedura, ou 0,5 mL de uma solução estéril de polietilenoglicol a 50% e 0,1 M LiOAc31. Combine a mistura de células, DNA de plasmídeo e solução de transformação completamente por pipetagem.
- Incubar a mistura de transformação estaticamente por 30-60 min a 30 ° C, mistura de vórtice em intervalos de 15 min. Colha as células centrifugando a 1.000 × g por 3 min.
- Prepare um tubo de cultura de 14 mL com 4,5 mL de meio SD-CAA. Ressuspenda as células contendo o plasmídeo desejado em 500 μL de SD-CAA e inocule os 4,5 mL preparados.
- Tomando cuidado para não perfurar o ágar, distribuir 50 μL dos 5 ml de cultura inoculada em uma placa de SD-CAA e incubar estaticamente a 30 °C por 48 h para determinar a eficiência da transformação.
- Incubar os 5 ml de cultura de células SD-CAA numa incubadora agitada a 30 °C, 300 rpm durante pelo menos 18 h. Monitore a densidade óptica (OD600nm) após 16 h e 20 h. Quando a amostra atingir uma densidade óptica suficiente não superior a 6, centrifugue a cultura por 3 min a 2.500 × g. Descarte o sobrenadante sem mexer no pellet de fermento.
- Ressuspenda o pellet de levedura em SRG-CAA até um OD finalde 600 nm menor que 1 (<1 × 107 levedura/mL).
- Incubar a cultura de levedura numa estufa agitada a 30 °C, 300 rpm durante, pelo menos, 8 h mas não mais de 24 h.
NOTA: A indução da expressão proteica em células de levedura pode variar entre 20-37 °C. 30 °C é adequado para a síntese de pares quinase/substrato29,33, mas pode ser ajustado se considerado necessário para as proteínas específicas em estudo. - Meça o OD600nm para determinar a densidade celular.
NOTA: O protocolo pode ser interrompido neste ponto armazenando as culturas de levedura a 4 °C.
2. Biotinilação do anticorpo 4G10 antifosfotirosina
- Ressuspenda um frasco de 2 mg de PEG4-NHS-Biotina até uma concentração final de 5 mM, adicionando 680 μL de PBS estéril.
NOTA: A ressuspensão de PEG4-NHS-Biotina deve ser feita imediatamente antes da reação de biotinilação ser realizada. NHS hidrolisa em solução aquosa. O uso de PBS estéril e frascos para injetáveis é importante para a preparação de reagentes para armazenamento de longo prazo e para uso em ensaios baseados em células para mitigar quaisquer efeitos potenciais de contaminantes na viabilidade do reagente ou nos ensaios sensíveis que estão sendo realizados. - Com base na concentração de anticorpos 4G10, adicione 100 μg de anticorpo em um frasco estéril de 1,7 mL.
- Adicione 1 μL do 5 mM PEG4-NHS-Biotina preparado da etapa 2.1 ao frasco contendo os 100 μg de anticorpo 4G10 para atingir uma proporção molar de biotina para anticorpo de 7,5:1. Pipete a mistura suavemente para homogeneizar a reação.
- Incubar a reação à temperatura ambiente com rotação constante durante pelo menos 2 h.
- Siga o protocolo do fabricante para colunas de dessalinização por centrifugação de 0,5 mL para trocar o tampão do 4G10 biotinilado (B-4G10) por PBS.
NOTA: As colunas de dessalinização com um corte de peso molecular (MWCO) de 7 kDa são comumente usadas para biotinilação de anticorpos para permitir que a biotina não reagida e outras moléculas pequenas sejam removidas, mantendo o anticorpo maior. - Diluir o anticorpo B-4G10 até uma concentração final de 1 μM em PBSA (PBS com 1 g/L de albumina sérica bovina). Aliquota o anticorpo B-4G10 em volumes menores para evitar ciclos repetidos de congelamento/descongelamento.
NOTA: Os anticorpos biotinilados podem ser armazenados a 4 °C para uso diário por até 3 meses sem perder eficiência significativa. Conservar as restantes alíquotas que não sejam utilizadas a -20 °C durante um período máximo de 2 anos.
3. Desfosforilação de substratos expressos na superfície da célula de levedura
- Prepare a solução tampão de trabalho 2x conforme descrito anteriormente na literatura34.
NOTA: 2x é recomendado para facilitar a medição dos ingredientes necessários. - Prepare o tampão de trabalho para as amostras em um frasco de 1,7 mL diluindo a solução tampão 2x preparada na etapa 3.1 1:2 em água deionizada.
NOTA: O volume total de reação para cada amostra será de 20 μL, e cada amostra exigirá entre 10 μL e 18 μL de tampão de trabalho. Prepare tampão de trabalho suficiente para todas as amostras ou controles. - Após a preparação recomendada da amostra descrita na Tabela 2, rotule os frascos de 1,7 mL com o controle correspondente ou nome da amostra.
- Com base no OD600nm medido na etapa 1.9, calcule o volume de cultura necessário para recuperar dois milhões (2 × 106) de células de levedura da cultura de levedura correspondente para cada amostra.
- Adicione o volume de cultura de levedura calculado na etapa anterior no frasco de 1,7 mL para cada amostra.
- Centrifugue o frasco para injetáveis durante 1 min a 4.500 × g. Remova cuidadosamente o sobrenadante usando uma micropipeta e descarte como resíduo de risco biológico.
- Ressuspenda as células peletizadas em 1 mL de PBSA e repita a etapa 3.6.
NOTA: É importante remover o máximo de sobrenadante possível sem perturbar as células peletizadas. - Com base em sua concentração de estoque, calcule o volume de SHP-2 humano recombinante necessário para ter uma concentração final de 1.000 nM em um volume total de reação de 20 μL.
NOTA: O SHP-2 recombinante deve ser aliquotado em pequenos volumes para que qualquer proteína usada em cada ensaio não tenha passado por mais de dois ciclos de congelamento e descongelamento. Qualquer SHP-2 que sobrar de uma alíquota depois de todas as amostras terem sido preparadas para um ensaio deve ser eliminada.
As concentrações de estoque de enzimas recombinantes podem variar dependendo do número do lote. O SHP-2 recombinante é comumente formulado em uma concentração de estoque de 0,2-0,4 mg/mL. Para uma concentração estoque de 0,324 mg / mL SHP-2, isso corresponde a uma concentração estoque de 4,696 μM (SHP-2 tem um peso molecular de 69 kDa). 4,26 μL do estoque de SHP-2 em uma reação de 20 μL resulta em uma concentração final de reação de 1.000 nM SHP-2. - Adicione 7,7 mg de DTT em 10 mL de água deionizada preparada em um cônico de 15 mL para criar uma solução de DTT de 5 mM. Se as limitações do equipamento dificultarem a pesagem de miligramas, adicione 0,77 g de DTT a 10 mL de água deionizada e, em seguida, execute uma diluição de 100x para criar a solução de DTT de 5 mM usada para o ensaio.
NOTA: A solução de TDT pode ser preparada em soluções de estoque de concentração mais alta para serem diluídas a 5 mM se não for possível medir quantidades em escala de miligramas usando o equipamento disponível. A solução de TDT precisa ser preparada fresca antes da ressuspensão celular em tampão de trabalho devido à sua tendência à hidrólise, tornando-a instável por longos períodos quando diluída em água. - Ressuspender as células peletizadas no tampão de trabalho preparado na etapa 3.2 de modo que o volume final de reação em cada amostra ou controle seja de 20 μL.
NOTA: A quantidade de tampão de trabalho adicionada deve ser calculada com base na quantidade de DTT (2 μL) e SHP-2 (calculada na etapa 3.8) que estará em cada amostra. - Adicionar 2 μL da solução de DTT de 5 mM preparada na etapa 3.9 a cada amostra ou controle para uma concentração de reação final de 0,5 mM de DTT.
- Adicione o volume de SHP-2 calculado na etapa 3.8 a cada amostra para um volume final de 20 μL e misture suavemente usando uma micropipeta.
- Envolva as tampas dos frascos de amostra em parafilme para evitar vazamento ou contaminação cruzada.
- Incubar as amostras a 37 °C durante 2 h num rotor a uma velocidade constante.
- Remova as amostras do rotor e interrompa a reação adicionando 1 mL de PBSA a cada amostra.
- Repita a etapa 3.6.
4. Marcação celular e análise por citometria de fluxo de substratos desfosforilados
- Ressuspenda as amostras da etapa 3.16 em uma mistura de 20 μL de seus reagentes primários correspondentes, conforme descrito na Tabela 2. Incube as amostras por 20 min em temperatura ambiente.
NOTA: Todas as concentrações de reagentes utilizadas foram calculadas como excedentes em relação ao número de proteínas expressas na superfície das células de levedura. O cálculo pressupõe a expressão de 10.000 proteínas/célula de todas as leveduras 2 x 106 em uma amostra35, quando rotineiramente apenas ~ 50% delas o fazem. A Tabela 3 mostra a taxa de reagentes de marcação em excesso para cada um dos reagentes expressa na Tabela 2, calculada da seguinte forma:
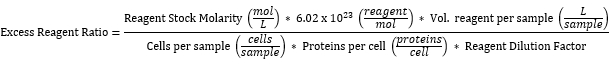
- Centrifugue amostras a 4.500 × g por 1 min e descarte o sobrenadante como resíduo de risco biológico.
- Lave as células uma vez, ressuspendendo em 1 mL de PBSA. Repita a etapa 4.2.
- Ressuspender as amostras numa mistura de 20 μL dos seus reagentes secundários correspondentes, conforme descrito no quadro 2. Incubar amostras por 15 min na ausência de luz.
- Repita a etapa 4.2.
- Repita a etapa 4.3.
- Ressuspenda as amostras lavadas em 300-500 μL de PBSA e transfira para tubos de poliestireno de 5 mL para análise imediata usando um citômetro de fluxo apropriado.
NOTA: Se for necessário transportar amostras, mantenha em gelo úmido. Não é recomendado, mas as amostras podem ser armazenadas a 4 °C por no máximo 2 h como pellets úmidos. - Depois de executar a inicialização e a preparação necessárias do citômetro para um novo experimento, clique no botão Novo experimento no menu Arquivo , nomeie o experimento e clique em Salvar para garantir que os dados adquiridos sejam salvos no caminho de arquivo desejado.
- Selecione o ícone de gráfico de pontos na barra de ferramentas superior para criar dois ou mais gráficos de pontos para cada exemplo a ser executado. Para um dos gráficos de pontos, selecione o nome do eixo X para exibir o canal FSC-A e o nome do eixo Y para exibir o canal SSC-A. Este gráfico mostra o Side Scatter - Área versus Forward Scatter - Area e é usado para bloquear células de levedura para análise posterior.
- Em outro gráfico de pontos, selecione o nome do eixo X para exibir o canal no qual o reagente secundário direcionado ao anticorpo anti-epítopo primário fluoresce. Selecione o nome do eixo Y para exibir o canal no qual o reagente secundário da estreptavidina fluoresce. Este gráfico mostrará apenas os eventos fechados do lado versus o gráfico de dispersão direta como células de levedura e é usado para exibir a fosforilação da tirosina no eixo Y e a expressão da superfície do substrato no eixo X.
NOTA: O reagente secundário direcionado ao anticorpo anti-epítopo primário fluoresce no canal FITC (AF-488) e o reagente secundário estreptavidina fluoresce no canal AF-647 neste exemplo. Os canais usados podem variar dependendo dos reagentes primários e secundários usados durante a rotulagem. - Coloque cada sample tubo no suporte do tubo do citômetro e selecione Executar para o citômetro para começar a carregar a amostra e adquirir dados. Ajuste os eventos a serem exibidos, os eventos a serem registrados, o tempo a ser registrado e a taxa de fluxo de amostra, conforme necessário.
- Defina uma porta ao redor das células de levedura saudáveis no gráfico SSC-A vs FSC-A criado na etapa 4.9. A Figura 1 ilustra uma representação descritiva da estratégia de gating a ser aplicada.
NOTA: No gráfico SSC-A versus FSC-A, 100.000 eventos para exibir e 50.000 eventos para registrar dentro da porta de levedura definida é uma boa diretriz para visualizar e coletar dados suficientes para análise posterior. Estratégias de gating mais rigorosas para selecionar levedura singlete podem ser aplicadas com base em um gráfico de Forward Scatter-Height versus Forward Scatter-Area, conforme relatado recentemente36. A estratégia de gating mostrada na Figura 1 corresponde a uma abordagem menos rigorosa, incluindo singleto e algumas leveduras duplas por meio de gating de dispersão. - Registre a fluorescência de todas as amostras de controle usando um analisador de citometria de fluxo. As amostras de controle são coletadas rotineiramente primeiro para ajudar a definir uma estratégia de gating, conforme descrito abaixo na etapa 4.14.
- Defina uma estratégia de gating para seu gráfico criado na etapa 4.10 antes de analisar as amostras tratadas. A Figura 1 ilustra uma representação descritiva da estratégia de gating a ser aplicada.
- Registar a fluorescência das amostras desfosforiladas utilizando o citómetro de fluxo e a estratégia de passagem definida nos passos 4.12 e 4.14.
- Analise os dados de citometria de fluxo adquiridos usando um software de análise de citometria de fluxo.
- Avalie a desfosforilação medindo e comparando a mediana do eixo Y das células que expressam proteínas em sua superfície e a fosforilação basal fornecida por células não exibidoras entre amostras e controles. Calcule a diferença de fosforilação mediana percentual da seguinte forma:

Resultados
A análise de citometria de fluxo de uma réplica individual de nosso sistema modelo incubado por 2 h sem (Figura 1A) e com (Figura 1B) 1.000 nM SHP-2 revela uma diferença mediana de fosforilação de 63,6%, que é definida como a razão da mediana do eixo Y (fosforilação) de todos os eventos exibidos na superfície menos o sinal de fosforilação da linha de base definido como a mediana do eixo Y dos eventos não exibidos entre a amostra tratada e o controle não tratado, conforme descrito na equação definido na etapa 4.12 do protocolo. Antes da análise das medianas, as amostras foram fechadas com base em seu tamanho (Forward Scatter) e complexidade (Side Scatter) para abranger um grupo saudável de células. Nas condições definidas, a desfosforilação da amostra deve ser distinguível à vista de todos (Figura 1C).
O ensaio é definido por um procedimento simples que consiste em quatro métodos principais (Figura 2A). O sistema de exibição da superfície da levedura que usamos e publicamos anteriormente é baseado em um plasmídeo contendo um promotor bidirecional com capacidade de expressão induzível simultânea de um par quinase-substrato consistindo na cauda citoplasmática de CD28 e na tirosina quinase LCK (Figura 2B) 29 . A co-localização das proteínas traduzidas é dirigida por um retículo endoplasmático direcionado ao peptídeo sinal e a modificação pós-traducional forçada pelo aumento do tempo de residência resultante de uma sequência de retenção de ER C-terminal. A secreção do substrato fosforilado fundido a Aga2p leva à expressão superficial ( Figura 2C ). O substrato é projetado para ser flanqueado por duas etiquetas de epítopos, o que permite a confirmação extracelular da tradução bem-sucedida e subsequente expressão de superfície. A incubação do substrato exibindo células de levedura com a fosfatase de interesse (neste caso, tirosina fosfatase SHP-2) permite a análise da modificação enzimática através da diminuição do fosfato ligado ao substrato (Figura 2D).
Embora as condições ótimas tenham sido definidas para o sistema modelo apresentado, a generalização do ensaio permite a diversificação das proteínas a serem analisadas. As condições ótimas para o ensaio foram definidas por meio de uma série de titulações onde diferentes combinações de tempo e concentração de fosfatase foram avaliadas em quadruplicata (Figura 3). Os dados analisados demonstraram significância estatística por meio de uma ANOVA de duas vias com replicação (p < 0,05). As condições escolhidas de 2 h e 1.000 nM (48,8% ± 10%) ofereceram uma diferença mediana de fosforilação aproximada de 50%, mantendo significância estatística quando comparadas à sua contraparte de 750 nM em 2 h (p < 0,05) com base em uma análise de teste t com variância desigual. O teste t também não revelou diferença significativa em relação ao resultado de 2 h e 1.000 nM, aumentando o tempo em 1 h sob qualquer uma das concentrações que forneceram uma porcentagem de diferença de fosforilação mediana aproximada (p > 0,05 para 500 nM, 750 nM e 1.000 nM em 3 h).
A análise post hoc de Tukey HSD revela que todas as comparações médias entre os períodos de incubação em todas as concentrações são significativamente diferentes, exceto de 1 h a 2 h. Ao comparar as múltiplas concentrações testadas, observamos apenas diferenças médias estatisticamente significativas ao comparar 250 nM com todas as outras concentrações, indicando que níveis comparáveis de atividade da fosfatase são esperados dentro dos grupos, exceto para 250 nM. Apesar de observar uma diferença de 20% quando as amostras foram tratadas em 4 h e 1.000 nM SHP-2 (22,1% ± 5,5%), em comparação com as condições ótimas (teste t, p < 0,05), decidimos não buscar essa combinação devido à redução da expressão superficial e diminuição da saúde da levedura devido à longa incubação com DTT. Nossa hipótese é que isso é causado pelas condições de redução do tampão de trabalho, que é necessário para a função adequada da fosfatase SHP-2.

Figura 1: Análise de citometria de fluxo do sistema modelo. Gráficos de densidade exibindo Forward Scatter (eixo X) versus Side Scatter gating (eixo Y) (esquerda) e gráficos de pontos exibindo expressão de superfície por meio de marcação de tag de epítopo C-terminal do substrato (eixo X) versus fosforilação do substrato (eixo Y) (centro) do domínio citoplasmático CD28 incubado por 2 h (A) sem SHP-2 e (B) com a concentração ideal de SHP-2 definida como 1.000 nM. A mediana Y foi medida dentro das portas definidas abrangendo células expressas na superfície (verde) apenas como uma medição de fosforilação relativa. Eventos de sinal não exibidos foram fechados para definir a medição Y-Mediana de fundo (cinza). (C) Sobreposição de gráfico de pontos de amostras mostrando diferença à vista na mediana Y. Clique aqui para ver uma versão maior desta figura.

Figura 2: Modificação enzimática da superfície da levedura exibindo proteínas. (A) Esquema do ensaio mostrando as etapas críticas dentro dos quatro métodos descritos: preparação de amostras com fosfatase (tan) para ensaio enzimático, incubação para atividade enzimática desejada, lavagem e marcação celular para detecção de atividade e análise de citometria de fluxo e coleta de dados. (B) Esquema genético que representa uma estrutura geral do usado para sequestro do retículo endoplasmático de um par enzima-substrato e expressão superficial do substrato. (C) Representação gráfica da co-localização do retículo endoplasmático de um par quinase-substrato (esquerda) seguida pela secreção do substrato pós-traduzilmente modificado exibido na superfície da levedura e marcado com um anticorpo anti-fosfotirosina (azul) e um anticorpo anti-epítopo (rosa) para confirmar a expressão da superfície (direita). (D) A incubação de células de levedura com fosfatase (tan) remove o grupo fosfato do substrato exibido, interrompendo a marcação de anticorpos antifosfotirosina, facilitando a análise de modificação enzimática por citometria de fluxo. Clique aqui para ver uma versão maior desta figura.

Figura 3: Atividade da fosfatase e titulação do tempo. As células de levedura foram expostas a múltiplas combinações de tempo e concentração de fosfatase, seguidas de análise por citometria de fluxo. Todas as amostras tratadas foram comparadas a um controle incubado pelo mesmo período de tempo e condições tampão sem SHP-2. A diferença percentual de fosforilação mediana foi definida como a razão entre a mediana Y dos eventos exibidos na superfície menos o sinal de ruído basal fornecido pela mediana Y em eventos não exibidos em amostras contendo SHP-2, dividido pela mesma relação em seus respectivos controles. A hipótese nula é rejeitada quando diferenças significativas são observadas na comparação das mudanças entre os grupos de tempo e concentração usando ANOVA de duas vias (p < 0,05). O efeito das variáveis descritas sobre a diferença percentual de fosforilação mediana é independente entre si (Interação p > 0,05). O teste HSD de Tukey foi realizado para análise post hoc para obter mais informações sobre a significância da diferença entre o tempo geral de incubação e os grupos de concentração, e uma série de testes t assumindo variância desigual foram usados para definir a significância estatística de grupos individuais em um tempo e concentração especificados. Os dados são apresentados como média ± desvio padrão de quatro repetições. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Diretrizes de preparação de meios de indução de leveduras e proteínas. Descrição tabular da massa necessária de cada componente químico para formular 1 L de Meio de Crescimento Seletivo de Levedura, Meio de Indução de Proteína de Levedura Seletiva e Meio de Dextrose de Peptona de Extrato de Levedura. Uma vez que o meio descrito tenha sido misturado corretamente, filtre-esterilize antes de seu uso. Instruções adicionais estão incluídas para fazer placas de mídia de crescimento de levedura seletiva. Clique aqui para baixar esta tabela.
Tabela 2: Preparação de amostra recomendada para desfosforilação de substrato e estratégia de marcação de anticorpos. Descrição tabular das amostras necessárias para a medição das modificações enzimáticas dos substratos fosforilados exibidos na superfície da levedura. A preparação da amostra e a estratégia de marcação de anticorpos a seguir são especificadas para os controles necessários e cada amostra a ser analisada, incluindo diluições correspondentes de reagentes de marcação. Clique aqui para baixar esta tabela.
Tabela 3: Cálculos da taxa de excesso de anticorpos para proteínas. Descrição tabular do número teórico de anticorpos disponíveis por proteína expressa na superfície de uma célula de levedura enquanto é marcada para citometria de fluxo. O número teórico expresso pressupõe que 100% das células de levedura expressam 10.000 proteínas em sua superfície para garantir o excesso de anticorpos e os cálculos da taxa de excesso são baseados na concentração de estoque de reagentes exibida na tabela e adquirida do fornecedor. Clique aqui para baixar esta tabela.
Tabela Suplementar S1: Sequência de aminoácidos de de construção. Representação tabular da sequência de aminoácidos para os de construção localizados em ambos os lados do promotor Gal 1-10. As sequências destacadas correspondem às suas descrições codificadas por cores. O lado Gal-10 do plasmídeo é representado como uma tradução reversa da sequência de aminoácidos para facilitar sua compreensão. Todos os caracteres deixados em preto correspondem à tradução de aminoácidos dos locais de digestão da enzima de restrição usados para fornecer modularidade à construção. Clique aqui para baixar este arquivo.
Discussão
O protocolo apresentado permite a análise de interações enzimáticas utilizando a exibição extracelular de proteínas na superfície da levedura. A incorporação do sequestro do retículo endoplasmático no plasmídeo de exibição de superfície usado introduz a capacidade de analisar interações específicas entre enzimas e substratos modificados pós-traducionalmente extracelularmente devido às interações intracelulares que podem ser projetadas para ocorrer27,29. Os ensaios de interação enzimática previamente estabelecidos usando a exibição da superfície da levedura envolvem a expressão das proteínas de interesse intracelularmente, sendo a exibição da superfície apenas uma ferramenta para a detecção de interações intracelulares que ocorrem entre as proteínas-alvo 23,25,29.
Este protocolo se baseia nessa plataforma, movendo as interações enzimáticas direcionadas para o ambiente extracelular, o que introduz flexibilidade adicional nas enzimas que podem ser estudadas e no ambiente em que sua atividade é monitorada. As interações estudadas que ocorrem extracelularmente dão aos pesquisadores a oportunidade de adaptar o ambiente de incubação para ser mais ideal para a atividade enzimática, expandindo as enzimas que poderiam ser estudadas cujas atividades são prejudicadas no retículo endoplasmático da levedura, que é um ambiente altamente oxidante37. Além disso, a capacidade de titular a concentração de enzimas em relação a um determinado substrato permite ensaios de atividade enzimática específicos que não poderiam ser realizados intracelularmente devido à saturação hipotética da taxa de reação durante o sequestro.
Dentro do protocolo, existem várias etapas críticas a serem observadas para garantir que o resultado desejado seja observado. A transformação bem-sucedida das construções estudadas em levedura é essencial para obter os melhores resultados das etapas a seguir. O monitoramento preciso da densidade óptica deve ser feito para rastrear o crescimento saudável das culturas e garantir que elas não cresçam demais antes da indução de proteínas ou preparação da amostra. A fase logarítmica do crescimento das células de levedura abrange o período em que a produção de proteínas é mais alta, enquanto na fase estacionária, os mecanismos responsáveis pela produção de proteínas são interrompidos38. Tendo isso em mente, as medições de densidade óptica fornecem uma medição precisa das fases de crescimento em que as culturas de levedura estão, e etapas como indução de proteínas e preparação para ensaios devem ser feitas fora da fase estacionária ou quando as culturas crescem demais (OD600nm < 6).
Para o ensaio de modificação enzimática, o ambiente de incubação descrito foi específico para a enzima estudada, SHP-2 e a atividade enzimática que estava sendo realizada, a desfosforilação. A TDT foi utilizada para o ambiente redutor que proporciona durante a incubação com SHP-234. Portanto, é importante medir com precisão as concentrações dos produtos químicos usados para modificar o ambiente de incubação nos ensaios enzimáticos para garantir uma atividade enzimática consistente entre amostras e experimentos. A SHP-2 foi usada como uma proteína recombinante e é fundamental regular a temperatura durante as diferentes etapas de manuseio da enzima. Para um ensaio bem-sucedido, a enzima não deve ter passado por mais de dois ciclos de congelamento e descongelamento e deve estar no gelo durante a preparação de cada amostra. É então imperativo aliquotar a enzima recombinante em um volume suficiente para satisfazer os requisitos do ensaio. Durante a incubação real, a temperatura precisa ser estritamente controlada na temperatura ideal para a enzima, 37 ° C neste caso, com movimento constante de um rotor para garantir a homogeneidade da mistura de incubação.
O método geral para a análise com enzimas recombinantes exigiu modificações específicas para a interação entre SHP-2 e o substrato fosforilado exibido na superfície. A adaptação do protocolo a outras interações enzima-substrato extracelular envolve a modificação das sequências usadas, do ambiente do tampão de atividade e do reagente usado para detecção. Para testar outras interações quinase-substrato-fosfatase, a adaptação envolve a substituição das sequências de proteínas por um par quinase-substrato em suas respectivas posições no de construção descrito na Tabela Suplementar S1. A sequência proteica do substrato, juntamente com pelo menos o domínio quinase da quinase de interesse, deve ser incluída no plasmídeo, e a fosfatase direcionada ao substrato fosforilado produzido deve estar na forma de uma proteína recombinante. A interação representativa entre LCK, CD28 e SHP-2 fornece um exemplo de uso do sequestro de retículo endoplasmático projetado no de construção como uma ferramenta para produzir proteínas modificadas pós-tradução a serem investigadas extracelularmente com sua enzima alvo. Substratos de interesse que não precisam passar por modificações pós-traducionais (por exemplo, substratos que podem ser fosforilados extracelularmente usando quinase adicionada) podem ser expressos na superfície da levedura sem uma enzima emparelhada dentro do de construção. Nesse caso, a sequência de proteínas para a quinase descrita na Tabela Suplementar S1 seria removida com apenas a sequência do substrato sendo incluída no de construção. Observamos em nossa experiência anterior que a co-localização de uma serina-treonina quinase com um substrato conhecido resultou na exibição de substrato que não foi fosforilado (Ezagui e Stern, dados não publicados), portanto, testes rigorosos de modificação enzimática bem-sucedida devem ser realizados antes da aplicação da fosfatase extracelular. Publicamos anteriormente um protocolo para co-localização quinase-substrato que pode ser útil para esta etapade qualificação 39.
Quinases e fosfatases geralmente contêm resíduos de cisteína não pareados que, quando oxidados, formam ligações dissulfeto dentro da proteína ou entre proteínas, o que pode interromper a atividade catalítica da proteína devido à mudança conformacional40,41. A compreensão dessa bioquímica proteica é essencial para determinar o ambiente de reação adequado para modificação enzimática. Como resultado, um agente redutor precisa ser suplementado ao ambiente de incubação para garantir que a proteína recombinante usada permaneça ativa. O DTT é um agente redutor comum usado para esses fins, mas a concentração no ensaio deve ser otimizada. O uso de uma concentração muito alta de TDT dificulta a exibição do substrato na superfície da levedura, pois as âncoras Aga1p e Aga2p são mantidas uma na outra por meio de ligações dissulfeto, que são reduzidas na presença de DTT42. A concentração de DTT foi ajustada para a concentração mínima que permitiria uma atividade de fosfatase máxima relativa sem ter efeitos prejudiciais na exibição da superfície dos substratos42. O ambiente de incubação para qualquer enzima que esteja sendo analisada deve ser otimizado para garantir a retenção da atividade enzimática quando usado neste ensaio. Se uma enzima requer um ambiente redutor consideravelmente mais forte do que o DTT de 0,5 mM usado neste ensaio, a plataforma é limitada pela redução na exibição da superfície e pode não ser ideal para o ensaio enzimático específico desejado. Da mesma forma, o tampão 2x usado durante a etapa de incubação neste protocolo foi incluído em pesquisas anteriores sobre tampões aceitáveis que promovem a atividade de SHP-2, e pesquisas semelhantes devem ser realizadas para formular um tampão de incubação para qualquer outra enzima usada para este ensaio34. Os pontos de partida para a elaboração desses tampões podem incluir a pesquisa bibliográfica de aplicações in vitro bem-sucedidas da enzima de interesse ou a recomendação do fabricante da enzima para um tampão de atividade. A enzima recombinante que está sendo usada deve ser titulada especificamente para este ensaio para identificar uma concentração e tempo de incubação aceitáveis, o que permite que a atividade enzimática direcionada ocorra antes da coleta de dados.
Para a adaptação deste protocolo a outros tipos de interação enzima-substrato, novos reagentes de detecção fluorescente precisarão ser selecionados e titulados quanto à sensibilidade. Outros estudos demonstraram exemplos disso, incluindo o uso de anticorpos direcionados a marcadores de epítopos para detectar a presença ou ausência de substratos peptídicos após o tratamento com protease23 , 25 e anticorpos sensíveis à acetilação para detectar modificações nas proteínas histonas43. Para qualificar esses reagentes, um controle positivo (que demonstre de forma verificável a modificação de interesse) e um controle negativo (um controle que demonstre de forma verificável a falta de modificação de interesse) devem ser estabelecidos. Isso pode ser feito por meio da exibição da superfície da levedura de uma construção que demonstrou anteriormente a modificação de interesse ou, em alguns casos, pode ser estabelecida por meio da imobilização de proteínas ou peptídeos recombinantes. Por exemplo, no caso de fosforilação (e muitas outras modificações pós-traducionais de interesse), peptídeos de sequência conhecida podem ser sintetizados com fosfotirosina (controle positivo) ou tirosina não modificada (controle negativo) e uma biotina C-terminal que permitiria a imobilização dos peptídeos em esferas revestidas com estreptavidina. Os grânulos revestidos com peptídeos podem ser marcados com o anticorpo específico de modificação e avaliados quanto à especificidade e sensibilidade de detecção usando citometria de fluxo com métodos semelhantes aos descritos na Seção 4. Diferentes diluições de anticorpos devem ser usadas para encontrar uma concentração que forneça sinal máximo do controle positivo, sinal mínimo do controle negativo e equilibre excesso suficiente de excesso de anticorpo por proteína modificada (usando a equação na nota da Etapa 4.1) com considerações econômicas para o número de experimentos a serem realizados por alíquota de anticorpo obtido.
Descrevemos um protocolo para adaptar a facilidade da plataforma de exibição da superfície da levedura para ensaios de atividade enzimática extracelular. O método é demonstrado usando CD28 fosforilado exibido na superfície da levedura para ser desfosforilado por SHP-2 recombinante durante a incubação, mas é generalizável para muitos tipos de modificação enzimática por meio da modificação do tampão de trabalho e do par enzima-substrato usado.
Divulgações
Os autores não têm conflitos de interesse relacionados a este trabalho a divulgar.
Agradecimentos
Este trabalho foi apoiado por um prêmio NSF CAREER para L.A.S. (CBET - 2339172) e fundos iniciais da University of South Florida.
Na Figura 2A, o ícone translúcido aberto de microtubo da Servier https://smart.servier.com/ está licenciado sob CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/. As modificações incluem a adição de tampão e uma célula de levedura (esquerda) e a adição de anticorpo (centro-direita).
O tubo de ensaio, a incubadora e o citômetro de fluxo da Figura 2A foram fornecidos por meio de www.bioicons.com em acesso aberto.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
Referências
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados