Method Article
효모 표면에 표시된 단백질의 Enzymatic modification and flow cytometry assessment (효소 변형 및 유세포 분석 평가)
요약
여기에서는 효소 변형 분석을 위해 효모 표면 표시 기질을 사용하기 위한 프로토콜을 소개합니다. 이 플랫폼은 대표적인 효소 변형 분석법으로 기질 중 하나에 대한 티로신 인산가수분해효소 SHP-2의 탈인산화 활성 분석을 사용하여 시연되었습니다.
초록
효모 표면 디스플레이는 단백질 기능에 대한 고처리량 스크리닝을 가능하게 하는 유전자형-표현형 연결 전략입니다. 전통적으로 효모 표면 디스플레이는 새로운 결합 단백질의 진화에 적용되었으며, 결합 강도 수준별로 평가하고 분류하는 데 사용되는 유세포 분석법이 사용되었습니다. 최근에는 유세포 분석으로 판독할 수 있는 표현형을 제공하는 첨가제(예: 인산화) 또는 감산(예: 단백질 분해) 변형을 사용하여 기질 변형체의 효소 변형을 스크리닝하기 위해 효모 표면 디스플레이를 적용하는 것에 대한 관심이 높아지고 있습니다. 이러한 변형은 세포 내 공동 국소화(intracellular co-localization)를 사용하여 정기적으로 적용되지만, 표시된 기질의 세포 외 효소 변형을 달성할 수 있는 능력은 조사에 더 많은 반응을 일으킬 수 있습니다. 여기에서는 효모 표면에 표시된 후보 기질에 대한 세포 외 효소 변형을 위한 스크리닝 분석을 설계 및 적용하고 유세포 분석을 사용하여 후속 평가를 수행하는 기술에 대해 설명합니다. 당사는 인산화효소(phosphatase), 탈인산화(dephosphorylating), 효모, 인산화된 티로신 잔기를 함유한 기질(lumine)의 맥락에서 이러한 프로토콜을 제공하며, 이 적용된 프레임워크가 다른 효소-기질 쌍에 대한 스크리닝 분석을 개발하는 데 어떻게 적용될 수 있는지에 대해 설명합니다.
서문
효소와 효소 표적 간의 상호 작용에 대한 이해는 세포 항상성 및 질병 발병을 제어하는 경로의 생물학적 특성화의 필요성으로 인해 점점 더 흥미로운 연구 분야가 되었습니다 1,2. 효소는 생물학적 생명을 유지하는 많은 반응의 촉매 작용을 담당하며, 세포 대사 3,4,신호 전달5과 같은 필요한 경로를 제어하고, 심지어 게놈 복구6,7와 같은 기본 과정도 조절합니다. 이러한 과정에서의 역할로 인해, 이들의 상호작용은 많은 질병의 발병에도 중요한 역할을 하는데, 그 이유는 암세포의 활성이 이탈하면 세포 활동의 심각한 조절 장애를 일으켜 세포사멸 또는 해로운 암세포의 증식을 유발할 수 있기 때문입니다2. 효소 활성에 대한 연구는 새로운 치료제 개발에 중요한 응용을 가졌으며 8,9 각 특정 효소-기질 상호 작용10에 맞춤화된 분석이 필요했습니다. 여러 효소 분석법은 이러한 상호 작용의 평가 및 특성화를 위한 표준 프로토콜로 확립되었습니다. 효소 상호작용을 분석하기 위해 개발된 분석법은 활성화/억제11에 대한 결합을 모니터링하는 검출 분석법 또는 효소12에 의한 기질 변형을 모니터링하는 분석법으로 분류됩니다.
효소의 주요 역할 중 하나는 세포의 행동을 조절하는 것입니다. 신호 전달(Signal transduction)은 세포외 유발인자(extracellular trigger) 13에 대한 세포의 세포 내 반응으로, 세포의 생존과 기능에 영향을 미칩니다. 세포 증식, 분화 및 기타 많은 기능적 과정은 모두 이를 관장하는 효소 상호 작용과 함께 신호 경로를 포함합니다14,15. 효소는 번역 후 변형(post-translational modification)을 촉매하는데, 이는 종종 세포외 메시지의 올바른 전달을 담당하는 대규모 신호 전달 네트워크를 조절합니다16. 단백질 인산화(protein phosphorylation)는 가장 일반적인 번역 후 변형(post-translational modification)으로, 세포 신호 전달 및 기타 여러 세포 경로에서 흔히 볼 수 있습니다. 결과적으로, 단백질 키나아제는 중요한 조절 역할로 인해 잠재적인 치료 표적의 중요한 부분으로 부상했습니다17. 인산염 효소는 인산염 기반 세포 신호 복합체18,19에 대한 천연 조절 분자로, 표적 단백질20에서 인산염 잔류물을 제거할 수 있는 능력을 가지고 있습니다. 지난 10년 동안 인산가수분해효소는 여러 세포 유형에서 다운스트림 신호 전달 경로의 조절에 관여하는 암 치료21 및 염증성 질환22의 주요 치료 표적이 되었습니다. 단백질 키나아제(protein kinase)와 인산가수분해효소(phosphatase)는 함께 광범위한 상호작용을 제공하며, 이는 특정 효소 분석 프로토콜의 개발을 통해 연구할 수 있습니다.
효모 표면 디스플레이는 효소 활성의 특성화 및 평가를 위한 도구로 사용되어 왔습니다23,24. 이는 소포체 격리 전략과 결합할 때 번역 후 변형 과정의 스크리닝을 위한 고처리량 플랫폼을 제공합니다25,26. 이를 통해 kinase-substrate pair는 KDEL 수용체27에 대한 결합을 통해 소포체에서 공동 국소화되고 유지될 수 있으며, 여기서 기질의 인산화는 kinase와 그 표적 사이의 근접성으로 인해 증가된 속도로 발생할 수 있습니다. KDEL 수용체 결합은 다른 HDEL 서열25,28보다 더 강한 보유 능력을 갖는 것으로 나타난 C-말단 FEHDEL 소포체 보유 서열에 의해 매개됩니다. 그런 다음 인산화된 기질은 유세포 분석29를 통한 후속 평가를 위해 효모 표면에 고정됩니다. 현재로서는 효모 표면에 표시되는 기질의 효소 변형을 위해 확립된 일반화 가능한 프로토콜은 없습니다. 우리는 세포 외에서 발현된 인산화된 기질 변이체를 활용하고 알려진 인산가수분해효소에 의한 탈인산화를 통해 변형함으로써 효모 표면 디스플레이의 용량을 확장합니다. 그런 다음 유세포 분석 분석은 알려진 인산가수분해효소(phosphatase)를 사용한 배양의 결과로 인한 인산화 중앙값의 변경 측정을 통해 앞서 언급한 기질의 표현형 평가를 위한 플랫폼을 제공합니다. 이는 표면 표시 단백질의 번역 후 변형을 위한 적응 가능한 방법을 제공하는 동시에 효모 표면 디스플레이 플랫폼을 사용할 때 상호 작용의 효소 변형 분석을 위한 방법을 제공합니다.
효모 표면 디스플레이 플랫폼에 kinase-substrate 상호 작용의 도입, 발현된 인산화 기질과 재조합 phosphatase의 공동 배양 및 유세포 분석을 통한 탈인산화 활성의 후속 분석을 설명하는 효소 변형 분석의 개발 및 적용을 위한 기술을 제시합니다. 이 보고서에서 이는 효모 소포체에서 CD28의 세포질 도메인과 림프구 키나아제(LCK)를 공동 국소화한 후 효모 표면에 인산화된 CD28을 표시하고 Src 상동성 영역 2 도메인 함유 인산가수분해효소-2(SHP-2)에 의한 탈인산화를 통해 수행됩니다. 다양한 펩타이드 서열에서 인산화된 티로신 잔기를 검출하는 팬 항인산화티로신 항체(본 연구에서는 4G10)가 인산가수분해효소 처리의 함수로 인산화량 수준의 정량화에 사용됩니다. 상세한 프로세스는 효소-기질 상호 작용을 조사하기 위한 일반화 가능한 접근 방식을 제공합니다. 정제된 방식으로 효소와 기질을 연구하는 전향적 방법.
프로토콜
1. 플라스미드를 품고 있는 효모의 세포 성장과 단백질 발현 유도
- 표 1에 설명된 레시피에 따라 비플라스미드 함유 효모(YPD), 플라스미드 함유 효모 세포 성장(SD-CAA) 및 단백질 발현 유도(SRG-CAA) 및 SD-CAA 플레이트의 성장에 필요한 배지를 준비합니다.
- 다양한 제조업체의 효모 형질전환 키트에 사용하기 위해 일반적으로 채택되는 리튬 양이온 기반 방법30,31을 통해 키나아제-기질 쌍을 포함하는 효모 디스플레이 플라스미드 DNA를 EBY-100 효모 세포로 변환합니다.
참고: Electroporation32 또는 다른 선호되는 효모 플라스미드 형질전환 기법은 형질전환되는 플라스미드 구조체에 따라 사용할 수 있습니다. - 10mL의 YPD 배지가 있는 14mL 배양 튜브를 준비합니다. EBY-100 세포를 접종하고 배양액이 0.8-1.0(8 x 106- 1 x 107 cells/mL)의 광학 밀도(OD600nm)에 도달할 때까지 30°C, 300rpm의 진탕 인큐베이터에서 성장합니다.
참고: OD600nm 는 각각의 배지에 효모 배양액을 1:10으로 희석한 3mL 샘플 큐벳과 샘플 희석에 사용되는 배지를 포함하는 3mL 블랭크 큐벳을 준비하여 측정합니다. 분광 광도계의 OD600nm 프로그램은 먼저 블랭크 큐벳을 측정한 다음 각 샘플에 대해 준비된 해당 희석액을 설정하여 각 샘플 큐벳을 측정하는 데 사용됩니다. 1 OD600nm 는 1 × 107 yeast/mL에 해당합니다. - 1,000 x g에서 3 분 동안 배양액을 원심 분리하여 세포를 수확하고 효모 형질 전환 키트 또는 TE (10 mM Tris-HCl 및 1.0 mM EDTA) 31에 제공된 1차 세척 용액으로 세척합니다.
- 1,000 x g 에서 3분 동안 세포를 다시 펠렛화하고 효모 형질전환 키트에 제공된 형질전환 완충액 1mL 또는 멸균수에 재현탁합니다. 세포는 50 μL 분취액으로 분주되어야 하며 -80 °C에서 최대 6개월 동안 보관할 수 있습니다.
- 플라스미드 형질전환을 위해 플라스미드 당 하나의 부분 표본을 준비하고 얼음에서 해동한 다음 효모 디스플레이 구조체를 포함하는 0.5-1.5 μg의 플라스미드 DNA를 세포에 직접 첨가합니다. 효모 형질전환 키트에 제공된 형질전환 용액 0.5mL 또는 멸균 50% 폴리에틸렌 글리콜 0.5mL 및 0.1M LiOAc 용액31을 첨가합니다. 피펫팅을 통해 세포, 플라스미드 DNA 및 형질전환 용액의 혼합물을 철저히 결합하십시오.
- 30 ° C에서 30-60 분 동안 형질 전환 혼합물을 정적으로 배양하고 15 분 간격으로 와류 혼합물을 배양합니다. 1,000 × g에서 3분 동안 원심분리하여 세포를 수확합니다.
- 4.5mL의 SD-CAA 배지가 있는 14mL 배양 튜브를 준비합니다. 원하는 플라스미드를 함유한 세포를 500μL의 SD-CAA에 재현탁시키고 제조된 4.5mL를 접종합니다.
- 한천을 뚫지 않도록 주의하면서 접종된 배양액 5mL 중 50μL를 SD-CAA 플레이트에 분배하고 30°C에서 48시간 동안 정적으로 배양하여 형질전환 효율을 확인합니다.
- 5mL의 SD-CAA 세포 배양을 30°C, 300rpm의 진탕 인큐베이터에서 최소 18시간 동안 배양합니다. 16시간 및 20시간 후 광학 밀도(OD600nm)를 모니터링합니다. 샘플이 6을 초과하지 않는 충분한 광학 밀도로 성장하면 2,500 × g에서 3분 동안 배양을 원심분리합니다. 효모 펠릿을 방해하지 않고 상등액을 버리십시오.
- SRG-CAA의 효모 펠릿을 최종 OD600nm 에서 1(<1 × 107 yeast/mL) 미만으로 재현탁합니다.
- 30°C, 300rpm의 진탕 인큐베이터에서 효모 배양을 최소 8시간 동안 24시간 이하로 배양합니다.
참고: 효모 세포에서 단백질 발현 유도는 20-37°C에서 다양할 수 있습니다. 30°C는 키나아제/기질 쌍29,33의 합성에 적합하지만 연구 중인 특정 단백질에 필요하다고 판단되는 경우 조정할 수 있습니다. - OD600nm 를 측정하여 세포 밀도를 측정합니다.
참고: 이 시점에서 프로토콜은 효모 배양액을 4°C에서 보관하여 중지할 수 있습니다.
2. 4G10 항 포스포티로신 항체의 비오틴닐화
- 680μL의 멸균 PBS를 첨가하여 PEG4-NHS-비오틴 2mg 바이알을 최종 농도 5mM로 재현탁합니다.
참고: PEG4-NHS-비오틴의 재현탁은 비오틴화 반응이 수행되기 직전에 새로 수행해야 합니다. NHS는 수용액에서 가수분해됩니다. 멸균 PBS 및 바이알의 사용은 장기 보관을 위한 시약 준비 및 세포 기반 분석에 사용하여 시약 생존력 또는 수행 중인 민감한 분석에 대한 오염 물질의 잠재적 영향을 완화하는 데 중요합니다. - 4G10 항체 농도에 따라 멸균 1.7mL 바이알에 항체 100μg을 첨가합니다.
- 2.1단계에서 준비된 5mM PEG4-NHS-비오틴 1μL를 4G10 항체 100μg이 들어 있는 바이알에 첨가하여 7.5:1의 항체에 대한 비오틴의 몰 비율을 달성합니다. 반응을 균질화하기 위해 혼합물을 부드럽게 피펫팅합니다.
- 최소 2시간 동안 일정한 회전으로 실온에서 반응을 배양합니다.
- 0.5mL 스핀 탈염 컬럼에 대한 제조업체의 프로토콜에 따라 비오틴화된 4G10(B-4G10)의 완충액을 PBS로 교환합니다.
참고: 분자량 차단(MWCO)이 7kDa인 탈염 컬럼은 일반적으로 항체의 비오틴화에 사용되어 더 큰 항체를 유지하면서 미반응 비오틴 및 기타 작은 분자를 제거할 수 있습니다. - B-4G10 항체를 PBSA(1g/L 소 혈청 알부민이 포함된 PBS)에서 최종 농도 1μM로 희석합니다. B-4G10 항체를 더 작은 부피로 분취하여 반복되는 동결/해동 주기를 방지합니다.
참고: 비오틴화 항체는 효율성을 크게 잃지 않고 최대 3개월 동안 매일 사용하기 위해 4°C에서 보관할 수 있습니다. 사용하지 않는 나머지 부분 표본은 -20 °C에서 최대 2년 동안 보관하십시오.
3. 효모 세포 표면에 발현된 기질의 탈인산화
- 문헌34에 앞서 기술된 바와 같이 2x 작업 완충 용액을 준비합니다.
알림: 필요한 성분의 측정을 용이하게 하기 위해 2x를 사용하는 것이 좋습니다. - 단계 3.1 1:2에서 준비한 2x 버퍼 용액을 탈이온수에 희석하여 1.7mL 바이알의 샘플에 대한 작업 버퍼를 준비합니다.
참고: 각 샘플의 총 반응 부피는 20 μL이며 각 샘플에는 10 μL에서 18 μL 사이의 작업 버퍼가 필요합니다. 모든 샘플 또는 대조군에 대해 충분한 작업 버퍼를 준비합니다. - 표 2에 설명된 권장 샘플 준비에 따라 1.7mL 바이알에 해당 대조군 또는 샘플 이름으로 라벨을 부착합니다.
- 1.9단계에서 측정한 OD600nm 를 기반으로 각 샘플에 대한 해당 효모 배양액에서 200만 개(2 × 106)의 효모 세포를 회수하는 데 필요한 배양량을 계산합니다.
- 이전 단계에서 계산한 효모 배양량의 부피를 각 샘플에 대해 1.7mL 바이알에 추가합니다.
- 바이알을 4,500 × g에서 1분 동안 원심분리합니다. 마이크로피펫을 사용하여 상등액을 조심스럽게 제거하고 생물학적 유해 폐기물로 폐기합니다.
- 펠릿화된 세포를 PBSA 1mL에 재현탁하고 3.6단계를 반복합니다.
참고: 펠릿화된 세포를 방해하지 않고 가능한 한 많은 상층액을 제거하는 것이 중요합니다. - 재고 농도를 기반으로 20 μL 총 반응 부피에서 1,000 nM의 최종 농도를 갖는 데 필요한 재조합 인간 SHP-2의 부피를 계산합니다.
참고: 재조합 SHP-2는 각 분석에 사용된 단백질이 두 번 이상의 동결-해동 주기를 거치지 않도록 소량으로 분주해야 합니다. 분석을 위해 모든 샘플이 준비된 후 부분 표본에서 남은 SHP-2는 폐기해야 합니다.
재조합 효소의 스톡 농도는 로트 번호에 따라 달라질 수 있습니다. 재조합 SHP-2는 일반적으로 0.2-0.4 mg/mL의 스톡 농도로 제형화됩니다. 0.324 mg/mL SHP-2의 스톡 농도의 경우 이는 4.696 μM의 스톡 농도에 해당합니다(SHP-2의 분자량은 69 kDa). 20 μL 반응에서 SHP-2 스톡의 4.26 μL는 1,000 nM SHP-2의 최종 반응 농도를 나타냅니다. - 15mL 원뿔형에서 준비된 10mL의 탈이온수에 7.7mg의 DTT를 첨가하여 5mM DTT 용액을 만듭니다. 장비 제한으로 인해 밀리그램의 무게를 측정하기 어려운 경우 10mL의 탈이온수에 0.77g의 DTT를 첨가한 다음 100배 희석을 수행하여 분석에 사용되는 5mM DTT 용액을 생성합니다.
참고: DTT 용액은 사용 가능한 장비를 사용하여 밀리그램 단위의 양을 측정할 수 없는 경우 5mM로 희석할 수 있는 고농도 원액에서 준비할 수 있습니다. DTT 용액은 가수분해 경향으로 인해 물에 희석될 때 장기간 불안정해지기 때문에 작업 완충액에서 세포 재현탁 전에 신선하게 준비해야 합니다. - 3.2단계에서 준비한 작업 완충액에 펠릿화된 세포를 재현탁시켜 각 샘플 또는 대조군의 최종 반응 부피가 20μL가 되도록 합니다.
참고: 추가된 작업 버퍼의 양은 각 샘플에 포함될 DTT(2μL) 및 SHP-2(3.8단계에서 계산)의 양을 기준으로 계산해야 합니다. - 3.9단계에서 준비한 5mM DTT 용액 2μL를 각 샘플 또는 대조군에 추가하여 최종 반응 농도를 0.5mM DTT로 만듭니다.
- 3.8단계에서 계산한 SHP-2의 부피를 각 샘플에 추가하여 최종 부피 20μL를 만들고 마이크로피펫을 사용하여 부드럽게 혼합합니다.
- 누출 또는 교차 오염을 방지하기 위해 샘플 바이알 뚜껑을 파라필름으로 감쌉니다.
- 37°C의 로터에서 일정한 속도로 2시간 동안 샘플을 배양합니다.
- 로터에서 샘플을 제거하고 각 샘플에 PBSA 1mL를 추가하여 반응을 중지합니다.
- 3.6단계를 반복합니다.
4. 탈인산화된 기질의 세포 라벨링 및 유세포 분석
- 표 2에 설명된 대로 3.16단계의 샘플을 해당 1차 시약의 20μL 혼합물에 재현탁합니다. 실온에서 20분 동안 샘플을 배양합니다.
참고: 사용된 모든 시약 농도는 효모 세포 표면에서 발현되는 단백질의 수와 관련하여 과도하게 계산되었습니다. 계산은 샘플35에서 모든 2 x 106 효모에서 10,000 개의 단백질 / 세포의 발현을 가정하지만, 일상적으로 그 중 ~ 50 % 만 발현합니다. 표 3은 다음과 같이 계산 된 표 2에 발현 된 각 시약에 대한 초과 라벨링 시약 비율을 보여줍니다.
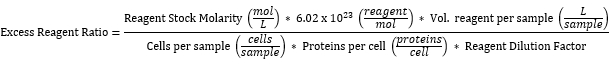
- 4,500 × g 에서 1분 동안 원심분리기 시료를 추출하고 상등액을 생물학적 유해 폐기물로 간주하여 폐기합니다.
- PBSA 1mL에 재현탁하여 세포를 한 번 세척합니다. 4.2단계를 반복합니다.
- 표 2에 설명된 대로 해당 2차 시약의 20μL 혼합물에서 샘플을 재현탁합니다. 빛이 없는 곳에서 15분 동안 샘플을 배양합니다.
- 4.2단계를 반복합니다.
- 4.3단계를 반복합니다.
- 세척된 샘플을 300-500 μL의 PBSA에 재현탁하고 5 mL 폴리스티렌 튜브로 옮겨 적절한 유세포 분석기를 사용하여 즉시 분석합니다.
알림: 샘플을 운송해야 하는 경우 젖은 얼음에 보관하십시오. 권장되지는 않지만 샘플은 습식 펠릿으로 최대 2시간 동안 4°C에서 보관할 수 있습니다. - 새로운 실험을 위해 필요한 세포 분석기 시작 및 준비를 수행한 후 File 메뉴에서 New Experiment 버튼을 클릭하고 실험 이름을 지정한 다음 Save를 클릭하여 획득한 데이터가 원하는 파일 경로에 저장되었는지 확인합니다.
- 위쪽 도구 모음에서 점도표 아이콘을 선택하여 실행할 각 샘플에 대해 두 개 이상의 점도표를 만듭니다. 점도표 중 하나의 경우 X축 이름을 선택하여 FSC-A 채널을 표시하고 Y축 이름을 선택하여 SSC-A 채널을 표시합니다. 이 플롯은 Side Scatter - Area 대 Forward Scatter - Area를 보여주며 추가 분석을 위해 효모 세포를 게이트하는 데 사용됩니다.
- 다른 점도표에서 X축 이름을 선택하여 1차 항항원결정기 태그 항체를 표적으로 하는 2차 시약이 형광을 발하는 채널을 표시합니다. Y축 이름을 선택하여 streptavidin 2차 시약이 형광을 발하는 채널을 표시합니다. 이 플롯은 측면 대 전방 산점도 플롯에서 게이트된 이벤트만 효모 세포로 표시하며 Y축에 티로신 인산화를 표시하고 X축에 기질 표면 발현을 표시하는 데 사용됩니다.
참고: 이 예에서 1차 항항원결정기 태그 항체를 표적으로 하는 2차 시약은 FITC(AF-488) 채널에서 형광을 발하고 스트렙타비딘 2차 시약은 AF-647 채널에서 형광을 발합니다. 사용되는 채널은 라벨링 중에 사용되는 1차 및 2차 시약에 따라 달라질 수 있습니다. - 각 샘플 튜브를 세포분석기의 튜브 홀더에 놓고 세포분석기에 대해 Run을 선택하여 샘플 로딩 및 데이터 수집을 시작합니다. 표시할 이벤트, 기록할 이벤트, 기록할 시간 및 필요에 따라 샘플 유량을 조정합니다.
- 4.9단계에서 생성된 SSC-A 대 FSC-A 플롯에서 건강한 효모 세포를 둘러싼 게이트를 정의합니다. 그림 1 은 적용할 게이팅 전략의 설명 표현을 보여줍니다.
참고: SSC-A 대 FSC-A 플롯에서 100,000개의 이벤트를 표시하고 정의된 효모 게이트 내에서 50,000개의 이벤트를 기록하는 것은 추가 분석을 위해 충분한 데이터를 시각화하고 수집하기 위한 좋은 지침입니다. 일중항 효모에 대해 선택하기 위한 보다 엄격한 게이팅 전략은 최근에 보고된 바와 같이 Forward Scatter-Height 대 Forward Scatter-Area의 플롯을 기반으로 적용할 수 있습니다36. 그림 1 에 표시된 게이팅 전략은 산란 게이팅을 통한 singlet 및 일부 doublet 효모를 포함하는 덜 엄격한 접근 방식에 해당합니다. - 유세포 분석기를 사용하여 모든 대조군 샘플의 형광을 기록합니다. 제어 샘플은 아래 4.14단계에서 설명하는 대로 게이팅 전략을 정의하는 데 도움이 되도록 먼저 정기적으로 수집됩니다.
- 처리된 샘플을 분석하기 전에 4.10단계에서 생성한 플롯에 대한 게이팅 전략을 정의합니다. 그림 1 은 적용할 게이팅 전략의 설명 표현을 보여줍니다.
- 유세포분석기와 4.12단계 및 4.14단계에서 정의된 게이팅 전략을 사용하여 탈인산화된 샘플의 형광을 기록합니다.
- 유세포 분석 소프트웨어를 사용하여 획득한 유세포 분석 데이터를 분석합니다.
- 표면에서 단백질을 발현하는 세포의 Y축 중앙값과 샘플과 대조군 사이에 표시되지 않는 세포가 제공하는 기준선 인산화를 측정하고 비교하여 탈인산화를 평가합니다. 다음과 같이 백분율 중앙값 인산화 차이를 계산합니다.

결과
1,000nM SHP-2 없이(그림 1A) 및 (그림 1B) 2시간 동안 배양된 모델 시스템의 개별 복제에서 유세포 분석 결과, 63.6%의 인산화 차이 중앙값이 63.6%로 나타났으며, 이는 모든 표면 표시 이벤트의 Y축 중앙값(인산화)에서 기준선 인산화 신호를 뺀 값으로 정의되며, 이는 방정식에 설명된 대로 처리된 샘플과 처리되지 않은 대조군 사이의 표시되지 않은 이벤트의 Y축 중앙값으로 정의되었습니다 프로토콜 단계 4.12에 정의되어 있습니다. 중앙값을 분석하기 전에 샘플은 건강한 세포 그룹을 포함하기 위해 크기(Forward Scatter)와 복잡성(Side Scatter)에 따라 게이트를 지정했습니다. 정의된 조건에서 시료 탈인산화는 눈에 잘 띄지 않게 구별할 수 있어야 합니다(그림 1C).
분석은 4가지 주요 방법으로 구성된 간단한 절차로 정의됩니다(그림 2A). 우리가 사용하고 이전에 발표한 효모 표면 디스플레이 시스템은 CD28의 세포질 꼬리와 티로신 키나아제 LCK로 구성된 키나아제-기질 쌍의 동시 유도 발현 능력을 가진 양방향 프로모터를 포함하는 플라스미드를 기반으로 합니다(그림 2B)29. 번역된 단백질의 공동 국소화는 신호 펩타이드를 표적으로 하는 소포체와 C-말단 ER 보유 서열로 인한 체류 시간 증가에 의해 강제되는 번역 후 변형에 의해 지시됩니다. Aga2p에 융합된 인산화된 기질의 분비는 표면 발현을 유도합니다(그림 2C). 기질은 두 개의 에피토프 태그(epitope tag)가 측면에 배치되도록 설계되어 있어 성공적인 번역 및 후속 표면 발현에 대한 세포 외 확인을 할 수 있습니다. 관심 있는 인산가수분해효소(이 경우 티로신 인산가수분해효소 SHP-2)가 포함된 효모 세포를 표시하는 기질을 배양하면 기질 결합 인산염의 감소를 통한 효소 변형을 분석할 수 있습니다(그림 2D).
제시된 모델 시스템에 대해 최적의 조건이 정의되었음에도 불구하고 분석의 일반화 가능성으로 인해 단백질의 다양화를 분석할 수 있습니다. 분석을 위한 최적의 조건은 일련의 적정을 통해 정의되었으며, 여기서 인산가수분해효소의 시간과 농도의 다양한 조합이 4배로 평가되었습니다(그림 3). 분석된 데이터는 복제를 동반한 양방향 ANOVA(p < 0.05)를 통해 통계적 유의성을 보여주었습니다. 2시간 및 1,000nM(48.8% ± 10%)의 선택된 조건은 불균등 분산을 가진 t-테스트 분석을 기반으로 2시간(p < 0.05)에서 750nM의 조건과 비교할 때 통계적 유의성을 유지하면서 약 50%의 중앙값 인산화 차이를 제공했습니다. t-테스트는 또한 대략적인 중앙값 인산화 차이 퍼센트(3h에서 500nM, 750nM 및 1,000nM의 경우 p > 0.05)를 제공하는 농도 중 하나에서 시간을 1시간 증가시킴으로써 2시간 및 1,000nM 결과와 유의미한 차이가 없음을 보여주었습니다.
Tukey HSD 사후 분석에 따르면 모든 농도에서 잠복 기간 간의 모든 평균 비교는 1시간에서 2시간을 제외하고는 유의하게 다릅니다. 테스트된 여러 농도를 비교할 때 250nM을 다른 모든 농도와 비교할 때 통계적으로 유의한 평균 차이만 관찰했으며, 이는 250nM을 제외하고 그룹 내에서 유사한 수준의 인산가수분해효소 활성이 예상됨을 나타냅니다. 샘플을 4시간 및 1,000nM SHP-2에서 처리했을 때 최적 조건(±t-test, p < 0.05)과 비교하여 20%의 차이를 관찰했음에도 불구하고 DTT를 사용한 긴 배양으로 인한 표면 발현 감소 및 효모 건강 저하로 인해 이 조합을 추구하지 않기로 결정했습니다. 우리는 이것이 적절한 SHP-2 인산가수분해효소 기능에 필요한 작동 완충액의 감소 조건에 의해 발생한다는 가설을 세웠습니다.

그림 1: 모델 시스템의 유세포 분석 전방 산란(X축) 대 측면 산란 게이팅(Y축)(왼쪽)을 보여주는 밀도 플롯 및 2시간 동안 배양된 CD28 세포질 도메인의 기질 C-말단 에피토프 태그 라벨링(X축) 대 기질 인산화(Y축)(가운데)를 보여주는 점도표(A) SHP-2 없이 (B) 최적 농도가 1,000nM로 정의됨. Y-Median은 표면 발현 세포(녹색)를 포함하는 정의된 게이트 내에서 상대적 인산화 측정으로만 측정되었습니다. 표시되지 않는 신호 이벤트는 배경 Y-Median 측정(회색)을 정의하기 위해 게이트되었습니다. (C) Y-중앙값에서 명백한 시야 차이를 보여주는 샘플의 점도표 오버레이. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 효모 표면의 효소 변형은 단백질을 표시했습니다. (A) 설명된 4가지 방법 내에서 중요한 단계를 보여주는 분석 개략도: 효소 분석을 위한 phosphatase(tan)를 사용한 샘플 준비, 원하는 효소 활성을 위한 배양, 활성 검출을 위한 세포 세척 및 라벨링, 유세포 분석 및 데이터 수집. (B) 효소-기질 쌍의 소포체 격리 및 기질의 표면 발현에 사용되는 카세트의 일반적인 구조를 나타내는 유전자 개략도. (C) 소포체의 그래픽 표현, 키나아제-기질 쌍의 공동 국소화(왼쪽), 효모 표면에 표시되고 표면 발현을 확인하기 위해 항인산성 항체(파란색) 및 항항원결부 태그 항체(분홍색)로 라벨링된 번역 후 변형된 기질의 분비(오른쪽). (D) phosphatase(tan)를 사용한 효모 세포의 배양은 표시된 기질에서 인산기를 제거하여 anti-phosphotyrosine 항체 표지를 방해하고 유세포 분석을 통한 효소 변형 분석을 용이하게 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: 인산가수분해효소 활성도 및 시간 적정. 효모 세포는 시간과 인산가수분해효소 농도의 여러 조합에 노출된 후 유세포 분석 분석을 수행했습니다. 처리된 모든 샘플은 SHP-2 없이 동일한 시간 길이 및 완충액 조건에서 배양된 대조군과 비교되었습니다. 퍼센트 중앙값 인산화 차이는 표면 표시 이벤트의 Y-중앙값에서 샘플을 포함하는 SHP-2의 비표시 이벤트에서 Y-중앙값이 제공하는 기준선 노이즈 신호를 뺀 비율로 정의되었으며, 이를 해당 대조군에서 동일한 관계로 나눈 값으로 정의되었습니다. 귀무 가설은 이원 분산 분석(p < 0.05)을 사용하여 시간과 농도 그룹 간의 변화를 비교할 때 유의한 차이가 관찰되면 기각됩니다. 백분율 중앙값 인산화 차이에 대해 설명된 변수의 효과는 서로 독립적입니다(교호작용 p > 0.05). 전체 배양 시간 및 농도 그룹 간의 차이 유의성에 대한 추가 정보를 얻기 위해 사후 분석을 위해 Tukey의 HSD 테스트를 수행했으며, 지정된 시간 및 농도에서 개별 그룹의 통계적 유의성을 정의하기 위해 불균등 분산을 가정하는 일련의 t-테스트를 사용했습니다. 데이터는 4회 반복의 평균 ± 표준 편차로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: 효모 성장 및 단백질 유도 배지 준비 지침. 1 L의 선택적 효모 성장 배지, 선택적 효모 단백질 유도 배지 및 효모 추출물 펩톤 덱스트로스 배지를 공식화하기 위해 각 화학 성분에서 필요한 질량에 대한 표 형식의 설명입니다. 설명된 매체가 적절하게 혼합되면 사용하기 전에 필터 멸균하십시오. 선택적 효모 성장 배지 플레이트를 만들기 위한 추가 지침이 포함되어 있습니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 2: 기질 탈인산화 및 항체 라벨링 전략을 위한 권장 시료 준비. 효모 표면에 표시된 인산화된 기질의 효소 변형을 측정하는 데 필요한 샘플에 대한 표 형식의 설명입니다. 샘플 준비 및 다음 항체 라벨링 전략은 라벨링 시약의 해당 희석을 포함하여 필요한 대조군과 분석할 각 샘플 모두에 대해 지정됩니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 3: 항체 대 단백질 과잉 비율 계산. 효모 세포 표면에서 발현되는 단백질당 사용할 수 있는 항체의 이론적 수에 대한 표 형식의 설명이며 유세포 분석을 위해 표지됩니다. 표현된 이론적 수치는 효모 세포의 100%가 항체 과잉을 보장하기 위해 표면에 10,000개의 단백질을 발현한다고 가정하며, 초과 비율 계산은 표에 표시된 시약 재고 농도를 기반으로 하고 공급자로부터 획득합니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
보충 표 S1: 구성물 카세트의 아미노산 서열. Gal 1-10 프로모터의 양쪽에 위치한 구조체 카세트에 대한 아미노산 서열의 표 표현. 강조 표시된 시퀀스는 색상으로 구분된 설명에 해당합니다. 플라스미드의 Gal-10 측은 이해를 돕기 위해 역 아미노산 서열 번역으로 표시됩니다. 검은색으로 표시된 모든 문자는 구조체에 모듈성을 제공하는 데 사용되는 제한 효소 분해 부위의 아미노산 번역에 해당합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
제시된 프로토콜은 효모 표면에서 단백질의 세포 외 표시를 사용하여 효소 상호 작용을 분석할 수 있도록 합니다. 사용된 표면 디스플레이 플라스미드에 소포체 격리를 통합하면 발생하도록 설계할 수 있는 세포 내 상호 작용으로 인해 효소와 번역 후 변형된 기질 간의 특정 상호 작용을 세포 외에서 분석할 수 있는 능력이 도입됩니다27,29. 효모 표면 표시를 사용하여 이전에 확립된 효소 상호 작용 분석은 관심 단백질의 세포 내 발현을 포함하며, 표면 표시는 표적 단백질 23,25,29 사이에서 발생하는 세포 내 상호 작용을 검출하기 위한 도구일 뿐입니다.
이 프로토콜은 표적 효소 상호 작용을 세포 외 환경으로 이동시켜 해당 플랫폼을 기반으로 하며, 이는 연구할 수 있는 효소와 효소의 활성을 모니터링하는 환경 모두에 추가적인 유연성을 도입합니다. 세포 밖에서 발생하는 연구된 상호 작용은 연구자들에게 효소 활성에 더 최적화되도록 배양 환경을 조정할 수 있는 기회를 제공하며, 심하게 산화되는 환경인 효모 소포체에서 활성을 방해하는 연구를 할 수 있는 효소를 확장할 수 있습니다37. 또한, 주어진 기질에 대한 효소의 농도를 적정하는 능력은 격리 중 가설된 반응 속도 포화로 인해 세포 내에서 수행할 수 없는 특정 효소 활성 분석을 가능하게 합니다.
프로토콜 내에는 원하는 결과를 관찰하기 위해 주목해야 할 몇 가지 중요한 단계가 있습니다. 연구된 구조체를 효모로 성공적으로 변환하는 것은 다음 단계에서 최적의 결과를 얻는 데 필수적입니다. 배양물의 건강한 성장을 추적하고 단백질 유도 또는 시료 전처리 전에 과도하게 증식하지 않도록 하기 위해 광학 밀도를 정확하게 모니터링해야 합니다. 효모 세포 성장의 로그 단계는 단백질 생산이 가장 높은 기간을 포함하는 반면, 정지 단계에서는 단백질 생산을 담당하는 메커니즘이 정지됩니다38. 이 점을 염두에 두고 광학 밀도 측정은 효모 배양이 있는 성장 단계를 정확하게 측정하며, 단백질 유도 및 분석 준비와 같은 단계는 고정상 밖에서 또는 배양이 과도하게 자랄 때(OD600nm < 6) 수행해야 합니다.
효소 변형 분석의 경우, 설명된 배양 환경은 연구된 효소인 SHP-2 및 수행되고 있는 효소 활성인 탈인산화에 특이적이었습니다. DTT는 SHP-234로 배양하는 동안 제공하는 환원 환경을 위해 사용되었습니다. 따라서 샘플과 실험 간의 일관된 효소 활성을 보장하기 위해 효소 분석에서 배양 환경을 수정하는 데 사용되는 화학 물질의 정확한 농도를 측정하는 것이 중요합니다. SHP-2는 재조합 단백질로 사용되었으며, 효소를 처리하는 여러 단계에서 온도를 조절하는 것이 중요합니다. 성공적인 분석을 위해서는 효소가 두 번 이상의 동결-해동 주기를 거치지 않아야 하며 각 샘플을 준비하는 동안 얼음 위에 있어야 합니다. 그런 다음 재조합 효소를 분석 요구 사항을 충족하기에 충분한 부피로 분취해야 합니다. 실제 배양 중에는 효소에 대한 최적의 온도(이 경우 37°C)에서 온도를 엄격하게 제어해야 하며, 배양 혼합물의 균질성을 보장하기 위해 로터에서 지속적으로 움직여야 합니다.
재조합 효소를 사용한 분석을 위한 전반적인 방법은 SHP-2와 표면에 표시된 인산화 기질 사이의 상호 작용에 특이적인 변형이 필요했습니다. 프로토콜을 다른 세포외 효소-기질 상호 작용에 적용하려면 사용된 염기서열, 활성 완충액 환경 및 검출에 사용되는 시약의 수정이 포함됩니다. 다른 kinase-substrate-phosphatase 상호작용을 분석하기 위해, adaptation은 kinase-substrate pair에 대한 단백질 서열을 보충 표 S1에 기술된 construct cassette의 해당 위치로 대체하는 것을 포함합니다. 적어도 관심있는 키나아제의 키나아제 도메인과 함께 기질의 단백질 서열은 플라스미드에 포함되어야 하며, 생성된 인산화기질을 표적으로 하는 인산가수분해효소는 재조합 단백질의 형태여야 합니다. LCK, CD28 및 SHP-2 간의 대표적인 상호 작용은 표적 효소로 세포 외에서 조사할 번역 후 변형 단백질을 생산하기 위한 도구로 구성 카세트에서 설계된 소포체 격리를 사용하는 예를 제공합니다. post-translational modification을 거칠 필요가 없는 관심 기질(예: 추가된 키나아제를 사용하여 세포 외에서 인산화될 수 있는 기질)은 구조체 카세트 내에서 쌍을 이루는 효소 없이 효모 표면에서 발현될 수 있습니다. 이 경우에, 보충표 S1 에 기재된 키나아제에 대한 단백질 서열은 구축물 카세트에 포함되는 기질의 서열만을 가지고 제거될 것이다. 우리는 이전 경험에 비추어 볼 때, 알려진 기질과 세린-트레오닌 키나아제의 공동 국소화로 인해 인산화되지 않은 기질이 표시되었으므로(Ezagui and Stern, 미발표 데이터), 세포외 포스파타아제를 적용하기 전에 성공적인 효소 변형에 대한 엄격한 테스트를 수행해야 합니다. 당사는 이전에 이 적격화 단계39에 도움이 될 수 있는 kinase-substrate co-localization에 대한 프로토콜을 발표했습니다.
Kinase 및 phosphatase는 종종 산화될 때 단백질 내부 또는 단백질 전체에 걸쳐 이황화 결합을 형성하는 짝을 이루지 않은 시스테인 잔기를 함유하고 있으며, 이는 구조적 변화로 인해 단백질의 촉매 활성을 방해할 수 있습니다40,41. 이 단백질 생화학에 대한 이해는 효소 변형을 위한 적절한 반응 환경을 결정하는 데 필수적입니다. 결과적으로, 사용된 재조합 단백질이 활성 상태를 유지하도록 하기 위해 환원제를 배양 환경에 보충해야 합니다. DTT는 이러한 목적으로 사용되는 일반적인 환원제이지만 분석의 농도를 최적화해야 합니다. 너무 높은 DTT 농도를 사용하면 Aga1p 및 Aga2p 앵커가 DTT42의 존재 하에서 감소하는 이황화 결합을 통해 서로 유지되기 때문에 효모 표면에 기판이 표시되는 것을 방해합니다. DTT의 농도는 기질의 표면 디스플레이에 해로운 영향을 미치지 않으면서 상대적인 최대 인산가수분해효소 활성을 허용하는 최소 농도로 조정되었습니다(42). 분석되는 효소에 대한 배양 환경은 이 분석에 사용될 때 효소 활성의 유지를 보장하기 위해 최적화되어야 합니다. 효소가 이 분석에 사용된 0.5mM DTT보다 상당히 강한 환원 환경을 필요로 하는 경우, 플랫폼은 표면 디스플레이의 감소로 인해 제한되며 원하는 특정 효소 분석에 최적이 아닐 수 있습니다. 유사하게, 이 프로토콜의 배양 단계 동안 사용된 2x 완충액은 SHP-2 활성을 촉진하는 허용 가능한 완충액에 대한 선행 연구로부터 포함되었으며, 이 분석에 사용되는 임의의 다른 효소에 대한 배양 완충액을 공식화하기 위해 유사한 연구가 수행되어야 한다34. 이러한 완충액을 만들기 위한 출발점에는 관심 효소의 성공적인 in vitro 적용을 위한 문헌 검색 또는 활성 완충액에 대한 효소 제조업체의 권장 사항이 포함될 수 있습니다. 사용 중인 재조합 효소는 허용 가능한 농도 및 배양 시간을 식별하기 위해 이 분석을 위해 특별히 적정되어야 하며, 이를 통해 데이터 수집 전에 표적 효소 활성이 발생할 수 있습니다.
이 프로토콜을 다른 유형의 효소-기질 상호 작용에 적용하려면 새로운 형광 검출 시약을 선택하고 감도를 위해 적정해야 합니다. 다른 연구에서는 프로테아제 처리 후 펩타이드 기질의 존재 또는 부재를 감지하기 위해 에피토프 태그 표적 항체를 사용하는 것23,25 및 히스톤 단백질에 대한 변형을 감지하기 위해 아세틸화에 민감한 항체를 사용하는 것을 포함하여 이에 대한 예를 입증했습니다43. 이러한 시약을 검증하려면 positive control(관심의 변형을 입증할 수 있는 것)과 negative control(관심의 modification이 없음을 입증할 수 있는 것)을 설정해야 합니다. 이는 이전에 관심 변형을 입증한 구조체의 효모 표면 표시를 통해 수행될 수 있으며, 경우에 따라 재조합 단백질 또는 펩티드의 고정화를 통해 확립될 수 있습니다. 예를 들어, 인산화(및 관심있는 다른 많은 번역 후 변형)의 경우, 알려진 서열의 펩타이드는 포스포티로신(양성 대조군) 또는 변형되지 않은 티로신(음성 대조군) 및 스트렙타비딘 코팅 비드에서 펩타이드의 고정화를 가능하게 하는 C-말단 비오틴과 합성할 수 있습니다. 펩타이드로 코팅된 비드는 변형 특이적 항체로 라벨링할 수 있으며 섹션 4에 설명된 것과 유사한 방법으로 유세포 분석을 사용하여 검출의 특이성 및 민감도를 평가할 수 있습니다. 항체의 다양한 희석액을 사용하여 양성 대조군의 최대 신호, 음성 대조군의 최소 신호를 제공하고 변형된 단백질당 충분한 항체 폴드 과잉(4.1단계의 메모에 있는 방정식 사용)과 얻은 항체 분취액당 수행할 실험 수에 대한 경제적 고려 사항의 균형을 유지하는 농도를 찾아야 합니다.
세포 외 효소 활성 분석을 위해 효모 표면 디스플레이 플랫폼의 용이성을 조정하기 위한 프로토콜에 대해 설명합니다. 이 방법은 배양 중 재조합 SHP-2에 의해 탈인산화되도록 효모 표면에 표시된 인산화된 CD28을 사용하여 시연되지만 사용되는 작업 완충액 및 효소-기질 쌍의 변형을 통해 많은 유형의 효소 변형에 대해 일반화할 수 있습니다.
공개
저자는 이 작업과 관련하여 공개할 이해 상충이 없습니다.
감사의 말
이 작업은 L.A.S.(CBET - 2339172)에 대한 NSF CAREER 상과 사우스 플로리다 대학교의 스타트업 펀드의 지원을 받았습니다.
그림 2A에서 Servier https://smart.servier.com/ 의 microtube-open-translucent 아이콘은 CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/ 에 따라 라이선스가 부여됩니다. 변형에는 완충액과 효모 세포의 추가(왼쪽)와 항체의 추가(가운데-오른쪽)가 포함됩니다.
그림 2A의 시험관, 인큐베이터 및 유세포 분석기는 개방형 액세스의 www.bioicons.com 통해 제공되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
참고문헌
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유