Method Article
Modifica enzimatica e citometria a flusso: valutazione delle proteine visualizzate sulla superficie del lievito
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, introduciamo un protocollo per l'utilizzo di substrati di lievito visualizzati sulla superficie per saggi di modifica enzimatica. La piattaforma è stata dimostrata utilizzando l'analisi dell'attività di defosforilazione della tirosina fosfatasi SHP-2 contro uno dei suoi substrati come saggio di modifica enzimatica rappresentativa.
Abstract
La visualizzazione della superficie del lievito è una strategia di collegamento genotipo-fenotipo che consente lo screening ad alto rendimento della funzione proteica. Tradizionalmente, il display della superficie del lievito è stato applicato all'evoluzione di nuove proteine leganti, con la citometria a flusso utilizzata per valutare e ordinare in base ai livelli di forza di legame. Recentemente, c'è stato un crescente interesse nell'applicazione del display di superficie del lievito per lo screening di modifiche enzimatiche di varianti di substrato, con modifiche additive (ad esempio, fosforilazione) o sottrattive (ad esempio, proteolisi) che forniscono un fenotipo leggibile mediante citometria a flusso. Tali modifiche vengono regolarmente applicate utilizzando la co-localizzazione intracellulare, ma la capacità di ottenere modificazioni enzimatiche extracellulari dei substrati visualizzati potrebbe aprire molte più reazioni allo studio. Qui, descriviamo le tecniche per la progettazione e l'applicazione di saggi di screening per la modifica enzimatica extracellulare ai substrati candidati visualizzati sulla superficie del lievito e la successiva valutazione mediante analisi di citometria a flusso. Forniamo questi protocolli nel contesto delle fosfatasi defosforilanti dei lieviti sottoposti a substrati contenenti residui di tirosina fosforilata e commentiamo come questo quadro applicato possa essere adattato allo sviluppo di saggi di screening per altre coppie enzima-substrato.
Introduzione
La comprensione delle interazioni tra gli enzimi e i loro bersagli è diventata un'area di ricerca sempre più interessante a causa della sua necessità nella caratterizzazione biologica delle vie che controllano l'omeostasi cellulare e lo sviluppo della malattia 1,2. Gli enzimi sono responsabili della catalisi di molte delle reazioni che mantengono la vita biologica, controllando percorsi necessari come il metabolismo cellulare 3,4, la segnalazione5 e persino processi fondamentali come la riparazione del genoma 6,7. A causa del loro ruolo in questi processi, le loro interazioni svolgono anche un ruolo nello sviluppo di molte malattie, poiché le deviazioni nella loro attività possono causare una grave disregolazione dell'attività cellulare, causando apoptosi o proliferazione di cellule tumorali dannose2. Lo studio dell'attività enzimatica ha avuto importanti applicazioni nello sviluppo di nuove terapie 8,9, che richiedono saggi personalizzati per ogni specifica interazione enzima-substrato10. Saggi enzimatici multipli sono stati stabiliti come protocolli standard per la valutazione e la caratterizzazione di queste interazioni. I saggi sviluppati per analizzare le interazioni enzimatiche sono classificati in saggi di rilevamento che monitorano il legame per l'attivazione/inibizione11 o saggi che monitorano la modifica del substrato da parte degli enzimi12.
Uno dei ruoli principali degli enzimi è la regolazione del comportamento cellulare. La trasduzione del segnale, la risposta intracellulare di una cellula a un innesco extracellulare13, è responsabile della sopravvivenza e della funzionalità cellulare. La proliferazione cellulare, la differenziazione e molti altri processi funzionali coinvolgono tutti vie di segnalazione con interazioni enzimatiche che le governano14,15. Gli enzimi catalizzano le modificazioni post-traduzionali, che spesso modulano le massicce reti di segnalazione responsabili della corretta trasmissione dei messaggi extracellulari16. La fosforilazione delle proteine è la più comune modificazione post-traduzionale, ubiquitaria nella segnalazione cellulare e in molte altre vie cellulari. Di conseguenza, le protein chinasi sono emerse come una percentuale significativa di potenziali bersagli terapeutici a causa del loro ruolo regolatore critico17. Le fosfatasi sono le molecole modulatorie naturali per i complessi di segnalazione cellulare a base di fosfato18,19, avendo la capacità di rimuovere i residui di fosfato dalle loro proteine bersaglio20. Nell'ultimo decennio, le fosfatasi sono diventate un importante bersaglio terapeutico per il trattamento del cancro21 e delle malattie infiammatorie22 sulla base del loro coinvolgimento nella regolazione delle vie di segnalazione a valle in diversi tipi di cellule. Insieme, le protein chinasi e le fosfatasi forniscono un'ampia gamma di interazioni, che possono essere studiate attraverso lo sviluppo di specifici protocolli di analisi enzimatica.
Il display della superficie del lievito è stato utilizzato come strumento per la caratterizzazione e la valutazione dell'attività enzimatica23,24. Fornisce una piattaforma ad alto rendimento per lo screening dei processi di modificazione post-traduzionale se combinata con strategie di sequestro del reticolo endoplasmatico25,26. Ciò consente alle coppie chinasi-substrato di essere co-localizzate e trattenute nel reticolo endoplasmatico attraverso il legame con i recettori KDEL27, dove la fosforilazione del substrato può avvenire a velocità maggiori a causa della vicinanza tra le chinasi e i loro bersagli. Il legame con il recettore KDEL è mediato da una sequenza di ritenzione del reticolo endoplasmatico FEHDEL C-terminale che ha dimostrato di avere una capacità di ritenzione più forte rispetto ad altre sequenze HDEL25,28. Il substrato fosforilato viene quindi ancorato alla superficie del lievito per la sua successiva valutazione attraverso la citometria a flusso29. Attualmente, non esistono protocolli generalizzabili stabiliti per la modifica enzimatica dei substrati visualizzati sulla superficie del lievito. Ampliamo le capacità di visualizzazione della superficie del lievito sfruttando le varianti del substrato fosforilato espresse extracellulare e modificandole attraverso la defosforilazione da parte della loro fosfatasi nota. L'analisi di citometria a flusso fornisce quindi una piattaforma per la valutazione fenotipica dei suddetti substrati attraverso la misurazione delle alterazioni della fosforilazione mediana come conseguenza dell'incubazione con la fosfatasi nota. Ciò fornisce un metodo adattabile per la modifica post-traduzionale delle proteine visualizzate sulla superficie, fornendo anche un metodo per l'analisi delle modifiche enzimatiche delle interazioni quando si utilizza la piattaforma di visualizzazione della superficie del lievito.
Presentiamo tecniche per lo sviluppo e l'applicazione di un saggio di modificazione enzimatica che descrive l'introduzione di un'interazione chinasi-substrato nella piattaforma di visualizzazione della superficie del lievito, la co-incubazione del substrato fosforilato espresso con una fosfatasi ricombinante e la successiva analisi dell'attività di defosforilazione attraverso la citometria a flusso. In questo rapporto, ciò si ottiene co-localizzando il dominio citoplasmatico del CD28 con la chinasi linfocitaria (LCK) nel reticolo endoplasmatico del lievito, seguito dalla visualizzazione del CD28 fosforilato sulla superficie del lievito e dalla successiva defosforilazione da parte della fosfatasi-2 contenente il dominio di omologia Src 2 (SHP-2). Un anticorpo anti-fosfotirosina pan (in questo studio, 4G10), che rileva residui di tirosina fosforilata in un'ampia varietà di sequenze peptidiche, viene utilizzato per la quantificazione del livello di fosforilazione in funzione del trattamento con fosfatasi. Il processo dettagliato fornisce un approccio generalizzabile per studiare le interazioni enzima-substrato; Un modo prospettico di studiare enzimi e substrati in modo purificato.
Protocollo
1. Crescita cellulare di plasmidi di lievito e induzione dell'espressione proteica
- Seguendo la ricetta descritta nella Tabella 1, preparare i terreni necessari per la crescita di lieviti non contenenti plasmidi (YPD), la crescita di cellule di lievito contenenti plasmidi (SD-CAA) e l'induzione dell'espressione proteica (SRG-CAA), nonché le piastre SD-CAA.
- Trasformare il DNA plasmidico del lievito contenente la coppia chinasi-substrato in cellule di lievito EBY-100 attraverso il metodo30,31 basato sui cationi di litio, comunemente adottato per l'uso nei kit di trasformazione del lievito di una varietà di produttori.
NOTA: L'elettroporazione32 o altre tecniche di trasformazione plasmidico di lievito preferite possono essere utilizzate a seconda del costrutto plasmidico da trasformare. - Preparare una provetta di coltura da 14 mL con 10 mL di terreno YPD. Inoculare le cellule EBY-100 e farle crescere in un incubatore agitatore a 30 °C, 300 giri/min fino a quando la coltura raggiunge una densità ottica (OD600 nm) di 0,8-1,0 (8 x 106- 1 x 107 cellule/mL).
NOTA: Il diametro esternodi 600 nm viene misurato preparando cuvette per campioni da 3 mL contenenti diluizioni 1:10 di colture di lievito nei rispettivi terreni e cuvette bianche da 3 mL contenenti i terreni utilizzati per le diluizioni dei campioni. Il programma OD600nm sullo spettrofotometro viene utilizzato per misurare prima la cuvetta bianca, quindi ogni cuvetta campione impostando la diluizione corrispondente preparata per ciascun campione. 1 OD600nm corrisponde a 1 × 107 lievito/mL. - Raccogliere le cellule centrifugando la coltura a 1.000 x g per 3 minuti e lavare con la1a soluzione di lavaggio fornita nel kit di trasformazione del lievito, o TE (10 mM Tris-HCl e 1,0 mM EDTA)31.
- Pellettare nuovamente le cellule a 1.000 x g per 3 minuti e risospendere in 1 mL del tampone di trasformazione fornito nel kit di trasformazione del lievito o in acqua sterile. Le celle devono essere aliquotate in aliquote da 50 μL e possono essere conservate a -80 °C per un massimo di 6 mesi.
- Per la trasformazione plasmidica, viene preparata un'aliquota per plasmide e scongelata con ghiaccio, quindi 0,5-1,5 μg di DNA plasmidico contenente il costrutto di visualizzazione del lievito vengono aggiunti direttamente alle cellule. Vengono aggiunti 0,5 mL della soluzione di trasformazione fornita nel kit di trasformazione del lievito, o 0,5 mL di una soluzione sterile di polietilenglicole al 50% e 0,1 M di LiOAc31. Combinare accuratamente la miscela di cellule, DNA plasmidico e soluzione di trasformazione mediante pipettaggio.
- Incubare staticamente la miscela di trasformazione per 30-60 minuti a 30 °C, la miscela a vortice a intervalli di 15 minuti. Raccogliere le cellule mediante centrifugazione a 1.000 × g per 3 min.
- Preparare una provetta per coltura da 14 mL con 4,5 mL di terreno SD-CAA. Risospendere le cellule contenenti il plasmide desiderato in 500 μL di SD-CAA e inoculare i 4,5 mL preparati.
- Facendo attenzione a non perforare l'agar, distribuire 50 μL dei 5 mL di coltura inoculata su una piastra SD-CAA e incubare staticamente a 30 °C per 48 ore per determinare l'efficienza di trasformazione.
- Incubare i 5 mL di coltura cellulare SD-CAA in un incubatore con agitazione a 30 °C, 300 giri/min per almeno 18 ore. Monitorare la densità ottica (OD600nm) dopo 16 ore e 20 ore. Una volta che il campione ha raggiunto una densità ottica sufficiente non superiore a 6, centrifugare la coltura per 3 minuti a 2.500 × g. Scartare il surnatante senza disturbare il pellet di lievito.
- Risospendere il pellet di lievito in SRG-CAA a un OD finale di600 nm inferiore a 1 (<1 × 107 lievito/mL).
- Incubare la coltura di lievito in un'incubatrice con agitazione a 30 °C, 300 giri/min per almeno 8 ore ma non più di 24 ore.
NOTA: L'induzione dell'espressione proteica nelle cellule di lievito può essere variata da 20 a 37 °C. 30 °C è adatto per la sintesi di coppie chinasi/substrato29,33, ma può essere regolato se ritenuto necessario per le proteine specifiche in studio. - Misura OD600nm per determinare la densità cellulare.
NOTA: A questo punto il protocollo può essere interrotto conservando le colture di lievito a 4 °C.
2. Biotinilazione dell'anticorpo anti-fosfotirosina 4G10
- Risospendere un flaconcino da 2 mg di PEG4-NHS-Biotina a una concentrazione finale di 5 mM aggiungendo 680 μL di PBS sterile.
NOTA: La risospensione di PEG4-NHS-Biotina deve essere eseguita di fresco immediatamente prima che venga eseguita la reazione di biotinilazione. NHS si idrolizza in soluzione acquosa. L'uso di PBS e fiale sterili è importante per la preparazione di reagenti per la conservazione a lungo termine e per l'uso in saggi basati su cellule per mitigare eventuali effetti potenziali dei contaminanti sulla vitalità dei reagenti o sui saggi sensibili eseguiti. - In base alla concentrazione di anticorpi 4G10, aggiungere 100 μg di anticorpo in un flaconcino sterile da 1,7 ml.
- Aggiungere 1 μl dei 5 mM PEG4-NHS-Biotina preparati dal passaggio 2.1 alla fiala contenente i 100 μg di anticorpo 4G10 per ottenere un rapporto molare tra biotina e anticorpo di 7,5:1. Pipettare delicatamente la miscela per omogeneizzare la reazione.
- Incubare la reazione a temperatura ambiente con rotazione costante per almeno 2 ore.
- Seguire il protocollo del produttore per le colonne di desalinizzazione centrifughe da 0,5 mL per sostituire il tampone dal 4G10 biotinilato (B-4G10) al PBS.
NOTA: Le colonne di desalinizzazione con un cutoff del peso molecolare (MWCO) di 7 kDa sono comunemente utilizzate per la biotinilazione degli anticorpi per consentire la rimozione della biotina non reagita e di altre piccole molecole mantenendo l'anticorpo più grande. - Diluire l'anticorpo B-4G10 a una concentrazione finale di 1 μM in PBSA (PBS con 1 g/L di albumina sierica bovina). Aliquotare l'anticorpo B-4G10 in volumi più piccoli per evitare ripetuti cicli di congelamento/scongelamento.
NOTA: Gli anticorpi biotinilati possono essere conservati a 4 °C per l'uso quotidiano fino a 3 mesi senza perdere una significativa efficienza. Conservare il resto delle aliquote non utilizzate a -20 °C per un massimo di 2 anni.
3. Defosforilazione dei substrati espressi sulla superficie cellulare del lievito
- Preparare la soluzione tampone di lavoro 2x come descritto in precedenza in letteratura34.
NOTA: Si consiglia 2x per facilitare la misurazione degli ingredienti necessari. - Preparare il tampone di lavoro per i campioni in una fiala da 1,7 mL diluendo la soluzione tampone 2x preparata al punto 3.1 1:2 in acqua deionizzata.
NOTA: Il volume totale di reazione per ciascun campione sarà di 20 μl e ogni campione richiederà tra 10 μl e 18 μl di tampone di lavoro. Preparare una quantità sufficiente di tampone di lavoro per tutti i campioni o i controlli. - Seguendo la preparazione del campione raccomandata descritta nella Tabella 2, etichettare i flaconcini da 1,7 ml con il nome del controllo o del campione corrispondente.
- Sulla base dell'OD600nm misurato nella fase 1.9, calcolare il volume di coltura necessario per recuperare due milioni (2 × 106) di cellule di lievito dalla coltura di lievito corrispondente per ciascun campione.
- Aggiungere il volume di coltura di lievito calcolato nel passaggio precedente nella fiala da 1,7 ml per ciascun campione.
- Centrifugare il flaconcino per 1 minuto a 4.500 × g. Rimuovere con cautela il surnatante utilizzando una micropipetta e gettarlo come rifiuto a rischio biologico.
- Risospendere le cellule pellettate in 1 mL di PBSA e ripetere il passaggio 3.6.
NOTA: È importante rimuovere la maggior quantità possibile di surnatante senza disturbare le celle pellettate. - Sulla base della sua concentrazione di riserva, calcolare il volume di SHP-2 umano ricombinante necessario per avere una concentrazione finale di 1.000 nM in un volume totale di reazione di 20 μL.
NOTA: L'SHP-2 ricombinante deve essere aliquotato in piccoli volumi in modo che qualsiasi proteina utilizzata in ciascun test non sia passata attraverso più di due cicli di congelamento-scongelamento. L'eventuale residuo di SHP-2 da un'aliquota dopo che tutti i campioni sono stati preparati per un saggio deve essere scartato.
Le concentrazioni di stock degli enzimi ricombinanti possono variare a seconda del numero di lotto. L'SHP-2 ricombinante è comunemente formulato a una concentrazione standard di 0,2-0,4 mg/mL. Per una concentrazione stock di 0,324 mg/mL di SHP-2, ciò corrisponde a una concentrazione stock di 4,696 μM (SHP-2 ha un peso molecolare di 69 kDa). 4,26 μL di SHP-2 in una reazione da 20 μL producono una concentrazione finale di reazione di 1.000 nM SHP-2. - Aggiungere 7,7 mg di DTT in 10 mL di acqua deionizzata preparata in un conico da 15 mL per creare una soluzione DTT da 5 mM. Se le limitazioni dell'apparecchiatura rendono difficile pesare i milligrammi, aggiungere 0,77 g di DTT a 10 mL di acqua deionizzata, quindi eseguire una diluizione 100x per creare la soluzione DTT da 5 mM utilizzata per il saggio.
NOTA: La soluzione DTT può essere preparata in soluzioni madre a concentrazione più elevata da diluire a 5 mM se non è possibile misurare quantità su scala millimetrica utilizzando l'attrezzatura disponibile. La soluzione di DTT deve essere preparata fresca prima della risospensione cellulare in tampone di lavoro a causa della sua tendenza all'idrolisi, che la rende instabile per lunghi periodi quando diluita in acqua. - Risospendere le cellule pellettate nel tampone di lavoro preparato al punto 3.2 in modo che il volume di reazione finale in ciascun campione o controllo sia di 20 μl.
NOTA: La quantità di tampone di lavoro aggiunta deve essere calcolata in base alla quantità di DTT (2 μL) e SHP-2 (calcolata al punto 3.8) che sarà presente in ciascun campione. - Aggiungere 2 μl della soluzione DTT da 5 mM preparata al punto 3.9 a ciascun campione o controllo per ottenere una concentrazione finale di reazione di 0,5 mM DTT.
- Aggiungere il volume di SHP-2 calcolato al punto 3.8 a ciascun campione per ottenere un volume finale di 20 μl e mescolare delicatamente con una micropipetta.
- Avvolgere i coperchi delle fiale dei campioni in parafilm per evitare perdite o contaminazioni incrociate.
- Incubare i campioni a 37 °C per 2 ore su un rotore a velocità costante.
- Rimuovere i campioni dal rotore e arrestare la reazione aggiungendo 1 mL di PBSA a ciascun campione.
- Ripetere il passaggio 3.6.
4. Marcatura cellulare e analisi in citometria a flusso di substrati defosforilati
- Risospendere i campioni del passaggio 3.16 in una miscela da 20 μl dei reagenti primari corrispondenti, come descritto nella Tabella 2. Incubare i campioni per 20 minuti a temperatura ambiente.
NOTA: Tutte le concentrazioni di reagenti utilizzate sono state calcolate in eccesso rispetto al numero di proteine espresse sulla superficie delle cellule di lievito. Il calcolo presuppone l'espressione di 10.000 proteine/cellula da tutti i 2 x 106 lieviti in un campione35, quando di routine solo ~50% di essi lo fa. La Tabella 3 mostra l'eccesso di rapporto dei reagenti di marcatura per ciascuno dei reagenti espresso nella Tabella 2, calcolato come segue:
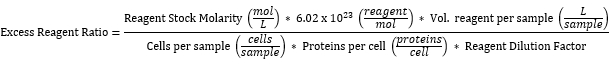
- Centrifugare i campioni a 4.500 × g per 1 minuto e scartare il surnatante come rifiuto a rischio biologico.
- Lavare le cellule una volta risospendendole in 1 mL di PBSA. Ripetere il passaggio 4.2.
- Risospendere i campioni in una miscela da 20 μl dei reagenti secondari corrispondenti, come descritto nella tabella 2. Incubare i campioni per 15 minuti in assenza di luce.
- Ripetere il passaggio 4.2.
- Ripetere il passaggio 4.3.
- Risospendere i campioni lavati in 300-500 μL di PBSA e trasferirli in provette di polistirene da 5 mL per analizzarli immediatamente utilizzando un citometro a flusso appropriato.
NOTA: Se è necessario trasportare campioni, tenerli su ghiaccio umido. Non è consigliato, ma i campioni possono essere conservati a 4 °C per un massimo di 2 ore come pellet umido. - Dopo aver eseguito l'avvio e la preparazione necessari del citometro per un nuovo esperimento, fare clic sul pulsante Nuovo esperimento nel menu File , assegnare un nome all'esperimento e fare clic su Salva per assicurarsi che i dati acquisiti vengano salvati nel percorso del file desiderato.
- Selezionare l'icona del grafico a punti nella barra degli strumenti superiore per creare due o più grafici a punti per ogni campione da eseguire. Per uno dei dot-plot, selezionare il nome dell'asse X per visualizzare il canale FSC-A e il nome dell'asse Y per visualizzare il canale SSC-A. Questo grafico mostra l'area Side Scatter rispetto all'area Forward Scatter e viene utilizzato per bloccare le cellule di lievito per ulteriori analisi.
- Su un altro dot-plot, selezionare il nome dell'asse X per visualizzare il canale in cui il reagente secondario che ha come bersaglio l'anticorpo primario anti-epitopo tag è fluorescente. Selezionare il nome dell'asse Y per visualizzare il canale in cui il reagente secondario della streptavidina diventa fluorescente. Questo grafico mostrerà solo gli eventi controllati dal lato rispetto al grafico di dispersione in avanti come cellule di lievito e viene utilizzato per visualizzare la fosforilazione della tirosina sull'asse Y e l'espressione della superficie del substrato sull'asse X.
NOTA: In questo esempio, il reagente secondario che ha come bersaglio l'anticorpo primario anti-epitopo è fluorescente nel canale FITC (AF-488) e il reagente secondario streptavidina è fluorescente nel canale AF-647. I canali utilizzati possono variare a seconda dei reagenti primari e secondari utilizzati durante l'etichettatura. - Posizionare ciascuna provetta del campione nel supporto della provetta del citometro e selezionare Esegui per il citometro per iniziare a caricare il campione e acquisire i dati. Regolare gli eventi da visualizzare, gli eventi da registrare, il tempo di registrazione e la portata del campione, se necessario.
- Definisci un cancello che circonda le cellule di lievito sane nel grafico SSC-A vs FSC-A creato nel passaggio 4.9. La Figura 1 illustra una rappresentazione descrittiva della strategia di gating da applicare.
NOTA: Sul grafico SSC-A rispetto a FSC-A, 100.000 eventi da visualizzare e 50.000 eventi da registrare all'interno del cancello di lievito definito sono una buona linea guida per visualizzare e raccogliere dati sufficienti per ulteriori analisi. Strategie di gating più rigorose per selezionare il lievito singoletto possono essere applicate sulla base di un grafico dell'altezza di dispersione in avanti rispetto all'area di dispersione in avanti, come recentemente riportato36. La strategia di gating mostrata nella Figura 1 corrisponde a un approccio meno rigoroso, che include singoletto e alcuni lieviti doppietti attraverso il gating a dispersione. - Registrare la fluorescenza di tutti i campioni di controllo utilizzando un analizzatore di citometria a flusso. I campioni di controllo vengono raccolti regolarmente per primi per aiutare a definire una strategia di gating, come descritto di seguito nel passaggio 4.14.
- Definire una strategia di gating per il grafico creato nel passaggio 4.10 prima di analizzare i campioni trattati. La Figura 1 illustra una rappresentazione descrittiva della strategia di gating da applicare.
- Registrare la fluorescenza dei campioni defosforilati utilizzando il citometro a flusso e la strategia di gating definita nei passaggi 4.12 e 4.14.
- Analizza i dati di citometria a flusso acquisiti utilizzando un software di analisi della citometria a flusso.
- Valutare la defosforilazione misurando e confrontando la mediana dell'asse Y delle cellule che esprimono proteine sulla loro superficie e la fosforilazione di base fornita dalle cellule non visualizzanti tra campioni e controlli. Calcolare la differenza di fosforilazione mediana percentuale come segue:

Risultati
L'analisi della citometria a flusso da una replica individuale del nostro sistema modello incubato per 2 ore senza (Figura 1A) e con (Figura 1B) 1.000 nM SHP-2 rivela una differenza di fosforilazione mediana del 63,6%, che è definita come il rapporto tra la mediana dell'asse Y (fosforilazione) di tutti gli eventi visualizzati sulla superficie meno il segnale di fosforilazione basale definito come la mediana dell'asse Y degli eventi non visualizzati tra il campione trattato e il controllo non trattato come descritto nell'equazione Definito nel passaggio del protocollo 4.12. Prima dell'analisi delle mediane, i campioni sono stati controllati in base alle loro dimensioni (Forward Scatter) e alla complessità (Side Scatter) per comprendere un gruppo sano di cellule. Nelle condizioni definite, la defosforilazione del campione dovrebbe essere distinguibile in bella vista (Figura 1C).
Il test è definito da una procedura semplice composta da quattro metodi principali (Figura 2A). Il sistema di visualizzazione della superficie del lievito che utilizziamo e che abbiamo pubblicato in precedenza si basa su un plasmide contenente un promotore bidirezionale con la capacità di espressione inducibile simultanea di una coppia chinasi-substrato costituita dalla coda citoplasmatica di CD28 e dalla tirosina chinasi LCK (Figura 2B)29. La co-localizzazione delle proteine tradotte è diretta da un reticolo endoplasmatico che ha come bersaglio il peptide segnale e la modificazione post-traduzionale è forzata dall'aumento del tempo di permanenza derivante da una sequenza di ritenzione del reticolo endoplasmatico C-terminale. La secrezione del substrato fosforilato fuso con Aga2p porta all'espressione superficiale (Figura 2C). Il substrato è progettato per essere affiancato da due tag epitopi, che consentono la conferma extracellulare del successo della traduzione e della successiva espressione superficiale. L'incubazione del substrato che mostra cellule di lievito con la fosfatasi di interesse (in questo caso, tirosina fosfatasi SHP-2) consente l'analisi della modificazione enzimatica attraverso la diminuzione del fosfato legato al substrato (Figura 2D).
Anche se sono state definite le condizioni ottimali per il sistema modello presentato, la generalizzabilità del saggio consente la diversificazione delle proteine da analizzare. Le condizioni ottimali per il saggio sono state definite attraverso una serie di titolazioni in cui sono state valutate diverse combinazioni di tempo e concentrazione di fosfatasi in quadruplicato (Figura 3). I dati analizzati hanno dimostrato la significatività statistica attraverso un'ANOVA bidirezionale con replicazione (p < 0,05). Le condizioni scelte di 2 h e 1.000 nM (48,8% ± 10%) hanno offerto una differenza di fosforilazione mediana di circa il 50%, pur mantenendo la significatività statistica rispetto alla sua controparte di 750 nM a 2 h (p < 0,05) sulla base di un'analisi t-test con varianza disuguale. Il test t non ha inoltre rivelato alcuna differenza significativa rispetto al risultato di 2 ore e 1.000 nM aumentando il tempo di 1 ora sotto una qualsiasi delle concentrazioni che fornivano una percentuale di differenza di fosforilazione mediana approssimativa (p > 0,05 per 500 nM, 750 nM e 1.000 nM a 3 ore).
L'analisi post hoc di Tukey HSD rivela che tutti i confronti medi tra i periodi di incubazione in tutte le concentrazioni sono significativamente diversi, a parte 1 h a 2 h. Quando si confrontano le concentrazioni multiple testate, si osservano solo differenze medie statisticamente significative quando si confrontano 250 nM con tutte le altre concentrazioni, indicando che livelli comparabili di attività della fosfatasi sono attesi all'interno dei gruppi, ad eccezione di 250 nM. Nonostante l'osservazione di una differenza del 20% quando i campioni sono stati trattati a 4 ore e 1.000 nM SHP-2 (22,1% ± 5,5%), rispetto alle condizioni ottimali (t-test, p < 0,05), abbiamo deciso di non perseguire questa combinazione a causa della ridotta espressione superficiale e della ridotta salute del lievito dovuta alla lunga incubazione con DTT. Ipotizziamo che ciò sia causato dalle condizioni riducenti del tampone di lavoro, necessario per la corretta funzione della fosfatasi SHP-2.

Figura 1: Analisi in citometria a flusso del sistema modello. Grafici di densità che mostrano la dispersione diretta (asse X) rispetto al gating di dispersione laterale (asse Y) (a sinistra) e grafici a punti che mostrano l'espressione della superficie attraverso l'etichettatura dell'etichetta dell'epitopo C-terminale del substrato (asse X) rispetto alla fosforilazione del substrato (asse Y) (centro) del dominio citoplasmatico CD28 incubato per 2 ore (A) senza SHP-2 e (B) con la concentrazione ottimale di SHP-2 definita come 1.000 nM. L'Y-mediana è stata misurata all'interno delle porte definite che comprendono le cellule espresse in superficie (verde) solo come misura di fosforilazione relativa. Gli eventi di segnale non visualizzati sono stati controllati per definire la misurazione Y-mediana di fondo (grigio). (C) Sovrapposizione di punteggi di campioni che mostrano una differenza di vista semplice in Y-mediana. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Modifica enzimatica delle proteine visualizzate sulla superficie del lievito. (A) Schema del saggio che mostra le fasi critiche all'interno dei quattro metodi descritti: preparazione di campioni con fosfatasi (tan) per il saggio enzimatico, incubazione per l'attività enzimatica desiderata, lavaggio e marcatura delle cellule per il rilevamento dell'attività, analisi della citometria a flusso e raccolta dei dati. (B) Schema genico che rappresenta una struttura generale della cassetta utilizzata per il sequestro del reticolo endoplasmatico di una coppia enzima-substrato e l'espressione superficiale del substrato. (C) Rappresentazione grafica della co-localizzazione del reticolo endoplasmatico di una coppia chinasi-substrato (a sinistra) seguita dalla secrezione del substrato modificato post-traduzionalmente visualizzato sulla superficie del lievito e marcato con un anticorpo anti-fosfotirosina (blu) e un anticorpo tag anti-epitopo (rosa) per confermare l'espressione superficiale (a destra). (D) L'incubazione di cellule di lievito con fosfatasi (tan) rimuove il gruppo fosfato dal substrato visualizzato, interrompendo la marcatura degli anticorpi anti-fosfotirosina, facilitando l'analisi delle modificazioni enzimatiche attraverso la citometria a flusso. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Attività della fosfatasi e titolazione nel tempo. Le cellule di lievito sono state esposte a più combinazioni di tempo e concentrazione di fosfatasi seguite da analisi di citometria a flusso. Tutti i campioni trattati sono stati confrontati con un controllo incubato per lo stesso periodo di tempo e condizioni tampone senza SHP-2. La differenza percentuale di fosforilazione mediana è stata definita come il rapporto tra la Y-mediana dagli eventi visualizzati in superficie meno il segnale di rumore di base fornito dalla Y-mediana negli eventi non visualizzabili in campioni contenenti SHP-2, diviso per la stessa relazione nel rispettivo controllo. L'ipotesi nulla viene rifiutata quando si osservano differenze significative confrontando i cambiamenti tra i gruppi di tempo e di concentrazione utilizzando l'ANOVA a due vie (p < 0,05). L'effetto delle variabili descritte sulla differenza percentuale di fosforilazione mediana è indipendente l'una dall'altra (Interazione p > 0,05). Il test HSD di Tukey è stato eseguito per l'analisi post hoc per ulteriori informazioni sulla significatività della differenza tra il tempo di incubazione complessivo e i gruppi di concentrazione, e una serie di t-test che assumono una varianza disuguale sono stati utilizzati per definire la significatività statistica dei singoli gruppi in un momento e una concentrazione specificati. I dati sono presentati come media ± deviazione standard di quattro repliche. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1: Linee guida per la preparazione dei terreni di crescita e induzione proteica del lievito. Descrizione tabellare della massa richiesta da ciascun componente chimico per formulare 1 litro di terreno di crescita selettivo, terreno di induzione selettiva delle proteine del lievito e terreno di estrazione di lievito peptone destrosio. Una volta che il mezzo descritto è stato miscelato correttamente, filtrare e sterilizzare prima del suo utilizzo. Sono incluse ulteriori istruzioni per la realizzazione di piastre per terreni di crescita selettivi per lievito. Clicca qui per scaricare questa tabella.
Tabella 2: Preparazione del campione consigliata per la defosforilazione del substrato e la strategia di marcatura degli anticorpi. Descrizione tabellare dei campioni necessari per la misurazione delle modificazioni enzimatiche dei substrati fosforilati visualizzati sulla superficie del lievito. La preparazione del campione e la successiva strategia di marcatura degli anticorpi sono specificate sia per i controlli richiesti che per ciascun campione da analizzare, comprese le corrispondenti diluizioni dei reagenti di marcatura. Clicca qui per scaricare questa tabella.
Tabella 3: Calcoli del rapporto tra anticorpi e eccesso di proteine. Descrizione tabellare del numero teorico di anticorpi disponibili per proteina espressi sulla superficie di una cellula di lievito durante la marcatura per citometria a flusso. Il numero teorico espresso presuppone che il 100% delle cellule di lievito esprima 10.000 proteine sulla loro superficie per garantire un eccesso di anticorpi e i calcoli del rapporto di eccesso si basano sulla concentrazione di stock di reagenti visualizzata nella tabella e acquisita dal fornitore. Clicca qui per scaricare questa tabella.
Tabella supplementare S1: Sequenza amminoacidica delle cassette dei costrutti. Rappresentazione tabellare della sequenza amminoacidica per le cassette dei costrutti situate su entrambi i lati del promotore Gal 1-10. Le sequenze evidenziate corrispondono alle loro descrizioni codificate a colori. Il lato Gal-10 del plasmide è rappresentato come una traduzione inversa della sequenza di amminoacidi per facilitarne la comprensione. Tutti i caratteri lasciati in nero corrispondono alla traduzione amminoacidica dei siti di digestione dell'enzima di restrizione utilizzati per fornire modularità al costrutto. Clicca qui per scaricare questo file.
Discussione
Il protocollo presentato consente l'analisi delle interazioni enzimatiche utilizzando la visualizzazione extracellulare delle proteine sulla superficie del lievito. L'incorporazione del sequestro del reticolo endoplasmatico nel plasmide surface-display utilizzato introduce la capacità di analizzare extracellulari interazioni specifiche tra enzimi e substrati modificati post-traduzionali a causa delle interazioni intracellulari che possono essere progettate per verificarsi27,29. I saggi di interazione enzimatica precedentemente stabiliti utilizzando il display di superficie del lievito coinvolgono l'espressione delle proteine di interesse a livello intracellulare, con il display di superficie che è esclusivamente uno strumento per il rilevamento delle interazioni intracellulari che si verificano tra le proteine bersaglio 23,25,29.
Questo protocollo si basa su quella piattaforma spostando le interazioni enzimatiche mirate nell'ambiente extracellulare, il che introduce una maggiore flessibilità sia negli enzimi che possono essere studiati, sia nell'ambiente in cui viene monitorata la loro attività. Le interazioni studiate che si verificano a livello extracellulare offrono ai ricercatori l'opportunità di adattare l'ambiente di incubazione per essere più ottimale per l'attività enzimatica, espandendo gli enzimi che potrebbero essere studiati le cui attività sono ostacolate nel reticolo endoplasmatico del lievito, che è un ambiente fortemente ossidante37. Inoltre, la capacità di titolare la concentrazione di enzimi rispetto a un dato substrato consente saggi di attività enzimatica specifici che non potrebbero essere eseguiti a livello intracellulare a causa dell'ipotizzata saturazione della velocità di reazione durante il sequestro.
All'interno del protocollo, ci sono diversi passaggi critici da notare per garantire che il risultato desiderato venga rispettato. La riuscita trasformazione dei costrutti studiati in lievito è essenziale per ottenere risultati ottimali dalle fasi successive. È necessario eseguire un monitoraggio accurato della densità ottica per monitorare la crescita sana delle colture e garantire che non crescano eccessivamente prima dell'induzione delle proteine o della preparazione del campione. La fase logaritmica della crescita delle cellule di lievito comprende il periodo in cui la produzione di proteine è più alta, mentre nella fase stazionaria, i meccanismi responsabili della produzione di proteine si arrestano38. Tenendo presente questo, le misurazioni della densità ottica forniscono una misurazione accurata delle fasi di crescita in cui si trovano le colture di lievito e fasi come l'induzione delle proteine e la preparazione per i saggi dovrebbero essere eseguite al di fuori della fase stazionaria o quando le colture diventano troppo cresciute (OD600 nm < 6).
Per il saggio di modifica enzimatica, l'ambiente di incubazione descritto era specifico per l'enzima studiato, SHP-2 e l'attività enzimatica che veniva eseguita, la defosforilazione. La DTT è stata utilizzata per l'ambiente riducente che fornisce durante l'incubazione con SHP-234. Pertanto, è importante misurare con precisione le concentrazioni delle sostanze chimiche utilizzate per modificare l'ambiente di incubazione nei saggi enzimatici per garantire un'attività enzimatica coerente tra i campioni e gli esperimenti. SHP-2 è stato utilizzato come proteina ricombinante ed è fondamentale regolare la temperatura durante le diverse fasi di manipolazione dell'enzima. Per un test di successo, l'enzima non deve essere stato sottoposto a più di due cicli di congelamento-scongelamento e deve essere in ghiaccio durante la preparazione di ciascun campione. È quindi imperativo aliquotare l'enzima ricombinante in un volume sufficiente per soddisfare i requisiti del saggio. Durante l'incubazione vera e propria, la temperatura deve essere rigorosamente controllata alla temperatura ottimale per l'enzima, 37 °C in questo caso, con un movimento costante da un rotore per garantire l'omogeneità della miscela di incubazione.
Il metodo generale per l'analisi con enzimi ricombinanti ha richiesto modifiche specifiche all'interazione tra SHP-2 e il substrato fosforilato visualizzato in superficie. L'adattamento del protocollo ad altre interazioni extracellulari enzima-substrato comporta la modifica delle sequenze utilizzate, dell'ambiente tampone di attività e del reagente utilizzato per il rilevamento. Per il saggio di altre interazioni chinasi-substrato-fosfatasi, l'adattamento comporta la sostituzione delle sequenze proteiche per una coppia chinasi-substrato nelle rispettive posizioni nella cassetta del costrutto descritta nella Tabella supplementare S1. La sequenza proteica del substrato insieme ad almeno il dominio chinasico della chinasi di interesse dovrebbe essere inclusa nel plasmide e la fosfatasi che ha come bersaglio il substrato fosforilato prodotto dovrebbe essere sotto forma di proteina ricombinante. L'interazione rappresentativa tra LCK, CD28 e SHP-2 fornisce un esempio di utilizzo del sequestro del reticolo endoplasmatico progettato nella cassetta del costrutto come strumento per la produzione di proteine modificate post-traduzionali da studiare extracellulare con il loro enzima target. I substrati di interesse che non necessitano di subire modifiche post-traduzionali (ad esempio, substrati che potrebbero essere fosforilati extracellulare utilizzando chinasi aggiunta) possono essere espressi sulla superficie del lievito senza un enzima accoppiato all'interno della cassetta del costrutto. In questo caso, la sequenza proteica per la chinasi descritta nella Tabella Supplementare S1 verrebbe rimossa con solo la sequenza del substrato inclusa nella cassetta del costrutto. Notiamo dalla nostra precedente esperienza che la co-localizzazione di una serina-treonina chinasi con un substrato noto ha portato alla visualizzazione di un substrato che non era fosforilato (Ezagui e Stern, dati non pubblicati), quindi è necessario condurre test rigorosi di modifica enzimatica riuscita prima di applicare la fosfatasi extracellulare. Abbiamo precedentemente pubblicato un protocollo per la co-localizzazione chinasi-substrato che potrebbe essere utile per questa fasedi qualificazione 39.
Le chinasi e le fosfatasi contengono spesso residui di cisteina spaiati che, una volta ossidati, formano legami disolfuro all'interno della proteina o attraverso le proteine, che possono interrompere l'attività catalitica della proteina a causa del cambiamento conformazionale40,41. La comprensione di questa biochimica delle proteine è essenziale per determinare l'ambiente di reazione appropriato per la modifica enzimatica. Di conseguenza, un agente riducente deve essere integrato nell'ambiente di incubazione per garantire che la proteina ricombinante utilizzata rimanga attiva. Il DTT è un agente riducente comune utilizzato per questi scopi, ma la concentrazione nel saggio deve essere ottimizzata. L'uso di una concentrazione troppo elevata di DTT ostacola la visualizzazione del substrato sulla superficie del lievito, poiché le ancore Aga1p e Aga2p sono tenute l'una all'altra attraverso legami disolfuro, che vengono ridotti in presenza di DTT42. La concentrazione di DTT è stata regolata alla concentrazione minima che consentirebbe un'attività fosfatasica massima relativa senza avere effetti dannosi sulla visualizzazione superficiale dei substrati42. L'ambiente di incubazione per qualsiasi enzima analizzato deve essere ottimizzato per garantire il mantenimento dell'attività enzimatica quando viene utilizzato in questo saggio. Se un enzima richiede un ambiente riducente considerevolmente più forte del DTT da 0,5 mM utilizzato in questo test, la piattaforma è limitata dalla riduzione della visualizzazione della superficie e potrebbe non essere ottimale per lo specifico test enzimatico desiderato. Allo stesso modo, il tampone 2x utilizzato durante la fase di incubazione in questo protocollo è stato incluso da ricerche precedenti sui tamponi accettabili che promuovono l'attività di SHP-2, e ricerche simili dovrebbero essere eseguite per la formulazione di un tampone di incubazione per qualsiasi altro enzima utilizzato per questo test34. I punti di partenza per la creazione di questi tamponi possono includere la ricerca in letteratura per applicazioni in vitro di successo dell'enzima di interesse o la raccomandazione del produttore dell'enzima per un tampone di attività. L'enzima ricombinante utilizzato deve essere titolato specificamente per questo test per identificare una concentrazione e un tempo di incubazione accettabili, che consentano l'attività enzimatica mirata prima della raccolta dei dati.
Per l'adattamento di questo protocollo ad altri tipi di interazione enzima-substrato, sarà necessario selezionare e titolare nuovi reagenti di rivelazione fluorescenti per la sensibilità. Altri studi hanno dimostrato esempi di ciò, tra cui l'uso di anticorpi mirati all'epitopo per rilevare la presenza o l'assenza di substrati peptidici dopo il trattamento con proteasi23,25 e anticorpi sensibili all'acetilazione per rilevare le modifiche alle proteine istoniche43. Per qualificare questi reagenti, devono essere stabiliti un controllo positivo (che dimostri in modo verificabile la modifica di interesse) e un controllo negativo (che dimostri in modo verificabile una mancanza di modifica di interesse). Ciò potrebbe essere fatto attraverso la visualizzazione sulla superficie del lievito di un costrutto che ha precedentemente dimostrato la modifica di interesse, o in alcuni casi potrebbe essere stabilito attraverso l'immobilizzazione di proteine o peptidi ricombinanti. Ad esempio, nel caso della fosforilazione (e di molte altre modificazioni post-traduzionali di interesse), peptidi di sequenza nota potrebbero essere sintetizzati con fosfotirosina (controllo positivo) o tirosina non modificata (controllo negativo) e una biotina C-terminale che consentirebbe l'immobilizzazione dei peptidi su perle rivestite di streptavidina. Le perle rivestite di peptidi possono essere marcate con l'anticorpo specifico per la modifica e valutate per la specificità e la sensibilità di rilevamento utilizzando la citometria a flusso con metodi simili a quelli descritti nella Sezione 4. Diverse diluizioni di anticorpi devono essere utilizzate per trovare una concentrazione che fornisca il segnale massimo del controllo positivo, il segnale minimo del controllo negativo e bilanci un sufficiente eccesso di ripiegamento dell'anticorpo per proteina modificata (utilizzando l'equazione nella nota del passaggio 4.1) con considerazioni economiche per il numero di esperimenti da condurre per aliquota di anticorpo ottenuto.
Descriviamo un protocollo per adattare la facilità della piattaforma di visualizzazione della superficie del lievito per i saggi di attività enzimatica extracellulare. Il metodo è stato dimostrato utilizzando CD28 fosforilato visualizzato sulla superficie del lievito per essere defosforilato da SHP-2 ricombinante durante l'incubazione, ma è generalizzabile per molti tipi di modificazione enzimatica attraverso la modifica del tampone di lavoro e della coppia enzima-substrato utilizzata.
Divulgazioni
Gli autori non hanno conflitti di interesse relativi a questo lavoro da divulgare.
Riconoscimenti
Questo lavoro è stato supportato da un premio NSF CAREER a L.A.S. (CBET - 2339172) e da fondi di avvio dalla University of South Florida.
Nella Figura 2A, l'icona microtube-open-translucent di Servier https://smart.servier.com/ è sotto licenza CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/. Le modifiche includono l'aggiunta di tampone e una cellula di lievito (a sinistra) e l'aggiunta di anticorpi (al centro a destra).
La provetta, l'incubatore e il citometro a flusso nella Figura 2A sono stati forniti tramite www.bioicons.com ad accesso libero.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
Riferimenti
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon