Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Pulse-chase Analyse des chaînes de sucre N-glycoprotéines liées à des cellules de mammifères
Dans cet article
Résumé
Nous décrivons une méthode pour l'analyse de l'altération des N-glycanes par le début de la vie des glycoprotéines après leur biosynthèse dans les cellules de mammifères. Ceci est réalisé par pulse-chase analyse des glycanes métaboliquement étiquetés, libération enzymatique des glycoprotéines et l'examen par HPLC.
Résumé
Fixation du Glc
Protocole
Le protocole suivant est destiné à l'analyse des chaînes de sucre des glycoprotéines totale ou d'une glycoprotéine d'intérêt spécifiques. La procédure est quasiment identique pour les deux cas, avec quelques modifications comme indiqué dans le protocole.
- Pour marquage métabolique des N-glycanes on a besoin d'utiliser une culture de cellules de mammifères subconfluentes cultivées pendant une nuit dans un plat de 90 mm pour chaque échantillon (les échantillons peuvent représenter différents points de temps ou de traitements). Ce protocole est optimisé pour les cellules NIH 3T3.
- Affamer les cellules pour le glucose par incubation dans 5 ml d'fraîchement préparés préalablement chauffé (37 ° C) sans glucose moyen, fourni avec 10% de FCS dialysées et 4 mM pyruvate de sodium pendant 5-15 minutes.
- Remplacez le support de famine avec 1 ml de pré-chauffée (37 ° C) sans glucose milieu contenant 400 pCi de [2 - 3 H] mannose-étiquetées (concentration plus faible de 65 pCi / ml est utilisé pour l'étiquetage des glycoprotéines total), et incuber les cellules dans les conditions normales de culture tissulaire pendant 1 heure.
- Retirez le support d'étiquetage de chaque échantillon, et ajoutez avec précaution 2 ml de PBS à 4 ° C à l'échantillons correspondant à impulsions et de les placer sur la glace (ces échantillons sont considérés comme des légumineuses), tandis que les échantillons correspondant à la chasse doivent être rincés trois fois avec 2 ml de la pré-chauffée (37 ° C) moyenne normale de culture complet, et ensuite placé dans un incubateur à CO 2 à 37 ° C avec 5 ml de pré-chauffé milieu régulier pour les périodes souhaitées chasse.
- Rincer les échantillons d'impulsion (précédemment mis sur la glace) 3 fois avec 2 ml de PBS glacé. Les cellules sont ensuite raclé le plat dans 2 ml de PBS en utilisant un grattoir à cellules, et placé dans un tube Eppendorf de 2 ml.
- Short-spin les cellules à 16000Xg (6-10 sec), éliminer le surnageant et congeler les cellules à -80 ° C à granulés
- Effectuez les étapes 5 et 6 également pour les échantillons courir à la fin de l'époque chasse.
- Retirer les cellules du congélateur et les lyser par addition de 300 ul de tampon A, vortex brièvement et incuber pendant 20 min sur la glace, ou dans le cas de l'analyse des glycoprotéines total, par incubation de 20 min sur la glace avec le tampon B, suivie par sonication (4 fois, 10 sec, amplitude maximale), puis faire bouillir les échantillons pendant 5min.
- Centrifuger les lysats à 16000Xg pendant 20 min à 4 ° C. Transférer le surnageant dans un nouveau tube Eppendorf et jeter le culot.
- Si l'analyse des glycoprotéines totales, procéder à la filtration moléculaire et déglycosylation (étape 17). Pour immunoprécipitation de la glycoprotéine H2a démontré ici, ajouter des 20l/sample (Repligen) protéine A-agarose perles 01h01 suspension et 3 pi / échantillon de nos anticorps de lapin polyclonal anti H2a pour la glycoprotéine souhaitée (dans notre exemple d'anticorps anti-H2a ), dans le surnageant de l'étape 9.
- Incuber les échantillons pendant 4-16 heures à 4 ° C, en permanence le mélange par rotation lente.
- Isoler les perles au 16000Xg pendant 30 secondes, et retirez soigneusement l'aspirateur surnageant à l'aide.
- Rincez les perles, en ajoutant 500 pi de tampon D et vortex. Spin comme dans l'étape 12 et retirer le surnageant. Répétez les 3 lavez fois.
- Ajouter 10 ul de tampon de dénaturation (fourni avec l'ONE endo-H kit) pour le culot de perles et faire bouillir les échantillons pendant 5 min (lorsque les protéines totales sont analysées, la déglycosylation est effectuée sur le filtre Microcon, étape 17).
- Isoler les perles (16000Xg pendant 30 secondes), et le transfert du surnageant dans un nouveau tube Eppendorf. Puis 0.5μl de l'endo H enzyme sont ajoutés à chaque échantillon avec 10 ul du tampon de réaction (également fourni avec le kit ONE endo H), et les échantillons sont incubés à 37 ° C pendant 3 heures.
- Pour séparer les glycanes libérés à partir des protéines, l'échantillon est dilué 5 fois avec de l'eau déminéralisée et placé sur le dessus d'un filtre moléculaire (Microcon Ultracel YM30), avec une coupure 30 kDa, puis centrifugés à 14000Xg pendant 3 min. Appliquer 50 pl d'eau déminéralisée et répétez la centrifugation des échantillons. Ce lavage est répété deux de plus, garder le flux continu.
- Si vous analysez les glycoprotéines totale, appliquer le surnageant des lysats (de l'étape 9) pour le filtre Microcon. Laver l'extrait avec 100 pi de tampon E, 3 fois par centrifugation répétée à 14000Xg pendant 3 min. Ajouter 3 pi de tampon de réaction 10X endo H et 1,5 pl de l'enzyme endo H au rétentat Microcon, et incuber pendant 3 heures à 37 ° C. Les glycanes libérés sont élués avec de l'eau déminéralisée par 3 centrifugations répétées, comme dans l'étape 16.
- Si désiré, rétentats des étapes 16 et 17, contenant l'endo H résistant glycoprotéines sont ensuite incubés avec 200mU de la N-glycosidase F dans 15 ul de tampon C à 37 ° C pendant 16 h. Élution avec de l'eau déminéralisée se fait comme dans l'étape 16.
- Placer le tube contenant la réaction endo H accréditives (les étapes 16 et 17) dans un concentrateur SpeedVacEt sécher les échantillons complètement (cela peut être fait jusqu'à 45 ° C pour accélérer ce processus).
- Pour préparer les échantillons pour l'HPLC, remettre en suspension les granulés secs dans 12 ul du solvant HPLC (acétonitrile: eau, 60:40, acide phosphorique à 1%).
- Réglez l'appareil CLHP (reliée à la colonne Spherisorb) au flux constant de solvant (1 ml / min) et de pression (une valeur particulière entre 1000 et 2000 psi), et placer un collecteur de fraction de changer un tube à chaque minute 1.
- Charger les échantillons dans l'appareil CLHP et démarrer le collecteur de fractions simultanément.
- Recueillir 48 fractions de 1 ml de chaque échantillon et exécuté d'une exécution d'un mélange standard oligosaccharides.
- Transférer 500 ul de chaque fraction dans un flacon à scintillation, et mélanger le contenu avec 3 ml de liquide scintillant miscible à l'eau.
- Charger les flacons et les lire dans un compteur à scintillation.
- La lecture cpm est tracée en fonction du nombre de fractions.
- Les valeurs cpm dans un pic de représenter une espèce spécifique glycane comme suit: 18 à 21 correspondent aux fractions M5, les fractions 22-26 à M6, les fractions 27-31 à M7, les fractions 32-35 à M8, les fractions 36-39 à M9, fractions 40-42 à 43-45 G1M9 et des fractions d'G2M9.
- La somme des valeurs cpm pour les fractions au sein de chaque pic reflète la quantité absolue d'une espèce glycane spécifiques. Afin de compenser le fait que les espèces contenant plusieurs résidus mannose acquérir plus de l'étiquette, le "montant molaire relative» de chaque espèce glycane est calculé comme suit: la quantité absolue de chaque espèce glycane est divisé par le nombre de résidus mannose qu'elle contient . Cette valeur est ensuite divisée par la somme des quantités relatives molaire de toutes les espèces glycane obtenu pour obtenir le "pour cent du total" pour chaque espèce glycane spécifiques.
| Chase (h) | 0 | 4 |
| Publié avec l'endo H (cpm) une | 90000 | 16800 |
| Sortie avec la N-glycosidase F (cpm) b | 20400 | 3900 |
| Glycoprotéines dans le rétentat (cpm) c | 371700 | 127695 |
| Dolichol-oligosaccharides dans le rétentat (cpm) d | 4350 | 540 |
| Dolichol-oligosaccharides dans le rétentat (%) e | 1,4 ± 0,7 | 0,2 ± 0,2 |
Tableau 1. Analyse de la libération des chaînes de sucre N-lié à deglycosidases et de la présence de dolichol-oligosaccharides dans les échantillons de glycoprotéine.
Nous avons appliqué la procédure de pulse-chase à un lysat de cellules NIH 3T3. Après filtration Microcon, étiquetés oligosaccharides N-liés ont été libérés principalement avec l'endo H après impulsion de l'étiquetage (type haute mannose) et avec d'autres traitements avec la N-glycosidase F après une période de chasse, ce qui correspondrait à un type complexe (Golgi-traitées glycanes), endo H oligosaccharides résistants. Cette analyse quantitative a déterminé que dolichol-oligosaccharides, représentaient une fraction insignifiante de l'étiquette dans le rétentat initiale.
Notes:
- Les cellules NIH 3T3 ont été étiquetés avec impulsions [2 - 3 H] mannose et pourchassé avec un milieu complet. Après la lyse des cellules et filtration à travers Microcon YM30, haute mannose oligosaccharides N-liés ont été libérés de rétentats par incubation avec l'endo H.
- Endo H matériau résistant au rétentats Microcon de (a) a ensuite été incubées avec la N-glycosidase F et publié oligosaccharides mesurée dans un compteur à scintillation.
- Rétentats Microcon obtenu comme dans (a) avant l'endo H traitement, ont été extraite 4 fois avec du chloroforme: méthanol: eau pour enlever 10:10:03 glycolipides précurseur, et le matériel non extraits solubilisés dans NaOH 1N et comptés dans un compteur à scintillation.
- Dolichol-oligosaccharides sont purifiés à partir d'extraits de (c) tel que décrit dans 1 et comptés dans un compteur à scintillation.
- Moyenne de 3 expériences pour les pour cent de dolichol-oligosaccharides (d) du total des cpm en rétentat (c + d).
Les résultats représentatifs
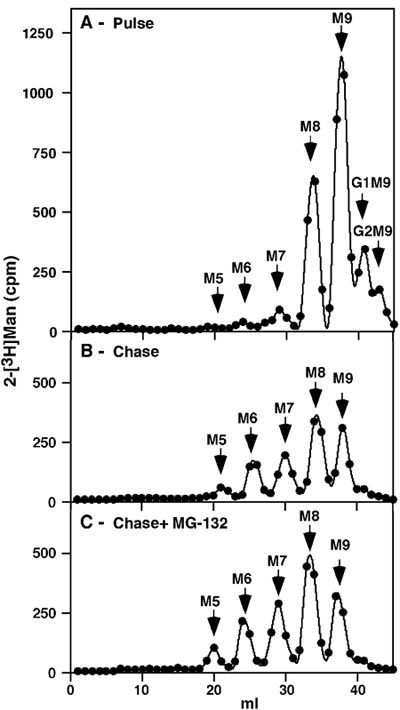
Figure 1. Pulse-chase analyse des glycoprotéines NIH 3T3 totale dans les cellules non traitées et sur l'inhibition du protéasome Après 1h d'impulsion-marquage avec. [2 - 3 H], nous avons obtenu un profil attendu avec une certaine glucosylésprécurseurs restants (2 Glc Man 9 GlcNAc 2 (G2M9) et G1M9), mais la plupart de l'étiquette présente dans M9 et M8 espèces, ce dernier étant le résultat d'une coupe de tous les résidus de glucose et d'une mannose (Figure 1). Non sans mannose ou d'autres précurseurs de petits ont été détectées, attestant de la rigueur de la purification. Après une assez longue course 8h il y avait plus vaste coupe à M7-6 et une petite quantité de M5, mais les principales espèces ont continué à être M9 et M8 (figure 1 B). Si la chasse a été fait en présence d'un inhibiteur du protéasome (30 uM MG-132), il y avait seulement une accumulation mineure de ces mêmes espèces taillés (figure 1 C).
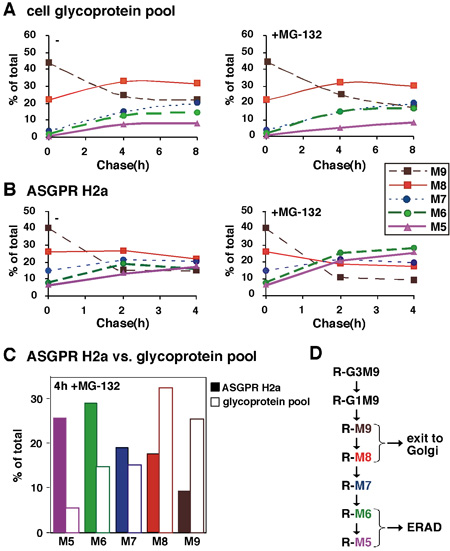
Figure 2. L'analyse quantitative des chaînes de sucre d'un substrat par rapport à ERAD glycoprotéines totales. (A) Dans une expérience similaire à celle de la figure 1, relative quantités molaires de chaque espèce d'oligosaccharides ont été calculés en fonction du contenu du mannose. Nous avons converti les valeurs cpm obtenus pour chaque espèce glycane, le pour cent de chaque espèce par rapport à la somme totale des montants relatifs molaire de tous les espèces présentes a ensuite été tracée en fonction du temps Chase pour une moyenne de deux expériences (B). Similaires à (A) sauf que les glycanes libérés de l'ERAD substrat ASGPR H2a ont été analysés après l'impulsion de l'étiquetage et chasser pour un maximum de 4h, en présence ou en absence de l'inhibiteur du protéasome MG-132. (C) Les valeurs de 4h chasse dans la présence de MG-132 de (A) et (B) sont comparés pour ASGPR H2a et la piscine glycoprotéine. (D) Schéma de la chaîne de sucre N-liés coupe processus dans les SU. Sucre processus qui mènent à la coupe M6-5 liés à des protéines (R) qui sont ciblés pour ERAD par rapport à M9-8 sur ceux qui la sortie à l'appareil de Golgi et au-delà. Les résultats indiquent que le processus ERAD est associée à la coupe du mannose N-glycanes pour produire des espèces avec 5-6 résidus mannose restantes.
Discussion
L'analyse pulse-chase de glycanes dans des cellules vivantes avec une séparation par HPLC fournit une méthode pour étudier la dynamique de l'oligosaccharide modifications structurelles à travers la vie d'une glycoprotéine. Il ya plus de preuves que ces altérations sont impliquées dans la production des signaux pour le pliage ER, contrôle de la qualité et la traite des systèmes de 2-5. La méthode peut être appliquée non seulement pour une glycoprotéine d'intérêt particuliers, ma...
Remerciements
Nous remercions Zehavit Frenkel et Sandra Tolchinsky d'assistance technique. Les recherches liées à ce travail est soutenu par des subventions de la Fondation Sciences d'Israël (1229-1207) et de la Coopération allemande Project-israélien (DIP-DFG).
matériels
| Name | Company | Catalog Number | Comments |
| 600E Multisolvent delivery System controller | Waters | WAT062710 | |
| Acetonitrile LiChrosolv (gradient grade for liquid chromatography) | Merck & Co., Inc. | 1.0003 | |
| Microcon Amicon Ultra 0.5 ml 30K or Centricon ultracel YM-30 | EMD Millipore | UFC503024 or 4208 | |
| Concentrator 5301, incl. 48 x 1.5 / 2.0 ml fixed-angle rotor | Eppendorf | 5301 000.016 | |
| Dialyzed F–tal Calf Serum | Biological Industries | 04-011-1A | |
| Dulbecco’s Modified Eagle’s Medium | GIBCO, by Life Technologies | 41965039 | |
| Dulbecco’s Modified Eagle’s Medium - glucose free | Sigma-Aldrich | D5030 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Sigma-Aldrich | D1408 | |
| EDTA disodium salt(Titriplex Iii) | Merck & Co., Inc. | 1084180250 | |
| Endo Hf Kit (1,000,000 units/ml) | New England Biolabs | p0703S | |
| F–tal Calf Serum (FCS) | Biological Industries | 04-001-1A | |
| Frac-100 fraction collector | Amersham | 18-1000-77 | |
| LS 6500 Liquid Scintillation Counting systems | Beckman Coulter Inc. | 510720 | |
| Mannose, D-[2-3H(N)]- (Specific Activity: 15-30Ci/mmol) | PerkinElmer, Inc. | NET570A | |
| N-carbobenzoxyl-leucinyl-leucinyl-leucinal (MG-132) | Calbiochem | 474790 | |
| N-glycosidase F (1000 units/ml) | Roche Group | 11365177 | |
| N-octylglucoside | Sigma-Aldrich | O3757 | |
| NIH 3T3 cells | American Type Culture Collection | CRL-1658 | |
| Opti-Flour | PerkinElmer, Inc. | 6013199 | |
| Phosphoric acid solution ( 49-51%, for HPLC) | Fluka | 79607 | |
| Protease Inhibitor Cocktail | Sigma-Aldrich | P2714 | |
| Protein A-Sepharose beads | Repligen | IPA300 | |
| Rabbit polyclonal anti-H2 carboxy-terminal 6 | |||
| Sodium deoxycholate | Sigma-Aldrich | 30970 | |
| Sodium dodecyl sulfate | Bio-Rad | 161-0301 | |
| Sodium phosphate | Sigma-Aldrich | 342483 | |
| Sodium pyruvate solution (100mM) | Sigma-Aldrich | S8636 | |
| Spherisorb NH2 Column, 5 μm, 4.6 x 250 mm | Waters | PSS831115 | |
| Triton X-100 | VWR | 306324N | |
| Vibra-Cell ultrasonic processors VCX 750 | Sonics and Materials, Inc. | 690-003 | |
| Buffer A: 1% Triton X-100, 0.5% w/v sodium deoxycholate, Protease inhibitor cocktail 2% v/v in PBS | |||
| Buffer B: 0.5% w/v SDS, Protease inhibitor cocktail 2% v/v in PBS | |||
| Buffer C: N-glycosidase F reaction buffer: 200mM Na3PO4, pH 8.0, 4mM EDTA, 1.5% N-octylglucoside | |||
| NIH 3T3 stably expressing H2a 6 | |||
| Buffer D: 0.5% Triton X-100, 0.25% w/v sodium deoxycholate,0.5% w/v SDS, Protease inhibitor cocktail 2% v/v in PBS | |||
| Buffer E: 0.5% w/v SDS, 1% v/v 2-Mercapt–thanol, Protease inhibitor cocktail 2% v/v in PBS | |||
| 2-mercapt–thanol | Sigma-Aldrich | M3148 |
Références
- Parodi, A. J., Behrens, N. H., Leloir, L. F., Carminatti, H. The role of polyprenol-bound saccharides as intermediates in glycoprotein synthesis in liver. Proc Natl Acad Sci U S A. 69 (11), 3268-3272 (1972).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Frenkel, Z., Gregory, W., Kornfeld, S., Lederkremer, G. Z. Endoplasmic reticulum-associated degradation of mammalian glycoproteins involves sugar chain trimming to Man6-5GlcNAc2. J Biol Chem. 278 (36), 34119-34124 (2003).
- Hosokawa, N. Enhancement of endoplasmic reticulum (ER) degradation of misfolded Null Hong Kong alpha1-antitrypsin by human ER mannosidase I. J Biol Chem. 278 (28), 26287-26294 (2003).
- Jakob, C. A., Burda, P., Roth, J., Aebi, M. Degradation of misfolded endoplasmic reticulum glycoproteins in Saccharomyces cerevisiae is determined by a specific oligosaccharide structure. J Cell Biol. 142 (5), 1223-1233 (1998).
- Tolchinsky, S., Yuk, M. H., Ayalon, M., Lodish, H. F., Lederkremer, G. Z. Membrane-bound versus secreted forms of human asialoglycoprotein receptor subunits. Role of a juxtamembrane pentapeptide. J Biol Chem. 271 (24), 14496-14503 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon