È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Pulse-chase Analisi di N-linked catene di zuccheri da glicoproteine in cellule di mammifero
In questo articolo
Riepilogo
Descriviamo un metodo per l'analisi dell'alterazione di N-glicani legati attraverso la vita inizi di glicoproteine dopo la loro biosintesi in cellule di mammifero. Ciò si ottiene pulse-chase analisi di metabolicamente glicani etichettati, rilascio enzimatico da glicoproteine e l'esame mediante HPLC.
Abstract
Fissaggio del Glc
Protocollo
Il seguente protocollo è stato progettato per l'analisi delle catene di zuccheri di glicoproteine totale o di una glicoproteina di interesse specifico. La procedura è sostanzialmente la stessa per entrambi i casi con alcune modifiche come affermato in tutto il protocollo.
- Per l'etichettatura metabolico di N-glicani legati uno ha bisogno di usare una cultura subconfluent di cellule di mammifero coltivate durante la notte in un piatto da 90 mm per ogni campione (i campioni possono rappresentare diversi momenti o trattamenti). Questo protocollo è ottimizzato per le cellule NIH 3T3.
- Affamare le cellule di glucosio da parte incubazione in 5 ml di preparati al momento pre-riscaldato (37 ° C) glucosio senza media, in dotazione con il 10% FCS dializzati e 4 mM di piruvato di sodio per 5-15 minuti.
- Sostituire il supporto fame con 1 ml di pre-riscaldato (37 ° C) glucosio senza supporto contenente 400 μCi di [2 - 3 H]-marcato mannosio (la concentrazione più bassa di 65 μCi / ml viene usato per l'etichettatura di glicoproteine totale), e incubare le cellule in condizioni normali coltura dei tessuti per 1 ora.
- Rimuovere il supporto di etichettatura da ogni campione, ed aggiungere con cautela 2 ml di PBS a 4 ° C per i campioni corrispondenti a impulsi e disporli sul ghiaccio (questi campioni sono indicati come legumi), mentre i campioni corrispondenti alla caccia devono essere risciacquati 3 volte con 2 ml di pre-riscaldato (37 ° C) media normale cultura completa, e quindi posto in un incubatore CO 2 a 37 ° C con 5 ml di pre-riscaldato medio regolare per i periodi di caccia desiderato.
- Sciacquare i campioni di impulsi (precedentemente immessi sul ghiaccio) 3 volte con 2 ml di PBS ghiacciata. Le cellule sono poi raschiato il piatto in 2 ml di PBS usando un raschietto cellula, e messo in una provetta da 2 ml Eppendorf.
- Short-spin le cellule a 16000Xg (6-10 sec), scartare il supernatante e congelare il pellet di cellule a -80 ° C.
- Eseguire i punti 5 e 6 anche per i campioni inseguimento al termine del tempo di caccia.
- Rimuovere le cellule dal congelatore e lisare loro con l'aggiunta di 300 ml di tampone A, vortex brevemente e incubando per 20 minuti sul ghiaccio, o nel caso di totale analisi glicoproteine, mediante incubazione 20 min in ghiaccio con tampone B, seguita dalla sonicazione (4 volte, 10 sec, massima ampiezza), poi far bollire i campioni per 5 minuti.
- Centrifugare i lisati a 16000Xg per 20 minuti a 4 ° C. Trasferire il surnatante in una nuova provetta Eppendorf e scartare il pellet.
- Se l'analisi di glicoproteine totale, procedere alla filtrazione molecolare e deglycosylation (punto 17). Per immunoprecipitazione della glicoproteina H2a dimostrato qui, aggiungere 20l/sample di (Repligen) proteina A-agarosio perline 01:01 sospensione e 3 ml / campione del nostro coniglio policlonale anticorpo anti H2a per la glicoproteina desiderato (nel nostro esempio anticorpi anti-H2a ), per il surnatante dal punto 9.
- Incubare i campioni per 4-16 ore a 4 ° C, mescolando continuamente con rotazione lenta.
- Spin giù le perle al 16000Xg per 30 secondi, e rimuovere con attenzione il vuoto surnatante utilizzando.
- Sciacquare le perline, con l'aggiunta di 500 microlitri di buffer D e vortex. Spin, come nel passaggio 12 e rimuovere il surnatante. Ripetere il lavaggio per 3 volte.
- Aggiungere 10μl di buffer denaturazione (fornito con il endo-H NEB kit) al pellet tallone e bollire i campioni per 5 minuti (quando le proteine totali sono analizzati, il deglycosylation viene eseguita sul filtro microcontrollore, punto 17).
- Spin giù le perline (16000Xg per 30 secondi), e trasferire il surnatante in un nuovo tubo Eppendorf. Poi 0.5μl di enzima endo H vengono aggiunti ad ogni campione con 10 ml di buffer di reazione (anche in dotazione con il kit endo ONA H), ei campioni sono incubati a 37 ° C per 3 ore.
- Per separare i glicani rilasciati dalle proteine, il campione viene diluito 5 volte con acqua deionizzata e posto sulla cima di un filtro molecolare (microcontrollore Ultracel YM30), con un 30kDa cut-off, poi centrifugato a 14000Xg per 3 min. Applicare 50μl di acqua deionizzata e ripetere la centrifugazione dei campioni. Questo lavaggio viene ripetuto altre 2 volte, mantenendo la flow-through.
- Se si sta analizzando glicoproteine totale, applicare il sovranatante della lisati (dal punto 9) per il filtro microcontrollore. Lavare l'estratto con 100 l di tampone E, 3 volte per centrifugazione ripetuto a 14000Xg per 3 min. Aggiungere 3 ml di buffer di reazione 10X endo H e 1,5 l di enzima endo H al retentato microcontrollore, e incubare per 3 ore a 37 ° C. I glicani rilasciati vengono eluiti con acqua deionizzata da 3 centrifugazioni ripetute, come al punto 16.
- Se lo si desidera, retentates da passaggi da 16 e 17, contenenti endo H-resistenti glicoproteine vengono poi incubate con 200mU di N-glicosidasi F in 15 ml di tampone C, a 37 ° C per 16 h. Eluizione con acqua deionizzata è fatto come nel passaggio 16.
- Collocare la provetta contenente la reazione endo H flow-through (passaggi da 16 e 17) in un concentratore SpeedVacE asciugare completamente i campioni (questo può essere fatto fino a 45 ° C per accelerare questo processo).
- Per preparare i campioni per HPLC, risospendere il pellet asciutto in 12 ml di solvente per HPLC (acetonitrile: acqua, 60:40, acido fosforico 1%).
- Regolare il dispositivo di HPLC (collegato alla colonna Spherisorb) a flusso costante del solvente (1 ml / min) e pressione (un particolare valore tra 1000 e 2000 psi), e inserire un raccoglitore di frazioni di cambiare un tubo ogni 1 minuto.
- Caricare i campioni nel dispositivo HPLC e avviare il raccoglitore di frazioni contemporaneamente.
- Raccolgono 48 frazioni di 1 ml di ogni campione e correre da una corsa di una miscela standard di oligosaccaridi.
- Trasferire 500 microlitri di ogni frazione di una fiala di scintillazione, e mescolare il contenuto con 3 ml di acqua-liquido miscibile scintillazione.
- Caricare le fiale e leggere in un contatore a scintillazione.
- La lettura cpm viene tracciata in funzione del numero di frazione.
- I valori di cpm all'interno di un picco di rappresentare una determinata specie glycan come segue: frazioni 18-21 corrispondono a M5, M6 frazioni di 22-26, 27-31 frazioni di M7, M8 per 32-35 frazioni, frazioni 36-39 a M9, frazioni 40-42 a 43-45 G1M9 e le frazioni di G2M9.
- La somma dei valori di cpm per le frazioni all'interno di ogni picco riflette la quantità assoluta di una specie glicano specifico. Al fine di compensare il fatto che le specie che contengono più residui di mannosio acquisire più etichetta, la "quantità relativa molare" di ogni specie glicano è calcolato come segue: la quantità assoluta di ciascuna specie glicano è diviso per il numero di residui di mannosio che contiene . Questo valore viene quindi diviso per la somma degli importi relativi molare di tutte le specie glicano ottenute per ottenere il "per cento del totale" per ciascuna specie glycan specifico.
| Chase (h) | 0 | 4 |
| Rilasciato con endo H (cpm) un | 90000 | 16800 |
| Rilasciato con N-glicosidasi F (cpm) b | 20400 | 3900 |
| Glicoproteine in retentato (cpm) c | 371700 | 127695 |
| Dolichol-oligosaccaridi a retentato (cpm) d | 4350 | 540 |
| Dolichol-oligosaccaridi a retentato (%) e | 1,4 ± 0,7 | 0,2 ± 0,2 |
Tabella 1. Analisi del rilascio di N-linked catene di zuccheri con deglycosidases e della presenza di dolichol-oligosaccaridi in campioni glicoproteina.
Abbiamo applicato la procedura di pulse-chase ad un lisato di cellule NIH 3T3. Dopo filtrazione microcontrollore, etichettato oligosaccaridi N-linked sono stati rilasciati principalmente con endo H dopo impulso etichettatura (alta tipo di mannosio) e con un ulteriore trattamento con N-glicosidasi F dopo un periodo di caccia, il che corrisponderebbe a tipo complesso (Golgi-lavorati glicani), endo H oligosaccaridi resistenti. Questa analisi quantitativa ha determinato che dolichol-oligosaccaridi, rappresentano una frazione insignificante del marchio nel retentato iniziale.
Note:
- Cellule NIH 3T3 sono stati etichettati con impulsi [2 - 3 H] mannosio e inseguito con terreno completo. Dopo la lisi cellulare e filtrazione attraverso microcontrollore YM30, alta mannosio oligosaccaridi N-linked sono stati rilasciati da retentates da incubazione con endo H.
- Endo H-materiale resistente in retentates microcontrollore da (a) è stato poi incubato con N-glicosidasi F e rilasciato oligosaccaridi misurata in un contatore a scintillazione.
- Retentates microcontrollore ottenuto come in (a) prima del trattamento endo H, sono stati estratti 4 volte con cloroformio: metanolo: acqua 10:10:03 rimuovere glicolipidi precursore, e il materiale non estratto solubilizzate in NaOH 1N e contate in un contatore a scintillazione.
- Dolichol-oligosaccaridi sono state purificate da estratti (c), come descritto al punto 1 e contate in un contatore a scintillazione.
- Media di 3 esperimenti per cento dei dolichol-oligosaccaridi (d) del cpm totale retentato (c + d).
Rappresentante Risultati
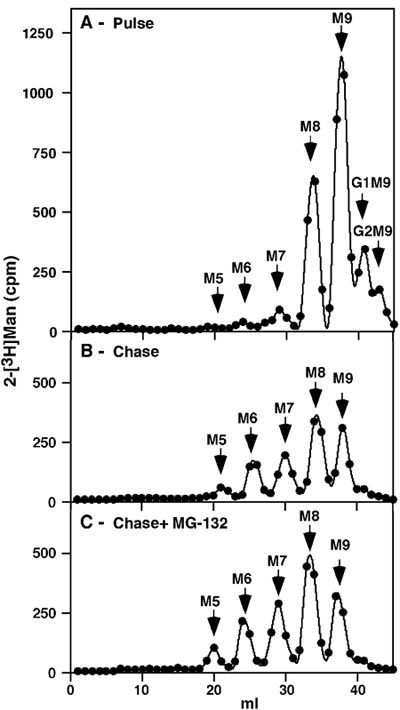
Figura 1. Pulse-chase analisi del totale delle glicoproteine NIH 3T3 in cellule non trattate e sulla inibizione del proteasoma Dopo 1h impulsi etichettatura con. [2 - 3 H] abbiamo ottenuto un profilo atteso con una certa glucosilatiprecursori rimanenti (Glc 2 Man 9 GlcNAc 2 (G2M9) e G1M9), ma la maggior parte delle etichette presenti nella M9 e M8 specie, quest'ultima essendo il risultato di taglio di tutte le glucosio e un residuo mannosio (Figura 1 A). Nessun libero precursori mannosio o altri piccoli sono stati rilevati, che attesta la completezza della purificazione. A seguito di un inseguimento piuttosto lungo 8h c'era più ampio taglio di M7-6 e una piccola quantità di M5, ma la specie più importanti hanno continuato ad essere M9 e M8 (Figura 1 B). Se l'inseguimento è stato fatto in presenza di un inibitore del proteasoma (30 mM MG-132), c'era solo un minore accumulo di queste stesse specie rifilato (Figura 1 C).
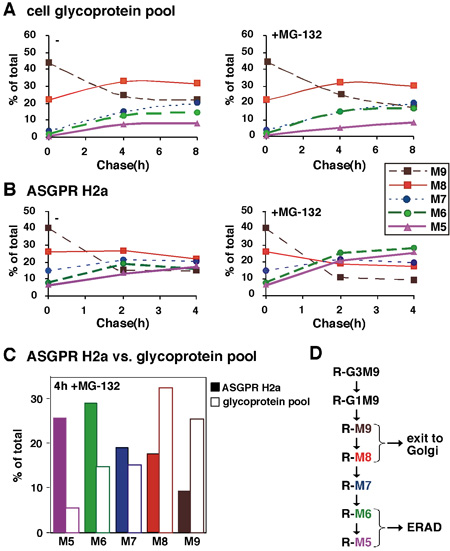
Figura 2. Analisi quantitativa delle catene di zuccheri di un substrato ERAD glicoproteine rispetto al totale. (A) In un esperimento simile a quello nella Figura 1, relativa quantità molare di ogni specie oligosaccaridi sono stati calcolati in base al contenuto mannosio. Abbiamo convertito i valori di cpm ottenuti per ogni specie glicani, la percentuale di ciascuna specie rispetto alla somma totale degli importi relativi molare di tutte le specie presenti è stata poi rappresentata in funzione del tempo la caccia per una media di due esperimenti. (B) simili a (A), tranne che glicani rilasciati dal substrato ERAD ASGPR H2a sono stati analizzati dopo l'etichettatura di impulsi e la caccia per un massimo di 4 ore, in presenza o assenza di inibitori del proteasoma MG-132. (C) Valori per 4h caccia in presenza di MG-132 da (A) e (B) vengono confrontati per ASGPR H2a e la piscina glicoproteina. (D) Schema di N-linked catena zucchero taglio processi al pronto soccorso. Processi di zucchero taglio che portano alla M6-5 legati alle proteine (R) che sono indirizzate a ERAD rispetto a M9-8 su quelle che escono al Golgi e oltre. I risultati indicano che il processo ERAD è associato con il mannosio taglio di N-glicani cedere specie con 5-6 residui di mannosio rimanenti.
Discussione
L'impulso-chase analisi dei glicani in cellule vive con la separazione HPLC fornisce un metodo per studiare la dinamica di oligosaccaridi alterazioni strutturali per tutta la vita di una glicoproteina. È sempre più evidente che tali alterazioni sono coinvolte nella produzione dei segnali per la piegatura ER, controllo qualità e il traffico di 2-5 sistemi. Il metodo può essere applicato non solo per una glicoproteina di interesse specifico, ma anche per analizzare le dinamiche della struttura di glicop...
Riconoscimenti
Ringraziamo Zehavit Frenkel e Sandra Tolchinsky per l'assistenza tecnica. Ricerca nel settore di questo lavoro è supportato anche da finanziamenti la Israel Science Foundation (1229-1207) e il tedesco-israeliano di cooperazione del progetto (DIP-DFG).
Materiali
| Name | Company | Catalog Number | Comments |
| 600E Multisolvent delivery System controller | Waters | WAT062710 | |
| Acetonitrile LiChrosolv (gradient grade for liquid chromatography) | Merck & Co., Inc. | 1.0003 | |
| Microcon Amicon Ultra 0.5 ml 30K or Centricon ultracel YM-30 | EMD Millipore | UFC503024 or 4208 | |
| Concentrator 5301, incl. 48 x 1.5 / 2.0 ml fixed-angle rotor | Eppendorf | 5301 000.016 | |
| Dialyzed F–tal Calf Serum | Biological Industries | 04-011-1A | |
| Dulbecco’s Modified Eagle’s Medium | GIBCO, by Life Technologies | 41965039 | |
| Dulbecco’s Modified Eagle’s Medium - glucose free | Sigma-Aldrich | D5030 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Sigma-Aldrich | D1408 | |
| EDTA disodium salt(Titriplex Iii) | Merck & Co., Inc. | 1084180250 | |
| Endo Hf Kit (1,000,000 units/ml) | New England Biolabs | p0703S | |
| F–tal Calf Serum (FCS) | Biological Industries | 04-001-1A | |
| Frac-100 fraction collector | Amersham | 18-1000-77 | |
| LS 6500 Liquid Scintillation Counting systems | Beckman Coulter Inc. | 510720 | |
| Mannose, D-[2-3H(N)]- (Specific Activity: 15-30Ci/mmol) | PerkinElmer, Inc. | NET570A | |
| N-carbobenzoxyl-leucinyl-leucinyl-leucinal (MG-132) | Calbiochem | 474790 | |
| N-glycosidase F (1000 units/ml) | Roche Group | 11365177 | |
| N-octylglucoside | Sigma-Aldrich | O3757 | |
| NIH 3T3 cells | American Type Culture Collection | CRL-1658 | |
| Opti-Flour | PerkinElmer, Inc. | 6013199 | |
| Phosphoric acid solution ( 49-51%, for HPLC) | Fluka | 79607 | |
| Protease Inhibitor Cocktail | Sigma-Aldrich | P2714 | |
| Protein A-Sepharose beads | Repligen | IPA300 | |
| Rabbit polyclonal anti-H2 carboxy-terminal 6 | |||
| Sodium deoxycholate | Sigma-Aldrich | 30970 | |
| Sodium dodecyl sulfate | Bio-Rad | 161-0301 | |
| Sodium phosphate | Sigma-Aldrich | 342483 | |
| Sodium pyruvate solution (100mM) | Sigma-Aldrich | S8636 | |
| Spherisorb NH2 Column, 5 μm, 4.6 x 250 mm | Waters | PSS831115 | |
| Triton X-100 | VWR | 306324N | |
| Vibra-Cell ultrasonic processors VCX 750 | Sonics and Materials, Inc. | 690-003 | |
| Buffer A: 1% Triton X-100, 0.5% w/v sodium deoxycholate, Protease inhibitor cocktail 2% v/v in PBS | |||
| Buffer B: 0.5% w/v SDS, Protease inhibitor cocktail 2% v/v in PBS | |||
| Buffer C: N-glycosidase F reaction buffer: 200mM Na3PO4, pH 8.0, 4mM EDTA, 1.5% N-octylglucoside | |||
| NIH 3T3 stably expressing H2a 6 | |||
| Buffer D: 0.5% Triton X-100, 0.25% w/v sodium deoxycholate,0.5% w/v SDS, Protease inhibitor cocktail 2% v/v in PBS | |||
| Buffer E: 0.5% w/v SDS, 1% v/v 2-Mercapt–thanol, Protease inhibitor cocktail 2% v/v in PBS | |||
| 2-mercapt–thanol | Sigma-Aldrich | M3148 |
Riferimenti
- Parodi, A. J., Behrens, N. H., Leloir, L. F., Carminatti, H. The role of polyprenol-bound saccharides as intermediates in glycoprotein synthesis in liver. Proc Natl Acad Sci U S A. 69 (11), 3268-3272 (1972).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Frenkel, Z., Gregory, W., Kornfeld, S., Lederkremer, G. Z. Endoplasmic reticulum-associated degradation of mammalian glycoproteins involves sugar chain trimming to Man6-5GlcNAc2. J Biol Chem. 278 (36), 34119-34124 (2003).
- Hosokawa, N. Enhancement of endoplasmic reticulum (ER) degradation of misfolded Null Hong Kong alpha1-antitrypsin by human ER mannosidase I. J Biol Chem. 278 (28), 26287-26294 (2003).
- Jakob, C. A., Burda, P., Roth, J., Aebi, M. Degradation of misfolded endoplasmic reticulum glycoproteins in Saccharomyces cerevisiae is determined by a specific oligosaccharide structure. J Cell Biol. 142 (5), 1223-1233 (1998).
- Tolchinsky, S., Yuk, M. H., Ayalon, M., Lodish, H. F., Lederkremer, G. Z. Membrane-bound versus secreted forms of human asialoglycoprotein receptor subunits. Role of a juxtamembrane pentapeptide. J Biol Chem. 271 (24), 14496-14503 (1996).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon