Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Thermodynamique du repliement des protéines membranaires mesuré par fluorescence Spectroscopie
Dans cet article
Résumé
Cet article détaille la vidéo la procédure expérimentale pour l'obtention du énergie libre de Gibbs du repliement des protéines membranaires par le tryptophane de fluorescence.
Résumé
Repliement des protéines membranaires est un sujet émergent ayant une importance fondamentale et liés à la santé. L'abondance des protéines membranaires dans les cellules sous-tend la nécessité d'étude exhaustive sur le pliage de cette famille de protéines ubiquitaires. En outre, les progrès dans notre capacité à caractériser les maladies associées à des protéines mal repliées ont motivé d'importants efforts expérimentaux et théoriques dans le domaine du repliement des protéines. Des progrès rapides dans ce domaine important est malheureusement entravée par les défis inhérents associés à des protéines membranaires et la complexité du mécanisme de pliage. Ici, nous présentons une procédure expérimentale pour mesurer les propriétés thermodynamiques de l'énergie libre de Gibbs se déroule en l'absence de dénaturant, Δ G ° H 2 O, pour une protéine membranaire intégrale représentant de E. coli. Ce protocole met l'accent sur l'application de la spectroscopie de fluorescence pour déterminer l'équilibre des populations d'états plié et déplié comme une fonction de la concentration de dénaturant. Considérations expérimentales pour la préparation de vésicules lipidiques synthétiques ainsi que des principales étapes de la procédure d'analyse de données sont mis en surbrillance. Cette technique est polyvalente et peut être poursuivie avec différents types de dénaturant, y compris la température et le pH, ainsi que dans divers environnements de pliage des lipides et des micelles. Le protocole actuel est celui qui peut être généralisé à toute membrane ou de protéines solubles qui répond à la série de critères discutés ci-dessous.
Protocole
1. Préparation de ~ 50 nm de diamètre de petites vésicules unilamellaires (SUV) pour Folding des protéines membranaires
- Une solution de 1,2-dimyristoyl-sn-glycéro-3-phosphocholine (DMPC) lipides dans le chloroforme est acheté et aliquotes dans des flacons en verre propre dans 20 mg par flacon pour le stockage de quantités. Une couche de gaz d'azote est ajouté à chaque flacon pour éviter l'oxydation des lipides, et les flacons sont scellés avec des bouchons et de parafilm. Les flacons sont conservés dans un congélateur à -20 ° C jusqu'à utilisation.
- Une seule fiole qui contient une aliquote de 20 mg de lipides dans le chloroforme est utilisé pour chaque expérience. Le contenu du flacon sont séchés en utilisant un flux d'azote gazeux pendant 1 heure jusqu'à ce qu'il ne reste solvable.
- Lipides séchés sont remis en suspension dans 1 ml de tampon phosphate 20 mM de potassium (pH = 7,3) en utilisant un sonicateur bain-marie pendant environ 30 secondes. Cette solution de lipides en suspension apparaîtra nuageux, et est transféré à un plastique de 15 ml tube avec un fond conique. Une autre aliquote de 1 ml de tampon phosphate est ajouté à la fiole de verre vide qui contenait à l'origine des lipides, et sonication est répété, la solution est ensuite ajoutée à la même durée de 15 ml tube. Ce processus est répété un total de 4 fois jusqu'à ce que le volume final dans le tube est de 4 ml, ce qui entraîne une concentration lipidique de 5 mg / ml de cette solution de vésicules stock.
- Le volume 4 ml de solution de vésicules lipidiques est placé dans un endroit chaud (~ 30 ° C), bain d'eau, et soniquées utilisant une micropointe ultrasonicator pendant 1 heure à 50% de cycle. Le but de l'eau du bain est double. D'abord, il empêche la solution lipidique de devenir trop chaud en raison du processus de sonication. Deuxièmement, le bain chaud assure que la température de la solution aqueuse de lipides demeure au-dessus de la température de transition de phase bicouche pendant la sonication; pour DMPC, cette température de transition est ~ 23 ° C.
- La solution soniquée est passé à travers une seringue de 0,22 um de filtre pour enlever les débris de la pointe sonicateur, et cette solution filtrée est équilibré nuit à 37 ° C.
2. Préparation des échantillons pour la formation initiale Curve Dépliage fluorescence
- Une solution mère de 10 M d'urée dans un tampon phosphate 20 mM (à pH 7,3) est faite. Il est essentiel que l'eau soit ajoutée à la matière solide (et non vice-versa), car la grande quantité d'urée occupe un volume significatif dans la solution. Cette solution d'urée peut être fait par l'ajout de solutés à un nettoyage, bouteille vide et en ajoutant de l'eau jusqu'au volume final prévu. Cette solution peut exiger le réchauffement dans un bain d'eau ou sur une plaque chauffante avec agitation pour dissoudre le soluté. L'urée est hygroscopique et par conséquent, la concentration réelle de la solution stock d'urée devrait être déterminée en utilisant l'indice de réfraction 1.
- Une solution stock tampon de 20 mM de phosphate (pH 7,3) est faite.
- Une solution stock de déplié protéine est préparée. Cette solution de protéines stock doit avoir une concentration de protéine de ~ 200 pM en urée 8 M, 20 mM de tampon phosphate.
- Les échantillons pour les études de fluorescence sont préparés de la manière suivante. Des volumes appropriés de protéines d'actions (article 2.3), solution lipidique des stocks (5 mg / ml de lipides, section 1), une solution d'urée stock 10 M (section 2.1), et un tampon phosphate de stock (article 2.2) sont combinés pour faire des échantillons contenant ~ 4 protéines uM, 1 mg / ml de lipides, et de 0 à 8 M d'urée par incréments de 1 M. Le volume total de chaque échantillon est de 200 mL. Un tableur par exemple (tableau 1) est inclus que les volumes de listes qui doivent utiliser une solution 200 uM stocks de protéines.
- Échantillons de contrôle sont réalisés comme décrit dans la section 2.4, cependant, les protéines ne sont pas ajoutées. En lieu et place de protéines, 4 pi d'une solution d'urée 8 M peuvent être ajoutées afin concentrations des autres composants sont identiques à celles de la section 2.4. Ces espaces sont utilisés pour soustraire de diffusion et un signal de fond qui apparaît dans d'autres les spectres de fluorescence de la protéine.
- Des échantillons provenant des sections 2.4 et 2.5 sont autorisés à incuber à 37 ° C pendant au moins 2 heures avant la mesure de spectres de fluorescence pour assurer pliage complet et d'équilibration.
3. Mesure des spectres de fluorescence
Fluorescence de tryptophane de chaque échantillon et blanc est mesuré en utilisant un fluorimètre état stable. Les spectres de fluorescence doit être enregistré avec longueur d'onde d'excitation de 290 nm pour éviter l'excitation des résidus tyrosine, et numérisé 305 à 500 nm. Entrée typique et passe-bande de sortie est de 3 nm. L'incrément de longueur d'onde et le temps d'intégration peut être optimisé pour signal-bruit. Les valeurs typiques pour incrémenter longueur d'onde et le temps d'intégration sont à 1 nm / étape et 0,5 sec / step, respectivement. Protéines membranaires en général plier en lipides synthétiques seulement lorsque la température est supérieure à la température de transition de phase bicouche. Par conséquent, dans ce cas de DMPC, la température de l'échantillon est maintenue constante à 30 ° C. Une cuvette microvolume silice fondue avec 160 microlitres est utilisé dans ces exriences.
4. Génération de la courbe initiale et dépliage Estimation de énergie libre de Gibbs dépliage des protéines membranaires
- Les spectres de fluorescence sous la forme d'un ensemble de données xy sont chargés dans des logiciels tels que Igor Pro, Matlab, origine, ou Excel.
- Signal du fond des vésicules lipidiques de l'urée et doit être soustraite suivant la procédure suivante:
Spectre Le spectre de fluorescence = premières de protéines dans la présence de vésicules lipidiques et de concentration spécifique de l'urée.
Spectre B = spectre brut de fluorescence des échantillons à blanc correspondant qui contient des vésicules lipidiques et la concentration d'urée même que dans le spectre de A; aucune protéine est dans les échantillons à blanc.
Spectre corrigé = spectre A - C * B. Spectre C est un scalaire typiquement égal à 1,0. Dans certains cas, C est inférieur ou supérieur à 1,0 en raison de problèmes de dispersion qui causent sur-ou sous-soustraction près de la région ~ 305-320 nm. - La longueur d'onde de fluorescence maximale, λ max, est totalisées à partir de spectres corrigés pour chaque concentration d'urée. Une valeur typique pour complètement dépliée protéine membranaire est de 350 nm tandis que celle de la protéine repliée est ~ 330 nm. Longueurs d'ondes tabulées sont convertis à la fraction déplié en convertissant la gamme des valeurs max λ (~ 330 à ~ 350 nm) à la gamme de 0,0 à 1,0 pour corréler la valeur λ max à la fraction de la population s'est déroulée. Par exemple, une valeur λ max de 330 nm correspond à 0,0, à 350 nm correspond à 1,0, et 340 nm correspond à 0,5 en termes de fraction dépliée. Cette fraction de protéine est alors déplié fonction de la concentration d'urée. Comme indiqué plus haut, la concentration d'urée doit être déterminée expérimentalement en mesurant l'indice de réfraction.
- Nous supposons que la protéine peut exister dans deux états: pliée ou dépliée. L'intrigue de la fraction déplié, f, fonction de la concentration d'urée, C, peut être adapté à l'équation: 2

Le coefficient m correspond à la vitesse de changement de l'énergie libre par rapport à la concentration de dénaturant, et C m correspond à la concentration d'urée milieu au cours de laquelle la population est égale à la population repliée dépliée. Ces valeurs sont les variables montage obtenu à partir des moindres carrés montage par le logiciel d'analyse de données. R (0.001987 kcal / mol • K) est la loi constante des gaz et T est la température (303 K). - Les coefficients obtenus à partir de l'ajustement sont utilisés pour déterminer l'énergie libre de Gibbs (kcal / mol) de dépliage en l'absence de l'urée:
 .
.
5. Optimisé Dépliage Courbe pour une mesure plus précise de l'énergie libre de Gibbs dépliage.
- La courbe de déroulement décrit ci-dessus révèle la gamme des concentrations d'urée qui provoque le plus grand changement dans le λ max. Cette gamme est généralement faible, et la majorité de déroulement se produit dans cette gamme de petite taille. Afin de déterminer précisément l'énergie libre du déroulement, des échantillons supplémentaires sont analysés dans cette région afin d'obtenir une parcelle qui contient de nombreux points de données nécessaires pour optimiser les moindres carrés de montage. Par exemple, si la protéine se déroule entre 2 et 4 M d'urée, les échantillons peuvent être faites qui contiennent de 2 à 4 M d'urée par incréments de 0,2 M de manière plus précise révèle la forme de cette région importante dans la courbe de déploiement. Il n'est pas nécessaire de mesurer les spectres vierge à chaque concentration d'urée, il suffit de mesurer un spectre vierge tous les ~ 0,5 M étape.
- L'énergie libre de dépliage obtenus à partir de ce complot peut différer légèrement de celle obtenue dans la courbe originale se déroule, mais reflète une valeur plus précise.
6. Les résultats représentatifs:
| Exemple | final [urée], M | protéines de stock (uL) | lipides stock (uL) | stock de 10 M d'urée (uL) | stock tampon (uL) |
| 1 | ~ 0.16 | 4 | 40 | 0 | 156 |
| 2 | 1 | 4 | 40 | 20 | 136 |
| 3 | 2 | 4 | 40 | 40 | 116 |
| 4 | 3 | 4 | 40 | 60 | 96 |
| 5 | 4 | 4 | 40 | 80 | 76 |
| 6 | 5 | 4 | 40 | 100 | 56 |
| 7 | 6 | 4 | 40 | 120 | 36 |
| 8 | 7 | 4 | 40 | 140 | 16 |
Tableau 1. Volume de solutions de stock nécessaire de faire des échantillons de fluorescence
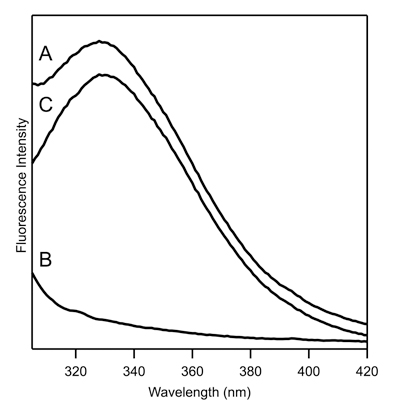
Figure 1. Tryptophane spectres de fluorescence de la protéine de la membrane ~ 5 uM représentant qui contient un résidu tryptophane unique. (A) des spectres de fluorescence de la protéine brute (spectre A partir de 4.2), (B) des spectres de fluorescence des premières vierges (spectre B de 4,2), (C) rectifié spectre de 4,2.

Figure 2. Corrigé tryptophane spectres de fluorescence de la protéine de membrane de la figure 1 pour des concentrations d'urée nombreux.

Figure 3. Dépliage courbe obtenue à partir des données de la figure 2, avec un ajustement de l'équation en 4.4. L'énergie libre de Gibbs est calculé conformément à la section 4.5.
Discussion
Le protocole actuel décrit la génération de courbes de déroulement associée à la membrane des protéines et des peptides qui contiennent des résidus tryptophane. Ici, il est supposé que la fluorescence du tryptophane indique si la protéine est pliée et insérée dans les vésicules lipidiques synthétiques, ou dépliée en solution. D'autres hypothèses, telles que les deux état de pliage et de la dépendance linéaire de l'énergie libre avec la concentration de dénaturant, sont faites dans le...
Déclarations de divulgation
Remerciements
Nous remercions Pékin Wu pour l'utilisation de ses données. Ce travail a été soutenu par une bourse de carrière NSF JEK
matériels
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| DMPC | Avanti Polar Lipids | 850345C | ||
| Urea | MP Biochemicals | 04821527 | ||
| Potassium Phosphate Dibasic | Fisher | P288 | ||
| Potassium Phosphate Monobasic | Fisher | P285 |
Références
- Shirley, B. A., Shirley, B. A. . Protein Folding and Stability. , 177-190 (1995).
- Pace, C. N. Determination and Analysis of Urea and Guanidine Hydrochloride Denaturation Curves. Methods Enzymol. 131, 266-279 (1986).
- Lau, F. W., Bowie, J. U. A Method for Assessing the Stability of a Membrane Protein. Biochemistry. 36, 5884-5892 (1997).
- Burgess, N. K., Dao, T. P., Stanley, A. M., Fleming, K. G. β-Barrel Proteins that Reside in the Escherichia coli Outer Membrane in Vivo Demonstrate Varied Folding Behavior in Vitro. J. Biol. Chem. 283, 26748-26758 (2008).
- Booth, P. J., Curnow, P. Folding scene investigation: membrane proteins. Curr. Opin. Struct. Biol. 19, 8-13 (2009).
- Hong, H., Tamm, L. K. Elastic coupling of integral membrane protein stability to lipid bilayer forces. Proc. Natl. Acad. Sci. U.S.A. 101, 4065-4070 (2004).
- Sanchez, K. M., Gable, J. E., Schlamadinger, D. E., Kim, J. E. Effects of Tryptophan Microenvironment, Soluble Domain, and Vesicle Size on the Thermodynamics of Membrane Protein Folding: Lessons from the Transmembrane Protein OmpA. Biochemistry. 47, 12844-12852 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon