É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Termodinâmica de enovelamento de proteínas de membrana Medido por espectroscopia de fluorescência
Neste Artigo
Resumo
Este artigo detalha o procedimento de vídeo experimental para a obtenção da energia livre de Gibbs da dobradura de proteína de membrana por fluorescência de triptofano.
Resumo
Enovelamento de proteínas da membrana é um tema emergente com significado fundamental e saúde. A abundância de proteínas da membrana das células está subjacente a necessidade de estudo detalhado da dobragem desta família de proteínas onipresente. Além disso, os avanços na nossa capacidade de caracterizar doenças associadas com proteínas deformadas ter motivado significativos esforços experimentais e teóricos no campo de enovelamento de proteínas. Progresso rápido neste domínio importante é, infelizmente, dificultada pela desafios inerentes associados com proteínas da membrana e da complexidade do mecanismo de dobramento. Aqui, descrevemos um procedimento experimental para medir a propriedade termodinâmica da energia livre de Gibbs de desdobramento, na ausência de desnaturante, Δ G ° H 2 O, por uma proteína de membrana representante integrante da E. coli. Este protocolo incide sobre a aplicação da espectroscopia de fluorescência para determinar as populações dos estados de equilíbrio dobrada e desdobrada em função da concentração de desnaturante. Considerações experimentais para a preparação de vesículas lipídicas sintéticas, bem como etapas fundamentais no processo de análise de dados são realçados. Esta técnica é versátil e pode ser perseguido com diferentes tipos de desnaturante, incluindo temperatura e pH, bem como em vários ambientes de dobramento de lipídios e micelas. O protocolo atual é aquele que pode ser generalizada para qualquer membrana ou proteína solúvel que atenda o conjunto de critérios discutidos abaixo.
Protocolo
1. Preparação de ~ 50 nm de diâmetro pequenas vesículas Unilamelares (SUVs) para Folding proteína de membrana
- Uma solução de 1,2-dimiristoil-sn-glicero-3-fosfocolina (DMPC) lipídios em clorofórmio é comprado e aliquotado em frascos de vidro limpo em 20 mg por frasco para armazenamento de quantidades. A camada de gás nitrogênio é adicionado a cada frasco para evitar a oxidação lipídica, e os frascos são selados com tampões e parafilme. Os frascos são armazenados em um freezer -20 ° C até o uso.
- Um único frasco que contém uma alíquota de 20 mg de lipídios em clorofórmio é usado para cada experimento. O conteúdo do frasco são secados usando um fluxo de gás nitrogênio por 1 hora até que não permanece solvente.
- Lipídios secas são ressuspendidos em 1 mL de tampão fosfato 20 mM de potássio (pH = 7.3) usando um sonicador banho-maria durante ~ 30 segundos. Esta solução de lipídios suspenso aparecerá nublado, e é transferido para um tubo de 15 mL de plástico com um fundo cônico. Outra alíquota de 1 mL de tampão fosfato é adicionado ao frasco de vidro vazio que originalmente continha lipídios e sonicação é repetido, a solução é então adicionado ao tubo de 15 mL mesma. Este processo é repetido um total de 4 vezes até que o volume final no tubo é de 4 mL, resultando em uma concentração de lipídios de 5 mg / mL desta solução vesícula estoque.
- O volume de 4 mL de solução vesícula lipídica é colocada num local quente (~ 30 ° C) banho de água e sonicado usando um MICROTIP ultrasonicator por 1 hora em ciclo de trabalho de 50%. A finalidade do banho de água é duplo. Primeiro, ela impede a solução de lipídios de tornar-se demasiado quente devido ao processo de sonicação. Segundo, o banho quente garante que a temperatura da solução aquosa de lipídios permanece acima da temperatura bicamada fase de transição durante sonicação; para DMPC, esta temperatura de transição é de aproximadamente 23 ° C.
- A solução sonicado é passado através de uma seringa 0,22 mM de filtro para remover detritos da ponta sonicador, e esta solução é filtrada equilibrada durante a noite a 37 ° C.
2. Preparação de amostra para Curva de fluorescência inicial Unfolding
- A solução estoque de 10 M de uréia em 20 mM tampão fosfato (pH 7,3) é feita. É essencial que a água ser adicionado ao sólido (e não vice-versa), pois a grande quantidade de uréia ocupa um volume significativo na solução. Esta solução de uréia pode ser feita pela adição de solutos para uma garrafa limpa e vazia e adicionando água até ao volume final pretendido. Esta solução pode exigir o aquecimento em banho-maria ou em uma chapa quente, mexendo para dissolver o soluto. Uréia é higroscópico e, portanto, a concentração real da solução estoque de uréia deve ser determinada através do índice de refração 1.
- Uma solução tampão estoque de 20 mM de fosfato (pH 7,3) é feita.
- Uma solução estoque de proteína é desdobrada preparado. Esta solução de proteína de ações deve ter uma concentração de proteína M ~ 200 em 8 de uréia M, tampão fosfato 20 mM.
- Amostras para estudos de fluorescência são preparados da seguinte maneira. Volumes adequados de proteína de ações (seção 2.3), a solução estoque de lipídeos (5 lipídica mg / mL, seção 1), estoque 10 solução de uréia M (seção 2.1) e tampão fosfato de estoque (seção 2.2) são combinados para fazer amostras contendo ~ 4 proteína M, 1 mg / mL de lipídios, e 0-8 M de uréia em incrementos de 1 M. O volume total de cada amostra é de 200 mL. Uma planilha exemplo (Tabela 1) está incluído o que volumes listas exigidas usando uma solução de proteína M 200 ações.
- Amostras em branco são feitas conforme descrito na seção 2.4, no entanto, a proteína não é adicionado. No lugar de proteína, 4 mL de uma solução de uréia 8 M podem ser adicionados para as concentrações de outros componentes são idênticos aos do ponto 2.4. Estes espaços são utilizados para subtrair espalhamento e sinal de fundo outra que aparece nos espectros de fluorescência da proteína.
- Amostras de seções 2.4 e 2.5 são autorizados a incubar a 37 ° C por pelo menos 2 horas antes da medição de espectros de fluorescência para garantir dobrar completa e equilíbrio.
3. Medição de espectros de fluorescência
Triptofano fluorescência de cada amostra e em branco é medida usando um fluorímetro steady-state. Espectros de fluorescência deve ser gravado com comprimento de onda de excitação de 290 nm para evitar a excitação de resíduos de tirosina, e digitalizados 305-500 nm. Entrada típica e bandpass de saída é de 3 nm. O incremento de comprimento de onda e tempo de integração pode ser otimizada para sinal-ruído. Valores típicos para o incremento de comprimento de onda e tempo de integração são 1 nm / etapa e 0,5 seg / step, respectivamente. Membrana proteínas em geral dobrar em lipídios sintéticos somente quando a temperatura está acima da temperatura bicamada fase de transição. Portanto, neste caso de DMPC, a temperatura da amostra é mantida constante em 30 ° C. A microvolume cubeta de sílica fundida com capacidade de 160 mL é utilizado nestes exexperimentos.
4. Geração de Curve Unfolding inicial e Estimativa da energia livre de Gibbs da proteína de membrana Unfolding
- Espectros de fluorescência na forma de um conjunto de dados xy são carregados no software, tais como Igor Pro, Matlab, Origin, ou Excel.
- Sinal do fundo da vesícula uréia e lipídeos deve ser subtraído utilizando o seguinte procedimento:
Spectrum A = espectro de fluorescência-prima de proteína na presença de vesículas lipídicas e de concentração específicos de uréia.
B = espectro espectro primas fluorescência de amostra em branco correspondente que contém vesículas lipídicas e concentração de uréia mesma que no espectro de A; nenhuma proteína nas amostras está em branco.
Spectrum Spectrum corrigido = A - C * espectro B. C é um escalar normalmente igual a 1,0. Em alguns casos, C é menor ou maior que 1.0 por causa de problemas de dispersão que causam sobre-ou sub-subtração perto da região nm ~ 305-320. - O comprimento de onda de máxima de fluorescência, λ max, é tabulados a partir de espectros corrigidos para cada concentração de uréia. Um valor típico para a plena desdobrado proteína de membrana é de 350 nm, enquanto a de proteína dobrada é de aproximadamente 330 nm. Comprimentos de onda tabelados são convertidos em fração desdobrado convertendo o intervalo de valores λ max (~ 330 a ~ 350 nm) para a faixa 0,0-1,0 correlacionar a λ valor máximo a fração da população se desenrolava. Por exemplo, um valor máximo de λ 330 nm corresponde a 0,0, a 350 nm corresponde a 1,0, e 340 nm corresponde a 0,5 em termos de fração desdobrado. Esta fração de proteína é, então, desdobrado plotado contra a concentração de uréia. Como dito acima, a concentração de uréia deve ser determinada experimentalmente através da medição do índice de refração.
- Supomos que a proteína pode existir em um dos dois estados: dobrado ou desdobrado. O enredo da fração desdobrado, f, versus concentração de uréia, C, pode estar em forma para a equação: 2

O coeficiente de m corresponde à taxa de variação da energia livre com respeito à concentração desnaturante e C m corresponde à concentração de uréia no ponto médio em que a população dobrada é igual a população se desenrolava. Estes valores são as variáveis encaixe obtido a partir de ajuste dos mínimos quadrados por dados software de análise. R (0.001987 kcal / mol • K) é a lei do gás constante, e T é a temperatura (303 K). - Os coeficientes obtidos a partir do ajuste são utilizadas para determinar a energia livre de Gibbs (kcal / mol) de desdobramento, na ausência de uréia:
 .
.
5. Otimizado Unfolding Curva para medições mais precisas de energia livre de Gibbs de desdobramento.
- A curva de desdobramento descrito acima revela a gama de concentrações de uréia que faz com que a maior mudança no λ max. Esta gama é normalmente pequeno, ea maioria de desdobramento ocorre dentro desta faixa de pequeno porte. , A fim de determinar com precisão a energia livre de desdobramento, amostras adicionais são analisados nesta região para obter um lote que contém muitos pontos dados necessários para a montagem dos mínimos quadrados otimizado. Por exemplo, se a proteína se desdobra entre 2 e 4 de uréia M, as amostras podem ser feitas que contêm 2-4 M de uréia em incrementos de 0,2 M para revelar mais precisamente a forma desta importante região da curva de desenvolvimento. Não é necessário medir espectros em branco em cada concentração de uréia, é suficiente para medir um espectro em branco a cada passo ~ 0,5 M.
- A energia livre de desdobramento obtido a partir deste enredo pode ser ligeiramente diferente do obtido na curva original desdobramento, mas reflete um valor mais preciso.
6. Resultados representativos:
| Amostra | final [uréia], M | proteína de ações (mL) | lipídica de ações (mL) | estoque 10 M uréia (mL) | estoque regulador (mL) |
| 1 | ~ 0,16 | 4 | 40 | 0 | 156 |
| 2 | 1 | 4 | 40 | 20 | 136 |
| 3 | 2 | 4 | 40 | 40 | 116 |
| 4 | 3 | 4 | 40 | 60 | 96 |
| 5 | 4 | 4 | 40 | 80 | 76 |
| 6 | 5 | 4 | 40 | 100 | 56 |
| 7 | 6 | 4 | 40 | 120 | 36 |
| 8 | 7 | 4 | 40 | 140 | 16 |
Tabela 1. Volume das soluções de reserva necessária para fazer amostras de fluorescência
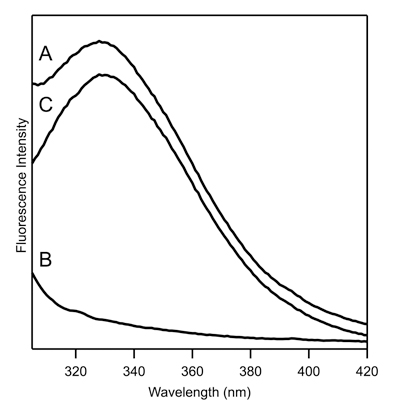
Figura 1. Triptofano espectros de fluorescência de ~ 5 proteína de membrana M representante que contém um resíduo de triptofano único. (A) Espectros de fluorescência da proteína Raw (Um espectro de 4,2), (B) Raw espectros de fluorescência de branco (espectro B de 4,2), (C) Corrigido espectro de 4,2.

Figura 2. Corrigida triptofano espectros de fluorescência da proteína de membrana a partir da Figura 1 para as concentrações de uréia numerosos.

Figura 3. Unfolding curva obtida a partir de dados na Figura 2, com ajuste para a equação em 4,4. A energia livre de Gibbs é calculado de acordo com a secção 4.5.
Discussão
O protocolo atual descreve a geração de curvas de desdobramento da membrana-associados proteínas e peptídeos que contêm resíduos de triptofano. Aqui, presume-se que a fluorescência de triptofano reflete se a proteína é dobrado e inserido em vesículas lipídicas sintéticas, ou desdobrado em solução. Suposições adicionais, tais como dois estados dobrar e dependência linear de energia livre com a concentração de desnaturante, são feitas no presente relatório;. Modificação destes pressupostos resultado...
Divulgações
Agradecimentos
Agradecemos a Beijing Wu para uso de seus dados. Este trabalho foi financiado por uma concessão de CARREIRA NSF para Jek
Materiais
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| DMPC | Avanti Polar Lipids | 850345C | ||
| Urea | MP Biochemicals | 04821527 | ||
| Potassium Phosphate Dibasic | Fisher | P288 | ||
| Potassium Phosphate Monobasic | Fisher | P285 |
Referências
- Shirley, B. A., Shirley, B. A. . Protein Folding and Stability. , 177-190 (1995).
- Pace, C. N. Determination and Analysis of Urea and Guanidine Hydrochloride Denaturation Curves. Methods Enzymol. 131, 266-279 (1986).
- Lau, F. W., Bowie, J. U. A Method for Assessing the Stability of a Membrane Protein. Biochemistry. 36, 5884-5892 (1997).
- Burgess, N. K., Dao, T. P., Stanley, A. M., Fleming, K. G. β-Barrel Proteins that Reside in the Escherichia coli Outer Membrane in Vivo Demonstrate Varied Folding Behavior in Vitro. J. Biol. Chem. 283, 26748-26758 (2008).
- Booth, P. J., Curnow, P. Folding scene investigation: membrane proteins. Curr. Opin. Struct. Biol. 19, 8-13 (2009).
- Hong, H., Tamm, L. K. Elastic coupling of integral membrane protein stability to lipid bilayer forces. Proc. Natl. Acad. Sci. U.S.A. 101, 4065-4070 (2004).
- Sanchez, K. M., Gable, J. E., Schlamadinger, D. E., Kim, J. E. Effects of Tryptophan Microenvironment, Soluble Domain, and Vesicle Size on the Thermodynamics of Membrane Protein Folding: Lessons from the Transmembrane Protein OmpA. Biochemistry. 47, 12844-12852 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados