È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Termodinamica di Folding delle proteine di membrana Misurato in spettroscopia di fluorescenza
In questo articolo
Riepilogo
Questo articolo dettagli video la procedura sperimentale per ottenere l'energia libera di Gibbs di ripiegamento delle proteine della membrana da triptofano fluorescenza.
Abstract
Ripiegamento delle proteine della membrana è un tema emergente di significati fondamentali e relativi alla salute. L'abbondanza di proteine di membrana delle cellule in base alla necessità per lo studio completo della piegatura di questa famiglia ubiquitaria di proteine. Inoltre, i progressi nella capacità di caratterizzare malattie associate con le proteine mal ripiegate hanno motivato notevoli sforzi teorici e sperimentali nel campo del ripiegamento delle proteine. Rapidi progressi in questo importante settore è purtroppo ostacolata dalle sfide inerenti associate proteine di membrana e la complessità del meccanismo di ripiegamento. Qui, delineare una procedura sperimentale per la misura di proprietà termodinamiche di energia libera di Gibbs di dispiegarsi in assenza di denaturante, Δ G ° H 2 O, per una proteina di membrana integrale rappresentante da E. coli. Questo protocollo si concentra sull'applicazione della spettroscopia di fluorescenza per determinare l'equilibrio le popolazioni degli stati piegato e spiegato in funzione della concentrazione di denaturante. Considerazioni sperimentali per la preparazione di vescicole lipidiche sintetici, nonché alle fasi principali della procedura di analisi dei dati sono evidenziati. Questa tecnica è versatile e può essere perseguito con diversi tipi di denaturante, tra cui la temperatura e pH, così come in vari ambienti ripiegamento dei lipidi e delle micelle. Il protocollo attuale è quella che può essere generalizzato a qualsiasi membrana o proteina solubile che soddisfa l'insieme dei criteri discussi di seguito.
Protocollo
1. Preparazione di ~ 50 nm Diametro piccole vescicole unilamellari (SUV) per Folding delle proteine di membrana
- Una soluzione di 1,2-dimyristoyl-sn-glicero-3-phosphocholine (DMPC) lipidi in cloroformio viene acquistato e aliquotted in fiale di vetro pulita in quantità di 20 mg per fiala per la conservazione. Uno strato di gas azoto viene aggiunto a ogni fiala per prevenire l'ossidazione dei lipidi, e le fiale sono chiusi con tappi e parafilm. I flaconcini sono conservati in un congelatore di -20 ° C fino al momento dell'uso.
- Un singolo flaconcino che contiene una aliquota di 20 mg di lipidi in cloroformio viene utilizzato per ogni esperimento. Il contenuto della fiala vengono essiccati utilizzando un flusso di azoto per 1 ora fino a quando non rimane solvente.
- Lipidi secchi sono risospesi in 1 ml di tampone fosfato di potassio 20 mM (pH = 7,3) con un sonicatore bagnomaria per circa 30 secondi. Questa soluzione di lipidi sospesi apparirà nuvoloso, e viene trasferito in una plastica da 15 ml tubo con un fondo conico. Un altro 1 aliquota mL di tampone fosfato viene aggiunta al flacone di vetro vuote che in origine conteneva dei lipidi, e sonicazione si ripete, la soluzione viene quindi aggiunto allo stesso tubo da 15 ml. Questo processo viene ripetuto per un totale di 4 volte fino a quando il volume finale nel tubo è di 4 ml, con una conseguente concentrazione dei lipidi di 5 mg / ml di questa soluzione vescicola stock.
- I 4 ml di soluzione delle vescicole lipidiche è collocato in un ambiente caldo (~ 30 ° C) bagno d'acqua, e sonicato utilizzando un microtip ultrasonicatore per 1 ora a duty cycle del 50%. Lo scopo del bagnomaria è duplice. In primo luogo, impedisce la soluzione dei lipidi di diventare troppo caldo a causa del processo di sonicazione. In secondo luogo, il bagno caldo assicura che la temperatura della soluzione acquosa lipidica rimane al di sopra della temperatura di transizione di fase doppio strato durante la sonicazione, per DMPC, questa temperatura di transizione è ~ 23 ° C.
- La soluzione sonicato è passato attraverso una siringa da 0,22 micron filtro per rimuovere i detriti dalla punta sonicatore, e questa soluzione filtrata viene equilibrata durante la notte a 37 ° C.
2. Preparazione del campione per la curva iniziale fluorescenza Unfolding
- Una soluzione madre di 10 M urea in tampone fosfato 20 mM (pH 7,3) è fatto. E 'essenziale che l'acqua da aggiungere al solido (e non viceversa) perché la grande quantità di urea occupa un volume significativo nella soluzione. Questa soluzione di urea può essere fatta con l'aggiunta di soluti ad un ambiente pulito, una bottiglia vuota e l'aggiunta di acqua fino al volume di destinazione finale. Questa soluzione potrebbe richiedere il riscaldamento a bagnomaria o su una piastra calda con mescolando per sciogliere il soluto. L'urea è igroscopico e quindi, la concentrazione effettiva della soluzione di riserva urea devono essere determinate con indice di rifrazione 1.
- Una soluzione tampone stock di fosfato 20 mM (pH 7,3) è fatto.
- Una soluzione di riserva della dispiegato proteina è preparato. Questa soluzione di proteine di riserva dovrebbe avere una concentrazione di circa 200 proteine mM in 8 M urea, 20 mM tampone fosfato.
- Campioni per studi di fluorescenza sono preparati nel modo seguente. Volumi adeguati di proteine stock (paragrafo 2.3), lipidi soluzione madre (5 mg / ml lipidi, sezione 1), fotografia di soluzione di urea 10 M (sezione 2.1), e tampone fosfato stock (sezione 2.2) sono combinati per rendere campioni contenenti ~ 4 proteina mM, 1 mg / mL lipidi, e 0-8 M urea in incrementi di 1 M. Il volume totale di ogni campione è di 200 microlitri. Un foglio di esempio (Tabella 1) è compreso che i volumi di liste di desiderata con uno stock 200 mM soluzione proteica.
- Campioni in bianco sono realizzate come descritto nel paragrafo 2.4, tuttavia, la proteina non viene aggiunto. Al posto di proteine, 4 ml di una soluzione di urea 8 M possono essere aggiunti in modo da concentrazioni di altri componenti sono identici a quelli nella sezione 2.4. Questi spazi vengono utilizzati per sottrarre dispersione del segnale e background che appare negli spettri di fluorescenza di proteine.
- I campioni di sezioni 2.4 e 2.5 sono autorizzati a incubare a 37 ° C per almeno 2 ore prima misura di spettri di fluorescenza per assicurare pieghevole completo e equilibrio.
3. Misura di spettri di fluorescenza
Triptofano fluorescenza di ciascun campione e vuota è misurata con un fluorimetro steady-state. Spettri di fluorescenza devono essere registrati con lunghezza d'onda di eccitazione di 290 nm per evitare di eccitazione dei residui di tirosina e digitalizzati 305-500 nm. Ingresso tipici e banda passante uscita è di 3 nm. L'incremento lunghezza d'onda e tempo di integrazione può essere ottimizzato per segnale-rumore. Valori tipici per incrementare lunghezza d'onda e tempo di integrazione sono 1 nm / passo e 0,5 sec / step, rispettivamente. Proteine di membrana in genere piega in lipidi di sintesi solo quando la temperatura è al di sopra della temperatura di transizione di fase doppio strato. Pertanto, in questo caso di DMPC, la temperatura del campione è mantenuta costante a 30 ° C. Una cuvetta silice fusa microvolumi con 160 microlitri è utilizzato in questi experiments.
4. Generazione di curve iniziali Unfolding e stima dell'energia libera di Gibbs di proteine di membrana Unfolding
- Spettri di fluorescenza nella forma di un insieme di dati xy vengono caricati in software come Igor Pro, Matlab, Origin o Excel.
- Segnale dal fondo vescicola urea e lipidico deve essere sottratto con la seguente procedura:
Spettro A = spettro di fluorescenza prime di proteine in presenza di vescicole lipidiche e di concentrazione specifici di urea.
Spettro B = spettro prime fluorescenza corrispondente campione in bianco che contiene vescicole lipidiche e la concentrazione di urea identica a quella dello spettro A, nessuna proteina nei campioni è vuoto.
Spettro corretta = Spectrum A - C * spettro B. C è uno scalare tipicamente pari a 1,0. In alcuni casi, C è inferiore o superiore a 1,0 a causa di problemi di dispersione che causano sovra o sotto-sottrazione vicino alla regione 305-320 nm ~. - La lunghezza d'onda del massimo di fluorescenza, λ max, è tabulati da spettri corretti per ciascuna concentrazione di urea. Un valore tipico per la piena spiegato proteina di membrana è di 350 nm, mentre quello della proteina ripiegata è ~ 330 nm. Lunghezze d'onda tabulati vengono convertiti in frazione spiegato convertendo la gamma di valori di λ max (~ 330 a ~ 350 nm) per la gamma 0,0-1,0 per correlare il valore λ max a frazione della popolazione spiegato. Per esempio, un valore λ massima di 330 nm corrisponde a 0,0, 350 nm corrisponde a 1,0 e 340 nm corrisponde a 0,5 in termini di frazione spiegato. Questa frazione di proteine è spiegato poi complottato contro la concentrazione di urea. Come detto sopra, la concentrazione di urea deve essere determinato sperimentalmente misurando indice di rifrazione.
- Partiamo dal presupposto che la proteina può esistere in uno dei due stati: ripiegati o aperti. La trama della frazione spiegato, f, in funzione della concentrazione di urea, C, può essere in forma per l'equazione: 2

Il coefficiente m corrisponde al tasso di variazione della energia libera rispetto alla concentrazione di denaturante e C m corrisponde alla concentrazione di urea punto medio in cui la popolazione piegata uguale alla popolazione spiegato. Questi valori sono le variabili montaggio ottenuto da minimi quadrati da un software di analisi dati. R (0.001987 kcal / mol • K) è la legge costante dei gas, e T è la temperatura (303 K). - I coefficienti ottenuti dal fit sono utilizzati per determinare l'energia libera di Gibbs (kcal / mol) di dispiegarsi in assenza di urea:
 .
.
5. Ottimizzato Unfolding Curva Per una misurazione più precisa della energia libera di Gibbs di Unfolding.
- La curva di svolgimento sopra descritta rivela l'intervallo di concentrazioni di urea che provoca il più grande cambiamento nella massima λ. Questa gamma è in genere piccolo, e la maggior parte di svolgimento avviene all'interno di questa gamma di piccole dimensioni. Al fine di determinare con precisione l'energia libera di dispiegarsi, campioni supplementari vengono analizzati in questa regione per ottenere una trama che contiene molti punti dati necessari per ottimizzare minimi quadrati. Per esempio, se la proteina si svolge tra 2 e 4 M urea, i campioni possono essere fatte che contengono 2-4 M urea in incrementi di 0,2 M per rivelare più precisamente la forma di questa importante regione della curva di svolgimento. Non è necessario misurare spettri vuoto ad ogni concentrazione di urea, ma è sufficiente per misurare uno spettro vuoto ogni passo ~ 0,5 M.
- L'energia libera di dispiegarsi ottenuti da questo complotto possono differire leggermente da quello ottenuto nella curva originale svolgimento, ma riflette un valore più preciso.
6. Rappresentante dei risultati:
| Campione | finale [Urea], M | magazzino proteine (mL) | magazzino dei lipidi (mL) | stock 10 M urea (mL) | magazzino tampone (mL) |
| 1 | ~ 0,16 | 4 | 40 | 0 | 156 |
| 2 | 1 | 4 | 40 | 20 | 136 |
| 3 | 2 | 4 | 40 | 40 | 116 |
| 4 | 3 | 4 | 40 | 60 | 96 |
| 5 | 4 | 4 | 40 | 80 | 76 |
| 6 | 5 | 4 | 40 | 100 | 56 |
| 7 | 6 | 4 | 40 | 120 | 36 |
| 8 | 7 | 4 | 40 | 140 | 16 |
Tabella 1. Volume delle soluzioni madre tenuto a fluorescenza di campioni
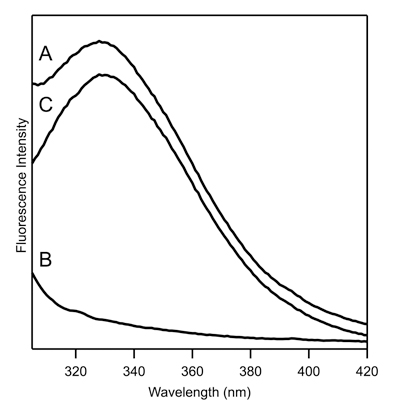
Figura 1. Triptofano spettri di fluorescenza di ~ 5 proteina di membrana rappresentante mM che contiene un singolo residuo triptofano. (A) spettri Raw fluorescenza di proteine (spettro da 4,2 A), (B) spettri Raw fluorescenza bianca (spettro di B da 4,2), (C) rettificato spettro da 4,2.

Figura 2. Corretto triptofano spettri di fluorescenza di proteine di membrana dalla Figura 1 per le concentrazioni di urea numerosi.

Figura 3. Unfolding curva ottenuta dai dati in figura 2, con forma di equazione in 4.4. L'energia libera di Gibbs è calcolato in base al paragrafo 4.5.
Discussione
L'attuale protocollo descrive la generazione di curve di svolgimento associata alla membrana proteine e peptidi che contengono residui di triptofano. Qui, si presume che la fluorescenza triptofano riflette se la proteina viene piegato e inserito in vescicole lipidiche sintetico, o si è svolto in soluzione. Ipotesi aggiuntive, come due Stati piegatura e dipendenza lineare di energia libera con concentrazione denaturante, sono contenute nella relazione corrente;. Modifica di tali presupposti risultato in divers...
Divulgazioni
Riconoscimenti
Ringraziamo Wu Pechino per l'utilizzo dei suoi dati. Questo lavoro è stato supportato da un riconoscimento alla CARRIERA NSF a JEK
Materiali
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| DMPC | Avanti Polar Lipids | 850345C | ||
| Urea | MP Biochemicals | 04821527 | ||
| Potassium Phosphate Dibasic | Fisher | P288 | ||
| Potassium Phosphate Monobasic | Fisher | P285 |
Riferimenti
- Shirley, B. A., Shirley, B. A. . Protein Folding and Stability. , 177-190 (1995).
- Pace, C. N. Determination and Analysis of Urea and Guanidine Hydrochloride Denaturation Curves. Methods Enzymol. 131, 266-279 (1986).
- Lau, F. W., Bowie, J. U. A Method for Assessing the Stability of a Membrane Protein. Biochemistry. 36, 5884-5892 (1997).
- Burgess, N. K., Dao, T. P., Stanley, A. M., Fleming, K. G. β-Barrel Proteins that Reside in the Escherichia coli Outer Membrane in Vivo Demonstrate Varied Folding Behavior in Vitro. J. Biol. Chem. 283, 26748-26758 (2008).
- Booth, P. J., Curnow, P. Folding scene investigation: membrane proteins. Curr. Opin. Struct. Biol. 19, 8-13 (2009).
- Hong, H., Tamm, L. K. Elastic coupling of integral membrane protein stability to lipid bilayer forces. Proc. Natl. Acad. Sci. U.S.A. 101, 4065-4070 (2004).
- Sanchez, K. M., Gable, J. E., Schlamadinger, D. E., Kim, J. E. Effects of Tryptophan Microenvironment, Soluble Domain, and Vesicle Size on the Thermodynamics of Membrane Protein Folding: Lessons from the Transmembrane Protein OmpA. Biochemistry. 47, 12844-12852 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon