Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Thermodynamik der Membrane Protein Folding durch Fluoreszenz-Spektroskopie
In diesem Artikel
Zusammenfassung
Dieses Video Artikel beschreibt die experimentellen Verfahren zur Erlangung der Gibbs-Membran Proteinfaltung durch Tryptophan-Fluoreszenz.
Zusammenfassung
Membrane Proteinfaltung ist ein aufstrebendes Thema sowohl mit Grundlagen-und gesundheitsbezogene Bedeutung. Die Fülle von Membranproteinen in der Zelle zugrunde liegt die Notwendigkeit einer umfassenden Studie über die Faltung von dieser allgegenwärtigen Familie von Proteinen. Darüber hinaus haben Fortschritte in unserer Fähigkeit, Krankheiten, die mit falsch gefalteten Proteinen assoziiert kennzeichnen signifikante experimentelle und theoretische Anstrengungen auf dem Gebiet der Proteinfaltung motiviert. Schnelle Fortschritte in diesem wichtigen Bereich ist leider durch die inhärenten Herausforderungen mit Membranproteinen und der Komplexität der Klappmechanismus verbunden behindert. Hier skizzieren wir ein experimentelles Verfahren zur Messung der thermodynamischen Eigenschaft der Gibbs-Energie der Entfaltung in Abwesenheit von Denaturierungsmittel, Δ G ° H 2 O, für eine repräsentative integrales Membranprotein von E. coli. Dieses Protokoll konzentriert sich auf die Anwendung der Fluoreszenz-Spektroskopie, um das Gleichgewicht Populationen von gefalteten und ungefalteten Zuständen als Funktion der Denaturierungsmittel Konzentration zu bestimmen. Experimentelle Überlegungen für die Herstellung von synthetischen Lipidvesikeln sowie wichtige Schritte in der Datenanalyse Verfahren sind hervorgehoben. Diese Technik ist vielseitig und kann mit verschiedenen Arten von Denaturierungsmittel, einschließlich Temperatur und pH-Wert, sowie in verschiedenen Umgebungen Faltung von Lipiden und Mizellen verfolgt werden. Das aktuelle Protokoll ist eine, die verallgemeinert werden, um eine Membran oder lösliches Protein, das die Kriterien erfüllt, kann unten besprochen.
Protokoll
1. Vorbereitung von ~ 50 nm Durchmesser kleine unilamellare Vesikel (SUVs) für Membrane Protein Folding
- Eine Lösung von 1,2-Dimyristoyl-sn-glycero-3-phosphocholin (DMPC) Lipide in Chloroform gekauft und aliquotiert in saubere Glasflaschen in 20 mg pro Fläschchen Mengen für die Lagerung. Eine Schicht von Stickstoffgas wird jedem Fläschchen gegeben, um Lipid Oxidation zu verhindern, und die Ampullen sind mit Kappen und Parafilm versiegelt. Die Fläschchen sind in einem -20 ° C Gefrierschrank bis zur Verwendung gelagert.
- Ein einziges Fläschchen, dass ein Aliquot von 20 mg Lipid in Chloroform enthält, ist für jedes Experiment verwendet. Der Inhalt des Fläschchens werden getrocknet mit einem Strom von Stickstoffgas für 1 Stunde, bis kein Lösungsmittel bleibt.
- Getrocknete Lipide werden in 1 ml 20 mM Kaliumphosphat-Puffer (pH = 7,3) mit einem Wasser-Ultraschallbad für ca. 30 Sekunden. Diese Lösung suspendiert Lipid erscheint bewölkt, und ist mit einem Kunststoff-15-ml-Röhrchen mit konischem Boden übertragen. Ein weiteres 1 mL Aliquot Phosphatpuffer wird die leere Glasflasche, die ursprünglich Lipid enthaltenen hinzugefügt und Beschallung wird wiederholt, die Lösung wird dann auf den gleichen 15-ml-Röhrchen gegeben. Dieser Vorgang wird insgesamt 4-mal wiederholt, bis der letzte Band in das Rohr 4 mL, was zu einer Lipid-Konzentration von 5 mg / mL in diesem Bestand Vesikel-Lösung.
- Die 4 mL Volumen von Lipid-Vesikel-Lösung ist in einem warmen platziert (~ 30 ° C) Wasserbad, und beschallt mit einem Ultraschallgerät Mikrospitze für 1 Stunde bei 50% Einschaltdauer. Der Zweck des Wasserbades ist zweifach. Erstens verhindert sie die Lipid-Lösung zu heiß wegen der Beschallung Prozess. Zweitens sorgt das warme Bad, dass die Temperatur der wässrigen Lösung Lipid-Doppelschicht oberhalb der Phasenübergangstemperatur während der Beschallung bleibt; für DMPC, ist dieser Übergang Temperatur ~ 23 ° C.
- Die beschallte Lösung wird durch ein 0,22 um Spritzenfilter, um Schmutz aus dem Ultraschallgerät Spitze zu entfernen, und das filtrierte Lösung wird äquilibriert über Nacht bei 37 ° C bestanden
2. Probenvorbereitung für die anfängliche Fluoreszenz Unfolding Curve
- Eine Stammlösung von 10 M Harnstoff in 20 mM Phosphatpuffer (pH 7,3) hergestellt wird. Es ist wichtig, dass das Wasser in den festen (und nicht umgekehrt) hinzugefügt werden, da die große Menge an Harnstoff nimmt eine bedeutende Volumen in der Lösung. Diese Harnstofflösung kann durch Zugabe von gelösten Stoffen, um eine saubere, leere Flasche und das Hinzufügen von Wasser bis zu den beabsichtigten Endvolumen aufgefüllt werden. Diese Lösung kann verlangen, Erwärmung im Wasserbad oder auf einer Heizplatte unter Rühren auf den gelösten Stoff aufzulösen. Harnstoff ist hygroskopisch und deshalb die tatsächliche Konzentration der Harnstofflösung soll anhand Brechungsindex werden. 1
- Eine Aktie Pufferlösung von 20 mM Phosphat (pH 7,3) hergestellt wird.
- Eine Stammlösung von entfalteten Proteins hergestellt wird. Dieser Bestand Protein-Lösung soll eine Konzentration von ~ 200 nM Protein in 8 M Harnstoff, 20 mM Phosphat-Puffer haben.
- Die Proben für die Fluoreszenz-Studien sind in der folgenden Weise hergestellt. Entsprechende Mengen auf Lager Protein (Abschnitt 2.3), hat Lipid-Lösung (5 mg / ml Lipid, Abschnitt 1), hat 10 M Harnstoff-Lösung (Abschnitt 2.1) und Lager-Phosphat-Puffer (Abschnitt 2.2) kombiniert werden, um Proben mit ~ 4 machen uM Protein, 1 mg / mL Lipid und 0 bis 8 M Harnstoff in 1 m-Schritten. Das Gesamtvolumen jeder Probe wird 200 pl. Ein Beispiel-Tabelle (Tabelle 1) enthalten ist, die Listen von Volumes unter Verwendung einer 200 uM Lager Protein-Lösung erforderlich.
- Blank-Proben gemacht werden, wie in Abschnitt 2.4 beschrieben, ist aber Protein nicht hinzugefügt. An die Stelle des Proteins, 4. Mai ul einer 8 M Harnstoff-Lösung hinzugefügt, so Konzentrationen von anderen Komponenten identisch mit denen in Abschnitt 2.4 sind. Diese Rohlinge werden verwendet, um Streuung und andere Hintergrund-Signal, dass in der Fluoreszenz-Spektren von Protein scheint zu subtrahieren.
- Die Proben aus den Abschnitten 2.4 und 2.5 sind bei 37 ° C für mindestens 2 Stunden inkubieren, bevor die Messung von Fluoreszenz-Spektren, um komplette Falt-und Gleichgewichts zu gewährleisten.
3. Die Messung der Fluoreszenz-Spektren
Tryptophan-Fluoreszenz jeder Probe und Blank wird mit einem steady-state-Fluorometer. Fluoreszenzspektren sollte mit Anregungswellenlänge von 290 nm aufgezeichnet werden, um Anregung von Tyrosinresten zu vermeiden, und gescannte 305 bis 500 nm liegt. Typische Ein-und Ausgang Bandpass ist 3 nm. Die Wellenlänge Inkrement-und Integrationszeit für Signal-Rausch-Verhältnis optimiert werden. Typische Werte für Wellenlängen Inkrement-und Integrationszeit sind 1 nm / Schritt und 0,5 sec / Schritt jeweils. Membranproteine in der Regel falten sich in synthetischen Lipiden nur dann, wenn die Temperatur über die Doppelschicht Phasenübergangstemperatur ist. Daher wird in diesem Fall von DMPC, ist die Temperatur der Probe konstant gehalten ° bei 30 C. Ein Mikrovolumens Quarzglas-Küvette mit 160 ul beträgt in diesen ex genutztExperimente.
4. Generierung von Initial Unfolding Curve und Abschätzung der Gibbs-Membrane Protein Unfolding
- Fluoreszenzspektren in Form eines xy Dataset in Software wie Igor Pro, Matlab, Origin oder Excel geladen.
- Signal aus dem Harnstoff-und Lipid-Vesikel Hintergrund muss subtrahiert mit dem folgenden Verfahren werden:
Spectrum A = raw Fluoreszenzspektrum von Proteinen in Gegenwart von Lipid-Vesikel und spezifische Konzentration von Harnstoff.
Spectrum B = raw Fluoreszenzspektrum entsprechenden Blindprobe, die Lipid-Vesikel und gleichzeitig Harnstoffkonzentration enthält als die in Spektrum A; kein Protein ist in der Blindproben.
Korrigierte Spectrum = Spectrum A - C * Spektrum B. C ist ein Skalar typischerweise gleich 1,0. In einigen Fällen ist C kleiner oder größer als 1,0 aufgrund der Streuung eines Problems, bei Über-oder Unter-Subtraktion in der Nähe des ~ 305-320 nm-Bereich. - Die Wellenlänge der maximalen Fluoreszenz, λ max, ist von korrigierten Spektren für jeden Harnstoffkonzentration tabellarisch dargestellt. Ein typischer Wert für voll entfalteten Membranprotein ist 350 nm, während die von gefalteten Proteins ist ~ 330 nm. Tabellarische Wellenlängen sind Bruchteil umgewandelt entfaltet durch die Umwandlung der Palette von λ max-Werte (~ 330 bis ~ 350 nm) auf den Bereich von 0,0 bis 1,0, die λ max-Wert um Bruchteil entfaltet Bevölkerung korrelieren. Zum Beispiel kann ein λ max-Wert von 330 nm bis 0,0 entspricht, entspricht 350 nm bis 1,0, und 340 nm entspricht 0,5 in der Fraktion entfaltet. Diese Fraktion der entfalteten Proteins wird dann gegen Harnstoff-Konzentration aufgetragen. Wie bereits erwähnt, sollte die Harnstoffkonzentration experimentell durch Messung Brechungsindex bestimmt werden.
- Wir gehen davon aus, dass das Protein in einem von zwei Zuständen existieren: gefaltet oder entfaltet. Die Handlung von Teil entfaltet, f, im Vergleich zu Harnstoff-Konzentration, C, kann die Gleichung angepasst werden: 2

Der Koeffizient m entspricht der Geschwindigkeit der Änderung der freien Energie in Bezug auf Denaturierungsmittel Konzentration und C m entspricht dem Mittelpunkt Harnstoff Konzentration, bei der das gefaltete Bevölkerung entspricht entfalteten Bevölkerung. Diese Werte sind die passenden Variablen aus der kleinsten Quadrate durch Daten-Analyse-Software erhalten. R (0.001987 kcal / mol • K) wird das Gas-Konstante und T die Temperatur (303 K). - Die Koeffizienten aus der fit erhalten werden eingesetzt, um die Gibbs-Energie (kcal / mol) der Entfaltung in Abwesenheit von Harnstoff zu bestimmen:
 .
.
5. Optimierte Unfolding Curve für genauere Messung der Gibbs-Energie der Entfaltung.
- Die Entfaltung Kurve oben beschrieben zeigt die Palette von Harnstoff-Konzentrationen, die die größte Veränderung in der λ max Ursachen. Dieser Bereich ist in der Regel klein, und die Mehrheit der Entfaltung stattfindet, in diesem kleinen Bereich. Um genau zu bestimmen, die freie Energie der Entfaltung, werden zusätzliche Proben in dieser Region analysiert werden, um ein Grundstück, dass viele Datenpunkte, die für optimierte kleinsten Quadrate befinden, zu erlangen. Zum Beispiel, wenn das Protein entfaltet sich zwischen 2 und 4 M Harnstoff, können die Proben gemacht, dass enthalten 2 - 4 M Harnstoff in 0,2 M Schritten genauer zeigen die Form dieser wichtigen Region in der sich entfaltenden Kurve. Es ist nicht notwendig, um leere Spektren bei jedem Harnstoffkonzentration zu messen, es reicht aus, um eine leere Spektrum messen jede ~ 0,5 M Schritt.
- Die freie Energie der Entfaltung von diesem Grundstück erhalten können geringfügig von der in der ursprünglichen Entfaltung Kurve erhalten, sondern spiegelt einen genaueren Wert.
6. Repräsentative Ergebnisse:
| Probe | endg. [Urea], M | Lager-Protein (ul) | Lager Lipid (ul) | Sofort lieferbar 10 M Harnstoff (ul) | Lager-Puffer (ul) |
| 1 | ~ 0,16 | 4 | 40 | 0 | 156 |
| 2 | 1 | 4 | 40 | 20 | 136 |
| 3 | 2 | 4 | 40 | 40 | 116 |
| 4 | 3 | 4 | 40 | 60 | 96 |
| 5 | 4 | 4 | 40 | 80 | 76 |
| 6 | 5 | 4 | 40 | 100 | 56 |
| 7 | 6 | 4 | 40 | 120 | 36 |
| 8 | 7 | 4 | 40 | 140 | 16 |
Tabelle 1. Volume von Stammlösungen benötigt, um Fluoreszenz-Proben machen
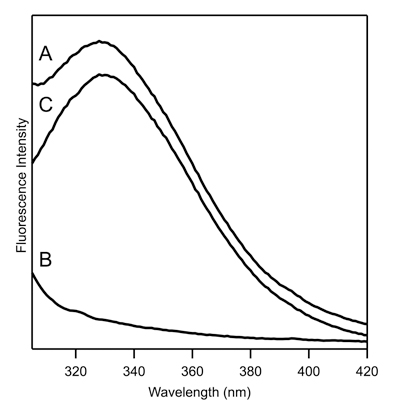
Abbildung 1. Tryptophan-Fluoreszenz-Spektren von ~ 5 uM Vertreter Membranprotein, dass eine einzige Tryptophanrest enthält. (A) Raw Fluoreszenzspektren von Protein (Spektrum A von 4,2), (B) Raw Fluoreszenzspektren blank (Spektrum B von 4,2), (C) Berichtigt Spektrum von 4,2.

Abbildung 2. Korrigierte Tryptophan Fluoreszenzspektren von Membranproteinen aus Abbildung 1 für eine Vielzahl von Harnstoff-Konzentrationen.

Abbildung 3. Unfolding Kurve aus den Daten in Abbildung 2 erhalten, mit fit Gleichung in 4.4. Die Gibbs-Energie ist nach Abschnitt 4.5 berechnet.
Diskussion
Das aktuelle Protokoll beschreibt die Generation der Entfaltung Kurven von Membran-assoziierte Proteine und Peptide, die Tryptophan enthalten. Hier wird davon ausgegangen, dass die Tryptophan-Fluoreszenz spiegelt, ob das Protein gefaltet ist und eingefügt in synthetische Lipidvesikel oder entfaltet in Lösung. Zusätzliche Annahmen, wie zB Zwei-Staaten-Faltung und lineare Abhängigkeit der freien Energie mit Denaturierungsmittel Konzentration, sind in dem aktuellen Bericht gemacht;. Modifikation von diesen Annahm...
Offenlegungen
Danksagungen
Wir danken Peking Wu für die Verwendung ihrer Daten. Diese Arbeit wurde durch einen NSF CAREER Award an JEK unterstützt
Materialien
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| DMPC | Avanti Polar Lipids | 850345C | ||
| Urea | MP Biochemicals | 04821527 | ||
| Potassium Phosphate Dibasic | Fisher | P288 | ||
| Potassium Phosphate Monobasic | Fisher | P285 |
Referenzen
- Shirley, B. A., Shirley, B. A. . Protein Folding and Stability. , 177-190 (1995).
- Pace, C. N. Determination and Analysis of Urea and Guanidine Hydrochloride Denaturation Curves. Methods Enzymol. 131, 266-279 (1986).
- Lau, F. W., Bowie, J. U. A Method for Assessing the Stability of a Membrane Protein. Biochemistry. 36, 5884-5892 (1997).
- Burgess, N. K., Dao, T. P., Stanley, A. M., Fleming, K. G. β-Barrel Proteins that Reside in the Escherichia coli Outer Membrane in Vivo Demonstrate Varied Folding Behavior in Vitro. J. Biol. Chem. 283, 26748-26758 (2008).
- Booth, P. J., Curnow, P. Folding scene investigation: membrane proteins. Curr. Opin. Struct. Biol. 19, 8-13 (2009).
- Hong, H., Tamm, L. K. Elastic coupling of integral membrane protein stability to lipid bilayer forces. Proc. Natl. Acad. Sci. U.S.A. 101, 4065-4070 (2004).
- Sanchez, K. M., Gable, J. E., Schlamadinger, D. E., Kim, J. E. Effects of Tryptophan Microenvironment, Soluble Domain, and Vesicle Size on the Thermodynamics of Membrane Protein Folding: Lessons from the Transmembrane Protein OmpA. Biochemistry. 47, 12844-12852 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten