Method Article
Dépistage du contenu haute sur les maladies neurodégénératives
Dans cet article
Résumé
Nous décrivons une méthodologie combinant la culture cellulaire automatisé à haute teneur en imagerie pour visualiser et quantifier les multiples processus cellulaires et des structures, de manière à haut débit. De telles méthodes peuvent aider dans l'annotation fonctionnelle des génomes d'autres ainsi que d'identifier des réseaux de gènes des maladies et de cibles thérapeutiques potentielles.
Résumé
L'annotation fonctionnelle des génomes, la construction de réseaux moléculaires et l'identification des cibles de nouveaux médicaments, sont des défis importants qui doivent être traitées comme une question de grande urgence 1-4. De multiples approches complémentaires en «omique» ont fourni des indices sur les facteurs de risque génétiques et les mécanismes pathogéniques sous-jacente de nombreuses maladies neurodégénératives, mais la plupart des conclusions ont encore besoin de validation fonctionnelle 5. Par exemple, une étude récente du génome entier pour l'association maladie de Parkinson (PD), a identifié plusieurs nouveaux loci comme facteurs de risque pour la maladie, mais la variante sous-jacente responsable (s) ou mécanisme pathogène n'est pas connue 6, 7. Comme chaque région associée peut contenir plusieurs gènes, l'évaluation fonctionnelle de chacun des gènes sur les phénotypes associés à la maladie, en utilisant les techniques traditionnelles de biologie cellulaire serait trop long.
Il ya aussi un besoin de comprendre les réseaux moléculaires qui pointentmutations génétiques à l'phénotypes qu'ils causent. Il est prévu que les phénotypes des maladies sont le résultat d'interactions multiples qui ont été perturbés. Reconstruction de ces réseaux moléculaires en utilisant des méthodes traditionnelles prendrait beaucoup de temps. Par ailleurs, les prévisions du réseau à partir d'études indépendant de composants individuels, l'approche réductionniste, ne sera probablement sous-estimer la complexité de 8 réseau. Cette sous-estimation pourrait, en partie, expliquer le faible taux de réussite de l'approbation des médicaments en raison d'effets secondaires indésirables ou toxiques. Gagner un point de vue du réseau de sentiers des maladies liées à l'aide HT / HC approches de dépistage cellulaire, et d'identifier les nœuds clés au sein de ces voies, pourrait conduire à l'identification de cibles qui sont plus adaptés pour l'intervention thérapeutique.
Criblage à haut débit (HTS) est une méthode idéale pour répondre à ces questions 9-12. mais les méthodes traditionnelles ont été unidimensionnel tests cellulaires entiers ainsi, celui utilisé simpliSTIC pour les affichages de processus biologiques complexes. Ils ont été incapables de quantifier simultanément les phénotypes observés dans de nombreuses maladies neurodégénératives telles que les déficits du transport axonal ou des altérations dans 13 propriétés morphologiques, 14. Cette approche ne pouvait pas être utilisée pour étudier la nature dynamique des processus cellulaires ou des événements pathogènes qui se produisent dans un sous-ensemble de cellules. Pour quantifier ces caractéristiques on doit passer au multi-dimensionnelle phénotypes appelés à haute teneur de dépistage (HCS) 4, 15-17. HCS est la quantification des cellules à base de plusieurs processus simultanément, ce qui fournit une représentation plus détaillée de la réponse cellulaire aux perturbations par rapport aux différentes HTS.
HCS a de nombreux avantages sur HTS 18, 19, mais faire un haut-débit (HT)-haut contenu (SC) l'écran dans des modèles neuronaux est problématique en raison de coûts élevés, la variation de l'environnement et l'erreur humaine. Afin de détecter les réponses cellulaires sur un «Phenomic« échelleen utilisant l'imagerie HC on doit réduire la variation et l'erreur, tout en augmentant la sensibilité et de reproductibilité.
Ici, nous décrivons une méthode précise et fiable pour mener des écrans shRNA utilisant la culture cellulaire automatisé 20 et HC imagerie dans des modèles cellulaires neuronales. Nous décrivons comment nous avons utilisé cette méthode pour identifier des modulateurs pour une protéine particulière, DJ1, qui, lorsqu'il est muté entraîne autosomique récessive parkinsonisme 21.
Alliant la polyvalence de l'imagerie avec des méthodes HC HT, il est possible de quantifier avec précision une pléthore de phénotypes. Ce pourrait ensuite être utilisé pour faire avancer notre compréhension du génome, les voies impliquées dans la pathogenèse de la maladie ainsi que d'identifier des cibles thérapeutiques potentielles.
Protocole
1. Processus automatisé adhérente de culture cellulaire (Figure 1) 20
- Préparer le système automatisé de culture cellulaire pour l'entrée des boîtes de culture cellulaire. Des consommables de charge (par exemple, des embouts de pipette, des plaques de culture cellulaire, plaques de dosage) dans le système en utilisant l'interface utilisateur graphique (GUI). S'assurer qu'il ya suffisamment de milieux de culture cellulaire, un tampon phosphate salin (PBS) et la trypsine dans le système robotisé.
- Manuellement plaques semences OmniTray deux avec 2 x 10 6 cellules par plaque, de la ligne de neuroblastome SH-SY5Y cellule. Maintenir les cellules de l'Opti-MEM avec 10% de sérum bovin foetal (FBS). Mettez les plaques dans le robot culture cellulaire en utilisant l'interface graphique. Les cellules seront incubées à 37 ° C et 5% de CO 2.
- Choisissez quelle cellule protocole de culture doit être initié 20. On peut choisir à partir d'un procédé de culture cellulaire adhérente 22, la culture et l'expansion des cellules souches embryonnaires (ES) sur les cellules cellules nourricières de souris 23 ou la culture de la suspension cells 22.
- Sélectionnez le protocole de culture de cellules adhérentes (figure 1) et assurer adhérente de cellules fichiers paramètre de ligne spécifiques sont ajustées de telle sorte que le seuil de la confluence (l'aire de la plaque OmniTray qui contient des cellules) est fixé à 70%. Régler l'heure trypsinisation total de deux minutes.
- Demander au robot pour préparer les plaques OmniTray nouvelle, avec un nombre de cellules de semis de 2 x 10 6 cellules par plaque.
- Plaques OmniTray entrée dans le système de culture cellulaire utilisant le protocole automatisé adhérente de culture cellulaire (figure 1). Ce protocole comporte les étapes suivantes: plaques sont incubées et imagé jusqu'à ce qu'ils atteignent le seuil de la confluence pré-définis. Si les cellules n'atteignent pas le seuil de la confluence dans les 5 lectures, les plaques sont retirés du système. Arrivé au seuil défini par l'utilisateur confluent, les cellules sont lavées, traitées à la trypsine et comptées. Un certain nombre de pré-défini de cellules sont ajoutées à des plaques OmniTray nouvelle et si il ya un nombre suffisant de cellules, un engourdissement spécifiéeer des plaques de dosage sont transportés sur le pont et un nombre défini de cellules distribué dans chaque puits. Plaques de dosage peut être directement imagés à l'aide du microscope intégré ou de sortie du système pour un traitement ultérieur.
- Demandez le système pour préparer quatre plaques de dosage par plaque OmniTray, avec un total de 5000 cellules par puits.
2. la production de virus shRNA et le placage en plaques de dosage (Temps requis: 6 jours)
- Cultivez stocks de glycérol contenant les vecteurs bactériens shRNA (Open Biosystems, TRC1) la nuit dans 2ml de médias milieu Luria-Bertani contenant 100 ug / ml d'ampicilline (Sigma-Aldrich).
- Extrait plasmides selon le protocole du fabricant (Promega Wizard MagneSil TFX).
- Produire de virus en utilisant l'ARNi Consortium haut débit de production Lentivirales (96 puits) du protocole 24. Travailler avec des lentivirus est relativement sûr car les particules de virus utilisés pour la transduction sont la réplication déficiente et split-stratégies de conditionnement génétique sont utilisées pour leur production. Cependant, quand on travaille avec des lentivirus, d'autres procédures de biosécurité sont nécessaires pour minimiser le risque pour soi et les autres 25. Toutes les expériences doivent être menées dans un laboratoire ou d'MLII BSL2 niveau de sécurité. Tous les plastiques (pipettes, des plats en plastique, les médias) qui ont été en contact avec des particules lentivirus doivent être incubées avec l'eau de Javel pour 24 heures avant l'élimination.
- Calculer la multiplicité d'infection (MOI) de lentivirus par la détermination du pourcentage de cellules GFP-positives à l'aide de la GFP pLKO.1 plasmide (Sigma-Aldrich).
- Lentivirus plaque dans des plaques d'essai, avec une MOI de 3.
3. Transduction lentiviraux et la différenciation neuronale des cellules SH-SY5Y (Temps requis: 6 jours)
- Les cellules sont ajoutés dans les plaques d'essai (voir étape 1.7). Charge plaques de dosage contenant des lentivirus shRNA dans le système de culture cellulaire automatisé.
- Après 24 heures, les médias sur leplaques de dosage sera changé pour Opti-MEM contenant 0,5% de FBS et 0,1 d'acide rétinoïque uM pour commencer le processus de différenciation. La différenciation des cellules SH-SY5Y permet la visualisation des structures neuritique et synchronise la division cellulaire.
- Poursuivre l'incubation des plaques d'essai dans les médias de différenciation pendant 5 jours. Cela garantit knockdown maximale de l'expression des gènes cibles.
- Au jour 5, ajouter 50 uM H 2 O 2 à la moitié des plaques d'essai pendant 24 heures pour stimuler la translocation de DJ1 aux mitochondries.
- Au jour 6, ajouter CmxROS Mitotracker (Invitrogen) pour les cellules, à une concentration finale de 200 nM par puits et incuber à 37 ° C pendant 30 minutes.
- On peut demander au système de plaques d'image directement en utilisant l'imageur HC ou plaques peuvent être exportés à partir du système pour un traitement ultérieur.
4. Immunomarquage automatisée des plaques d'essai (Temps requis: 2 jours)
Qualité d'image est paramount pour la réalisation d'une sensible et fiable HCS. Dommages à la monocouche cellulaire due à pipetage inexactes peuvent conduire à la qualité d'image médiocre et les résultats non reproductibles. Afin de minimiser les dommages couche de cellules, l'immunomarquage a été réalisée en utilisant une station robotique. La procédure est similaire à celui qui a été décrit précédemment, mais 26 a été personnalisé pour augmenter le débit et réduire l'utilisation des consommables.
- Fixer les cellules avec 100 pl de paraformaldéhyde à 4% de pré-chauffé à 37 ° C. Incuber pendant 20 minutes à température ambiante.
- Laver les cellules avec 200 pi de PBS pendant 5 minutes, 3 fois.
- Incuber les plaques d'essai avec 200 pi de PBS contenant 0,1% de Triton (PBST) pendant 10 minutes.
- Laver les cellules avec 200 pi de PBS pendant 5 minutes, 3 fois.
- Incuber les plaques d'essai avec 200 pi de tampon de blocage (PBST avec 5% de FBS) pendant 1 heure à température ambiante.
- Laver les cellules avec 200 pi de PBS pendant 5 minutes, 3 fois.
- Incuber avec le Follen raison des anticorps primaires nuit à 4 ° C:
- Chèvre DJ1 N20 (Santa Cruz, 5 pg / ml)
- Lapin β-tubuline III (Sigma-Aldrich, 1 ug / ml)
- Le lendemain, laver les cellules avec 200 pi de PBS pendant 5 minutes, 3 fois.
- Incuber les plaques de dosage avec les anticorps secondaires suivants 1 heure à température ambiante:
- AlexaFluor 488 d'âne anti-chèvre (Invitrogen, 2 pg / ml)
- AlexaFluor 647 chèvre anti-lapin (Invitrogen, 2 pg / ml
- Laver les cellules avec 200 pi de PBS pendant 5 minutes, 3 fois.
- Incuber les cellules avec Hoechst (Invitrogen; 1 ug / ml) pendant 10 minutes.
- Laver les cellules avec 200 ul de PBS pendant 5 minutes, 3 fois.
- Plaques Conserver à 4 ° C jusqu'à ce qu'ils puissent être imagé.
5. Haute images par le contenu d'acquisition et d'analyse d'image (Temps requis: 5 jours)
- Image d'un total de 30 champs par puits en utilisant la lentille de l'objectif 20x. Visualisez DJ1 avec l'IFTC jeu de filtres, les mitochondries avec l'ensemble TRITC filtre, β-tubuline III avec le jeu de Cy5 filtre et les noyaux en utilisant l'ensemble filtre UV (figure 3).
- Analyser les images en utilisant l'analyse compartimentale Bioapplication (Cellomics, ThermoFisher) pour déterminer l'intensité moyenne du signal Mitotracker dans les mitochondries. (Figure 4B, F).
- Pour déterminer le coefficient moyen de chevauchement entre DJ1 et les mitochondries, d'analyser les images en utilisant le bioapplication colocalisation Cellomics (Cellomics, ThermoFisher). Définir des régions d'intérêt (ROI) comme suit: Un retour sur investissement - le noyau (figure 4A, E), le ROI B - mitochondries (figure 4 B, F). Exclure ROI A partir de ROI B pour assurer l'analyse de seulement le cytoplasme. Définir les mitochondries que la région cible I et DJ1 comme cible la région II (figure 4C, G).
- Analyser les images en utilisant le profilage bioapplication neuronale (Cellomics, ThermoFisher) pour tracer la longueur moyenne des neurites de la tubuline β-coloration III (figure4D, H).
- Plaques d'image en utilisant l'Opéra LX automatisés confocale lecteur (Perkin-Elmer). Image d'un total de 30 champs par puits en utilisant la lentille de l'objectif 60x avec immersion dans l'eau. Visualisez mitochondries avec le laser 561 nM et noyaux avec excitation UV.
- Analyser les images en utilisant le Spot-Bord-Ridge (SER) la texture caractéristiques algorithme. Le filtre SER-Ridge transmet l'intensité de pixels formant des crêtes comme des modèles. Le plus fragmenté les mitochondries, plus le score SER-crête (figure 8).
6. La normalisation des données et analyse
- Importer les données du logiciel d'analyse d'image dans la BioConductor CellHTS2 paquet pour l'environnement logiciel R (R version 2.11.1, la version 2.6 du BioConductor).
- Logarithme de base (2) de transformer les données avant de plaque médiane de normalisation par base 27, 28. Ne pas appliquer l'ajustement de variance par plaque.
- Pour identifier les modificateurs d'un phénotype, utiliser une ANOVA à deux voies entre les différent le traitement des groupes à savoir les cellules infectées non traitées brouillés brouillés vs cellules toxine traitée vs cellules non traitées vs gène cible les cellules traitées gène cible (figures 5-7).
7. Les résultats représentatifs
Mutations au sein DJ1 donner lieu à l'apparition au début récessif parkinsonisme 21, mais on ne sait pas comment la perte de DJ1 donne lieu à phénotype de la maladie. Il est connu que les cellules déficientes des DJ1 sont plus sensibles à la mort cellulaire induite par le stress oxydatif et en réponse au stress oxydatif, DJ1 translocation du cytoplasme vers le 29 mitochondries, 30. En construisant des tests HC pour surveiller ces phénotypes, nous pouvons identifier les gènes qui réglementent ou affectent les phénotypes associés à DJ1. Cette approche peut aider à déchiffrer les voies dans lesquelles DJ1 fonctions et qui pourraient être impliquées dans la pathogenèse de la maladie.
Exemple d'une interaction avec les épistatique DJ1 (figure 5): Knockdown des DJ1 dans les cellules exposéesaux résultats de toxine dans une plus grande perte de viabilité cellulaire (BAR-B: image-B) par rapport aux cellules infectées par brouillés lentivirus (BAR-A: l'image-A). Knockdown d'un gène cible a un effet similaire à celui observé dans des cellules avec un effet de choc DJ1 (BAR-C: l'image-C). Knockdown des deux DJ1 et gène cible A en résulte une perte nettement supérieure de la viabilité cellulaire que la perte de gènes soit seul (BAR-D:-D l'image). Ceci suggère une interaction entre les épistatique DJ1 et gène cible A.
Exemple d'un gène régulant DJ1 translocation (Figure 6): Quand les cellules sont exposées à une toxine, DJ1 translocation du cytoplasme vers les mitochondries, qui est quantifiée par un coefficient plus élevé de chevauchement entre DJ1 et les mitochondries (BAR-A: image Un rapport BAR-C: image C). Dans les cellules où B gène cible a été réduite au silence, moins DJ1 translocation dans la mitochondrie où les cellules sont exposées à la toxine. Ceci suggère que B gène cible est impliqué dans le transport de DJ1 aux mitochondries. (BAR-B: l'image B et BAR-D: l'image D)
Exemple d'un gène impliqué dans excroissance neuronale (figure 7): Knockdown de C gène cible dans les cellules de type sauvage des résultats SH-SY5Y à une augmentation significative de la longueur des neurites (BAR-B: image-B) par rapport aux cellules infectées par des lentivirus exprimant brouillés shRNA (BAR-A: image A). Cet effet est perdu dans les cellules incubées avec de la toxine (BAR-C et D).
Exemple d'un gène impliqué dans la morphologie mitochondriale (figure 8): L'infection de type sauvage SH-SY5Y avec shRNA ciblage génique résultats D à une diminution de la valeur de segmentation SER-Ridge mitochondriale (figure 8, l'image C et D) par rapport à cellules infectées par brouillés lentivirus (figure 8, l'image A et B).
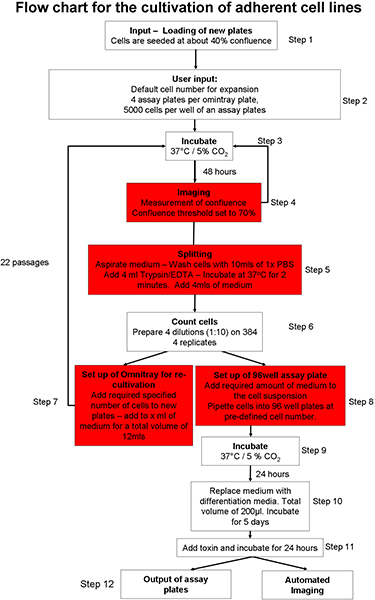
Figure 1 Schéma du protocole de culture cellulaire automatisé.:
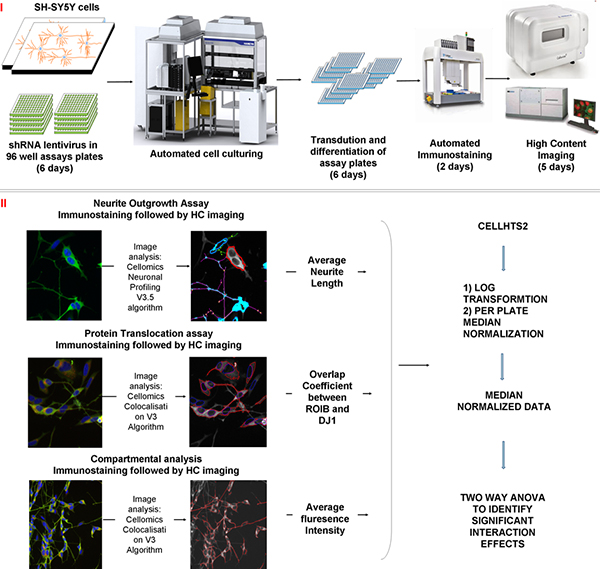
Figure 2 Vue schématique du processus de dépistage, l'analyse d'image et les méthodes statistiques utilisées lors du processus de dépistage:. I) Les cellules sont cultivées jusqu'à ce qu'elles soient confluentes et ensuite étalées dans des plaques de dosage contenant le lentivirus shRNA. Les cellules sont différenciées pendant 5 jours et la toxine est alors ajouté aux plaques pendant 24 heures. Plaques de dosage sont de sortie du système et immunocolorés. Le montant de temps requis pour chacun des processus est indiqué entre parenthèses. ii) Les données sont acquises en utilisant un imageur HC (viabilité des cellules, des protéines et translocation excroissance neuronale) et un imageur automatisés confocale (morphologie mitochondriale). Les données sont exportées vers CellHTS2 package dans R, de base (2) une transformation logarithmique et normalisée. Deux ANOVA est utilisé pour identifier les interactions significatives entre les différentes variables.

Figure 3. Les images composites de cellules acquis par HC imagerie. A) Des cellules non traitées, b) les cellules traitées avec H 2 O 2. DJ1 est marquée en vert, les mitochondries en rouge et les noyaux en bleu. Coloration neurites n'est pas mis en évidence.
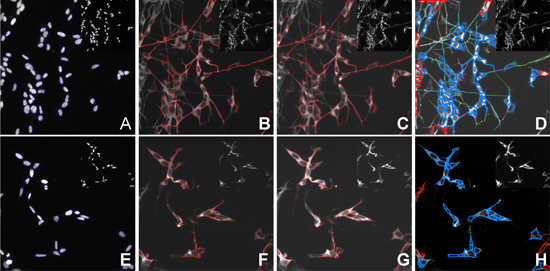
Figure 4. Quantification de plusieurs fonctions cellulaires obtenues à partir d'un HCS. JC) non traitée SH-SY5Y. EH) H 2 O 2 traités SH-SY5Y. Une segmentation, E noyaux) et la définition des ROI A, B, F) Identification et quantification des mitochondries B, ROI, C, G) Identification des DJ1, cible de la voie II, D, G) l'identification et le calcul de la longueur des neurites moyenne. Cellules près du bord de l'image sont exclus de l'analyse. Encart images sont des images avant l'analyse.

Figure 5. Identification d'un gène dans épistasie avec DJ1 (lettres sur les barres correspondent aux inscriptions sur les images). Images de A à D sont SH-SY5Y étiquetés avec Mitotracker CMXRos (rouge) qui a été utilisé pour la quantification de la santé des cellules.
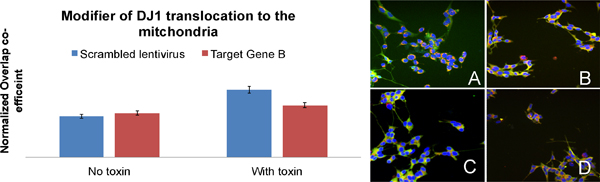
Figure 6. Identification d'un gène régulant DJ1 translocation (lettres sur les barres correspondent aux inscriptions sur les images). Images de A à D sont SH-SY5Y étiquetés pour DJ1 (vert), les mitochondries (en rouge) et des noyaux (bleu) qui ont été utilisées pour la quantification des DJ1 translocation vers la mitochondrie.
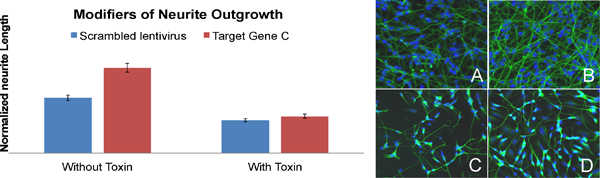
Figure 7. Identification d'un gène impliqué dans excroissance neuronale (lettres sur les barres correspondent aux inscriptions sur les images). Images de A à D sont SH-SY5Y étiquetés pour β-III tubuline (vert) et des noyaux (bleu) qui ont été utilisées pour la quantification de la longueur des neurites.
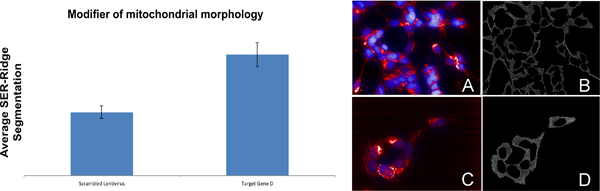
Figure 8. Identification d'un gène impliqué dans la régulation de la morphologie mitochondriale. Image A et C sont des images composites du SH-SY5Y cellules infectées par brouillés shRNA ou un shRNA ciblage génique D respectivement. Les mitochondries sont colorés en rouge, tandis que les noyaux sont colorés en bleu. Image B et D sont des visualisations de la quantification SER-Ridge.
Discussion
Avec la baisse des coûts de HT / HC systèmes de criblage cellulaire, combinée à la disponibilité de puissants outils de l'échelle du génome de modifier la fonction des gènes, HT / HC écrans deviennent monnaie courante dans les milieux universitaires. L'approche a déjà été appliquée avec succès dans divers domaines de recherche tels que l'identification des cibles médicamenteuses dans le cancer 9, 31-33 et 34-36 et le développement embryonnaire a même potentiel d'application dans le déchiffrement des voies impliquées dans les troubles neuropsychiatriques 37,38. Cependant la mise en œuvre d'un tel système nécessite un investissement important de temps et d'effort avec l'optimisation des processus prenant souvent un minimum de 6 mois. Toutes les étapes, comme les temps de traitement à la trypsine, les vitesses de pipetage et les densités de semis doivent être ajustées, afin de s'assurer que les cellules sont saines et une croissance régulière. Prévention de la contamination bactérienne est l'un des défis les plus difficiles de culture cellulaire automatisé avec des protocoles de nettoyage hebdomadaires dans combination avec constante de rinçage de toutes les lignes portant liquides avec de l'éthanol à 70% étant nécessaire pour les cultures sans contamination. Il sera également nécessaire d'améliorer la robotique afin que les instruments supplémentaires, tels que les microscopes confocaux pour une meilleure résolution et des congélateurs à -80 ° C pour le stockage composé peut être intégré.
Il ya aussi des limites qui doivent être abordées pour améliorer la sensibilité, la vitesse et l'utilité de cette méthode pour étudier les réseaux de gènes et d'identifier les gènes impliqués dans les voies pathogènes moléculaires.
Pour effectuer un écran HT / HC et s'assurer que les données fiables sont recueillis, plusieurs aspects doivent être optimisés. Tout d'abord, la fiabilité de la mesure est primordiale et dépend de la robustesse et la sensibilité du dosage. Par exemple, les tests décrits ci-dessus sont adaptés pour les petits écrans, mais sont difficiles à mettre en œuvre à l'échelle du génome entier, en raison d'étapes de traitement avant le nombre requis d'acquisition d'image.Ainsi, on aurait pu construire des lignées cellulaires stables exprimant le gène rapporteur, ce qui permettrait à l'imagerie directe et conduire à des variations diminué en raison du nombre réduit d'étapes de traitement. A l'heure actuelle, la conception d'un test qui décrit avec précision et quantifie de manière fiable un phénotype d'intérêt est un obstacle majeur dans le processus de dépistage HC.
De nombreux écrans sont menées dans les cellules de mammifères en utilisant différentes bibliothèques d'ARNi, qui souffrent d'effets hors-cible, l'efficacité limitée silençage génique et la couverture du génome incomplet. Ainsi les bibliothèques doivent être faites, qui sont plus spécifiques, puissants et ont une meilleure couverture. Des efforts sont en cours pour créer un tel il est à espérer de tels efforts permettront d'améliorer la reproductibilité des HT / HC SCRE fr hits.
Une limitation de nombre de grands écrans de cellules échelle basée sont qu'elles sont conduites dans les cellules de neuroblastome parce qu'elles peuvent être manipulées génétiquement et cultivé à un grand nombre avec une relative facilité. Cependant, la pertinence des «hits» identifiés dans des modèles ex vivo de culture cellulaire pour la fonction in vivo est discutable, surtout que le cerveau se compose de types de cellules hautement spécialisées qui forment un réseau dense et complexe de connexions synaptiques de fonctionner comme une unité hautement intégrée. En conséquence, il est fréquent que les coups identifiées en utilisant la méthode de dépistage décrite ci-dessus, sont validées en utilisant des techniques d'écrans secondaires additionnels et dans plus physiologique des modèles pertinents 39. Pour améliorer la traduction des hits identifiés lors de HCS, des modèles plus représentatifs et plus sophistiquées, telles que des cellules primaires et des cellules souches différenciées ou de co-culture de systèmes ont besoin d'être développé et adapté pour HT / HC approches.
ntent "> Avec une combinaison de culture cellulaire automatisé et HC imagerie, on peut rapidement gagner de nouveaux aperçus sur le fonctionnement des neurones et de déterminer quelles voies sont importantes pour le développement des maladies. Combinant HCS / HTS données avec des informations générées par d'autres approches en« omique », il sera alors possible de construire une vue d'ensemble la biologie des systèmes des maladies du cerveau, facilitant ainsi le développement thérapeutique.Déclarations de divulgation
Nous n'avons rien à divulguer
Remerciements
Nous tenons à remercier les programmeurs et les spécialistes de Hamilton pour le soutien continu et Blaas Eva pour l'assistance technique. Ce travail a été soutenu par deux subventions d'investissement NWO (911-07-031 et 40-00506-98-10011), Le Prinses Beatrix Fonds Wetenschapsprijs 2009 et les neurosciences Campus Amsterdam; SJ est soutenu par Ti-Pharma: T5-207.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | |
| AI.CELLHOST | HAMILTON | http://www.hamiltonrobotics.com/en-uk/applications/cellomics/ | |
| OPTI-MEM | INVITROGEN | 31985-054 | |
| ACIDE RETINOIQUE | SIGMA-ALDRICH | R2625 | |
| PLAQUES OmniTray | NUNC | 465219 | |
| 96 PLAQUES DE CULTURE BIEN | GRENIER | 655086 | |
| DJ1 ANTICORPS N20 | SANTA CRUZ | SC27004 | |
| ANTICORPS TUBULINE BETA-III | SIGMA-ALDRICH | T3952 | |
| CMXROS Mitotracker | INVITROGEN | M-7512 | |
| Hoechst-33342 | INVITROGEN | H1399 | |
| PEROXYDE D'HYDROGÈNE | SIGMA-ALDRICH | 216 763-100ml | |
| TRYPSINE | INVITROGEN | 25050014 | |
| Phosphate de Dulbecco solution saline tamponnée | INVITROGEN | 14190086 | |
| PROMEGA WIZARD MagneSil TFX | PROMEGA | A2380 | |
| CLONES shARN | OUVERT BIOSYSTEMS | http://www.openbiosystems.com / ARNi / shRNALibraries / TRCLibraryDetails / | |
| BIOAPPLICATIONS Cellomics | THERMO FISHER- | http://www.thermo.com/hcs |
Références
- Geschwind, D. H., Konopka, G. Neuroscience in the era of functional genomics and systems biology. Nature. 461, 908-915 (2009).
- Ge, H., Walhout, A. J., Vidal, M. Integrating 'omic' information: a bridge between genomics and systems biology. Trends. Genet. 19, 551-560 (2003).
- Zhu, H., Snyder, M. 'Omic' approaches for unraveling signaling networks. Curr. Opin. Cell. Biol. 14, 173-179 (2002).
- Jain, S., Heutink, P. From single genes to gene networks: high-throughput-high-content screening for neurological disease. Neuron. 68, 207-217 (2010).
- Manolio, T. A. Finding the missing heritability of complex diseases. Nature. 461, 747-753 (2009).
- Nalls, M. A. Imputation of sequence variants for identification of genetic risks for Parkinson's disease: a meta-analysis of genome-wide association studies. Lancet. 377, 641-649 (2011).
- Simon-Sanchez, J. Genome-wide association study confirms extant PD risk loci among the Dutch. Eur. J. Hum. Genet. 19 (6), 655-661 (2011).
- Van Regenmortel, M. H. Reductionism and complexity in molecular biology. Scientists now have the tools to unravel biological and overcome the limitations of reductionism. EMBO. Rep. 5, 1016-1020 (2004).
- Aherne, G. W., McDonald, E., Workman, P. Finding the needle in the haystack: why high-throughput screening is good for your health. Breast. Cancer. Res. 4, 148-154 (2002).
- An, W. F., Tolliday, N. J. Introduction: cell-based assays for high-throughput screening. Methods. Mol. Biol. 486, 1-12 (2009).
- Mayr, L. M., Bojanic, D. Novel trends in high-throughput screening. Curr. Opin. Pharmacol. 9, 580-588 (2009).
- Hertzberg, R. P., Pope, A. J. High-throughput screening: new technology for the 21st century. Curr. Opin. Chem. Biol. 4, 445-451 (2000).
- Conrad, C., Gerlich, D. W. Automated microscopy for high-content RNAi screening. J. Cell. Biol. 188, 453-461 (2010).
- Thomas, N. High-content screening: a decade of evolution. J. Biomol. Screen. 15, 1-9 (2010).
- Arrasate, M., Finkbeiner, S. Automated microscope system for determining factors that predict neuronal fate. Proc. Natl. Acad. Sci. U. S. A. 102, 3840-3845 (2005).
- Dragunow, M. High-content analysis in neuroscience. Nat. Rev. Neurosci. 9, 779-788 (2008).
- Varma, H., Lo, D. C., Stockwell, B. R. High throughput screening for neurodegeneration and complex disease phenotypes. Comb. Chem. High. Throughput. Screen. 11, 238-248 (2008).
- Durr, O. Robust hit identification by quality assurance and multivariate data analysis of a high-content, cell-based assay. J. Biomol. Screen. 12, 1042-1049 (2007).
- Miller, J. Quantitative relationships between huntingtin levels, polyglutamine length, inclusion body formation, and neuronal death provide novel insight into huntington's disease molecular pathogenesis. J. Neurosci. 30, 10541-10550 (2010).
- Jain, S., Sondervan, D., Rizzu, P., Bochdanovits, Z., Caminada, D., Heutink, P. The Complete Automation of Cell Culture. Journal of Biomolecular Screening. 16 (8), (2011).
- Bonifati, V. Mutations in the DJ-1 gene associated with autosomal recessive early-onset parkinsonism. Science. 299, 256-259 (2003).
- Ricardo, R., Phelan, K. Trypsinizing and Subculturing Mammalian Cells. J. Vis. Exp. (16), e755-e755 (2008).
- Kent, L. Culture and Maintenance of Human Embryonic Stem Cells. J. Vis. Exp. (34), e1427-e1427 (2009).
- Moffat, J. A lentiviral RNAi library for human and mouse genes applied to an arrayed viral high-content screen. Cell. 124, 1283-1298 (2006).
- Volksgezondheid, M. V. Integrale versie van de Regeling genetisch gemodificeerde organismen en het Besluit genetische gemodificeerde. Organismen. , (2004).
- Anderl, J. L., Redpath, S., Ball, A. J. A Neuronal and Astrocyte Co-Culture Assay for High Content Analysis of Neurotoxicity. J. Vis. Exp. (27), e1173-e1173 (2009).
- Wiles, A. M., Ravi, D., Bhavani, S., Bishop, A. J. An analysis of normalization methods for Drosophila RNAi genomic screens and development of a robust validation scheme. J. Biomol. Screen. 13, 777-784 (2008).
- Canet-Aviles, R. M. The Parkinson's disease protein DJ-1 is neuroprotective due to cysteine-sulfinic acid-driven mitochondrial localization. Proc. Natl. Acad. Sci. U. S. A. 101, 9103-9108 (2004).
- Blackinton, J. Formation of a stabilized cysteine sulfinic acid is critical for the mitochondrial function of the parkinsonism protein DJ-1. J. Biol. Chem. 284, 6476-6485 (2009).
- Gupta, P. B. Identification of selective inhibitors of cancer stem cells by high-throughput screening. Cell. 138, 645-659 (2009).
- Luo, J. A genome-wide RNAi screen identifies multiple synthetic lethal interactions with the Ras oncogene. Cell. 137, 835-848 (2009).
- Mouchet, E. H., Simpson, P. B. High-content assays in oncology drug discovery: opportunities and challenges. IDrugs. 11, 422-427 (2008).
- Vogt, A. Automated image-based phenotypic analysis in zebrafish embryos. Dev. Dyn. 238, 656-663 (2009).
- Pardo-Martin, C. High-throughput in vivo vertebrate screening. Nat. Methods. 7, 634-636 (2010).
- Vogt, A., Codore, H., Day, B. W., Hukriede, N. A., Tsang, M. Development of automated imaging and analysis for zebrafish chemical screens. J. Vis. Exp. (40), e1900-e1900 (2010).
- Ross, P. J., Ellis, J. Modeling complex neuropsychiatric disease with induced pluripotent stem cells. F1000. Biol. Rep. 2, 84-84 (2010).
- Ebert, A. D., Svendsen, C. N. Human stem cells and drug screening: opportunities and challenges. Nat. Rev. Drug. Discov. 9, 367-372 (2010).
- An, W. F., Tolliday, N. Cell-based assays for high-throughput screening. Mol. Biotechnol. 45, 180-186 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon