Method Article
Contenuto di screening ad alto contenuto di Malattie Neurodegenerative
In questo articolo
Riepilogo
Noi descriviamo una metodologia che combina coltura automatizzati cella con alto contenuto di imaging per visualizzare e quantificare molteplici processi cellulari e le strutture, in un high-throughput modo. Tali metodi possono aiutare nel ulteriore annotazione funzionale dei genomi, oltre a identificare le reti gene della malattia e potenziali bersagli farmacologici.
Abstract
L'annotazione funzionale dei genomi, la costruzione di reti molecolari e identificazione di droghe nuovo bersaglio, sono sfide importanti che devono essere affrontati come una questione di grande urgenza 1-4. Molteplici approcci complementari 'omiche' hanno fornito indizi utili a capire i fattori di rischio genetici e meccanismi patogenetici alla base di numerose malattie neurodegenerative, ma la maggior parte scoperte richiedono ancora validazione funzionale 5. Per esempio, un recente studio di associazione genome-wide per la malattia di Parkinson (PD), ha individuato loci molti nuovi fattori di rischio per la malattia, ma la variante sottostante causale (s) o meccanismo patogenetico non è noto 6, 7. Come ogni regione associata può contenere diversi geni, la valutazione funzionale di ciascuno dei geni sui fenotipi associati con la malattia, utilizzando le tradizionali tecniche di biologia cellulare sarebbe troppo lungo.
Vi è anche una necessità di comprendere le reti molecolari che colleganomutazioni genetiche per i fenotipi che provocano. Si prevede che i fenotipi della malattia sono il risultato di molteplici interazioni che sono state interrotte. La ricostruzione di queste reti con i tradizionali metodi molecolari sarebbe tempo. Inoltre, le previsioni di rete da studi indipendenti di singoli componenti, l'approccio di riduzionismo, probabilmente sottovaluta la complessità della rete 8. Questa sottovalutazione potrebbe, in parte, spiegare il basso tasso di successo di approvazione dei farmaci a causa degli effetti collaterali o tossici. Ottenere una prospettiva di rete di percorsi malattie correlate con HT / HC approcci di screening cellulare e identificare i nodi chiave all'interno di questi percorsi, potrebbe portare alla individuazione di obiettivi che sono più adatti per un intervento terapeutico.
High-throughput screening (HTS) è una metodologia ideale per affrontare tali questioni 9-12. ma i metodi tradizionali sono stati uni-dimensionale con tutto il bene saggi cellulari, che utilizzati semplificaSTIC letture per i complessi processi biologici. Essi sono stati in grado di quantificare simultaneamente i fenotipi osservati in molte malattie neurodegenerative, come il deficit di trasporto assonale o alterazioni nelle proprietà morfologia 13, 14. Questo approccio non potrebbe essere utilizzato per indagare la natura dinamica dei processi cellulari o eventi patogeni che si verificano in un sottogruppo di cellule. Per quantificare tali caratteristiche si deve passare al multi-dimensionale fenotipi definito alto contenuto di screening (HCS) 4, 15-17. HCS è la cellula-base quantificazione dei processi contemporaneamente, che fornisce una rappresentazione più dettagliata della risposta cellulare a perturbazioni diverse rispetto a HTS.
HCS ha molti vantaggi rispetto HTS 18, 19, ma conducendo una high-throughput (HT)-alto contenuto (HC) schermo nei modelli neuronali è problematico a causa dei costi elevati, variazioni ambientali ed errori umani. Al fine di individuare le risposte cellulari su scala 'phenomics'HC utilizzando immagini si deve ridurre le variazioni ed errori, aumentando la sensibilità e riproducibilità.
Qui descriviamo un metodo per condurre in modo accurato e affidabile schermi shRNA utilizzando colture di cellule automatizzati 20 e HC di imaging neuronale in modelli cellulari. Ci descrivono come abbiamo utilizzato questa metodologia per identificare modulatori per una particolare proteina, DJ1, che se mutato causa parkinsonismo autosomico recessivo 21.
Combinando la versatilità di HC imaging con metodi HT, è possibile quantificare con precisione una pletora di fenotipi. Questo potrebbe essere successivamente utilizzato per far avanzare la nostra comprensione del genoma, i meccanismi coinvolti nella patogenesi della malattia e identificare potenziali bersagli terapeutici.
Protocollo
1. Automatizzata aderente cultura processo cellulare (Figura 1) 20
- Preparare il sistema automatizzato di coltura cellulare per l'inserimento di piastre di coltura cellulare. Carico di consumo (es. puntali, piastre di coltura cellulare, piatti saggio) nel sistema utilizzando l'interfaccia grafica utente (GUI). Assicurarsi che ci sia sufficiente terreni di coltura cellulare, tampone fosfato (PBS) e tripsina nel sistema robotico.
- Manualmente seme due piastre omnitray con 2 x 10 6 cellule per piastra, delle SH-SY5Y linea di cellule di neuroblastoma. Mantenere le cellule in Opti-MEM con il 10% di siero fetale bovino (FBS). Mettere i piatti nel robot coltura cellulare utilizzando la GUI. Le cellule verranno incubate a 37 ° C e 5% di CO 2.
- Scegliere quale delle cellule protocollo cultura ha bisogno di essere iniziato 20. Si può scegliere tra un processo di coltura cellulare aderente 22, coltura ed espansione di staminali embrionali (ES), le cellule sulle cellule del mouse alimentatore 23 o la coltura di sospensione cells 22.
- Selezionare il protocollo aderente colture cellulari (Figura 1) e garantire aderente cellulare file di parametri specifica linea sono regolate in modo che la soglia di confluenza (l'area del piatto omnitray che contiene le cellule) è fissato al 70%. Impostare il tempo totale tripsinizzazione a due minuti.
- Istruire il robot per preparare piatti omnitray nuovo, con un numero di cellule semina di 2 x 10 6 cellule per piastra.
- Omnitray piastre di ingresso nel sistema di coltura cellulare utilizzando il protocollo automatizzato aderente colture cellulari (Figura 1). Questo protocollo prevede le seguenti fasi: le piastre vengono incubate e ripreso fino a raggiungere la soglia prestabilita, confluenza. Se le cellule non raggiungono la soglia di confluenza entro 5 letture, le piastre vengono rimossi dal sistema. Una volta raggiunta la soglia di confluenza definiti dall'utente, le cellule vengono lavate, trypsinized e contati. Un pre-definito numero di cellule sono aggiunti alle piastre omnitray nuovo e se ci sono un numero sufficiente di cellule, un insensibile specificatoer saggio di piastre vengono trasportati al ponte e un numero definito di cellule erogato in ciascun pozzetto. Piastre di test può essere ripreso direttamente utilizzando il microscopio integrato o di uscita dal sistema per ulteriori elaborazioni.
- Istruire il sistema per preparare 4 piatti test per piastra omnitray, con un totale di 5.000 cellule per pozzetto.
2. shRNA produzione di virus e placcatura in piastre di test (Tempo di percorrenza: 6 giorni)
- Crescono le scorte glicerolo batterico contenente i vettori shRNA (Open Biosystems, TRC1) pernottamento in 2 ml di Luria-Bertani supporti media contenente 100 mg / ml di ampicilin (Sigma-Aldrich).
- Estratto di plasmidi seguendo il protocollo del produttore (Promega Wizard MagneSil TFX).
- Produrre dei virus mediante RNAi Consorzio High-Throughput Produzione lentivirali (96 pozzetti) protocollo 24. Lavorare con i lentivirus è relativamente sicuro, perché le particelle del virus utilizzato per la trasduzione si replica con deficit e split-strategie di imballaggio gene sono utilizzati per la loro produzione. Tuttavia, quando si lavora con lentivirus, ulteriori procedure di biosicurezza sono necessarie per minimizzare il rischio per se stessi e gli altri 25. Tutti gli esperimenti devono essere condotti in un laboratorio di livello MLII o BSL2 sicurezza. Tutte le plastiche (pipette, piatti di plastica, i media) che sono stati in contatto con particelle di lentivirus devono essere incubate con candeggina per 24 ore prima dello smaltimento.
- Calcolare la molteplicità di infezione (MOI) del lentivirus determinando la percentuale di cellule GFP positive con il GFP pLKO.1 plasmide (Sigma-Aldrich).
- Lentivirus piastra in piastre di test, con una MOI di 3.
3. Trasduzione lentivirali e la differenziazione neuronale delle cellule SH-SY5Y (Tempo di percorrenza: 6 giorni)
- Le cellule sono aggiunti alle piastre test (vedi passo 1.7). Piastre di test di carico contenenti lentivirus shRNA nel sistema automatizzato di coltura cellulare.
- Dopo 24 ore, i media sulpiastre test verrà modificato in Opti-MEM contenente FBS 0,5% e 0,1 mM acido retinoico per iniziare il processo di differenziazione. Differenziazione delle cellule SH-SY5Y permette la visualizzazione di strutture neuritiche e sincronizza la divisione cellulare.
- Continua l'incubazione delle piastre saggio sui media differenziazione per 5 giorni. Questo assicura la massima knockdown di espressione del gene bersaglio.
- Il giorno 5, aggiungere 50 mM H 2 O 2 alla metà delle piastre test per 24 ore per stimolare la traslocazione di DJ1 ai mitocondri.
- Il giorno 6, aggiungere CmxROS Mitotracker (Invitrogen) alle celle, ad una concentrazione finale di 200 Nm per pozzetto e incubare a 37 ° C per 30 minuti.
- Si può istruire il sistema di piastre immagine direttamente mediante l'imager HC o piastre possono essere esportati dal sistema per ulteriori elaborazioni.
4. Immunocolorazione automatizzata delle piastre test (Tempo di percorrenza: 2 giorni)
La qualità dell'immagine è paramount per lo svolgimento di un sensibile e affidabile HCS. Danni al monostrato cellulare a causa di pipettaggio inesatte possono portare a scarsa qualità dell'immagine e dei risultati non riproducibili. Al fine di minimizzare i danni strato di cellule, il immunocolorazione è stata condotta utilizzando una stazione robotica. La procedura è simile a uno che è stato descritto in precedenza 26 ma è stato personalizzato per aumentare la produttività e ridurre l'utilizzo di materiali di consumo.
- Fissare le cellule con 100 ml di paraformaldeide 4% pre-riscaldato a 37 ° C. Incubare per 20 minuti a temperatura ambiente.
- Lavare le cellule con 200 ml di PBS per 5 minuti, 3 volte.
- Incubare le piastre test con 200 ml di PBS contenente 0,1% Triton (PBST) per 10 minuti.
- Lavare le cellule con 200 ml di PBS per 5 minuti, 3 volte.
- Incubare le piastre test con 200 ml di buffer di blocco (PBST con il 5% FBS) per 1 ora a temperatura ambiente.
- Lavare le cellule con 200 ml di PBS per 5 minuti, 3 volte.
- Incubare con il folla causa anticorpi primari notte a 4 ° C:
- Capra DJ1 N20 (Santa Cruz, 5 mcg / ml)
- Coniglio β-III tubulina (Sigma-Aldrich, 1 mg / ml)
- Il giorno seguente, lavare le cellule con 200 ml di PBS per 5 minuti, 3 volte.
- Incubare le piastre test con i seguenti anticorpi secondari 1 ora a temperatura ambiente:
- AlexaFluor 488 asino anti-capra (Invitrogen, 2 mg / ml)
- AlexaFluor 647 capra anti-coniglio (Invitrogen, 2 mg / ml
- Lavare le cellule con 200 ml di PBS per 5 minuti, 3 volte.
- Incubare le cellule con la Hoechst (Invitrogen, 1 mg / ml) per 10 minuti.
- Lavare le cellule con 200 l PBS per 5 minuti, 3 volte.
- Conservare le piastre a 4 ° C fino a che non possono essere esposte.
5. Alto contenuto di immagine acquisizione e analisi delle immagini (Tempo richiesto: 5 giorni)
- Immagine per un totale di 30 campi per pozzetto utilizzando la lente dell'obiettivo 20x. Visualizza DJ1 con la FITC filtro impostato, i mitocondri con il set TRITC filtro, β-tubulina III con l'insieme Cy5 filtro e dei nuclei utilizzando il set di filtro UV (Figura 3).
- Analizzare le immagini utilizzando la Bioapplication Compartimentale Analysis (Cellomics, ThermoFisher) per determinare l'intensità media del segnale Mitotracker all'interno dei mitocondri. (Figura 4B, F).
- Per determinare il coefficiente medio di sovrapposizione tra DJ1 ei mitocondri, analizzare le immagini utilizzando la bioapplication Cellomics Colocalisation (Cellomics, ThermoFisher). Definire le aree di interesse (ROI) nel modo seguente: ROI A - nucleo (4A, E), il ROI B - mitocondri (Figura 4 B, F). Escludi ROI ROI A da B per garantire l'analisi di soltanto nel citoplasma. Definire i mitocondri come regione obiettivo I e DJ1 come obiettivo regione II (Figura 4C, G).
- Analizzare le immagini utilizzando la bioapplication neuronale Profiling (Cellomics, Thermofisher) per tracciare la lunghezza media dei neuriti dal β-tubulina III colorazione (Figura4D, H).
- Piastre immagine utilizzando l'Opera LX automatizzato confocale lettore (Perkin-Elmer). Immagine per un totale di 30 campi per pozzetto utilizzando la lente 60x obiettivo con immersione in acqua. Visualizza mitocondri con il laser 561 nM e nuclei con eccitazione UV.
- Analizzare le immagini utilizzando il Spot-Edge-Ridge (SER) texture algoritmo caratteristiche. La SER-Ridge filtro trasmette intensità in pixel che formano cresta simile a modelli. La più frammentati i mitocondri, maggiore è il SER-Ridge score (Figura 8).
6. Normalizzazione dei dati e analisi
- Importare i dati dal software di analisi delle immagini nel pacchetto Bioconductor CellHTS2 per l'ambiente software R (R versione 2.11.1, la versione Bioconductor 2.6).
- Logaritmo in base (2) trasformare i dati prima di ogni normalizzazione piastra mediana a base 27, 28. Non applicare la regolazione varianza per piastra.
- Per identificare modificatori di un fenotipo, utilizzare due ANOVA strada tra la different i gruppi di trattamento ovvero strapazzate infettate cellule non trattate vs strapazzate tossina cellule trattate rispetto a cellule non trattate rispetto al gene bersaglio le cellule trattate gene bersaglio (figure 5-7).
7. Rappresentante risultati
Le mutazioni all'interno DJ1 dar luogo a esordio precoce-recessiva parkinsonismo 21, ma non è chiaro come la perdita di DJ1 dà origine al fenotipo della malattia. E 'noto che le cellule carenti di DJ1 sono più suscettibili a stress ossidativo indotto la morte cellulare e nella risposta allo stress ossidativo, DJ1 trasloca dal citoplasma ai mitocondri 29, 30. Mediante la costruzione di test HC di monitorare questi fenotipi, siamo in grado di identificare i geni che regolano o influenzano fenotipi associati con DJ1. Questo approccio può aiutare a decifrare i percorsi all'interno del quale DJ1 funzioni e che potrebbero essere coinvolti nella patogenesi della malattia.
Esempio di interazione con epistatico DJ1 (Figura 5): Atterramento di DJ1 in cellule esposteai risultati tossina in una maggiore perdita di vitalità cellulare (BAR-B: image-B) rispetto alle cellule infettate con lentivirus strapazzate (BAR-A: image-A). Knockdown del gene bersaglio A ha un effetto simile a quella osservata nelle cellule con un knockdown DJ1 (BAR-C: image-C). Knockdown sia DJ1 e gene bersaglio Un traduce in una perdita significativamente maggiore di vitalità cellulare di perdita di entrambi i geni da soli (BAR-D: image-D). Questo suggerisce una interazione tra epistatico DJ1 e target gene A.
Esempio di un gene che regola il DJ1 traslocazione (Figura 6): Quando le cellule sono esposte ad una tossina, DJ1 trasloca dal citoplasma ai mitocondri, che è quantificato da un coefficiente maggiore sovrapposizione tra DJ1 ei mitocondri (BAR-A: image A contro BAR-C: image C). Nelle cellule B in cui gene bersaglio è stato messo a tacere, meno DJ1 trasloca ai mitocondri quando le cellule sono esposte alla tossina. Ciò suggerisce che il gene B obiettivo è coinvolto nel trasporto di DJ1 ai mitocondri. (BAR-B: immagine B e BAR-D: image D)
Esempio di un gene coinvolto nella crescita neuronale (Figura 7): Atterramento di C gene bersaglio nel selvaggio tipo SH-SY5Y cellule si traduce in un significativo aumento della lunghezza dei neuriti (BAR-B: image-B) rispetto alle cellule infettate con lentivirus che esprimono strapazzate shRNA (BAR-A: image A). Questo effetto si perde nelle cellule incubate con tossina (BAR-C e D).
Esempio di un gene coinvolto nella morfologia mitocondriale (Figura 8): L'infezione di tipo selvaggio cellule SH-SY5Y con shRNA gene targeting D provocano una diminuzione della mitocondriale SER-Ridge valore di segmentazione (Figura 8, Immagine C e D) rispetto ai cellule infettate con lentivirus strapazzate (Figura 8, Immagine A e B).
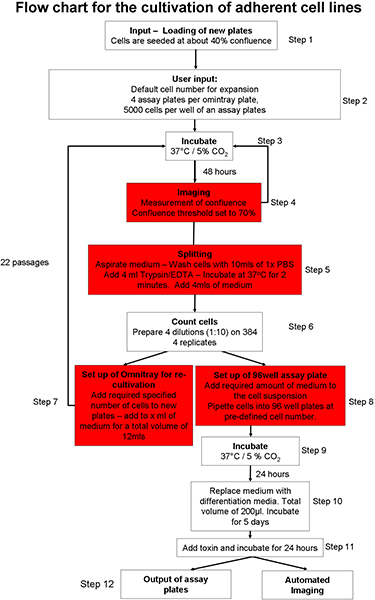
Figura 1 Schema di protocollo automatizzato colture cellulari.:
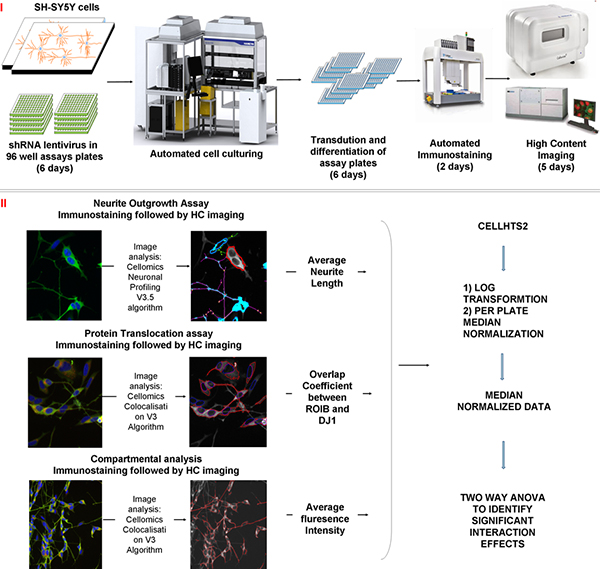
Figura 2 Schema del processo di screening, l'analisi delle immagini e dei metodi statistici utilizzati durante il processo di screening:. I) Le cellule sono coltivate fino a quando non sono confluenti e successivamente cromato nelle piastre test contenente il lentivirus shRNA. Le cellule sono differenziate per 5 giorni e la tossina viene quindi aggiunta alle piastre per 24 ore. Piastre di test sono uscita dal sistema e immunoistochimica. La quantità di tempo impiegato per ciascuno dei processi è indicato tra parentesi. ii) I dati sono acquisiti mediante un sensore HC (la vitalità delle cellule, proteine e traslocazione escrescenza neuronale) e un imager automatizzato confocale (morfologia mitocondriale). I dati vengono esportati in CellHTS2 pacchetto entro R, base (2) log trasformati e normalizzati. ANOVA a due vie viene utilizzato per identificare interazioni significative tra le diverse variabili.

Figura 3. Immagini composite di cellule acquisita da HC imaging. A) le cellule non trattate, B) Le cellule trattate con H 2 O 2. DJ1 è etichettato in verde, i mitocondri in rosso e in blu i nuclei. Colorazione dei neuriti non è evidenziato.
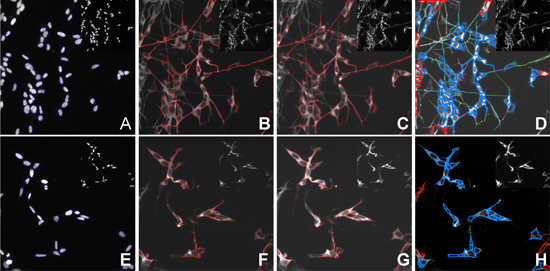
Figura 4. Quantificazione delle tante caratteristiche cellulari ottenute da un HCS. DC) non trattato cellule SH-SY5Y. EH) H 2 O 2 trattati con cellule SH-SY5Y. A, E) Nuclei segmentazione e la definizione del ROI A, B, F) Identificazione e quantificazione dei mitocondri, ROI B, C, G) Identificazione di DJ1, Target canale II, D, G) Identificazione e calcolo della durata media dei neuriti. Le cellule vicino al bordo dell'immagine sono esclusi dall'analisi. Inset immagini sono immagini prima dell'analisi.

Figura 5. Identificazione di un gene in epistasi con DJ1 (lettere sulle barre corrispondono alle scritte sulle immagini). Immagini dalla A alla D sono cellule SH-SY5Y etichettati con Mitotracker CMXRos (rosso) che è stato utilizzato per la quantificazione della salute delle cellule.
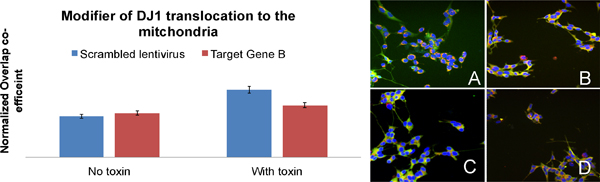
Figura 6. Identificazione di un gene che regola il DJ1 traslocazione (lettere sulle barre corrispondono alle scritte sulle immagini). Immagini dalla A alla D sono cellule SH-SY5Y etichettato per DJ1 (verde), mitocondri (rosso) e nuclei (blu) che sono stati utilizzati per la quantificazione della traslocazione DJ1 ai mitocondri.
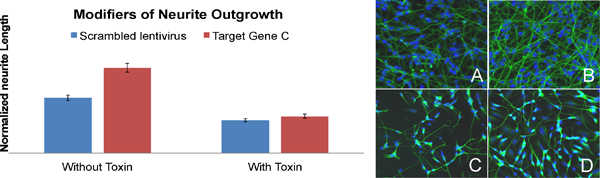
Figura 7. Identificazione di un gene coinvolto nella crescita neuronale (lettere sulle barre corrispondono alle scritte sulle immagini). Immagini dalla A alla D sono cellule SH-SY5Y etichettato per la β-III tubulina (verde) e nuclei (blu) che sono stati utilizzati per la quantificazione della lunghezza dei neuriti.
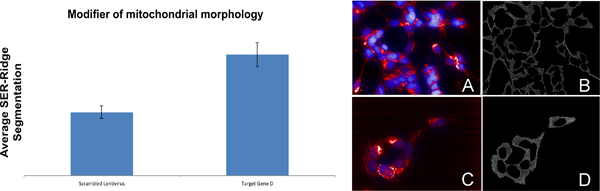
Figura 8. Identificazione di un gene coinvolto nella regolazione della morfologia mitocondriale. Immagine A e C sono immagini composite di SH-SY5Y cellule infettate con shRNA strapazzate o un gene targeting shRNA D rispettivamente. I mitocondri sono colorati in rosso, mentre i nuclei sono colorati in blu. Immagine B e D sono visualizzazioni di SER-Ridge quantificazione.
Discussione
Con la diminuzione dei costi di HT / HC sistemi di screening cellulari, unita alla disponibilità di potenti genoma strumenti per modificare la funzione del gene, HT / HC schermi stanno diventando comune nel mondo accademico. L'approccio è già stato applicato con successo a diverse aree di ricerca quali l'identificazione di bersagli farmacologici nel cancro 9, 31-33 e 34-36 lo sviluppo embrionale e ha anche potenzialità di applicazione nel decifrare i meccanismi coinvolti nei disordini neuropsichiatrici 37,38. Tuttavia implementazione di tale sistema richiede un investimento significativo di tempo e fatica con l'ottimizzazione dei processi che spesso prende un minimo di 6 mesi. Tutte le fasi, quali i tempi di tripsinizzazione, velocità di pipettaggio e densità di semina devono essere adeguati, per assicurare che le cellule sono sane e crescono costantemente. Prevenzione della contaminazione batterica è una delle sfide più difficili di fronte cultura automatizzata delle cellule con protocolli di pulizia settimanale in combination con risciacquo costante di tutte le linee di rilevamento liquidi con etanolo al 70% sia necessario per la contaminazione delle culture libero. Sarà inoltre necessario migliorare la robotica in modo che gli strumenti aggiuntivi, come microscopi confocale per una maggiore risoluzione e -80 ° C congelatori per la conservazione composto può essere integrato.
Ci sono anche limitazioni che devono essere affrontate per migliorare la sensibilità, la velocità e l'utilità di questo metodo per lo studio delle reti geniche e identificare i geni coinvolti nei meccanismi patogeni molecolari.
Per condurre un HT / HC schermo e garantire che dati affidabili raccolti, diversi aspetti devono essere ottimizzati. In primo luogo, l'affidabilità della misura è fondamentale e dipende dalla robustezza e la sensibilità del test. Ad esempio, il test sopra descritti sono adatti per schermi di piccole dimensioni, ma sono difficili da implementare su larga scala del genoma, a causa di fasi di lavorazione il numero richiesto prima di acquisizione delle immagini.Quindi, bisognerebbe costruire linee cellulari stabili che esprimono il gene reporter, che consentirebbe per l'imaging diretto e portare a variazioni diminuito a causa del ridotto numero di fasi di lavorazione. Attualmente, la progettazione di un saggio che descrive in modo accurato e affidabile quantifica un fenotipo di interesse è un serio ostacolo nel processo di selezione HC.
Molte schermate sono condotti in cellule di mammifero utilizzando diverse librerie di RNAi, che soffrono di off-target effetti, limitata efficienza silenziamento genico e la copertura del genoma incompleto. Così le biblioteche devono essere effettuate, che sono più specifici, potenti e hanno una migliore copertura. Si sta cercando di creare tale si spera tali sforzi a migliorare la riproducibilità di HT / HC SCRE it hits.
Una limitazione di scala molti schermi a grandi cellule base è che sono condotti in cellule di neuroblastoma perché possono essere geneticamente manipolate e colto ad un gran numero con relativa facilità. Tuttavia, la rilevanza delle 'hit' identificati in modelli ex vivo di coltura cellulare in funzione in vivo è discutibile, soprattutto perché il cervello è composto di tipi di cellule altamente specializzate che formano una fitta rete e complicato delle connessioni sinaptiche di funzionare come unità altamente integrata. Di conseguenza, è frequente che colpisce identificato utilizzando l'approccio di screening di cui sopra, sono convalidati in schermi secondari utilizzando tecniche aggiuntive e in più modelli fisiologici rilevanti 39. Per migliorare la traduzione di visite identificati durante HCS, i modelli più rappresentativi e sofisticati, come le cellule primarie e cellule staminali differenziate o co-coltura sistemi devono essere sviluppati e adattati per HT / HC approcci.
ntent "> Con una combinazione di coltura delle cellule automatizzati e HC di imaging si può rapidamente acquisire nuove conoscenze sul modo in cui i neuroni funzione e determinare quali percorsi sono importanti per lo sviluppo della malattia. Combinando HCS / HTS dati con le informazioni generate da altri approcci 'omiche', sarà poi sarà possibile costruire una visione d'insieme biologia dei sistemi di malattie cerebrali, facilitando così lo sviluppo terapeutico.Divulgazioni
Non abbiamo nulla da rivelare
Riconoscimenti
Ringraziamo i programmatori Hamilton e specialisti per il supporto continuo e Blaas Eva per l'assistenza tecnica. Questo lavoro è stato supportato da due sovvenzioni agli investimenti NWO (911-07-031 e 40-00506-98-10011), Il Prinses Beatrix Fonds Wetenschapsprijs 2009 e il Campus di Neuroscienze di Amsterdam; SJ è supportato da Ti-Pharma: T5-207.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
| AI.CELLHOST | HAMILTON | http://www.hamiltonrobotics.com/en-uk/applications/cellomics/ | |
| OPTI-MEM | Invitrogen | 31985-054 | |
| Acido Retinoico | SIGMA-ALDRICH | R2625 | |
| OMNITRAY PIASTRE | NUNC | 465219 | |
| 96 piastre di coltura BENE | GRENIER | 655086 | |
| DJ1 N20 ANTICORPI | SANTA CRUZ | SC27004 | |
| BETA-III TUBULINA ANTICORPI | SIGMA-ALDRICH | T3952 | |
| MITOTRACKER CMXROS | Invitrogen | M-7512 | |
| HOECHST-33342 | Invitrogen | H1399 | |
| Perossido di idrogeno | SIGMA-ALDRICH | 216763-100ML | |
| Tripsina | Invitrogen | 25050014 | |
| FOSFATO Dulbecco Buffered Saline | Invitrogen | 14190086 | |
| PROMEGA WIZARD MagneSil TFX | PROMEGA | A2380 | |
| ShRNA CLONI | APERTO BIOSISTEMI | http://www.openbiosystems.com / RNAi / shRNALibraries / TRCLibraryDetails / | |
| CELLOMICS BIOAPPLICATIONS | THERMO-FISHER | http://www.thermo.com/hcs |
Riferimenti
- Geschwind, D. H., Konopka, G. Neuroscience in the era of functional genomics and systems biology. Nature. 461, 908-915 (2009).
- Ge, H., Walhout, A. J., Vidal, M. Integrating 'omic' information: a bridge between genomics and systems biology. Trends. Genet. 19, 551-560 (2003).
- Zhu, H., Snyder, M. 'Omic' approaches for unraveling signaling networks. Curr. Opin. Cell. Biol. 14, 173-179 (2002).
- Jain, S., Heutink, P. From single genes to gene networks: high-throughput-high-content screening for neurological disease. Neuron. 68, 207-217 (2010).
- Manolio, T. A. Finding the missing heritability of complex diseases. Nature. 461, 747-753 (2009).
- Nalls, M. A. Imputation of sequence variants for identification of genetic risks for Parkinson's disease: a meta-analysis of genome-wide association studies. Lancet. 377, 641-649 (2011).
- Simon-Sanchez, J. Genome-wide association study confirms extant PD risk loci among the Dutch. Eur. J. Hum. Genet. 19 (6), 655-661 (2011).
- Van Regenmortel, M. H. Reductionism and complexity in molecular biology. Scientists now have the tools to unravel biological and overcome the limitations of reductionism. EMBO. Rep. 5, 1016-1020 (2004).
- Aherne, G. W., McDonald, E., Workman, P. Finding the needle in the haystack: why high-throughput screening is good for your health. Breast. Cancer. Res. 4, 148-154 (2002).
- An, W. F., Tolliday, N. J. Introduction: cell-based assays for high-throughput screening. Methods. Mol. Biol. 486, 1-12 (2009).
- Mayr, L. M., Bojanic, D. Novel trends in high-throughput screening. Curr. Opin. Pharmacol. 9, 580-588 (2009).
- Hertzberg, R. P., Pope, A. J. High-throughput screening: new technology for the 21st century. Curr. Opin. Chem. Biol. 4, 445-451 (2000).
- Conrad, C., Gerlich, D. W. Automated microscopy for high-content RNAi screening. J. Cell. Biol. 188, 453-461 (2010).
- Thomas, N. High-content screening: a decade of evolution. J. Biomol. Screen. 15, 1-9 (2010).
- Arrasate, M., Finkbeiner, S. Automated microscope system for determining factors that predict neuronal fate. Proc. Natl. Acad. Sci. U. S. A. 102, 3840-3845 (2005).
- Dragunow, M. High-content analysis in neuroscience. Nat. Rev. Neurosci. 9, 779-788 (2008).
- Varma, H., Lo, D. C., Stockwell, B. R. High throughput screening for neurodegeneration and complex disease phenotypes. Comb. Chem. High. Throughput. Screen. 11, 238-248 (2008).
- Durr, O. Robust hit identification by quality assurance and multivariate data analysis of a high-content, cell-based assay. J. Biomol. Screen. 12, 1042-1049 (2007).
- Miller, J. Quantitative relationships between huntingtin levels, polyglutamine length, inclusion body formation, and neuronal death provide novel insight into huntington's disease molecular pathogenesis. J. Neurosci. 30, 10541-10550 (2010).
- Jain, S., Sondervan, D., Rizzu, P., Bochdanovits, Z., Caminada, D., Heutink, P. The Complete Automation of Cell Culture. Journal of Biomolecular Screening. 16 (8), (2011).
- Bonifati, V. Mutations in the DJ-1 gene associated with autosomal recessive early-onset parkinsonism. Science. 299, 256-259 (2003).
- Ricardo, R., Phelan, K. Trypsinizing and Subculturing Mammalian Cells. J. Vis. Exp. (16), e755-e755 (2008).
- Kent, L. Culture and Maintenance of Human Embryonic Stem Cells. J. Vis. Exp. (34), e1427-e1427 (2009).
- Moffat, J. A lentiviral RNAi library for human and mouse genes applied to an arrayed viral high-content screen. Cell. 124, 1283-1298 (2006).
- Volksgezondheid, M. V. Integrale versie van de Regeling genetisch gemodificeerde organismen en het Besluit genetische gemodificeerde. Organismen. , (2004).
- Anderl, J. L., Redpath, S., Ball, A. J. A Neuronal and Astrocyte Co-Culture Assay for High Content Analysis of Neurotoxicity. J. Vis. Exp. (27), e1173-e1173 (2009).
- Wiles, A. M., Ravi, D., Bhavani, S., Bishop, A. J. An analysis of normalization methods for Drosophila RNAi genomic screens and development of a robust validation scheme. J. Biomol. Screen. 13, 777-784 (2008).
- Canet-Aviles, R. M. The Parkinson's disease protein DJ-1 is neuroprotective due to cysteine-sulfinic acid-driven mitochondrial localization. Proc. Natl. Acad. Sci. U. S. A. 101, 9103-9108 (2004).
- Blackinton, J. Formation of a stabilized cysteine sulfinic acid is critical for the mitochondrial function of the parkinsonism protein DJ-1. J. Biol. Chem. 284, 6476-6485 (2009).
- Gupta, P. B. Identification of selective inhibitors of cancer stem cells by high-throughput screening. Cell. 138, 645-659 (2009).
- Luo, J. A genome-wide RNAi screen identifies multiple synthetic lethal interactions with the Ras oncogene. Cell. 137, 835-848 (2009).
- Mouchet, E. H., Simpson, P. B. High-content assays in oncology drug discovery: opportunities and challenges. IDrugs. 11, 422-427 (2008).
- Vogt, A. Automated image-based phenotypic analysis in zebrafish embryos. Dev. Dyn. 238, 656-663 (2009).
- Pardo-Martin, C. High-throughput in vivo vertebrate screening. Nat. Methods. 7, 634-636 (2010).
- Vogt, A., Codore, H., Day, B. W., Hukriede, N. A., Tsang, M. Development of automated imaging and analysis for zebrafish chemical screens. J. Vis. Exp. (40), e1900-e1900 (2010).
- Ross, P. J., Ellis, J. Modeling complex neuropsychiatric disease with induced pluripotent stem cells. F1000. Biol. Rep. 2, 84-84 (2010).
- Ebert, A. D., Svendsen, C. N. Human stem cells and drug screening: opportunities and challenges. Nat. Rev. Drug. Discov. 9, 367-372 (2010).
- An, W. F., Tolliday, N. Cell-based assays for high-throughput screening. Mol. Biotechnol. 45, 180-186 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon