Method Article
Screening alto teor em Doenças Neurodegenerativas
Neste Artigo
Resumo
Nós descrevemos uma metodologia que combina cultura de células automatizado com alto conteúdo de imagem para visualizar e quantificar vários processos celulares e estruturas, de uma forma de alto rendimento. Tais métodos podem auxiliar na anotação de genomas ainda mais funcional, bem como identificar redes de doença genética e alvos terapêuticos potenciais.
Resumo
A anotação funcional de genomas, construção de redes e identificação molecular romance alvo da droga, são desafios importantes que precisam ser tratadas como uma questão de grande urgência 1-4. Múltiplas abordagens complementares a genómica forneceram pistas sobre os fatores de risco genéticos e mecanismos patogênicos subjacentes inúmeras doenças neurodegenerativas, mas a maioria dos achados ainda necessitam de validação funcional 5. Por exemplo, um recente estudo de associação genômica ampla para a Doença de Parkinson (DP), identificou muitos loci novos fatores de risco para a doença, mas a variante subjacente causal (s) ou mecanismo patogênico não é conhecida 6, 7. Como cada região associada pode conter vários genes, a avaliação funcional de cada um dos genes em fenótipos associados com a doença, utilizando técnicas de biologia celular tradicional levaria muito tempo.
Há também uma necessidade de compreender as redes moleculares que apontammutações genéticas para os fenótipos que elas causam. Espera-se que fenótipos da doença são o resultado de múltiplas interações que foram rompidas. Reconstrução dessas redes usando os tradicionais métodos moleculares seria demorado. Além disso, as previsões de rede de estudos independentes de componentes individuais, a abordagem reducionista, provavelmente subestimam a complexidade da rede 8. Essa subestimação poderia, em parte, explicar a baixa taxa de sucesso de aprovação da droga devido aos efeitos colaterais indesejáveis ou tóxicos. Ganhando uma perspectiva de rede de vias de doenças relacionadas com HT / HC abordagens de triagem celular, e identificar os nós-chave dentro dessas vias, poderiam levar à identificação de alvos que são mais adequados para a intervenção terapêutica.
High-throughput screening (HTS) é uma metodologia ideal para abordar estas questões 9-12. mas os métodos tradicionais foram unidimensional ensaios de todo o bem celular, que usava simplistic leituras de processos biológicos complexos. Eram incapazes de quantificar simultaneamente os fenótipos observados em muitas doenças neurodegenerativas, tais como déficits de transporte axonal ou alterações nas propriedades morfologia 13, 14. Esta abordagem não poderia ser usado para investigar a natureza dinâmica de processos celulares ou eventos patogênicos que ocorrem em um subconjunto de células. Para quantificar tais características tem de se mudar para multi-dimensional fenótipos denominado de alto teor de triagem (HCS) 4, 15-17. HCS é a quantificação baseada em células de vários processos simultaneamente, o que fornece uma representação mais detalhada da resposta celular a perturbações diversas em relação ao HTS.
HCS tem muitas vantagens sobre HTS 18, 19, mas a realização de um high-throughput (HT) de alta o conteúdo da tela (HC) em modelos neuronal é problemático devido ao alto custo, a variação ambiental e erro humano. A fim de detectar respostas celulares em uma escala 'phenomics'usando uma imagem HC tem que reduzir a variação e erro, enquanto aumentando a sensibilidade e reprodutibilidade.
Aqui nós descrevemos um método para realizar com precisão e confiabilidade telas shRNA utilizando cultura de células automatizado 20 e HC de imagem em modelos celulares neuronais. Descrevemos como nós usamos esta metodologia para identificar moduladores de uma determinada proteína, DJ1, que quando mutado causa parkinsonismo autossômica recessiva 21.
Combinando a versatilidade do HC de imagem com métodos de HT, é possível quantificar com precisão um grande número de fenótipos. Esta situação pode ser utilizada para fazer avançar a nossa compreensão do genoma, os caminhos envolvidos na patogênese da doença, bem como identificar potenciais alvos terapêuticos.
Protocolo
1. Processo de cultura de células aderentes automatizado (Figura 1) 20
- Preparar o sistema de cultura de células automatizado para a entrada das placas de cultura de células. Consumíveis de carga (por exemplo, pontas de pipeta, placas de cultura celular, placas de ensaio) para o sistema utilizando a interface gráfica do usuário (GUI). Garantir que há meios de cultura de células suficiente, tampão fosfato salino (PBS) e tripsina no sistema robótico.
- Propagar manualmente duas placas omnitray com 2 x 10 6 células por placa, da linhagem de células SH-SY5Y neuroblastoma. Manter as células em Opti-MEM com 10% de soro fetal bovino (FBS). Coloque as placas em cultura de células do robô usando o GUI. As células serão incubadas a 37 ° C e 5% CO 2.
- Escolha qual célula protocolo de cultura precisa ser iniciado 20. Pode-se escolher a partir de um processo de cultura de células aderentes 22, cultivo e expansão do tronco embrionárias (ES) células em células de camundongo alimentador 23 ou cultura de suspensão cells 22.
- Selecione o protocolo de cultura de células aderentes (Figura 1) e garantir a aderência de células line arquivos de parâmetros específicos são ajustados para que o limite de confluência (a área da placa omnitray que contém células) é fixado em 70%. Definir o tempo de tripsinização total para dois minutos.
- Instruir o robô a preparar pratos omnitray novo, com um número de celular semeadura de 2 x 10 6 células por placa.
- Placas de entrada omnitray no sistema de cultura celular utilizando o protocolo automatizado cultura de células aderentes (Figura 1). Este protocolo envolve as seguintes etapas: as placas são incubadas e fotografada até atingirem o limite de confluência pré-definidos. Se as células não atingir o limiar confluência dentro de 5 leituras, as placas são removidas do sistema. Ao chegar à confluência limiar definido pelo usuário, as células são lavadas, tripsinizados e contadas. Um número pré-definido de células são adicionados às placas omnitray novo e se houver um número suficiente de células, um numb especificadoer de placas de ensaio são transportados para o convés e um número definido de células dispensada em cada poço. Placas de ensaio pode ser diretamente fotografada usando o microscópio integrados ou saída do sistema para processamento posterior.
- Instruir o sistema para preparar 4 placas de ensaio por placa omnitray, com um total de 5.000 células por poço.
2. shRNA produção de vírus e revestimento em chapas de ensaio (Tempo necessário: 6 dias)
- Crescer stocks glicerol bacteriana contendo os vetores shRNA (Open Biosystems, TRC1) durante a noite em 2ml de mídia Luria-Bertani meio contendo 100 mg / ml de ampicilina (Sigma-Aldrich).
- Extrato de plasmídeos seguindo o protocolo do fabricante (Promega Assistente MagneSil TFX).
- Produzir vírus usando o RNAi Consórcio de Produção de Alto Throughput Lentivirus (96 placa bem) o protocolo 24. Trabalhando com lentivírus é relativamente seguro, pois as partículas do vírus utilizado para a transdução de replicação são deficientes e split-estratégias de embalagem gene são usados para a sua produção. No entanto, quando se trabalha com lentivírus, procedimentos adicionais de biossegurança são necessárias para minimizar o risco para si mesmo e os outros 25. Todos os experimentos devem ser realizados em um laboratório de nível MLII ou BSL2 segurança. Todos os plásticos (pipetas, pratos de plástico, mídia) que tenham estado em contacto com partículas de lentivírus devem ser incubadas com água sanitária por 24 horas antes da eliminação.
- Calcular a multiplicidade de infecção (MOI) do lentivírus por determinação da percentagem de células GFP positivas usando o GFP pLKO.1 plasmídeo (Sigma-Aldrich).
- Lentivírus placa em placas de ensaio, com uma MOI de 3.
3. Transdução lentiviral e diferenciação neuronal de células SH-SY5Y (Tempo necessário: 6 dias)
- Células são adicionados às placas de ensaio (ver passo 1.7). Carregar placas de ensaio contendo lentivirus shRNA no sistema automatizado de cultura de células.
- Após 24 horas, os meios de comunicação sobre oplacas de ensaio será alterado para Opti-MEM contendo 0,5% de SFB e 0,1 mM de ácido retinóico para iniciar o processo de diferenciação. Diferenciação de células SH-SY5Y permite a visualização de estruturas neuríticas e sincroniza a divisão celular.
- Continue a incubação das placas de ensaio na mídia diferenciação por 5 dias. Isso garante knockdown máxima da expressão do gene alvo.
- No dia 5, adicionar 50 mM H 2 O 2 a metade das placas de ensaio por 24 horas para estimular a translocação de DJ1 para as mitocôndrias.
- No dia 6, adicione CmxROS Mitotracker (Invitrogen) para as células, a uma concentração final de 200 nM por poço e incubar a 37 ° C por 30 minutos.
- Pode-se instruir o sistema para placas de imagem diretamente usando o gerador de imagens HC ou placas podem ser exportados a partir do sistema para processamento posterior.
4. Imunocoloração automatizada de placas de ensaio (Tempo necessário: 2 dias)
Qualidade da imagem é paramount para a realização de uma HCS sensível e confiável. Danos à monocamada celular devido à pipetagem imprecisa pode levar à má qualidade de imagem e os resultados irreproduzíveis. A fim de minimizar os danos camada de células, a imunocoloração foi realizado usando uma estação robótica. O procedimento é similar ao que foi descrito anteriormente 26, mas foi customizado para aumentar o rendimento e reduzir o uso de consumíveis.
- Fix células com 100 l de paraformaldeído 4% pré-aquecido a 37 ° C. Incubar por 20 minutos em temperatura ambiente.
- Lavar as células com 200 mL de PBS por 5 minutos, 3 vezes.
- Incubar as placas de ensaio com 200 mL de PBS contendo Triton 0,1% (PBST) por 10 minutos.
- Lavar as células com 200 mL de PBS por 5 minutos, 3 vezes.
- Incubar as placas de ensaio com 200 mL de buffer do bloco (PBST com 5% de FBS) por 1 hora em temperatura ambiente.
- Lavar as células com 200 mL de PBS por 5 minutos, 3 vezes.
- Incube com o exerdevido anticorpos primários durante a noite a 4 ° C:
- Cabra DJ1 N20 (Santa Cruz, 5 mg / ml)
- Tubulina coelho β-III (Sigma-Aldrich, 1 mg / ml)
- No dia seguinte, lave as células com 200 mL de PBS por 5 minutos, 3 vezes.
- Incubar as placas de ensaio com os seguintes anticorpos secundários 1 hora em temperatura ambiente:
- AlexaFluor burro anti-cabra 488 (Invitrogen, 2 mg / ml)
- AlexaFluor cabra 647 anti-coelho (Invitrogen, 2 mg / ml
- Lavar as células com 200 mL de PBS por 5 minutos, 3 vezes.
- Células incube com Hoechst (Invitrogen, 1 mg / ml) por 10 minutos.
- Lavar as células com 200 mL PBS por 5 minutos, 3 vezes.
- Placas armazenar a 4 ° C até que possam ser trabalhada.
5. Alto teor de análise de imagem e de aquisição de imagem (Tempo requerido: 5 dias)
- Imagem um total de 30 campos por bem usar a lente objetiva de 20x. Visualize DJ1 com o FITC conjunto de filtros, a mitocôndria com o conjunto TRITC filtro, β-tubulina III com o conjunto Cy5 filtro e os núcleos usando o conjunto de filtro UV (Figura 3).
- Analisar as imagens usando o Bioapplication Análise compartimental (Cellomics, ThermoFisher) para determinar a intensidade média do sinal Mitotracker dentro da mitocôndria. (Figura 4B, F).
- Para determinar o coeficiente médio de sobreposição entre DJ1 e as mitocôndrias, analisar as imagens usando o bioapplication Colocalisation Cellomics (Cellomics, ThermoFisher). Definir regiões de interesse (ROI) da seguinte forma: ROI A - núcleo (Figura 4A, E), ROI B - mitocôndria (Figura 4 B, F). A partir de excluir ROI ROI B para garantir a análise de apenas o citoplasma. Definir as mitocôndrias como região alvo I e DJ1 como alvo região II (Figura 4C, G).
- Analisar as imagens usando o bioapplication Profiling Neuronal (Cellomics, Thermofisher) para rastrear os comprimentos médios das neurites da coloração tubulina β-III (Figura4D, H).
- Placas de imagem usando o Opera LX automático confocal leitor (Perkin-Elmer). Imagem um total de 30 campos por bem usar a lente objetiva 60x com imersão em água. Visualize mitocôndrias com o laser 561 nM e núcleos com excitação UV.
- Analisar as imagens usando o ponto-Edge-Ridge (SER) algoritmo características de textura. O filtro SER-Ridge transmite intensidade em pixels formando cume-como testes padrões. Quanto mais fragmentada a mitocôndria, quanto maior a pontuação do SER-Ridge (Figura 8).
6. Normalização de dados e análise
- Importar os dados do software de análise de imagem no pacote BioConductor CellHTS2 para o ambiente de software R (R versão 2.11.1, versão BioConductor 2.6).
- Logaritmo base (2) transformar os dados antes de por a placa normalização mediana base 27, 28. Não aplicar o ajuste variância por placa.
- Para identificar modificadores de um fenótipo, use uma ANOVA de duas vias entre o diferetratamento nt grupos ou seja, as células infectadas sem tratamento Scrambled vs mexidos células toxina tratada versus células-alvo gene não tratada versus células-alvo gene tratados (Figuras 5-7).
7. Resultados representativos
Mutações dentro DJ1 dar origem ao início-início recessive parkinsonismo 21, mas não está claro como a perda de DJ1 dá origem ao fenótipo da doença. Sabe-se que as células deficiente de DJ1 são mais suscetíveis à morte celular induzida pelo estresse oxidativo e na resposta ao estresse oxidativo, DJ1 transloca do citoplasma para a mitocôndria 29, 30. Através da construção de ensaios HC para monitorar esses fenótipos, podemos identificar genes que regulam fenótipos ou afetar associados DJ1. Essa abordagem pode ajudar a decifrar os caminhos em que DJ1 funções e que poderiam estar envolvidos na patogênese da doença.
Exemplo de uma interação com epistasia DJ1 (Figura 5): Knockdown de DJ1 em células expostasaos resultados da toxina em uma maior perda de viabilidade celular (BAR-B: image-B) em comparação com as células infectadas com lentivirus mexidos (BAR-A: image-A). Knockdown do gene-alvo A tem um efeito similar ao observado em células com um knockdown DJ1 (BAR-C: image-C). Knockdown de ambos DJ1 e gene-alvo A resulta em uma perda significativamente maior de viabilidade celular do que a perda de qualquer gene sozinho (BAR-D: image-D). Isso sugere uma interação entre epistáticos DJ1 e alvo gene A.
Exemplo de um gene regulador DJ1 translocação (Figura 6): Quando as células são expostas a uma toxina, DJ1 transloca do citoplasma para a mitocôndria, que é quantificada por um coeficiente maior sobreposição entre DJ1 e as mitocôndrias (BAR-A: Uma imagem em relação BAR-C: image C). Nas células onde B gene-alvo tem sido silenciada, menos DJ1 transloca para as mitocôndrias, quando as células são expostas à toxina. Isto sugere que B gene-alvo está envolvida no transporte de DJ1 para as mitocôndrias. (BAR-B: imagem B e BAR-D: D imagem)
Exemplo de um gene envolvido na conseqüência neuronal (Figura 7): Knockdown do gene C-alvo no tipo selvagem resultados SH-SY5Y células em um aumento significativo no comprimento neurite (BAR-B: image-B) em comparação com as células infectadas com lentivirus expressar scrambled shRNA (BAR-A: Uma imagem). Este efeito é perdido em células incubadas com a toxina (BAR-C e D).
Exemplo de um gene envolvido na morfologia mitocondrial (Figura 8): Infecção do tipo selvagem SH-SY5Y células com shRNA visando resultados gene D em uma diminuição no valor segmentação mitocondrial SER-Ridge (Figura 8, Imagem C e D) quando comparado com células infectadas com lentivirus mexidos (Figura 8, Image A e B).
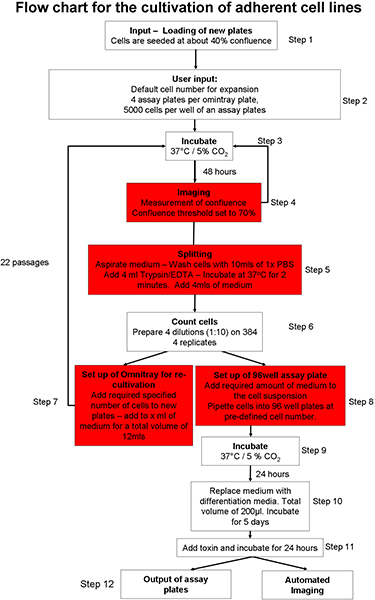
Figura 1 Esboço de protocolo automatizado de cultura de células.:
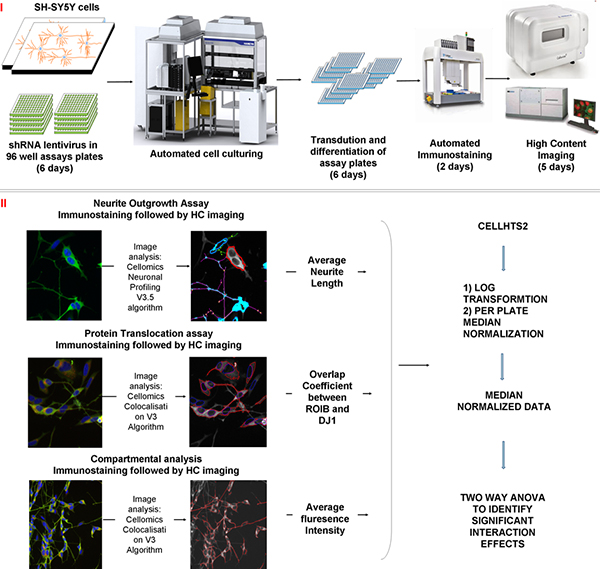
Figura 2 Visão esquemática do processo de seleção, análise de imagem e métodos estatísticos utilizados durante o processo de triagem:. I) As células são cultivadas até que sejam confluentes e, posteriormente, banhado nas placas de ensaio contendo o shRNA lentivírus. As células são diferenciadas por 5 dias e toxina é então adicionado as placas por 24 horas. Placas de ensaio estão de saída do sistema e histoquímica. A quantidade de tempo necessário para cada um dos processos está indicado entre parênteses. ii) Os dados são adquiridos utilizando um gerador de imagens HC (a viabilidade celular, a translocação de proteínas e crescimento neuronal) e um sensor de imagem confocal automatizado (morfologia mitocondrial). Os dados são exportados para o pacote dentro CellHTS2 R, base (2) log transformados e normalizados. Two-way ANOVA é usado para identificar interações significativas entre as diferentes variáveis.

Figura 3. Composite imagens de células adquiridas pela HC imagem. A) As células não tratadas, B) As células tratadas com H 2 O 2. DJ1 é rotulada de verde, as mitocôndrias em vermelho e os núcleos em azul. Neuritos coloração não é destacada.
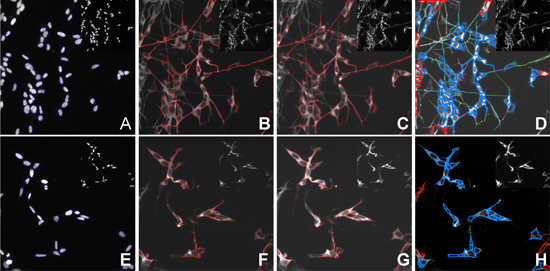
Figura 4. Quantificação de várias características celulares obtidos de uma HCS. AD) não tratada SH-SY5Y células. EH) H 2 O 2 tratados SH-SY5Y células. A, E segmentação Núcleos) e definição de ROI A, B, F) Identificação e quantificação das mitocôndrias, ROI B, C, G) Identificação de DJ1, Target canal II; D, G) Identificação e cálculo de comprimento neurite média. Células perto da borda da imagem são excluídos da análise. Inset imagens são imagens antes da análise.

Figura 5. Identificação de um gene em epistasia com DJ1 (letras nas barras correspondem às letras na imagem). Imagens de A a D são SH-SY5Y células marcadas com Mitotracker CMXRos (vermelho) que foi usado para quantificação de células de saúde.
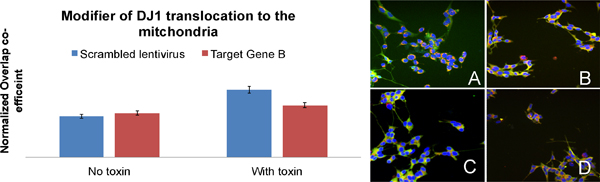
Figura 6. Identificação de um gene regulador DJ1 translocação (letras nas barras correspondem às letras na imagem). Imagens de A a D são SH-SY5Y células marcadas para DJ1 (verde), mitocôndrias (vermelho) e núcleos (azul) que foram utilizados para quantificação de DJ1 translocação para a mitocôndria.
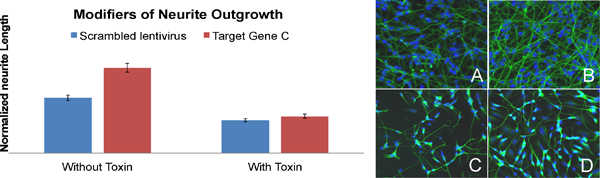
Figura 7. Identificação de um gene envolvido na conseqüência neuronal (letras nas barras correspondem às letras na imagem). Imagens de A a D são SH-SY5Y células marcadas para a β-tubulina III (verde) e núcleos (azul) que foram utilizadas para a quantificação de comprimento neurite.
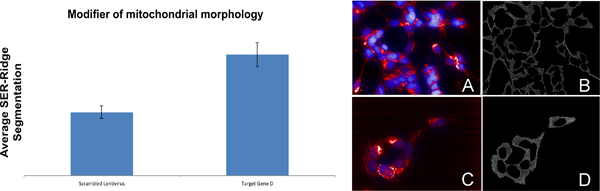
Figura 8. Identificação de um gene envolvido na regulação da morfologia mitocondrial. Uma imagem e C são imagens compostas de SH-SY5Y células infectadas com shRNA mexidos ou um shRNA alvo gene D, respectivamente. Mitocôndrias são coloridas em vermelho, enquanto os núcleos são coloridos em azul. Imagem B e D são visualizações da quantificação SER-Ridge.
Discussão
Com os custos decrescentes de HT / HC sistemas de rastreamento de celular, combinado com a disponibilidade de poderosos genome-wide ferramentas para modificar a função do gene, HT / HC telas estão se tornando comuns na academia. A abordagem já foi aplicado com sucesso em diversas áreas de pesquisa, como a identificação de alvos terapêuticos no câncer 9, 31-33 e 34-36 desenvolvimento embrionário e ainda tem potencial para aplicação em decifrar os caminhos envolvidos em distúrbios neuropsiquiátricos 37,38. No entanto a implementação de tal sistema exige um investimento significativo de tempo e esforço com otimização de processos, muitas vezes tendo um mínimo de 6 meses. Todas as etapas, tais como tripsinização vezes, velocidades e densidades de semeadura pipetagem tem que ser ajustado, para garantir que as células são saudáveis e crescem de forma consistente. Prevenção da contaminação bacteriana é um dos desafios mais difíceis enfrentados cultura de células automatizado semanalmente com os protocolos de limpeza em combination com a constante lavagem de todas as linhas de rolamento líquido com etanol 70% é necessário para a cultura livre de contaminação. Será igualmente necessário melhorar a robótica de forma que instrumentos adicionais, como microscópios confocal de alta resolução e -80 ° C freezers para armazenamento de composto pode ser integrado.
Há também limitações que precisam ser tratadas para melhorar a velocidade, sensível e utilidade deste método para estudar redes de gene e identificar genes envolvidos em vias patogênicas molecular.
Para conduzir uma tela HT / HC e assegurar que dados confiáveis são coletados, vários aspectos precisam ser otimizados. Primeiro, a confiabilidade da medição é primordial e é dependente da robustez e da sensibilidade do ensaio. Por exemplo, os ensaios acima descritos são adequados para telas menores, mas são difíceis de implementar em uma escala genoma de largura, devido ao número de passos de processamento necessária antes da aquisição da imagem.Assim, seria necessário construir linhas celulares estáveis expressando o gene repórter, o que permitiria a imagem direta e levar a variação diminuiu devido ao número reduzido de etapas de processamento. No presente, projetando um ensaio que retrata com precisão e confiabilidade quantifica um fenótipo de interesse é o principal entrave para o processo de triagem HC.
Muitas telas são realizados em células de mamíferos utilizando diferentes bibliotecas RNAi, que sofrem de efeitos fora do alvo, eficiência limitada silenciamento de genes do genoma e cobertura incompleta. Thus bibliotecas precisam ser feitas, que são mais específicos, potentes e têm uma melhor cobertura. Estão em curso esforços para criar tal espera-se tais empreendimentos irão melhorar a reprodutibilidade de HT / HC scre en hits.
A limitação de muitas telas de grande escala à base de células é que eles são realizados em células de neuroblastoma, porque eles podem ser manipulados geneticamente e cultivadas para grandes números com relativa facilidade. No entanto, a relevância dos 'hits' identificadas na ex vivo modelos de cultura de células para função in vivo é questionável, especialmente porque o cérebro é composto de tipos de células altamente especializadas que formam uma rede densa e complicada de conexões sinápticas funcionar como uma unidade altamente integrado. Como conseqüência, é comum que atinge identificados utilizando a abordagem de triagem descrito acima, são validados em telas secundárias usando técnicas adicionais e mais fisiológico modelos relevantes 39. Para melhorar a tradução de visitas identificadas durante HCS, os modelos mais representativo e sofisticadas, tais como pilhas e células-tronco diferenciadas ou co-cultura sistemas precisam ser desenvolvidos e adaptados para HT / HC abordagens.
> ntent "Com uma combinação de cultura de células automatizado e imagem HC pode rapidamente obter novos insights sobre como os neurônios de função e determinar quais as vias são importantes para o desenvolvimento da doença. Combinando HCS / HTS dados com informações geradas a partir de outras abordagens a genómica, que vai então, ser possível construir uma visão de biologia de sistemas de doenças cerebrais, facilitando assim o desenvolvimento terapêutico.Divulgações
Não temos nada a divulgar
Agradecimentos
Agradecemos aos programadores Hamilton e especialistas para o apoio contínuo e Blaas Eva para assistência técnica. Este trabalho foi apoiado por duas bolsas de Investimento NWO (911-07-031 e 40-00506-98-10011), The Prinses Beatrix Fonds Wetenschapsprijs 2009 eo Neuroscience Campus Amsterdam; SJ é suportado por Ti-Pharma: T5-207.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número Catálogo | |
| AI.CELLHOST | HAMILTON | http://www.hamiltonrobotics.com/en-uk/applications/cellomics/ | |
| OPTI-MEM | INVITROGEN | 31985-054 | |
| Ácido Retinóico | SIGMA-ALDRICH | R2625 | |
| PLACAS OMNITRAY | NUNC | 465219 | |
| 96 BEM placas de cultura | GRENIER | 655086 | |
| DJ1 ANTICORPO N20 | SANTA CRUZ | SC27004 | |
| BETA-III ANTICORPO tubulina | SIGMA-ALDRICH | T3952 | |
| CMXROS MITOTRACKER | INVITROGEN | M-7512 | |
| HOECHST-33342 | INVITROGEN | H1399 | |
| PERÓXIDO DE HIDROGÊNIO | SIGMA-ALDRICH | 216763-100ML | |
| TRIPSINA | INVITROGEN | 25050014 | |
| FOSFATO Dulbecco Tampão Salina | INVITROGEN | 14190086 | |
| PROMEGA WIZARD MAGNESIL TFX | PROMEGA | A2380 | |
| Plasmideo CLONES | Open Biosystems | http://www.openbiosystems.com / RNAi / shRNALibraries / TRCLibraryDetails / | |
| BIOAPPLICATIONS CELLOMICS | THERMO FISHER- | http://www.thermo.com/hcs |
Referências
- Geschwind, D. H., Konopka, G. Neuroscience in the era of functional genomics and systems biology. Nature. 461, 908-915 (2009).
- Ge, H., Walhout, A. J., Vidal, M. Integrating 'omic' information: a bridge between genomics and systems biology. Trends. Genet. 19, 551-560 (2003).
- Zhu, H., Snyder, M. 'Omic' approaches for unraveling signaling networks. Curr. Opin. Cell. Biol. 14, 173-179 (2002).
- Jain, S., Heutink, P. From single genes to gene networks: high-throughput-high-content screening for neurological disease. Neuron. 68, 207-217 (2010).
- Manolio, T. A. Finding the missing heritability of complex diseases. Nature. 461, 747-753 (2009).
- Nalls, M. A. Imputation of sequence variants for identification of genetic risks for Parkinson's disease: a meta-analysis of genome-wide association studies. Lancet. 377, 641-649 (2011).
- Simon-Sanchez, J. Genome-wide association study confirms extant PD risk loci among the Dutch. Eur. J. Hum. Genet. 19 (6), 655-661 (2011).
- Van Regenmortel, M. H. Reductionism and complexity in molecular biology. Scientists now have the tools to unravel biological and overcome the limitations of reductionism. EMBO. Rep. 5, 1016-1020 (2004).
- Aherne, G. W., McDonald, E., Workman, P. Finding the needle in the haystack: why high-throughput screening is good for your health. Breast. Cancer. Res. 4, 148-154 (2002).
- An, W. F., Tolliday, N. J. Introduction: cell-based assays for high-throughput screening. Methods. Mol. Biol. 486, 1-12 (2009).
- Mayr, L. M., Bojanic, D. Novel trends in high-throughput screening. Curr. Opin. Pharmacol. 9, 580-588 (2009).
- Hertzberg, R. P., Pope, A. J. High-throughput screening: new technology for the 21st century. Curr. Opin. Chem. Biol. 4, 445-451 (2000).
- Conrad, C., Gerlich, D. W. Automated microscopy for high-content RNAi screening. J. Cell. Biol. 188, 453-461 (2010).
- Thomas, N. High-content screening: a decade of evolution. J. Biomol. Screen. 15, 1-9 (2010).
- Arrasate, M., Finkbeiner, S. Automated microscope system for determining factors that predict neuronal fate. Proc. Natl. Acad. Sci. U. S. A. 102, 3840-3845 (2005).
- Dragunow, M. High-content analysis in neuroscience. Nat. Rev. Neurosci. 9, 779-788 (2008).
- Varma, H., Lo, D. C., Stockwell, B. R. High throughput screening for neurodegeneration and complex disease phenotypes. Comb. Chem. High. Throughput. Screen. 11, 238-248 (2008).
- Durr, O. Robust hit identification by quality assurance and multivariate data analysis of a high-content, cell-based assay. J. Biomol. Screen. 12, 1042-1049 (2007).
- Miller, J. Quantitative relationships between huntingtin levels, polyglutamine length, inclusion body formation, and neuronal death provide novel insight into huntington's disease molecular pathogenesis. J. Neurosci. 30, 10541-10550 (2010).
- Jain, S., Sondervan, D., Rizzu, P., Bochdanovits, Z., Caminada, D., Heutink, P. The Complete Automation of Cell Culture. Journal of Biomolecular Screening. 16 (8), (2011).
- Bonifati, V. Mutations in the DJ-1 gene associated with autosomal recessive early-onset parkinsonism. Science. 299, 256-259 (2003).
- Ricardo, R., Phelan, K. Trypsinizing and Subculturing Mammalian Cells. J. Vis. Exp. (16), e755-e755 (2008).
- Kent, L. Culture and Maintenance of Human Embryonic Stem Cells. J. Vis. Exp. (34), e1427-e1427 (2009).
- Moffat, J. A lentiviral RNAi library for human and mouse genes applied to an arrayed viral high-content screen. Cell. 124, 1283-1298 (2006).
- Volksgezondheid, M. V. Integrale versie van de Regeling genetisch gemodificeerde organismen en het Besluit genetische gemodificeerde. Organismen. , (2004).
- Anderl, J. L., Redpath, S., Ball, A. J. A Neuronal and Astrocyte Co-Culture Assay for High Content Analysis of Neurotoxicity. J. Vis. Exp. (27), e1173-e1173 (2009).
- Wiles, A. M., Ravi, D., Bhavani, S., Bishop, A. J. An analysis of normalization methods for Drosophila RNAi genomic screens and development of a robust validation scheme. J. Biomol. Screen. 13, 777-784 (2008).
- Canet-Aviles, R. M. The Parkinson's disease protein DJ-1 is neuroprotective due to cysteine-sulfinic acid-driven mitochondrial localization. Proc. Natl. Acad. Sci. U. S. A. 101, 9103-9108 (2004).
- Blackinton, J. Formation of a stabilized cysteine sulfinic acid is critical for the mitochondrial function of the parkinsonism protein DJ-1. J. Biol. Chem. 284, 6476-6485 (2009).
- Gupta, P. B. Identification of selective inhibitors of cancer stem cells by high-throughput screening. Cell. 138, 645-659 (2009).
- Luo, J. A genome-wide RNAi screen identifies multiple synthetic lethal interactions with the Ras oncogene. Cell. 137, 835-848 (2009).
- Mouchet, E. H., Simpson, P. B. High-content assays in oncology drug discovery: opportunities and challenges. IDrugs. 11, 422-427 (2008).
- Vogt, A. Automated image-based phenotypic analysis in zebrafish embryos. Dev. Dyn. 238, 656-663 (2009).
- Pardo-Martin, C. High-throughput in vivo vertebrate screening. Nat. Methods. 7, 634-636 (2010).
- Vogt, A., Codore, H., Day, B. W., Hukriede, N. A., Tsang, M. Development of automated imaging and analysis for zebrafish chemical screens. J. Vis. Exp. (40), e1900-e1900 (2010).
- Ross, P. J., Ellis, J. Modeling complex neuropsychiatric disease with induced pluripotent stem cells. F1000. Biol. Rep. 2, 84-84 (2010).
- Ebert, A. D., Svendsen, C. N. Human stem cells and drug screening: opportunities and challenges. Nat. Rev. Drug. Discov. 9, 367-372 (2010).
- An, W. F., Tolliday, N. Cell-based assays for high-throughput screening. Mol. Biotechnol. 45, 180-186 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados