Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Utilisation de la pHluorine pour évaluer la dynamique des récepteurs de guidage axonaux dans la culture cellulaire et dans l’embryon de poussin
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous décrivons ici l’utilisation d’une variante fluorescente verte pH-sensible de protéine, pHluorin, pour étudier la dynamique spatio-temporelle des récepteurs de guidage axon en trafic à la surface cellulaire. Le récepteur marqué par la pHluorine est exprimé à la fois en culture cellulaire et in vivo, enutilisant l’électroporation de l’embryon de poussin.
Résumé
Au cours du développement, les récepteurs de guidage axonaux jouent un rôle crucial dans la régulation de la sensibilité des axones aux signaux attrayants et répulsifs. En effet, l’activation des récepteurs de guidage est la première étape des mécanismes de signalisation permettant aux pointes axoniques, les cônes de croissance, de répondre aux ligands. En tant que tel, la modulation de leur disponibilité à la surface de la cellule est l’un des mécanismes qui participent à la définition de la sensibilité du cône de croissance. Nous décrivons ici une méthode pour visualiser avec précision la dynamique de surface spatio-temporelle de cellules d’un récepteur de guidage axone in vitro et in vivo dans la moelle épinière de poussin en développement. Nous avons tiré parti de la propriété de fluorescence pH-dépendante d’une variante verte de protéine fluorescente (GFP) pour détecter spécifiquement la fraction du récepteur de guidage axone qui est adressée à la membrane plasmique. Nous décrivons d’abord la validation in vitro de telles constructions pH-dépendantes et nous détaillons plus loin leur utilisation in vivo,dans la moelle épinière de poussin, pour évaluer la dynamique spatio-temporelle du récepteur axon de guidage d’intérêt.
Introduction
Lors de leur navigation, les axones intègrent de multiples indices environnementaux qui les guident vers leur cible. Ces signaux activent les récepteurs de guidage à la surface des bornes axoniques, les cônes de croissance, qui à leur tour initient une voie de signalisation appropriée. Ainsi, la régulation temporelle et spatiale de la distribution de surface cellulaire des récepteurs est essentielle pour définir la sensibilité du cône de croissance1. Dans ce contexte, le croisement de la ligne médiane par les axones commissuraux est un excellent modèle pour étudier la régulation des niveaux de surface des cellules réceptrices. Dans la moelle épinière en développement, les axones commissuraux sont initialement attirés vers la plaque ventrale du plancher où ils traversent la ligne médiane. Après la traversée, ils perdent leur réactivité aux attractifs de la plaque de plancher et gagnent en réponse aux répulsifs de plaque de plancher afin qu’ils puissent sortir de la plaque de plancher et naviguer vers leur destination finale dans le côté controlatéral du système nerveux2,3. La régulation de la disponibilité des récepteurs à la surface du cône de croissance est l’un des mécanismes sous-jacents au changement de réactivité aux signaux médians4,5. Ainsi, la surveillance sélective des récepteurs présents à la membrane plasmique des cônes de croissance est d’une importance primordiale. Nous décrivons ici une méthode basée sur la propriété de fluorescence pH-dépendante d’une variante verte de protéine fluorescente (GFP) pour visualiser spécifiquement les récepteurs de guidage axonaux qui sont adressés à la membrane plasmique in vitro et in vivo,dans la moelle épinière de poussin en développement.
Rothman et ses collègues ont conçu par des mutations ponctuelles des variantes sensibles au pH de la GFP, y compris le pHluorin écliptique6. Le pHluorin écliptique a la propriété d’être non fluorescent lorsqu’il est exposé à un pH acide (<6), tout en étant fluorescent à pH neutre. Cela permet de distinguer les récepteurs non fluorescents localisés dans les compartiments acides intracellulaires(c’est-à-dire endosomes, vésicules de trafic) des récepteurs fluorescents incorporés à la membrane plasmique et donc exposés au pH neutre extracellulaire7. Nous en avons profité pour surveiller la localisation de la membrane plasmique de la plexineA1, un récepteur de guidage axone médiant la réponse du cône de croissance à la sémaphorine répulsifique médiane 3B5 (figure 1A). Nous décrivons ici la caractérisation in vitro d’une construction pHluorin-plexinA1, avec l’électroporation8-10 in ovo de cette construction dans la moelle épinière de poussin se développante suivie de l’analyse microscopique des cryosections qui permettent de suivre la dynamique de récepteur de guidage d’axone in vivo avec des résolutions spatiales et temporelles.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Stratégie de clonage pour marquer le récepteur PlexinA1 avec pHluorin
- Choisissez un vecteur d’expression approprié comme épine dorsale(par exemple, le vecteur exprimant le récepteur de la souris plexineA1, un don gentil du Dr Andreas Puschel11).
Remarque: Ce vecteur plexinA1 a été conçu pour réaliser l’insertion efficace de récepteur HA- ou VSV-marqué dans la membrane plasmique. - Amplifier par PCR la séquence codante de la pHluorine écliptique en utilisant le plasmide adéquat comme modèle(par exemple, le récepteur GABA A marqué par la pHluorine, un don du Dr Jacob2). Si nécessaire, ajoutez un site de restriction à l’extrémité 5' de l’amorce afin de faciliter l’étape de clonage dans l’épine dorsale principale.
- Insérez la séquence pHluorine dans le cadre entre le peptide signal et la séquence codante du récepteur à l’aide de sites de restriction(par exemple les sites de restriction NruI/Kpn2I comme décrit à la figure 1B).
Remarque: Parce que le peptide signal qui assure le ciblage correct des récepteurs est clivé, le pHluorin doit être placé après elle. Ceci justifie la reconnaissance du peptide signal et empêche le clivage du pHluorin du récepteur d’intérêt. - Séquencez les constructions obtenues pour s’assurer qu’aucune mutation n’a été introduite par PCR.
2. Caractérisation in vitro du récepteur marqué par la pHluorine dans les cellules COS7
La capacité de la protéine de fusion à atteindre la membrane plasmique et sa perte réversible de fluorescence à mesure que le pH est abaissé peuvent être confirmées à l’aide de la procédure suivante.
- Jour 1. Plaquez 1,5 x 105 cellules COS7 dans un plat de 35 mm à fond de verre dans 2 ml de milieu Eagle modifié complet de Dulbecco (DMEM - 10 % sérum fœtal bovin - 1 mM de pyruvate de sodium - 25 U/ml pénicilline/stretomycine - 2,5 μg/ml d’amphotéricine B - pH 7,4).
- Jour 2. Cellules transfectées :
Remarque: Les cellules doivent être confluentes à 70-80%.- Préparer 200 μl de NaCl 150 mM et ajouter 3 μg d’ADN, c’est-à-dire le vecteur codant pour le récepteur marqué par la pHluorine. Vortex doucement et tournez brièvement vers le bas.
Remarque : Une carte du vecteur pHluorin-plexinA1 utilisé dans ces expériences est illustrée à la figure 1B. - Ajouter 10 μl de réactif de transfection (ou la quantité appropriée du réactif utilisé). Vortex immédiatement.
- Incuber 10 min à TA.
- Ajouter 200 μl du mélange réactif de transfection/ADN aux cellules.
- Déplacez doucement la plaque pour obtenir le repartition du mélange et replacez les cellules dans l’incubateur à 37 °C.
- Préparer 200 μl de NaCl 150 mM et ajouter 3 μg d’ADN, c’est-à-dire le vecteur codant pour le récepteur marqué par la pHluorine. Vortex doucement et tournez brièvement vers le bas.
- Jour 3. Retirez le milieu de transfection et remplacez-le par 2 ml de DMEM complet frais.
- Jour 4. Effectuer l’imagerie cellulaire en direct des cellules transfectées COS7 pHluorin-plexineA1:
- Préparer deux aliquotes de milieu complet DMEM et ajuster le pH à 3,5 et à 9,5, respectivement.
Remarque: Pour une plaque de 35 mm, 1,5 ml de chaque solution est nécessaire pour effectuer l’expérience. - Retirez le milieu cellulaire et remplacez-le par 1 ml de DMEM complet milieu pH 7,4.
- Préparez une seringue de 5 ml avec un type approprié de tube afin d’injecter divers composants directement dans le milieu de culture cellulaire sans ouvrir la chambre du microscope.
- Utilisez un module qui permet le maintien d’une atmosphère de travail humide à 37 °C, 5 % deCO2.
Remarque : Une autre approche de l’utilisation d’une chambre à CO2 consiste à utiliser un milieu tamponné hepes (généralement de l’ordre de 10 à 25 mM selon le type de cellule). - Placez les cellules dans la chambre et ajustez le tube et la seringue.
Remarque : Le microscope doit être équilibré avant de commencer à éviter la dérive mécanique pendant l’enregistrement. - Ouvrez le logiciel d’imagerie et sélectionnez le programme d’acquisition multidimensionnel.
- Trouvez des cellules COS7 transfectées avec l’objectif 40X et marquez la position dans le logiciel pour chacune d’elles.
- Configurez la pile Z afin d’avoir une acquisition de profondeur de 15 μm (la mise au point peut changer lors de l’ajout de média dans la plaque).
- Définissez l’exposition pour le filtre GFP et la phase.
- Configurez le calendrier d’acquisition.
Note: Pour l’ensemble de l’expérience avec 5 champs d’intérêt, l’acquisition toutes les 20 secondes pendant 10 min devrait être suffisante. - Commencez l’acquisition et prenez 5 images de contrôle dans un milieu DMEM pH 7,4.
- Suspendre l’acquisition, injecter 1,25 ml de PH 3,5 DMEM complet pour atteindre un pH de 5,5 dans le milieu de culture, marquer l’événement dans le logiciel et reprendre l’acquisition pour 5 points de temps supplémentaires.
Remarque: La fluorescence verte devrait progressivement disparaître. - Suspendre l’acquisition, injecter 1,2 ml de PH 9,5 DMEM complet pour atteindre un pH 7,4 dans le milieu de culture, marquer l’événement dans le logiciel et reprendre l’acquisition pour 5 points de temps supplémentaires.
Remarque: La fluorescence verte devrait réapparaître au niveau de la membrane plasmique. - Analyser les images.
La figure 2 montre des images représentatives obtenues avec un tel protocole avec la construction pHluorin-plexinA1.
- Préparer deux aliquotes de milieu complet DMEM et ajuster le pH à 3,5 et à 9,5, respectivement.
3. In ovo Electroporation de pHluorin-plexinA1 Construire
- Manipulation des oeufs avant électroporation:
- Conservez les œufs fécondés au réfrigérateur à 14 °C jusqu’à une semaine avant l’incubation.
- Incuber les œufs à 38,5 °C (101,3 °F) dans un incubateur avec une humidité saturée pendant 50-52 heures jusqu’à ce que les embryons atteignent le stade HH1412.
Remarque: Les œufs doivent être placés horizontalement pendant l’incubation afin que l’embryon soit correctement positionné pour l’électroporation, flottant sur le dessus du jaune. Le stade HH14 convient pour obtenir l’expression des plasmides dans les neurones différenciés de la moelle épinière et dans le ganglion de la racine dorsale avec un taux de survie approprié.
- Embryons électroporates8-10:
- Préparer l’électroporation:
- Préparer des plasmides d’ADN sans endotoxine avec une concentration supérieure à 2 μg/μl, pour pouvoir le diluer comme concentration désirée.
- Tirez suffisamment de capillaires en verre pour injecter les différentes solutions d’ADN.
- Préparer du PBS stérile(-Ca2+;-Mg2+) - 100 U/ml de pénicilline/streptomycine et équilibrer à 38,5 °C.
- Stérilisez le capot, les ciseaux incurvés et les pinces fines.
- Contrôlez l’espacement des électrodes.
Remarque: Un espace de 4 mm entre les électrodes est généralement utilisé.
- Fenêtre de l’œuf13 (figure 3A):
- Utilisez des ciseaux incurvés pour percer la coquille sur le côté émoussé de l’œuf.
- Retirer 2 ml d’albumine à l’aide d’une aiguille de 0,9 mm x 25 mm et d’une seringue de 5 ml. Orientez l’aiguille verticalement afin d’éviter d’endommager le sac vitellin.
- Couvrez le dessus de l’œuf avec du ruban adhésif pour maintenir l’intégrité de la coquille.
- À l’aide de ciseaux incurvés, percez la coquille au milieu du ruban adhésif pour égaliser la pression lors du retrait de 2 ml d’albumine de l’œuf. Ensuite, coupez une fenêtre assez grande pour visualiser l’embryon et pouvoir travailler dessus.
- Ajouter ~2 ml de PBS chaud stérile(-Ca2+;-Mg2+)- 100 U/ml de pénicilline/streptomycine pour éviter la déshydratation de l’embryon et le rendre plus accessible au manipulateur.
- Injecter de l’ADN et électroporater l’embryon
- Diluer le plasmide dans le PBS(-Ca2+;-Mg2+) à une concentration comprise entre 0,5 et 2 μg/μl et ajouter le colorant vert rapide pour atteindre une concentration finale de 0,025 %. Chargez le mélange d’ADN dans un capillaire. L’utilisation d’un injecteur est recommandée.
Remarque : Vérifiez que la résistance capillaire n’est ni trop grande (ce qui signifie qu’il pourrait y avoir des difficultés lors de l’injection d’embryons) ni trop petite (ce qui signifie que le capillaire pourrait être trop gros et pourrait endommager l’embryon). De plus, une concentration d’acides nucléiques supérieure à 2 μg/μl peut causer des effets non spécifiques et doit être contrôlée. - Percer le sac vitellin et le tube neural du côté caudal avec le capillaire chargé. Entrez dans le tube neural avec un angle peu profond et remplissez la lumière de la queue à la tête avec le mélange d’ADN (Figure 3B).
Remarque: Le vert rapide permet de contrôler la précision de l’injection. - Placez rapidement les électrodes de platine de 4 mm de chaque côté du tube neural au niveau que vous souhaitez électroporater et appliquez 3 impulsions à 31 V pendant 50 ms avec des intervalles de 500 ms(Figure 3C).
Remarque : Évitez de placer les électrodes sur le cœur ou sur de gros vaisseaux embryonnaires supplémentaires pour éviter d’endommager l’embryon en développement. Des bulles devraient se former sur les électrodes. - Avec l’aiguille, retirez 2 ml d’albumen pour diminuer le niveau dans la coquille.
- Scellez la fenêtre et le côté émoussé hermétiquement avec du ruban adhésif.
- Remettez les œufs à 38,5 °C dans l’incubateur jusqu’à ce qu’ils atteignent le stade souhaité.
- Diluer le plasmide dans le PBS(-Ca2+;-Mg2+) à une concentration comprise entre 0,5 et 2 μg/μl et ajouter le colorant vert rapide pour atteindre une concentration finale de 0,025 %. Chargez le mélange d’ADN dans un capillaire. L’utilisation d’un injecteur est recommandée.
- Préparer l’électroporation:
4. Incorporation et cryosection d’embryons
- 48 heures après l’électroporation, prélever soigneusement des embryons électroporés (stade HH24). Couper le ruban adhésif et la moitié de la membrane chorioallantoïde. Pour empêcher les embryons de s’enfoncer dans le jaune, placez une passoire sous l’embryon et coupez la seconde moitié de la membrane chorioallantoïde.
- Transférer l’embryon dans une parabole de dissection remplie de PBS glacé.
- Vérifiez l’efficacité de l’électroporation en recherchant la fluorescence dans le tube neural avec un stéréomicroscope à fluorescence.
Note : La coélectroporation d’un plasmide témoin codant pour la RFP peut aider à visualiser la zone électroporée. - Disséquer les embryons à l’aide d’un microscalpel afin de sélectionner la zone électroporée de la moelle épinière.
- Transférer les embryons disséqués sur une plaque de 24 puits et fixer dans un pH de 7,4 4 % de paraformaldéhyde (PFA) - tampon phosphate salin (PBS), O/N à 4 °C.
Remarque: L’étape de fixation est cruciale pour permettre la stabilisation de la pHluorine dans sa conformation « vivante » et ainsi pouvoir utiliser la pHluorine dans les tissus fixes / perméabilisés. Bien que la fixation ralentisse considérablement le changement pH-dépendant de la fluorescence, il faut prendre en compte que les changements conformationnels/protonationnels peuvent encore se produire après fixation. Ainsi, le protocole suivant (incorporation, cryosections et observation) doit être réalisé dans les 3 jours suivant l’étape de fixation, avec tous les tampons à pH 7. Si la fixation n’est pas nécessaire, il est recommandé d’effectuer une observation sur les sections de tissus vivants. - Enlever 4 % de PFA et laver les embryons à un pH de 7,4 PBS.
- Incuber les embryons dans du saccharose PBS-15 % et les conserver à 4 °C jusqu’à ce que les embryons coulent.
- Incuber des embryons fixes dans un pH de 7,4 gélatine à 7,5 % et 15 % de saccharose pendant 45 min à 37 °C afin que les embryons soient complètement incorporés.
- Placez des moules d’encastrement sur de la glace et ajoutez 400 μl de pH 7,4 gélatine à 7,5% et 15% de saccharose pour obtenir une base solide de 2 mm.
- Aspirer l’embryon incorporé avec une pointe coupée et placer l’embryon sur la base de gélatine solide.
- Couvrir avec un pH de 7,4 gélatine à 7,5% et 15% de saccharose et positionner l’embryon avec une pince avant que la gélatine ne se solidifie.
- Une fois que la gélatine est solide, préparez un bain d’isopropanol à -40 °C (utilisez de la glace carbonique ou de l’azote liquide) et congelez le bloc de gélatine pendant 5 min.
- Gardez les blocs congelés à -80 °C.
- Placer le bloc congelé à -20 °C pendant 1 h.
- Retirez le moule et fixez le bloc sur un mandrin avec un milieu de polyéthylène glycol.
- Une fois que le bloc est bien fixé, placez le mandrin dans le système cryostat.
Remarque : Utilisez des lames enrobées pour éviter la perte de tissu pendant la coloration. - Effectuer des cryosections en série (des cryosections de 20 μm sont habituellement effectuées).
- Laisser sécher les cryosections pendant 15 min à TA.
Remarque: des cryosections devraient être protégées contre l’exposition inutile à la lumière afin d’éviter le blanchiment de la fluorescence de GFP.
5. Analyse microscopique des cryosections
- Réhydratez les cryosections dans le pH 7,4 PBS à droite pendant 10 minutes.
Remarque: Si nécessaire, les noyaux peuvent être colorés avec Hoechst. - Utiliser une solution de Hoechst à 0,5 μg/ml dans du PBS et incuber des cryosections pendant 15 min.
- Rincer les lames 3x avec un pH 7,4 PBS pendant 5 min.
- Passez au montage par glissement. Une solution de montage d’alcool polyvinylique à pH 7,4 (ou plus basique) qui durcit O/N peut être utilisée : positionner soigneusement la lame de couverture pour éviter la formation de bulles d’air entre la lame et la lame de couverture.
- Laisser le milieu de montage durcir O/N à 4 °C dans l’obscurité.
- Utilisez un microscope confocal inversé pour visualiser avec précision la protéine de fusion pHluorin-plexineA1 : effectuez une z-stack à la résolution optimale du trou d’épingle et optique et utilisez des lentilles 20X (NA 0,75) ou 40X (NA 1,3).
Remarque : La pHluorine est détectable avec les mêmes paramètres que ceux utilisés pour détecter la GFP(c.-à-d. le pic d’émission à 509 nm). Les paramètres d’excitation et de filtre de détection de longueur d’onde sont définis de manière optimale par le logiciel d’imagerie. Hoechst est détecté entre 425-460 nm (l’excitation est à 405 nm), GFP ou pHluorin est détecté entre 485-545 nm (excitation est à 473 nm) et RFP est détecté entre 575-675 nm (excitation est à 559 nm).
Des images représentatives de l’expression de la pHluorine-plexineA1 et de l’eGFP dans la moelle épinière de l’embryon de poussin sont illustrées à la figure 4.
Access restricted. Please log in or start a trial to view this content.
Résultats
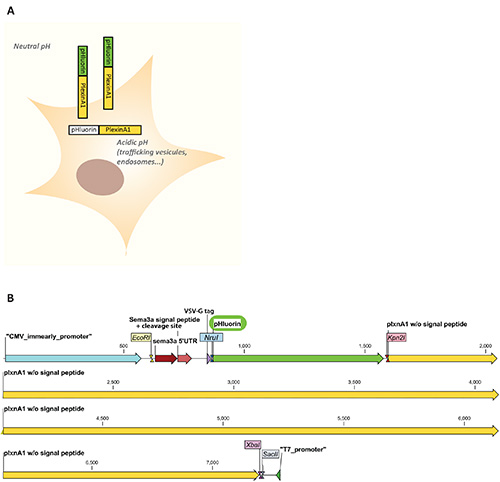
Figure 1. A. Schéma des propriétés de fluorescence de la pHluorine-plexineA1 dans un contexte cellulaire. La pHluorine est non fluorescente dans les compartiments intracellulaires où le pH est acide (<6) comme dans les vésicules en trafic ou dans les endosomes et est fluorescente lorsqu’elle est expos...
Access restricted. Please log in or start a trial to view this content.
Discussion
Ce protocole fournit une procédure étape par étape pour suivre la dynamique d’un récepteur de guidage axone à la fois dans la culture cellulaire et dans le contexte développemental de la moelle épinière de l’embryon de poussin.
Pour concevoir une protéine marquée par la pHluorine de novo, deux points doivent être pris en compte concernant la stratégie de clonage. Tout d’abord, la balise de pHluorin devrait être exposée à la lumière des endosomes acides, et par co...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Homaira Nawabi, Frederic Moret et Isabelle Sanyas pour leur aide. Ces travaux sont soutenus par le CNRS, Association Française contre les Myopathies (AFM), ANR YADDLE, Labex DevWeCan, Labex Cortex, ERC YODA à V.C. ; C.D-B et A.J sont soutenus respectivement par les bourses De la Ligue contre le cancer et du Labex DevWeCan.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| COS7 cells | ATCC | CRL-1651 | |
| DMEM GlutaMAX | GIBCO | 61965-026 | |
| Sodium pyruvate | GIBCO | 11360-039 | |
| Amphotericin B | Sigma | A2942 | |
| Fetal bovine serum | GIBCO | 10270-106 | |
| Penicillin/Streptomycin | GIBCO | 15140-122 | |
| Exgen500 reagent | Euromedex Fermentas | ET0250 | |
| PBS -Ca2+ -Mg2+ | GIBCO | 14190-094 | |
| Fast green dye | Sigma | F7252 | |
| 32% Paraformaldehyde aqueous solution | Electron Microscopy | 15714-S | Dilute extemporaneously in PBS to achieve a 4% solution |
| Gelatin from cold water fish skin | Sigma | G7041 | |
| Sucrose | Sigma | S0389 | |
| Cryomount | Histolab | 00890 | |
| Hoechst 34580 | Invitrogen | H21486 | |
| Mowiol 4-88 | Fluka | 81381 | |
| Consumables | |||
| Bottom-glass 35 mm dish | MatTek | P35G-1.5-14-C | |
| 5 ml Syringe | Terumo | SS-05S | |
| Needles 0.9 mm x 25 mm | Terumo | NN-2025R | |
| Capillaries | CML | PP230PO | capillaries are stretched manually in the flame |
| Superfrost Plus Slides | Thermo Scientific | 4951PLUS | |
| Material | |||
| Curved scissors | FST | 129-10 | |
| Microscalpel | FST | 10316-14 | |
| Forceps | FST | Dumont #5 REF#11254 | |
| Equipment/software | |||
| Time lapse microscope | Zeiss | Observer 1 | |
| Temp module S | PECON for Zeiss | ||
| CO2 module S | PECON for Zeiss | ||
| Metamorph software | Metamorph | ||
| Eggs incubator | Sanyo | MIR154 | |
| Electroporator apparatus | Nepa Gene CO., LTD | CUY21 | |
| Electrodes | Nepa Gene CO., LTD | CUY611P7-4 | 4 mm platinum electrodes |
| Fluorescence stereomicroscope | LEICA | MZ10F | |
| Cryostat | MICROM | HM550 | |
| Confocal microscope | Olympus | FV1000, X81 | |
| Fluoview software | Olympus | ||
| CLC Main Workbench software | CLC Bio |
Références
- Winckler, B., Mellman, I. Trafficking guidance receptors. Cold Spring Harb. Perspect. Biol. 2, (2010).
- Jacob, T. C., et al. J. Neurosci. 25, 10469-10478 (2005).
- Nawabi, H., Castellani, V. Axonal commissures in the central nervous system: how to cross the midline. Cell Mol. Life Sci. 68, 2539-2553 (2011).
- Keleman, K., Ribeiro, C., Dickson, B. J. Comm function in commissural axon guidance: cell-autonomous sorting of Robo in vivo. Nat. Neurosci. 8, 156-163 (2005).
- Nawabi, H., et al. A midline switch of receptor processing regulates commissural axon guidance in vertebrates. Genes Dev. 24, 396-410 (2010).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394, 192-195 (1998).
- Miesenbock, G. Synapto-pHluorins: genetically encoded reporters of synaptic transmission. Cold Spring Harb. Protoc.. 2012, 213-217 (2012).
- Avraham, O., Zisman, S., Hadas, Y., Vald, L., Klar, A. Deciphering axonal pathways of genetically defined groups of neurons in the chick neural tube utilizing in ovo electroporation. J. Vis. Exp. (39), 1792-17 (2010).
- Blank, M. C., Chizhikov, V., Millen, K. J. In ovo electroporations of HH stage 10 chicken embryos. J. Vis. Exp. (9), (2007).
- Wilson, N. H., Stoeckli, E. T. In ovo electroporation of miRNA-based plasmids in the developing neural tube and assessment of phenotypes by DiI injection in open-book preparations. J. Vis. Exp. (68), (2012).
- Rohm, B., Ottemeyer, A., Lohrum, M., Puschel, A. W. Plexin/neuropilin complexes mediate repulsion by the axonal guidance signal semaphorin 3A. Mech. Dev. 93, 95-104 (2000).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. 1951. Dev. Dyn.. 195, 231-272 (1992).
- Korn, M. J., Cramer, K. S. Windowing chicken eggs for developmental studies. J. Vis. Exp. (8), (2007).
- Alberts, P., et al. Cdc42 and actin control polarized expression of TI-VAMP vesicles to neuronal growth cones and their fusion with the plasma membrane. Mol. Biol. Cell. 17, 1194-1203 (2006).
- Perret, E., Lakkaraju, A., Deborde, S., Schreiner, R., Rodriguez-Boulan, E. Evolving endosomes: how many varieties and why. Curr. Opin. Cell Biol. 17, 423-434 (2005).
- Li, Y., et al. Imaging pHluorin-tagged receptor insertion to the plasma membrane in primary cultured mouse neurons. J. Vis. Exp. (69), (2012).
- Tojima, T., Itofusa, R., Kamiguchi, H. Asymmetric clathrin-mediated endocytosis drives repulsive growth cone guidance. Neuron. 66, 370-377 (2010).
- Matsui, A., Yoshida, A. C., Kubota, M., Ogawa, M., Shimogori, T. Mouse in utero electroporation: controlled spatiotemporal gene transfection. J. Vis. Exp. (54), 3024-30 (2011).
- Falk, J., et al. Electroporation of cDNA/Morpholinos to targeted areas of embryonic CNS in Xenopus. BMC Dev. Biol. 7 (107), (2007).
- Holzhausen, L. C., Lewis, A. A., Cheong, K. K., Brockerhoff, S. E. Differential role for synaptojanin 1 in rod and cone photoreceptors. J. Comp. Neurol. 517, 633-644 (2009).
- Shang, Y., Claridge-Chang, A., Sjulson, L., Pypaert, M., Miesenbock, G. Excitatory local circuits and their implications for olfactory processing in the fly antennal lobe. Cell. 128, 601-612 (2007).
- Dittman, J. S., Kaplan, J. M. Factors regulating the abundance and localization of synaptobrevin in the plasma membrane. Proc. Natl. Acad. Sci. U.S.A. 103, 11399-11404 (2006).
- Bozza, T., McGann, J. P., Mombaerts, P., Wachowiak, M. In vivo imaging of neuronal activity by targeted expression of a genetically encoded probe in the mouse. Neuron. 42, 9-21 (2004).
- Sankaranarayanan, S., Ryan, T. A. Real-time measurements of vesicle-SNARE recycling in synapses of the central nervous system. Nat. Cell. Biol. 2, 197-204 (2000).
- Stark, D. A., Kasemeier-Kulesa, J. C., Kulesa, P. M. Photoactivation cell labeling for cell tracing in avian development. CSH Protoc.. 2008, (2008).
- Hildick, K. L., Gonzalez-Gonzalez, I. M., Jaskolski, F., Henley, J. M. Lateral diffusion and exocytosis of membrane proteins in cultured neurons assessed using fluorescence recovery and fluorescence-loss photobleaching. J. Vis. Exp. (60), (2012).
- Hanson, G. T., et al. Green fluorescent protein variants as ratiometric dual emission pH sensors. 1. Structural characterization and preliminary application. Biochemistry. 41, 15477-15488 (2002).
- Rose, T., Schoenenberger, P., Jezek, K., Oertner, T. G. Developmental refinement of vesicle cycling at schaffer collateral synapses. Neuron. 77, 1109-1121 (2013).
- Li, Y., Tsien, R. W. pHTomato, a red, genetically encoded indicator that enables multiplex interrogation of synaptic activity. Nat. Neurosci. 15, 1047-1053 (2012).
- de Wit, J., Toonen, R. F., Verhage, M. Matrix-dependent local retention of secretory vesicle cargo in cortical neurons. J. Neurosci. 29, 23-37 (2009).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon