È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Uso di pHluorin per valutare la dinamica dei recettori di guida axon nella coltura cellulare e nell'embrione del pulcino
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Descriviamo qui l'uso di una variante di proteina fluorescente verde sensibile al pH, la pHluorina, per studiare la dinamica spazio-temporale dei recettori di guida dell'assone che trafficano sulla superficie cellulare. Il recettore taggato con pHluorina è espresso sia in coltura cellulare che in vivo, utilizzandol'elettroporazione dell'embrione del pulcino.
Abstract
Durante lo sviluppo, i recettori di guida dell'assone svolgono un ruolo cruciale nella regolazione della sensibilità degli assoni a segnali attraenti e ripugnanti. Infatti, l'attivazione dei recettori di guida è il primo passo dei meccanismi di segnalazione che consentono alle punte dell'assone, ai coni di crescita, di rispondere ai ligandi. Come tale, la modulazione della loro disponibilità sulla superficie cellulare è uno dei meccanismi che partecipano all'impostazione della sensibilità del cono di crescita. Descriviamo qui un metodo per visualizzare con precisione la dinamica superficiale della cellula spazio-temporale di un recettore di guida dell'assone sia in vitro che in vivo nel midollo spinale del pulcino in via di sviluppo. Abbiamo approfittato della proprietà di fluorescenza dipendente dal pH di una variante di proteina fluorescente verde (GFP) per rilevare specificamente la frazione del recettore di guida dell'assone che è indirizzata alla membrana plasmatica. Descriviamo prima la convalida in vitro di tali costrutti dipendenti dal pH e ne dettagliamo ulteriormente l'uso in vivo,nella corda spinale del pulcino, per valutare la dinamica spazio-temporale del recettore della guida dell'assone di interesse.
Introduzione
Durante la loro navigazione, gli assoni integrano più segnali ambientali che li guidano verso il loro obiettivo. Questi segnali attivano recettori di guida sulla superficie dei terminali di axon, i coni di crescita, che a loro volta avviano un'appropriata via di segnalazione. Pertanto, la regolazione temporale e spaziale della distribuzione della superficie cellulare dei recettori è fondamentale per impostare la sensibilità del cono di crescita1. In questo contesto, l'attraversamento della linea mediana da parte degli assoni commissurali è un modello eccellente per indagare la regolazione dei livelli superficiali delle cellule recettoriali. Nel midollo spinale in via di sviluppo, gli assoni commissurali sono inizialmente attratti verso la piastra del pavimento ventrale dove attraversano la linea mediana. Dopo l'attraversamento, perdono la loro reattività agli attrattori della piastra del pavimento e ottengono risposta ai repellenti per piastre del pavimento in modo che possano uscire dalla piastra del pavimento e navigare verso la loro destinazione finale nel lato contralaterale del sistemanervoso 2,3. La regolazione della disponibilità del recettore sulla superficie del cono di crescita è uno dei meccanismi alla base del passaggio di reattività ai segnali della lineamediana 4,5. Pertanto, il monitoraggio selettivo dei recettori presenti nella membrana plasmatica dei coni di crescita è di primaria importanza. Descriviamo qui un metodo basato sulla proprietà di fluorescenza dipendente dal pH di una variante di proteina fluorescente verde (GFP) per visualizzare specificamente i recettori di guida dell'assone che sono indirizzati alla membrana plasmatica in vitro e in vivo,nel midollo spinale del pulcino in via di sviluppo.
Rothman e colleghi progettati da mutazioni puntili varianti sensibili al pH della GFP tra cui la pHluorina eclittica6. La pHluorina eclittica ha la proprietà di essere non fluorescente se esposta al pH acido (<6), pur essendo fluorescente a pH neutro. Ciò consente di distinguere i recettori non fluorescenti localizzati nei compartimenti acidiintracellulari (cioè endosomi, vescicole di traffico) dai recettori fluorescenti incorporati nella membrana plasmatica e quindi esposti al pH neutro extracellulare7. Ne abbiamo approfittato per monitorare la localizzazione della membrana plasmatica della plexinA1, un recettore di guida dell'assone che media la risposta del cono di crescita alla semaphorin repellente della linea mediana 3B5 (Figura 1A). Descriviamo qui la caratterizzazione in vitro di un costrutto pHluorin-plexinA1, insieme all'elettroporazione ovo 8-10 di questo costrutto nel midollo spinale del pulcino in via di sviluppo seguito dall'analisi microscopica delle criosezioni che consentono di seguire la dinamica del recettore della guida dell'assone in vivo con risoluzioni spaziali e temporali.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Strategia di clonazione per taggare il recettore PlexinA1 con pHluorin
- Scegli un vettore di espressione appropriato come spinadorsale (ad esempio il vettore di espressione plexinA1 del recettore del topo, un dono gentile del Dr. Andreas Puschel11).
Nota: Questo vettore plexinA1 è stato progettato per ottenere un efficiente inserimento del recettore con tag HA o VSV nella membrana plasmatica. - Amplificare da PCR la sequenza di codifica della pHluorina eclittica utilizzando il plasmide adeguato come modello(ad esempio il recettore GABA A con tag pHluorin, un dono gentile del Dr. Jacob2). Se necessario, aggiungere un sito di restrizione all'estremità 5 ' del primer per facilitare la fase di clonazione nella spina dorsale.
- Inserire la sequenza di pHluorina nel fotogramma tra il peptide del segnale e la sequenza di codifica delrecettore utilizzando siti di restrizione ( ad esempio siti di restrizione NruI/Kpn2I come descritto nella figura 1B).
Nota: Poiché il peptide del segnale che garantisce il corretto targeting dei recettori è scisto, la pHluorina deve essere posizionata dopo di essa. Ciò garantisce il riconoscimento del peptide del segnale e impedisce la scissione della pHluorina dal recettore di interesse. - Sequenziare i costrutti ottenuti per garantire che nessuna mutazione sia stata introdotta dalla PCR.
2. Caratterizzazione del recettore taggato con pHluorina in vitro nelle cellule COS7
La capacità della proteina di fusione di raggiungere la membrana plasmatica e la sua reversibile perdita di fluorescenza man mano che il pH viene abbassato possono essere confermate utilizzando la seguente procedura.
- Giorno 1. Piastra 1,5 x 105 cellule COS7 in un piatto con fondo di vetro da 35 mm in 2 ml di mezzo aquila modificato di Dulbecco completo (DMEM - siero bovino fetale al 10% - 1 mM di piruvato di sodio - 25 U/ml penicillina/stretomicina - 2,5 μg/ml Amphotericina B - pH 7,4).
- Giorno 2. Celle transfette:
Nota: le cellule devono essere confluenti al 70-80%.- Preparare 200 μl NaCl 150 mM e aggiungere 3 μg di DNA, cioè il recettore vettoriale codificante con tag pHluorina. Delicatamente vortice e girare brevemente verso il basso.
Nota: Una mappa del vettore pHluorin-plexinA1 utilizzato in questi esperimenti è mostrata nella figura 1B. - Aggiungere 10 μl di reagente di trasfezione (o la quantità appropriata del reagente utilizzato). Vortice immediatamente.
- Incubare 10 minuti a RT.
- Aggiungere 200 μl del reagente di trasfezione/miscela DNA alle cellule.
- Spostare delicatamente la piastra per ottenere la ripartizione della miscela e posizionare le cellule nell'incubatore a 37 °C.
- Preparare 200 μl NaCl 150 mM e aggiungere 3 μg di DNA, cioè il recettore vettoriale codificante con tag pHluorina. Delicatamente vortice e girare brevemente verso il basso.
- Giorno 3. Rimuovere il mezzo di trasfezione e sostituirlo con 2 ml di DMEM fresco completo.
- Giorno 4. Eseguire immagini di cellule vive di cellule trasfetate COS7 pHluorin-plexinA1:
- Preparare due aliquote di dmem mezzo completo e regolare il pH rispettivamente a 3,5 e a 9,5.
Nota: Per una piastra da 35 mm, sono necessari 1,5 ml di ogni soluzione per eseguire l'esperimento. - Rimuovere il supporto cellulare e sostituirlo con 1 ml di DMEM pH medio completo 7.4.
- Preparare una siringa da 5 ml con un tipo appropriato di tubi per iniettare vari componenti direttamente nel mezzo di coltura cellulare senza aprire la camera del microscopio.
- Utilizzare un modulo che consenta la manutenzione di un'atmosfera di lavoro umida di 37 °C, 5% CO2.
Nota: Un approccio alternativo all'uso di una camera di CO2 è quello di utilizzare un mezzo tamponato con HEPES (di solito nell'intervallo di 10-25 mM a seconda del tipo di cella). - Posizionare le cellule nella camera e regolare il tubo e la siringa.
Nota: Il microscopio deve essere equilibrato prima di iniziare a evitare deriva meccanica durante la registrazione. - Aprire il software di imaging e selezionare il programma di acquisizione multidimensionale.
- Trova celle COS7 trasfette con l'obiettivo 40X e segna la posizione nel software per ognuna di esse.
- Configurare la pila Z in modo da avere un'acquisizione di profondità di 15 μm (la messa a fuoco può cambiare quando si aggiungono supporti nella piastra).
- Impostare l'esposizione per il filtro GFP e la fase.
- Configurare i tempi di acquisizione.
Nota: Per l'intero esperimento con 5 campi di interesse, l'acquisizione ogni 20 secondi per 10 minuti dovrebbe essere sufficiente. - Inizia l'acquisizione e scatta 5 immagini di controllo in dmem pH 7.4 medium.
- Sospendere l'acquisizione, iniettare 1,25 ml di pH 3.5 dmem completo per ottenere un pH di 5,5 nel mezzo di coltura, contrassegnare l'evento nel software e riprendere l'acquisizione per altri 5 punti di tempo.
Nota: la fluorescenza verde dovrebbe scomparire progressivamente. - Sospendere l'acquisizione, iniettare 1,2 ml di pH 9,5 dmem completo per ottenere un pH 7.4 nel mezzo di coltura, contrassegnare l'evento nel software e riprendere l'acquisizione per altri 5 punti di tempo.
Nota: La fluorescenza verde dovrebbe riapparire sulla membrana plasmatica. - Analizza le immagini.
La figura 2 mostra le immagini rappresentative ottenute con tale protocollo con il costrutto pHluorin-plexinA1.
- Preparare due aliquote di dmem mezzo completo e regolare il pH rispettivamente a 3,5 e a 9,5.
3. In ovo Elettroporazione del costrutto pHluorin-plexinA1
- Manipolazione delle uova prima dell'elettroporazione:
- Conservare le uova fecondate in frigorifero a 14 °C fino a una settimana prima dell'incubazione.
- Incubare le uova a 38,5 °C (101,3 °F) in un'incubatrice con umidità satura per 50-52 ore fino a quando gli embrioni raggiungono lo stadio HH1412.
Nota: Le uova devono essere posizionate orizzontalmente durante l'incubazione in modo che l'embrione sia correttamente posizionato per l'elettroporazione, galleggiando sulla parte superiore del tuorlo. Lo stadio HH14 è adatto per ottenere l'espressione dei plasmidi nei neuroni differenziati nel midollo spinale e nel ganglio della radice dorsale con un adeguato tasso di sopravvivenza.
- Embrioni elettroporati8-10:
- Preparare l'elettroporazione:
- Preparare plasmidi di DNA senza endotossine con una concentrazione superiore a 2 μg/μl, per poterlo diluire come concentrazione desiderata.
- Tirare abbastanza capillari di vetro per iniettare le diverse soluzioni di DNA.
- Preparare PBS sterile (-Ca2+; -Mg2+) - 100 U/ml penicillina/streptomicina ed equilibrare a 38,5 °C.
- Sterilizzare il cappuccio, le forbici curve e le forcep fini.
- Controllare la spaziatura degli elettrodi.
Nota: viene generalmente utilizzato uno spazio di 4 mm tra gli elettrodi.
- Finestral'uovo 13 (Figura 3A):
- Usa forbici curve per perforare il guscio sul lato smussato dell'uovo.
- Rimuovere 2 ml di albume utilizzando un ago da 0,9 mm x 25 mm e una siringa da 5 ml. Orientare l'ago verticalmente per evitare di danneggiare il sacco del tuorlo.
- Coprire la parte superiore dell'uovo con nastro adesivo per mantenere l'integrità del guscio.
- Usando forbici curve, perforare il guscio al centro del nastro per equalizzare la pressione quando si rimuovono 2 ml di albume dall'uovo. Quindi, taglia una finestra abbastanza grande da visualizzare l'embrione ed essere in grado di lavorarti.
- Aggiungere ~2 ml di PBS caldo sterile (-Ca2+; -Mg2+) - 100 U/ml penicillina/streptomicina per evitare la disidratazione dell'embrione e renderlo più accessibile al manipolatore.
- Iniettare DNA ed elettroporare l'embrione
- Diluire il plasmide in PBS (-Ca2+; -Mg2+) ad una concentrazione compresa tra 0,5-2 μg/μl e aggiungere colorante verde veloce per raggiungere una concentrazione finale dello 0,025%. Caricare il mix di DNA in un capillare. Si consiglia l'uso di un iniettore.
Nota: Verificare che la resistenza capillare non sia né troppo grande (il che significa che potrebbero esserci difficoltà durante l'iniezione di embrioni) né troppo piccola (il che significa che il capillare potrebbe essere troppo grande e potrebbe danneggiare l'embrione). Inoltre, la concentrazione di acidi nucleici superiori a 2 μg/μl può causare effetti non specifici e deve essere controllata. - Forare il tuorlo-sacco e il tubo neurale sul lato caudale con il capillare caricato. Entra nel tubo neurale con un angolo poco profondo e riempi il lume dalla coda alla testa con il mix di DNA(Figura 3B).
Nota: Il verde veloce consente di controllare l'accuratezza dell'iniezione. - Posizionare rapidamente gli elettrodi di platino da 4 mm su entrambi i lati del tubo neurale al livello che si desidera elettroporare e applicare 3 impulsi a 31 V per 50 msec con intervalli di 500 msec(Figura 3C).
Nota: Evitare di posizionare gli elettrodi sul cuore o su grandi vasi embrionali extra per evitare di danneggiare l'embrione in via di sviluppo. Le bolle dovrebbero formarsi sugli elettrodi. - Con l'ago, rimuovere 2 ml di albume per diminuire il livello nel guscio.
- Sigillare la finestra e il lato smussato ermeticamente con del nastro adesivo.
- Rimettere le uova a 38,5 °C nell'incubatrice fino a raggiungere lo stadio desiderato.
- Diluire il plasmide in PBS (-Ca2+; -Mg2+) ad una concentrazione compresa tra 0,5-2 μg/μl e aggiungere colorante verde veloce per raggiungere una concentrazione finale dello 0,025%. Caricare il mix di DNA in un capillare. Si consiglia l'uso di un iniettore.
- Preparare l'elettroporazione:
4. Incorporamento e criosezione degli embrioni
- 48 ore dopo l'elettroporazione, raccogliere con cura embrioni elettroporati (stadio HH24). Tagliare il nastro e metà della membrana corioallantoide. Per evitare che gli embrioni sprofondano nel tuorlo, posizionare un colino sotto l'embrione e tagliare la seconda metà della membrana corioallantoide.
- Trasferire l'embrione in un piatto di dissezione riempito con PBS ghiacciato.
- Controlla l'efficienza dell'elettroporazione cercando la fluorescenza nel tubo neurale con un microscopio stereo a fluorescenza.
Nota: La coelettroporazione di un plasmide di controllo che codifica per la RFP può aiutare a visualizzare l'area elettroporata. - Sezionare embrioni utilizzando un microscalpel per selezionare l'area elettroporata del midollo spinale.
- Trasferire embrioni sezionati su una piastra a 24 po', fissarli in pH 7,4 4% Paraformaldeide (PFA) - Phosphate Buffer Saline (PBS), O/N a 4 °C.
Nota: La fase di fissazione è fondamentale per consentire la stabilizzazione della pHluorina nella sua conformazione "viva" e quindi essere in grado di utilizzare la pHluorina nel tessuto fisso / permeabilizzato. Sebbene la fissazione rallenti drasticamente il cambiamento di fluorescenza dipendente dal pH, si deve tenere conto del fatto che i cambiamenti conformazionali / protonazione possono ancora verificarsi dopo la fissazione. Pertanto, il seguente protocollo (incorporamento, criosezioni e osservazione) deve essere eseguito entro 3 giorni dalla fase di fissazione, con tutti i buffer a pH 7. Se la fissazione non è necessaria, si consiglia di eseguire osservazioni su sezioni di tessuto vivo. - Rimuovere il 4% di PFA e lavare gli embrioni in pH 7.4 PBS.
- Incubare gli embrioni in saccarosio PBS-15% e conservarsi a 4 °C fino a quando gli embrioni affondano.
- Incubare embrioni fissi in pH 7,4 7,5% gelatina- 15% saccarosio per 45 min a 37 °C in modo che gli embrioni siano completamente incorporati.
- Posizionare gli stampi di incorporamento sul ghiaccio e aggiungere 400 μl di pH 7,4 7,5% di gelatina- 15% di saccarosio per ottenere una base solida di 2 mm.
- Aspirare l'embrione incorporato con una punta tagliata e posizionare l'embrione sulla base di gelatina solida.
- Coprire con pH 7,4 7,5% gelatina- 15% saccarosio e posizionare l'embrione con pini prima che la gelatina si solidifichi.
- Una volta che la gelatina è solida, preparare un bagno isopropanolo -40 °C (utilizzare ghiaccio secco o azoto liquido) e congelare il blocco di gelatina per 5 minuti.
- Mantenere i blocchi congelati a -80 °C.
- Posizionare il blocco congelato a -20 °C per 1 ora.
- Rimuovere lo stampo e fissare il blocco su un mandrino con un mezzo di polietilene glicole.
- Una volta che il blocco è ben fissato, posizionare il mandrino nel sistema criostato.
Nota: utilizzare diapositive rivestite per evitare la perdita di tessuto durante la colorazione. - Eseguire criosezioni seriali (di solito vengono eseguite criosezioni da 20 μm).
- Lasciare asciugare le criosezioni per 15 minuti a RT.
Nota: le criosezioni devono essere protette da un'esposizione alla luce non necessaria per evitare lo sbiancamento della fluorescenza GFP.
5. Analisi microscopica delle criosezioni
- Criosezioni reidrate in pH 7.4 PBS a RT per 10 min.
Nota: se necessario, i nuclei possono essere macchiati con Hoechst. - Utilizzare una soluzione di Hoechst da 0,5 μg/ml in PBS e incubare criosezioni per 15 minuti.
- Risciacquare le diapositive 3 volte con pH 7.4 PBS per 5 min.
- Procedere al montaggio dello scivolo. È possibile utilizzare una soluzione di montaggio alcoliche polivinilico pH 7.4 (o più basico) che indurisce O/N: posizionare con cura il coverslip per evitare la formazione di bolle d'aria tra lo scivolo e il coverslip.
- Lasciare indurire il mezzo di montaggio O/N a 4 °C al buio.
- Utilizzare un microscopio confocale invertito per visualizzare con precisione la proteina fusion pHluorin-plexinA1: eseguire z-stack al foro di spillo ottimale e risoluzione ottica e utilizzare lenti 20X (NA 0.75) o 40X (NA 1.3).
Nota: la pHluorina è rilevabile con gli stessi parametri utilizzati per rilevare la GFP(cioè picco di emissione a 509 nm). Le impostazioni del filtro di eccitazione e rilevamento della lunghezza d'onda sono definite in modo ottimale dal software di imaging. Hoechst viene rilevato tra 425-460 nm (l'eccitazione è a 405 nm), GFP o pHluorin viene rilevato tra 485-545 nm (l'eccitazione è a 473 nm) e RFP viene rilevato tra 575-675 nm (l'eccitazione è a 559 nm).
Immagini rappresentative dell'espressione pHluorin-plexinA1 ed eGFP nel midollo spinale dell'embrione del pulcino sono mostrate nella figura 4.
Access restricted. Please log in or start a trial to view this content.
Risultati
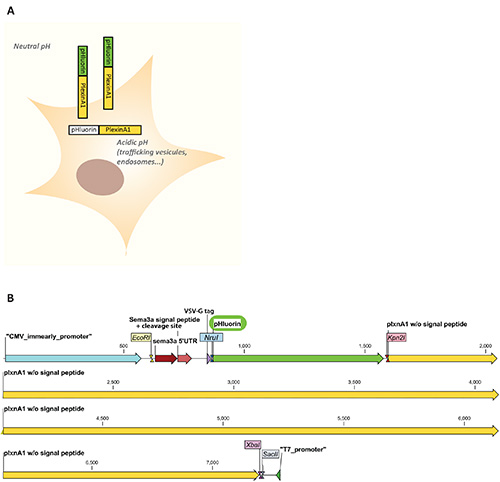
Figura 1. A. Schema delle proprietà di fluorescenza pHluorin-plexinA1 in un contesto cellulare. La pHluorina non è fluorescente nei compartimenti intracellulari dove il pH è acido (<6) come nelle vesciche di traffico o negli endosomi ed è fluorescente quando esposto al mezzo extracellulare dove il pH è ...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo protocollo fornisce una procedura passo-passo per seguire la dinamica di un recettore di guida dell'assone sia nella coltura cellulare che nel contesto dello sviluppo del midollo spinale dell'embrione del pulcino.
Per progettare una proteina etichettata con pHluorina de novo, è necessario considerare due punti per quanto riguarda la strategia di clonazione. In primo luogo, l'etichetta pHluorina deve essere esposta al lume degli endosomi acidi e, di conseguenza, al compartiment...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Homaira Nawabi, Frederic Moret e Isabelle Sanyas per il loro aiuto. Questo lavoro è supportato da CNRS, Association Francaise contre les Myopathies (AFM), ANR YADDLE, Labex DevWeCan, Labex Cortex, ERC YODA to V.C.; C.D-B e A.J sono supportati rispettivamente da borse di studio La Ligue contre le cancer e Labex DevWeCan.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| COS7 cells | ATCC | CRL-1651 | |
| DMEM GlutaMAX | GIBCO | 61965-026 | |
| Sodium pyruvate | GIBCO | 11360-039 | |
| Amphotericin B | Sigma | A2942 | |
| Fetal bovine serum | GIBCO | 10270-106 | |
| Penicillin/Streptomycin | GIBCO | 15140-122 | |
| Exgen500 reagent | Euromedex Fermentas | ET0250 | |
| PBS -Ca2+ -Mg2+ | GIBCO | 14190-094 | |
| Fast green dye | Sigma | F7252 | |
| 32% Paraformaldehyde aqueous solution | Electron Microscopy | 15714-S | Dilute extemporaneously in PBS to achieve a 4% solution |
| Gelatin from cold water fish skin | Sigma | G7041 | |
| Sucrose | Sigma | S0389 | |
| Cryomount | Histolab | 00890 | |
| Hoechst 34580 | Invitrogen | H21486 | |
| Mowiol 4-88 | Fluka | 81381 | |
| Consumables | |||
| Bottom-glass 35 mm dish | MatTek | P35G-1.5-14-C | |
| 5 ml Syringe | Terumo | SS-05S | |
| Needles 0.9 mm x 25 mm | Terumo | NN-2025R | |
| Capillaries | CML | PP230PO | capillaries are stretched manually in the flame |
| Superfrost Plus Slides | Thermo Scientific | 4951PLUS | |
| Material | |||
| Curved scissors | FST | 129-10 | |
| Microscalpel | FST | 10316-14 | |
| Forceps | FST | Dumont #5 REF#11254 | |
| Equipment/software | |||
| Time lapse microscope | Zeiss | Observer 1 | |
| Temp module S | PECON for Zeiss | ||
| CO2 module S | PECON for Zeiss | ||
| Metamorph software | Metamorph | ||
| Eggs incubator | Sanyo | MIR154 | |
| Electroporator apparatus | Nepa Gene CO., LTD | CUY21 | |
| Electrodes | Nepa Gene CO., LTD | CUY611P7-4 | 4 mm platinum electrodes |
| Fluorescence stereomicroscope | LEICA | MZ10F | |
| Cryostat | MICROM | HM550 | |
| Confocal microscope | Olympus | FV1000, X81 | |
| Fluoview software | Olympus | ||
| CLC Main Workbench software | CLC Bio |
Riferimenti
- Winckler, B., Mellman, I. Trafficking guidance receptors. Cold Spring Harb. Perspect. Biol. 2, (2010).
- Jacob, T. C., et al. J. Neurosci. 25, 10469-10478 (2005).
- Nawabi, H., Castellani, V. Axonal commissures in the central nervous system: how to cross the midline. Cell Mol. Life Sci. 68, 2539-2553 (2011).
- Keleman, K., Ribeiro, C., Dickson, B. J. Comm function in commissural axon guidance: cell-autonomous sorting of Robo in vivo. Nat. Neurosci. 8, 156-163 (2005).
- Nawabi, H., et al. A midline switch of receptor processing regulates commissural axon guidance in vertebrates. Genes Dev. 24, 396-410 (2010).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394, 192-195 (1998).
- Miesenbock, G. Synapto-pHluorins: genetically encoded reporters of synaptic transmission. Cold Spring Harb. Protoc.. 2012, 213-217 (2012).
- Avraham, O., Zisman, S., Hadas, Y., Vald, L., Klar, A. Deciphering axonal pathways of genetically defined groups of neurons in the chick neural tube utilizing in ovo electroporation. J. Vis. Exp. (39), 1792-17 (2010).
- Blank, M. C., Chizhikov, V., Millen, K. J. In ovo electroporations of HH stage 10 chicken embryos. J. Vis. Exp. (9), (2007).
- Wilson, N. H., Stoeckli, E. T. In ovo electroporation of miRNA-based plasmids in the developing neural tube and assessment of phenotypes by DiI injection in open-book preparations. J. Vis. Exp. (68), (2012).
- Rohm, B., Ottemeyer, A., Lohrum, M., Puschel, A. W. Plexin/neuropilin complexes mediate repulsion by the axonal guidance signal semaphorin 3A. Mech. Dev. 93, 95-104 (2000).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. 1951. Dev. Dyn.. 195, 231-272 (1992).
- Korn, M. J., Cramer, K. S. Windowing chicken eggs for developmental studies. J. Vis. Exp. (8), (2007).
- Alberts, P., et al. Cdc42 and actin control polarized expression of TI-VAMP vesicles to neuronal growth cones and their fusion with the plasma membrane. Mol. Biol. Cell. 17, 1194-1203 (2006).
- Perret, E., Lakkaraju, A., Deborde, S., Schreiner, R., Rodriguez-Boulan, E. Evolving endosomes: how many varieties and why. Curr. Opin. Cell Biol. 17, 423-434 (2005).
- Li, Y., et al. Imaging pHluorin-tagged receptor insertion to the plasma membrane in primary cultured mouse neurons. J. Vis. Exp. (69), (2012).
- Tojima, T., Itofusa, R., Kamiguchi, H. Asymmetric clathrin-mediated endocytosis drives repulsive growth cone guidance. Neuron. 66, 370-377 (2010).
- Matsui, A., Yoshida, A. C., Kubota, M., Ogawa, M., Shimogori, T. Mouse in utero electroporation: controlled spatiotemporal gene transfection. J. Vis. Exp. (54), 3024-30 (2011).
- Falk, J., et al. Electroporation of cDNA/Morpholinos to targeted areas of embryonic CNS in Xenopus. BMC Dev. Biol. 7 (107), (2007).
- Holzhausen, L. C., Lewis, A. A., Cheong, K. K., Brockerhoff, S. E. Differential role for synaptojanin 1 in rod and cone photoreceptors. J. Comp. Neurol. 517, 633-644 (2009).
- Shang, Y., Claridge-Chang, A., Sjulson, L., Pypaert, M., Miesenbock, G. Excitatory local circuits and their implications for olfactory processing in the fly antennal lobe. Cell. 128, 601-612 (2007).
- Dittman, J. S., Kaplan, J. M. Factors regulating the abundance and localization of synaptobrevin in the plasma membrane. Proc. Natl. Acad. Sci. U.S.A. 103, 11399-11404 (2006).
- Bozza, T., McGann, J. P., Mombaerts, P., Wachowiak, M. In vivo imaging of neuronal activity by targeted expression of a genetically encoded probe in the mouse. Neuron. 42, 9-21 (2004).
- Sankaranarayanan, S., Ryan, T. A. Real-time measurements of vesicle-SNARE recycling in synapses of the central nervous system. Nat. Cell. Biol. 2, 197-204 (2000).
- Stark, D. A., Kasemeier-Kulesa, J. C., Kulesa, P. M. Photoactivation cell labeling for cell tracing in avian development. CSH Protoc.. 2008, (2008).
- Hildick, K. L., Gonzalez-Gonzalez, I. M., Jaskolski, F., Henley, J. M. Lateral diffusion and exocytosis of membrane proteins in cultured neurons assessed using fluorescence recovery and fluorescence-loss photobleaching. J. Vis. Exp. (60), (2012).
- Hanson, G. T., et al. Green fluorescent protein variants as ratiometric dual emission pH sensors. 1. Structural characterization and preliminary application. Biochemistry. 41, 15477-15488 (2002).
- Rose, T., Schoenenberger, P., Jezek, K., Oertner, T. G. Developmental refinement of vesicle cycling at schaffer collateral synapses. Neuron. 77, 1109-1121 (2013).
- Li, Y., Tsien, R. W. pHTomato, a red, genetically encoded indicator that enables multiplex interrogation of synaptic activity. Nat. Neurosci. 15, 1047-1053 (2012).
- de Wit, J., Toonen, R. F., Verhage, M. Matrix-dependent local retention of secretory vesicle cargo in cortical neurons. J. Neurosci. 29, 23-37 (2009).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon