Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Génération de cellules T humaines allo-spécifiques du sang périphérique
Dans cet article
Résumé
This article describes a method for the generation and propagation of human T cell clones that specifically respond to a defined alloantigen. This protocol can be adapted for cloning human T cells specific for a variety of peptide-MHC ligands.
Résumé
The study of human T lymphocyte biology often involves examination of responses to activating ligands. T cells recognize and respond to processed peptide antigens presented by MHC (human ortholog HLA) molecules through the T cell receptor (TCR) in a highly sensitive and specific manner. While the primary function of T cells is to mediate protective immune responses to foreign antigens presented by self-MHC, T cells respond robustly to antigenic differences in allogeneic tissues. T cell responses to alloantigens can be described as either direct or indirect alloreactivity. In alloreactivity, the T cell responds through highly specific recognition of both the presented peptide and the MHC molecule. The robust oligoclonal response of T cells to allogeneic stimulation reflects the large number of potentially stimulatory alloantigens present in allogeneic tissues. While the breadth of alloreactive T cell responses is an important factor in initiating and mediating the pathology associated with biologically-relevant alloreactive responses such as graft versus host disease and allograft rejection, it can preclude analysis of T cell responses to allogeneic ligands. To this end, this protocol describes a method for generating alloreactive T cells from naive human peripheral blood leukocytes (PBL) that respond to known peptide-MHC (pMHC) alloantigens. The protocol applies pMHC multimer labeling, magnetic bead enrichment and flow cytometry to single cell in vitro culture methods for the generation of alloantigen-specific T cell clones. This enables studies of the biochemistry and function of T cells responding to allogeneic stimulation.
Introduction
Les lymphocytes T sont des composantes essentielles du système immunitaire adaptatif. Les cellules T sont responsables de la médiation directement non seulement des réponses immunitaires protectrices à des agents pathogènes à travers une variété de mécanismes effecteurs, mais également le maintien activement auto-tolérance immunologique et diriger les réponses des autres cellules dans le système immunitaire. Ces fonctions sont dirigés à travers un certain nombre de signaux intégrés, y compris des récepteurs de cellule T (TCR) ligature, les cytokines et les chimiokines, et les métabolites 1. Parmi ces signaux, le TCR est d'une importance particulière, car il fournit la spécificité caractéristique qui définit le rôle de la cellule T dans l'immunité adaptative. Un TCR interagit avec les antigènes peptidiques linéaires présentés par le CMH (HLA de orthologue humain) molécules (complexes de pMHC) d'une manière hautement spécifique et sensible pour fournir les signaux qui déclenchent la fonction T effectrice des cellules. Les paramètres biochimiques des interactions TCR avec des ligands PMHC fournissent non seulement la spécificité de Tl'activation des cellules, mais aussi avoir un impact qualitatif sur la fonction des cellules T ultérieur 2. Ainsi, l'étude de la fonction des cellules T nécessite souvent l'examen des réponses de lymphocytes T clonaux ayant une spécificité antigénique déterminée.
Le compartiment des cellules T humaines, contenant environ 10 12 cellules T αβ, contient environ 7 octobre au 8 octobre αβTCRs distinctes 3-4. Ce répertoire très diversifié offre la possibilité de reconnaissance de la vaste gamme de peptides de pathogènes potentiels qui nécessiterait une réponse des cellules T à une immunité protectrice. On estime que la fréquence de la réponse des cellules T à un antigène étranger présenté donné par auto-MHC est de l'ordre de 10 -4 à 10 -7 en l'absence de réponse immunitaire à cet antigène avant 5. Le répertoire des lymphocytes T naïfs est formée par sélection thymique à assurer la capacité de reconnaître des auto-antigènes MHC de présentation de peptides et de limite réagissentivité contre les antigènes auto-peptide, la maximisation de l'utilité potentielle de la médiation de l'immunité protectrice 2. Cependant, en violation de cette réactivité conçu, une fréquence relativement importante, 10 -3 à 10 -4, de cellules T d'individus immunologiquement naïfs répondre à une stimulation avec des cellules allogéniques, reconnaissant à la fois les molécules du CMH étrangers ainsi que les peptides endogènes qu'ils présentent 6. La reconnaissance de ligands PMHC allogéniques est structurellement similaire à la reconnaissance d'antigènes étrangers présentés par CMH du soi; du TCR permet d'interactions biochimiques essentiels à la fois avec la molécule MHC allogénique aussi bien que le peptide présenté 7. La robustesse de la réponse des cellules T aux résultats de stimulation allogéniques de la diversité de complexes de pMHC présent sur la surface des cellules allogéniques 8. On estime que chaque CMH présente à environ 2 x 10 4 différents antigènes peptidiques endogènes 9. Ce breadth de réponse à une stimulation allogénique est un aspect important de la pathologie cliniquement pertinentes, telles que le rejet d'allogreffe ou la réaction du greffon contre l'hôte (GVHD), résultant de T alloréactivité cellulaire.
Étude de cellules T réponses alloréactives humaines a toujours compté sur l'examen des réponses polyclonaux de cellules T naïves après stimulation avec des cellules allogéniques. Stimulation répétée avec la même lignée cellulaire allogénique combiné avec dilution limitante analyse est capable de générer des cellules T clonales de reconnaissance définis allogénique HLA de 10. Cependant, cette approche est problématique pour examiner les réponses aux différents ligands PMHC allogéniques, comme le répertoire vaste et diversifié de endogène pMHC complexes présente un allogénique HLA donné stimule un large répertoire de cellules T. Cette stimulation de la population en vrac et en limitant approche de dilution, il faudrait dépistage d'un grand nombre de clones pour isoler les cellules T avec la Reacti souhaitéevité contre un seul ligand pMHC. En outre, la fréquence des cellules T répondant à un ligand pMHC allogénique individu est relativement faible chez les populations de cellules T naïves, qui présente un obstacle à la génération efficace de clones de lymphocytes T humains en réponse à un antigène donné.
L'identification et l'isolement des cellules T spécifiques de l'antigène à partir de populations polyclonales ont été activées par le développement de multimères pMHC 11 marqué par un fluorophore. Cette approche utilise des antigènes peptidiques spécifiques chargés dans des molécules de CMH solubles recombinants biotinylés, qui sont marqués par liaison à un fluorophore de la streptavidine marquée. Multimérisation de pMHC augmente l'avidité, la compensation de l'affinité intrinsèquement faible (M) de TCR pour ligands PMHC solubles. Les cellules marquées peuvent être identifiées et isolées par cytométrie de flux. Cependant, cette approche est encore limitée par la faible fréquence des cellules T spécifiques de l'antigène chez les populations de cellules T naïves, qui sont typiquementordres de grandeur inférieure à la limite de l'identification et quantification précise sur la plupart des cytomètres de flux. Pour remédier à cette limitation, un procédé de marquage pMHC de tétramère et d'enrichissement par billes magnétiques subséquente des cellules tétramère marqué a été mis au point 12. Cette méthode a montré une détection fiable, le dénombrement et l'isolement des cellules T spécifiques de l'antigène à basse fréquence.
Ce protocole décrit un protocole efficace pour la génération de clones de cellules T humains qui réagissent spécifiquement aux ligands individuels pMHC allogéniques. Le protocole applique pMHC (HLA) l'étiquetage des multimères et d'enrichissement pour l'isolement de cellules T humaines spécifiques à l'alloantigène de cytométrie de flux de tri cellulaire et un procédé robustes pour la culture in vitro de cellules T humaines pour permettre la production de clones de cellules triées individuelles de cellules T ( Vue d'ensemble de la figure 1).
Protocole
NOTE: Ce protocole nécessite l'utilisation d'échantillons de sang périphérique chez des volontaires humains. Toutes les recherches sur des sujets humains doit être examiné et approuvé par un comité d'examen institutionnel études humaines pour assurer la conformité avec la Déclaration d'Helsinki (2013) et la Health Insurance Portability and Accountability Act de 1996.
1. Isolement de cellules de sang total T
- Avant de commencer, chauffer le milieu à gradient de densité à la température ambiante. Aliquote de 4 ml de milieu de gradient de densité dans 2-4 stériles tubes de 15 ml à centrifuger coniques (1 tube sera utilisé pour chaque 10 ml de volume total de sang dilué).
- Obtenir 10-20 ml de sang dans l'héparine 1-2 sodium enrobés par pulvérisation (vert-haut) tubes de prélèvement de sang veineux. Recueillir des échantillons humains sous la supervision de personnes qualifiées et selon un Institutional Review Board protocole approuvé.
- Essuyez l'extérieur des tubes avec 70% d'éthanol. Retirez délicatement les sommets de la firempli les tubes de prélèvement et décanter le sang dans un tube de 50 ml à centrifuger stérile.
- Ajouter 10 ml de solution saline tamponnée au phosphate stérile (PBS) dans chaque tube de collecte de sang. Recueillir le PBS, ajouter le sang tout décanté, et mélanger doucement.
- Ajouter 25 ul T Cellule humaine enrichissement cocktail / 2 ml volume total. Incuber à température ambiante pendant 20 min.
- L'utilisation d'une pipette de 10 ml, la couche de 10 ml de 1: 1 de sang dilué légèrement au-dessus du milieu à gradient de densité. Attention à ne pas perturber la surface du milieu de gradient de densité.
- Centrifugeuse sang en couches et le milieu de gradient de densité à 1200 g pendant 20 minutes à 20 o C.
- Retirer les tubes de la centrifugeuse, en prenant soin de ne pas perturber l'interface entre le milieu à gradient de densité et la couche de globules blancs à l'interface entre le milieu à gradient de densité et le plasma dilué. L'utilisation d'un pipette de 5 ml, de recueillir soigneusement la couche de leucocytes et de les transférer à un nouveau 50 ml baignoire conique stérilee.
- Ajouter PBS pour porter le volume de la PBL recueilli 50 ml et mélanger doucement.
- Centrifuger à 600 g pendant 5 min à 20 ° C. Décanter.
- Remettre en suspension les cellules en culot dans 10 ml flux stérile cytométrie tampon (PBS stérile filtrée contenant 1% de BSA) de tri. Échantillon de 10 pi de suspension cellulaire, diluer 01h10 (ajouter 90 pi) au bleu trypan à compter les cellules en utilisant un hématimètre (rendement attendu: 1 - 4 x 10 6 cellules / tubes de sang total).
- Retirer aliquote de 1 ml, transférer à 5 ml tube, et garder sur la glace pour l'analyse des cellules T non étiquetés par tétramère. Gardez la suspension cellulaire sur la glace.
2. Enrichissement magnétique de cellules T allo-antigène-spécifiques
- Diluer allo pMHC tétramère à 1: 100 dans le tampon de tri stérile.
- Centrifuger la suspension de cellules (9 ml de l'étape 1.11) à 600 g pendant 5 min à 20 ° C. Décanter.
- Ajouter 50 ul de dilution allo pMHC tétramère à granulésles cellules (à 10 7 cellules). Mélanger par pipetage doux. Transférer dans un tube de 5 ml stérile. Incuber pendant 30 min à température ambiante.
- Ajouter 2 ml sorte tampon. Centrifuger à 600 g pendant 5 min à 20 ° C. Décanter.
- cellules de remettre en suspension dans 100 ul de tampon de tri. Ajouter 10 ul biotine sélection Cocktail et incuber pendant 15 min à température ambiante.
- Ajouter 5 ul magnétique Nanoparticules et incuber pendant 10 min à température ambiante.
- Ajouter 2,5 ml sorte de tampon et mélanger doucement à la pipette. Retirer 100 pi aliquote, transférer à 5 ml tube, et garder sur la glace pour l'analyse des cellules pré-enrichissement.
- Placer le tube de 5 ml contenant la suspension de cellules dans l'aimant de séparation de cellules. Le plafond devrait être vaguement au sommet du tube. Incuber pendant 5 min à température ambiante.
- Retirez délicatement le bouchon du tube. En tenant le tube et aimant ensemble, décanter le contenu du tube dans un nouveau tube de 5 ml. Ne touchez pas ou ne le secouez le contenu du tube àéliminer les dernières gouttes de liquide.
- Enlevez le tube de l'aimant. Ajouter 2 ml de tampon de tri froid et mélanger doucement à la pipette. Échantillon de 10 pi de suspension cellulaire, diluer 1: 2 (ajouter 10 pi) au bleu trypan à compter les cellules en utilisant un hématimètre.
3. Préparation des cellules T pour une seule cellule cytométrie en flux Tri cellulaire
- Ajouter tampon de tri froid à tous les tubes de cellules (tétramère non marquée, tétramère marqué non enrichi, et tétramère marqué enrichi) pour porter le volume à 3 ml.
- Centrifugeuse à 600 g pendant 5 min à 4 ° C et tampon décanter pour récupérer les cellules.
- Reprendre culot de cellules dans 25 tampon de tri ul froid. Ajouter 5 ul de bloc Fc humaine. Incuber sur de la glace pendant 20 min.
- Préparer des anticorps pour la cytométrie en flux tri. Mélanger 15 pi de tampon de tri, 15 pl anti-CD5, 15 pl anti-CD14, et 15 pi anti-CD19.
- Ajouter 20 ul de mélange d'anticorps à des cellules de chaque tube. Incuber sur de la glace pendant 20 min.
- Ajouter 2 ml de tampon de tri froid dans chaque tube.
- Centrifugeuse à 600 g pendant 5 min à 4 ° C et tampon décanter pour récupérer les cellules.
- Remettre en suspension les cellules à une concentration de 1 à 2 x 10 6 cellules / ml dans du tampon de tri.
- Aliquote de 100 ul de milieu de culture de cellules humaines T (DMEM d'Iscove complété avec 2 mM de Glutamax, HEPES 10 mM, 50 ug / ml de gentamycine, 50 uM de 2-mercaptoéthanol, 10% de sérum humain AB inactivé par la chaleur, et de 2,5 ng ml humaine recombinante / IL-2) à chaque puits d'une plaque ronde à 96 puits de culture de cellules de base. Garder sur la glace.
4. Isolement de cellules T tétramère marqué par l'unité de cytométrie en flux des cellules Tri
- Établir cytométrie en flux paramètres de déclenchement pour identifier les cellules allo-pMHC tétramères marqués T (Figure 2.A). Utilisez la fraction de tétramère de pré-enrichissement marqué à établir gating des stratégies pour éliminer les doublets, porte sur les lymphocytes, les cellules exprimant CD19 exclure B et CD14exprimant les monocytes, et identifier les cellules T par l'expression CD5. Utilisez l'échantillon non étiqueté avec tétramère comme une fluorescence moins un contrôle d'établir le déclenchement pour l'identification de cellules tétramère contraignant T (avec 0% de CD5 + CD14 - CD19 - les cellules de l'échantillon ne sont pas étiquetés par tétramère doivent se situer dans cette porte) .
REMARQUE: un noise gate stratégies qui ne sont pas suffisamment strictes seront conduire à l'isolement de cellules non-T ou cellules T qui ne sont pas l'antigène spécifique, tandis que les stratégies de déclenchement trop restrictives permettront de réduire le nombre de cellules T isolées. - Programmer les paramètres de la plaque de la sorte d'une seule cellule. Dirigez la trieuse de placer 1 CD5 + CD14 - CD19 - tétramère + cellule dans chaque puits. Dirigez la plaque mis en place pour contenir 1 puits du contrôle négatif (pas de cellules triées dans le puits) et 1 puits du contrôle positif (100 CD5 + CD14 - CD19 - tétramère - cellules) dans chaque rangée (Figure 2.B ).
- Utilisation de l'écoulement établi cytométrie stratégie de déclenchement et de la configuration de la plaque, isoler des cellules individuelles de tétramère contraignant T directement dans la plaque de 96 puits en utilisant un trieur de cellules par cytométrie en flux.
5. La culture et l'expansion de clones allo-spécifiques des cellules T
- Après l'achèvement de tri cellulaire, la plaque centrifuge à 600 g pendant 5 min à 20 ° C. Placer la plaque de culture à 37 ° C dans un incubateur à CO2 de 6%.
- Vortex stimulation microbilles anti-CD3 / anti-CD28 pour 30 secondes pour remettre en suspension les perles.
- Calculer le volume de billes d'activateur nécessaires pour la stimulation des cellules T recueillies (0,5 ul de microbilles activateur / puits). Transférer le volume calculé de billes stimulatrices dans un tube de 5 ml stérile. Ajouter 1 ml de milieu de culture humaine de cellules T et de vortex.
- Placez le tube avec microbilles suspension dans l'aimant. Le plafond devrait être vaguement au sommet du tube. Incuber pendant 2 min à la température ambiante.
- Retirez délicatementle bouchon du tube. En tenant le tube et aimant ensemble, le contenu du tube de décantation dans un nouveau tube de 5 ml. Ne touchez pas ou ne le secouez le contenu du tube.
- Enlevez le tube de l'aimant. Ajouter milieu de culture de cellules humaines T (100 ul de milieu / 0,5 ul de microbilles ajoutées initialement).
- Aliquote de 100 pi d'activateur suspension de billes dans chaque puits de la plaque de 96 puits. Incuber la plaque de culture à 37 ° C dans un incubateur à CO 2 à 6%.
- Surveiller la croissance des cellules quotidiennement par l'examen microscopique. Petits amas de cellules proliférantes peuvent être observées au microscope après 5 - 7 jours de culture (Figure 3.A).
REMARQUE: Visualisation des cultures de cellules T à ce point peut être difficile, et l'absence d'amas cellulaires observables à ce stade ne peut être le signe d'un manque de cellules T croissance. - 14 jours après l'isolement cellulaire, les cultures d'alimentation en retirant soigneusement 100 pi de milieu off de la partie supérieure de la culture et de la remplacer par 100 μ; L de milieu de culture de cellules T humaines fraîches. Continuer l'incubation de la plaque de culture à 37 ° C dans un incubateur à CO 2 à 6%.
- Surveiller la croissance cellulaire tous les jours par examen et l'évaluation de la couleur du milieu de culture microscopique. Les grands groupes de cellules doivent être visibles au microscope 2 - 3 jours après le changement de milieu. Macroscopiquement, culots cellulaires devraient devenir visible pendant les 14 jours suivants le changement moyen.
- A 28 jours suivants l'isolement cellulaire, identifier les clones de plus en plus par l'examen microscopique. Transférer le volume de 200 ul de la plaque à 96 puits de cultures de croissance positive à des puits individuels d'une plaque de culture tissulaire à 48 puits contenant 200 milieu de culture de cellules T humaines ul.
- Ajouter 100 ul contenant 12,5 ul de microbilles anti-CD3 / anti-CD28 de stimulation (préparé comme décrit dans les sections 05.02 à 05.06). Incuber la plaque de culture à 37 ° C dans un incubateur à CO 2 à 6%.
- Surveiller la croissance de la culture quotidienne par ex microscopiqueamination et l'évaluation de la couleur du milieu de culture. Quand la culture de cellules T atteint> 2x10 6 / ml (généralement 3-5 jours après re-stimulation, peut être estimée par la journée que les médias commence à se tourner jaune paille), la culture de transfert à un puits d'une plaque de culture de 24 puits tissu et ajouter 500 milieu de cellule T humaine ul.
- À 10-14 jours suivre re-stimulation, recueillir 200 pi de culture de cellules T à évaluer allo spécificité par cytométrie en flux de pMHC tétramère contraignant (en utilisant la stratégie de l'étiquetage et de déclenchement décrites dans les sections 3 et 4.1).
6. long terme Re-stimulation et la culture des clones de cellules T
- Continuellement re-stimuler et de développer les cultures de cellules T clonales avec la spécificité désirée peut être tous les 14 jours. Recueillir les cultures de cellules T dans un tube de 5 ml stérile à 14 jours suivant la dernière stimulation de microbilles anti-CD3 / CD28. Combinaison de plusieurs puits d'un même clone de cellules T dans un seul tube, jusqu'à un volume de 2,5 ml.
- Ajouter moyen pour amener le volume final de 2,5 ml et mélanger doucement à la pipette.
- Placer le tube 5 ml de la culture contenant des cellules T dans l'aimant de séparation de cellules pour éliminer les vieilles microbilles de la culture. Le plafond devrait être vaguement au sommet du tube. Incuber pendant 5 min à température ambiante.
- Retirez délicatement le bouchon du tube. En tenant le tube et aimant ensemble, décanter la suspension de cellules dans un nouveau tube de 5 ml. Ne touchez pas ou ne le secouez le contenu du tube pour éliminer les dernières gouttes de liquide.
- Ajouter moyen pour amener le volume final de 4 ml. Échantillon de 10 pi de compter les cellules en utilisant un hématimètre.
- Récupérer les cellules T par centrifugation à 600 xg pendant 5 min à température ambiante.
- Décanter le surnageant. Remettre en suspension les cellules T à 10 6 cellules / ml de milieu de culture de cellules T humaines. Transfert des aliquotes de 1 ml à chaque puits d'une plaque de culture tissulaire à 24 puits.
- Re-stimuler les cellules T par l'ajout de 100 médias ul contenant 12,5 pi de stimulation anti-CD3 / amicrobilles nti-CD28 (tels que décrits dans les sections 5.2 à 5.6). Incuber à 37 ° C dans un incubateur à CO 2 à 6%.
Résultats
Ce protocole décrit la génération de cultures de cellules T humaines clonale défini allo spécificité par un enrichissement par billes magnétiques et flux unicellulaire cytométrie tri stratégie. La figure 1 donne un aperçu du processus.
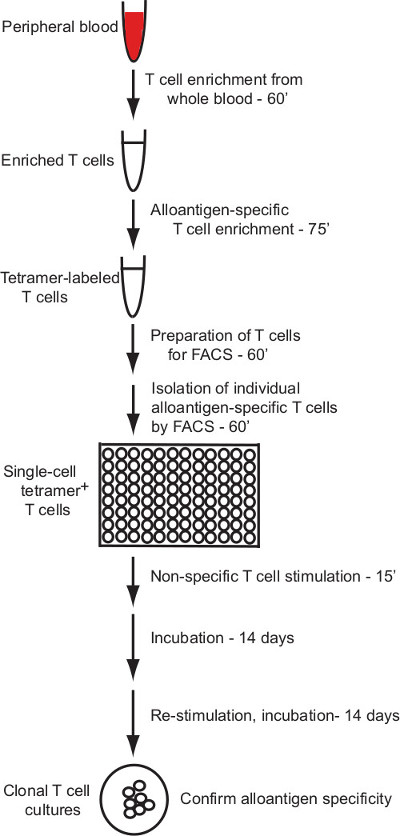
Figure 1: vue d'ensemble Protocole Le protocole décrit ici fournit une méthode fiable pour la...
Discussion
T cell alloreactivity is a long-studied and clinically-relevant phenomenon. The robust proliferative and effector responses of T cells to allogeneic stimulation has enabled extensive analyses of human T cell responses in vitro through relatively straightforward mixed lymphocyte reactions of peripheral blood T cells against inactivated allogeneic cells. However, these primary alloreactive T cell responses are oligoclonal, comprised of a large number of individual T cells responding to specific alloantigens. This ...
Déclarations de divulgation
The authors declare no competing financial interests.
Remerciements
The author would like to thank the NIH Tetramer Core Facility for tetramer production. The author would also like to thank E.D. O’Connor and K.E. Marquez at the UCSD Human Embryonic Stem Cell Core Facility flow cytometry laboratory for assistance in cell sorting. This work was funded by National Institutes of Health grant K08AI085039 (G.P.M.).
matériels
| Name | Company | Catalog Number | Comments |
| Sodium heparin venous blood collection tube 16 x 100 mm | Becton, Dickenson and Company | 366480 | |
| Lymphoprep | Stemcell Technologies | 7801 | |
| Rosette Sep Human T Cell Enrichment Kit | Stemcell Technologies | 15061 | |
| Dulbecco's PBS, 1x without Ca or Mg | Corning | 21-031-CV | |
| Bovine serum albumin | Sigma-Aldrich | A7906 | |
| EDTA | Sigma-Aldrich | E6635 | |
| Fluorophore-labeled pMHC tetramer | NIH Tetramer Facility | NA | |
| EasySep Biotin Selection Kit | Stemcell Technologies | 18553 | |
| EasySep Selection magnet | Stemcell Technologies | 18000 | |
| TruStain FcX Human Fc blocking solution | Biolegend | 422301 | |
| Anti-CD5 PE-Cy7 (clone UCHT2) | Biolegend | 300621 | |
| Anti-CD14 FITC (clone HCD14) | Biolegend | 325603 | |
| Anti-CD19 FITC (clone HIB19) | Biolegend | 302205 | |
| Iscove's DMEM, without b-ME or L-glutamine | Corning | 15-016-CV | |
| HEPES | Corning | 25-060-CI | |
| b-Mercaptoethanol | Life Technologies | 21985-023 | |
| Glutamax | Life Technologies | 35050061 | |
| Gentamicin sulfate (50 mg/ml) | Omega Scientific | GT-50 | |
| Human AB serum, male donor | Omega Scientific | HS-30 | |
| Recombinant human IL-2 | Peprotech | AF 200-02 | |
| Dynabeads Human T-Activator CD3/CD28 | Life Technologies | 11131D | |
| Media | |||
| Cell sorting buffer | |||
| PBS, pH 7.4 | 1 L | ||
| BSA | 10 g | ||
| EDTA (0.5 M) | 2 ml | ||
| Human T Cell Culture Medium | |||
| Iscove's DMEM | 351.6 ml | ||
| Heat-inactivated human AB serum | 40 ml | ||
| HEPES (1 M) | 4 ml | ||
| Glutamax (100x) | 4 ml | ||
| Gentamicin (50 mg/ml) | 0.4 ml | ||
| b-mercaptoethanol (14.3 M) | 1.4 ml | ||
| Recombinant human IL-2 (1 mg/ml) | 1 ml |
Références
- Smith-Garvin, J. E., Koretzky, G. A., Jordan, M. S. T cell activation. Annu. Rev. Immunol. 27 (1), 591-619 (2009).
- Morris, G. P., Allen, P. M. How the TCR balances sensitivity and specificity for the recognition of self and pathogens. Nat. Immunol. 13 (2), 121-128 (2012).
- Arstilla, T. P., et al. A direct estimation of the human αβ T cell receptor diversity. Science. 286 (5441), 958-961 (1999).
- Robbins, H. S., et al. Comprehensive assessment of T-cell receptor β-chain diversity in αβ T cells. Blood. 114 (19), 4099-4107 (2009).
- Alanio, C., Lemaitre, F., Law, H. K. W., Hasan, M., Albert, M. L. Enumeration of human antigen-specific naive CD8+ T cells reveals conserved precursor frequencies. Blood. 115 (18), 3718-3725 (2010).
- Suchin, E. J., et al. Quantifying the frequency of alloreactive T cells in vivo: new answers to an old question. J. Immunol. 166 (2), 973-981 (2001).
- Felix, N. J., Allen, P. M. Specificity of T-cell alloreactivity. Nat. Rev. Immunol. 7 (12), 942-953 (2007).
- Morris, G. P., Ni, P. P., Allen, P. M. Alloreactivity is limited by the endogenous peptide repertoire. Proc. Natl. Acad. Sci. USA. 108 (9), 3695-3700 (2011).
- Suri, A., et al. In APCs, the autologous peptides selected by the diabetogenic I-Ag7 molecule are unique and determined by the amino acid changes in the P9 pocket. J. Immunol. 168 (3), 1235-1243 (2002).
- Yssl, H., Spits, H. Generation and maintenance of cloned human T cell lines. Curr. Protoc. Immunol. 7, (2002).
- Altman, J. D., et al. Phenotypic analysis of antigen-specific T lymphocytes. Science. 274 (5284), 94-96 (1996).
- Moon, J. J., et al. Naive CD4+ T cell frequency varies for different epitopes and predicts repertoire diversity and response magnitude. Immunity. 27 (2), 203-213 (2007).
- Chicz, R. M., et al. Specificity and promiscuity among naturally processed peptides bound to HLA-DR alleles. J. Exp. Med. 178 (1), 27-47 (1993).
- Ni, P. P., Allen, P. M., Morris, G. P. The ability to rearrange dual TCRs enhances positive selection, leading to increased allo- and autoreactive T cell repertoires. J. Immunol. In press, (2014).
- Morris, G. P., Uy, G. L., Donermeyer, D., DiPersio, J. F., Allen, P. M. Dual receptor T cells mediate pathologic alloreactivity in patients with acute graft-versus-host disease. Sci. Transl. Med. 5 (188), (2013).
- Altman, J. D., Reay, P. A., Davis, M. M. Formation of functional peptide complexes of class II major histocompatibility complex proteins from subunits produced in Escherichia coli. Proc. Natl. Acad. Sci. USA. 90 (21), 10330-10334 (1993).
- Sabatino, J. J., Huang, J., Zhu, C., Evavold, B. D. High prevalence of low affinity peptide-MHC II tetramer-negative effectors during polyclonal CD4+ T cell responses. J. Exp. Med. 208 (1), 81-90 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon