Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Ciblage des Thiols de cystéine pour in Vitro in situ Glycosylation des protéines recombinantes
Dans cet article
Résumé
Des analyses biochimiques et structurales des protéines glycosylées exigent des quantités relativement importantes des échantillons homogènes. Nous présentons ici une méthode chimique efficace pour in site glycosylation des protéines recombinantes purifiée à partir de bactéries en ciblant les réactifs Cys thiols.
Résumé
Stromale interaction molécule-1 (STIM1) est un type-j’ai une protéine transmembranaire située sur le réticulum endoplasmique (re) et la membrane plasmique (PM). STIM1 ER-résident réglemente l’activité des canaux PM Orai1 dans un processus connu comme stocker entrée de commande de calcium (Ca2 +) qui est le principal Ca2 + signalisation processus qui entraîne la réponse immunitaire. STIM1 subit post-traductionnelles N- glycosylation sur deux sites de crash luminales au sein du Ca2 + détection domaine de la molécule. Toutefois, la biochimiques, biophysiques, et effets biologiques structure des N- glycosylées STIM1 ont été mal compris jusqu'à ce que récemment en raison de l’incapacité d’obtenir facilement des niveaux élevés de homogène N- glycosylées protéine. Nous décrivons ici la mise en œuvre d’une approche in vitro chimique qui s’attache à moitiés de glucose aux sites de protéine spécifique applicables à la compréhension des effets sous-jacents de N- glycosylation sur le mécanisme et la structure des protéines. En utilisant la spectroscopie de résonance magnétique nucléaire de solution, nous évaluons les deux l’efficacité de la modification, ainsi que les conséquences structurelles de l’attachement de glucose avec un seul échantillon. Cette approche peut facilement être adaptée pour étudier les protéines glycosylées myriad trouvées dans la nature.
Introduction
Stocker les exploités de calcium (Ca2 +) entrée (SOCE) est la principale voie par laquelle les cellules immunitaires prennent Ca2 + de l’espace extracellulaire dans le cytosol. Dans les lymphocytes T, lymphocytes T récepteurs situés sur la membrane plasmique (PM) lient antigènes qui activent la protéine-tyrosine kinases (évaluées à 1,2,3). Une cascade de phosphorylation entraîne l’activation de la phospholipase-γ (PLCγ) qui intervient par la suite dans l’hydrolyse de la membrane phosphatidylinositol 4, 5-bisphosphate (PIP2) en diacylglycérol et inositol 1,4,5-trisphosphate (IP3 ). IP3 est un petit Messager diffusible qui se lie aux IP3 récepteurs (IP3R) sur le réticulum endoplasmique (re) ainsi ouvrir ce canal récepteur et permis Ca2 + s’écouler dans le gradient de concentration de l’ER Lumen dans le cytosol (évaluée à 4). Signalisation des protéines G couplées et récepteurs à tyrosine kinase dans une variété d’autre cellules excitables et non-excitables types plomb pour la même production d’IP3 et l’activation des IP3Rs de récepteur.
En raison de la Ca2 + stockage capacité finie de l’urgence, l’IP3-communiqué de la médiation et augmentation résultante cytosolique Ca2 + est seulement transitoire ; Toutefois, cette diminution de l’ER luminale Ca2 + effets profondément stromales interaction molécule-1 (STIM1), un type-je transmembranaires (TM) protéines surtout présents sur la membrane de ER 5,6,7. STIM1 contient un lumen orienté Ca2 + détection domaine composé d’une paire de main EF et du α-motif (EFSAM). Coiled-coil de cytosolique axé sur trois domaines sont séparés des EFSAM par le seul domaine de TM (évaluée à 8). Sur ER luminale Ca2 + l’appauvrissement, EFSAM subit une oligomérisation couplés déstabilisation 7,9 , qui provoque des réarrangements structuraux de la TM et domaines coiled-coil 10. Ces changements structurels aboutissent à un piégeage de STIM1 ER-PM jonctions 11,12,13,14 , grâce à des interactions avec les PM phosphoinositides 15, 16 et Orai1 sous-unités 17,18. Orai1 protéines sont les sous-unités de PM qui assemblent pour former Ca2 + canaux 19,20,21,22. Les interactions de STIM1-Orai1 au niveau des jonctions ER-PM facilitent une Ca2 + déblocage activé Ca2 + (CRAC) canal conformation ouverte qui permet le mouvement de Ca2 + dans le cytosol de la concentration élevée de la espace extracellulaire. Dans les cellules immunitaires, les soutenue cytosolique Ca2 + élévations via les canaux CRAC induisent la Ca2 +- calmoduline/calcineurine dépendante déphosphorylation du facteur nucléaire des lymphocytes T activés qui par la suite entre le noyau et commence la régulation transcriptionnelle des gènes favorisant l’activation de lymphocytes T 1,3. Le processus d’activation canal CRAC par STIM1 23,24 via induite par l’agoniste ER luminale Ca2 + l’appauvrissement et la résultante soutenue cytosolique Ca2 + élévation est collectivement appelé SOCE 25. Le rôle vital de SOCE en lymphocytes T est évident par les études qui ont démontré que des mutations héréditaires dans les STIM1 et les Orai1 peuvent causer des déficit immunitaire combiné sévère syndromes 3,19,26, 27. EFSAM initie SOCE après détection ER-luminale Ca2 + épuisement par la perte de Ca2 + coordination à la main EF canonique, conduisant finalement à la déstabilisation couplés auto-association 7, 28,,29.
La glycosylation est la covalente et la transformation des structures oligosaccharide, également connu sous le nom de glycanes, à travers différentes étapes biosynthétiques de l’ER et Golgi (évaluée à 30,32,,33). Il existe deux types prédominants de glycosylation chez les eucaryotes : N-liés et O-liée, selon les acides aminés spécifiques et l’atome de la liaison de raccordement. Dans la N- glycosylation, glycanes sont attachés à l’amide de chaîne latérale de l’Asn, et dans la plupart des cas, l’étape d’initiation se produit dans la salle d’urgence comme le polypeptide chaîne se déplace dans le lumen 34. La première étape de la N- glycosylation est le transfert d’une structure de base de quatorze-sucre composé de glucose (Glc), mannose (Man) et N- acétylglucosamine (GlcNAc) (c.-à-d. Glc3Man9GlcNAc2) de l’ER membrane lipidique par un oligosaccharyltransferase 35,36. Des mesures supplémentaires, telles que le clivage ou le transfert des résidus de glucose, sont catalysées aux urgences par des glycosyltransférases et glycosidases spécifiques. Certaines protéines qui quittent la salle d’urgence et passer dans l’appareil de Golgi peuvent être encore transformés 37. O- glycosylation se réfère à l’ajout des glycanes, habituellement au groupe hydroxyle côté chaîne de résidus Ser ou Thr, et cette modification a été apportée tout à fait dans l’appareil de Golgi 33,34. Il y a plusieurs structures de O- glycanes qui peuvent être constitués de N- acétylglucosamine, fucose, galactose et l’acide sialique avec chaque monosaccharide ajouté séquentiellement 33.
Alors qu’aucune séquence spécifique n’a été identifié comme condition sine qua non pour de nombreux types de O- glycosylation, une séquence consensus commun a été associée avec le N-liée modification : Asn-X-Ser/Thr/Cys, où X peut être n’importe quel acide aminé sauf Pro 33. STIM1 EFSAM contient deux de ces sites de glycosylation - consensus N: Asn131-Trp132-Thr133 et Asn171-Thr172-Thr173. En effet, des études antérieures ont montré que EFSAM peut être N- glycosylées en cellules mammifères Asn131 et Asn171 38,39,40,41. Toutefois, des études antérieures des conséquences de la N- glycosylation sur SOCE ont été incongrues, suggérant supprimée, potentialise ou pas d’effet de cette modification post-traductionnelle SOCE activation 38,= « xref » > 39,40,41. Ainsi, la recherche sur les conséquences structurels, biochimiques et biophysiques sous-jacents de EFSAM N- glycosylation est essentielle pour comprendre les effets régulateurs de cette modification. En raison de l’exigence pour les niveaux élevés de protéines homogènes dans ces expériences in vitro , a appliqué une approche sélective des site pour attacher par covalence moitiés de glucose à EFSAM. Fait intéressant, Asn131 et Asn171 de glycosylation causé des changements structurels qui convergent dans le cœur EFSAM et d’améliorer les propriétés biophysiques qui favorisent la médiation STIM1 SOCE 42.
La fixation chimique du glycosyl groupes thiols Cys a été bien établie par un ouvrage qui tout d’abord démontré l’utilité de cette approche sans enzymes pour comprendre les effets propres au site de glycosylation sur protéine fonction 43 , 44. plus récemment et en ce qui concerne les STIM1, les résidus Asn131 et Asn171 ont été mutés à Cys et glucose-5-(methanethiosulfonate) [glucose-5-(MTS)] a été utilisé de façon covalente lier glucose à thiols libres 42. Nous décrivons ici, cette approche qui non seulement utilise mutagénèse d’incorporer les résidus Cys spécifiques de site pour modification, mais s’applique également à spectroscopie de résonance magnétique nucléaire (RMN) solution pour évaluer rapidement l’efficacité modification tant structurels perturbations suite à la glycosylation. Notamment, cette méthodologie générale est facilement adaptable pour étudier les effets de l' O- ou N- glycosylation d’une inoculation produit protéique.
Protocole
1. réaction en chaîne par polymérase (PCR)-médiée par mutagénèse dirigée pour l’incorporation de Cys dans un vecteur d’expression bactérienne pET-28 a.
- Déterminer la concentration du vecteur animal-28 a (c'est-à-dire ADN bicaténaire) en utilisant un coefficient d’extinction ultraviolet (UV) de 0,020 (μg/mL) cm -1 à 260 nm.
- Synthétiser une paire d’amorces mutagéniques complémentaires pour chaque mutation Cys telle j’ai) il y a un minimum de 15 nucléotides complémentaires pour le modèle avant l’incompatibilité de la première base et 15 nucléotides complémentaires pour le modèle après le incompatibilité de base finale, ii) longueur totale apprêt n’excède pas 45 nucléotides et iii) une guanine ou cytosine est situé à la position du premier et le dernier nucléotide de chaque amorce (tableau 1). Assurer la synthèse de l’apprêt est effectuée en utilisant une purification d’échelle et la cartouche de 0,025 μmol.
- à l’aide d’une haute fidélité ADN polymérase, mis en place deux 20 µL PCR réaction des mélanges : celui contenant l’amorce vers l’avant et le second contenant l’amorce de marche arrière. Préparer chaque mélange pour contenir la concentration finale de 1 x PCR tampon avec 1,5 mM MgCl 2, dNTPs mM 0,2, 0,5 μM primer, 0,4 μL DMSO, modèle de ng/μL 1.25 0,02 U/μl haute fidélité ADN polymérase ADN.
- Cycle thermiquement les mélanges distincts à l’aide d’un protocole de trois étapes : 98 ° C pendant 30 s (dénaturation), 53-56 ° C pendant 30 s (recuit), 72 ° C 30 s kilobase(kb) -1 (extension) du modèle de l’ADN. Répéter le programme de température pour 5 cycles et ajouter une étape d’extension finale 72 ° C pendant 7,5 min.
- Après l’initiale PCR avec des amorces et inverses dans des tubes distincts, combiner les produits dans un tube unique (c'est-à-dire 40 μL de total) et continuer la réaction PCR pendant une 20 cycles supplémentaires en utilisant les mêmes paramètres de cyclisme comme indiqué au point 1.4.
- Electrophorese 15 μl du mélange réactionnel PCR sur un gel d’agarose de 1 % (p/v) de gel à l’aide de 0,5 x Tris, l’acide acétique, acide acétique éthylène diamine tétra (EDTA) tampon (TAE). En tant que contrôles, electrophorese une quantité équivalente d’ADN qui n’a pas été amplifiés par PCR et une portion de l’échelle d’ADN de référence qui contient des bandes de marquage fois supérieur et inférieur à la taille prévue du produit PCR.
- Après électrophorèse à 120 V pendant 40 min, submerger le gel dans l’eau contenant 0,5 bromure d’éthidium μg/mL et agiter pendant 30 min à température ambiante. Confirmer le modèle pleine longueur a été amplifié par les amorces mutagéniques comme une augmentation dans l’intensité de fluorescence de bromure d’éthidium relative de la bande amplifiée par rapport à la bande de modèle de contrôle sous la lumière ultraviolette (302 nm).
- Si aucune amplification n’est apparente, répéter la PCR après avoir réglé la température de recuit entre la plage de température de 53-56 ° C par incréments de 0,5 ° C.
- Lors de la confirmation de l’amplification du modèle par les amorces mutagènes, traiter le μL de ~ 25 restant du mélange réactionnel PCR avec l’enzyme de restriction DpnI pour digérer le modèle méthylé ADN. Utilisez DpnI à 0,5 µL (10 unités) par 25 μL PCR réaction mélange et une concentration finale de 1 × DpnI tampon de réaction. Incuber pendant 2,5 heures à 37 ° C.
- Après la digestion de modèle, ajoutez environ 5-10 μL du mélange digéré à 100 µL de chaleur choc compétente DH5α Escherichia coli cellules dans un tube de microcentrifuge de 1,75 mL. Incuber le mélange cellulaire-ADN sur la glace pour 60 min.
- Choc thermique le mélange de l’ADN cellulaire dans le tube de microcentrifuge à 42 ° C pendant 45 s sur un bloc de chaleur sèche. Après incubation le mélange sur la glace pendant 3 min, ajouter 900 μL de bouillon de la température de la pièce Luria-Bertani (LB) dans les cellules et transférer la suspension totale de cellules dans un tube à fond rond stérile de 14 mL.
- Incuber la suspension de cellules à 37 ° C pendant 90 min avec une agitation constante à 190 tr/min.
- Par la suite, transfert de la suspension de cellules dans un tube de microcentrifuge de 1,75 mL et centrifuger à 10 000 x g pendant 5 min à température ambiante.
- Après la centrifugation, supprimer 900 μl du liquide surnageant et remettre en suspension les cellules bactériennes par pipetage doux dans le reste de 100 μL de lb
- Transférer la suspension concentrée des cellules résultantes sur un LB-gélose contenant antibiotique qui est sélectif pour le vecteur d’expression (c'est-à-dire 60 μg/mLKanamycin). Étendre la suspension uniformément sur la gélose de façon aseptique et incuber pendant environ 16 heures à 37 ° C.
- Le lendemain, ensemencer une seule colonie de la plaque dans 5 mL de liquide LB contenant la pression de sélection des antibiotiques (c.-à-d. 60 μg/mL kanamycine). Cultiver la culture liquide pendant une nuit à 37 ° C sous agitation constante à 37 ° C.
- Isoler et purifier le plasmide propagé par les cellules d’e. coli à l’aide d’un kit disponible dans le commerce basé sur la procédure de lysis alkalin 45.
- Confirmer la mutation d’intérêt est présent et dans le cadre de lecture adéquat par séquençage de l’ADN Sanger du plasmide 46.
2. Uniforme de 15 expression de la protéine N-étiquetés dans BL21 ΔE3 Escherichia coli.
Remarque : différentes protéines recombinantes exigent des conditions d’expression différente. Voici la procédure optimisée pour l’expression de la protéine humaine de STIM1 EFSAM.
- Transformer le vecteur d’expression contenant les mutations Cys (c.-à-d. pET-28 a-EFSAM) en cellules compétentes ΔE3 choc codon (+) BL21 thermiques et plaque d’identification sur LB-géloses contenant la pression de sélection antibiotique comme décrit aux étapes 1,9-1.14) avec la après modifications : plaque directement une partie aliquote de 150 μL de la suspension cellulaire totale de ~ 1 000 μL sur la gélose LB sans la nécessité de concentrer les cellules dans un tube de microcentrifuge par centrifugation.
- Le lendemain, transvaser aseptiquement une seule colonie dans un erlenmeyer contenant 20 mL de LB additionné de l’antibiotique approprié (c.-à-d. 60 de kanamycine μg/mL pour animal de compagnie-28 a-EFSAM) de 200 mL. Se développer cette culture starter liquide pendant la nuit (soit environ 16 h) à 37 ° C sous agitation constante à ~ 190 tr/min.
- Le même jour que l’étape 2.2, préparer M9 moyenne pour 15 expression de la protéine N-étiqueté à l’autoclave 1 L de M9 tampon sels (42 mM Na 2 HPO 4, 22 mM KH 2 PO 4, 8,6 mM NaCl, pH 7,4) dans une fiole d’Erlenmeyer de 4 L. Une fois refroidi, filtrer un mélange de 20 % (p/v) de D-glucose, 1 M CaCl 2, thiamine 1 M, 1 M MgSO 4, biotine 1 mg/mL et 0,2 g/mL 15 N-NH 4 Cl à travers un filtre de seringue stérile 0,2 μm dans la solution saline du M9 stérile 1 L pour que la la concentration finale de ces composants est de 0,2 % (p/v) de D-glucose, 100 μM CaCl 2, 50 thiamine μM, 1 mM MgSO 4, 1 biotine μg/mL et 1 mg/mL 15 N-NH 4 CL.
- Le lendemain, transférer stérilement la culture 20 mL de liquide démarreur pendant la nuit dans un tube conique stérile de 50 mL et centrifuger à 2 400 × g pendant 15 min pour les cellules de granuler.
- Après décantation le milieu LB, resuspendre le culot cellulaire résultant dans 10 mL de milieu minimal M9 et transvaser le mélange de l’extrait concentré dans 1 L de milieu minimal M9 avec antibiotique (c.-à-d. 60 kanamycine de μg/mL).
- Croître le 1 L de milieu minimal M9 contenant la culture bactérienne démarreur à 37 ° C et constante ~ 190 tr/mn, secouant jusqu'à ce que la densité optique à 600 nm (DO600) atteint ~0.6-0,8.
- Lorsque la gamme de specfified DO600 est atteinte, ajouter 200 μM d’isopropyl β-D-1-thiogalactopyranoside (IPTG) pour induire l’expression de la protéine.
- Après l’addition de l’IPTG, continuer en incubant les cellules pour l’expression de la protéine à température ambiante en agitant constamment à ~ 190 tr/min pendant environ 16 h (c'est-à-dire la nuit).
- Le lendemain, récolter les bactéries par centrifugation à ~ 10 000 × g, 4 ° C pendant 30 min.
- Décanter la LB et transférer le culot cellulaire dans un tube conique de 50 mL. Stocker les granulés à-80 ° C jusqu'à purification.
3. Purification de protéine recombinante d’e. coli.
Remarque : différentes protéines recombinantes nécessitent des procédures de purification distinct. Voici le protocole pour 6 & #215; Purification de EFSAM son étiquette de corps d’inclusion exprimée à partir de la construction de l’animal de compagnie-28 a.
- Homogénéiser manuellement le culot bactérien congelés en guanidine-HCl 6 M, 20 mM Tris-HCl (pH 8) et β-mercaptoéthanol de 5 mM à l’aide d’une pipette de transfert motorisé 10 mL. Ajouter environ 40 mL de guanidine-HCl par 5 mL de pellet hydro-électriques pour cette étape. Incubation
- suivant un 90 min à température ambiante avec une rotation constante dans un four à hybridation, centrifuger le mélange à ~ 15 000 × g, 8 ° C pendant 40 min séparer les débris cellulaires insolubles (c.-à-d. pellet) de la protéine soluble mélange (c.-à-d. surnageant).
- Ajouter 750 µL de 50 % (v/v) Ni 2 +-agarose acide nitrilotriacétique lisier à la clarification de lysat de billes et incuber pendant 90 min à température ambiante avec inversion dans un four à hybridation.
- Capture par la suite, la protéine de His-tag 6 × liée à la Ni 2 + en recueillant les perles d’agarose dans une colonne de purification de protéine de flux gravité. Permettre le lysat de circuler à travers la colonne complètement avant de passer à l’étape 3.5.
- Laver les perles collectées trois fois avec 10 mL d’urée 6 M, 20 mM Tris-HCl pH 8 et 5 mM β-mercaptoéthanol. S’assurer que l’ensemble 10 mL traverse la colonne avant chaque 10 mL suivante Wash.
- Éluer les protéines dans une série de fractions de 2 mL à l’aide de 6 M urée, 20 mM Tris-HCl pH 8, imidazole de 300 mM et 5 mM β-mercaptoéthanol avec un temps d’incubation s 90 entre les fractions. S’assurer que l’ensemble 2 mL traverse la colonne avant chaque étape ultérieure élution.
- à ce stade, s’assurer de la protéine d’intérêt dans les fractions éluées par Coomassie teinté bleu sodium dodécyl sulfate sur gel de polyacrylamide (SDS-PAGE) en utilisant la méthode de Laemmli 47. Évaluer la taille de la protéine, la quantité et la pureté par comparaison contre des bandes de marqueur de poids moléculaire standard qui sont à la fois inférieur à et supérieur au poids moléculaire attendu de la protéine d’intérêt.
- Centraliser les fractions éluées protéines dans une membrane de dialyse dont le poids moléculaire Da 3 500 coupure et incuber dans un tampon de repliement 1 L (20 mM Tris, 300 mM NaCl, 1 millimètre DTT, 5 mM CaCl 2, pH 8) à 4 ° C durant la nuit tandis que la mémoire tampon est actuellement agité par un magneti agitateur c.
- Après environ 16 h de temps de repliement, ajouter 1 ~ U de la thrombine par mg de protéine directement dans le sac de dialyse et incuber à 4 ° C pour une supplémentaire ~ 24 h.
- Vérifier l’étendue de la 6 × son clivage tag par coloration au bleu de Coomassie de ~ 15 μL protéine prélèvements du sac de dialyse avant et après incubation avec la thrombine qui sont plusieurs à des gels de polyacrylamide (SDS-PAGE) à l’aide de dénaturation la méthode de Laemmli 47. Si un décalage de 2 ~ kDa dans la migration est observé correspondant au poids moléculaire du × 6 clivées sa balise, passez à l’étape 3.11 ; Si une fraction des restes non digérés de protéine qui est détectable par coloration au bleu de Coomassie, versez ~0.2 U de la thrombine par mg de protéine directement dans le sac de dialyse et incuber à 4 ° C pendant une h ~ 24 supplémentaires
- Chromatographie utilisation exclusion ou échange d’ions pour purifier davantage la protéine. Pour chromatographie échangeuse d’anion de EFSAM, retirer la solution protéique du sac de dialyse et concentré de ~ 10 fois en utilisant un concentrateur centrifuge de l’ultrafiltration avec 10 000, un poids moléculaire de Da coupure. Par la suite, re-diluer la solution ~ 20 fois dans un tampon sans NaCl (20 mM Tris, 5 mM CaCl 2, 1 mM DTT, pH 8).
- Équilibrer une colonne échangeuse d’anion préemballés avec 10 volumes de colonne du tampon sans NaCl décrit à l’étape 3.11. Equilibrer utilisant le tampon chargé dans une seringue luer lock ne contenant aucune bulle d’air en appuyant sur la solution à travers la colonne de manière goutte à goutte et en évitant les pressions seringue qui provoquent un flux constant de solution de sortie de la colonne. Utiliser un échangeur d’anions forts (par exemple d’agarose réticulé avec groupes fonctionnels d’ammonium quaternaire).
- Charger la solution protéique diluée dans le tampon sans NaCl (étape 3.11) sur la colonne comme indiqué au point 3.12.
- Éluer les protéines dans un dégradé [c'est-à-dire 0 - 60 % (v/v)] de NaCl de plus en plus de la mémoire tampon (20 mM Tris, 1 M NaCl, 5 mM CaCl 2, 1 mM DTT, pH 8) à l’aide d’un système de chromatographie en phase liquide (FPLC) protéine rapide de deux pompes. Régler le système FPLC pour collecter des fractions de mL ~1-1.5 et surveiller le profil d’élution de protéine à l’aide d’absorbance de UV 280 nm et un débit de 0,5 mL/min.
- Identifier les pics d’élution et fractions contenant la protéine d’intérêt ainsi que les gels de pureté de la protéine par teinté bleu de Coomassie SDS-PAGE à l’aide de la méthode de Laemmli 47.
- Fractions de piscine montrant > 95 % (soit pris sous forme de fractions qui ne montrent qu’une seule bande protéique sur la coloration des gels de bleu de Coomassie) dans un sac de dialyse et de la conversion en tampon expérimentale d’intérêt par la dialyse comme indiqué au point 3.8.
4. Fixation chimique du glucose-5-MTS à la protéine par dialyse.
Solution mère- Prepare un 55 mM de N-(β-D-glucopyranosyl)-N '-[2-methanethiosulfonyl) éthyl] urée (glucose-5-MTS) dissoudre 10 mg du composé dans 500 μl de 100 % (v/v) de DMSO. Stocker le glucose-5-MTS inutilisés solubilisées dans le DMSO à -20 ° C.
- Préparer l’échantillon de protéine pour modification par dialyse 1,5 mL de ~ 60 μM protéine dans 1 L de modification tampon composé de 20 mM MOPS, NaCl 150 mM, 5 mM CaCl 2 et 0,1 mM TCEP, pH 8,3. Utiliser un seuil de poids moléculaire de membrane de dialyse qui est plus petit que la taille de la protéine en cours de modification (par exemple utiliser une fréquence de coupure 3 500 Da pour le ~ 17 500 Da EFSAM).
- Après 24 h à 4 ° C, transférer l’échantillon dans le sac de dialyse dans un tube de microcentrifuge. Ajouter le DMSO solubilisée glucose-5-MTS à une concentration finale de 2 mM.
- Incuber l’échantillon dans l’obscurité pendant 1 h à température ambiante. Au cours de la période d’incubation de 1 h, mélanger la solution par le tapement doux du tube chaque 10 min.
- Re-échanger par la suite, la protéine dans le dernier tampon expérimentaux ne contenant aucun agent réducteur en dialyse à 4 ° C, tel que décrit à l’étape 4.2 ou par ultrafiltration centrifuge. Pour la procédure de l’ultrafiltration, concentrer l’échantillon de protéine ~1.5 mL à < 0,5 mL et diluer par la suite dans le concentrateur même avec le tampon expérimental. Répétez cette étape de dilution concentration deux fois supplémentaires afin que l’échange total est d’au moins 30 × 30 × 30 = 27,000-fold. Pour EFSAM, utiliser 20 mM Tris, 150 mM NaCl, 5 mM CaCl 2, pH 7.5 comme tampon expérimentale.
- Préparation des échantillons pour la spectrométrie de masse à ionisation par électronébulisation par dialyse ou ultrafiltration exchange comme décrit aux points 4.2 et 4.5, respectivement, en 25 mM d’ammonium bicarbonate ou acétate d’ammonium 25 mM. Si à l’aide de dialyse, assurez-vous que vous l’échanger au moins trois fois pour enlever les sels résiduels de 2 NaCl et ACIC.
- Déterminer la masse exacte (i.e. ± 1 Da) de la protéine d’intérêt à l’aide d’ionisation par électronébulisation mass spectrometry 48 , 49. Attendre chaque addition covalente de glucose à un thiol Cys, par l’intermédiaire de produit chimique méthanethiosulfonate pour ajouter 281,3 Da à la protéine de masse (c'est-à-dire ajouter 360.4 Da pour le glucose-5-MTS et soustraire 79,1 Da pour le CH 3 donc 2 laissant groupe pendant covalente pièce jointe).
5. Évaluation de la solution NMR de modifdes perturbations structurelles et efficacité ication.
- S’assurer que la concentration de la mis à jour le protéine est > 100 μM après la fixation du glucose et l’échange tampon final. Pour EFSAM, estimer la concentration de protéines à l’aide d’un coefficient d’extinction UV à 280 nm de 1,54 (mg mL – 1) cm – 1.
- Supplément la solution protéique avec 60 μM d’acide 4,4-dimethyl-4-silapentane-1-sulfonic (DSS) pour le calage et l’impulsion d’étalonnage et de 10 % (v/v) D 2 O le verrou de signal. Pour utilisation de signal-bruit élevé de 600 échantillons μL dans des tubes de même fréquence 5 mm NMR, inséré dans un spectromètre de 600 MHz minimum, équipé d’une sonde à triple résonance HCN cryogénique.
- Recueillir les spectres de HSQC N standard 1 H - 15 comme précédemment détaillées 50 , 51 à température, 1 H et largeurs de balayage 15 N, transitoire et est incrémenté paramètres appropriés pour l’échantillon particulier. Pour les spectres EFSAM, utilisation de 20 ° C, 256 1 H transitoires, 64 incréments de dimension 15 N et 1 H et 15 N sweep largeurs fixés respectivement à 8 000 et 1 800 Hz.
- Suite à l’acquisition de la gamme de protéines glycosylées, ajouter le dithiothréitol (DTT) à l’échantillon de NMR provenant d’un stock de 1 M à une concentration finale de 15 mM. La TNT supprime la portion de glucose de la protéine par réduction de l’accessoire induite par le disulfure.
- Acquérir un deuxième 1 H - 15 N HSQC dans ces conditions réduit/non modifié, offrant un éventail de référence afin d’évaluer les perturbations structurelles et de l’efficacité de modification provoquées par l’attachement de glucose.
- Traiter les données de la RMN à l’aide de NMRPipe tel qu’indiqué précédemment 52. S’assurer que le traitement minimalement comprend conversion de données, progressive, solvant suppression, transformation de Fourier et initiale visualisation des spectres.
- Evaluer l’efficacité modification en mesurant les intensités maximales amide et les valeurs des déplacements chimiques dans les spectres réduites et modifiés en utilisant le plugin NEASY sur CARA 53. S’assurer pour évaluer l’intensité du pic de l’amide de Cys tant dans le glucose attaché et réduit les spectres. Si l’amide Cys ne peut pas être fiable situé dans les deux spectres, utiliser les intensités des résidus adjacents à la Cys comme une lecture.
- Calculer le rendement comme l’intensité de l’amide du spectre Cys modifiés divisée par l’intensité de l’amide de la Cys-réduit (c'est-à-dire traités DTT) spectrum, multiplié par 100 :
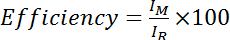 , où j’ai M est l’intensité de l’amide dans le spectre de Cys modifiés et j’ai R est l’intensité de l’amide dans le spectre de Cys-réduit. Vous pouvez également évaluer l’efficacité moyenne sur plusieurs sommets amide :
, où j’ai M est l’intensité de l’amide dans le spectre de Cys modifiés et j’ai R est l’intensité de l’amide dans le spectre de Cys-réduit. Vous pouvez également évaluer l’efficacité moyenne sur plusieurs sommets amide :
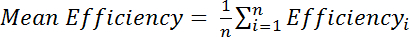 , où efficacité i est le rendement déterminé séparément calculé pour chacun résidus, i et n est le nombre total des résidus utilisés dans le calcul de.
, où efficacité i est le rendement déterminé séparément calculé pour chacun résidus, i et n est le nombre total des résidus utilisés dans le calcul de. - Calcul des perturbations de déplacement chimique (CSP) de la différence de déplacement chimique entre les deux spectres observés dans les 15 N et 1 H dimensions de chaque pic et normalisation pour la plus grande et 15 N déplacement chimique utilisée par les suivant l’équation :
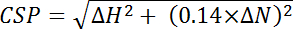 , où ΔH est le changement de ppm dans la dimension de proton et ΔN est le changement de ppm dans la dimension de l’azote.
, où ΔH est le changement de ppm dans la dimension de proton et ΔN est le changement de ppm dans la dimension de l’azote.
Résultats
La première étape de cette approche nécessite la mutagénèse des résidus de glycosylation candidat aux résidus Cys qui peuvent être modifiables à l’aide de la EFSAM de glucose-5-MTS. n’a aucun résidu Cys endogènes, donc aucune des considérations particulières ne doivent être apportées avant la mutagenèse. Toutefois, les résidus Cys natives doivent être mutés aux résidus non modifiable avant d’effectuer la chimie décrite. Pour au minimum réaliser la structure nat...
Discussion
Glycosylation des protéines est une modification post-traductionnelle où les sucres sont covalente aux polypeptides principalement par le biais de liens avec des chaînes latérales d’acides aminés. Jusqu'à 50 % de protéines de mammifères sont glycosylés 54, où les protéines glycosylées peuvent avoir par la suite une gamme variée d’effets de dérégler l’affinité de liaison biomoléculaire, influençant la protéine pliage, altérant l’activité du canal, ciblage molécules de ...
Remerciements
Cette recherche a été financée par les Sciences naturelles et l’ingénierie Conseil de recherches du Canada (05239 à PBS), la Fondation canadienne pour l’Innovation de l’Ontario Research Fund (à PBS), la Prostate Cancer Foundation Fight - Telus Ride for Dad (à PBS) et de l’Ontario Bourses d’études supérieures (à Y.J.C. et n.-é).
matériels
| Name | Company | Catalog Number | Comments |
| Phusion DNA Polymerase | Thermo Fisher Scientific | F530S | Use in step 1.3. |
| Generuler 1kb DNA Ladder | Thermo Fisher Scientific | FERSM1163 | Use in step 1.6. |
| DpnI Restriction Enzyme | New England Biolabs, Inc. | R0176 | Use in step 1.8. |
| Presto Mini Plasmid Kit | GeneAid, Inc. | PDH300 | Use in step 1.16. |
| BL21 DE3 codon (+) E. coli | Agilent Technologies, Inc. | 230280 | Use in step 2.1. |
| DH5a E. coli | Invitrogen, Inc. | 18265017 | Use in step 1.9. |
| 0.22 mm Syringe Filter | Millipore, Inc. | SLGV033RS | Use in step 2.3. |
| HisPur Ni2+-NTA Agarose Resin | Thermo Fisher Scientific | 88221 | Use in step 3.3. |
| 3,500 Da MWCO Dialysis Tubing | BioDesign, Inc. | D306 | Use in step 3.8, 3.16, 4.2, 4.5 and 4.6. |
| Bovine Thrombin | BioPharm Laboratories, Inc. | SKU91-055 | Use in step 3.9. |
| 5 mL HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5156-01 | Use in step 3.11. |
| Glucose-5-MTS | Toronto Research Chemicals, Inc. | G441000 | Use in step 4.1. |
| Vivaspin 20 Ultrafiltration Centrifugal Concentrators | Sartorius, Inc. | VS2001 | Use in step 3.11, 4.2, 4.5 and 4.6. |
| PageRuler Unstained Broad Protein Ladder | Thermo Fisher Scientific | 26630 | Use in step 3.7, 3.10 and 3.15 |
| HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5053-01 | Use in step 3.12. |
| AKTA Pure Fast Protein Liquid Chromatrography System | GE Healthcare, Inc. | 29018224 | Use in step 3.14. |
| 600 MHz Varian Inova NMR Spectrometer | Agilent Technologies, Inc. | Use in step 5.2 and 5.5. |
Références
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Feske, S., Skolnik, E. Y., Prakriya, M. Ion channels and transporters in lymphocyte function and immunity. Nat Rev Immunol. 12 (7), 532-547 (2012).
- Shaw, P. J., Feske, S. Physiological and pathophysiological functions of SOCE in the immune system. Front Biosci (Elite Ed). 4, 2253-2268 (2012).
- Seo, M. D., Enomoto, M., Ishiyama, N., Stathopulos, P. B., Ikura, M. Structural insights into endoplasmic reticulum stored calcium regulation by inositol 1,4,5-trisphosphate and ryanodine receptors. Biochim Biophys Acta. 1853 (9), 1980-1991 (2015).
- Stathopulos, P. B., Ikura, M. Structural aspects of calcium-release activated calcium channel function. Channels (Austin). 7 (5), 344-353 (2013).
- Stathopulos, P. B., Ikura, M. Structure and function of endoplasmic reticulum STIM calcium sensors. Curr Top Membr. 71, 59-93 (2013).
- Stathopulos, P. B., Li, G. Y., Plevin, M. J., Ames, J. B., Ikura, M. Stored Ca2+ depletion-induced oligomerization of stromal interaction molecule 1 (STIM1) via the EF-SAM region: An initiation mechanism for capacitive Ca2+ entry. J Biol Chem. 281 (47), 35855-35862 (2006).
- Stathopulos, P. B., Ikura, M. Store operated calcium entry: From concept to structural mechanisms. Cell Calcium. , (2016).
- Stathopulos, P. B., Ikura, M. Structurally delineating stromal interaction molecules as the endoplasmic reticulum calcium sensors and regulators of calcium release-activated calcium entry. Immunol Rev. 231 (1), 113-131 (2009).
- Muik, M., et al. STIM1 couples to ORAI1 via an intramolecular transition into an extended conformation. EMBO J. 30 (9), 1678-1689 (2011).
- Luik, R. M., Wang, B., Prakriya, M., Wu, M. M., Lewis, R. S. Oligomerization of STIM1 couples ER calcium depletion to CRAC channel activation. Nature. 454 (7203), 538-542 (2008).
- Luik, R. M., Wu, M. M., Buchanan, J., Lewis, R. S. The elementary unit of store-operated Ca2+ entry: local activation of CRAC channels by STIM1 at ER-plasma membrane junctions. J Cell Biol. 174 (6), 815-825 (2006).
- Wu, M. M., Buchanan, J., Luik, R. M., Lewis, R. S. Ca2+ store depletion causes STIM1 to accumulate in ER regions closely associated with the plasma membrane. J Cell Biol. 174 (6), 803-813 (2006).
- Liou, J., Fivaz, M., Inoue, T., Meyer, T. Live-cell imaging reveals sequential oligomerization and local plasma membrane targeting of stromal interaction molecule 1 after Ca2+ store depletion. Proc Natl Acad Sci U S A. 104 (22), 9301-9306 (2007).
- Calloway, N., et al. Stimulated association of STIM1 and Orai1 is regulated by the balance of PtdIns(4,5)P(2) between distinct membrane pools. J Cell Sci. 124 (Pt 15), 2602-2610 (2011).
- Korzeniowski, M. K., et al. Dependence of STIM1/Orai1-mediated calcium entry on plasma membrane phosphoinositides. J Biol Chem. 284 (31), 21027-21035 (2009).
- Park, C. Y., et al. STIM1 clusters and activates CRAC channels via direct binding of a cytosolic domain to Orai1. Cell. 136 (5), 876-890 (2009).
- Yuan, J. P., et al. SOAR and the polybasic STIM1 domains gate and regulate Orai channels. Nat Cell Biol. 11 (3), 337-343 (2009).
- Feske, S., et al. A mutation in Orai1 causes immune deficiency by abrogating CRAC channel function. Nature. 441 (7090), 179-185 (2006).
- Prakriya, M., et al. Orai1 is an essential pore subunit of the CRAC channel. Nature. 443 (7108), 230-233 (2006).
- Vig, M., et al. CRACM1 multimers form the ion-selective pore of the CRAC channel. Curr Biol. 16 (20), 2073-2079 (2006).
- Vig, M., et al. CRACM1 is a plasma membrane protein essential for store-operated Ca2+ entry. Science. 312 (5777), 1220-1223 (2006).
- Liou, J., et al. STIM is a Ca2+ sensor essential for Ca2+-store-depletion-triggered Ca2+ influx. Curr Biol. 15 (13), 1235-1241 (2005).
- Roos, J., et al. STIM1, an essential and conserved component of store-operated Ca2+ channel function. J Cell Biol. 169 (3), 435-445 (2005).
- Putney, J. W. A model for receptor-regulated calcium entry. Cell Calcium. 7 (1), 1-12 (1986).
- Feske, S. CRAC channelopathies. Pflugers Arch. 460 (2), 417-435 (2010).
- Maus, M., et al. Missense mutation in immunodeficient patients shows the multifunctional roles of coiled-coil domain 3 (CC3) in STIM1 activation. Proc Natl Acad Sci U S A. 112 (19), 6206-6211 (2015).
- Stathopulos, P. B., Zheng, L., Li, G. Y., Plevin, M. J., Ikura, M. Structural and mechanistic insights into STIM1-mediated initiation of store-operated calcium entry. Cell. 135 (1), 110-122 (2008).
- Stathopulos, P. B., Ikura, M. Partial unfolding and oligomerization of stromal interaction molecules as an initiation mechanism of store operated calcium entry. Biochem Cell Biol. 88 (2), 175-183 (2010).
- Dennis, J. W., Lau, K. S., Demetriou, M., Nabi, I. R. Adaptive regulation at the cell surface by N-glycosylation. Traffic. 10 (11), 1569-1578 (2009).
- Nilsson, T., Au, C. E., Bergeron, J. J. Sorting out glycosylation enzymes in the Golgi apparatus. FEBS Lett. 583 (23), 3764-3769 (2009).
- Stanley, P. Golgi glycosylation. Cold Spring Harb Perspect Biol. 3 (4), (2011).
- Moremen, K. W., Tiemeyer, M., Nairn, A. V. Vertebrate protein glycosylation: diversity, synthesis and function. Nat Rev Mol Cell Biol. 13 (7), 448-462 (2012).
- Gerlach, J., Sharma, S., Leister, K., Joshi, L., Agostinis, P., Afshin, S. . Endoplasmic Reticulum Stress in Health and Disease. , 23-39 (2012).
- Pearse, B. R., Hebert, D. N. Lectin chaperones help direct the maturation of glycoproteins in the endoplasmic reticulum. Biochim Biophys Acta. 1803 (6), 684-693 (2010).
- Stanley, P., Sundaram, S. Rapid assays for lectin toxicity and binding changes that reflect altered glycosylation in mammalian cells. Curr Protoc Chem Biol. 6 (2), 117-133 (2014).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Csutora, P., et al. Novel role for STIM1 as a trigger for calcium influx factor production. J Biol Chem. 283 (21), 14524-14531 (2008).
- Kilch, T., et al. Mutations of the Ca2+-sensing stromal interaction molecule STIM1 regulate Ca2+ influx by altered oligomerization of STIM1 and by destabilization of the Ca2+ channel Orai1. J Biol Chem. 288 (3), 1653-1664 (2013).
- Williams, R. T., et al. Stromal interaction molecule 1 (STIM1), a transmembrane protein with growth suppressor activity, contains an extracellular SAM domain modified by N-linked glycosylation. Biochim Biophys Acta. 1596 (1), 131-137 (2002).
- Mignen, O., Thompson, J. L., Shuttleworth, T. J. STIM1 regulates Ca2+ entry via arachidonate-regulated Ca2+-selective (ARC) channels without store depletion or translocation to the plasma membrane. J Physiol. 579 (Pt 3), 703-715 (2007).
- Choi, Y. J., Zhao, Y., Bhattacharya, M., Stathopulos, P. B. Structural perturbations induced by Asn131 and Asn171 glycosylation converge within the EFSAM core and enhance stromal interaction molecule-1 mediated store operated calcium entry. Biochim Biophys Acta. 1864 (6), 1054-1063 (2017).
- Davis, B. G., Lloyd, R. C., Jones, J. B. Controlled site-selective protein glycosylation for precise glycan structure-catalytic activity relationships. Bioorg Med Chem. 8 (7), 1527-1535 (2000).
- Gamblin, D. P., van Kasteren, S. I., Chalker, J. M., Davis, B. G. Chemical approaches to mapping the function of post-translational modifications. FEBS J. 275 (9), 1949-1959 (2008).
- Ehrt, S., Schnappinger, D. Isolation of plasmids from E. coli by alkaline lysis. Methods Mol Biol. 235, 75-78 (2003).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 94 (3), 441-448 (1975).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bell, D. J. Mass spectrometry. Methods Mol Biol. 244, 447-454 (2004).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312 (5771), 212-217 (2006).
- Farrow, N. A., et al. Backbone Dynamics of a Free and a Phosphopeptide-Complexed Src Homology-2 Domain Studied by N-15 Nmr Relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Kay, L. E., Keifer, P., Saarinen, T. Pure Absorption Gradient Enhanced Heteronuclear Single Quantum Correlation Spectroscopy with Improved Sensitivity. Journal of the American Chemical Society. 114 (26), 10663-10665 (1992).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Masse, J. E., Keller, R. AutoLink: automated sequential resonance assignment of biopolymers from NMR data by relative-hypothesis-prioritization-based simulated logic. J Magn Reson. 174 (1), 133-151 (2005).
- Monticelli, M., Ferro, T., Jaeken, J., Dos Reis Ferreira, V., Videira, P. A. Immunological aspects of congenital disorders of glycosylation (CDG): a review. J Inherit Metab Dis. 39 (6), 765-780 (2016).
- An, H. J., Kronewitter, S. R., de Leoz, M. L., Lebrilla, C. B. Glycomics and disease markers. Curr Opin Chem Biol. 13 (5-6), 601-607 (2009).
- Wani, W. Y., Chatham, J. C., Darley-Usmar, V., McMahon, L. L., Zhang, J. O-GlcNAcylation and neurodegeneration. Brain Res Bull. , (2016).
- Haines, A. M., Tobe, S. S., Kobus, H. J., Linacre, A. Properties of nucleic acid staining dyes used in gel electrophoresis. Electrophoresis. 36 (6), 941-944 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon