È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Targeting dei tioli cisteina per in Vitro site-specific glicosilazione delle proteine ricombinanti
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Le analisi biochimiche e strutturali delle proteine glicosilate richiedono relativamente grandi quantità di campioni omogenei. Qui, presentiamo un efficiente metodo chimico per site-specific glicosilazione delle proteine ricombinanti purificate da batteri prendendo di mira i tioli reattivi di Cys.
Abstract
Stromal interazione molecola-1 (STIM1) è un tipo-io la proteina del transmembrane situato sul reticolo endoplasmatico (ER) e membrane plasmatiche (PM). ER-residente STIM1 regola l'attività dei canali PM Orai1 in un processo noto come memorizzare voce azionata calcio (Ca2 +) che è il principale Ca2 + segnalazione processo che spinge il sistema immunitario. STIM1 subisce post-traduzionali N- glicosilazione in due siti di Asn luminal all'interno del Ca2 + rilevamento dominio della molecola. Tuttavia, la biochimica, biofisica, e gli effetti biologici di struttura di N- glicosilata STIM1 erano capiti male fino a recentemente dovuto un'incapacità di facilmente ottenere alti livelli di omogeneo N- glicosilata proteina. Qui, descriviamo l'implementazione di un in vitro approccio chimico che attribuisce moiety glucosio ai siti di proteina specifica applicabili alla comprensione degli effetti sottostanti di N- glicosilazione il meccanismo e la struttura della proteina. Usando la spettroscopia a risonanza magnetica nucleare soluzione valutiamo sia l'efficienza della modificazione nonché le conseguenze strutturali del collegamento del glucosio con un singolo campione. Questo approccio può essere adattato facilmente per studiare le proteine glicosilate una miriade che si trovano in natura.
Introduzione
Negozio gestito calcio (Ca2 +) voce (SOCE) è la via principale, in cui le cellule immunitarie prendono il Ca2 + dallo spazio extracellulare nel cytosol. Nei linfociti T, cellule T recettori situati sulla membrana plasmatica (PM) legano gli antigeni che attivano proteine tirosin chinasi (rivisto in 1,2,3). Una cascata di fosforilazione conduce all'attivazione di fosfolipasi-γ (PLCγ), che successivamente ha trasmesso l'idrolisi di membrana fosfatidilinositolo 4,5-bisfosfato (PIP2) in diacilglicerolo e inositolo 1,4,5-trisfosfato (IP3 ). IP3 è un piccolo messaggero diffusibile che si lega ai IP3 recettori (IP3R) il reticolo endoplasmatico (ER) quindi aprire questo canale del ricevitore e permettendo Ca2 + a fluire verso il basso la pendenza di concentrazione dall'ER lumen al cytosol (rivisto in 4). Segnalazione da accoppiati a proteine G e recettori tirosina-chinasi in una varietà di altre cellule eccitabili e non eccitabili tipi piombo per la produzione stessa di IP3 e l'attivazione di IP3Rs del ricevitore.
A causa del Ca2 + deposito capacità limitata dell'ER, IP3-rilascio mediato e aumento risultante in Ca citosolico2 + è solo temporaneo; Tuttavia, questo svuotamento del ER luminal Ca2 + effetti profondamente stromal interazione molecola-1 (STIM1), un tipo-io transmembrana (TM) proteina trovata principalmente sul ER membrana 5,6,7. STIM1 contiene un lume-oriented Ca2 + rilevamento dominio costituito da una coppia di EF-hand e sterile di α-motivo (EFSAM). Tre citosolico orientato arrotolato-arrotola domini sono separati da EFSAM dal singolo dominio TM (rivisto in 8). Al momento di svuotamento2 + ER luminal Ca, EFSAM subisce una destabilizzazione-accoppiato oligomerizzazione 7,9 che provoca riarrangiamenti strutturali della TM e arrotolato-arrotola domini 10. Questi cambiamenti strutturali culminano in un intrappolamento di STIM1 ER-PM giunzioni 11,12,13,14 attraverso interazioni con PM phosphoinositides 15, 16 e Orai1 subunità 17,18. Orai1 proteine sono le subunità di PM che assemblano per formare Ca2 + canali 19,20,21,22. Facilitano le interazioni di STIM1-Orai1 alle giunzioni di ER-PM una Ca2 + sblocco attivato Ca2 + (CRAC) canale conformazione aperta che consente lo spostamento di Ca2 + nel citosol da alte concentrazioni della spazio extracellulare. In cellule del sistema immunitario, il citosolico Ca2 + elevazioni continue attraverso canali di CRAC inducono il Ca2 +- calmodulina/calcineurina dipendente defosforilazione del fattore nucleare delle cellule di T attivate che successivamente entra nel nucleo e inizia la regolazione trascrizionale di geni promuovendo l'attivazione delle cellule T 1,3. Il processo di attivazione del canale CRAC di STIM1 23,24 via indotta da agonisti ER luminal Ca2 + lo svuotamento e la risultante citosolico Ca2 + elevazione continua è collettivamente denominato SOCE 25. Il ruolo vitale dei SOCE in cellule di T è evidente da studi che dimostrano che le mutazioni ereditabili in Orai1 e STIM1 possono causare immunodeficienza combinata grave sindromi 3,19,26, 27. EFSAM avvia SOCE dopo rilevamento luminal ER Ca2 + svuotamento tramite la perdita di Ca2 + coordinamento presso la canonica EF-hand, infine conducendo alla destabilizzazione-accoppiato autoassociazione 7, 28,29.
La glicosilazione è il legame covalente ed elaborazione di strutture oligosaccaride, noto anche come glicani, attraverso vari passaggi biosintetici in ER e Golgi (rivisto in 30,32,33). Ci sono due tipi predominanti di glicosilazione negli eucarioti: N-collegati e O-collegato, a seconda delle specifiche dell'aminoacido e atomo colmare il sollevatore. In N- glicosilazione, glicani sono associati l'ammide di catena laterale di Asn, e nella maggior parte dei casi, il passo di iniziazione si verifica nell'ER come polipeptide catena si muove nel lume 34. Il primo passo di N- glicosilazione è il trasferimento di una struttura di nucleo di quattordici-zucchero composto da glucosio (cromatografia gaseoliquido), mannosio (uomo) e N- acetilglucosamina (GlcNAc) (cioè Glc3Man9GlcNAc2) da un ER dei lipidi di membrana da un oligosaccharyltransferase 35,36. Ulteriori passi, come la scissione o il trasferimento di residui di glucosio, vengono catalizzate al pronto soccorso di glicosiltrasferasi e specifiche glicosidasi. Alcune proteine che lasciano l'ER e spostare nel Golgi possono essere ulteriormente trasformati 37. O- glycosylation si riferisce all'aggiunta di glicani, solitamente per il gruppo dell'idrossile di catena laterale dei residui Ser o Thr, e questa modificazione avviene interamente nel complesso di Golgi 33,34. Ci sono diverse strutture di - glycan Oche possono essere costituiti da N- acetilglucosamina, fucosio, galattosio e acido sialico con ogni monosaccaride aggiunto in sequenza 33.
Mentre nessuna sequenza specifica è stata identificata come prerequisito per molti tipi di O- glicosilazione, una sequenza di consenso comune è stata associata con la N-collegato modifica: Asn-X-Ser/Thr/Cys, dove X può essere qualsiasi amminoacido tranne Pro 33. STIM1 EFSAM contiene due di questi siti di consenso N- glicosilazione: Asn131-Trp132-Thr133 e Asn171-Thr172-Thr173. Infatti, studi precedenti hanno dimostrato che la EFSAM può essere N- glicosilata in cellule di mammifero in Asn131 e Asn171 38,39,40,41. Tuttavia, gli studi precedenti delle conseguenze di N- glicosilazione su SOCE sono state incongruenti, suggerendo soppresso, ha rafforzato o alcun effetto di questa modifica alberino-translational su SOCE attivazione 38,= "xrif" > 39,40,41. Così, la ricerca sulle conseguenze biofisiche, biochimiche e strutturali sottostanti di EFSAM N- glicosilazione è fondamentale per comprendere gli effetti normativi di questa modifica. A causa del requisito per alti livelli di proteine omogenei in questi esperimenti in vitro , è stato applicato un approccio selettivo sito cui connettersi covalentemente moiety glucosio EFSAM. Interessante, Asn131 e Asn171 glicosilazione causato cambiamenti strutturali che convergono all'interno del nucleo EFSAM e che permettono di migliorare le proprietà biofisiche che promuovono STIM1-mediata SOCE 42.
L'allegato chimica dei gruppi glicosilici Cys tioli è stata consolidata da un lavoro seminale che in primo luogo ha dimostrato l'utilità di questo enzima-libero approccio alla comprensione degli effetti specifici del sito di glicosilazione su proteina funzione 43 , 44. più recentemente e per quanto riguarda STIM1, i residui Asn131 e Asn171 erano mutati di Cys e glucose-5-(methanethiosulfonate) [glucose-5-(MTS)] è stato usato per covalenza collegarsi glucosio i tioli liberi 42. Qui, Descriviamo questo approccio che non solo utilizza mutagenesi per incorporare i residui di Cys specifici sito per la modifica, ma si applica anche la spettroscopia di risonanza magnetica nucleare (NMR) soluzione per valutare rapidamente strutturale ed efficienza di modifica perturbazioni a causa la glicosilazione. In particolare, questa metodologia generale è facilmente adattabile per studiare gli effetti di entrambi O- o N- glicosilazione di qualsiasi recombinantly prodotto della proteina.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. reazione a catena della polimerasi (PCR)-mediata di mutagenesi sito-diretta per l'incorporazione di Cys in un vettore di espressione batterica pET-28a.
- Determinare la concentrazione del vettore animale-28a (cioè DNA incagliato doppio) utilizzando un coefficiente di estinzione (UV) ultravioletto di 0.020 (μg/mL) cm -1 a 260 nm.
- Sintetizzare un paio degli iniettori mutageni complementari per ogni mutazione Cys tale che io) ci sono un minimo di 15 nucleotidi complementari al modello prima della mancata corrispondenza prima base e 15 nucleotidi complementari per il modello dopo la mancata corrispondenza di base finale, ii) della lunghezza totale dell'iniettore non superi 45 nucleotidi e iii) una guanina o citosina si trova alla posizione del primo e l'ultimo nucleotide di ogni primer (tabella 1). Garantire la sintesi di primer viene eseguita utilizzando una purificazione di scala e cartuccia di 0.025 μmol.
- Utilizzando un'alta fedeltà DNA polimerasi, impostare di miscele di reazione di PCR due 20 µ l: uno contenente il primer in avanti e la seconda contenente il primer reverse. Preparare ogni miscela per contenere le concentrazioni finali di 1 x PCR tampone con 1,5 mM MgCl 2, 0.2 mM dNTPs, 0,5 μM primer, 0,4 μL DMSO, modello di 1,25 ng/μL del DNA, 0.02 U/μL ad alta fedeltà della polimerasi del DNA.
- Ciclo termicamente le miscele separate utilizzando un protocollo di tre fasi: 98 ° C per 30 s (denaturazione), 53-56 ° C per 30 s (ricottura), 72 ° C per 30 s kilobase(kb) -1 (estensione) di DNA della mascherina. Ripetere il programma di temperatura per 5 cicli e aggiungere un passaggio di estensione finale 72 ° C per un minimo di 7,5
- Dopo l'iniziale PCR con primer forward e reverse in provette separate, combinare i prodotti in un unico tubo (cioè 40 μL totale) e proseguire la reazione di PCR per un ulteriore 20 cicli utilizzando gli stessi parametri in bicicletta come descritto al punto 1.4.
- Electrophorese 15 μL di miscela di reazione PCR su un agarosio all'1% (p/v) gel usando 0,5 x Tris, acido acetico, etilene diammina tetra acido acetico (EDTA) (TAE) tampone di corsa. Come controlli, elettroforesi una quantità equivalente di modello di DNA che non è stato amplificato dalla PCR e un'aliquota della scala di riferimento del DNA che contiene le bande dell'indicatore sia maggiore e minore la dimensione prevista del prodotto PCR.
- Dopo l'elettroforesi a 120 V per 40 min, immergere il gel in acqua contenente 0,5 μg/mL di bromuro di etidio e agitare per 30 min a temperatura ambiente. Confermare il modello full-length è stato amplificato dai primer del mutageno come un aumento nell'intensità relativa etidio bromuro fluorescente della band amplificata rispetto alla band di modello di controllo sotto la luce UV (302 nm).
- Se nessuna amplificazione è apparente, ripetere la PCR dopo aver regolato la temperatura di ricottura con incrementi di 0,5 ° C tra la gamma di temperatura di 53-56 ° C.
- Al momento della conferma dell'amplificazione del modello dai primer del mutagene, trattare il restante ~ 25 μL della miscela di reazione PCR con l'enzima di restrizione DpnI per digerire il modello metilato di DNA. Utilizzare DpnI a 0,5 µ l (10 unità) per la miscela di reazione PCR μL 25 e una concentrazione finale di 1 × DpnI tampone di reazione. Incubare per 2,5 h a 37 ° C.
- Dopo digestione modello, aggiungere ~ 5-10 µ l della miscela digerita a 100 µ l di calore shock cellule DH5α Esherichia coli competenti in un tubo del microcentrifuge 1,75 mL. Incubare la miscela di DNA delle cellule sul ghiaccio per 60 min.
- Scossa di calore la miscela di DNA delle cellule nel tubo del microcentrifuge a 42 ° C per 45 s su un blocco a calore secco. Dopo incubazione la miscela sul ghiaccio per 3 min, aggiungere 900 μL di brodo temperatura Luria-Bertani (LB) alle cellule e trasferire la sospensione totale delle cellule in una provetta di turno-fondo sterile mL 14.
- Incubare la sospensione cellulare a 37 ° C per 90 min con agitazione costante a 190 giri/min.
- Successivamente, trasferire la sospensione cellulare nuovamente dentro un tubo del microcentrifuge 1,75 mL e centrifugare a 10.000 x g per 5 min a temperatura ambiente.
- Dopo la centrifugazione, rimuovere 900 μL del surnatante e risospendere le cellule batteriche pipettando delicato nei restanti 100 μL di lb.
- Trasferire la sospensione concentrata di cellule risultante su un piatto di LB-agar che contiene antibiotico che è selettiva per il vettore di espressione (cioè 60 μg/mLKanamycin). Asetticamente diffondere la sospensione uniformemente sulla piastra agar e incubare per ~ 16 h a 37 ° C.
- Il giorno seguente, inoculare una singola Colonia dalla piastra in 5 mL di liquido LB contenente la pressione di selezione antiobiotic (cioè 60 μg/mL kanamicina). Far crescere la cultura liquida durante la notte a 37 ° C con agitazione costante a 37 ° C.
- Isolare e purificare il plasmide propagato dalle cellule e. coli utilizzando un kit disponibile in commercio basato sul Lisi alcalina procedura 45.
- Confermare la mutazione di interesse è presente e nel telaio della lettura corretta mediante sequenziamento Sanger del plasmide 46.
2. Uniforme 15 espressione di proteina N-labeled in BL21 ΔE3 Escherichia coli.
Nota: diverse proteine ricombinanti richiedono condizioni di espressione diversa. Quanto segue è la procedura ottimizzata per l'espressione della proteina umana STIM1 EFSAM.
- Trasformare il vettore di espressione che harboring le mutazioni di Cys (cioè animali-28a-EFSAM) in cellule competenti di BL21 ΔE3 codone (+) calore shock e piastra sulle piastre di LB-agar contenente la pressione di selezione antibiotica come descritto nei passaggi 1,9-1.14) con la successive modifiche: piastra direttamente un'aliquota di 150 μL fuori la sospensione totale delle cellule di ~ 1.000 μL sulla piastra LB-agar senza la necessità di concentrare le cellule in un tubo del microcentrifuge mediante centrifugazione.
- Il giorno seguente, Trasferire asetticamente una singola Colonia in una beuta contenente 20 mL di LB completati con l'antibiotico adatto (cioè kanamicina μg/mL 60 per animale domestico-28a-EFSAM) da 200 mL. Far crescere questa liquido avviamento cultura durante la notte (cioè ~ 16 h) a 37 ° C con agitazione costante alle ~ 190 giri/min.
- Lo stesso giorno come passo 2.2, preparare M9 medio per 15 espressione di proteina N-labeled in autoclave 1 L di M9 tampone sali (cioè 42mm Na 2 HPO 4, 22 mM KH 2 PO 4, 8,6 mM NaCl, pH 7.4) in un matraccio di Erlenmeyer 4L. Una volta fredda, filtrare su una miscela di 20% (p/v) D-glucosio, 1m CaCl 2, tiamina di 1 M, 1 M MgSO 4, biotina 1 mg/mL e 0,2 g/mL 15 N-NH 4 Cl 0,2 μm siringa sterile filtro nella soluzione 1 L sterile M9 sale affinché il le concentrazioni finali di questi componenti sono 0,2% (w/v) D-glucosio, 100 μM CaCl 2, 50 tiamina μM, 1mm MgSO 4, 1 biotina μg/mL e 1 mg/mL 15 N-NH 4 cl.
- Il giorno successivo, Trasferire asetticamente la coltura starter liquido durante la notte di 20 mL in una provetta conica sterile da 50 mL e centrifugare a 2.400 × g per 15 min agglomerare le cellule.
- Dopo decantazione il mezzo LB, risospendere il pellet cellulare risultante in 10 mL di terreno minimo M9 e trasferire il composto di sedimento risospeso in 1 L di terreno minimo M9 con antibiotico (cioè 60 kanamicina μg/mL).
- Crescere i 1 L di terreno minimo M9 contenente la coltura starter batterici a 37 ° C e ~ 190 giri/min costante agitazione fino a quando la densità ottica a 600 nanometro (OD600) raggiunge ~0.6-0.8.
- Quando viene raggiunto l'intervallo di specfified OD600, aggiungere 200 μM di isopropile β-D-1-thiogalactopyranoside (IPTG) per indurre l'espressione della proteina.
- Dopo l'aggiunta di IPTG, continuare incubando le cellule per l'espressione della proteina a temperatura ambiente con agitazione costante a ~ 190 rpm per ~ 16 h (cioè durante la notte).
- Il giorno successivo, raccogliere i batteri mediante centrifugazione a ~ 10.000 × g, 4 ° C per 30 min.
- Decantare il LB e trasferire il pellet cellulare in una provetta conica da 50 mL. Conservare il pellet a-80 ° C fino al momento di purificazione.
3. Purificazione della proteina ricombinante da e. coli.
Nota: diverse proteine ricombinanti richiedono procedure di purificazione distinti. Di seguito è riportato il protocollo per 6 & n. 215; Purificazione di EFSAM sua etichetta dai corpi di inclusione espressa dal costrutto pET-28a.
- Manualmente omogeneizzare il pellet di cellule batteriche congelati in 6m guanidina-HCl, 20 mM Tris-HCl (pH 8) e 5 mM β-mercaptoetanolo utilizzando una pipetta di trasferimento motorizzata da 10 mL. Aggiungere circa 40 mL di guanidina-HCl ogni 5 mL di pellet cellulare bagnato per questo passaggio.
- Seguenti a 90 min di incubazione a temperatura ambiente con rotazione costante in un forno di ibridazione, centrifugare la miscela a ~ 15.000 × g, 8 ° C per 40 min separare i detriti cellulari insolubile (cioè pellet) dalla proteina solubile miscela (cioè surnatante).
- Aggiungere 750 µ l di un 50% (v/v) Ni 2 +-agarosio acido nitrilotriacetico perlina liquami per il chiarificato lisato e incubare per un altro 90 min a temperatura ambiente con inversione in un forno di ibridazione.
- Catturare successivamente, la proteina His-tag 6 × associata alla Ni 2 + raccogliendo le sfere di agarosio in una colonna di purificazione della proteina di gravità flusso. Consentire il lisato di fluire attraverso la colonna completamente prima di passare alla fase 3.5.
- Lavare le perle raccolte tre volte con 10ml di 6 M urea, 20 mM Tris-HCl pH 8 e 5 mM β-mercaptoetanolo. Assicurarsi che l'intero mL 10 passi attraverso la colonna prima di ogni successivo 10 mL nello stato di Washington
- Eluire le proteine in una serie di frazioni di 2ml utilizzando 6 M urea, 20 mM Tris-HCl a pH 8, imidazolo di 300 mM e 5 mM β-mercaptoetanolo con un tempo di incubazione di s 90 tra frazioni. Assicurarsi che l'intero mL 2 passi attraverso la colonna prima di ogni fase successiva eluizione.
- In questa fase, confermare la proteina di interesse è presente nelle frazioni eluite di Coomassie blu-macchiato elettroforesi dodecilica del sodio solfato del gel di poliacrilammide (SDS-PAGE) utilizzando il metodo di Laemmli 47. Valutare la dimensione di proteine, la quantità e la purezza in confronto contro bande di marcatore di peso molecolare standard che sono entrambi minore e maggiore del previsto peso molecolare della proteina di interesse.
- Piscina le frazioni eluite proteina in una membrana di dialisi con 3.500 peso molecolare Da taglio e incubare nel buffer di refolding 1 L (20 mM Tris, 300 mM NaCl, 1 millimetro DTT, 5mm CaCl 2, pH 8) a 4 ° C durante la notte mentre il buffer è agitato da un magneti agitatore di c.
- Dopo ~ 16 h di refolding tempo, aggiungere ~ 1 U di trombina per mg di proteina direttamente per il sacchetto di dialisi e incubare a 4 ° C per un ulteriore ~ 24 h.
- Verificare la portata di 6 × sua fenditura di tag-azzurro di Coomassie macchiando di ~ 15 proteine aliquote del μL prelevate dal sacchetto di dialisi prima e dopo incubazione con trombina che sono electrophoresed su denaturare i gel di poliacrilammide (SDS-PAGE) utilizzando il metodo di Laemmli 47. Se uno spostamento di ~ 2 kDa nella migrazione è osservato corrisponde al peso molecolare della fenduti 6 × suo tag, procedere al passaggio 3.11; Se una frazione di resti di proteine non digerite che è rilevabile dalla macchiatura azzurro di Coomassie, aggiungere ~0.2 U di trombina per mg di proteina direttamente il sacchetto di dialisi e incubare a 4 ° C per un ulteriore ~ 24 h.
- Cromatografia di esclusione o scambio ionico di formato uso per purificare ulteriormente la proteina. Per cromatografia a scambio anionico di EFSAM, rimuovere la soluzione della proteina dal sacchetto di dialisi e concentrato ~ 10 volte usando un concentratore centrifugo di ultrafiltrazione con 10.000 peso molecolare Da taglio. Successivamente, re-diluire la soluzione ~ 20 volte in un tampone privo di NaCl (20 mM Tris, 5mm CaCl 2, 1mm DTT, pH 8).
- Equilibrare una colonna di scambio anionico preconfezionati con 10 volumi di colonna del buffer privo di NaCl al punto 3.11. Equilibrare utilizzando buffer caricato in una siringa luer lock non contenente bolle d'aria spingendo la soluzione attraverso la colonna in modo goccia a goccia ed evitando pressioni di siringa che causano flussi costante della soluzione uscendo la colonna. Utilizzare uno scambiatore di anioni forti (ad es. il reticolato agarosio con gruppi funzionali di ammonio quaternario).
- Caricare la soluzione di proteine diluita in tampone privo di NaCl (punto 3.11) sulla colonna come descritto nel passaggio 3.12.
- Eluire le proteine in una sfumatura [cioè 0 - 60% (v/v)] di NaCl in aumento del buffer (20 mM Tris, 1 M di NaCl, 5 mM CaCl 2, 1mm DTT, pH 8) utilizzando un sistema di cromatografia in fase liquida (FPLC) proteine veloci di due pompe. Impostare il sistema FPLC per raccogliere le frazioni ~1-1.5 mL e monitorare il profilo di eluizione di proteina utilizzando la capacità di assorbimento di UV 280 nm e una portata nominale di 0,5 mL/min
- Identificare i picchi di eluizione e le frazioni contenenti la proteina d'interesse così come la purezza della proteina di Coomassie blu-macchiato SDS-PAGE gel utilizzando il metodo di Laemmli 47.
- Frazioni piscina mostrando > 95% (ossia assunto come frazioni che mostrano solo una banda singola proteina su macchiato gel di Coomassie) in un sacchetto di dialisi e cambio nel buffer sperimentale di interesse tramite dialisi come descritto al punto 3.8.
4. Allegato chimica del glucosio-5-MTS alla proteina tramite dialisi.
Soluzione di riserva- preparare un 55 millimetri di N.-(β-D-glucopyranosyl)-N '-[2-methanethiosulfonyl) etil] urea (glucosio-5-MTS) sciogliendo 10 mg del composto in 500 μl di 100% (v/v) DMSO. Immagazzinare glucosio-5-MTS inutilizzati solubilizzato in DMSO a -20 ° C.
- Preparare il campione della proteina per la modifica di dialisi 1,5 mL di proteina μM ~ 60 in 1 L di modificazione buffer costituito da MOPS di 20 mM, 150 mM NaCl, 5 mM CaCl 2 e 0,1 mM TCEP, pH 8.3. Utilizzare un taglio peso molecolare di membrana di dialisi che è inferiore alla dimensione della proteina viene modificata (ad esempio utilizzare un cut-off Da 3.500 per il EFSAM Da ~ 17.500).
- Dopo 24 h a 4 ° C, trasferire il campione dal sacchetto di dialisi in una microcentrifuga. Aggiungere il glucosio-5-MTS solubilizzato in DMSO ad una concentrazione finale di 2 mM.
- Incubare il campione al buio per 1 h a temperatura ambiente. Durante il periodo di incubazione di 1 h, miscelare la soluzione di picchiettando del tubo ogni 10 min.
- Successivamente, ri-scambiare la proteina con il buffer finale sperimentale non contenente nessun agente riduttore di dialisi a 4 ° C, come descritto al punto 4.2 o mediante ultrafiltrazione centrifugo. Per la procedura di ultrafiltrazione, concentrare il campione di proteina mL di ~1.5 di < 0,5 mL e successivamente diluire nel concentratore stesso con il buffer di sperimentale. Ripetere questo passaggio di concentrazione-diluizione due volte in modo che lo scambio totale è un minimo di 30 × 30 × 30 = 27,000-fold. Per EFSAM, utilizzare 20 mM Tris, 150 mM NaCl, 5 mM CaCl 2, pH 7.5 del buffer sperimentale.
- Preparare il campione per la spettrometria totale di ionizzazione electrospray di dialisi o ultrafiltrazione scambio come descritto ai punti 4.2 e 4.5, rispettivamente, in bicarbonato di ammonio di 25 mM o 25 mM ammonio acetato. Se si utilizza la dialisi, assicurarsi si scambiano almeno tre volte per rimuovere qualsiasi residuo NaCl e CaCl 2 sali.
- Determinare la massa accurata (cioè ± 1 Da) della proteina di interesse utilizzando la ionizzazione electrospray di massa spettrometria 48 , 49. Si aspettano ogni aggiunta di glucosio covalente di un tiolo di Cys via il prodotto chimico methanethiosulfonate per aggiungere Da 281.3 alla proteina massa (cioè aggiungere 360.4 Da per il glucosio-5-MTS e sottrarre Da 79,1 per CH 3 modo lasciando 2 gruppo durante covalente allegato).
5. Valutazione di soluzione NMR di modifperturbazioni di efficienza e strutturali icazione.
Proteina- verificare la concentrazione del modificati è > 100 μM dopo l'attacco di glucosio e lo scambio di buffer finale. Per EFSAM, stimare la concentrazione nella proteina utilizzando un coefficiente di estinzione UV a 280 nm di 1,54 (mg mL – 1) cm – 1.
- Integratore soluzione della proteina con 60 μM di acido 4,4-dimethyl-4-silapentane-1-sulfonic (DSS) per la calibrazione di spessoramento e impulso e 10% (v/v) D 2 O per il blocco del segnale. Per i campioni di alto segnale-rumore uso 600 μL in tubi di frequenza-abbinati 5 mm NMR, inserito in uno spettrometro di 600 MHz minimo dotato di una sonda criogenica di tripla risonanza HCN.
- Raccogliere gli spettri standard 1 H - 15 N HSQC come precedentemente dettagliate 50 , 51 a temperatura, 1 H e 15 N sweep larghezze, transitori e incremento impostazioni adatte per il particolare campione. Per EFSAM spectra, uso 20 ° C, 256 1 H transitori, 64 incrementi di dimensione 15 N e 1 H e 15 N spazzare larghezze impostate rispettivamente su 8.000 e 1.800 Hz,.
- a seguito dell'acquisizione dello spettro della proteina glicosilata, aggiungere ditiotreitolo (DTT) al campione NMR da uno stock di 1m a una concentrazione finale di 15 mM. Il DTT rimuove la molecola di glucosio dalla proteina di riduzione del pignoramento bisolfuro-mediata.
- Acquisire una seconda 1 H - 15 N HSQC in queste condizioni di ridotto/non modificato, fornendo uno spettro di riferimento per valutare le perturbazioni di modificazione strutturale ed efficienza causati tramite il collegamento di glucosio.
- Elaborare i dati NMR utilizzando NMRPipe come dettagliato in precedenza 52. Assicurarsi che l'elaborazione minimamente includa conversione dati, phasing, solvente soppressione, di Fourier e visualizzazione iniziale degli spettri.
- Valutare l'efficienza di modifica misurando l'intensità del picco di ammide e valori di spostamento chimico negli spettri modificati e ridotti utilizzando il plugin NEASY su CARA 53. Garantire per valutare l'intensità di picco del ammide Cys entrambi nel glucosio attaccato e ridotto gli spettri. Se non può trovarsi in modo affidabile l'ammide Cys in due spettri, utilizzare le intensità dei residui adiacenti la Cys come una lettura a.
- Calcola l'efficienza come l'intensità del ammide dallo spettro Cys-modificato diviso per l'intensità del ammide dallo spettro della Cys-ridotto (cioè DTT-trattato), moltiplicato per 100:
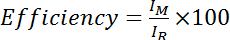 , dove M è l'intensità del ammide nello spettro Cys-modificato e I R è l'intensità del ammide nello spettro Cys-ridotto. In alternativa, valutare l'efficienza media sopra parecchi picchi di ammide:
, dove M è l'intensità del ammide nello spettro Cys-modificato e I R è l'intensità del ammide nello spettro Cys-ridotto. In alternativa, valutare l'efficienza media sopra parecchi picchi di ammide:
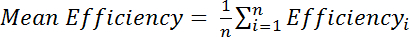 , dove l'efficienza i è l'efficienza separatamente determinato calcolato per ogni residuo, i e n è il numero totale di residui utilizzati nel calcolo.
, dove l'efficienza i è l'efficienza separatamente determinato calcolato per ogni residuo, i e n è il numero totale di residui utilizzati nel calcolo. - Calcolare perturbazioni di spostamento chimico (CSP) dalle differenze di spostamento chimico tra i due spettri osservati nel 15 N e 1 H dimensioni di ogni picco e normalizzante per il più grande 15 N spostamento chimico gamma utilizzando il seguendo l'equazione:
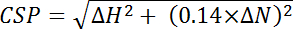 , dove ΔH è il cambiamento di ppm nella dimensione del protone e ΔN è il cambiamento di ppm nella dimensione azoto.
, dove ΔH è il cambiamento di ppm nella dimensione del protone e ΔN è il cambiamento di ppm nella dimensione azoto.
Access restricted. Please log in or start a trial to view this content.
Risultati
Il primo passo di questo approccio richiede la mutagenesi dei residui glicosilazione candidato ai residui di Cys che possono essere modificabile utilizzando il EFSAM di glucosio-5-MTS. non ha nessun residui di Cys endogeni, quindi particolari considerazioni non devono essere fatte prima dei mutagenesi. Tuttavia, residui di Cys natali devono essere mutati in residui non modificabile prima di eseguire la chimica descritta. Per effetto minimamente la struttura nativa, suggeriamo di eseguire ...
Access restricted. Please log in or start a trial to view this content.
Discussione
Glicosilazione della proteina è una modificazione post-traduzionale dove gli zuccheri sono covalente di polipeptidi principalmente attraverso collegamenti con catene laterali dell'amminoacido. Oltre il 50% delle proteine di mammiferi sono glicosilata 54, dove le proteine glicosilate successivamente possono avere una vasta gamma di effetti da alterazione biomolecolare affinità di legame, che influenzano la proteina pieghevole, alterando l'attività del canale, il targeting molecole per degradazio...
Access restricted. Please log in or start a trial to view this content.
Riconoscimenti
Questa ricerca è stata sostenuta dalle scienze naturali e ingegneria Research Council of Canada (05239 di P.B.S), Fondazione canadese per l'innovazione/Ontario Research Fund (per P.B.S), Prostate Cancer Foundation combattere - Telus Ride per papà (di P.B.S) e Ontario Borsa di studio laureato (per Y.J.C. e N.S.).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Phusion DNA Polymerase | Thermo Fisher Scientific | F530S | Use in step 1.3. |
| Generuler 1kb DNA Ladder | Thermo Fisher Scientific | FERSM1163 | Use in step 1.6. |
| DpnI Restriction Enzyme | New England Biolabs, Inc. | R0176 | Use in step 1.8. |
| Presto Mini Plasmid Kit | GeneAid, Inc. | PDH300 | Use in step 1.16. |
| BL21 DE3 codon (+) E. coli | Agilent Technologies, Inc. | 230280 | Use in step 2.1. |
| DH5a E. coli | Invitrogen, Inc. | 18265017 | Use in step 1.9. |
| 0.22 mm Syringe Filter | Millipore, Inc. | SLGV033RS | Use in step 2.3. |
| HisPur Ni2+-NTA Agarose Resin | Thermo Fisher Scientific | 88221 | Use in step 3.3. |
| 3,500 Da MWCO Dialysis Tubing | BioDesign, Inc. | D306 | Use in step 3.8, 3.16, 4.2, 4.5 and 4.6. |
| Bovine Thrombin | BioPharm Laboratories, Inc. | SKU91-055 | Use in step 3.9. |
| 5 mL HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5156-01 | Use in step 3.11. |
| Glucose-5-MTS | Toronto Research Chemicals, Inc. | G441000 | Use in step 4.1. |
| Vivaspin 20 Ultrafiltration Centrifugal Concentrators | Sartorius, Inc. | VS2001 | Use in step 3.11, 4.2, 4.5 and 4.6. |
| PageRuler Unstained Broad Protein Ladder | Thermo Fisher Scientific | 26630 | Use in step 3.7, 3.10 and 3.15 |
| HiTrap Q FF Anion Exchange Column | GE Healthcare, Inc. | 17-5053-01 | Use in step 3.12. |
| AKTA Pure Fast Protein Liquid Chromatrography System | GE Healthcare, Inc. | 29018224 | Use in step 3.14. |
| 600 MHz Varian Inova NMR Spectrometer | Agilent Technologies, Inc. | Use in step 5.2 and 5.5. |
Riferimenti
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Feske, S., Skolnik, E. Y., Prakriya, M. Ion channels and transporters in lymphocyte function and immunity. Nat Rev Immunol. 12 (7), 532-547 (2012).
- Shaw, P. J., Feske, S. Physiological and pathophysiological functions of SOCE in the immune system. Front Biosci (Elite Ed). 4, 2253-2268 (2012).
- Seo, M. D., Enomoto, M., Ishiyama, N., Stathopulos, P. B., Ikura, M. Structural insights into endoplasmic reticulum stored calcium regulation by inositol 1,4,5-trisphosphate and ryanodine receptors. Biochim Biophys Acta. 1853 (9), 1980-1991 (2015).
- Stathopulos, P. B., Ikura, M. Structural aspects of calcium-release activated calcium channel function. Channels (Austin). 7 (5), 344-353 (2013).
- Stathopulos, P. B., Ikura, M. Structure and function of endoplasmic reticulum STIM calcium sensors. Curr Top Membr. 71, 59-93 (2013).
- Stathopulos, P. B., Li, G. Y., Plevin, M. J., Ames, J. B., Ikura, M. Stored Ca2+ depletion-induced oligomerization of stromal interaction molecule 1 (STIM1) via the EF-SAM region: An initiation mechanism for capacitive Ca2+ entry. J Biol Chem. 281 (47), 35855-35862 (2006).
- Stathopulos, P. B., Ikura, M. Store operated calcium entry: From concept to structural mechanisms. Cell Calcium. , (2016).
- Stathopulos, P. B., Ikura, M. Structurally delineating stromal interaction molecules as the endoplasmic reticulum calcium sensors and regulators of calcium release-activated calcium entry. Immunol Rev. 231 (1), 113-131 (2009).
- Muik, M., et al. STIM1 couples to ORAI1 via an intramolecular transition into an extended conformation. EMBO J. 30 (9), 1678-1689 (2011).
- Luik, R. M., Wang, B., Prakriya, M., Wu, M. M., Lewis, R. S. Oligomerization of STIM1 couples ER calcium depletion to CRAC channel activation. Nature. 454 (7203), 538-542 (2008).
- Luik, R. M., Wu, M. M., Buchanan, J., Lewis, R. S. The elementary unit of store-operated Ca2+ entry: local activation of CRAC channels by STIM1 at ER-plasma membrane junctions. J Cell Biol. 174 (6), 815-825 (2006).
- Wu, M. M., Buchanan, J., Luik, R. M., Lewis, R. S. Ca2+ store depletion causes STIM1 to accumulate in ER regions closely associated with the plasma membrane. J Cell Biol. 174 (6), 803-813 (2006).
- Liou, J., Fivaz, M., Inoue, T., Meyer, T. Live-cell imaging reveals sequential oligomerization and local plasma membrane targeting of stromal interaction molecule 1 after Ca2+ store depletion. Proc Natl Acad Sci U S A. 104 (22), 9301-9306 (2007).
- Calloway, N., et al. Stimulated association of STIM1 and Orai1 is regulated by the balance of PtdIns(4,5)P(2) between distinct membrane pools. J Cell Sci. 124 (Pt 15), 2602-2610 (2011).
- Korzeniowski, M. K., et al. Dependence of STIM1/Orai1-mediated calcium entry on plasma membrane phosphoinositides. J Biol Chem. 284 (31), 21027-21035 (2009).
- Park, C. Y., et al. STIM1 clusters and activates CRAC channels via direct binding of a cytosolic domain to Orai1. Cell. 136 (5), 876-890 (2009).
- Yuan, J. P., et al. SOAR and the polybasic STIM1 domains gate and regulate Orai channels. Nat Cell Biol. 11 (3), 337-343 (2009).
- Feske, S., et al. A mutation in Orai1 causes immune deficiency by abrogating CRAC channel function. Nature. 441 (7090), 179-185 (2006).
- Prakriya, M., et al. Orai1 is an essential pore subunit of the CRAC channel. Nature. 443 (7108), 230-233 (2006).
- Vig, M., et al. CRACM1 multimers form the ion-selective pore of the CRAC channel. Curr Biol. 16 (20), 2073-2079 (2006).
- Vig, M., et al. CRACM1 is a plasma membrane protein essential for store-operated Ca2+ entry. Science. 312 (5777), 1220-1223 (2006).
- Liou, J., et al. STIM is a Ca2+ sensor essential for Ca2+-store-depletion-triggered Ca2+ influx. Curr Biol. 15 (13), 1235-1241 (2005).
- Roos, J., et al. STIM1, an essential and conserved component of store-operated Ca2+ channel function. J Cell Biol. 169 (3), Epub 2005 May 2002 435-445 (2005).
- Putney, J. W. A model for receptor-regulated calcium entry. Cell Calcium. 7 (1), 1-12 (1986).
- Feske, S. CRAC channelopathies. Pflugers Arch. 460 (2), 417-435 (2010).
- Maus, M., et al. Missense mutation in immunodeficient patients shows the multifunctional roles of coiled-coil domain 3 (CC3) in STIM1 activation. Proc Natl Acad Sci U S A. 112 (19), 6206-6211 (2015).
- Stathopulos, P. B., Zheng, L., Li, G. Y., Plevin, M. J., Ikura, M. Structural and mechanistic insights into STIM1-mediated initiation of store-operated calcium entry. Cell. 135 (1), 110-122 (2008).
- Stathopulos, P. B., Ikura, M. Partial unfolding and oligomerization of stromal interaction molecules as an initiation mechanism of store operated calcium entry. Biochem Cell Biol. 88 (2), 175-183 (2010).
- Dennis, J. W., Lau, K. S., Demetriou, M., Nabi, I. R. Adaptive regulation at the cell surface by N-glycosylation. Traffic. 10 (11), 1569-1578 (2009).
- Nilsson, T., Au, C. E., Bergeron, J. J. Sorting out glycosylation enzymes in the Golgi apparatus. FEBS Lett. 583 (23), 3764-3769 (2009).
- Stanley, P. Golgi glycosylation. Cold Spring Harb Perspect Biol. 3 (4), (2011).
- Moremen, K. W., Tiemeyer, M., Nairn, A. V. Vertebrate protein glycosylation: diversity, synthesis and function. Nat Rev Mol Cell Biol. 13 (7), 448-462 (2012).
- Gerlach, J., Sharma, S., Leister, K., Joshi, L. Endoplasmic Reticulum Stress in Health and Disease. Agostinis, P., Afshin, S. , Bristol-Myers Squibb. Syracuse, NY. 23-39 (2012).
- Pearse, B. R., Hebert, D. N. Lectin chaperones help direct the maturation of glycoproteins in the endoplasmic reticulum. Biochim Biophys Acta. 1803 (6), 684-693 (2010).
- Stanley, P., Sundaram, S. Rapid assays for lectin toxicity and binding changes that reflect altered glycosylation in mammalian cells. Curr Protoc Chem Biol. 6 (2), 117-133 (2014).
- Avezov, E., Frenkel, Z., Ehrlich, M., Herscovics, A., Lederkremer, G. Z. Endoplasmic reticulum (ER) mannosidase I is compartmentalized and required for N-glycan trimming to Man5-6GlcNAc2 in glycoprotein ER-associated degradation. Mol Biol Cell. 19 (1), 216-225 (2008).
- Csutora, P., et al. Novel role for STIM1 as a trigger for calcium influx factor production. J Biol Chem. 283 (21), 14524-14531 (2008).
- Kilch, T., et al. Mutations of the Ca2+-sensing stromal interaction molecule STIM1 regulate Ca2+ influx by altered oligomerization of STIM1 and by destabilization of the Ca2+ channel Orai1. J Biol Chem. 288 (3), 1653-1664 (2013).
- Williams, R. T., et al. Stromal interaction molecule 1 (STIM1), a transmembrane protein with growth suppressor activity, contains an extracellular SAM domain modified by N-linked glycosylation. Biochim Biophys Acta. 1596 (1), 131-137 (2002).
- Mignen, O., Thompson, J. L., Shuttleworth, T. J. STIM1 regulates Ca2+ entry via arachidonate-regulated Ca2+-selective (ARC) channels without store depletion or translocation to the plasma membrane. J Physiol. 579 (Pt 3), 703-715 (2007).
- Choi, Y. J., Zhao, Y., Bhattacharya, M., Stathopulos, P. B. Structural perturbations induced by Asn131 and Asn171 glycosylation converge within the EFSAM core and enhance stromal interaction molecule-1 mediated store operated calcium entry. Biochim Biophys Acta. 1864 (6), 1054-1063 (2017).
- Davis, B. G., Lloyd, R. C., Jones, J. B. Controlled site-selective protein glycosylation for precise glycan structure-catalytic activity relationships. Bioorg Med Chem. 8 (7), 1527-1535 (2000).
- Gamblin, D. P., van Kasteren, S. I., Chalker, J. M., Davis, B. G. Chemical approaches to mapping the function of post-translational modifications. FEBS J. 275 (9), 1949-1959 (2008).
- Ehrt, S., Schnappinger, D. Isolation of plasmids from E. coli by alkaline lysis. Methods Mol Biol. 235, 75-78 (2003).
- Sanger, F., Coulson, A. R. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 94 (3), 441-448 (1975).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bell, D. J. Mass spectrometry. Methods Mol Biol. 244, 447-454 (2004).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312 (5771), 212-217 (2006).
- Farrow, N. A., et al. Backbone Dynamics of a Free and a Phosphopeptide-Complexed Src Homology-2 Domain Studied by N-15 Nmr Relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Kay, L. E., Keifer, P., Saarinen, T. Pure Absorption Gradient Enhanced Heteronuclear Single Quantum Correlation Spectroscopy with Improved Sensitivity. Journal of the American Chemical Society. 114 (26), 10663-10665 (1992).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Masse, J. E., Keller, R. AutoLink: automated sequential resonance assignment of biopolymers from NMR data by relative-hypothesis-prioritization-based simulated logic. J Magn Reson. 174 (1), 133-151 (2005).
- Monticelli, M., Ferro, T., Jaeken, J., Dos Reis Ferreira, V., Videira, P. A. Immunological aspects of congenital disorders of glycosylation (CDG): a review. J Inherit Metab Dis. 39 (6), 765-780 (2016).
- An, H. J., Kronewitter, S. R., de Leoz, M. L., Lebrilla, C. B. Glycomics and disease markers. Curr Opin Chem Biol. 13 (5-6), 601-607 (2009).
- Wani, W. Y., Chatham, J. C., Darley-Usmar, V., McMahon, L. L., Zhang, J. O-GlcNAcylation and neurodegeneration. Brain Res Bull. , (2016).
- Haines, A. M., Tobe, S. S., Kobus, H. J., Linacre, A. Properties of nucleic acid staining dyes used in gel electrophoresis. Electrophoresis. 36 (6), 941-944 (2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon