Method Article
Une approche impartiale de l’échantillonnage des Sections TEM en neurosciences
Dans cet article
Résumé
Nous introduisons un roman "workflow" pour microscopie électronique enquêtes du tissu cérébral. La méthode permet à l’utilisateur d’examiner les fonctions neuronales de façon impartiale. Pour l’analyse élémentaire, nous présentons également un script qui automatizes la majeure partie du flux de travail pour l’échantillonnage aléatoire.
Résumé
Enquêtes des caractéristiques ultrastructurales des neurones et leurs synapses ne sont possibles avec la microscopie électronique. Surtout pour des études comparatives des changements dans la densité et les distributions de ces caractéristiques, un protocole d’échantillonnage non biaisé est vital pour des résultats fiables. Nous présentons ici un "workflow" pour l’acquisition de l’image des échantillons de cerveau. Le workflow permet un échantillonnage aléatoire systématique uniform dans une région du cerveau définie, et les images peuvent être analysées à l’aide d’un disector. Cette technique est beaucoup plus rapide qu’un examen approfondi des coupes sériées, mais présente toujours une approche possible pour estimer les densités et les distributions des caractéristiques de l’ultrastructure. Avant l’intégration, tachée vibratome sections ont été utilisées comme référence pour identifier la zone du cerveau sous enquête, qui a contribué à accélérer la préparation de l’échantillon global du processus. Cette approche a été utilisée pour des études comparatives sur les effets d’un environnement enrichi-logement sur plusieurs paramètres ultrastructurales dans le cerveau de souris. Basé sur l’utilisation efficace du flux de travail, nous avons adapté aux fins de l’analyse élémentaire des échantillons de cerveau. Nous avons optimisé le protocole en fonction du temps d’interaction utilisateur. Automatisation de toutes les étapes fastidieuses à compiler un script pour le logiciel open source SerialEM permet à l’utilisateur de se concentrer sur l’essentiel du travail d’acquérir les cartes élémentaires. Comme dans le flux de travail original, nous avons prêté attention à l’approche d’échantillonnage non biaisé pour garantir des résultats fiables.
Introduction
En microscopie électronique, il est souvent difficile de goûter des régions représentatives au sein des sections. Nous, en qualité d’observateur, sommes souvent biaisées pour ressembler à des régions spécifiques portées à notre attention par remarquables caractéristiques de l’échantillon, empêchant un échantillonnage non biaisé bien réparti. Échantillonnage de partialité ne peut être évité que si toutes les parties de la région d’intérêt obtient les mêmes chances de se retrouver dans une micrographie électronique1. Il est possible d’éviter les biais d’échantillonnage sans une solution de logiciel, par exemple, en appuyant sur la trackball du microscope manuellement sans regarder l’image, afin de sélectionner les régions d’échantillonnage où la scène s’arrête. Mais, à proprement parler, ce n’est pas une procédure aléatoire, parce que, consciemment ou inconsciemment, l’utilisateur peut avoir une influence sur le mouvement de la scène, et, en outre, ce n’est pas une manière sophistiquée de sélection des régions d’échantillonnage. Échantillonnage aléatoire devient particulièrement important si les paires des sections sont utilisés pour évaluer le nombre de structures dans un certain volume, par exemple, stéréologie1, qui nécessite des paires des sections, une distance connue apart. Il serait également possible de regarder seulement une seule section et estimer le nombre de structures spécifiques2, mais avec cette approche enquêteurs ont tendance à surestimer la densité numérique de structures plus grandes, à moins que les structures sont de très petites comparaison de l’épaisseur de coupe. Approches alternatives doivent reconstituer des volumes de tissus provenant de coupes sériées et ainsi obtenir les données souhaitées3. Mais c’est très longue et pas une approche possible pour les études comparatives (plus grands).
Pour résoudre ces problèmes, nous avons développé un workflow qui permet au chercheur de sélectionner automatiquement des échantillons pour l’obtention de micrographies à espacement régulier au sein des sections ultra-minces. La position de la microscopie électronique est aléatoire, permettant d’échantillonnage non biaisé. L’approche est adaptée aussi bien pour la détermination de la densité numérique des structures (par exemple, les synapses dans un certain neuropile volume4,5) et les dimensions des caractéristiques structurelles (par exemple, la largeur de la fente synaptique, (ou le diamètre de la densité postsynaptique4,,5).
Le flux de travail utilise un point aléatoire sur mesure, d’échantillonnage (RPS) logiciel (écrit en Java script à l’aide de logiciel de script fourni avec notre microscope) qui calcule automatiquement les places sur la grille dans une région prédéfinie d’intérêt dans une section ultra-mince. Le logiciel RPS déplace la scène du microscope électronique à ces points prédéfinis, afin qu’un microscope électronique peut être effectué à chaque point. Tout d’abord, l’utilisateur définit une zone d’intérêt au sein de la section mince. Ensuite, le logiciel RPS calcule places sur la grille au sein de cette région. Le x / coordonnées de la première position sont créées aléatoirement, et les positions restantes sont placées à intervalles réguliers de grille en ce qui concerne la première position. Parce que toutes les parties de la région d’intérêt a les mêmes chances d’être examinés, cela permet la collecte de données minimale. Cette approche de l’échantillonnage est aussi appelée un échantillonnage aléatoire systématique uniform (voir référence6,,7 , pour plus de détails).
Pour déterminer la densité numérique des structures, nous travaillons avec paires de sections qui sont une distance connue apart. Après avoir obtenu une micrographie électronique de la première section dans une des positions prédéterminées, logiciel TEM Section série (partie du package logiciel fourni avec notre microscope électronique) se déplace vers le point correspondant dans la deuxième section, afin de obtenir un micrographe de l’emplacement correspondant. Cela est répété pour chaque emplacement dans la grille prédéterminée. Dans notre approche, un disector est utilisé pour compter le nombre de particules dans chaque paire de micrographies8,9. Un disector se compose d’une paire d’images de comptage, un pour chaque section8,9. La densité numérique des objets est déterminée en comptant seulement les objets visibles sur la première section (ou section de référence), mais pas à la deuxième section (section de recherche). Cela permet d’estimer la densité numérique des objets dans une manière rapide et efficace8,9. En outre sur des coupes simples, deux dimensions caractéristiques structurelles peuvent être mesurés.
Nous avons appliqué ce flux de travail avec succès afin d’évaluer les différences de nombre de synapse dans l’hippocampe de souris exposées à l’environnement enrichi (EE) par rapport à l’environnement standard (SE) logement conditions4,5, de conditions de logement et aussi pour évaluer les différences ultrastructurales entre les souris de type sauvage (WT) et le neuropeptide Y (NPY) KO souris maintenues sous soi et EE5. Notre objectif consistait à examiner plus précisément les caractéristiques structurales des neurones, comme la densité numérique synaptique, les longueurs de la zone active dans les coupes et de la densité postsynaptique, la largeur de la fente synaptique et le nombre de vésicules synaptiques, afin d’évaluer les changements de connectivité neuronale et l’activation entre les différentes conditions expérimentales. En outre, nous étions intéressés par la densité numérique des vésicules de noyau dense (DCV) dans les neurones pour déterminer le montant des neuropeptides stockée dans une certaine région du cerveau.
Basé sur le succès de notre approche pour les études décrites ci-dessus, dans notre prochaine étape, nous avons adapté notre workflow pour sélectionner les zones pour les analyses élémentaires non biaisées dans des échantillons de cerveau humain. Cela a été fait au fer d’image, qui est stocké dans les molécules de ferritine dans les neurones et les cellules gliales. Pour ce faire, nous avons compilé un script qui nous a permis d’automatiser la plupart des opérations pour un processus de sélection aléatoire des sections de cerveau dans une zone définie.
Protocole
Toutes les expériences ont été approuvés par un comité d’éthique au ministère fédéral des sciences et recherche de la République d’Autriche (BMWF-80.104/2-BrGT/2007 et BMWF-66.010/0037-II/3b/2013), ou la commission d’éthique de l’université médicale de Graz, numéro 28-549 ex 15/16 et effectué conformément à la Directive des communautés européennes Conseil du 24 novembre 1986 (86/609/CEE) et la Directive du Parlement européen et du Conseil de September2010 22 (2010/63/UE). L’expérimentation animale ont été conçue et réalisée de telle sorte que le nombre d’animaux utilisés a été réduit au minimum.
1. dissection et la Fixation du tissu
- Euthanasier souris (21 semaines vieux, femelle souris C57BL/6J et C57BL/6N souris ; les souris mâles WT et NPY KO sur un mixte C57BL / 6:129 / SvJ (1:1) contexte respectivement) en injectant pentobarbital de 150 mg/kg par voie intrapéritonéale.
- Retirer le cerveau du crâne et immédiatement, coupez-les en deux (qui sépare la gauche de l’hémisphère droit) - à l’aide d’un scalpel.
- Placer immédiatement les moitiés dans des récipients en verre avec 2 % de formaldéhyde et glutaraldéhyde à 2,5 % en cacodylate de 0,1 M de tampon (CH3)2AsO2Na·3H2O, pH ajusté à 7,4 avec 1 M NaCl), pH 7,4 à 4 ° C.
Attention : Les fixateurs sont toxiques et doivent être manipulés avec soin. Utiliser seulement avec des gants de protection et sous la hotte. - Difficulté les moitiés de cerveau pendant 2 jours à 4 ° C et de rinçage dans le même tampon pendant au moins 24 h, également à 4 ° C. Le volume de la mémoire tampon doit être à environ dix fois le volume de spécimen.
2. identifier la zone d’intérêt dans le cerveau en comparant les Sections Vibratome avec référence des Sections de la souris Brain Atlas10
- Placer l’échantillon de cerveau sur un vibratome. Assurez-vous que l’orientation du cerveau est correcte (dans ce cas, une orientation coronaire a été choisie pour l’hippocampe).
- La garniture jusqu'à ce que la région d’intérêt (ROI) dans le cerveau est atteint. Les coupes sur une grande épaisseur (p. ex.., 100 µm) jusqu'à ce que la partie du cerveau avec la région d’intérêt est atteint et jeter toutes les sections.
- Commencer à couper les sections vibratome avec 20 µm d’épaisseur au début de la partie du cerveau avec le retour sur investissement et placez les sections vibratome sur une lame de verre. Tacher les (coloration de Nissl) en plaçant les sections en acétate thionine 0,05 % dans le tampon d’acétate de sodium, pH 4,2 pendant 1 min (Iadela Figure 1).
- Comparer les sections colorées avec l’atlas de cerveau de souris à l’aide d’un microscope optique et continuer la coupe et la coloration jusqu'à ce que les coordonnées de cerveau souhaités sont atteints.
3. obtention d’une échantillon de région pour l’enrobage
- Dès que la zone de droite est identifiée, commencer une coupe à 150 µm d’épaisseur avec le vibratome.
- Microdissect cette section vibratome avec une lame de rasoir sous le microscope stéréo. Découper les pièces autour du ROI. La section devrait avoir une taille appropriée pour les étapes suivantes de la préparation pour la microscopie électronique à transmission (TEM) (pas plus grande que 1 x 1 mm2).
4. préparation TEM - incorporation, ultra-mince, coupes et coloration
- Enrobage
- Après correction pendant 2 h à 2 % le tétroxyde d’osmium à température ambiante (RT). Utiliser un volume pour couvrir l’échantillon avec au moins 10 fois le volume de l’échantillon, mais évitez les excès fixateur pour empêcher les déchets toxiques inutiles.
Attention : le tétroxyde d’Osmium est très toxique et doit être manipulé avec beaucoup de soin. Utiliser seulement avec des gants de protection et sous la hotte. - Déshydrater à l’aide de l’alcools EM Grade par incréments de 20 min. utilisation un volume plus important que celui à l’étape précédente (50 %, 70 %, 80 %, 96 % et 100 % éthanol).
- Placer dans l’oxyde de propylène pendant 40 min à RT. Place dans un mélange de résine / intégration de l’oxyde de propylène (1:2) pour 2 h à RT et nuit de 1:3 à 4 ° C.
Attention : L’oxyde de propylène est très toxique et doit être manipulé avec beaucoup de soin. Utiliser seulement avec des gants de protection et sous la hotte. - Incorporer les échantillons en résine 100 % en changeant la résine 2 fois après 60 min et une fois après 90 min (tous à 45 ° C).
- Enfin, placer les échantillons dans les moules appropriés et laissez la résine de polymériser à 90 ° C pendant 3 jours (Figure 1Aii).
- Après correction pendant 2 h à 2 % le tétroxyde d’osmium à température ambiante (RT). Utiliser un volume pour couvrir l’échantillon avec au moins 10 fois le volume de l’échantillon, mais évitez les excès fixateur pour empêcher les déchets toxiques inutiles.
- Parage et sectionnement sur un ultramicrotome.
- Couper le bloc, en veillant à ce que les côtés sont plus en douceur possible pour les parties d’adhérer les uns aux autres (Figure 1Aiii).
- Produit 55 nm ultra-mince sériées (devrait être argent gris) en utilisant un ultramicrotome. Utiliser une grille de fente (slot largeur 1 x 2 mm) recouvert de pioloform (Figure 1Aiv).
- Contre-coloration ultra minces sur les grilles de la fente à l’aide de l’acétate d’uranyle 2 % pendant 30 min et citrate de plomb pendant 30 s à la droite (il s’agit de méthode de microscopie standard11).
Attention : L’acétate d’uranyle est hautement toxique et Eviter tout contact direct. Manipuler avec des gants de protection. Nitrate de plomb est toxique si ingéré ou inhalé et doivent être manipulés avec soin.
5. imagerie du ROIs correspondants à la référence et des articles de recherche sur TEM avec les logiciels
- Examiner les sections sur la grille avec le TEM en utilisant un grossissement faible (selon la taille des sections), d’orientation et d’évaluer la qualité des sections.
- Lancez le logiciel (un paquet de logiciel d’analyse Image TEM ou TIA, fourni avec le microscope) pour générer des images virtuelles des sections en stockant les angles des sections. Qui permet au logiciel de trouver aux emplacements correspondants du ROI sur chaque section.
- Allez dans la Section et choisissez insérer pour ajouter les points d’angle pour la section de référence et de recherche. Suivez les instructions de la fenêtre contextuelle. Commencez par la section de référence et continuez avec la section de recherche. Assurez-vous que les bords de la section qui sont parallèles à la section suivante sous les points 1 et 2 (Figure 1Bi) sont entrés.
- Visualiser la section de référence sous un grossissement faible pour identifier la zone d’intérêt et de déplacer la platine du microscope à l’aide de TIA sur la section de référence à plusieurs points d’angle du ROI pour créer un plan du ROI.
- Enregistrer les coordonnées du polygone résultant à l’aide de logiciels RPS . Pour ce faire, appuyez sur ajouter coordonnées dans la boîte de dialogue du logiciel RPS à chaque point du polygone qui est nécessaire pour décrire le retour sur investissement dans la section (Figure 1Bii).
- Définir et entrez une taille appropriée pour les zones d’échantillonnage et les distances entre les zones dans le logiciel RPS (dans l’étude présentée, la taille des zones d’échantillonnage est de 7 µm et la distance entre eux est de 20 µm, pour au moins 20 zones d’échantillonnage au sein de la RETOUR SUR INVESTISSEMENT). Appuyez sur Calculer Raster, puis le logiciel génère des coordonnées de zone d’échantillonnage d’une manière aléatoire uniforme systématique pour les postes de la micrographie dans le polygone (Figure 1Biii).
Remarque : Pour une meilleure orientation, le polygone et les zones d’échantillonnage dans le polygone peuvent être affichées par le logiciel RPS (Figure 1Biv).
- Stocker les points de prélèvement sur la section de référence et de recherche en utilisant le logiciel RPS et TEM série Section logiciel. Ce sont les coordonnées des montages qui sont enregistrés par la suite11.
- Dans le logiciel RPS, appuyez sur aller à la position suivante pour déplacer la platine du microscope à x / coordonnées de chaque échantillonnage de zone dans la section de référence. Allez à l’emplacement et Insert pour importer ces coordonnées dans le logiciel dans la section de référence. Répétez cette opération pour toutes les coordonnées.
- Afin de refléter les coordonnées de la section de référence sur la section look-up, allez à la Section , puis appuyez sur aller à la Section. Entrez le numéro de la section de recherche (généralement ' 2') dans la fenêtre de dialogue.
- L’inscription des montages (voir étape suivante), de basculer entre la section de référence et de recherche, comme décrit précédemment et changer la position sur la section de référence avec emplacement et accédez au numéro. Choisissez la coordonnée suivante dans la fenêtre de dialogue.
- Pour les montages de SerialEM à chaque échantillonnage, coordonner, allez dans fichier et choisissez Nouveau Montage dans le menu déroulant. Dans la fenêtre de dialogue, sélectionnez le bon nombre de tuiles et le pourcentage de chevauchement. Pour la présente étude, un grossissement de 5000 X est suffisant pour reconnaître les fonctionnalités synaptiques sous étude, mais le champ de vision limité de la caméra CCD requise de faire des montages d’images de 2 x 2. Les montages sont faits avec la SerialEM.
- Avant d’enregistrer chaque montage, réajuster le focus (ou si possible, activez l’option de mise au point automatique dans le logiciel d’enregistrement). Choisissez le dossier pour enregistrer le fichier de montage et lancer l’enregistrement du montage en appuyant sur Start dans le sous-menu montage à gauche dans SerialEM.
6. analyser les images TEM avec ImageJ pour Document caractéristiques ultrastructurales.
Remarque : Pour ce faire, il est important de créer une pile d’images alignées de la section de référence et de recherche.
- Convertir les couples d’images avec ImageJ Images à pile dans un fichier de pile et aligner les images avec StackReg (doit être installé de BIG-EPFL http://sites.imagej.net/BIG-EPFL/; dans le cas présent, l’algorithme Affine a été utilisé). Pour obtenir la densité numérique des caractéristiques de structure cellulaires, la macro d’ImageJ sur-mesure Disector génère une trame de comptage avec une taille de 5,5 x 5,5 µm placés au hasard sur l’image avec un angle aléatoire.
- Démarrer la macro Disector (emplacement dans le menu ImageJ dépend le répertoire dans lequel il est enregistré) et définir les paramètres (taille de la trame de comptage, nombre de segments) comme nécessaire (Figure 1Cii).
- Compter le nombre de synapses/µm2 à l’aide de la Disector. Compter chaque synapse dans le cadre de comptage qui est visible dans la section références, mais non visible dans la section de recherche. Omettez ces synapses qui croise les deux «raies interdites» de la disector ; mais comptez synapses sur l’opposé «lignes d’acceptation». Pour ce faire, utilisez l' Outil Multipoint dans les Toolsbar dans ImageJ (Figure 2A, Figure 2B).
- Pour mesurer des paramètres de la synapse, sélectionnez uniquement les synapses avec une fente synaptique orientée en section transversale (sur la section de référence, dans les mêmes cadres d’image utilisés pour le disector).
- Démarrer le plugin ObjectJ dans ImageJ dans le menu déroulant (Figure 2A) via Plugins | Analyser. Ouvrez un nouveau projet de la boîte de dialogue déroulante ObjectJ. Cela ouvrira une fenêtre qui permet à l’utilisateur à des structures contour et marque avec Outil marqueur (Figure 2B, cercle rouge). Les paramètres synaptiques utilisés pour les expériences présentées ici sont décrites dans les étapes suivantes.
- Mesurer la longueur de la membrane présynaptique et densité postsynaptique en traçant une ligne le long de la structure à l’aide de l' Outil marqueur (Figure 2B, cercle rouge).
- Obtenir la moyenne largeur de la fente synaptique en dessinant un polygone qui couvre aussi bien la membrane pre- et post-synaptiques, à côtés parallèles, rectilignes et un mi-chemin segmentée, équidistant de deux membranes.
- Pour déterminer le nombre de vésicules ancrées, compter toutes les vésicules qui ont une distance maximale de la membrane présynaptique d’un diamètre de vésicule ou moins.
- Pour déterminer le nombre de vésicules non ancrées, compter ces vésicules avec une distance maximum de diamètre d’une vésicule de vésicules non ancrées ancrées ou autres à la même synapse.
7. produisant des Sections minces semi pour la microscopie de lumière au Document caractéristiques macroscopiques (dans le cas présent, le nombre de cellules) dans le même ROI choisi pour l’enquête de TEM.
- Immédiatement après avoir coupé les sections ultra-minces pour TEM, couper deux sections semi minces, 0,5 µm épais et juste à côté les uns aux autres. Déposer sur une lame de verre et les taches à l’aide de solution de bleu de toluidine de 0,5 %. Placez une lamelle couvre-objet sur les sections (avec DPX milieu de montage, consistant en distyrene, un plastifiant (phosphate de tricrésyle) et xylène). Ces deux sections sont utilisées pour comptabiliser le nombre de cellules.
- Photographier les sections semi minces à faible grossissement, à l’aide d’un microscope optique.
Remarque : Les structures incriminé doivent être identifiables dans les images. Dans la présente étude, un grossissement de 20 X servait à identifier les corps cellulaires. Si nécessaire, plusieurs images peuvent être piqués avec une logiciel de traitement d’image. - Aligner les paires de deux sections consécutives de semi minces avec une logiciel de traitement d’image (e.g., ImageJ).
Remarque : Les différences de rotation et taille (peut se produire en raison des artefacts de compression introduites par le processus de découpe) doivent être ajustées pour une superposition parfaite des deux sections. - Marquent la frontière du ROI sur l’image de microscopie photonique. Le ROI doit correspondre à la ROI utilisée pour les images TEM. À cet effet, charger les images dans ImageJ et choisissez la sélection polygone dans la Toolsbar (Figure 2A).
- Générer une grille dans toute l’image à l’aide de ImageJ en ouvrant Analyze | Outils | Grille et réglez les paramètres selon les besoins.
- Calculer l’aire et le volume du polygone du nombre de points de grille dans la zone d’intérêt et la section d’épaisseur (Volume = NCS * unGS * hST ; NCS = compté Cross sections ; UneGS = taille de la grille ; hST = épaisseur de coupe).
- Compter le nombre de noyaux à partir des corps cellulaires neurones dans le retour sur investissement si elles ne sont présentes que sur une seule section. Sélectionnez Outil Multipoint en ImageJ dans le Toolsbar et marquer les cellules. Après avoir compté les cellules sur chaque section, calculer le nombre de cellules par volume (
 ; Cla densité = densité cellulaire ; Cnombre = nombre d’éléments).
; Cla densité = densité cellulaire ; Cnombre = nombre d’éléments). - Enregistrer toutes les mesures dans la fenêtre de Résultats de ObjectJ (Figure 2C). L’exportation et les enregistrer pour une utilisation ultérieure.
8. utilisation du SerialEM Script pour optimiser l’analyse élémentaire dans des échantillons de cerveau en association avec DigitalMicrograph (DM, livré avec filtre d’imagerie).
- Réglage du filtre (GIF) d’imagerie.
- Charger l’échantillon dans le TEM et aller à un trou dans l’échantillon pour le tuning de GIF en utilisant le trackball ou le joystick du microscope.
- Passez à l' Énergie filtré TEM (EFTEM) dans et/ou de logiciel DM DM et sélectionnez caméra CCD en DM.
- Choisissez l’agrandissement pour l’analyse élémentaire (le grossissement dépend de la structure/zone d’intérêt, en l’espèce, le grossissement est réglé de 76 k jusqu'à 125 k). 0,001 s et commencer à voirla valeur du temps d’exposition.
- Focaliser le faisceau soigneusement jusqu'à ce que les bords sont visibles et centrer la poutre dans l’image. Réduire ouverture C2 / l’objectif jusqu'à ce que les bords de l’ouverture sont visibles et centre de la poutre avec le trackball pour la poutre.
- Défocalisation de la poutre (autour de C2 46,2 %, durée d’exposition pour l’accord devrait être autour de 0,05 s) et trouver le zéro pic de perte (ZLP) en appuyant sur le bouton ZLP . Démarrer la procédure de réglage en appuyant sur le bouton Full tune .
- Obtenez des points aléatoires de l’acquisition.
- Revenir au TEM mode d’imagerie et de sélectionner la caméra et de vérifier le focus à un grossissement de 10 k (ajuster l’axe z et défocalisation à zéro).
- Sélectionnez LM 75 X grossissement et vérifiez si l’appareil n’est pas insérée (autrement il ne peut pas être contrôlée par SerialEM software).
- Lancez SerialEM et ouvrez un nouveau projet.
- Ouvrez le navigateur. Vérifiez les paramètres de la caméra en caméra & contrôles de Script (pour affichage et enregistrement) et démarrer l’affichage (caméra est inséré automatiquement).
- Pour réserver un coin carte de la section ultra-mince, choisissez ajouter des points dans la fenêtre du navigateur.
- Définissez les points d’angle sur les bords d’une section ultra-mince. Déplacer la scène à un point d’angle en maintenant enfoncée la touche de souris-droite. Une fois atteint un angle de la section, ajoutez un point d’angle avec la souris-clic-gauche. Répétez la procédure pour ajouter des points d’angle plus 3. Assurez-vous que dans la fenêtre de navigateur la boite C pour les points d’angle est cochée pour les points stockées.
- Pour commencer le montage de coin (préférable à un grossissement de 75 LM), aller au navigateur dans la barre de menus SerialEM et choisissez Montaging & grilles et Montage d’angle Setup dans le menu déroulant.
Remarque : Le meilleur ajustement des réglages pour les paramètres d’enregistrement choisies est affiché. Changez-les si nécessaire. En l’espèce, seul le pourcentage de chevauchement a été changé à 20 %.
- Aperçu de la section/ROI sur l’image du montage. Choisissez Ajouter polygone dans la fenêtre du navigateur et esquisser la section en droit-clics multiples. Choisissez ajouter grille de points dans le menu déroulant navigator et définir une distance entre les points (la distance dépend la question expérimentale ; e.g., 10 µm, Figure 3).
- Suivez les commandes de script. Entrez le nombre de points de grille comme indiqué dans le navigateur et entrez le nombre de points d’acquisition (e.g., 20).
- Définissez le seuil de l’illumination afin que le script peut éviter les barres de la grille. Suivez les commandes de script et de déplacer la scène manuellement pour couvrir le champ de vision d’un quart avec un grille-bar. Selon la valeur affichée, entrez une valeur d’éclairement de seuil (devrait être supérieure à la valeur affichée).
- Attendez que les points d’acquisition sont sélectionnées et la routine de la cuisson est terminée.
Remarque : Cela prend beaucoup de temps et peut être du jour au lendemain ; le script envoie le microscope en veille après avoir terminé la cuisson routine ; la routine de cuisson est importante pour stabiliser les sections dans le faisceau d’électrons.
- L’énergie filtré l’analyse élémentaire de TEM (EFTEM)
- Choisissez le mode EFTEM TIA/DM et sélectionnez caméra CCD en DM.
- Centrez le faisceau et suivre les commandes de script qu’empruntera la scène à chaque coordonnée d’échantillonnage.
Remarque : L’utilisateur sera demandé si le stade doit être déplacé à la prochaine coordonnée. - Aligner le zéro pic de perte (ZLP) et la valeur C2 ~ 46,2 %.
- La valeur de l’énergie à 60 eV (dans ce cas, la valeur est définie pour la détection de la Fe M-edge), fente au moment 10 eV et l’exposition à 0,4 s. insérer la fente, puis affectez-lui C2 ~ 43,9 %.
- Démarrer l’affichage et mettre l’image.
- La valeur de C2 à 46,2 %, aligner ZLP et la valeur de C2 à 43,9 %.
- Acquérir la carte élémentaire avec des paramètres spécifiques élémentaires.
Remarque : Les paramètres par défaut peuvent être utilisés dans la boîte de dialogue acquisition, mais il est recommandé de tester ces au préalable, si possible ; exemple filtré image sont affichées à la Figure 4. - La valeur de C2 à 46,2 % et acquérir la carte de l’épaisseur.
- Répétez pour tous les points d’acquisition déterminés (étape 8.3.2-8.3.12).
- Suivez les instructions du script et obtenez les micrographies unique ou les montages des points acquisition. Choisissez le grossissement afin que toutes les (ultra-) informations structurelles soient visible/identifiable.
- Enregistrer fichier LOG, navigator et fenêtre de résultats et enlever la fente.
- Fermer le robinet, retirer l’échantillon et fermer le TEM.
Résultats
Nous décrivons ici un flux de travail pour sélectionner les zones pour TEM de façon automatique et non biaisée. L’utilisateur sélectionne la zone d’intérêt dans une section ultra mince sous le TEM et utilise plusieurs solutions logicielles pour le flux de travail y compris notre logiciel RPS qui calcule automatiquement les coordonnées des 20 zones d’échantillonnage au sein de la zone de intérêt. Alors, la scène TEM est déplacée à chaque zone de prélèvement pour la photographie. Cela est possible pour l’analyse des sections unique tant pour l’analyse des paires des sections, une distance connue, qui permet la sélection des zones pour l’imagerie sans le biais de l’enquêteur.
Ce flux de travail s’est avéré précieux documenter les caractéristiques ultrastructurales dans le cerveau de souris pour les sections4,5. Dans ces études, la couche de cellules polymorphes du gyrus denté (DGpl) a été examinée dans un domaine d’intérêt. Notre méthode s’est avérée appropriée pour l’examen de la DG, parce qu’une section coronale de la dorsale DG (−1, 3 Bregma) s’inscrit dans une section ultra-mince et ses contours sont facilement reconnaissables à faible grossissement. Si d’autres régions du cerveau devaient être examinée, il serait important de vérifier leur taille et caractéristiques distinctives avant les expériences de planification.
Appliquant le workflow décrit ici a montré que le logement de EE augmente la largeur de la fente synaptique significativement à la DGpl4, en ce qui concerne les conditions de logement standard. En outre, le nombre de vésicules de noyau dense a diminué significativement dans les animaux logés en EE4, indiquant les changements dans le ménage des neuropeptides. Lorsque des souris knockout WT et NPY ont été détenus dans des conditions EE, le nombre de neurones/µm³ dans la DGpl augmenté tandis que la densité du DCV a diminué par rapport à la SE-logement5. En revanche, knockout NPY a entraîné une augmentation significative des vésicules synaptiques dans le pool de réserve (fin des vésicules synaptiques) indépendant du logement conditions5. L’effet de l’ere sur la largeur de la fente synaptique a été renversé dans le groupe NPY KO, ce qui entraîne une différence significative entre EE logées WT et NPY KO souris5. Pris ainsi que des études comportementales de WT et NPY animaux knock-out tenue en vertu de ces deux types de conditions de logement, ce qui indique un rôle crucial de NPY les effets neurobiologiques des EE5.
Pour visualiser le fer stocké dans le cerveau humain, un script a été compilé pour le logiciel SerialEM de choisir au hasard les points d’acquisition de dans une section entière ultramince, dans le but d’obtenir une énergie filtré micrographe à chacun de ces points . Après avoir chargé la section dans le microscope électronique, le script interprété la stabilisation de la section (i.e., la routine « spécimen-cuisine ») pour la nuit. Ensuite, le script sélectionné au hasard quelques points prédéfinis par l’utilisateur à partir de celles qui sont marquées avec un caillebotis à la Figure 3. En produisant une micrographie de test et vérifier ses niveaux de gris, le script a vérifié si les points choisis étaient situés sur une partie visible de la section, en rejetant les points qui ont atterri sur les barres. La plupart des flux de travail a été gérée par le script avec une interaction minimale de l’utilisateur. Cependant, il n’était pas possible d’enregistrer automatiquement les micrographies énergie filtrée (avec cette configuration), car il y avait trop peu de lumière pour la routine de mise au point automatique de SerialEM. Par conséquent, nous sommes passés à DM logiciel dès que le script avait déplacé la scène à chaque point, régler la mise au point à la main et fait une image EFTEM de fer. Un exemple d’une image de fer EFTEM du cerveau humain post-mortem est illustré à la Figure 4.
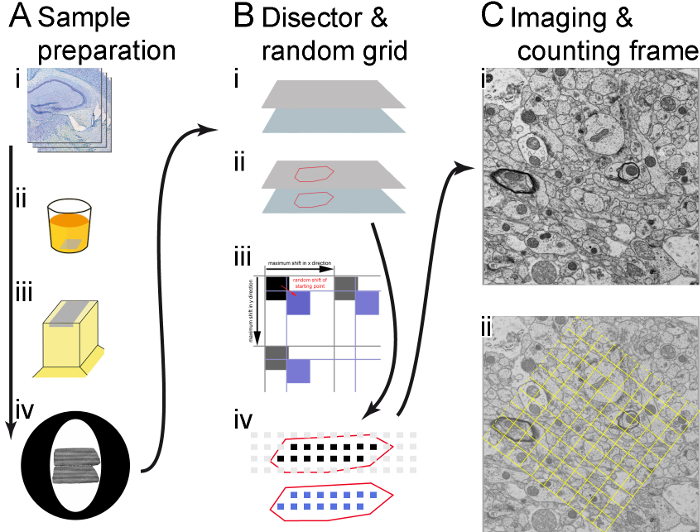
La figure 1. Illustration du flux de travail. (A) les principales étapes de la préparation de l’échantillon sont indiqués (dans le sens de la flèche) : vibratome sections (i), embedding(ii), couper les sections semi minces et ultra-minces (iii) et attraper les paires des sections ultra-minces sur des grilles de cuivre (iv). (B) l’utilisation du fournisseur microscope logiciel sur mesure RPS logiciel et créer des coordonnées de zone d’échantillonnage qui déterminent les sites d’enregistrement. Les points d’angle des sections sont stockés dans le logiciel de la microscopie et le retour sur investissement est décrit (identifié avec l’aide des sections minces semi) (i) ; une grille de zones d’échantillonnage est générée sur le retour sur investissement, un changement aléatoire (flèche rouge) est introduit dans le x et y. Le changement est plus petit que la distance entre les zones d’échantillonnage, (flèches noires), afin qu’au lieu des carrés noirs, qui n’ont pas de changement, les carrés bleus, qui sont déplacées au hasard, sont utilisées pour déterminer les sites d’enregistrement (ii) ; seules les zones d’échantillonnage dans les frontières du ROI (tiré comme des carrés noirs et bleus) sont utilisés pour l’enregistrement. Micrographies sont faites dans la section de référence (carrés noirs) et dans les emplacements correspondants dans la section de recherche (cases bleues) (iii). (C) une micrographie est faite à chaque zone d’échantillonnage à un grossissement suffisant ; un montage composé de plusieurs images fusionnées est effectué si nécessaire (dans ce cas, les images ont été fusionnés ensemble de 2 x 2) (i) ; un cadre de comptage est généré et montré en surimpression sur l’image, les structures dans le cadre et sur les «lignes d’acceptation» (lignes en pointillés cadre) seront comptés, mais pas ceux sur les «lignes interdites» (lignes de trame solide) (ii). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
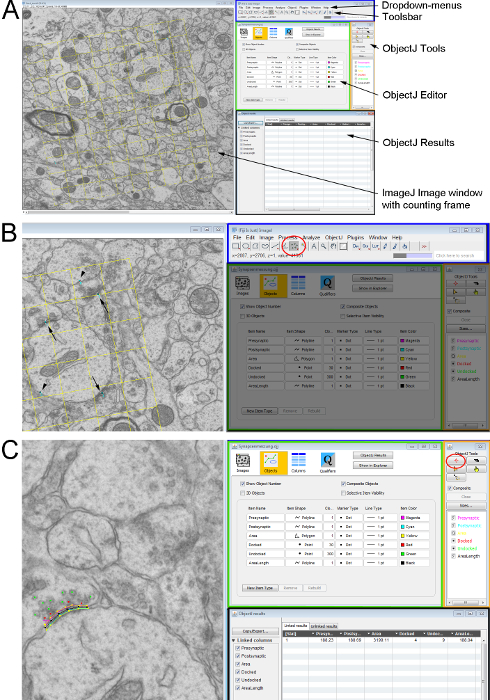
Figure 2 . Mesure des paramètres ultrastructurales sur une paire de sections ultra-minces avec une trame de comptage non biaisée. (A) vue d’ensemble des fenêtres ImageJ utilisé pour compter et analyser les structures cellulaires. Côté gauche montre la micrographie électronique avec l’armature de comptage en surimpression. Sur le côté droit : l’interface utilisateur de ImageJ (rectangle bleu) avec le menu déroulant (en haut) et la barre d’outils (ci-dessous) ; l' éditeur d’objets de ObjectJ (rectangle vert) ; la fenêtre d’Outils d’ObjectJ (rectangle orange) où les outils de mesure peuvent être sélectionnés et la fenêtre de résultats de ObjectJ (rectangle noir) où toutes les mesures sont documentés. (B) détail d’une montrant les synapses marquées sur la microscopie électronique (synapses sur les deux sections - flèches ; synapse uniquement sur les section de recherche-pointes de flèche). Ceux qui sont marqués à l’aide de l’outil Multipoint (cercle rouge) de la barre d’outils dans ImageJ. (C) les noms et les paramètres de mesure des caractéristiques synaptiques sont définies en utilisant les Outils de ObjectJ. Marqueur-outil et la fonctionnalité d’intérêt sont sélectionnés et le plan dessiné à l’image de gauche-clics de souris le long de la structure. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 3. Sélection de zones pour la cartographie élémentaire dans un retour sur investissement pour la microscopie électronique à transmission énergétique filtré d’échantillonnage. SerialEM est utilisé pour sélectionner une région d’intérêt et de proposer un grand nombre de zones d’échantillonnage à un espacement régulier (rose croix dans les images de A et B). Un script puis sélectionne au hasard des zones d’échantillonnage (à partir de ces coordonnées prédéfinies) pour l’acquisition. Les zones d’échantillonnage avec éclairage pauvre sont rejetés pour éviter des barres et des arrêts de script dès 20 zones de bien éclairée d’échantillonnage ont été sélectionnés dans le retour sur investissement. (A) vue d’ensemble de toute la zone d’intérêt, décrite par un polygone, avec un grand nombre de zones d’échantillonnage possible marquée avec x. (B) détail de l’A montrant les zones d’échantillonnage avant sélection aléatoire. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 4. Exemple d’acquisition de carte élémentaire (EFTEM). (A) champ lumineux TEM électronique des environs de point d’acquisition. Une zone sélectionnée au hasard a été utilisée pour obtenir un plan élémentaire et est marquée par un rectangle noir. (B) EFTEM edge pré image fenêtre réalisé dans le domaine choisi au hasard. (C) EFTEM post-bord image de fenêtre fait à la même zone. (D) le plan élémentaire de fer de la même région (M-edge). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Le flux de travail présentée ici permet au chercheur d’obtenir des données sur les caractéristiques ultrastructurales de façon impartiale. C’est beaucoup moins de temps que les enquêtes de volume de coupes sériées. Plusieurs applications différentes sont utilisées pour atteindre cet objectif. Dans un premier temps, notre logiciel RPS sur mesure (pour plus de détails sur la disponibilité, veuillez contacter l’auteur-correspondant) est utilisé pour introduire un décalage aléatoire-étape pour sélectionner les coordonnées de zone d’échantillonnage. Cela permet un échantillonnage aléatoire systématique et uniform du ROI. Ensuite, pour le dépouillement de structures spécifiques, nous avons adapté la méthode de disector, où sont comparées les 2 sections consécutives avec distance connue, d’une manière nouvelle par rapport aux précédentes études13,14,15 , que nous avons utilisé notre logiciel RPS sur mesure pour un échantillonnage aléatoire systématique uniform. Cela fait gagner du temps par rapport à la 3D-reconstruction des volumes entiers de coupes sériées. Le logiciel RPS sur mesure est développé spécifiquement pour un type de microscope qui est un facteur limitant pour la reproduction du flux de travail. Une alternative de ce logiciel spécifique serait une application qui permet aux scripts et est compatible avec d’autres modèles de microscope.
Nous avons utilisé avec succès cette approche pour nos études comparatives4,5. Sur les sections ultra-minces de tissu neuronal, la zone d’intérêt a été exposée et images ont été prises par un échantillonnage aléatoire systématique et uniform dans ce domaine. Il est à noter que la zone d’intérêt, la couche polymorphe du gyrus denté, est une zone assez petite pour enquêter sur qui pourrait être bénéfique pour notre approche. Dans un disector placé au hasard, nous avons évalué le nombre de DCV et plusieurs caractéristiques ultrastructurales des synapses dans le guidage de souris adultes logés dans la SE et EE, en plus d’adulte WT souris par rapport à des souris knockout NPY adultes. Grâce à notre approche, les données recueillies ont montré des changements dans certains des paramètres étudiés. Ces résultats confirment ceux d’autres études similaires chez les jeunes animaux2.
L’inconvénient de l’utilisation expérimentale de ce flux de travail peut être que cette approche multi-demande n’est pas idéale en termes de facilité d’utilisation, comme les utilisateurs doivent se familiariser avec différentes interfaces utilisateur (dans notre cas, l’interface utilisateur, Section Serial TEM le logiciel RPS et le logiciel SerialEM). Apprendre à gérer toutes les demandes de façon efficace prend du temps et doit être pris en compte. Cependant, investir dans le temps à apprendre à utiliser ce flux de travail est toujours clairement favorable au fil du temps qui est nécessaire pour analyser des volumes entiers avec serial section TEM. La méthode d’utilisation d’un disector passée un échantillonnage aléatoire systématique et uniform dans la zone d’intérêt est suffisante pour présenter des données fiables1 sans la nécessité d’enquêter sur une grande quantité de sections/volume.
Afin de maximiser les résultats de nos études, il était vital de prendre bien soin durant la préparation de l’échantillon, comme la préservation des tissus et les structures n’est pas seulement indispensable pour l’évaluation des caractéristiques structurelles, mais aussi pour identifier le domaine d’intérêt sans ambiguïté. Un facteur crucial et peut-être un autre inconvénient de cette méthode, est qu’il faut une haute qualité des paires ultra minces : il ne doit y avoir aucune perforation ou les rides qui couvrent la zone incriminés dans une des sections, et l’épaisseur de coupe doit être maintenue homogène. Le chercheur doit être bien formé en ultramicrotomy. Soin doit être pris lors de l’imagerie, les sections dans le TEM, comme les sections sont sensibles aux dommages de faisceau d’électrons et peuvent facilement déchirer. En outre, il est important de choisir le bon nombre de zones d’échantillonnage dans le retour sur investissement. Selon le but expérimental, le grossissement de la microscopie électronique doit être définie avec précaution. Pour nos expériences spécifiquement, comptage des synapses dans le système nerveux central, 20 régions d’intérêt sur une partie avec la zone de 30,25 µm2 sont optimales. Il est conseillé de former le personnel bien en reconnaissant les caractéristiques en question (dans notre cas de synapses, fonctions synaptiques et DCV) pour obtenir des résultats fiables. Afin d’identifier les synapses, les vésicules synaptiques doivent être identifiables et cela nécessite une résolution d’au moins 10 nm. Pour ce faire, un grossissement de 5000 X était optimal, mais il est à noter que le grossissement dépend de paramètres matériels tels que le type et la position des caméras et devra être adapté pour d’autres types de microscope ou l’appareil photo. Il faut également noter que le protocole utilise des applications spécifiques à une TEM et que les utilisateurs avec d’autres modèles devront examiner les différences dans les paramètres.
Nous croyons que notre flux de travail peut être adapté pour de nombreuses autres applications non seulement en neurosciences, mais dans un large champ de sciences biologiques et sciences des matériaux (lorsque la haute résolution d’un TEM est nécessaire) chaque fois que la question de la recherche exige un uniforme systématique un échantillonnage aléatoire et la quantité d’échantillons à examiner demande un moyen efficace de temps d’analyse. Par exemple, nous nous intéressons actuellement dans la localisation des réserves de fer dans le cerveau humain. Pour ce faire, nous avons récemment adapté notre flux de travail, afin de permettre l’analyse élémentaire sur les sections ultra-minces dans des domaines choisis au hasard. Afin de minimiser le nombre de demandes qui sont nécessaires pour le flux de travail, nous avons cherché à appliquer en utilisant le logiciel de SerialEM seulement, car il peut être programmé pour passer à l’étape de pré-définir des points qui peuvent être sélectionnés de façon aléatoire. Nous avons créé des scripts personnalisés pour contrôler le TEM, dans le but d’automatiser le flux de travail entièrement. Cela s’avère possible, à l’exception de l’autofocus en mode imagerie filtré, qui n’ont pas donné de résultats satisfaisants. Ainsi, nous avons utilisé logiciel DM pour mise au point et pour obtenir les images filtrées énergie.
En résumé, nous présentons des solutions logicielles qui aident à obtenir les micrographies de manière impartiale.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Financé par le numéro de projet Fonds autrichien des sciences, FWF, P 29370 B27
matériels
| Name | Company | Catalog Number | Comments |
| Pentobarbital | SigmaAldrich | P3761 | |
| Formaldehyde | Merck | 1040051000 | 1kg |
| Glutardialdehyde | Science Services | E 16210 | 25%; 100ml; EM grade |
| cacodylate buffer | Merck | C4945 | 250g; Dimethylarsinic acid sodium trihydrate |
| Thionine acetate/Ceristain | Merck | 861340 | |
| acetic acid | Merck | 1000631000 | 1 L |
| Sodium hydroxide | Merck | 1064951000 | 1 kg, pellets |
| osmium tetraoxide | Science Services | E 19110 | 10x1g |
| TAAB embedding resin | Science Services | TAT001 | 500g |
| DMP-30 | Science Services | TAD024 | 100g |
| DDSA | Science Services | TAD025 | 500g |
| Uranyl acetate dihydrate | Plano GmbH | 19481 | depleted, 25g |
| Ultrastain 2 | Leica | 16707235 | Lead citrate |
| Toluidine blue solution | Agar Scientific | AGR1727 | 10g |
| Pioloform | Plano GmbH | R1275 | 10g Powder |
| Proylenoxide | SigmaAldrich | 82320-1L | 1L |
| DPX embedding medium | Plano GmbH | R1320 | embedding medium for semi-thin sections on glass slide, 50 ml |
| Vibratome, Leica VT 1000 | Leica Microsystems, Vienna, Austria | ||
| Leica Ultracut UCT, ultramicrotom | Leica Microsystems, Wetzlar, Germany | ||
| Tecnai G2 20 | FEI,Eindhoven, Netherlands | ||
| Megaview wide angle camera | Olympus Soft Imaging Solution, Münster, Germany | ||
| US 1000 digital camera | Gatan, Pleasanton, USA | ||
| TEM Imaging Analysis Software | FEI,Eindhoven, Netherlands | ||
| FEI Serial Section Software | FEI,Eindhoven, Netherlands | ||
| Fiji, ImageJ 1.52e | National Institute of Health, USA | ||
| SPSS 20.0 | SPSS Inc., Chicago, IL, USA | ||
| SerialEM | Regents of the University of Colorado | ||
| RPS (random point sampling) software 0.9a | custom-made | ||
| Disector v1.0.2 (ImageJ macro) | custom-made | ||
| EFTEMSerialEM (SerialEM script) | custom-made |
Références
- Howard, V., Reed, M. . Unbiased Stereology: Three-Dimensional Measurement in Microscopy. , (1998).
- Nakamura, H., Kobayashi, S., Ohashi, Y., Ando, S. Age-changes of brain synapses and synaptic plasticity in response to an enriched environment. Journal of Neuroscience Research. 56 (3), 307-315 (1999).
- Landers, M. S., Knott, G. W., Lipp, H. P., Poletaeva, I., Welker, E. Synapse formation in adult barrel cortex following naturalistic environmental enrichment. Neuroscience. 199, 143-152 (2011).
- Reichmann, F., et al. A novel unbiased counting method for the quantification of synapses in the mouse brain. Journal of Neuroscience Methods. 240, 13-21 (2015).
- Reichmann, F., et al. Environmental enrichment induces behavioural disturbances in neuropeptide Y knockout mice. Scientific Report. 6, 28182 (2016).
- Mayhew, T. M. Taking Tissue Samples from the Placenta: An Illustration of Principles and Strategies. Placenta. 29, 1-14 (2008).
- Ferguson, S., Steyer, A. M., Mayhew, T. M., Schwab, Y., Lucocq, J. M. Quantifying Golgi structure using EM: combing volume-SEM and stereology for higher throughput. Histochemistry and Cell Biology. 147, 653-669 (2017).
- Sterio, D. C. The unbiased estimation of number and sizes of arbitrary particles using the disector. Journal of Microscopy. 134 (2), 127-136 (1984).
- Gundersen, H. J., et al. The new stereological tools: disector, fractionator, nucleator and point sampled intercepts and their use in pathological research and diagnosis. APMIS. 96 (10), 857-881 (1988).
- Franklin, K. B. J., Paxinos, G. . The Mouse Brain in Stereotaxic Coordinates. , (2008).
- Lewis, P. R., Knight, D. P. . Staining Methods for Sectioned Material. , (1988).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. J Struct Biol. 152 (1), 36-51 (2005).
- Rampon, C., Tang, Y. P., Goodhouse, J., Shimizu, E., Kyin, M., Tsien, J. Z. Enrichment induces structural changes and recovery from nonspatial memory deficits in CA1 NMDAR1-knockout mice. Nature Neuroscience. 3 (3), 238-244 (2000).
- Xu, X., Ye, L., Ruan, Q. Environmental enrichment induces synaptic structural modification after transient focal cerebral ischemia in rats. Experimental Biology and Medicine. 234 (3), 296-305 (2009).
- Lonetti, G., et al. Early environmental enrichment moderates the behavioral and synaptic phenotype of MeCP2 null mice. Biological Psychiatry. 67 (7), 657-665 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon