Method Article
Enrichissement de particules de lipoprotéines indigènes avec microRNA et détermination subséquente de leur teneur absolue/relative de microRNA et de leur taux de transfert cellulaire
Dans cet article
Résumé
Ici, un protocole quantitatif en temps réel basé sur la réaction en chaîne de la polymérase est présenté pour la détermination de la teneur en micro-ARN native (absolue/relative) des particules de lipoprotéines. En outre, une méthode pour augmenter le niveau de micro-ARN, ainsi qu’une méthode pour déterminer le taux d’absorption cellulaire des particules de lipoprotéines, est démontrée.
Résumé
Les particules de lipoprotéines sont principalement des transporteurs de lipides et de cholestérol dans la circulation sanguine. En outre, ils contiennent de petites quantités de brins de microRNA non codant (miRNA). En général, miRNA modifie le profil d’expression protéique en raison des interactions avec l’ARN messager (mRNA). Ainsi, la connaissance de la teneur en miRNA relative et absolue des particules de lipoprotéines est essentielle pour estimer l’effet biologique de l’absorption des particules cellulaires. Ici, un protocole quantitatif en temps réel de réaction en chaîne de la polymérase (qPCR) est présenté pour déterminer la teneur absolue en miRNA des particules de lipoprotéines — illustré pour les particules de lipoprotéines natives et enrichifiées par le miRNA. Le contenu miRNA relatif est quantifié à l’aide de cartes multipuits microfluidiques. En outre, ce protocole permet aux scientifiques d’estimer le miRNA cellulaire et, par conséquent, le taux de capture des particules lipoprotéiques. Une augmentation significative du niveau de miRNA cellulaire est observable lors de l’utilisation de particules de lipoprotéines de haute densité (HDL) chargées artificiellement avec le miRNA, tandis que l’incubation avec des particules de HDL indigènes ne donne aucun effet significatif en raison de leur teneur assez faible en miRNA. En revanche, l’absorption cellulaire des particules de lipoprotéines de basse densité (LDL) — ni avec le miRNA natif, ni avec elle, ne modifie pas le niveau de miRNA cellulaire.
Introduction
Les particules de lipoprotéines sont composées d’une monocouche de lipides amphiphiles et de cholestérol renfermant un noyau d’esters cholestérylés et de graisses triglycérides. La particule entière est stabilisée par des apolipoprotéines intégrées à la membrane, qui définissent la fonctionnalité biologique de la particule. Les particules de lipoprotéines peuvent être distinguées en fonction de leur densité croissante respective et, par conséquent, de la taille décroissantes, à savoir les lipoprotéines de très basse densité (VLDL), les lipoprotéines de densité intermédiaire (IDL), les LDL et les particules de HDL. Malgré le transport de composants insolubles dans l’eau dans la circulation sanguine, il a été démontré que les particules de HDL transportent des brins non codant de mirna1,2. Les micro-RNAs sont une classe de brins d’ARN courts (généralement deux douzaines de nucléotides), qui dégradent les brins d’ARNm complémentaires intracellulares et, par conséquent, modifient le profil d’expression de certaines protéines3,4,5, le 6. En outre, les modifications du profil miRNA ont été trouvées dans une variété de maladies et, par conséquent, le profil est applicable en tant que biomarqueur pour le diagnostic et le pronostic. Le transport extracellulaire de miRNAs entre les cellules par l’intermédiaire de particules de lipoprotéines peut servir de mécanisme supplémentaire pour la modulation de niveau d’ARNm intercellulaire. Pour estimer quantitativement l’effet biologique, il est nécessaire de connaître la teneur en miRNA absolue et relative des particules de lipoprotéines.
La PCR quantitative en temps réel est une méthode appropriée et relativement rapide pour obtenir cette information. Par conséquent, la valeur relative de quantification (RQ) peut être calculée, et les différences relatives entre les différents échantillons et les fractions de lipoprotéine sont estimables. Les cartes multipuits microfluidiques sont une méthode rapide et facile à utiliser pour déterminer la présence relative (équivaut à la valeur RQ) des miRNAs dans un échantillon. Les cartes multipuits microfluidiques sont constituées de 96 ou 384 chambres de réaction individuelles pour les réactions de qPCR individuelles incorporées dans un dispositif microfluidique. Chaque chambre contient la sonde d’hydrolyse requise et des amorces spécifiques de qPCR pour un miRNA individuel. Les avantages sont un temps de manipulation court en raison de la normalisation, un workflow simple, et un nombre réduit d’étapes de pipetage. De plus, le volume d’échantillonnage requis est minimisé. Contrairement à la quantification relative, le contenu absolu de miRNA nécessite une comparaison des résultats de l’échantillon de qPCR avec des courbes standard de nombres absolus connus de brins de miRNA. Il convient de noter que, en raison de leur teneur relativement faible en miRNA, la norme et, en outre, même les techniques d’imagerie sensibles à une seule molécule ne sont pas réalisables — l’enrichissement artificiel des particules de lipoprotéine avec miRNA est inévitable pour étudier les cellules cellulaires l’interaction des particules de lipoprotéines et le transfert de miRNA. En ce qui concerne cette, la déconcentration de la particule de HDL suivie de la relipidation subséquente7 permet l’incorporation de et, par conséquent, l’enrichissement avec des brins de mirna. L’enrichissement similaire des particules de LDL avec le miRNA n’est pas réalisable en raison de l’hydrophobicité de la protéine apoB-100, qui est le constituant principal de la particule de LDL. Cependant, par addition du diméthyl sulfoxyde de solvant polaire (DMSO), qui est capable de pénétrer les membranes lipidiques, les particules de LDL peuvent être chargées artificiellement avec des brins de miRNA aussi bien.
La microscopie à force atomique à grande vitesse (HS-AFM) est un outil puissant pour la caractérisation des spécimens biologiques offrant une résolution temporelle de subnanomètre spatiale et sous-seconde8. Par conséquent, il s’agit d’une technique bien adaptée pour le contrôle de la qualité des particules de lipoprotéines modifiées, car les particules de lipoprotéines natives/reconstituées/étiquetées peuvent être imagées dans un environnement quasi physiologique.
Ici, un protocole basé sur qPCR est présenté étape par étape pour déterminer la teneur absolue/relative en miRNA des particules de lipoprotéines et des échantillons de cellules, ce qui permet une estimation du taux de capture des particules de lipoprotéines cellulaires. En outre, une méthode pour l’enrichissement des particules de lipoprotéine avec miRNA est démontrée. Cette méthode peut être adaptée pour la manipulation générale de la teneur en lipoprotéines et, par conséquent, démontre l’applicabilité des particules de lipoprotéines comme cibles pour la délivrance de médicaments.
Protocole
Les dons de sang ont été approuvés par le Comité d’éthique de l’Université médicale de Vienne (EK-Nr. 511/2007, EK-Nr. 1414/2016). La nomenclature est conforme à l’information minimale pour la publication d’expériences quantitatives en temps réel de PCR (MIQE)9 lignes directrices.
1. isolement des particules de lipoprotéine du sang humain
- Prérefroidir un ultracentrifuge à 4 ° c. Prélever du sang de volontaires sains après un jeûne de nuit.
NOTE: les trois donneurs sont généralement requis, faisant un don de 80 mL chacun, et des tubes de prélèvement sanguin contenant de l’acide éthylenediaminetétraacétique (EDTA) comme anticoagulant. - Centrifuger à 2 000 x g pendant 20 min à 4 ° c et récolter le plasma (phase supérieure); éviter les forces de cisaillement. Déterminez le volume total V et ajustez, si nécessaire, un multiple du volume du tube de centrifugation à l’aide d’une solution saline tamponnée au phosphate (PBS). Mesurez la masse de 1 mL 3x et calculez la densité moyenne ρ.
- Calculer la quantité requise de bromure de potassium (KBr) pour l’ajustement de la densité avec l’équation10 suivante (pour la densité désirée en grammes/millilitre, utiliser A = 1,019, et pour le volume spécifique
 de KBr, utiliser = 0,364 ml/g). Ajouter KBr au plasma et remuer doucement pour éviter les forces de cisaillement jusqu’à ce que KBr soit totalement dissous.
de KBr, utiliser = 0,364 ml/g). Ajouter KBr au plasma et remuer doucement pour éviter les forces de cisaillement jusqu’à ce que KBr soit totalement dissous.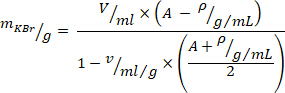
- Mesurez la densité comme décrit à l’étape 1,2 et ajustez de nouveau, si nécessaire, en ajoutant plus de tubes de centrifugation KBr. Fill et Seal appropriés pour l’ultracentrifution avec le plasma. Évitez les bulles d’air; Sinon, le tube peut s’effondrer. Placer les tubes dans le rotor selon les instructions du fabricant et centrifuger à 214 000 x g pendant 20 h à 4 ° c.
- Ouvrir les tubes selon les instructions du fabricant et jeter la phase supérieure contenant des VLDL et des IDL. Déterminez le volume total V et ajustez, si nécessaire, un multiple du volume du tube de centrifugation à l’aide de PBS.
- Déterminez la densité ρ de la fraction inférieure. Calculez la quantité requise de KBr pour le réglage de la densité (utilisez A = 1,063). Remuer doucement pour éviter les forces de cisaillement jusqu’à dissolution du KBr. Répétez l’étape 1,4.
- Retirer et collecter la phase supérieure contenant des particules de LDL. Conserver la solution de particules de LDL sous atmosphère gazeuse inerte à 4 ° c. Déterminez le volume total V et ajustez, si nécessaire, un multiple du volume du tube de centrifugation à l’aide de PBS. Déterminez la densité ρ de la fraction inférieure comme décrit à l’étape 1,2.
- Calculez la quantité requise de KBr pour le réglage de la densité (utilisez A = 1,220) et ajoutez-la. Remuer doucement pour éviter les forces de cisaillement jusqu’à dissolution du KBr. Répétez l’étape 1,4.
- Retirez et collectez la phase supérieure contenant des particules de HDL. Déterminez son volume total V et ajustez, si nécessaire, un multiple du volume du tube de centrifugation à l’aide de PBS. Déterminez la densité ρ. Une deuxième étape de centrifugation de la phase supérieure à 214 000 x g pendant 20 h à 4 ° c est recommandée pour éliminer l’albumine. Si nécessaire, répétez l’étape 1,8. Retirez et collectez la phase supérieure contenant des particules de HDL.
- Préparer et prérefroidir au moins 20 L de tampon de dialyse (0,9% de NaCl, 0,1% d’EDTA [pH 7,4]) à 4 ° c. Tubes de dialyse préhumides (coupure de poids moléculaire: 12 – 14 kDa) et ajouter la solution de particules de LDL et de HDL selon les instructions du fabricant. Dialysez contre 5 L de la mémoire tampon de dialyse à 4 ° c et changez la mémoire tampon après 1, 2 et 4 h.
- Après 24 h, récupérez les solutions de particules de lipoprotéine des tubes de dialyse et déterminez la concentration de protéine utilisant le dosage de Bradford11 ou un autre approprié. Stockez les solutions de particules de HDL et de LDL sous atmosphère gazeuse inerte à 4 ° c.
2. aliquotes de miRNA synthétiques
Remarque: lorsque vous manipulez des oligonucléotides d’ARN, travaillez sans RNase. Ne travaillez qu’avec des consommables en plastique frais et jetables et portez toujours des gants, qui doivent être changés fréquemment. N’utilisez que des solutions exemptes de nucléases. Toujours travailler sur la glace.
- Faire tourner le flacon acquis du fabricant à la force maximale pour former une pastille de miRNA synthétique lyophilisé. Ajouter un volume approprié de 10 mM de tris (hydroxyméthyl) aminométhane (TRIS) tampon, pH 7,5, pour une concentration finale de 10 μM (concentration en stock) miRNA.
- Pipetter doucement de haut en bas quelques fois pour la remise en suspension. Préparer des aliquotes de 100 μL chacun dans des tubes stériles. Conservez-les à-20 ° c s’ils ne sont pas utilisés immédiatement. Évitez le dégel et le surgélation répétés.
3. reconstitution des particules de HDL
-
Délipidation
- Préparer le tampon A (150 mM NaCl, 0,01% EDTA, 10 mM Tris/HCl [pH 8,0]). Prérefroidir la centrifugeuse à-10 ° c. Prérefroidir 100 mL d’un mélange d’éthanol: éther diéthylique (3:2) à-20 ° c.
ATTENTION: Portez un équipement de protection individuelle approprié et travaillez dans une hotte tout en manipulant l’éther diéthylique car il est extrêmement inflammable et nocif pour la peau. - Mélanger un volume correspondant à 5 mg de particules de HDL avec 50 mL de mélange d’éthanol prérefroidi: éther diéthylique (3:2) et incuber pendant 2 h à-20 ° c. Centrifuger à 2 500 x g pendant 10 min à-10 ° c.
- Jeter le surnageant, resuspendre le culot dans 50 mL d’éthanol prérefroidi: mélange d’éther de diéthyle par vortexing, et incuber une seconde fois pendant 2 h à-20 ° c. Centrifugez à nouveau à 2 500 x g pendant 10 min à-10 ° c.
Remarque: Si vous le souhaitez, Lyophilisez le surnageant pour une analyse de la teneur en miRNA dans la fraction lipidique des particules de HDL. - Sécher le culot sous le débit de gaz N2 et le remettre en suspension dans 250 μl de tampon A (voir étape 3.1.1). Déterminer la concentration protéique en utilisant le dosage de la protéine Bradford ou un autre approprié et diluer jusqu’à une concentration finale de 1 mg de protéine/250 μL de tampon A.
Remarque: le protocole peut être suspendu ici. Conserver la solution pendant la nuit à 4 ° c sous atmosphère gazeuse inerte.
- Préparer le tampon A (150 mM NaCl, 0,01% EDTA, 10 mM Tris/HCl [pH 8,0]). Prérefroidir la centrifugeuse à-10 ° c. Prérefroidir 100 mL d’un mélange d’éthanol: éther diéthylique (3:2) à-20 ° c.
-
reconstitution
- Préparer une solution d’inventaire de la phosphatidylcholine (PC) à l’aide d’un mélange de chloroforme: méthanol (2:1) à une concentration de 5,6 mg de PC/mL. De même, préparer des solutions d’inventaire d’oléate de cholestéryle (5 mg de CO/mL) et de cholestérol (5 mg de C/mL). Stockez toutes les solutions à-20 ° c.
- Dans un tube de verre propre, mélanger 500 μL de PC, 100 μL de CO et 13,5 μL de C. Ces volumes correspondent à un rapport molaire approximatif de 100 PC: 22 CO: 4.8 C. sécher le mélange sous N2 débit de gaz tout en tournant le tube pour donner une couche de surface homogène.
Remarque: le protocole peut être suspendu ici. Conserver le flacon en verre (si désiré, le stockage est possible) sous atmosphère gazeuse inerte à-20 ° c. - Préparer une solution de spermine fraîche de 30 mM dans le tampon A. mélanger une aliquote (100 μL, 10 μM) de miRNA synthétique (Voir l’étape 2,2) avec 100 μL de solution de spermine et incuber pendant 30 min à 30 ° c.
Remarque: pour les expériences de contrôle négatif, remplacez la solution miRNA et/ou spermine par le même volume de tampon A. - Dissoudre une aliquote de mélange maître PC/CO/C dans le mélange à partir de l’étape 3.2.3.
- Préparer une solution de 30 mg/mL de désoxycholate de sodium dans le tampon A et ajouter 50 μL à la solution à partir de l’étape 3.2.4. Mélanger à 4 ° c pendant 2 h.
- Ajouter 250 μL de la solution HDL delipidatée à l’étape 3.1.4. Ce volume correspond à un rapport molaire approximatif de 100 PC: 22 CO: 4.8 C:1 protéines HDL dépidatés. Remuer à 4 ° c pendant la nuit.
-
dialyse
- Prérefroidir au moins 15 L de PBS à 4 ° c. Ajouter 50 g de billes adsorbées à 800 mL d’eau double distillée (ddH2O) et remuer pendant 1 min. attendre 15 min, décanter le surnageant, et répéter la procédure avec PBS.
- Les cassettes de dialyse préhumides (coupure de poids moléculaire: 20 kDa) ou les tubes de dialyse appropriés et ajouter la solution de l’étape 6, à l’aide d’une seringue selon les instructions du fabricant.
- Ajouter les perles adsorbées de l’étape 3.3.1 à 3 L de PBS et dialyser à 4 ° c. Changez la mémoire tampon et les billes après 1 h et 2 h.
- Après 24 h, récupérez la solution de particules de HDL reconstituée (rHDL) et déterminez la concentration protéique à l’aide du dosage de Bradford. Conserver la solution de particules rHDL sous atmosphère gazeuse inerte à 4 ° c.
4. étiquetage des particules de LDL
- Préparer 10x tampon de LDL (1,5 M NaCl, 3 mM EDTA, 1 mM éthylène glycol-bis (β-aminoethyl éther)-N, N, N', n'-tétraacétique acide [EGTA, pH 7,4]) et le stocker à température ambiante.
- Préparer une solution de spermine fraîche de 30 mM dans de l’eau sans RNase. Mélanger une aliquote (100 μL, 10 μM) de miRNA synthétique (Voir l’étape 2,2) avec 100 μL de solution de spermine et incuber pendant 30 min à 30 ° c.
Remarque: pour les expériences de contrôle négatif, remplacez le miRNA et/ou la solution de spermine par le même volume de 1 tampon LDL. - Ajouter 100 μL de DMSO à la solution miRNA/spermine à partir de l’étape 4,2 et diluer-le plus loin avec 1,2 mL de tampon LDL 1.
- Diluer la solution de particules de LDL à une concentration finale d’environ 4 mg/mL avec du PBS et mélanger 450 μL avec 50 μL de tampon de LDL 10x. Incuber pendant 10 min sur la glace.
- Combiner la solution de particules de LDL de l’étape précédente et la solution miRNA/spermine/DMSO (à partir de l’étape 4,3) et l’incuber pendant 2 h à 40 ° c.
- Effectuer une dialyse similaire comme décrit dans la section 3,3 et stocker la solution de particules de LDL étiquetée en conséquence.
5. contrôle de qualité des particules de lipoprotéines reconstituées/étiquetées
Remarque: pour le contrôle de la qualité, le diamètre et la forme générale des particules de lipoprotéines peuvent être déterminés en utilisant, par exemple, l’AFM ou la microscopie électronique (EM). Ici, HS-AFM est utilisé pour mesurer la distribution de taille des particules de lipoprotéines natives/reconstituées/étiquetées.
- Diluer la solution de particules de HDL/LDL dans PBS (1:100 – 1:1000) et l’incuber sur du mica fraîchement clivé pendant 5 min. Pour le clivage du mica12, appuyez sur le ruban adhésif contre le substrat et enlevez les couches de mica supérieures en tirant la bande.
NOTE: selon l’instrument particulier de l’AFM, la zone d’observation et la concentration initiale des particules, le facteur de dilution doit être ajusté pour observer les particules individuelles. - Après incubation, rincer l’échantillon avec du PBS et effectuer une imagerie HS-AFM en PBS et en mode de taraudage avec des cantileviers ayant des constantes de ressort de kcant ≪ 0,2 N/m. Il est recommandé d’utiliser des tailles de balayage < 1 μM2 et de garder les forces d’imagerie aussi basses que possible.
- Déterminez la hauteur des particules imagées par rapport à la surface de mica avec un logiciel approprié.
- Chargez les données dans Gwynedd (freeware), détectez les particules par l’analyse du grain (Marquez les grains par seuil) et réglez le seuil au-dessus du fond du substrat. Aplatir l’image (Supprimer l’arrière-plan polynôme) en choisissant l’option exclure la région masquée .
- Exportez les valeurs de hauteur des particules détectées (répartition des différentes caractéristiques du grain) et répétez ces étapes pour toutes les images enregistrées. Effectuer une analyse statistique en créant des histogrammes ou en calculant les fonctions de densité de probabilité des hauteurs de particules obtenues.
- Répétez les étapes 5.1 à 5.3 avec des particules de lipoprotéines indigènes ainsi que reconstituées/étiquetées et comparez les résultats obtenus pour vérifier la qualité des particules. Si vous observez des débris et/ou des conglomérats, jetez l’échantillon.
6. culture cellulaire
- Cultiver des cellules adhérentes selon un protocole établi (p. ex., ldlA7-SRBI13) jusqu’à atteindre la confluence.
NOTE: plusieurs chambres indépendantes en fonction du nombre d’expériences (recommandées sont deux chambres par cadre expérimental) et des expériences de contrôle négatif (recommandées sont deux chambres sans addition de particules de lipoprotéines) sont nécessaires. En outre, deux chambres sont nécessaires pour la détermination du numéro de cellule. - Lavez délicatement les cellules 3x avec la solution de sel équilibrée de Hank (HBSS) préchauffée pour éliminer les débris cellulaires et couvrir la couche cellulaire avec un volume approprié de milieu de croissance sans sérum. Ajouter un volume approprié de solution de particules de lipoprotéines pour atteindre une concentration finale de 50 μg/mL de particules de lipoprotéines. Incuber à 37 ° c et 5% CO2 pendant 16 h.
NOTE: selon la conception expérimentale et la lignée cellulaire, le temps d’incubation doit être adapté pour obtenir une augmentation suffisante (c.-à-d. mesurable) du niveau de miRNA cellulaire. - Lavez doucement les cellules 3x avec du HBSS préchauffé (37 ° c) pour éliminer les particules de débris cellulaires/lipoprotéines et couvrez la couche cellulaire avec un volume approprié de milieu sans sérum.
- Déterminer la densité de la cellule à l’aide d’une méthode appropriée (p. ex., hemocytomètre, compteur automatisé de cellules) dans au moins deux chambres indépendantes pour calculer le nombre de cellules dans le volume de l’échantillon à partir de l’étape 6,3. Ce numéro est utilisé pour la normalisation.
7. extraction de miRNA à partir d’échantillons de particules de cellules et de lipoprotéines
Remarque: l’extraction du miRNA des cellules est effectuée à l’aide du kit d’extraction miRNA avec les modifications suivantes.
-
Échantillons de cellules
- Prérefroidir la centrifugeuse à 4 ° c. Ajouter 350 μL de réactif de lyse à deux alvéoles contenant des cellules. En tant qu’expérience de contrôle négatif, utilisez une chambre sans cellules.
ATTENTION: Portez un équipement de protection individuelle approprié et travaillez dans une hotte tout en manipulant le réactif de lyse car il contient du phénol et du thiocyanate. - Attendre 3 à 5 min (en fonction de la lignée cellulaire) pour le détachement de cellules. Si nécessaire, vérifiez le détachement de cellules avec la microscopie en champ clair. Faire la piscine du contenu des deux chambres dans un tube de 1,5 mL.
- À l’aide d’une aiguille de 20 G et d’une seringue de 5 mL, homogénéiser/perturber l’échantillon 5x – 10x par aspiration et incuber pendant 5 min. Ajouter 140 μL de chloroforme (CHCl3), agiter vigoureusement pendant 15 s et incuber pendant 3 min.
- Centrifuger à 12 000 x g pendant 15 min à 4 ° c. Ensuite, arrêtez de refroidir la centrifugeuse.
- Transférer la phase aqueuse supérieure vers un nouveau tube de 1,5 mL; éviter le mélange de phase/contamination car l’interphase contient de l’ADN et la phase inférieure contient des protéines. Ajouter 1,5 x le volume de 100% d’éthanol et mélanger soigneusement par pipetage.
- Placer une colonne de spin dans un tube collecteur de 2 mL et ajouter 700 μL du mélange à partir de l’étape 7.1.5. Centrifuger à 8 000 x g pendant 15 s à température ambiante. Jetez le flux et répétez cette étape avec le volume d’échantillon résiduel.
- Jetez le flux à travers et ajoutez 700 μL du premier tampon de lavage à la colonne de spin. Centrifugez-le à 8 000 x g pendant 15 s à température ambiante.
- Jetez le flux et ajoutez 500 μL du deuxième tampon de lavage à la colonne spin. Centrifugez-le à 8 000 x g pendant 15 s à température ambiante. Répétez cette étape une deuxième fois avec 2 min de temps de centrifugation.
- Placez la colonne de spin dans un nouveau tube collecteur de 2 mL et Centrifugez-la à pleine vitesse pendant 1 min pour sécher la membrane.
- Placez la colonne de spin dans un tube collecteur de 1,5 mL. Ajouter 30 μL d’eau sans RNase au centre de la membrane pour l’élution et la centrifuger à 8 000 x g pendant 1 min. répéter cette étape une deuxième fois avec le premier écoulement à travers comme solution d’élution. L’étape de transcription inversée se fait immédiatement après l’extraction; dans le cas contraire, stockez les échantillons de miRNA extraits à-20 ° c.
- Prérefroidir la centrifugeuse à 4 ° c. Ajouter 350 μL de réactif de lyse à deux alvéoles contenant des cellules. En tant qu’expérience de contrôle négatif, utilisez une chambre sans cellules.
-
Particules de lipoprotéine
- Réglez le volume de l’échantillon avec la plus faible concentration de protéine à 100 μL (= volume d’échantillon maximal). Calculez les volumes d’échantillonnage des autres échantillons en fonction de cette normalisation, inversement à leur concentration. Pour les expériences de contrôle négatif, utiliser 100 μL d’eau sans RNase.
Remarque: la normalisation n’est pas nécessaire, mais simplifie la comparaison directe des résultats au cours de l’étape et de l’analyse de qPCR. - Prérefroidir la centrifugeuse à 4 ° c. Ajouter 700 μL de réactif de lyse au volume d’échantillonnage de l’étape 7.2.1.
- Effectuer l’extraction miRNA selon les étapes 7.1.3 – 7.1.10.
- Réglez le volume de l’échantillon avec la plus faible concentration de protéine à 100 μL (= volume d’échantillon maximal). Calculez les volumes d’échantillonnage des autres échantillons en fonction de cette normalisation, inversement à leur concentration. Pour les expériences de contrôle négatif, utiliser 100 μL d’eau sans RNase.
8. transcription inversée
Remarque: la transcription inverse de miRNA est effectuée à l’aide d’un kit de transcription inversée avec les modifications suivantes.
- Décongeler les réactifs du kit et les amorces de transcription inversée sur la glace. Préparer le mélange maître suivant dans un tube de réaction sur la glace: 45,7 μl de H2O sans rnase, 16,5 μl de tampon de transcription inverse 10x, 11 μL d’enzyme de transcription inversée, 2,1 μl d’inhibiteur de rnase et 1,7 μl de mélange de dNTP. Mélanger doucement, ne pas tourbillonner.
Remarque: l’échelle dépend de la quantité d’échantillon; ici, il est calculé pour 10 réactions. - Étiqueter 0,2 tubes mL en conséquence et mélanger 7 μL du mélange principal de l’étape 8,1 avec 5 μL de l’échantillon extrait de l’étape 7.1.10 et 3 μL d’apprêt de transcription inversée. Mélanger doucement, centrifuger sous peu, et ranger le mélange sur la glace.
- Afin d’utiliser le même numéro de cellule pour chaque ligne de cellule, réduisez le volume de l’échantillon de la ligne de cellule avec un numéro de cellule global plus élevé; utiliser ce volume résiduel pour atteindre le volume total d’échantillon de 5 μL d’eau sans RNase.
Remarque: les échantillons de particules de lipoprotéine sont déjà normalisés à l’étape 7.2.1. Pour la préparation de la courbe standard, diluer une aliquote de miRNA séquentiellement dans de l’eau sans RNase et calculer le nombre de brins par volume d’échantillon. Requis sont au moins cinq points de données dans la plage des valeurs de cycle de quantification d’échantillon (cq) obtenues. Habituellement, les facteurs de dilution totaux allant de 102 à 106 sont appropriés. - Placer les tubes dans la machine thermocycleuse et commencer le programme suivant: 30 min à 16 ° c (pas de recuit), 30 min à 42 ° c (transcription inversée), 5 min à 85 ° c (pas de fusion), et pause à 4 ° c (stockage). Effectuez l’étape qPCR immédiatement après la transcription inversée; Sinon, stockez l’ADN complémentaire (cDNA) synthétisé à partir des échantillons de miRNA à-20 ° c.
9. qPCR
- Effectuer une qPCR de l’ADNc (transcription inversée de miRNA) à l’aide d’un dosage disponible dans le commerce (voir le tableau des matériaux) avec les modifications suivantes.
- Décongeler tous les réactifs (Supermix, sans RNase H2O), les échantillons d’ADNc (à partir de l’étape 8,4) et les amorces sur la glace. Préparer le mélange maître suivant dans un tube de réaction sur la glace: 75 μL de Supermix, 47,5 μL de H2O sans rnase, 7,56 μL d’apprêt. Mélanger doucement, ne pas tourbillonner.
Remarque: l’échelle dépend de la quantité d’échantillon; ici, il est calculé pour 10 réactions. - Étiqueter 0,2 tubes mL en conséquence et ajouter 2 μL de l’échantillon d’ADNc à 13 μL du mélange principal et mélanger doucement. En général, mesurez chaque échantillon 2x.
Remarque: au moins deux échantillons de contrôle négatif sont requis en plus — utilisez de l’eau sans RNase comme échantillon. Si la courbe d’étalonnage n’est pas déterminée dans la même course que l’échantillon, un échantillon de la mesure de la courbe d’étalonnage est également requis. Il est utilisé pour calibrer chaque course individuelle à la même efficacité de réaction. - Placer les tubes dans la machine PCR et commencer le programme suivant: 2 min à 50 ° c, 10 min à 95 ° c, 15 s à 95 ° c, et 60 s à 60 ° c. Répétez les deux dernières étapes du programme jusqu’à 50x.
- Pour une analyse des valeurs de cq avec le progiciel de la machine de PCR, activez la normalisation de dynamictube (pour la compensation des différents niveaux d’arrière-plan utilisant le deuxième dérivé de chaque trace d’échantillon) et pente de bruit Correction (normalisation au niveau du bruit).
Remarque: la valeur de cq du contrôle négatif doit être de plusieurs cycles supérieur à la valeur cq la plus élevée de l’échantillon.- Pour une analyse de la courbe d’étalonnage, déterminez le seuil pour le calcul de la cq pour chaque Mirna à partir des courbes standard de chaque Mirna individuellement, en utilisant la fonction de seuil de recherche automatique du progiciel, et maintenez-la constante pour chaque miRNA spécifique.
- Pour l’analyse d’échantillon, si nécessaire, compenser les différentes efficacités de réaction de l’échantillon exécuté avec le point de données de l’échantillon de courbe d’étalonnage. Le logiciel calcule les valeurs cq des échantillons à partir du niveau de seuil de la mesure de la courbe d’étalonnage respective.
10. calcul du contenu miRNA
-
Courbe d’étalonnage
- Calculer, à partir du nombre initial de brins de miRNA par aliquote (100 μL de miRNA 10 μM, masse moléculaire à partir de la feuille de calcul) et les étapes de dilution série ultérieures, le nombre de brins de miRNA dans le volume de l’échantillon (le volume d’échantillon de 5 μL de l’étape 8,3).
Remarque: en supposant une efficacité de transcription inversée de 1, ce nombre est égal au nombre de brins d’ADNc. - Calculer le nombre de brins d’ADNc dans le volume de l’échantillon de 2 μL à partir de l’étape 9,3. Considérez ainsi le facteur de dilution supplémentaire de 7,5 (le volume d’échantillon de 2 μL de l’étape 9,4 du volume d’échantillon de 15 μL de l’étape 8,4).
- Tracer la valeur de cq à partir de l’étape 9.5.1 par rapport au nombre de brins nbrins calculés à l’étape 2 dans un tracé semilogarithmique de base-10, et ajuster les points de données avec la courbe de régression suivante (M = la pente du linéaire courbe de régression, B = offset).
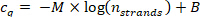
Confirmez que le coefficient de corrélation (R2) de la ligne est > 0,99.
- Calculer, à partir du nombre initial de brins de miRNA par aliquote (100 μL de miRNA 10 μM, masse moléculaire à partir de la feuille de calcul) et les étapes de dilution série ultérieures, le nombre de brins de miRNA dans le volume de l’échantillon (le volume d’échantillon de 5 μL de l’étape 8,3).
-
Particules de lipoprotéine
- Calculer le nombre de brins de miRNA par volume d’échantillon à l’aide de la valeur mesurée de l’échantillon cq (étape) et de l’équation suivante (M et B sont les paramètres de la courbe d’étalonnage du Mirna spécifique).
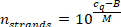
- Calculer le nombre correspondant de particules de lipoprotéines dans le volume de l’échantillon, à partir du volume à l’étape 7.2.1 (100 μL), de sa concentration connue et des étapes de dilution ultérieures (100 μL-> 30 μL de volume d’échantillon à l’étape 7.1.10-> 5 μL [dilution 1:6] échantillon dans le volume total de 15 μL à l’étape 8,4-> 2 μL [dilution 1:7.5] à l’étape 9,4). Supposons un poids moléculaire de 250 kDa pour les particules de HDL et 3 MDa pour les particules de LDL et une récupération de 100% du miRNA au cours de l’étape d’extraction du miRNA (ignorer toute contribution lipidique au poids moléculaire donne une légère surestimation du nombre de brins de miRNA par particules de lipoprotéines).
- Diviser le nombre de brins de miRNA de l’étape 10.2.1 par le nombre de particules calculées à l’étape précédente pour donner le nombre de brins de miRNA par particule de lipoprotéine.
- Calculer le nombre de brins de miRNA par volume d’échantillon à l’aide de la valeur mesurée de l’échantillon cq (étape) et de l’équation suivante (M et B sont les paramètres de la courbe d’étalonnage du Mirna spécifique).
-
Cellules
- Calculez le nombre de brins de miRNA par volume d’échantillon selon l’étape 10.2.1.
- Calculez le nombre correspondant de cellules dans le volume de l’échantillon selon l’étape 10.2.2, en commençant par la concentration initiale de numéro de cellule mesurée à l’étape 6,4.
- Divisez le nombre de brins de miRNA de 10.3.1 par le nombre de cellules calculées à l’étape précédente pour donner le nombre de brins de miRNA par cellule.
- Calculer le taux d’absorption des particules de lipoprotéines en divisant la quantité totale de brins de miRNA de l’étape précédente après correction pour le niveau de miRNA de fond des cellules obtenues à partir d’une expérience de contrôle négatif par le rapport miRNA/particule (étape ) et le temps d’incubation (16 h, voir l’étape 6,2).
11. matrices microfluidiques multipuits
-
extraction de miRNA
- Effectuez l’extraction de miRNA comme décrit à l’étape 7.
-
Transcription inversée
- Décongeler les amorces de transcription inversée, les composants du kit de transcription inversée et le MgCl2 (25 mm) sur la glace. Pour huit échantillons, mélanger 8 μL d’amorces de transcription inversée (10x), 2,25 μL de dNTPs avec dTTP (100 mM), 16,88 μL de transcriptase inverse (50 U/μL), 9,00 μL de tampon de transcription inverse 10x, 10,12 μL de MgCl2, 1,12 μL d’inhibiteur de RNase (20 U/μl) et 1 μL d’eau exempte de nucléases.
- Mélanger doucement et centrifuger brièvement. Ajouter 4,3 μL de mélange de réaction de transcription inverse à 3,5 μL de miRNA extrait dans un tube et mélanger, tourner vers le bas et incuber sur la glace pendant 5 min. Placer les tubes dans une machine thermocycleuse et commencer le programme suivant: 16 ° c pendant 2 min, 42 ° c pendant 1 min , et 50 ° c pendant 1 s répété 40x au total, puis, comme réaction d’arrêt, 85 ° c pendant 5 min et maintenir à 4 ° c jusqu’à l’arrêt.
-
Préamplification
- Décongeler les amorces sur la glace et inverser et centrifuger brièvement. Mélanger le mélange principal de préamplification (2x) en tourbillonnant la bouteille. Préparer le mélange de réaction de préamplification selon les instructions suivantes pour huit échantillons: 112,5 μL de mélange principal de préamplification (2x), 22,5 μL d’amorces de préamplification et 67,5 μL d’eau exempte de nucléases. Inverser et centrifuger brièvement.
- Mélanger 2,5 μL du produit de la réaction de transcription inverse de l’étape 11.2.2 avec 22,5 μL de mélange de réaction de préamplification de l’étape précédente et inverser et centrifuger brièvement. Incuber les échantillons sur la glace pendant 5 min.
- Placer les tubes dans une machine thermocycleuse et incuber aux réglages suivants: activation enzymatique à 95 ° c pendant 10 min, recuit à 55 ° c pendant 2 min, s’étendant à 72 ° c pendant 2 min, répété 12x: dénaturation à 95 ° c pendant 15 s et recuit/allongement à 60 ° c pendant 4 min , inactivation enzymatique à 99,9 ° c pendant 10 min et 4 ° c en attente.
- Tourner vers le bas, ajouter 7,5 μL de 1x TE (pH 8,0) et 67,5 μL d’eau exempte de nucléases, inverser et centrifuger brièvement. Les échantillons peuvent être stockés à-20 ° c pendant une semaine.
-
qPCR
- Mélanger le mélange principal en tourbillonnant la bouteille. Préparer le mélange de réaction PCR pour une carte: 450 μL de mélange principal, 441 μL d’eau exempte de nucléases et 9 μL de l’échantillon de préamplification de l’étape 11.3.4. Inverser et centrifuger brièvement.
- Chargez chaque réservoir de remplissage de la carte multipuits microfluidique avec 100 μL de mélange de réaction PCR préparée selon les instructions du fabricant et Centrifugez 2x pendant 1 min à 3 000 x g. L’accélération pendant les deux étapes de centrifugation consécutives est importante pour remplir correctement la carte. Sceller la carte conformément aux instructions du fabricant.
- Utiliser un système de PCR avec le programme suivant: activation enzymatique à 95 ° c pendant 10 min puis, répétée 40x: dénaturation à 95 ° c pendant 15 s et recuit/allongement à 60 ° c pendant 1 min.
- Importez le fichier de résultats à partir du système de PCR et calculez les valeurs RQ à l’aide du progiciel du système avec les paramètres d’analyse suivants: une valeur cq maximale autorisée de 40,0, des valeurs de cq maximales dans les calculs inclus et des points aberrants Parmi les réplicats exclus. Activez le taux de découverte fausse de Benjamini – Hochberg comme option pour l’ajustement de la valeur p(correction de l’occurrence des faux positifs14) et la normalisation globale comme méthode de normalisation (en utilisant une valeur de seuil médiane pour tous les échantillons 15).
Résultats
Un schéma général de l’isolement des particules lipoprotéiques
La figure 1 montre le schéma général de l’isolement des particules de lipoprotéines à partir de sang total, en utilisant la flottaison séquentielle ultracentrifugation16. Si désiré, d’autres fractions de particules de lipoprotéines comme les particules de VLDL et d’IDL peuvent être récoltées pendant ce protocole. Le rotor en titane à angle fixe en combinaison avec des tubes à joint rapide en polypropylène est adapté pour résister aux forces de centrifugation. Pour éviter l’effondrement du tube, il est important d’éviter les bulles d’air dans le tube. La centrifugation est effectuée à 4 ° c pour minimiser la dégradation des protéines. Habituellement à partir du plasma (60-80 mL par donneur) de dons de sang groupés de trois volontaires, on peut s’attendre à un rendement des volumes de solution de particules de LDL et de HDL de 3 mL chacun avec des concentrations comprises dans la fourchette de 1-3 mg/mL. L’ensemble de la procédure, à partir de don de sang, a pris environ 7 jours.
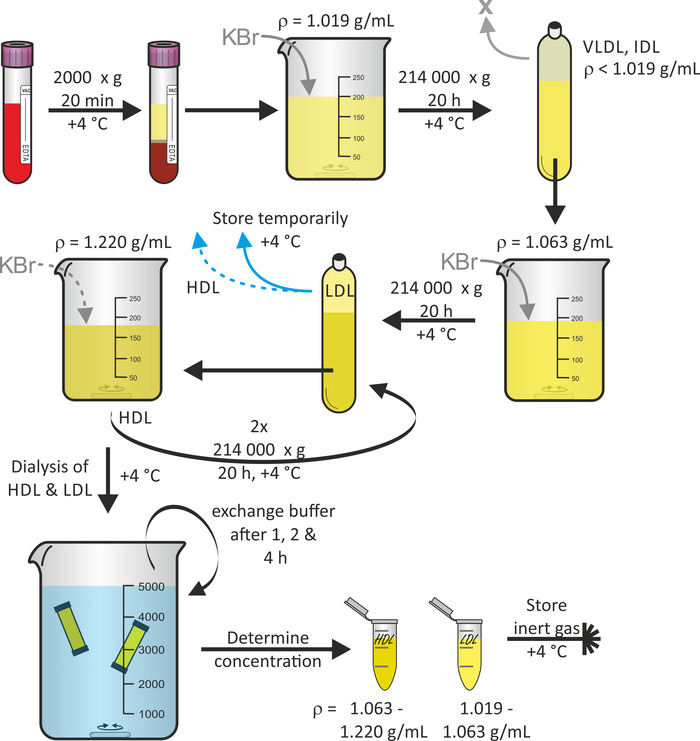
Figure 1: Organigramme de l’isolement des lipoprotéines. Centrifuger le sang de volontaires sains dans les tubes de récipient sous vide et collecter le plasma (phase supérieure). Après l’ajustement de sa densité à ρ = 1,019 g/ml à l’aide de KBr, Centrifugez la solution à 214 000 x g pendant 20 h à 4 ° c. Après l’ajustement de la densité de la fraction inférieure à ρ = 1,063 g/ml à l’aide de KBr, Centrifugez la solution à nouveau à 214 000 x g pendant 20 h à 4 ° c. Conserver la fraction supérieure contenant des particules de LDL temporaires à 4 ° c. Après l’ajustement de la densité de la fraction inférieure à ρ = 1,220 g/ml à l’aide de KBr, Centrifugez la solution deux fois à 214 000 x g pendant 20 h à 4 ° c. Collectez la fraction supérieure contenant des particules de HDL, dialysez les solutions de particules de HDL et de LDL et échangez la mémoire tampon après 1, 2 et 4 h. Après 24 h, déterminer la concentration protéique et stocker les échantillons sous gaz inerte à 4 ° c. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Reconstitution des particules de HDL
La reconstitution des particules de HDL a été réalisée selon un protocole précédemment publié par Jonas7. La première étape a été la délétion des particules de HDL, comme le montre la figure 2a, suivie de la deuxième étape de la relipidation (c.-à-d. reconstitution), comme le montre la figure 2b, en utilisant des lipides PC, Co et C en plus d’un mélange de mirna et de spermine. Nous avons choisi l’homme mature miR-223 et miR-155 parce que miR-223 montre une abondance élevée et miR-155 est rare dans les particules de lipoprotéine17. Habituellement, les deux étapes sont exécutées sur deux jours séquentiels. Lors de la reconstitution, d’autres composants lipophiles et/ou amphiphiles pourraient être ajoutés comme désiré. L’évaporation complète de l’éthanol/éther diéthylique et du solvant méthanol/chloroforme du PC, du CO et du C était critique. La dernière étape, comme le montre la Figure 2c, était la procédure de dialyse pour séparer les particules de HDL reconstituées (rHDL) des lipides libres/Mirna/détergent. Cela a duré 1-2 jours supplémentaires. L’addition de billes absorbantes à la solution de dialyse a maintenu le gradient de densité le long de la constante de la membrane de dialyse. On peut s’attendre à un rendement de 50% des particules de rHDL.
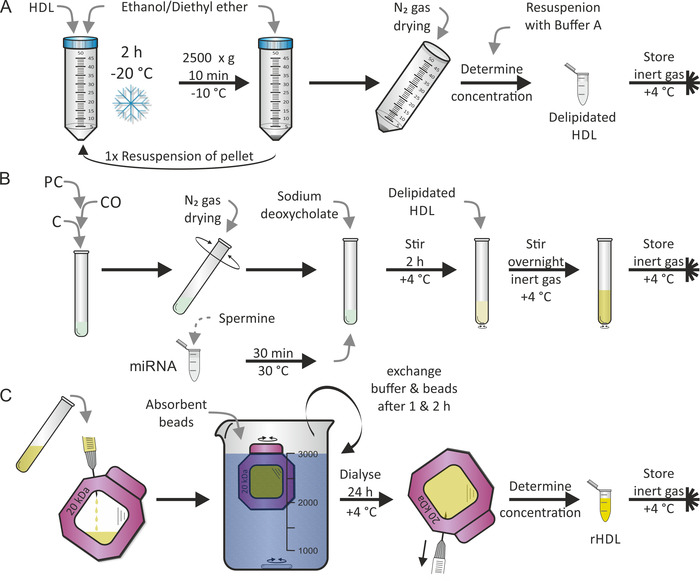
Figure 2: Organigramme de reconstitution des particules de HDL. A) delipidation: mélanger la solution de particules de HDL avec de l’éther d’éthanol/diéthyle prérefroidi et incuber à-20 ° c pendant 2 h. Après avoir jeté le surnageant, Resuspendez le culot et répétez la procédure. Sécher le culot avec du gaz N2 et le remettre en suspension dans le tampon A. Après la détermination de la concentration, stocker le HDL dédaté sous atmosphère gazeuse inerte à 4 ° c. (B) reconstitution: après avoir mélangé la phosphatidyl-choline (PC), l’oléate de cholestéryle (CO) et le cholestérol (C), évaporez le solvant à l’aide de gaz N2 tout en tournant le tube. Incuber le miRNA aliquote avec une solution de spermine pendant 30 min à 30 ° c, ajouter le désoxycholate de sodium et Resuspendre le film lipidique séché. Mélanger l’échantillon pendant 2 h à 4 ° c, ajouter la solution de HDL délédée et mélanger à nouveau l’échantillon, cette fois pendant la nuit à 4 ° c sous atmosphère gazeuse inerte. (C) dialyse: transférer la solution du panneau B contenant des particules de HDL reconstituées (rHDL) dans une chambre à membrane de dialyse (coupure de poids moléculaire: 20 kDa) et DIALYSER contre le PBS et les billes absorbantes à 4 ° c. Échangez la mémoire tampon et les billes après 1 h et 2 h. récupérer la solution de particules rHDL après 24 h, déterminer la concentration et stocker l’échantillon sous atmosphère gazeuse inerte à 4 ° c. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Étiquetage des particules de LDL
L’étiquetage des particules de LDL avec le miRNA (figure 3) comme démontré pour les particules de HDL n’était pas réalisable en raison de l’hydrophobicité de la protéine apob-100, qui est le constituant principal de la particule de LDL. Le DMSO a été utilisé pour la pénétration de la monocouche lipidique de la particule de LDL et, par conséquent, a médié l’Association miRNA. L’ensemble de la procédure a pris 1-2 jours avec un rendement proche de 100%.
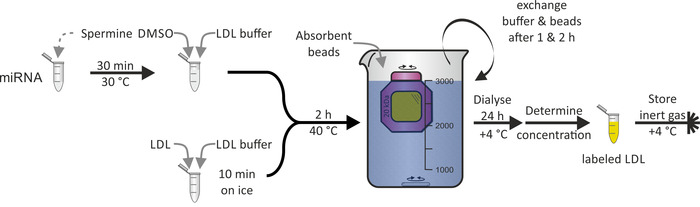
Figure 3: Organigramme de l’étiquetage des particules de LDL. Incuber le miRNA aliquote avec une solution de spermine pendant 30 min à 30 ° c et ajouter le DMSO et le tampon LDL. Incuber l’échantillon de LDL avec le tampon LDL pendant 10 min sur la glace et ajouter le mélange miRNA/spermine/DMSO. Après incubation à 40 ° c pendant 2 h, transférer la solution dans une chambre à membrane de dialyse (coupure de poids moléculaire: 20 kDa) et dialyser contre le PBS et les billes absorbantes à + 4 ° c. Échangez le tampon et les billes après 1 & 2 h. récupérez la solution de particules de LDL étiquetées après 24 h, déterminez la concentration et stockez-la sous atmosphère gazeuse inerte à + 4 ° c. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Contrôle de qualité des particules de lipoprotéines
HS-AFM peut être utilisé pour examiner la taille et la forme des particules de lipoprotéines indigènes et reconstituées/étiquetées sur le mica. Juste avant l’utilisation, le mica doit être fraîchement clivé (utiliser du ruban adhésif pour enlever la couche supérieure [s]) afin de fournir une surface propre et plane. Lors de l’incubation des particules de HDL/LDL sur le mica, le facteur de dilution (et/ou le temps d’incubation) doit être ajusté pour observer les particules individuelles. Les clusters ne permettent pas de déterminer les dimensions des particules. Les particules de HDL sont mobiles sur le mica. Lors de l’utilisation de l’AFM conventionnel au lieu de HS-AFM, le protocole d’immobilisation doit être adapté en conséquence (tampon, revêtement de surface) pour réduire la mobilité des particules latérales. Lors de la numérisation de l’échantillon, la force d’imagerie doit être maintenue basse (mode de taraudage) pour éviter toute déformation des particules, ce qui affectera les valeurs mesurées. Pour l’analyse des données, des particules ont été détectées par l’intermédiaire d’un algorithme de seuil (par exemple, dans Gwynedd: Grains > marque par seuil) et leur hauteur a été déterminée par rapport à la surface du mica. La mesure de la hauteur des particules est le moyen le plus précis de déterminer la taille des particules, car les dimensions latérales apparentes sont élargies par la forme de la pointe (voir les images exemplaires de la figure 4). Les fonctions de densité de probabilité (PDF) des hauteurs de particule ont été calculées pour l’évaluation statistique et la comparaison des distributions de taille des différentes particules de lipoprotéine. Une comparaison des particules de LDL marquées par des miRNA et des natifs, comme illustré à la figure 4 , permet de vérifier la similitude principale entre les particules de lipoprotéines étiquetées et non étiquetées (c.-à-d. indigènes) (particules de LDL étiquetées sans addition de mirna/ les mélanges de spermine sont montrés comme un contrôle pour la procédure d’étiquetage elle-même). L’ensemble de la procédure a duré environ 1 jour.
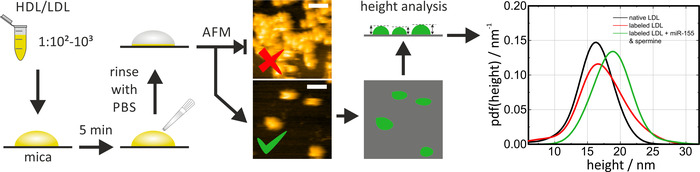
Figure 4: organigramme et résultats représentatifs des mesures HS-AFM. Diluer l’échantillon de particules de HDL/LDL dans du PBS (1:102-1:103) et l’incuber sur du mica fraîchement clivé pendant 5 min, suivi d’un rinçage soigneux avec PBS pour éliminer les particules libres (non adsorbée électrostatiquement). Effectuez l’imagerie HS-AFM et vérifiez la densité des particules sur la surface. Effectuer les mesures en PBS à température ambiante. L’image supérieure de cette figure montre une densité de particules trop élevée; l’image inférieure convient à l’analyse. La hauteur des particules individuelles a été analysée après que les particules de seuil et de courbe native (courbe noire) et reconstituées/étiquetées (courbes rouge et verte) ont été comparées dans une évaluation statistique. La barre d’échelle = 100 nm. Ce chiffre a été modifié d’Axmann et coll.19. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
extraction de miRNA, transcription inversée et qPCR
L’extraction de miRNA à partir d’échantillons de cellules ou de lipoprotéines indigènes/artificiellement enrichis a été effectuée à l’aide d’un kit d’extraction miRNA, comme illustré à la figure 5A. Par la présente, un environnement sans RNase était critique. Cette étape a duré environ 1 h. la transcription inversée de l’échantillon de miRNA extrait (figure 5B) a été réalisée à l’aide de procédures biochimiques normalisées telles que décrites par le fabricant. Cette étape a pris environ 1,5 h. Enfin, la quantité d’ADNc obtenue au cours de la dernière étape a été déterminée à l’aide de qPCR (Figure 5C). Une courbe standard, qui relie les valeurs cq obtenues au nombre absolu de torons Mirna, a donné la teneur absolue en Mirna de l’échantillon initial. Cela a pris environ 2,5 h.
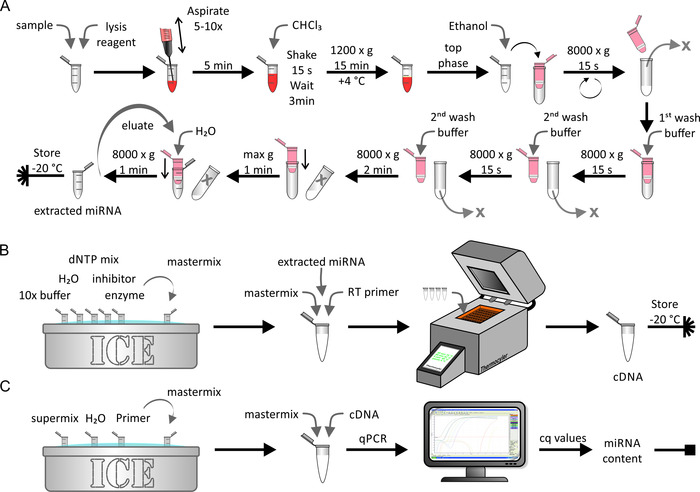
Figure 5: organigramme de l’extraction de miRNA, transcription inversée et qPCR. (A) extraction de mirna: mélanger l’échantillon avec un réactif de lyse et le lyser par aspiration à l’aide d’une seringue. Incuber pendant 5 min et ajouter CHCl3. Agiter vigoureusement pendant 15 s et incuber pendant 3 min. Après centrifugation à 1 200 x g pendant 15 min à 4 ° c, recueillir la phase supérieure et la mélanger avec de l’éthanol. Transférer la solution dans une colonne de spin (volume maximum < 700 μL) et la centrifuger à 8 000 x g pendant 15 s. jetez l’éluent et répétez la dernière étape avec le reste de la solution. Ajouter le premier tampon de lavage et centrifuger à 8 000 x g pendant 15 s. jeter l’éluent, ajouter le deuxième tampon de lavage, et centrifuger à 8 000 x g pendant 15 s. répéter la dernière étape avec un temps de centrifugation de 2 min. Sécher davantage la membrane par centrifugation à la vitesse maximale pendant 1 min. éluer le miRNA avec H2O et centrifuger à 80 000 x g pendant 1 min. conserver l’échantillon de mirna extrait à-20 ° c. (B) transcription inversée: décongeler la mémoire tampon 10x, H2O, le mélange dNTP, l’inhibiteur et l’enzyme sur la glace et préparer le mélange principal. Ajouter le miRNA extrait du panneau A au mélange principal et à l’amorce de transcription inversée et effectuer une transcription inversée à l’aide d’une machine thermocycleuse. Conserver l’échantillon d’ADNc à-20 ° c. (C) qPCR: décongeler le Supermix, H2O, et primer sur la glace et préparer le mélange principal. Ajoutez le cDNA du panneau B au mixage principal et exécutez qPCR. Analysez les données pour obtenir des valeurs cq et calculez le contenu Mirna absolu de l’échantillon (voir la figure 6 et les résultats représentatifs pour plus de détails). S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Contenu absolu miRNA et taux de transfert
On a calculé la teneur absolue en miRNA des particules de HDL et de LDL riches en éléments natifs et artificiellement, à partir des valeurs cq des échantillons et d’une courbe standard du Mirna respectif, comme le montre la figure 6. La figure 6A montre les données calculées par le logiciel d’analyse (avec la normalisation de la dynamictube activée [pour la compensation de différents niveaux de fond en utilisant la deuxième dérivée de chaque trace d’échantillon] et la correction de la pente de bruit [normalisation au niveau du bruit]). cq les valeurs des courbes standard ont été déterminées à l’aide de la fonction de seuil de recherche automatique du progiciel sur le signal de fluorescence normalisé mesuré par la machine qPCR. Par la présente, le logiciel a maximisé la valeur R de l’ajustement de la courbe standard. Le niveau de seuil a été maintenu constant pour chaque analyse d’échantillon de miRNA spécifique. Par la suite, les valeurs de cq ont été tracées en fonction du nombre de brins de Mirna, et une ligne de régression a été calculée. Les valeurs de l’échantillon cq ont été déterminées avec le même niveau de seuil, comme illustré à la figure 6B; les différences d’efficacité de réaction entre les différentes exécutions de qPCR ont été compensées automatiquement par le logiciel en utilisant un échantillon de courbe d’étalonnage supplémentaire inclus dans chaque course. À l’aide de l’équation de la ligne de régression, la quantité inconnue de miRNA dans l’échantillon peut être calculée. Le nombre de particules de lipoprotéines a été estimé à partir de la concentration initiale en protéines et de son poids moléculaire moyen (MWHDL ~ 250 kDa). Ainsi, aucune contribution lipidique au poids moléculaire n’a été assumée — ainsi, le nombre de brins de miRNA par particule de lipoprotéine a été légèrement surestimée. De plus, on a supposé un taux de récupération de 100% de miRNA pendant l’étape d’extraction du miRNA. En outre, la teneur en miRNA des cellules avant et après l’incubation avec des particules de HDL a été déterminée et le taux de transfert de miRNA a été calculé comme illustré à la figure 6C.
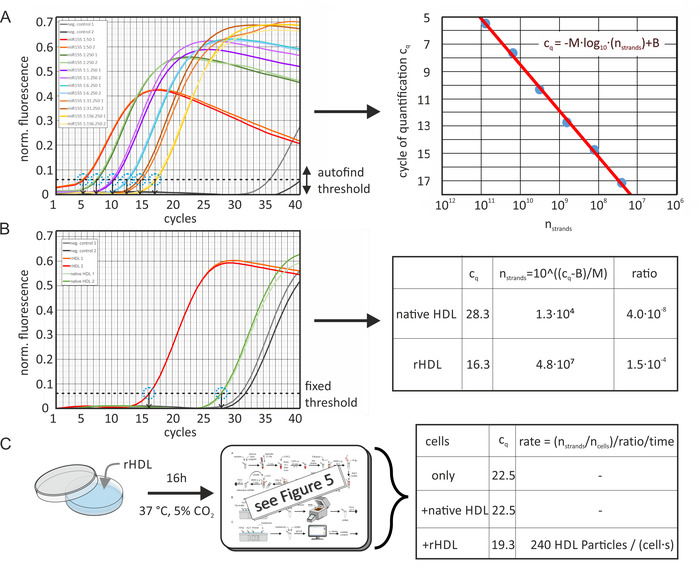
Figure 6: organigramme de calcul du contenu absolu de miRNA et taux de transfert. (A) courbe standard pour le mir-155: une aliquote de mir-155 (100 μl, 10 μM) a été diluée en série avec de l’eau sans ARN comme indiqué. qPCR a donné des valeurs cq pour chaque échantillon de dilution en série (mesuré deux fois) à l’aide de la fonction de seuil de recherche automatique du progiciel. Les expériences de contrôle négatif (sans addition de miRNA) ont donné des valeurs cq de > 35. Les points de données des valeurs cq en fonction du nombre de brins de mirna par volume d’échantillonnage (calculés à partir de la concentration initiale et des dilutions sérielles) ont été équipés de l’équation présentée (ligne rouge, image droite), produisant M =-3,36 et B = 42,12. L’efficacité de la PCR déterminée était de 0,98. Les barres d’erreur ont été calculées à partir des résultats des répétitions expérimentales et étaient plus petites que le diamètre du cercle de points de données. Bles valeurs de cq des particules de HDL natives ou artificiellement enrichies ont été déterminées avec le même seuil que celui déterminé dans le groupe A et converties en nombre de brins de mirna dans le volume de l’échantillon de qPCR. Le ratio absolu de miRNA de l’échantillon original a été calculé à partir du nombre (concentration) de particules de HDL dans le volume de l’échantillon (3,2 x 1011 particules). (C) les échantillons cellulaires (lignée cellulaire LDLA7-SRBI) ont été incubés pendant 16 h avec des particules de HDL enrichiées artificiellement (50 μg/ml) et analysés de façon similaire. Les valeurs déterminées de cq étaient 22,5, 22,5 et 19,3 pour les cellules seulement, pour les cellules incubées avec le HDL natif, ou pour les cellules incubées avec la solution de particules rHDL (50 μg/ml), respectivement. Ces valeurs ont été converties au nombre de brins de miRNA comme fait dans le panneau B. Le nombre de brins de miRNA après incubation (7,3 x 106) a été corrigé par soustraction du nombre de brins de mirna avant l’incubation (8,6 x 105). Le résultat a été divisé par le nombre de cellules dans le volume de l’échantillon (3 100), le miRNA-Particle-ratio (1,5 x 10-4), et la période d’incubation (16 h). Cela a donné le taux de transfert des particules de lipoprotéines par absorption de miRNA (240 événements de capture de particules de HDL par cellule et seconde). Ce chiffre a été modifié d’Axmann et coll.19. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
MultiWell microfluidique Array
En raison des faibles rendements de l’extraction de miRNA, la transcription inversée du miRNA extrait a été suivie d’une étape de préamplification. Enfin, la qPCR, comme illustré à la figure 6, a été exécutée. Pour toutes les étapes, des procédures biochimiques normalisées ont été utilisées comme décrit par le fabricant. Ici, une partie du profil global de miRNA sur les particules de HDL des patients urémiques recrutés pour une étude sur l’influence du CRF sur l’efflux de cholestérol des macrophages18 est montrée. Dans cette étude, la capacité d’accepteur de cholestérol de HDL ou de sérum dans — outre d’autres — 17 jeunes adultes urémiques (CKD stades 3-5) et 14 jeunes adultes hémodialysés patients sans maladies associées et des contrôles appariés a été mesurée. Pour analyser les données, les paramètres par défaut ont été utilisés (valeur CT maximale admissible: 40,0, y compris les valeurs maximales de CT dans les calculs et excluant les points aberrants parmi les répliques). Les valeurs Pont été ajustées à l’aide du taux de fausse découverte de Benjamini-Hochberg (correction de l’occurrence de faux positifs), et comme méthode de normalisation, la normalisation globale a été choisie, qui trouve les dosages communs parmi tous les échantillons pour utiliser sa médiane CT pour la normalisation. Dans les résultats représentatifs, certains RQs de miRNAs isolés des HDLs des patients urémiques sont représentés (les RQs des témoins sont 1). De toute évidence, les miR-122 et miR-224 sont fortement exprimés dans les HDLs des patients urémiques. Toute cette étape a duré environ 1 jour.
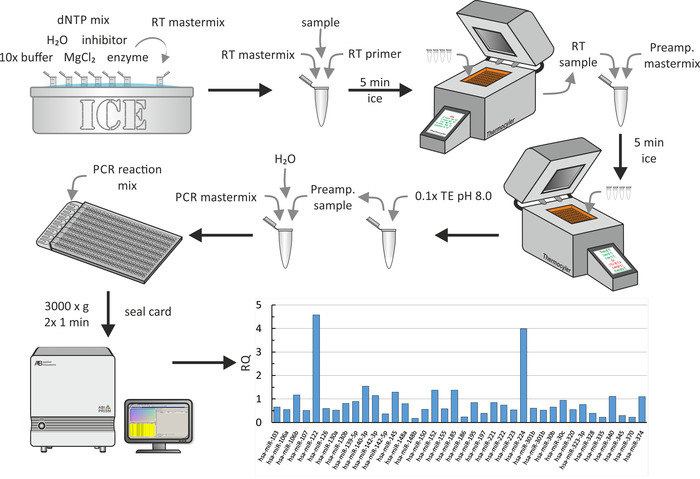
Figure 7: organigramme et résultats représentatifs du réseau microfluidique multipuits. Après l’extraction de miRNA comme illustré à la figure 5A, mélanger l’échantillon Mirna avec l’amorce de transcription inversée et un mélange principal contenant le tampon 10x, H2O, mélange de dNTP, inhibiteur, MgCl2, et enzyme. Après incubation sur glace pendant 5 min, effectuer la transcription inverse à l’aide d’une machine thermocycleuse. Ajouter le mélange principal de préamplification, incuber pendant 5 min sur la glace et effectuer une préamplification à l’aide d’une machine thermocycleuse. Ajouter 0,1 x TE (pH 8,0) et mélanger une aliquote avec PCR Master Mix et H2O. Pipetter le mélange de réaction PCR dans le port de remplissage de la matrice microfluidique multipuits et tourner deux fois à 3 000 x g pendant 1 min chacun. Effectuer qPCR en utilisant un système de PCR et d’analyser les données pour donner des valeurs RQ (ici, la figure montre les valeurs de RQ des particules de HDL des patients d’urémie par rapport à un groupe de contrôle sain18). S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Discussion
Ici, l’isolement des fractions de particules de lipoprotéine du sang humain et la détermination de leur contenu individuel de miRNA est décrit étape par étape. Il est essentiel de travailler dans un environnement exempt de RNase tout en manipulant le miRNA isolé et synthétisé — le miRNA incorporé aux particules est manifestement à l’abri de la dégradation enzymatique. Comme le rapport miRNA/particule des particules de lipoprotéines indigènes est assez faible, l’enrichissement artificiel avec miRNA est nécessaire pour étudier l’absorption des particules Holo des cellules. Ainsi, la reconstitution des particules de HDL comme décrit précédemment7 est modifiée pour incorporer les brins de mirna. En outre, la séparation de la fraction lipidique et protéique au cours de cette procédure permet aux scientifiques d’examiner les composants associés aux lipides et aux protéines de la particule de lipoprotéine19. De la même manière, la procédure d’étiquetage des particules de LDL est adaptée. Il est intéressant de noter que l’addition de spermine — un stabilisateur naturel des nucléotides — n’a pas influencé le rapport miRNA/particule. Il convient de noter que, en principe, la méthode permet l’inpliage d’autres substances que le miRNA dans une particule de lipoprotéine. Bien sûr, il y a une limite en ce qui concerne la taille physique de la substance en fonction de la taille globale du HDL (diamètre: 5-12 nm) et des particules de LDL (diamètre: 18-25 nm).
En ce qui concerne le contrôle de la qualité des particules de lipoprotéines reconstituées/étiquetées, HS-AFM est une méthode applicable pour caractériser les particules de HDL/LDL au niveau de la particule unique. Par rapport à l’EM, il permet des temps de préparation plus courts et des conditions quasi-physiologiques (humides, température ambiante).
En raison de sa sensibilité inhérente et de son amplification, la qPCR est la méthode de choix pour détecter les faibles concentrations de miRNA. Alternativement, la microscopie à fluorescence à une seule molécule, qui est capable de détecter même des molécules individuelles, ne serait pas appropriée en raison des faibles concentrations, par exemple, de brins de miRNA marqués par fluorescence par particule. Ainsi, le ratio des brins de miRNA par particule de lipoprotéine native s’est révélé être 10-8. L’enrichissement artificiel augmente le ratio d’un facteur de 10 000, ce qui facilite l’estimation du taux d’absorption des lipoprotéines cellulaires (aucune différence significative n’est détectée à l’aide de particules de lipoprotéines indigènes 19). La sensibilité élevée de la qPCR permet de déterminer ce taux d’absorption en mesurant le nombre de brins de miRNA après le temps d’incubation et le rapport miRNA/particule. Il convient de noter que la valeur calculée ignore la dégradation cellulaire et la libération de miRNA et, par conséquent, représente au moins une limite inférieure pour le ratio d’absorption des particules de lipoprotéine.
À l’avenir, la méthode peut être adaptée pour transférer des substances pharmaceutiques (notamment des lipophiles) dans les cellules et corréler leur effet biologique à la concentration intracellulaire (déterminée par le taux d’absorption des particules de lipoprotéines).
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été appuyé par le projet du Fonds scientifique autrichien P29110-B21, le projet «Hochschuljubiläumsstiftung der Stadt Wien zur Förderung der Wissenschaft» H-3065/2011, le Fonds européen pour le développement régional (EFRE, IWB2020), l’État fédéral de la haute L’Autriche, et le «Land OÖ Basisfinanzierung».
matériels
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge | Beckman | Optima L-100 XP | Lipoprotein isolation |
| Ultracentrifuge rotor | Beckman | TI 55.2 | Lipoprotein isolation |
| Vacutainer (EDTA) | Becton Dickinson | 366643 | Lipoprotein isolation |
| Kaliumbromid | Sigma Aldrich | 2110 | Lipoprotein isolation |
| Ultracentrifuge tubes | Beckman | 342414 | Lipoprotein isolation |
| Ultracentrifuge tube sealer | Beckman | 342428 | Lipoprotein isolation |
| Sodiumchlorid | Carl Roth GmbH | P029.2 | Lipoprotein isolation |
| EDTA | Fisher Scientific | D/0650/50 | Lipoprotein isolation |
| Dialysis tubes, MWCO 12 - 14k | Spectra | 132700 | Lipoprotein isolation |
| synthetic miRNA | microSynth | - | synthetic miRNA |
| TRIS buffer pH 7 | Ambion | AM9850G | synthetic miRNA |
| TRIS buffer pH 8 | Ambion | AM9855G | synthetic miRNA |
| sterile tubes | Carl Roth GmbH | ENE8.1 | synthetic miRNA |
| Photometer | Eppendorf | Eppendorf Biophotometer | Bradford assay |
| Cuvette | Carl Roth GmbH | XK20 | Bradford assay |
| Coomassie G-250 Stain | BioRad GmbH | 161-0786 | Bradford assay |
| Sodiumchlorid | Carl Roth GmbH | 3957.1 | Delipitation |
| EDTA | Fluka Analytical | 03690-100ML | Delipitation |
| TRIS buffer pH 8.0 | Ambion | AM9855G | Delipitation |
| Ethanol 100% | AustrAlco | Ethanol Absolut 99.9% | Delipitation |
| Diethyl ether | Carl Roth GmbH | 3942.1 | Delipitation |
| Centrifuge | Thermo Scientific | Multifuge X3R | Delipitation |
| Chloroform | Carl Roth GmbH | 3313.1 | Reconstitution |
| Methanol | Carl Roth GmbH | 4627.1 | Reconstitution |
| Phosphatidylcholine | Sigma Aldrich GmbH | P3556-25mg | Reconstitution |
| Cholesterol oleate | Sigma Aldrich GmbH | C-9253-250mg | Reconstitution |
| cholesterol | Sigma Aldrich GmbH | C8667-500mg | Reconstitution |
| glass tubes | Carl Roth GmbH | K226.1 | Reconstitution |
| spermine | Sigma Aldrich GmbH | S3256-1G | Reconstitution |
| Thermomixer | Eppendorf | Thermomixer comfort | Reconstitution |
| Sodium deoxycholate | Sigma Aldrich GmbH | 3097-25G | Reconstitution |
| Magnetic stir bar | Carl Roth GmbH | 0955.2 | Reconstitution |
| 100µL micropipettes | Carl Roth GmbH | A762.1 | Reconstitution |
| PBS | Carl Roth GmbH | 9143.2 | Dialysis |
| Amberlite XAD-2 beads | Sigma Aldrich GmbH | 10357 | Dialysis |
| Slide-A-Lyzer casette (cut-off 20 kDa) | Thermo Scientific | 66003 | Dialysis |
| Syringe | Braun | 9161406V | Dialysis |
| Syringe needle | Braun | 465 76 83 | Dialysis |
| EGTA | Carl Roth GmbH | 3054.2 | Labeling of LDL particles |
| DMSO | life technologies | D12345 | Labeling of LDL particles |
| Atomic Force Microscope | RIBM | SS-NEX | Quality control of reconstituted/labeled lipoprotein particles |
| Muscovite Mica (V-1 Grade) | Christine Gröpl | G250-1/V1 | Quality control of reconstituted/labeled lipoprotein particles |
| AFM Cantilever | Nanoworld | USC-F1.2-k0.15 | Quality control of reconstituted/labeled lipoprotein particles |
| Gwyddion 2.49 | Czech Metrology Institute | http://gwyddion.net | Quality control of reconstituted/labeled lipoprotein particles |
| Chamber slides, Nunc Lab-Tek | VWR | 734-2122 | Cell culture |
| HBSS | Carl Roth GmbH | 9117.1 | Cell culture |
| Cell counter | Omni Life Science GmbH | Casy | Cell culture |
| miRNeasy Mini Kit | QIAGEN | 217004 | miRNA extraction |
| Centrifuge | Eppendorf | 5415R | miRNA extraction |
| 20G needle | Braun | Sterican 465 75 19 | miRNA extraction |
| 5ml syringe | Becton Dickinson | 309050 | miRNA extraction |
| PCR cabinet | Esco | PCR-3A1 | Reverse Transcription |
| TaqMan Reverse Transcription Kit | Thermo Scientific | 4366596 | Reverse Transcription |
| Rnase-free water | Carl Roth GmbH | T143 | Reverse Transcription |
| 0.2 ml tubes | Brand | 781305 | Reverse Transcription |
| Centrifuge | Carl Roth GmbH | Microcentrifuge AL | Reverse Transcription |
| Thermocycler | Sensoquest | Labcycler | Reverse Transcription |
| TaqMan Primer | Thermo Scientific | - | qPCR |
| iTaq Universal probe supermix | BioRad GmbH | 1725131 | qPCR |
| PCR machine | Corbett | Rotor-Gene RG-6000 | qPCR |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366596 | TaqMan Array |
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | TaqMan Array |
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 | TaqMan Array |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399933 | TaqMan Array |
| TaqMan Universal Master Mix II, no UNG | Applied Biosystems | 4440040 | TaqMan Array |
| TaqMan Advanced miRNA Human A Cards | Applied Biosystems | A31805 | TaqMan Array |
| Nuclease-free water | Thermo Scientific | R0581 | TaqMan Array |
| MgCl2 (25mM) | Thermo Scientific | R0971 | TaqMan Array |
| TE, pH 8.0 | Invitrogen | AM9849 | TaqMan Array |
| 7900HT Fast Real-Time PCR System | Applied Biosystems | 4351405 | TaqMan Array |
| TaqMan Array Card Sealer | Applied Biosystems | - | TaqMan Array |
Références
- Vickers, K. C., Palmisano, B. T., Shoucri, B. M., Shamburek, R. D., Remaley, A. T. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nature Cell Biology. 13 (4), 423-435 (2011).
- Tabet, F., et al. HDL-transferred microRNA-223 regulates ICAM-1 expression in endothelial cells. Nature Communications. 5, (2014).
- Wahid, F., Shehzad, A., Khan, T., Kim, Y. Y. MicroRNAs: Synthesis, mechanism, function, and recent clinical trials. Biochimica et Biophysica Acta - Molecular Cell Research. 1803 (11), 1231-1243 (2010).
- Carthew, R. W., Sontheimer, E. J. Origins and Mechanisms of miRNAs and siRNAs. Cell. 136 (4), 642-655 (2009).
- Fabian, M. R., Sonenberg, N., Filipowicz, W. Regulation of mRNA Translation and Stability by microRNAs. Annual Review of Biochemistry. 79 (1), 351-379 (2010).
- Filipowicz, W., Bhattacharyya, S. N., Sonenberg, N. Mechanisms of post-transcriptional regulation by microRNAs: Are the answers in sight?. Nature Reviews Genetics. 9 (2), 102-114 (2008).
- Jonas, A. Reconstitution of High-Density Lipoproteins. Methods in Enzymology. , 553-582 (1986).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Bustin, S. A., et al. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Patsch, J. R., Patsch, W. Zonal ultracentrifugation. Methods in Enzymology. 129 (1966), 3-26 (1986).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Yamamoto, D., et al. High-Speed Atomic Force Microscopy Techniques for Observing Dynamic Biomolecular Processes. Methods in Enzymology. 475, 541-564 (2010).
- Stangl, H., Cao, G., Wyne, K. L., Hobbs, H. H. Scavenger receptor, class B, type I-dependent stimulation of cholesterol esterification by high density lipoproteins, low density lipoproteins, and nonlipoprotein cholesterol. Journal of Biological Chemistry. 273 (47), 31002-31008 (1998).
- Hochberg, B. Controlling the False Discovery Rate: a Practical and Powerful Approach to Multiple Testing. Journal of the Royal Statistical Society. 57 (1), 289-300 (1995).
- Mestdagh, P., et al. A novel and universal method for microRNA RT-qPCR data normalization. Genome Biology. 10 (6), (2009).
- Schumaker, V. N., Puppione, D. L. 6] Sequential Flotation Ultracentrifugation. Methods in Enzymology. 128 (C), 155-170 (1986).
- Wagner, J., et al. Characterization of levels and cellular transfer of circulating lipoprotein-bound microRNAs. Arteriosclerosis, Thrombosis, and Vascular Biology. 33 (6), 1392-1400 (2013).
- Meier, S. M., et al. Effect of chronic kidney disease on macrophage cholesterol efflux. Life Sciences. 136, 1-6 (2015).
- Axmann, M., et al. Serum and Lipoprotein Particle miRNA Profile in Uremia Patients. Genes. 9 (11), 533 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon