Method Article
Enriquecimento de partículas de lipoproteína nativa com microRNA e subsequente determinação de seu conteúdo de microRNA absoluto/relativo e sua taxa de transferência celular
Neste Artigo
Resumo
Aqui, é apresentado um protocolo de reação em cadeia de polimerase em tempo real quantitativo para a determinação do conteúdo de microRNA nativo (absoluto/relativo) de partículas de lipoproteína. Além, um método para aumentar o nível do Micro-RNA, assim como um método para determinar a taxa de captação celular de partículas da lipoproteína, é demonstrado.
Resumo
As partículas de lipoproteína são predominantemente transportadores de lipídios e colesterol na corrente sanguínea. Além disso, eles contêm pequenas quantidades de fios de não-codificação microRNA (miRNA). Em geral, miRNA altera o perfil de expressão protéica devido a interações com o mensageiro-RNA (mRNA). Assim, o conhecimento do conteúdo relativo e absoluto de miRNA de partículas de lipoproteína é essencial para estimar o efeito biológico da captação de partículas celulares. Aqui, uma reacção em cadeia quantitativa do polymerase em tempo real (qPCR)-o protocolo baseado é apresentado para determinar o índice absoluto de miRNA das partículas da lipoproteína-exemplificada mostrada para partículas nativas e miRNA-enriquecidas da lipoproteína. O conteúdo relativo de miRNA é quantificado usando placas de matriz microfluídico multipoços. Além disso, este protocolo permite aos cientistas estimar o miRNA celular e, assim, a taxa de captação de partículas de lipoproteínas. Um aumento significativo do nível de miRNA celular é observável ao usar partículas de lipoproteína de alta densidade (HDL) carregadas artificialmente com miRNA, enquanto a incubação com partículas nativas de HDL não produz nenhum efeito significativo devido ao seu teor de miRNA bastante baixo. Em contrapartida, a captação celular de partículas de lipoproteína de baixa densidade (LDL) — nem com miRNA nativa nem carregada artificialmente — não alterou o nível de miRNA celular.
Introdução
As partículas de lipoproteína são compostas de uma monocamada de lipídios anfifílicos e colesterol que delimitam um núcleo de ésteres de do cholesteryl e gorduras de triglicérides. A partícula inteira é estabilizada por apolipoproteínas membrana-encaixadas, que definem a funcionalidade biológica da partícula. As partículas da lipoproteína podem ser distinguidas de acordo com sua densidade crescente respectiva e, assim, diminuindo o tamanho, a saber como a lipoproteína da muito-baixo-densidade (VLDL), a lipoproteína da intermediário-densidade (IDL), o LDL, e as partículas de HDL. Apesar do transporte de componentes insolúveis em água na corrente sanguínea, tem sido demonstrado que as partículas de HDL carregam fios não codificantes de Mirna1,2. As micro-RNAs são uma classe de fios de RNA curtos (geralmente de duas dúzias de nucleotídeos), que degradam as vertentes intracellularmente complementares do mRNA e, assim, alteram o perfil de expressão de certas proteínas3,4,5, seisanos. Além disso, as alterações do perfil de miRNA foram encontradas em uma variedade de doenças e, assim, o perfil é aplicável como um biomarcador para diagnóstico e prognóstico. O transporte extracelular de miRNAs entre pilhas através das partículas da lipoproteína pode serir como um mecanismo adicional para a modulação intercelular do nível do mRNA. Para estimar quantitativamente o efeito biológico, é necessário o conhecimento sobre o teor absoluto e relativo de miRNA de partículas de lipoproteína.
O PCR quantitativo em tempo real é um método adequado e relativamente rápido para obter essa informação. Assim, o valor da quantificação relativa (RQ) pode ser calculado, e diferenças relativas entre diferentes amostras e frações de lipoproteína são estimáveis. Os cartões de matriz microfluídico multiwell são um método rápido e fácil de usar para determinar a presença relativa (equivale ao valor RQ) de miRNAs em uma amostra. Os cartões microfluídicos da disposição de multiwell consistem em 96 ou em câmaras de reação individuais do 384 para reações individuais do qPCR encaixadas em um dispositivo microfluídicos. Cada câmara contém a sonda de hidrólise necessária e primers qPCR específicos para um miRNA individual. As vantagens são um tempo de manuseio curto devido à padronização, um fluxo de trabalho simples e um número reduzido de etapas de pipetagem. Além disso, o volume de amostra exigido é minimizado. Em contraste com a quantificação relativa, o teor absoluto de miRNA requer uma comparação dos resultados da amostra de qPCR com curvas padrão de números absolutos conhecidos de fios de miRNA. Deve-se notar que, devido ao seu teor relativamente baixo de miRNA, padrão e, além disso, mesmo as técnicas de imagem sensíveis de única molécula não são viáveis — o enriquecimento artificial de partículas de lipoproteína com miRNA é inevitável para estudar interação de partículas de lipoproteína e transferência de miRNA. Em relação a isso, a delipidação da partícula de HDL seguida com subsequente relipidation7 permite a incorporação e, assim, o enriquecimento com fios de Mirna. O enriquecimento semelhante de partículas de LDL com miRNA não é viável devido à hidrofobicidade da proteína apoB-100, que é o principal constituinte da partícula LDL. No entanto, por adição do solvente polar dimetil sulfóxido (DMSO), que é capaz de penetrar as membranas lipídicas, as partículas de LDL também podem ser carregadas artificialmente com fios de miRNA.
A microscopia de força atômica de alta velocidade (HS-AFM) é uma ferramenta poderosa para a caracterização de espécimes biológicos que oferecem subnanômetro de resolução espacial e subsegundo temporal8. Assim, é uma técnica bem adequada para o controle de qualidade de partículas de lipoproteína modificada como partículas de lipoproteína nativas/reconstituídas/rotuladas podem ser imaged um ambiente quase fisiológico.
Aqui, um protocolo baseado em qPCR é apresentado passo a passo para determinar o teor absoluto/relativo de miRNA de partículas de lipoproteína e amostras de células, o que permite estimar a taxa de captação de partículas de lipoproteína celular. Além disso, um método para o enriquecimento de partículas da lipoproteína com miRNA é demonstrado. Este método pode ser adaptado para a manipulação geral do conteúdo de lipoproteínas e, portanto, demonstra a aplicabilidade das partículas de lipoproteína como alvos para a entrega de medicamentos.
Protocolo
As doações de sangue foram aprovadas pelo Comitê de ética da universidade médica de Viena (EK-Nr. 511/2007, EK-Nr. 1414/2016). A nomenclatura é de acordo com a informação mínima para a publicação de experimentos quantitativos de PCR em tempo real (MIQE)9 diretrizes.
1. isolamento de partículas de lipoproteínas do sangue humano
- Precool um ultracentlee a 4 ° c. Tirar sangue de voluntários saudáveis após o jejum noturno.
Nota: tipicamente exigido são três doadores, doando 80 ml cada um, e os tubos da coleção do sangue que contêm o ácido etilenodiaminotetracético (EDTA) como o anticoagulante. - Centrifugue em 2.000 x g por 20 min a 4 ° c e colheita de plasma (fase superior); evitar forças de cisalhamento. Determine o volume total V e ajuste, se necessário, a um múltiplo do volume do tubo da centrifugação usando o soro fisiológico fosfato-tamponado (PBS). Meça a massa de 1 mL 3x e calcule a densidade média ρ.
- Calcule a quantidade necessária de brometo de potássio (KBr) para ajuste de densidade com a seguinte equação10 (para densidade desejada em gramas/mililitro, use a = 1, 19, e para o volume específico de kbr, use
 = 0,364 ml/g). Adicione KBr ao plasma e mexa suavemente para evitar forças de cisalhamento até que o KBr esteja totalmente dissolvido.
= 0,364 ml/g). Adicione KBr ao plasma e mexa suavemente para evitar forças de cisalhamento até que o KBr esteja totalmente dissolvido.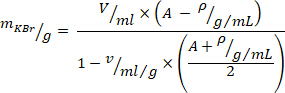
- Meça a densidade conforme descrito na etapa 1,2 e ajuste novamente, se necessário, adicionando mais KBr. Encha e sele os tubos de centrifugação apropriados para ultracentlee com plasma. Evite bolhas de ar; caso contrário, o tubo pode desmoronar. Coloque os tubos no rotor de acordo com as instruções do fabricante e centrifugue a 214.000 x g durante 20 h a 4 ° c.
- Abra os tubos de acordo com as instruções do fabricante e descarte a fase superior contendo VLDL e IDL. Determine o volume total V e ajuste, se necessário, a um múltiplo do volume do tubo de centrifugação usando PBS.
- Determine a densidade ρ da fração inferior. Calcule a quantidade necessária de KBr para ajuste de densidade (use A = 1, 63). Mexa suavemente para evitar forças de cisalhamento até que o KBr seja dissolvido. Repita a etapa 1,4.
- Retire e colete a fase superior contendo partículas de LDL. Armazene a solução de partículas de LDL uma atmosfera de gás inerte a 4 ° c. Determine o volume total V e ajuste, se necessário, a um múltiplo do volume do tubo de centrifugação usando PBS. Determine a densidade ρ da fração inferior conforme descrito na etapa 1,2.
- Calcule a quantidade necessária de KBr para ajuste de densidade (use A = 1,220) e adicione-a. Mexa suavemente para evitar forças de cisalhamento até que o KBr seja dissolvido. Repita a etapa 1,4.
- Retire e colete a fase superior contendo partículas de HDL. Determine seu volume total V e ajuste, se necessário, a um múltiplo do volume do tubo de centrifugação usando PBS. Determine a densidade ρ. Uma segunda etapa da centrifugação da fase superior em 214.000 x g por 20 h em 4 ° c é recomendada para remover a albumina. Se necessário, repita a etapa 1,8. Retire e colete a fase superior contendo partículas de HDL.
- Prepare e precool pelo menos 20 L de tampão de diálise (0,9% NaCl, 0,1% EDTA [pH 7,4]) a 4 ° c. Tubos de diálise pré-húmidos (corte de peso molecular: 12 – 14 kDa) e adicionar a solução de partículas de LDL e HDL de acordo com as instruções do fabricante. Dialyze de encontro a 5 litros do amortecedor da diálise em 4 ° c e mude o amortecedor após 1, 2, e 4 h.
- Após 24 h, recupere as soluções da partícula da lipoproteína dos tubos da diálise e determine a concentração da proteína usando o ensaio de Bradford11 ou outro apropriado. Armazene as soluções de partículas de HDL e LDL em atmosfera de gás inerte a 4 ° c.
2. alíquotas de miRNA sintéticas
Nota: ao manusear oligonucleotides de RNA, trabalhe sem RNase. Trabalhe somente com os consumíveis plásticos frescos, descartáveis e use sempre as luvas, que devem ser mudadas freqüentemente. Utilize apenas soluções sem nucleases. Trabalhe sempre no gelo.
- Gire para baixo o frasco adquirido do fabricante na força máxima para formar uma pelota do miRNA sintético liofilizado. Adicionar um volume apropriado de 10 mM TRIS (hidroximetil) aminometano (TRIS) tampão, pH 7,5, para uma concentração final de 10 μM (concentração de ações) miRNA.
- Pipetar suavemente para cima e para baixo um par de vezes para ressuscitá-lo. Prepare alíquotas de 100 μL cada em tubos estéreis. Guarde-os a-20 ° c se não for utilizado imediatamente. Evite a congelação e o congelamento repetidos.
3. reconstituição de partículas de HDL
-
Delipidação
- Prepare o tampão A (150 mM NaCl, 0, 1% EDTA, 10 mM Tris/HCl [pH 8,0]). Precool a centrífuga para-10 ° c. Precool 100 mL de uma mistura de etanol: éter dietílico (3:2) a-20 ° c.
PRECAUÇÃO: Utilize equipamento de protecção individual adequado e trabalhe num exaustor enquanto manuseando éter dietílico, uma vez que é extremamente inflamável e prejudicial para a pele. - Misturar um volume correspondente a 5 mg de partículas de HDL com 50 mL da mistura de etanol pré-arrefecido: éter dietílico (3:2) e incubar durante 2 h a-20 ° c. Centrifugue a 2.500 x g durante 10 min a-10 ° c.
- Descarte o sobrenadante, Ressuspender o pellet em 50 mL de etanol pré-resfriado: mistura de éter dietílico por vortexing, e incubar uma segunda vez para 2 h a-20 ° c. Centrifugue novamente a 2.500 x g durante 10 min a-10 ° c.
Nota: se desejado, liofilizar o sobrenadante para uma análise do conteúdo de miRNA na fração lipídica das partículas de HDL. - Seque a pelota o fluxo de gás N2 e ressuspender-a em 250 μL de tampão a (ver passo 3.1.1). Determine a concentração de proteína utilizando o ensaio de proteína Bradford ou outra adequada e diluir para uma concentração final de 1 mg de proteína/250 μL de tampão A.
Observação: o protocolo pode ser pausado aqui. Armazene a solução durante a noite a 4 ° c em atmosfera de gás inerte.
- Prepare o tampão A (150 mM NaCl, 0, 1% EDTA, 10 mM Tris/HCl [pH 8,0]). Precool a centrífuga para-10 ° c. Precool 100 mL de uma mistura de etanol: éter dietílico (3:2) a-20 ° c.
-
Reconstituição
- Prepare uma solução conservada em estoque do fosfatidilcolina (PC) usando uma mistura do clorofórmio: metanol (2:1) em uma concentração de 5,6 mgs de PC/ml. Da mesma forma, prepare soluções de estoque de oleato de colesterilo (5 mg de CO/mL) e colesterol (5 mg de C/mL). Armazene todas as soluções a-20 ° c.
- Em um tubo de vidro limpo, misture 500 μL de PC, 100 μL de CO e 13,5 μL de C. Estes volumes correspondem a uma relação molar aproximada de 100 PC: 22 CO: 4.8 C. seque a mistura o fluxo do gás de N2 ao girar o tubo para render uma camada de superfície homogênea.
Observação: o protocolo pode ser pausado aqui. Guarde o frasco de vidro (se desejar, é possível estocar) atmosfera de gás inerte a-20 ° c. - Prepare uma solução fresca de 30 mm espermina no tampão a. Misture uma alíquota (100 μl, 10 μm) de Mirna sintética (ver passo 2,2) com 100 μl de solução de espermina e incubar durante 30 min a 30 ° c.
Nota: para experimentos de controle negativo, substitua a solução de Mirna e/ou espermina pelo mesmo volume de tampão a. - Dissolva uma aliquota de mistura mestra PC/CO/C na mistura da etapa 3.2.3.
- Prepare uma solução de 30 mg/mL de desoxicolato de sódio no tampão A e adicione 50 μL à solução a partir do passo 3.2.4. Agitar a 4 ° c durante 2 h.
- Adicione 250 μl da solução de HDL deslipemizado da etapa 3.1.4. Este volume corresponde a uma relação molar aproximada de 100 PC: 22 co: 4.8 c:1 proteínas de HDL deslipemizado. Mexa a 4 ° c durante a noite.
-
Diálise
- Precool pelo menos 15 L de PBS a 4 ° c. Adicionar 50 g de grânulos de adsorvente a 800 mL de água destilada dupla (ddH2O) e mexa por 1 min. aguarde 15 min, decantar o sobrenadante, e repita o procedimento com PBS.
- Gavetas pré-molhadas da diálise (corte do peso molecular: 20 kDa) ou tubos apropriados da diálise e adicionam a solução da etapa 3.2.6, usando uma seringa de acordo com as instruções do fabricante.
- Adicione os grânulos adsorventes do passo 3.3.1 para 3 L de PBS e diálise a 4 ° c. Mude o tampão e os grânulos após 1 h e 2 h.
- Após 24 h, recupere a solução de partícula reconstituída de HDL (rHDL) e determine a concentração de proteína usando o ensaio de Bradford. Armazene a solução de partículas rHDL em atmosfera de gás inerte a 4 ° c.
4. rotulagem de partículas de LDL
- Prepare o tampão do LDL 10x (1,5 M NaCl, EDTA de 3 milímetros, 1 milímetro etileno glicol-bis (éter do β-aminoethyl)-N, N, N, N, n'-o ácido Tetraacetic [EGTA, pH 7,4]) e armazene-o na temperatura ambiente.
- Prepare uma solução fresca da espermina de 30 milímetros na água RNase-livre. Misturar uma alíquota (100 μl, 10 μm) de Mirna sintética (ver passo 2,2) com 100 μl de solução espermina e incubar durante 30 min a 30 ° c.
Nota: para experimentos de controle negativo, substitua o Mirna e/ou a solução de espermina com o mesmo volume de tampão de LDL 1x. - Adicionar 100 μL de DMSO à solução de miRNA/spermine da etapa 4,2 e diluir-lo ainda mais com 1,2 mL de tampão de LDL 1x.
- Diluir a solução de partículas de LDL para uma concentração final de aproximadamente 4 mg/mL com PBS e misturar 450 μL com 50 μL de tampão LDL de 10x. Incubar-lo por 10 min no gelo.
- Combine a solução de partículas de LDL da etapa anterior e a solução de miRNA/spermine/DMSO (da etapa 4,3) e incubar-a por 2 h a 40 ° c.
- Realize a diálise similar como descrito na seção 3,3 e armazene a solução etiquetada da partícula de LDL conformemente.
5. controle da qualidade de partículas reconstituídas/etiquetadas da lipoproteína
Nota: para controle de qualidade, o diâmetro e a forma geral das partículas de lipoproteína podem ser determinados usando, por exemplo, AFM ou microscopia eletrônica (EM). Aqui, o HS-AFM é usado para medir a distribuição do tamanho de partículas nativas/reconstituídas/etiquetadas da lipoproteína.
- Diluir a solução de partículas de HDL/LDL em PBS (1:100 – 1:1000) e incubatê-la em mica recém-clivada por 5 min. Para clivagem a mica12, pressione a fita adesiva de encontro à carcaça e remova as camadas superiores de mica puxando a fita fora.
Nota: dependendo do instrumento AFM específico, da área de observação e da concentração inicial de partículas, o factor de diluição tem de ser ajustado para observar as partículas individuais. - Após a incubação, enxágüe a amostra com PBS e realize a imagem latente de HS-AFM em PBS e no modo de batida com os cantialavancas que têm constantes da mola de kcant ≪ 0,2 N/m. Recomenda-se o uso de tamanhos de digitalização < 1 μm2 e para manter as forças de imagem o mais baixo possível.
- Determine a altura das partículas imaged com respeito à superfície de mica com software apropriado.
- Carregar os dados em Gwyddion (freeware), detectar as partículas através de análise de grãos (marca grãos por limiar) e definir o limiar acima do fundo do substrato. Aplainar a imagem (remover o fundo polinomial) escolhendo a opção excluir região mascarada .
- Exporte os valores de altura das partículas detectadas (distribuição de várias características de grãos) e repita estas etapas para todas as imagens gravadas. Realize uma análise estatística criando histogramas ou calculando as funções de densidade de probabilidade das alturas de partícula obtidas.
- Repita as etapas 5.1 – 5.3 com partículas de lipoproteína nativas, bem como reconstituídas/rotuladas, e compare os resultados obtidos para verificar a qualidade das partículas. Se observar detritos e/ou conglomerados, descarte a amostra.
6. cultura celular
- Cresça pilhas aderentes de acordo com um protocolo estabelecido (por exemplo, ldlA7-SRBI13) até alcançar a confluência.
Nota: várias câmaras independentes, dependendo do número de experimentos (recomendado são duas câmaras por configuração experimental) e experimentos de controle negativo (recomendado são duas câmaras sem a adição de partículas de lipoproteína) são necessários. Adicionalmente, duas câmaras são exigidas para a determinação do número da pilha. - Lave suavemente as células 3x com a solução de sal balanceada de Hank (HBSS) para remover detritos celulares e cubra a camada celular com um volume adequado de meio de crescimento sem soro. Adicionar um volume adequado de solução de partículas de lipoproteína para atingir uma concentração final de 50 μg/mL de partículas de lipoproteína. Incubar a 37 ° c e 5% CO2 por 16 h.
Nota: dependendo do desenho experimental e da linha celular, o tempo de incubação deve ser adaptado para atingir um aumento suficiente (ou seja, mensurável) no nível de miRNA celular. - Lave suavemente as células 3x com pré-aquecido (37 ° c) HBSS para remover restos de células/partículas de lipoproteína e cubra a camada celular com um volume adequado de meio livre de soro.
- Determine a densidade da célula usando um método apropriado (por exemplo, Hemocytometer, contador de células automatizado) em pelo menos duas câmaras independentes para calcular o número de células no volume de amostra da etapa 6,3. Esse número é usado para normalização.
7. extração de miRNA de amostras de partículas de células e lipoproteínas
Nota: a extração de miRNA de células é realizada usando o kit de extração de miRNA com as seguintes modificações.
-
Amostras de células
- Precool a centrífuga para 4 ° c. Adicionar 350 μL de reagente de Lise a duas câmaras contendo células. Como um experimento de controle negativo, use uma câmara sem células.
Cuidado: Use equipamento de proteção pessoal apropriado e trabalhe em uma capa de fumaça ao manusear o reagente de Lise, pois contém fenol e tiocianato. - Aguarde 3 – 5 min (dependendo da linha celular) para o descolamento da célula. Se necessário, verifique o descolamento da célula com microscopia de campo. Pool o conteúdo das duas câmaras em um 1,5 mL tubo.
- Utilizando uma agulha de 20 G e uma seringa de 5 mL, homogeneizar/interromper a amostra 5x – 10x por aspiração e incubar durante 5 min. adicionar 140 μL de clorofórmio (CHCl3), agitar vigorosamente durante 15 s e incubar durante 3 min.
- Centrifugue a 12.000 x g durante 15 min a 4 ° c. Depois, pare de arrefecer a centrífuga.
- Transfira a fase aquosa superior para um novo tubo de 1,5 mL; Evite a mistura/contaminação da fase porque a interfase contem o ADN e a fase mais baixa contem proteínas. Adicionar 1,5 x o volume de 100% de etanol e misture completamente por pipetagem.
- Coloque uma coluna de spin em um tubo de coleta de 2 mL e adicione 700 μL da mistura da etapa 7.1.5. Centrifugue a 8.000 x g por 15 s à temperatura ambiente. Elimine o caudal e repita este passo com o volume de amostra residual.
- Descarte o fluxo e adicione 700 μL do primeiro tampão de lavagem à coluna de rotação. Centrifugue-o em 8.000 x g por 15 s à temperatura ambiente.
- Descarte o fluxo e adicione 500 μL do segundo tampão de lavagem à coluna de rotação. Centrifugue-o em 8.000 x g por 15 s à temperatura ambiente. Repita esta etapa inteira uma segunda vez com 2 minutos do tempo da centrifugação.
- Coloque a coluna de centrifugação num novo tubo de recolha de 2 mL e Centrifugue-o a toda a velocidade durante 1 min para secar a membrana.
- Coloque a coluna de rotação em um tubo de coleta de 1,5 mL. Adicione 30 μL de água sem RNase no centro da membrana para eluição e Centrifugue-o a 8.000 x g durante 1 min. Repita este passo inteiro uma segunda vez com o primeiro fluxo através de solução de eluição. A etapa de transcrição reversa é feita imediatamente após a extração; caso contrário, armazene as amostras de miRNA extraídas a-20 ° c.
- Precool a centrífuga para 4 ° c. Adicionar 350 μL de reagente de Lise a duas câmaras contendo células. Como um experimento de controle negativo, use uma câmara sem células.
-
Partículas de lipoproteína
- Definir o volume da amostra com a menor concentração de proteína para 100 μL (= volume máximo da amostra). Calcule os volumes amostrais das outras amostras de acordo com esta normalização, inversamente à sua concentração. Para experimentos de controle negativo, use 100 μL de água livre de RNase.
Nota: a normalização não é exigida mas simplifica a comparação direta dos resultados durante a etapa e a análise do qPCR. - Precool a centrífuga para 4 ° c. Adicionar 700 μL de reagente de Lise ao volume amostral da etapa 7.2.1.
- Realize a extração de miRNA de acordo com as etapas 7.1.3 – 7.1.10.
- Definir o volume da amostra com a menor concentração de proteína para 100 μL (= volume máximo da amostra). Calcule os volumes amostrais das outras amostras de acordo com esta normalização, inversamente à sua concentração. Para experimentos de controle negativo, use 100 μL de água livre de RNase.
8. transcrição reversa
Nota: a transcrição reversa de miRNA é realizada usando um kit de transcrição reversa com as seguintes modificações.
- Descongelar os reagentes do kit e os primers de transcrição reversa no gelo. Prepare a seguinte mistura mestra em um tubo de reação no gelo: 45,7 μL de RNase-livre H2O, 16,5 μl do tampão reverso da transcrição 10x, 11 μl da enzima reversa da transcrição, 2,1 μl do inibidor de RNase, e 1,7 μl da mistura de dNTP. Misture suavemente, não vórtice.
Nota: a escala depende da quantidade da amostra; aqui, é calculado para 10 reações. - Rotule 0,2 mL de tubos em conformidade e misture 7 μL da mistura mestra da etapa 8,1 com 5 μL da amostra extraída do passo 7.1.10 e 3 μL de primer de transcrição reversa. Misture suavemente, centrifugue em breve e guarde a mistura no gelo.
- A fim usar o mesmo número de pilha para cada linha de pilha, reduza o volume de amostra da linha de pilha com um número de pilha total mais elevado; Utilize-o como volume residual para atingir o volume total da amostra de 5 μL de água sem RNase.
Nota: as amostras de partículas de lipoproteína já estão normalizadas na etapa 7.2.1. Para a preparação da curva padrão, diluir uma alíquota de miRNA sequencialmente em água sem RNase e calcular o número de fios por volume amostral. São necessários pelo menos cinco pontos de dados dentro do intervalo dos valores resultantes do ciclo de quantificação da amostra (cq). Geralmente, os fatores de diluição totais que variam de 102 a 106 são apropriados. - Coloc os tubos na máquina do termociclador e comece o seguinte programa: 30 minutos em 16 ° c (etapa do annealing), 30 minutos em 42 ° c (transcrição reversa), 5 minutos em 85 ° c (etapa de derretimento), e pausa em 4 ° c (armazenamento). Realize a etapa qPCR imediatamente após a transcrição reversa; caso contrário, armazene o DNA complementar (cDNA) sintetizado a partir das amostras de miRNA a-20 ° c.
9. qPCR
- Realize um qPCR do cDNA (reverso transcrito de miRNA) usando um ensaio comercialmente disponível (veja a tabela de materiais) com as seguintes modificações.
- Descongelar todos os reagentes (Supermix, RNase-Free H2O), amostras de cDNA (da etapa 8,4), e os primers no gelo. Prepare a seguinte mistura mestra em um tubo de reação no gelo: 75 μL de Supermix, 47,5 μL de RNase-Free H2O, 7,56 μL de primer. Misture suavemente, não vórtice.
Nota: a escala depende da quantidade da amostra; aqui, é calculado para 10 reações. - Rotule 0,2 mL de tubos em conformidade e adicione 2 μL da amostra de cDNA a 13 μL da mistura mestra e misture suavemente. Em geral, meça cada amostra 2x.
Nota: pelo menos duas amostras de controlo negativo são necessárias adicionalmente — utilize água sem RNase como amostra. Se a curva de calibração não for determinada na mesma corrida que a amostra, uma amostra da medição da curva de calibração também será necessária. Ele é usado para calibrar cada corrida individual para a mesma eficiência de reação. - Coloque os tubos na máquina de PCR e inicie o seguinte programa: 2 min a 50 ° c, 10 min a 95 ° c, 15 s a 95 ° c e 60 s a 60 ° c. Repita as duas últimas etapas do programa até 50X.
- Para uma análise dos valores de cq com o pacote de software da máquina do PCR, ative a normalização de dynamictube (para a compensação de níveis de fundo diferentes usando a segunda derivada de cada traço da amostra) e a inclinação do ruído Correção (normalização para o nível de ruído).
Nota: o valor de cq do controlo negativo deve ser vários ciclos mais elevados do que a maior amostra c valorq .- Para uma análise da curva de calibração, determine o limiar para o cálculo do cq para cada Mirna a partir das curvas padrão de cada Mirna individualmente, utilizando a função de limiar de auto-Find do pacote de software e mantenha-a constante para cada miRNA específico.
- Para a análise da amostra, se necessário, compensar diferentes eficiências de reação da amostra executada com o ponto de dados da amostra da curva de calibração. O software calcula os valores de cq das amostras a partir do nível de limiar da respectiva medição da curva de calibração.
10. cálculo do conteúdo de miRNA
-
Curva de calibração
- Calcular, a partir do número inicial de fios de miRNA por alíquota (100 μL de miRNA de 10 μM, peso molecular da folha de dados) e as etapas subsequentes de diluição em série, o número de fios de miRNA no volume amostral (o volume amostral de 5 μL da etapa 8,3).
Nota: assumindo uma eficiência de transcrição reversa de 1, este número é igual ao número de vertentes do cDNA. - Calcule o número de fios de cDNA no volume amostral de 2 μL da etapa 9,3. Considere assim o factor de diluição adicional de 7,5 (o volume de amostra de 2 μL da etapa 9,4 do volume de amostra de 15 μL do passo 8,4).
- Plote o valor de cq da etapa 9.5.1 contra o número de vertentes nvertentes calculadas na etapa 10.1.2 em um gráfico semilogarítmico base-10 e ajuste os pontos de dados com a seguinte curva de regressão (M = a inclinação do linear curva de regressão, B = offset).
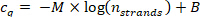
Confirme se o coeficiente de correlação (R2) para a linha é > 0,99.
- Calcular, a partir do número inicial de fios de miRNA por alíquota (100 μL de miRNA de 10 μM, peso molecular da folha de dados) e as etapas subsequentes de diluição em série, o número de fios de miRNA no volume amostral (o volume amostral de 5 μL da etapa 8,3).
-
Partículas de lipoproteína
- Calcule o número de fios de miRNA por volume de amostra usando o valor medido cq (Step 9.5.2) e a seguinte equação (M e B são os parâmetros da curva de calibração do Mirna específico).
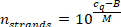
- Calcule o número correspondente de partículas de lipoproteína no volume amostral, a partir do volume na etapa 7.2.1 (100 μL), sua concentração conhecida e as etapas subsequentes de diluição (100 μL-> 30 μL de volume amostral na etapa 7.1.10-> 5 μL [diluição 1:6] amostra no volume total de 15 μL na etapa 8,4-> 2 μL [diluição 1:7.5] na etapa 9,4). Assumir um peso molecular de 250 kDa para as partículas de HDL e 3 MDa para partículas de LDL e uma recuperação de 100% de miRNA durante a etapa de extração de miRNA (ignorando qualquer contribuição lipídica para o peso molecular produz uma ligeira superestimativa do número de cordões de miRNA por partícula de lipoproteína).
- Divida o número de fios de miRNA do passo 10.2.1 pelo número de partículas calculadas na etapa anterior para produzir o número de fios de miRNA por partícula de lipoproteína.
- Calcule o número de fios de miRNA por volume de amostra usando o valor medido cq (Step 9.5.2) e a seguinte equação (M e B são os parâmetros da curva de calibração do Mirna específico).
-
Células
- Calcule o número de fios de miRNA por volume de amostra de acordo com a etapa 10.2.1.
- Calcule o número correspondente de células no volume de amostra de acordo com a etapa 10.2.2, começando com a concentração inicial do número da célula medida na etapa 6,4.
- Divida o número de fios de miRNA de 10.3.1 pelo número de células calculadas na etapa anterior para produzir o número de fios de miRNA por célula.
- Calcule a taxa de captação de partículas de lipoproteína dividindo a quantidade total de fios de miRNA da etapa anterior após correção para o nível de miRNA de fundo das células obtidas de um experimento de controle negativo pela razão de miRNA/partícula (etapa 10.2.3 ) e o tempo de incubação (16 h, ver passo 6,2).
11. matrizes microfluídicos do multiwell
-
extração de miRNA
- Execute a extração de miRNA conforme descrito na etapa 7.
-
Transcrição reversa
- Descongelar os primers de transcrição reversa, os componentes do kit de transcrição reversa e o MgCl2 (25 mm) no gelo. Para oito amostras, misturar 8 μL de primers de transcrição reversa (10x), 2,25 μL de dNTPs com dTTP (100 mM), 16,88 μL de transcriptase reversa (50 U/μL), 9, 0 μL de tampão de transcrição reversa de 10x, 10,12 μL de MgCl2, 1,12 μL de inibidor de RNase (20 u/μL) e 1 μL de água sem nuclease.
- Misture suavemente e centrifugue brevemente. Adicionar 4,3 μL de mistura de reacção de transcrição inversa a 3,5 μL de miRNA extraído num tubo e misturar, girar para baixo e incubar no gelo durante 5 min. Coloque os tubos em uma máquina termocicreadora e inicie o seguinte programa: 16 ° c por 2 min, 42 ° c por 1 min , e 50 ° c para 1 s repetiu 40x no total, e então, como a reação do batente, 85 ° c por 5 minutos e preensão em 4 ° c até parado.
-
Pré-amplificação
- Descongele os primers no gelo e inverta e centrifugue brevemente. Misture a mistura mestra de pré-amplificação (2x) rodando a garrafa. Preparar a mistura de reacção de pré-amplificação de acordo com as seguintes instruções para oito amostras: 112,5 μL de mistura mestra de pré-amplificação (2x), 22,5 μL de primers de pré-amplificação e 67,5 μL de água sem nuclease. Inverta e centrifugue brevemente.
- Misture 2,5 μL do produto de reacção de transcrição reversa do passo 11.2.2 com 22,5 μL de mistura de reacção de pré-amplificação do passo anterior e inverta e centrifugue brevemente. Incubar as amostras no gelo durante 5 min.
- Coloque os tubos em uma máquina termocicreadora e incubar nas seguintes configurações: ativação enzimática a 95 ° c por 10 min, recozimento a 55 ° c por 2 min, estendendo-se a 72 ° c por 2 min, repetido 12x: desnaturando a 95 ° c por 15 s e recozimento/estendendo-se a 60 ° c por 4 min , inactivação enzimática a 99,9 ° c durante 10 min e 4 ° c em espera.
- Gire para baixo, adicione 7,5 μL de 1x TE (pH 8,0) e 67,5 μL da água nuclease-livre, inverta, e centrifugue momentaneamente. As amostras podem ser armazenadas a-20 ° c por até 1 semana.
-
qPCR
- Misture a mistura mestra girando a garrafa. Prepare a mistura de reacção de PCR para um cartão: 450 μL de mistura máster, 441 μL de água sem nuclease e 9 μL da amostra de pré-amplificação a partir do passo 11.3.4. Inverta e centrifugue brevemente.
- Carregue cada reservatório de enchimento do cartão multipoços microfluídico array com 100 μL de mistura de reação de PCR preparada de acordo com as instruções do fabricante e centrifugue 2x por 1 min a 3.000 x g. A aceleração durante as duas etapas de centrifugação consecutivas é importante para o preenchimento adequado do cartão. Sele o cartão de acordo com as instruções do fabricante.
- Use um sistema do PCR com o seguinte programa: ativação da enzima em 95 ° c por 10 minutos e então, 40x repetido: desnaturando em 95 ° c para 15 s e recozimento/que estende em 60 ° c por 1 minuto.
- Importe o arquivo de resultados do sistema PCR e calcule os valores de RQ usando o pacote de software do sistema com as seguintes configurações de análise: um valor cq máximo permitido de 40,0, valores máximos cq em cálculos incluídos e outliers entre as repetições excluídas. Ative a taxa de descoberta falsa de benjamini – Hochberg como opção para ajuste do valor de p(correção da ocorrência de falsos positivos14) e normalização global como método de normalização (usando um valor de limiar mediano para todas as amostras 15).
Resultados
Um esquema geral de isolamento de partículas de lipoproteínas
A Figura 1 mostra o esquema geral de isolamento das partículas de lipoproteína a partir do sangue total, utilizando a ultracentração seqüencial de flotação16. Se desejar, outras frações de partículas de lipoproteína como VLDL e IDL podem ser colhidas durante este protocolo. O rotor de titânio de ângulo fixo em combinação com tubos de vedação rápida de polipropileno é adequado para resistir às forças de centrifugação. Para evitar o colapso do tubo, é importante evitar bolhas de ar no tubo. A centrifugação é realizada a 4 ° c para minimizar a degradação da proteína. Geralmente partindo do plasma (60-80 mL por doador) de doações de sangue agrupadas de três voluntários, um rendimento de LDL e volumes de solução de partículas de HDL de 3 mL cada um com concentrações na escala de 1-3 mg/mL pode ser esperado. Todo o procedimento, a partir de doação de sangue, demorou cerca de 7 dias.
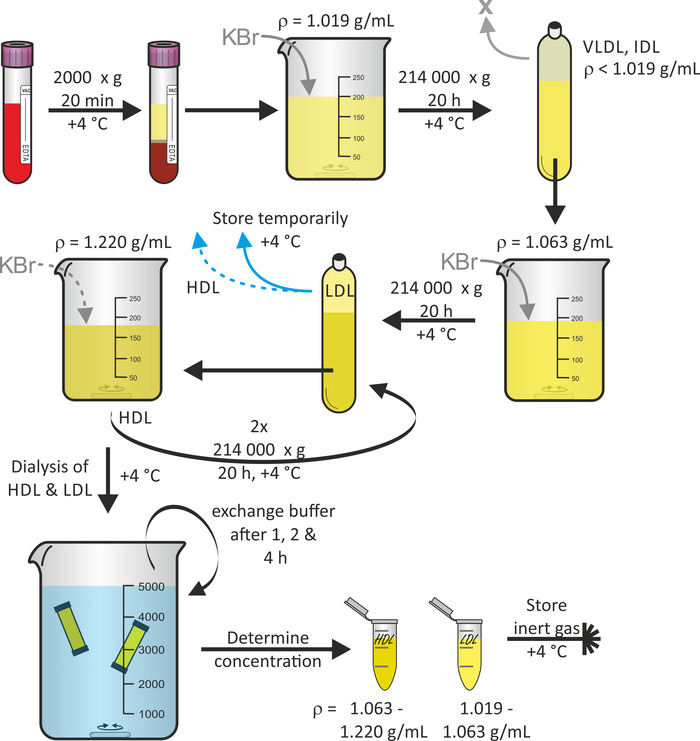
Figura 1: Fluxograma do isolamento da lipoproteína. Centrifugue o sangue dos voluntários saudáveis em tubos do recipiente do vácuo e colete o plasma (fase superior). Após o ajuste de sua densidade para ρ = 1, 19 g/ml usando KBR, centrifugue a solução em 214.000 x g por 20 h a 4 ° c. Após o ajuste da densidade da fração inferior para ρ = 1, 63 g/ml usando KBR, centrifugue a solução novamente em 214.000 x g por 20 h a 4 ° c. Armazene a fração superior contendo partículas de LDL temporárias a 4 ° c. Após o ajuste da densidade da fração inferior para ρ = 1,220 g/ml usando KBR, centrifugue a solução duas vezes a 214.000 x g por 20 h a 4 ° c. Colete a fração superior que contem partículas de HDL, diálise as soluções do HDL e da partícula de LDL e troque o amortecedor após 1, 2, e 4 h. Após 24 h, determine a concentração proteica e guarde as amostras gás inerte a 4 ° c. Estale por favor aqui para ver uma versão maior desta figura.
Reconstituição de partículas de HDL
A reconstituição das partículas de HDL foi realizada de acordo com um protocolo previamente publicado por Jonas7. O primeiro passo foi a delipidação de partículas de HDL, como mostrado na Figura 2a, seguida do segundo passo de relipidation (i.e., reconstituição), como mostrado na Figura 2b, utilizando o PC lipídico, co e C, além de uma mistura de Mirna e spermine. Nós escolhemos o homem maduro miR-223 e miR-155 porque miR-223 mostra uma abundância elevada e miR-155 é raro nas partículas da lipoproteína17. Normalmente, ambas as etapas são executadas em dois dias seqüenciais. Durante a reconstituição, outros componentes lipofílicos e/ou anfifílicos podem ser adicionados como desejado. A evaporação completa do etanol/éter dietílico e solvente metanol/clorofórmio de PC, CO e C foi crítico. A última etapa — como mostrado na Figura 2C— foi o procedimento de diálise para separar as partículas de HDL reconstituídas (rhdl) de lipídios livres/Mirna/detergente. Isso levou um adicional de 1-2 dias. A adição de grânulos absorventes à solução de diálise manteve o gradiente de densidade ao longo da constante da membrana Dialítica. Pode-se esperar um rendimento de 50% de partículas de rHDL.
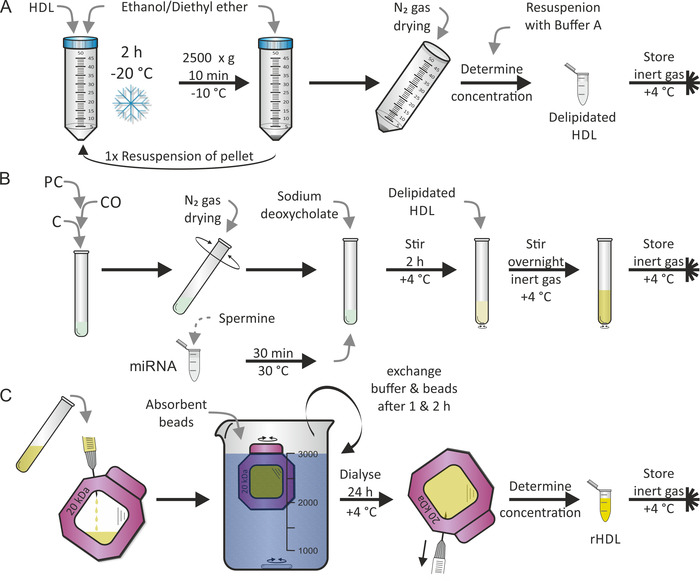
Figura 2: Fluxograma da reconstituição da partícula de HDL. (A) delipidation: Misture a solução de partículas de HDL com etanol pré-resfriado/éter dietílico e incubar a-20 ° c por 2 h. Depois de descartar o sobrenadante, Ressuspender o pellet e repita o procedimento. Seque a pelota com o gás N2 e ressuspender em tampão a. Após a determinação da concentração, armazene o HDL deslipemizado a atmosfera de gás inerte em 4 ° c. (B) reconstituição: após a mistura de fosfatidilcolina (PC), oleato de do cholesteryl (co), e colesterol (C), evaporar o solvente usando o gás N2 ao girar o tubo. Incubar a alíquota de Mirna com solução de espermina por 30 min a 30 ° c, adicionar desoxicolato de sódio e ressuscitar o filme lipídico seco. Agitar a amostra por 2 h a 4 ° c, adicionar a solução de HDL desipidada e agitar a amostra novamente, desta vez durante a noite a 4 ° c em atmosfera de gás inerte. (C) diálise: Transfira a solução do painel B contendo partículas reconstituídas de HDL (rhdl) para uma câmara de membrana de diálise (corte de peso molecular: 20 kDa) e DIALISADA contra PBS e grânulos absorventes a 4 ° c. Troque o tampão e os grânulos após 1 h e 2 h. recupere a solução da partícula de rHDL após 24 h, determine a concentração, e armazene a amostra a atmosfera de gás inerte em 4 ° c. Estale por favor aqui para ver uma versão maior desta figura.
Rotulagem de partículas de LDL
A rotulagem das partículas de LDL com miRNA (Figura 3) como demonstrado para as partículas de HDL não foi viável devido à hidrofobicidade da proteína apob-100, que é o principal constituinte da partícula LDL. O DMSO foi utilizado para a penetração da monocamada lipídica da partícula LDL e, assim, mediou a associação de miRNA. O procedimento inteiro fêz exame de 1-2 dias com um rendimento próximo a 100%.
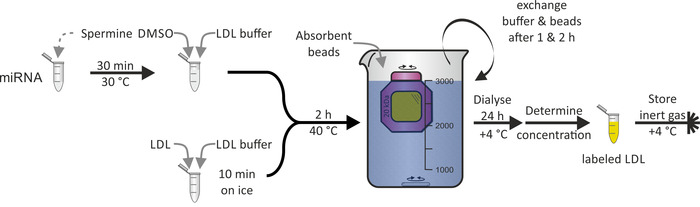
Figura 3: Fluxograma de rotulagem de partículas de LDL. Incubar a alíquota de Mirna com solução de espermina por 30 min a 30 ° c e adicionar tampão DMSO e LDL. Incubar o tampão LDL da sagacidade da amostra de LDL para 10 minutos no gelo e adiciona a mistura de miRNA/spermine/DMSO. Após a incubação a 40 ° c durante 2 h, transfira a solução para uma câmara de membrana de diálise (corte de peso molecular: 20 kDa) e diálise contra PBS e grânulos absorventes a + 4 ° c. Tampão e grânulos da troca após 1 & 2 h. recupere a solução etiquetada da partícula de LDL após 24 h, determine a concentração e armazene-a a atmosfera de gás inerte em + 4 ° c. Estale por favor aqui para ver uma versão maior desta figura.
Controle de qualidade de partículas de lipoproteínas
HS-AFM pode ser usado para examinar o tamanho e a forma das partículas nativas e reconstituídas/etiquetadas da lipoproteína na mica. Pouco antes de usar, mica tem que ser recém-clived (use fita adesiva para remover a camada superior [s]), a fim de fornecer uma superfície limpa e plana. Quando incubando partículas de HDL/LDL em mica, o fator de diluição (e/ou o tempo de incubação) precisa ser ajustado para observar partículas individuais. Os clusters não permitem uma determinação das dimensões das partículas. As partículas de HDL são móveis em mica. Ao usar o AFM convencional em vez do HS-AFM, o protocolo da imobilização precisa de ser adaptado conformemente (amortecedor, revestimento de superfície) para reduzir a mobilidade lateral da partícula. Durante a digitalização da amostra, a força de imagem deve ser mantida baixa (modo de batida) para evitar qualquer deformação das partículas, o que, consequentemente, afetará os valores medidos. Para a análise dos dados, foram detectadas partículas por meio de um algoritmo de limiar (por exemplo, em Gwyddion: grãos > marca por limiar) e sua estatura foi determinada em relação à superfície de mica. A medição da altura das partículas é a forma mais precisa de determinar os tamanhos das partículas, uma vez que as dimensões laterais aparentes são ampliadas pela forma da ponta (ver imagens exemplares na Figura 4). As funções de densidade de probabilidade (PDFs) das alturas de partícula foram calculadas para avaliação estatística e comparação de distribuições de tamanho das diversas partículas de lipoproteína. Uma comparação das partículas de LDL nativas e com rótulo de miRNA, como mostrado na Figura 4 , possibilita verificar a similaridade principal entre as partículas de lipoproteína rotuladas e não rotuladas (i.e., nativas) (identificadas como partículas de LDL sem a adição de Mirna/ as misturas do espermina são mostradas como um controle para o procedimento de rotulagem próprio). Todo o procedimento demorou aproximadamente 1 dia.
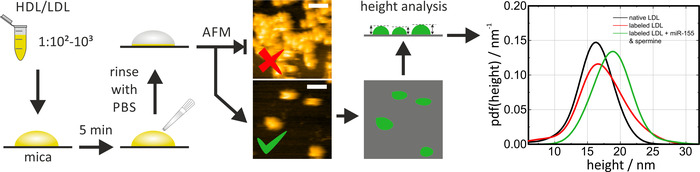
Figura 4: fluxograma e resultados representativos das medições HS-AFM. Diluir a amostra de partículas de HDL/LDL em PBS (1:102-1:103) e incubar-lo em mica recém-cliada por 5 min, seguido por uma enxaguadura cuidadosa com PBS para remover livre (não eletrostaticamente adsorveu) partículas. Realize a imagem latente de HS-AFM e verific a densidade de partícula na superfície. Realize as medições em PBS à temperatura ambiente. A imagem superior desta figura mostra uma densidade de partícula muito alta; a imagem inferior é adequada para análise. A altura das partículas únicas foi analisada após a departicipação e as partículas nativas (curva preta) e reconstituída/rotulada (curva vermelha e verde) foram comparadas em uma avaliação estatística. A barra de escala = 100 nm. Este número foi modificado a partir de Axmann et al.19. Estale por favor aqui para ver uma versão maior desta figura.
extração de miRNA, transcrição reversa e qPCR
A extração de miRNA de lipoproteína nativa/artificialmente enriquecida ou amostras de células foi realizada por meio de um kit de extração de miRNA, como mostra a Figura 5a. Por este meio, um ambiente livre de RNase foi crítico. Esta etapa demorou aproximadamente 1 h. a transcrição reversa da amostra de miRNA extraída (Figura 5b) foi realizada por meio de procedimentos bioquímicos padronizados, conforme descrito pelo fabricante. Esta etapa fêz exame de aproximadamente 1,5 h. Finalmente, a quantidade de cDNA obtida durante o último passo foi determinada por meio da qPCR (Figura 5C). Uma curva padrão, que relaciona os valores obtidos de cq ao número absoluto da vertente de Mirna, rendeu o teor absoluto de Mirna da amostra inicial. Isto fêz exame de aproximadamente 2,5 h.
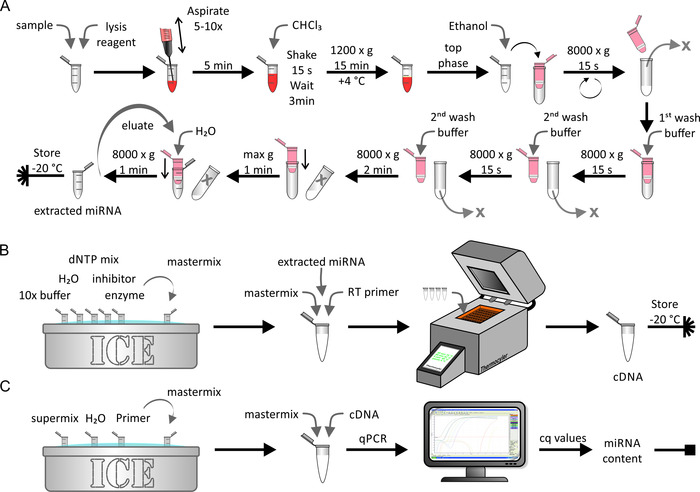
Figura 5: fluxograma de extração de Mirna, transcrição reversa e qPCR. (A) extração de Mirna: Misture a amostra com reagente de Lise e lyse-a através de aspiração usando uma seringa. Incubar por 5 min e adicionar CHCl3. Agitar vigorosamente durante 15 s e incubar durante 3 min. Após centrifugação a 1.200 x g durante 15 min a 4 ° c, recolher a fase de topo e misturá-la com etanol. Transfira a solução para uma coluna de rotação (volume máximo < 700 μL) e centrifugue-a em 8.000 x g por 15 s. descarte o eluente e repita o último passo com o resto da solução. Adicione o primeiro tampão de lavagem e centrifugue em 8.000 x g por 15 s. descarte o eluente, adicione o segundo tampão de lavagem, e centrifugue em 8.000 x g por 15 s. Repita a última etapa com uma época da centrifugação de 2 minutos. Secar ainda mais a membrana através da centrifugação à velocidade máxima durante 1 min. elute o miRNA com H2o e centrifugador em 80.000 x g por 1 min. armazene a amostra de Mirna extraída a-20 ° c. (B) transcrição reversa: descongelar o tampão 10x, H2o, dNTP mistura, inibidor, e enzima no gelo e prepare a mistura mestra. Adicione o Mirna extraído do painel a à mistura mestra e à primeira demão da transcrição reversa e realize a transcrição reversa usando uma máquina do termociclador. Armazene a amostra de cDNA a-20 ° c. (C) qPCR: descongelar o Supermix, H2o, e primer no gelo e preparar a mistura mestra. Adicione o cDNA do painel B à mistura mestra e execute o qPCR. Analise os dados para obter valores de cq e calcule o teor absoluto de Mirna da amostra (ver Figura 6 e os resultados representativos para detalhes). Estale por favor aqui para ver uma versão maior desta figura.
Teor absoluto de miRNA e taxa de transferência
O teor absoluto de miRNA de partículas de HDL e LDL nativas e artificialmente enriquecidas foi calculado a partir dos valores de cq das amostras e uma curva padrão do respectivo Mirna, como mostrado na Figura 6. A Figura 6a mostra os dados calculados pelo software de análise (com normalização dynamictube ativada [para a compensação de diferentes níveis de fundo usando a segunda derivada de cada traço de amostra] e correção de inclinação de ruído [normalização para o nível de ruído]). c valoresq das curvas padrão foram determinados usando a função de limiar de auto-Find do pacote de software no sinal de fluorescência normalizado medido pela máquina qPCR. Por este meio, o software maximizou o valor R do ajuste da curva padrão. O nível de limiar foi mantido constante para cada análise de amostra específica de miRNA. Posteriormente, os valores de cq foram plotados em função do número de fios de Mirna, e uma linha de regressão foi calculada. Os valores da amostra cq foram determinados com o mesmo nível de limiar, como mostrado na Figura 6B; as diferenças da eficiência da reação entre funcionamentos diferentes do qPCR foram compensadas automaticamente pelo software usando uma amostra adicional da curva da calibração incluída em cada funcionamento. Usando a equação da linha de regressão, a quantidade desconhecida de miRNA na amostra poderia ser calculada. O número de partículas de lipoproteína foi estimado a partir da concentração inicial de proteína e seu peso molecular médio (MWHDL ~ 250 kDa). Assim, não foi assumido qualquer contribuição lipídica para o peso molecular — assim, o número de fios de miRNA por partícula de lipoproteína foi ligeiramente superestimado. Além disso, uma taxa de recuperação de 100% de miRNA durante a etapa de extração de miRNA foi assumida. Além disso, o teor de miRNA das células antes e após a incubação com partículas de HDL foi determinado e a taxa de transferência de miRNA foi calculada conforme mostrado na Figura 6C.
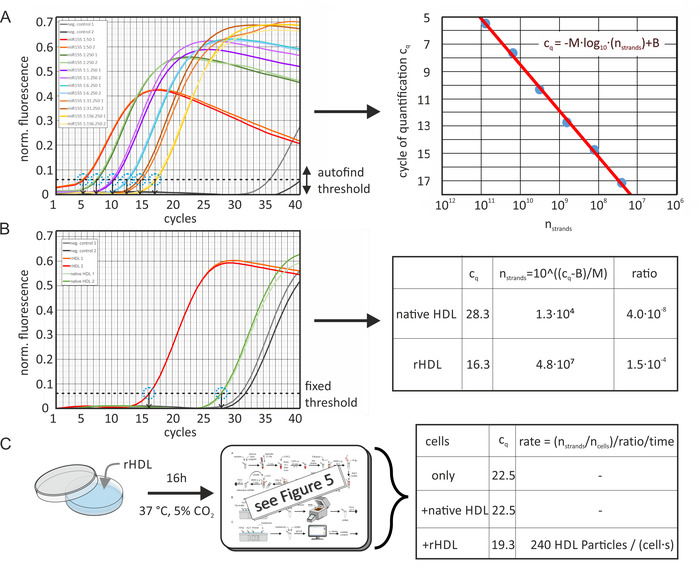
Figura 6: fluxograma de cálculo do teor absoluto de Mirna e taxa de transferência. (A) curva padrão para mir-155: uma alíquota mir-155 (100 μl, 10 μm) foi diluída em série com água sem RNA, conforme indicado. o qPCR rendeu valores de cq para cada amostra de diluição serial (medido duas vezes) usando a função de limite de encontrar automaticamente do pacote de software. Experimentos de controle negativo (sem adição de miRNA) produziram valores de cq de > 35. Os pontos de dados dos valores de cq como função do número de fios de Mirna por volume amostral (calculado a partir da concentração inicial e das diluições seriadas) foram ajustados com a equação apresentada (linha vermelha, imagem direita), produzindo M =-3,36 e B = 42,12. A eficiência determinada do PCR era 0,98. As barras de erro foram calculadas a partir dos resultados das repetições experimentais e foram menores do que o diâmetro do círculo de pontos de dados. (B) os valores de cq das partículas de HDL nativas/artificialmente enriquecidas foram determinados com o mesmo nível de limiar determinado no painel a e convertidos para o número de fios de Mirna no volume da amostra de qPCR. A razão absoluta de miRNA da amostra original foi calculada a partir do número (concentração) de partículas de HDL no volume amostral (3,2 x 1011 partículas). (C) amostras de células (linhagem celular LDLA7-SRBI) foram incubadas por 16 h com partículas de HDL enriquecidas artificialmente (50 μg/ml) e analisadas de forma semelhante. Os valores de cq determinados foram 22,5, 22,5 e 19,3 para células apenas, para células INCUBADAS com HDL nativo, ou para células incubadas com solução de partículas de rhdl (ambos 50 μg/ml), respectivamente. Esses valores foram convertidos para o número de fios de miRNA como feito no painel B. O número de fios de miRNA após incubação (7,3 x 106) foi corrigido por subtração do número de fios de Mirna antes da incubação (8,6 x 105). O resultado foi dividido pelo número de células no volume amostral (3.100), a relação miRNA-partícula (1,5 x 10-4) e o período de incubação (16 h). Isto rendeu a taxa de transferência de partículas da lipoproteína através da tomada de miRNA (240 eventos da captação da partícula de HDL por a pilha e segundo). Este número foi modificado a partir de Axmann et al.19. Estale por favor aqui para ver uma versão maior desta figura.
Multiwell microfluídicos array
Devido aos pequenos rendimentos da extração de miRNA, a transcrição reversa do miRNA extraído foi seguida por uma etapa de pré-amplificação. Finalmente, o qPCR, como mostrado na Figura 6, foi realizado. Para todas as etapas, os procedimentos bioquímicos padrão foram usados como descrito pelo fabricante. Aqui, uma parte do perfil global do miRNA em partículas de HDL de pacientes urêmica recrutados para um estudo na influência de CRF no efluxo do colesterol dos macrófagos18 é mostrada. Neste estudo, a capacidade do aceitador de colesterol de HDL ou soro em — além de outros — 17 pacientes urêmica adultos novos (CKD estágios 3-5) e 14 pacientes de hemodiálise adultos novos sem doenças associadas e controles combinados foi medido. Para analisar os dados, foram usadas as configurações padrão (valor máximo permitido de TC: 40,0, incluindo valores máximos de TC em cálculos e excluindo outliers entre réplicas). Os valores de Pforam ajustados utilizando-se a taxa de falsa descoberta de benjamini-Hochberg (correção da ocorrência de falsos positivos) e, como método de normalização, foi selecionada a normalização global , que encontra os ensaios comuns entre todos os amostras para usar sua mediana CT para normalização. Nos resultados representativos, alguns RQs de miRNAs isolados de HDLs de pacientes urêmicos são representados (RQs de controles são 1). Obviamente, Mir-122 e Mir-224 são expressos altamente nos HDLs de pacientes urêmica. Este passo inteiro demorou aproximadamente 1 dia.
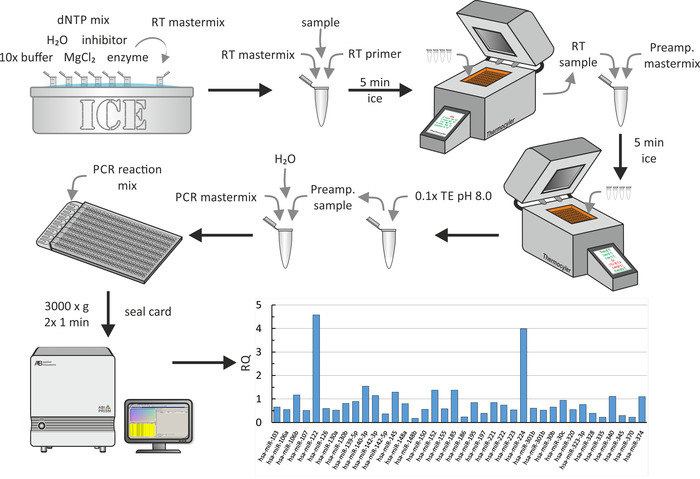
Figura 7: fluxograma e resultados representativos do array microfluídico multipoços. Após a extração de miRNA, como mostra a Figura 5a, misturar a amostra de Mirna com primer de transcrição reversa e uma mistura mestra contendo tampão 10x, H2O, dNTP Mix, inibidor, MgCl2e enzima. Após a incubação no gelo por 5 min, realize a transcrição reversa usando uma máquina termocicreadora. Adicione a mistura mestra da pré-amplificação, incubar por 5 minutos no gelo, e realize a pré-amplificação usando uma máquina do termociclador. Adicionar 0.1 x TE (pH 8,0) e misturar uma alíquota com mistura mestra de PCR e H2o. Pipetar a mistura de reação de PCR na porta de preenchimento do array microfluídico multipoços e girar duas vezes a 3.000 x g por 1 min cada. Realizar qPCR utilizando um sistema de PCR e analisar os dados para produzir valores de RQ (aqui, a figura mostra valores de RQ de partículas de HDL de pacientes com uremia em comparação com um grupo de controle saudável18). Estale por favor aqui para ver uma versão maior desta figura.
Discussão
Aqui, o isolamento das frações de partículas de lipoproteínas do sangue humano e a determinação de seu conteúdo individual de miRNA é descrito passo a passo. É crítico trabalhar em um ambiente livre de RNase ao segurar o Mirna isolado e sintetizado — o miRNA partícula-encaixado é protegido obviamente da degradação enzimática. Como a razão de miRNA/partícula de partículas de lipoproteína nativa é bastante baixa, o enriquecimento artificial com miRNA é necessário para estudar a captação de partículas de holo de células. Assim, a reconstituição das partículas de HDL como descrito anteriormente7 é modificada para incorporar os fios de Mirna. Adicionalmente, a separação da fração lipídica e protéica durante este procedimento permite que os cientistas examinem os componentes associados a lipídios e proteínas da partícula de lipoproteína19. De forma semelhante, o procedimento de rotulagem das partículas de LDL é adaptado. Curiosamente, a adição de spermine — um estabilizador natural de nucleotídeos — não influenciou a relação de miRNA/partícula. Deve-se notar que, em princípio, o método permite o indobramento de outras substâncias do que o miRNA dentro de uma partícula de lipoproteína. Naturalmente, há um limite no que diz respeito ao tamanho físico da substância com base no tamanho total de HDL (diâmetro: 5-12 nm) e partículas de LDL (diâmetro: 18-25 nm).
Em relação ao controle de qualidade das partículas de lipoproteína reconstituídas/rotuladas, o HS-AFM é um método aplicável para caracterizar as partículas de HDL/LDL no nível de partícula única. Em comparação com em, permite tempos de preparação mais curtos e condições quase fisiológicas (molhado, temperatura ambiente).
Devido à sua sensibilidade e amplificação inerentes, a qPCR é o método de escolha para detectar baixas concentrações de miRNA. Alternativamente, a microscopia sensível da fluorescência da único-molécula, que pode detectar mesmo moléculas individuais, não seria apropriada devido às baixas concentrações de, por exemplo, as costas fluorescente etiquetadas de Mirna por a partícula. Assim, a proporção de fios de miRNA por partícula de lipoproteína nativa foi encontrada para ser 10-8. O enriquecimento artificial aumenta a razão por um fator de 10.000, o que facilita a estimativa da taxa de captação da lipoproteína celular (não se detecta diferença significativa com as partículas de lipoproteína nativa 19). A alta sensibilidade da qPCR possibilita determinar essa taxa de captação medindo o número de cordões de miRNA após o tempo de incubação e a razão de miRNA/partícula. Deve-se notar que o valor calculado ignora a degradação celular e liberação de miRNA e, portanto, representa pelo menos um limite inferior para a relação de captação de partículas de lipoproteína.
No futuro, o método pode ser adaptado para transferir substâncias farmacêuticas (nomeadamente também as lipofílicas) nas células e correlacionar o seu efeito biológico à concentração intracelular (determinada através da taxa de absorção de partículas de lipoproteínas).
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi apoiado pelo projeto Austrian do fundo da ciência P29110-B21, o projeto H-3065/2011 do "Hochschuljubiläumsstiftung der Stadt Wien zur Förderung der Wissenschaft", o Fundo Europeu para o desenvolvimento regional (EFRE, IWB2020), o estado federal da parte superior Áustria, e o "Land OÖ Basisfinanzierung".
Materiais
| Name | Company | Catalog Number | Comments |
| Ultracentrifuge | Beckman | Optima L-100 XP | Lipoprotein isolation |
| Ultracentrifuge rotor | Beckman | TI 55.2 | Lipoprotein isolation |
| Vacutainer (EDTA) | Becton Dickinson | 366643 | Lipoprotein isolation |
| Kaliumbromid | Sigma Aldrich | 2110 | Lipoprotein isolation |
| Ultracentrifuge tubes | Beckman | 342414 | Lipoprotein isolation |
| Ultracentrifuge tube sealer | Beckman | 342428 | Lipoprotein isolation |
| Sodiumchlorid | Carl Roth GmbH | P029.2 | Lipoprotein isolation |
| EDTA | Fisher Scientific | D/0650/50 | Lipoprotein isolation |
| Dialysis tubes, MWCO 12 - 14k | Spectra | 132700 | Lipoprotein isolation |
| synthetic miRNA | microSynth | - | synthetic miRNA |
| TRIS buffer pH 7 | Ambion | AM9850G | synthetic miRNA |
| TRIS buffer pH 8 | Ambion | AM9855G | synthetic miRNA |
| sterile tubes | Carl Roth GmbH | ENE8.1 | synthetic miRNA |
| Photometer | Eppendorf | Eppendorf Biophotometer | Bradford assay |
| Cuvette | Carl Roth GmbH | XK20 | Bradford assay |
| Coomassie G-250 Stain | BioRad GmbH | 161-0786 | Bradford assay |
| Sodiumchlorid | Carl Roth GmbH | 3957.1 | Delipitation |
| EDTA | Fluka Analytical | 03690-100ML | Delipitation |
| TRIS buffer pH 8.0 | Ambion | AM9855G | Delipitation |
| Ethanol 100% | AustrAlco | Ethanol Absolut 99.9% | Delipitation |
| Diethyl ether | Carl Roth GmbH | 3942.1 | Delipitation |
| Centrifuge | Thermo Scientific | Multifuge X3R | Delipitation |
| Chloroform | Carl Roth GmbH | 3313.1 | Reconstitution |
| Methanol | Carl Roth GmbH | 4627.1 | Reconstitution |
| Phosphatidylcholine | Sigma Aldrich GmbH | P3556-25mg | Reconstitution |
| Cholesterol oleate | Sigma Aldrich GmbH | C-9253-250mg | Reconstitution |
| cholesterol | Sigma Aldrich GmbH | C8667-500mg | Reconstitution |
| glass tubes | Carl Roth GmbH | K226.1 | Reconstitution |
| spermine | Sigma Aldrich GmbH | S3256-1G | Reconstitution |
| Thermomixer | Eppendorf | Thermomixer comfort | Reconstitution |
| Sodium deoxycholate | Sigma Aldrich GmbH | 3097-25G | Reconstitution |
| Magnetic stir bar | Carl Roth GmbH | 0955.2 | Reconstitution |
| 100µL micropipettes | Carl Roth GmbH | A762.1 | Reconstitution |
| PBS | Carl Roth GmbH | 9143.2 | Dialysis |
| Amberlite XAD-2 beads | Sigma Aldrich GmbH | 10357 | Dialysis |
| Slide-A-Lyzer casette (cut-off 20 kDa) | Thermo Scientific | 66003 | Dialysis |
| Syringe | Braun | 9161406V | Dialysis |
| Syringe needle | Braun | 465 76 83 | Dialysis |
| EGTA | Carl Roth GmbH | 3054.2 | Labeling of LDL particles |
| DMSO | life technologies | D12345 | Labeling of LDL particles |
| Atomic Force Microscope | RIBM | SS-NEX | Quality control of reconstituted/labeled lipoprotein particles |
| Muscovite Mica (V-1 Grade) | Christine Gröpl | G250-1/V1 | Quality control of reconstituted/labeled lipoprotein particles |
| AFM Cantilever | Nanoworld | USC-F1.2-k0.15 | Quality control of reconstituted/labeled lipoprotein particles |
| Gwyddion 2.49 | Czech Metrology Institute | http://gwyddion.net | Quality control of reconstituted/labeled lipoprotein particles |
| Chamber slides, Nunc Lab-Tek | VWR | 734-2122 | Cell culture |
| HBSS | Carl Roth GmbH | 9117.1 | Cell culture |
| Cell counter | Omni Life Science GmbH | Casy | Cell culture |
| miRNeasy Mini Kit | QIAGEN | 217004 | miRNA extraction |
| Centrifuge | Eppendorf | 5415R | miRNA extraction |
| 20G needle | Braun | Sterican 465 75 19 | miRNA extraction |
| 5ml syringe | Becton Dickinson | 309050 | miRNA extraction |
| PCR cabinet | Esco | PCR-3A1 | Reverse Transcription |
| TaqMan Reverse Transcription Kit | Thermo Scientific | 4366596 | Reverse Transcription |
| Rnase-free water | Carl Roth GmbH | T143 | Reverse Transcription |
| 0.2 ml tubes | Brand | 781305 | Reverse Transcription |
| Centrifuge | Carl Roth GmbH | Microcentrifuge AL | Reverse Transcription |
| Thermocycler | Sensoquest | Labcycler | Reverse Transcription |
| TaqMan Primer | Thermo Scientific | - | qPCR |
| iTaq Universal probe supermix | BioRad GmbH | 1725131 | qPCR |
| PCR machine | Corbett | Rotor-Gene RG-6000 | qPCR |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366596 | TaqMan Array |
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | TaqMan Array |
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 | TaqMan Array |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399933 | TaqMan Array |
| TaqMan Universal Master Mix II, no UNG | Applied Biosystems | 4440040 | TaqMan Array |
| TaqMan Advanced miRNA Human A Cards | Applied Biosystems | A31805 | TaqMan Array |
| Nuclease-free water | Thermo Scientific | R0581 | TaqMan Array |
| MgCl2 (25mM) | Thermo Scientific | R0971 | TaqMan Array |
| TE, pH 8.0 | Invitrogen | AM9849 | TaqMan Array |
| 7900HT Fast Real-Time PCR System | Applied Biosystems | 4351405 | TaqMan Array |
| TaqMan Array Card Sealer | Applied Biosystems | - | TaqMan Array |
Referências
- Vickers, K. C., Palmisano, B. T., Shoucri, B. M., Shamburek, R. D., Remaley, A. T. MicroRNAs are transported in plasma and delivered to recipient cells by high-density lipoproteins. Nature Cell Biology. 13 (4), 423-435 (2011).
- Tabet, F., et al. HDL-transferred microRNA-223 regulates ICAM-1 expression in endothelial cells. Nature Communications. 5, (2014).
- Wahid, F., Shehzad, A., Khan, T., Kim, Y. Y. MicroRNAs: Synthesis, mechanism, function, and recent clinical trials. Biochimica et Biophysica Acta - Molecular Cell Research. 1803 (11), 1231-1243 (2010).
- Carthew, R. W., Sontheimer, E. J. Origins and Mechanisms of miRNAs and siRNAs. Cell. 136 (4), 642-655 (2009).
- Fabian, M. R., Sonenberg, N., Filipowicz, W. Regulation of mRNA Translation and Stability by microRNAs. Annual Review of Biochemistry. 79 (1), 351-379 (2010).
- Filipowicz, W., Bhattacharyya, S. N., Sonenberg, N. Mechanisms of post-transcriptional regulation by microRNAs: Are the answers in sight?. Nature Reviews Genetics. 9 (2), 102-114 (2008).
- Jonas, A. Reconstitution of High-Density Lipoproteins. Methods in Enzymology. , 553-582 (1986).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Bustin, S. A., et al. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Patsch, J. R., Patsch, W. Zonal ultracentrifugation. Methods in Enzymology. 129 (1966), 3-26 (1986).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Yamamoto, D., et al. High-Speed Atomic Force Microscopy Techniques for Observing Dynamic Biomolecular Processes. Methods in Enzymology. 475, 541-564 (2010).
- Stangl, H., Cao, G., Wyne, K. L., Hobbs, H. H. Scavenger receptor, class B, type I-dependent stimulation of cholesterol esterification by high density lipoproteins, low density lipoproteins, and nonlipoprotein cholesterol. Journal of Biological Chemistry. 273 (47), 31002-31008 (1998).
- Hochberg, B. Controlling the False Discovery Rate: a Practical and Powerful Approach to Multiple Testing. Journal of the Royal Statistical Society. 57 (1), 289-300 (1995).
- Mestdagh, P., et al. A novel and universal method for microRNA RT-qPCR data normalization. Genome Biology. 10 (6), (2009).
- Schumaker, V. N., Puppione, D. L. 6] Sequential Flotation Ultracentrifugation. Methods in Enzymology. 128 (C), 155-170 (1986).
- Wagner, J., et al. Characterization of levels and cellular transfer of circulating lipoprotein-bound microRNAs. Arteriosclerosis, Thrombosis, and Vascular Biology. 33 (6), 1392-1400 (2013).
- Meier, S. M., et al. Effect of chronic kidney disease on macrophage cholesterol efflux. Life Sciences. 136, 1-6 (2015).
- Axmann, M., et al. Serum and Lipoprotein Particle miRNA Profile in Uremia Patients. Genes. 9 (11), 533 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados