Method Article
Dissection, culture et analyse des cellules neurales primaires de crête neurale de la souris pour l'étude de la délamination et de la migration neurales de cellules de crête
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit la dissection et la culture des cellules de crête neurale crânienne des modèles de souris, principalement pour l'étude de la migration cellulaire. Nous décrivons les techniques d'imagerie en direct utilisées et l'analyse des changements de vitesse et de forme cellulaire.
Résumé
Au cours des dernières décennies, il y a eu une disponibilité accrue de modèles de souris génétiquement modifiés utilisés pour imiter les pathologies humaines. Cependant, la capacité d'étudier les mouvements cellulaires et la différenciation in vivo est encore très difficile. Les neurocristopathies, ou désordres de la lignée neurale de crête, sont particulièrement difficiles à étudier en raison d'un manque d'accessibilité des étapes embryonnaires clés et des difficultés en séparant le mesenchyme neural de crête du mésenchyme mésodermal adjacent. Ici, nous avons entrepris d'établir un protocole bien défini et de routine pour la culture des cellules neurales primaires de crête neurale. Dans notre approche, nous disséquons la bordure de plaque neurale de souris pendant l'étape initiale d'induction de crête neurale. La région frontalière de plaque neurale est explantée et cultivée. Les cellules de crête neurale forment dans une feuille épithéliale entourant la bordure de plaque neurale, et par 24 h après explantation, commencent à délaminer, subissant une transition épithéliale-mesenchymal (EMT) pour devenir des cellules entièrement motielles de crête neurale. En raison de notre approche de culture bidimensionnelle, les populations distinctes de tissu (plaque neurale contre crête neurale prémigratoire et migratoire) peuvent être facilement distinguées. À l'aide d'approches d'imagerie en direct, nous pouvons ensuite identifier les changements dans l'induction de la crête neuronale, l'EMT et les comportements migratoires. La combinaison de cette technique avec des mutants génétiques sera une approche très puissante pour comprendre la biologie normale et pathologique des cellules de crête neurale.
Introduction
La lignée de la crête neurale (NC) est une population transitoire, multipotente et migratrice de cellules qui apparaît exclusivement chez les vertébrés au début du développement embryonnaire1,2. Les dérivés de crête neurale sont extrêmement divers, et incluent la glie, le muscle lisse, les mlanocytes, les neurones et l'os craniofacial et le cartilage3,4. Parce que la crête neurale contribue à la fonction de nombreux systèmes d'organes, cette lignée est essentielle pour l'embryogenèse humaine. Le développement aberrant de NC est impliqué dans un large éventail des défauts de naissance humains les plus communs (c.-à-d., lèvre et palais de fissure)5, et également des désordres tels que Hirschsprung' la maladie de s (HSCR), syndrome de Wardensburg (WS), syndrome de CHARGE et syndrome de Williams6 ,7,8,9.
Le développement du NC a été exploré dans un certain nombre de modèles modèles non mammifères, y compris les modèles Xenopus,poussins et poissons zèbres. Chez les mammifères, le travail dans des modèles de souris a identifié certains des événements génétiques clés sous-jacents au développement de la crête neurale; cependant, il a été plus difficile de suivre la biologie cellulaire de la migration de crête neurale, en raison de l'inaccessibilité de l'embryon de souris (revue ailleurs10,11). En outre, alors que des études sur les poussins, les xenopus et les poissons zèbres ont établi un réseau de régulation génétique pour nC, la perte d'études fonctionnelles dans ces modèles animaux ne présente parfois pas un phénotype comparable chez la souris. Par exemple, chez Xenopu,poisson zèbre et poussin, la signalisation non canonique Wnt est l'un des mécanismes cellulaires qui permet au NC d'acquérir sa capacité migratoire12,13,14,15 . Cependant, chez la souris, la perte de signalisation Wnt non canonique ne semble pas affecter la migration16. Comme la migration in vivo NC a été difficile à suivre pendant de longues périodes chez la souris, il n'est pas clair si ces espèces-différences reflètent des modes de migration différents, ou des différences dans la régulation moléculaire.
Comme indiqué, les études NC chez la souris ont été très difficiles parce que la culture ex utero des embryons est laborieuse. En outre, le NC est constamment en contact intime avec les tissus adjacents tels que le mesoderm et le neurectoderm. L'utilisation récente de conducteurs de crête neurale spécifiques à la crête ou de colorants exogènes nous a permis d'étiqueter fluorescentement le NC migrateur; toutefois, ces approches sont encore limitées. Malgré de multiples rapports décrivant différentes techniques pour visualiser la migration NC17,18, il a été difficile de résoudre ces techniques dans une procédure simple et routinière.
Il est clair qu'il y a un besoin de techniques qui permettent la manipulation et la caractérisation des mammifères NC. Nous avons concentré nos efforts sur la souris crânienne NC car il est le modèle principal pour étudier le développement craniofacial humain et les neurocristopathies. Nous avons affiné notre approche basée sur plusieurs rapports intéressants décrivant la culture primaire des cellules NC19,20,21. Ici, nous décrivons soigneusement les techniques optimales de culture pour explanter les cellules primaires de NC. Nous démontrons la méthode d'imagerie cellulaire vivante et l'utilisation optimale de différentes matrices pour enrober les plaques de culture. Notre protocole décrit comment capturer la migration des cellules NC vivantes à l'aide d'un microscope inversé, qui est conçu comme une ligne directrice pour une utilisation avec d'autres microscopes, ainsi qu'un résumé détaillé de nos analyses cellulaires.
Le résultat attendu de l'explante devrait être une distribution magnifiquement aménagée des cellules qui sont clairement distinguées sous le microscope, où l'on peut voir trois populations différentes de cellules qui représentent (i) plaque neurale, (ii) prémigratoire, et, (iii) cellules de crête neurale migratrices. Nous démontrons comment analyser les comportements cellulaires à la frontière de la population prémigratoire des cellules pendant la transition épithéliale-mesenchymal. Nous avons également concentré nos efforts sur l'étude des cellules entièrement migratrices pour la vitesse, la distance et la morphologie cellulaire.
Protocole
Tous les travaux sur les animaux ont fait l'objet d'une approbation éthique par le King's College London Ethical Review Process et ont été effectués conformément à la licence de projet du Home Office du Royaume-Uni P8D5E2773 (KJL).
1. Préparation des réactifs
- Préparer des solutions et des outils généraux, y compris la saline stérile de tampon de phosphate (PBS), l'éthanol à 70 %, les outils de dissection (forceps et lames de dissection ou aiguilles stériles), les plaques en plastique ou les lames de verre recouvertes d'une matrice extracellulaire disponible dans le commerce ( ECM) à base d'hydrogel ou de fibronectin (voir le Tableau des matériaux), et des supports de crête neurale (voir ci-dessous).
- Préparer le milieu basal de crête neurale à l'aide du milieu d'aigle modifié de Dulbecco (DMEM, 4500 mg/L de glucose), 15 % de sérum bovin fœtal (FBS), 0,1 mM d'acides aminés essentiels moyens minimums non essentiels (MEM NEAA 100X), 1 mM de pyruvate de sodium, 55 m-mercaptoethanol, 100 unités/mL de pénicilline, 100 unités/mL de streptomycine et 2 mM de L-glutamine.
- Conditionner les médias du jour au lendemain à l'aide de cellules d'alimentation STO inhibées par la croissance21.
- Préparer les cellules STO (voir le tableau des matériaux)pour contenir le DMEM complété par 10 % de FBS et 100 u/mL de pénicilline, 100 streptomycine U/mL. Cultivez et détendez les cellules STO à la confluence en flacons de 25 cm2 recouverts de gélatine de 0,1 %. Appliquer 5000 rad d'irradiation gamma.
- Seed environ 3 x 106 cellules inhibées par la croissance sur un plat de 10 cm2 ou 25 cm2 flacon (à partir de l'étape 1.2.1.1). Ajouter environ 10 à 12 ml de milieu basal de crête neurale et incuber toute la nuit.
REMARQUE : Les cellules ensemencées peuvent être utilisées pour produire un milieu conditionnel jusqu'à 10 jours. Vérifier régulièrement l'apparence des cellules
- Filtrer le milieu (0,22 m de taille de pores) et compléter avec 25 ng/mL facteur de croissance de base de fibroblaste (bFGF) et 1000 U du facteur inhibiteur de leucémie (LIF).
REMARQUE : Conserver à 4 oC et l'utiliser dans un délai d'un mois ou le magasiner à -20 oC et l'utiliser dans les 3 mois.
- Conditionner les médias du jour au lendemain à l'aide de cellules d'alimentation STO inhibées par la croissance21.
- Enrober les surfaces de culture tissulaire d'une matrice extracellulaire.
REMARQUE : Selon la question biologique posée, la matrice peut être enduite sur des plats de culture à fond de verre, des plats de culture de tissus en plastique ou des feuillets de couverture en verre. Voir ci-dessous pour les différentes dilutions hydrogel à base d'ECM dépendant du substrat matriciel. La fibronectine a été testée sur des plats à fond de verre et les feuillets de couverture seulement aux concentrations spécifiées ci-dessous. Ici, nous nous référerons aux surfaces recouvertes de substrat sous le titre de « plaques enduites ».- Enrober les surfaces de culture tissulaire d'hydrogel à base d'ECM.
REMARQUE : Gardez le substrat froid jusqu'au placage, soit en refroidissant le support, soit en restant sur la glace.- Décongeler l'hydrogel à 4 oC pendant la nuit. Ajouter 5 mL de 10% FBS en DMEM à 5 ml d'hydrogel pour un volume final de 10 ml (voir le Tableau des Matériaux).
- Faire des aliquots de 0,5 à 1 ml comme pratique et conserver à -20 oC.
- Décongeler les aliquots hydrogel sur la glace.
- Utilisez une dilution 1:20 du stock d'hydrogel pour enrober le plastique.
- Utilisez une dilution de 1:5 du stock d'hydrogel pour enrober les lames de verre et les plaques de culture de tissu à fond de verre.
REMARQUE: Diluer l'hydrogel dans le DMEM froid. - Appliquer suffisamment d'hydrogel dilué pour couvrir la zone désirée sur les plaques/lames et couver pendant 30 à 45 min à 37 oC.
- Utilisez immédiatement des assiettes/glissières enduites ou rangez les lames enduites à 4 oC pendant la nuit.
- Enlever les excès et rincer les lames avec un DMEM à teneur élevée en glucose (facultatif) avant utilisation.
- Enrober les surfaces de culture tissulaire de fibronectin.
- Faire des aliquots de 1 mg/mL de cofibrose et stocker à -80 oC. Diluer la fibronectine avec le PBS (dPBS) de Dulbecco jusqu'à une concentration finale de 1 g/mL.
- Appliquer suffisamment de fibronectin pour couvrir la zone désirée et couver à température ambiante pendant 15 min.
- Retirer la fibronectine résiduelle et laisser sécher le verre de 30 à 45 min.
- Rincer les puits ou les borderles de couverture avec un DMEM à teneur élevée en glucose (facultatif) avant utilisation.
- Enrober les surfaces de culture tissulaire d'hydrogel à base d'ECM.
2. Jour 1 : Dissection des embryons de stade somite précoce
REMARQUE : Utilisez des outils stériles et des solutions stériles. Si le génotypage est nécessaire, recueillir le corps de l'embryon pour l'extraction de l'ADN.
- La dissection de la plaque neurale crânienne est limitée aux embryons à 8,5 jours après le coitum (dpc). Sélectionnez les embryons à l'étape de la somite 5 à 8. Disséquer l'utérus en PBS et couper le mesometrium pour séparer chaque embryon (Figure 1A). La paroi musculaire de l'utérus se contracte et le tissu décidual deviendra visible (Figure 1B).
REMARQUE : Maintenir les embryons dans l'utérus dans le PBS glacé pendant que les dissections sont exécutées un embryon à la fois. Déplacez les embryons avec une pipette Pasteur en verre dans un PBS stérile frais afin d'améliorer la visibilité et de réduire la contamination. - Faites glisser les forceps entre la couche musculaire et le tissu décidual et retirez la couche musculaire à l'appui d'une deuxième paire de forceps (Figure 1C).
- À l'aide de forceps, percer le déciduum sur les bords du mât méotrial et avec une deuxième paire de forceps déchirer pour s'ouvrir perpendiculairement au poteau.
- Pelez le tissu décidual avec les forceps pour visualiser la membrane du Reichert.
- Retirez soigneusement la membrane de Reichert. Le sac de jaune viscéral devient visible et l'embryon peut être vu à l'intérieur (Figure 1D).
- Retirez le sac viscéral de jaune et l'amnion (Figure 1E) et placez l'embryon afin de visualiser le pli de la tête (Figure 1F).
- Couper le pli de la tête au-dessus du cœur et gratter le mesoderm sous-jacent à l'aide de forceps et/ ou d'outils de cils pour obtenir une plaque neuronale propre (NP) (Figure 1H).
REMARQUE: Le NP peut être maintenu entier ou divisé sur l'axe antéropostérieur de sorte que chaque côté peut être plaqué individuellement. La bordure de plaque neurale peut être encore coupée loin de la plaque neurale afin de réduire au minimum les contributions neuronales aux explants. - Utilisez une pipette Pasteur en verre pour transférer la plaque neurale disséquée sur un plat recouvert d'hydrogel rempli de support de crête neurale conditionné.
- Faites tourbillonner doucement le plat pour positionner le NP au milieu du puits. Ceci est important pour maximiser la qualité de phase pour l'imagerie des cellules vivantes (le jour 2).
- Incuber la nuit (ou au point de temps désiré) à 37 oC dans 5 % de CO2. Les cellules de crête neurale devraient migrer visiblement hors de la plaque neurale.
REMARQUE : Les cellules se fixent habituellement dans les 6 à 8 h. Après l'explantation se fixe, laissez plus de temps pour visualiser les cellules migrantes. Habituellement par 24 h après l'explantation, nous pouvons trouver trois populations distinctives de cellules. La première population, au centre de l'explantation, est la plaque neurale (NP). La deuxième population, le NC prémigrateur (PNC), entoure le NP dans une feuille épithéliale de cellules. La troisième population, dans l'anneau extérieur, est formée de NC migrateur (mNC), qui sont de plus grande taille, et semblent entièrement mésenchymales (figure 2).
3. Jour 2 : Imagerie cellulaire vivante des cellules de crête neurale crânienne murine
REMARQUE : L'imagerie doit être exécutée à 24 h après l'explantation afin d'imager de manière optimale et de quantifier la migration des cellules de crête neurale. Les supports à induction NC n'ont pas besoin d'être rafraîchis avant l'imagerie cellulaire en direct. L'accès à un microscope inversé, avec une scène motorisée et une chambre d'environnement incorporée est nécessaire. Utiliser des plats de culture tissulaire multi-puits adaptés à l'imagerie (Tableau des matériaux).
-
Configuration du microscope
- Fixer la chambre de l'environnement à 37 oC et 5 % de CO2.
- Percer un trou dans le couvercle du couvercle de la plaque de culture tissulaire pour permettre à l'aiguille CO2, reliée à la chambre d'humidification CO2, de s'asseoir à l'intérieur de la plaque.
- Placez le plat de culture tissulaire dans le porte-échantillon et collez le couvercle de la plaque et l'aiguille co2 pour éviter les secousses lors de l'acquisition de plusieurs puits.
- Allumez le contrôleur de microscope, le contrôleur de scène et le logiciel d'imagerie.
- Concentrez-vous sur les cellules NC crâniennes au grossissement 10x (avec anneau de phase correspondant dans le condenseur sélectionné).
- Réglez le contraste de phase de haute qualité sur le microscope en ajustant le diaphragme d'iris de champ, le diaphragme d'iris d'ouverture et le télescope de centrage, comme spécifié dans le manuel de configuration de microscope.
-
Imagerie à cellules vivantes de contraste de phase
- Définir l'annuaire ou l'emplacement du fichier où les fichiers time-lapse seront enregistrés.
- Définir le temps d'exposition, le binning et la zone de la caméra.
- Définir le nombre de points de temps, la durée de l'imagerie et l'intervalle de temps entre les images.
- Pour quantifier la capacité migratoire des cellules NC, mettre le microscope à 10x grossissement, en prenant 1 cadre toutes les 5 min (217 points de temps sur 18 h). Pour quantifier la morphologie cellulaire, définir le grossissement à 40x, en prenant 1 cadre/min (61 points de temps sur 1 h). Pour quantifier la dynamique lamellipodial, mettre grossissement à 40x ou 60x grossissement, en prenant 1 cadre tous les 10 s (plus de 10 min).
- Pour l'imagerie multi-puits, installez la scène mécanique pour passer d'une position d'intérêt XY sélectionnée. Confirmez que les cellules NC crâniennes sont au point et les positions de scène sont correctes.
- Utilisez la commande Acquire pour démarrer l'imagerie time-lapse.
- Une fois l'imagerie en accéléré terminée, examinez les données multidimensionnelles et exportez les fichiers .stk pour analyse.
REMARQUE: .stk est un fichier de pile TIFF. - Sortez du logiciel, éteignez l'ordinateur et éteignez la scène, la caméra et les contrôleurs de microscope.
4. Analyse d'imagerie : quantification de la migration des cellules de crête neurale
REMARQUE : Pour mieux définir les comportements cellulaires exhibés par les cellules neurales neurales de cellules neurales murines migratrices, nous avons analysé une série de paramètres migratoires quantifiables, se concentrant spécifiquement sur la capacité migratoire et la dynamique de forme cellulaire. (1) La migration (distance accumulée) est la longueur totale du chemin empruntée par la cellule (m); (2) La migration (distance eucliden) est la distance en ligne droite entre la position initiale et la position finale de la cellule (m); (3) La migration (vitesse cellulaire) est la distance parcourue par cellule par unité de temps (m/min); (4) La forme cellulaire (zone cellulaire) est la surface totale couverte par la cellule. Définir l'échelle de pixel à l'échelle de micron selon le microscope d'imagerie. (A - Apx x Npx, où Une zonede pixels px et Npx - nombre de pixels. Nombre d'unités : m2; (5) La forme cellulaire (circularité cellulaire) est la déviation de la forme cellulaire par rapport à un cercle parfait qui est indiquée par une valeur de circularité de 1,0 (4 oA/P2)) où la zone A et le périmètre P.
-
Suivi des cellules individuelles
REMARQUE : Pour mesurer la migration des cellules NC, les coordonnées XY de cellules individuelles dans toutes les images de time-lapse sont générées. Cela permet une analyse ultérieure des mesures de la distance, de la vitesse et de la persistance de la migration cellulaire.- Ouvrez ImageJ et importez les données sous forme de fichiers TIFF stack.
- Cliquez sur Analyser Définir l'échelle pour calibrer les fichiers .stk en fonction des paramètres du microscope, en travaillant en pixel/m.
- Cliquez sur Plugins (fr) Suivi de l'année . Suivi manuel pour ouvrir le plugin manuel de suivi des cellules Image J. Pour commencer le suivi cellulaire, sélectionnez Ajouter la piste.
- Suivez les cellules à travers toutes les images de films time-lapse, en utilisant le noyau comme point de référence.
REMARQUE : 10 à 20 cellules doivent être suivies par explante, avec un total de 60 cellules suivies (n ' 3). Les cellules qui subissent une division cellulaire au cours du time-lapse devraient être exclues de l'analyse. - Enregistrer et exporter les résultats comme un fichier .csv. Les résultats représentent le nombre de pistes de cellules individuelles, le nombre de tranches et les coordonnées XY sur toutes les images.
-
Quantification de la capacité migratoire des cellules de crête neurale
- Ouvrez les données de suivi des cellules individuelles (voir ci-dessus). Convertissez les fichiers .csv en format de fichier .txt.
- Ouvrez le logiciel de migration (la Table des Matériaux). Cliquez sur l'onglet Données d'importation pour importer les données de suivi cellulaire comme fichier .txt.
- Sous datasets (fr) Initialisation, sélectionnez le nombre de tranches ou d'images à analyser et définir l'étalonnage XY et l'intervalle de temps entre les images. Sélectionnez Appliquer les paramètres pour enregistrer les paramètres.
- Sélectionnez le symbole De données de parcelle pour former des parcelles de trajectoire. Sélectionnez le symbole Statistiques pour quantifier les mesures de distance et de vitesse.
- Enregistrer les parcelles de trajectoire que bitmap (.bmp) fichiers, et la distance et les mesures de vitesse que les fichiers .txt. Sélectionnez le symbole Supprimer les données. Répétez l'opération pour d'autres fichiers time-lapse.
REMARQUE : Les parcelles de trajectoire peuvent être utilisées pour visualiser la franchise des chemins cellulaires individuels pour une condition cellulaire ou un état donné au cours de films en accéléré (figure 4A). Les données de distance et de vitesse stockées dans les fichiers .txt peuvent ensuite être utilisées pour une analyse plus approfondie.
-
Quantification de la surface cellulaire de crête neurale et de la circularité
- Ouvrez les fichiers time-lapse .stk dans ImageJ et étalonnez en fonction des réglages du microscope, en travaillant en pixel/m.
- Sous Analyze (en anglais) Définir Les mesures, cliquez pour sélectionner les paramètres de forme cellulaire : zone cellulaire, périmètre et descripteur de forme.
- Utilisez l'outil De sélection à main levée pour dessiner manuellement autour de chaque cellule, en utilisant les limites de la membrane cellulaire comme guide.
- Appuyez sur les touches Ctrl et B sur le clavier pour maintenir le contour de la cellule sur l'image. Répétez l'opération pour les cellules sur chaque cadre time-lapse.
- Utilisez l'Image La relaye (fr) Au gestionnaire de retour sur investissement pour stocker les valeurs.
- Une fois que toutes les cellules d'intérêt par image ont été décrites, cliquez sur Mesurer. Enregistrer les résultats comme un fichier .csv.
REMARQUE : 10 à 20 cellules par film doivent être décrites, avec un total de 30 à 60 cellules analysées par condition (n - 3). Les données sur la forme cellulaire (fichiers.csv) peuvent être utilisées pour quantifier l'évolution de la dynamique de la forme cellulaire au fil du temps(figure 4C)ou la façon dont la morphologie peut être modifiée dans le cadre de différents traitements cellulaires.
Résultats
En utilisant la procédure démontrée ici, les embryons de souris ont été disséqués de l'utérus, et les tissus extraembryonnaires ont été enlevés (Figure 1A-D). Les embryons ont été mis en scène somite (en utilisant uniquement des embryons à 5 à 8 somites (ss), Figure 1E,F). La plaque neurale crânienne a alors été disséquée et le neuroepithelium a été isolé. Les cellules mésodermales, identifiées comme des cellules lâches, circulaires et mésenchymales, ont été légèrement brossées(figure 1G-L). La plaque neurale antérieure peut être explantée entière, auquel cas le tissu de crête neurale émergera latéralement et se dilatera radialement autour de l'explantation, ou chaque bordure neurale de plaque (droite et gauche) peut être explantée séparément. Ceci est particulièrement utile lors de l'explantation de mutants génétiques.
Dans un délai de 24 h, une région de crête neuronale neurocrânienne prémigratoire (épithéliale) peut clairement être vue autour de l'explantation de la plaque neurale (Figure 2B). En outre, une sous-population de cellules de crête neurale ont subi une transition épithéliale à mésenchymale et semblent entièrement mésenchymales (Figure 2). Ainsi, nous avons plusieurs anneaux concentriques de cellules distinctes, avec la plaque neurale (NP) au centre, la crête neurale pré-migratoire (pNC) dans le cercle intermédiaire, et une population de crête neurale migratrice (mNC) dans l'anneau extérieur (Figure 2B). Afin de retracer les cellules NC, il est possible d'utiliser des modèles de souris génétiquement modifiés comme nous le montrons dans la figure 2C. Dans ce cas, nous avons utilisé la crête neuronale spécifique Wnt1::Cre; RosamTmG qui se traduit par des cellules NC étant étiquetés en vert. Chez ces souris, les cellules expriment la tomate membranaire (mT, en rouge) à moins qu'elles n'expriment Cre recombinase. La recombinaison conduit les cellules à exprimer des protéines fluorescentes vertes (GFP, en vert). Les globules rouges montrés au centre de l'explantation sont des cellules de plaque neurale. Quelques cellules neurales de plaque dorsal expriment également GFP ; pour la culture à long terme, nous excitirions toutes les cellules du centre. Pour nos besoins, la pureté de l'explantation est suffisante pour suivre les différentes populations de cellules de crête neurale. Lorsque la pureté plus élevée de la crête neurale est nécessaire, cette stratégie d'étiquetage génétique peut être combinée avec le tri des cellules activées fluorescentes (FACS) pour assurer la pureté de la population. Alternativement, il est possible de fixer les explants et d'identifier la population NC avec l'étiquetage des anticorps.
Il était également évident par 24 h que les anneaux concentriques caractéristiques des cellules NC prémigratoires et entièrement migratrices des cultures d'explantation n'étaient pas dépendants ni régis par le choix de matrice (figure 3). Les cultures d'explantation plaquées sur un hydrogel à base d'ECM et de fibronectin ont formé des structures d'explantation comparables, comprenant les trois populations cellulaires, NP, pNC et mNC(figure 3A,C). La morphologie des cellules à crête neuronale était également comparable entre celles qui étaient plaquées sur l'hydrogel à base d'ECM et la fibronectine(figure 3B,D). Cependant, les explants plaqués sur la fibronectine ont produit des cellules avec la lamellipodia plus proéminente au bord d'avant de cellules, apparemment plus polarisé dans la direction de la migration (figure 3B,D).
Une fois qu'une population de cellules de crête neurale migratrice est évidente, l'imagerie cellulaire vivante peut être accomplie. La microscopie time-lapse est fixée à 10x grossissement (18 h, 1 cadre/5 min) pour l'analyse ultérieure de la migration des cellules NC (figure 4A). ImageJ Manuel Tracking plug-in génère des coordonnées XY de cellules individuelles sur toutes les images des films time-lapse (Figure 4B). Ces coordonnées peuvent être traitées à l'aide du logiciel de migration. Ce logiciel permet la visualisation des pistes cellulaires individuelles au fil du temps (Figure 4B) et peut être utilisé pour quantifier la distance accumulée et Euclidin, ainsi que la vitesse des cellules.
Les données d'imagerie en accéléré fournissent également une mine d'informations sur la morphologie cellulaire pendant la migration des cellules de crête neurale crânienne(figure 4C). En décrivant les membranes cellulaires individuelles, les mesures de la surface cellulaire et du périmètre peuvent être calculées à partir de toutes les images des films (Figure 4C). Ces mesures permettent de quantifier ultérieurement la surface cellulaire et la circularité (figure 4D). La figure 4C montre une analyse des changements de forme cellulaire sur 18 h. Notez qu'à mesure que les cellules migrent loin de l'explante, la surface cellulaire augmente considérablement tandis que la circularité cellulaire demeure relativement constante (ANOVA à sens unique, test de comparaisons multiples de Tukey) ( Figure 4E,F). Ceci suggère que pendant que les cellules s'écartent du bord épithélial et perdent des contacts de cellule-cellule, elles montrent la zone accrue de diffusion de cellules. Les mesures de circularité cellulaire n'ont pas beaucoup changé au fil du temps; cependant, des changements à court terme de la circularité peuvent être observés si un nombre accru de points de temps sont quantifiés. Les mesures de circularité cellulaire peuvent également fournir des données intéressantes sur la dynamique de la forme cellulaire en présence d'un signal chimiotaxique ou dans des conditions confinées.
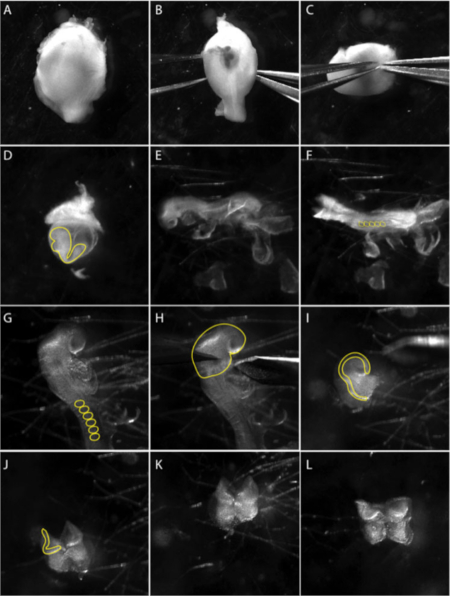
Figure 1 : Isolement des explantations de crête neurale crânienne d'un embryon e8.5.
Les images sont des images fixes d'une vidéo documentant la technique de micro-dissection. (A-C) Dissection de l'embryon de l'utérus. (B-C) À l'aide de deux forceps tranchants, démonter délicatement la couche musculaire. Le panneau (D) montre l'embryon à l'intérieur du sac de jaune viscéral (ligne jaune). Extraire l'embryon du sac de jaune viscéral. (E) Vues latérales de l'embryon au stade 8.5 latérale. (F) Vue dorsale de l'embryon au stade 8.5. Compter les somites (ss) pour déterminer l'âge des embryons; généralement 5 à 8 ss (cercles jaunes en F). (G) Regardez de près la région crânienne de l'embryon. Enlever les membranes extraembryonnaires de la région crânienne; somites sont marqués d'une ligne jaune. (H) Les dissections de la plaque neurale antérieure sont effectuées sous la première arche branchiale (ligne jaune). (I) Vue latérale de la dissection antérieure de plaque neurale. Les plis neuronaux, où les cellules de crête neurale apparaissent, sont marqués d'une ligne jaune. (J-L) Enlever le tissu mésodermal (cellules mésenchymales moelleuses) sous-jacents plis neuronaux antérieurs autant que possible avant de placage NP sur les plats de culture préparés. Le film a été pris à l'aide d'un stéréo-microscope avec une lentille apochromatique à large champ au zoom 3.0X (voir le tableau des matériaux). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
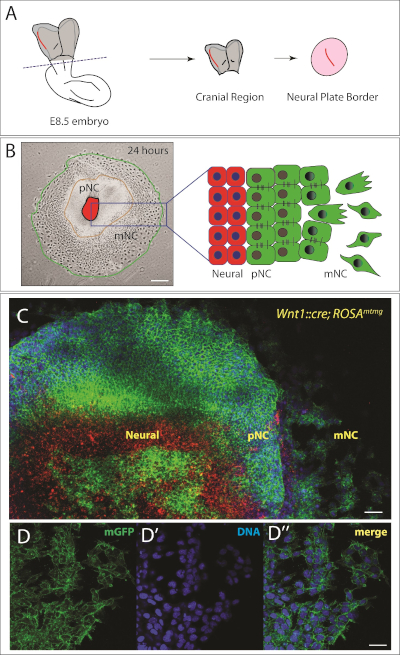
Figure 2 : Explantation de crête neurale crânienne de Murine.
(A) Représentation schématique de la vue dorsale d'un embryon de souris e8.5. La région crânienne de l'embryon est coupée à la ligne pointillée. La bordure de plaque neurale (surlignée en rouge) est isolée du tissu environnant de mesoderm et cultivée pendant 24 h pour permettre à la crête neurale crânienne d'émigrer. Schéma adapté de22,23. (B) Gauche : Image de champ lumineuse représentative d'une crête neurale crânienne explante 24 heures après le placage. Trois populations de cellules sont observées, qui sont également schématisées sur la droite. NP - plaque neurale, pNC - crête neurale pré-migratoire et mNC - crête neurale migratrice. Barre d'échelle de 250 m. (C) Images de grossissement plus élevées d'une explante d'une souris génétiquement étiquetée (Wnt1::cre; RosamTmG). Cellules sans le conducteur Cre express tomate membranaire (mT) en rouge. L'expression de Cre sous le contrôle d'un promoteur Wnt1 spécifique de crête neurale conduit à l'excision de la cassette mT et à l'expression de la membrane GFP (mG) en vert. Les noyaux sont tachés de Hoescht (en bleu). Barre d'échelle de 200 m. (D'D'' Images de grossissement plus élevées de cellules migratrices exprimant la membrane GFP (D). (D') L'ADN est étiqueté avec Hoescht (bleu). (D') Fusion de D et D'. Barre d'échelle de 20 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
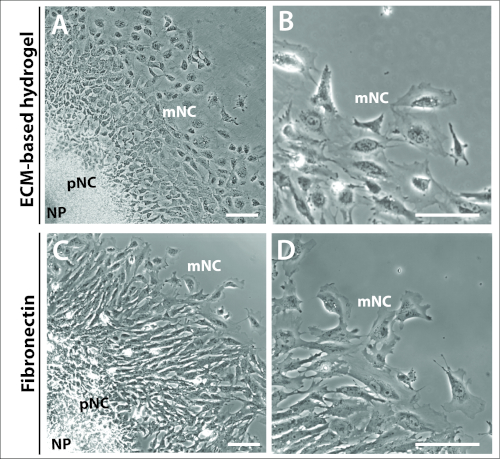
Figure 3 : Explantations cultivées sur différents substrats.
(A-B) Images de contraste de phase des explants cultivés sur eCM-hydrogel commercial. (C-D) Explantations cultivées sur 1 fibronectine de 1 g/mL. Les cellules de crête neurale (NP), prémigratoires (pNC) et migratoires (mNC) peuvent être distinguées par leurs différentes morphologies cellulaires. Barre d'échelle de 100 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
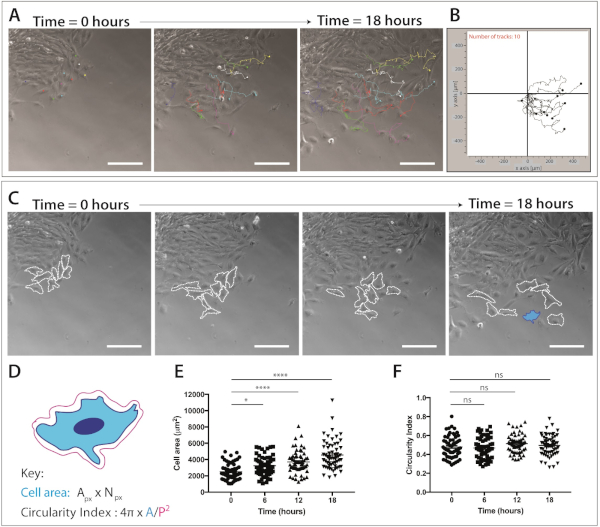
Figure 4 : Quantification de la migration des cellules de crête neurale crânienne et de la dynamiquede la forme cellulaire.
(A) Cadres de contraste de phase de l'imagerie en time-lapse des cultures d'explantation superposées avec des traces de cellules à crête neurale unique, à l'aide du plug-in de suivi manuel des cellules ImageJ/Fidji. Dix cellules mNC représentatives ont été suivies manuellement sur 18 h (217 images) et le XY coordonnées ont été exportées. Les données sont représentées comme un point de retouche et des parcelles de ligne. Les cellules ont été plaquées sur la fibronectine de 1 g/mL. Barre d'échelle de 200 m. (B) Parcelle de trajectoire représentative de 10 cellules mNC, générée à l'aide d'un logiciel de migration. (C) Cadres de contraste de phase tirés de l'analyse en time-lapse des cultures d'explantation. Les lignes en pointillés décrivent 8 cellules de crête neurale migratoire s'analysées pour la dynamique de forme cellulaire lorsqu'elles sont plaquées sur une fibronectine de 1 g/mL. Barre d'échelle de 200 m. (D) Représentation schématique des calculs utilisés pour quantifier la surface cellulaire et la circularité. La morphologie cellulaire du schéma est celle de la cellule mise en évidence en bleu (C). Une zonede pixels px, Npx, numéro de pixel, zone A, périmètre P (1 pixel 1,60772 m2). (E-F) Quantification de la surface cellulaire et des mesures de circularité cellulaire au fil du temps. Les données représentent la moyenne et le SEM. Chaque point représente une cellule (n - 60), tirée de 3 expériences indépendantes, et analysé à 0 h, 6 h, 12 h et 18 h (ns non-significatif, plt;0,05, p 'lt; 0,0001 à sens unique ANOVA, test de comparaisons multiples de Tukey). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
L'étude des cellules de crête neurale de mammifères a été un défi pour des scientifiques en raison de la nature in utero du développement de mammifère. Les études in vivo sont difficiles à mettre en place, car l'embryon doit être manipulé dans des conditions qui imitent la vie dans l'utérus. Dans la pratique, il est presque impossible de reproductiblement la culture de ces embryons (E8) pendant plus de 24 h, en particulier pour l'imagerie en direct. En outre, l'induction et la migration neurales de crête se produisent en même temps que la fermeture de tube neural et le tournant embryonnaire dans la souris ; il s'agit d'un événement morphogénétique crucial et stressant, qui échoue fréquemment lorsque les embryons sont cultivés ex utero. Ainsi, le taux de réussite des approches ex utero est généralement faible. L'utilisation de cellules NC immortalisées21 est un outil utile pour réduire l'utilisation des animaux et il peut fournir une meilleure source de cellules de crête neurale pour l'analyse à long terme, la transfection et les études d'enrichissement. Cependant, il est clairement nécessaire de culture fiable cellules de crête neurale primaire. Notre méthode s'applique aux modèles génétiques knock-out ou conditionnels de souris. Une méthode comparable à la nôtre a été décrite pour d'autres populations de crête sneuronal20; cependant, notre méthode décrit complètement l'isolement étape par étape des cellules ncinales murines de NC. Nous décrivons également en détail l'utilisation de différentes matrices ainsi que la procédure d'analyse de la migration.
Pour obtenir des résultats cohérents, nous avons constaté qu'une attention particulière était accordée à la mise en scène lors de la sélection des embryons. Comme on pouvait s'y attendre, le nombre de somites est en corrélation avec différentes étapes du développement crânien du NC. Par conséquent, la connaissance de l'anatomie embryonnaire est très importante avant d'acquérir des données expérimentales. Cette approche peut ensuite être adaptée à l'isolation des populations distinctes de cellules de crête neurale, selon la question biologique et les cellules cibles.
Une fois les embryons sélectionnés et disséqués, les cellules mésodermales peuvent être facilement distinguées et doivent être enlevées pour permettre une meilleure visualisation et réduire la contamination. Pour les cultures à plus long terme, le tissu de plaque neurale peut être enlevé à 24 h de placage afin d'empêcher la contamination par les tissus neuronaux. Un autre raffinement pourrait être l'utilisation de l'étiquetage fluorescent de lignée (par exemple, en utilisant un Wnt1::cre ou Sox10::creERT pilotes combinés avec des journalistes fluorescents24,25) pour distinguer les cellules de crête neurale de d'autres tissus comme le montre la figure 2C.
Des rapports précédents ont mis en évidence le potentiel de placage de souris NC explanter des cultures sur différentes matrices, le plus souvent sur les hydrogels commerciaux ECM, la fibronectine et le collagène I20,21,26. Dans nos mains, les cultures d'explantations NC crâniennes de souris sont cultivées avec succès sur les trois matrices, à des concentrations spécifiées dans les rapports originaux (données non montrées). L'approche raffinée initiale que nous avons adaptée pour nos cultures d'explantation s'est utilisée par un hydrogel commercial comme matrice de choix, qui se composait principalement de laminininet et de collagènes.21(Figure 3A-B). Cependant, la composition de cet hydrogel n'est pas clairement définie, avec un facteur de croissance inconnu et la teneur en protéines. En tant que tel, nous avons depuis changé notre approche à placage souris NC explanter les cultures sur la fibronectine (Figure 3C-D (en)). La fibronectine est bien définie et fortement exprimée dans les membranes ECM et sous-sol le long desquelles les cellules NC migrentin vivo28,29,30. Afin d'optimiser une matrice de fibronectine qui reproduit le mieux la migration et la morphologie des cellules de crête neurale telles qu'on l'a vu à l'aide de l'hydrogel, nous avons comparé les comportements cellulaires NC exposés sur l'hydrogel à une titration de la fibronectine de 0,25 à 30 g/mL, et définissions la fibronectine de 1 g/mL comme fournir des propriétés idéales (données non affichées). Nous croyons que ces travaux préliminaires peuvent aider à établir un cadre pour la comparaison systématique des matrices, telles que la fibronectine, avec ceux précédemment décrits, à savoir le collagène et la lamininine32,33,34. Il serait particulièrement intéressant de comparer la capacité migratoire des cellules NC de souris sur la fibronectine par rapport au collagène I, étant donné que le collagène-IA1 est endogènement sécrété par les cellules NC de souris, d'oiseaux et d'humains28,30,31,32. Le collagène I est donc aussi pertinent que la fibronectine dans l'examen du choix de matrice. Il est également intéressant de reconnaître que la biodisponibilité des facteurs de croissance dans les médias peut être modifiée par différents composants de matrice, en particulier compte tenu de la teneur élevée en sérum de nos médias. Pour surmonter cela, nous travaillons actuellement à produire des conditions de culture définies sans sérum. Ces médias définis sont utilisés avec succès dans les protocoles d'induction de crête neurale dans le champ pluripotent de cellules souches, mais exigent davantage d'optimisation pour notre système de culture d'explantation de NC33,34. Nos travaux peuvent également servir de point de départ pour le raffinage des conditions pour d'autres types de cellules NC telles que le cancer et le tronc NC, et pour les études ultérieures de la différenciation NC. Plus important encore, ce protocole permet l'isolement des cellules NC crâniennes pour une variété d'applications. Nous envisageons des études sur la migration dirigée, la migration 3D et l'invasion. Les cellules isolées de cette manière peuvent être traitéesin vitropour un certain nombre d'analyses. Par exemple, les cellules peuvent facilement être traitées à l'aide de différentes petites molécules pour cibler des protéines spécifiques, elles peuvent être traitées à des moments définis, et des expériences de lavage peuvent être conçues pour déterminer la récupération des comportements cellulaires (Figure 4). La culture à plus long terme pour les tests de transfection et de différenciation est possible, ainsi que le passage des cellules (données non montrées). Cependant, la viabilité, la capacité de renouvellement cellulaire et la multipotence devraient être validées après le passage. Les cellules plaquées sur des plaques de verre peuvent également être utilisées dans les protocoles de coloration immunofluorescente, après imagerie en direct. Enfin, cette approche représente un système extrêmement puissant pour étudier la migration du NC à partir de modèles de souris génétiques22,23,24,25.
Déclarations de divulgation
Les auteurs n'ont aucun conflit d'intérêts.
Remerciements
Nous sommes reconnaissants à l'unité des services biologiques du King's College de Londres, en particulier tiffany Jarvis et Lynsey Cashnella pour leur soutien continu. Nous remercions Derek Stemple, Mamoru Ishii et Robert Maxson pour leurs conseils et leur aide auprès des réactifs lors de l'établissement initial de ce protocole. Nous remercions Dheraj Taheem pour son aide en ce qui concerne l'irradiation gamma des cellules STO. Nous remercions les laboratoires Liu et Krause, en particulier Tommy Pallett, pour leur soutien. Ces travaux ont été financés par des subventions du BBSRC (BB/R015953/1 à KJL/MK), d'une formation du Programme de formation doctorale du MRC (LD), du Newland Pedley Fund (ALM) et du KRIPIS II, Secrétariat général de la recherche et de la technologie (GSRT), ministère de l'Éducation et de la Religion Grèce et la Fondation Santé (à la SGGM).
matériels
| Name | Company | Catalog Number | Comments |
| bFGF | R&D systems | 233-FB | |
| b-mercaptoethanol | Gibco | 31350-010 | |
| Tissue culture flasks | Corning | 430639 | 25cm2 culture flasks |
| DMEM (4500 mg/L glucose) | Sigma | D5671 | |
| Dulbecco's phosphate-buffered saline (dPBS) | Sigma | D8537 | |
| Ethanol | Fisher Chemicals | E/0650DF/C17 | |
| Fetal Bovine Serum | Sigma | TFS AA 10-155 | |
| Fetal Bovine Serum (ES Cell FBS) | Gibco | 26140079/16141-079 | |
| Fibronectin bovine plasma | Sigma | F1141 | |
| Gelatin from bovine skin | Sigma | G9382 | |
| L-glutamine | Sigma | G7513 | |
| Glass-bottomed, multi-well 24-well tissue culture plate | Ibidi | 82406 | Glass-bottomed, No 1.5, 24-well tissue culture dish with black sides |
| Cell migration analysis tool | Ibidi | v2.0 | Manuals for this software can be found at: https://ibidi.com/manual-image-analysis/171-chemotaxis-and-migration-tool.html. |
| Circularity Plug-in | ImageJ | v1.29 or later | The circularity plug-in is an extended version of ImageJ/Fiji’s Measure command, designed by Rasband, W., (2000) (wsr@nih.gov). |
| LIF | ESGRO by Millipore | ESG1106 | |
| Manual Cell Tracking Plug-in | ImageJ | v1.34k or later | ref Cordelieres F. Institut Curie, Orsay (France) 2005 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| MEM non-essential aminoacids 100X | Gibco | 11140-050 | |
| Matrigel (ECM-based Hydrogel) | Corning | 356234 | |
| Microscope | Olympus | 1X81 | Olympus 1X81 inverted microscope |
| Microscope camera | Photometrics | 512B | Photometrics Cascadell 512B camera (4x UPlanFL N, 10x UPlanFL N, 20x UPlanFL N, 40x UPlanFL N objectives) |
| Microscope controller | Olympus | 1X2-UCB | Olympus 1X2-UCB microscope controller |
| Microscope temperature controller | Solent Scientific | RS232 | Maintained at 37°C |
| Penicillin/streptomycin | Sigma | A5955 | |
| Sodium Pyruvate | Sigma | S8636 | |
| Statistics software | Graphpad Prism | v7.0 | |
| STO feeder cells | ATCC | CRL-1503 | |
| Stereomicroscope | Nikon | SMZ | |
| XY microscope stage controller | Applied Scientific Instrumentation (ASI) | MS-4400 |
Références
- Duband, J. L. Diversity in the molecular and cellular strategies of epithelium-to-mesenchyme transitions: Insights from the neural crest. Cell Adhesion & Migration. 4, 458-482 (2010).
- Mayor, R., Carmona-Fontaine, C. Keeping in touch with contact inhibition of locomotion. Trends in Cell Biology. 20, 319-328 (2010).
- Le Douarin, N., Kalcheim, C. . The neural crest. 2nd edn. , (1999).
- Trainor, P. A. . Neural Crest Cells : Evolution, Development, and Disease. , (2014).
- Jiang, R., Bush, J. O., Lidral, A. C. Development of the upper lip: morphogenetic and molecular mechanisms. Developmental dynamics : an official publication of the American Association of Anatomists. 235, 1152-1166 (2006).
- Ahola, J. A., et al. Increased incidence of Hirschsprung's disease in patients with hypoplastic left heart syndrome--a common neural crest-derived etiology?. Journal of Pediatric Surgery. 44, 1396-1400 (2009).
- Bajpai, R., et al. CHD7 cooperates with PBAF to control multipotent neural crest formation. Nature. 463, 958-962 (2010).
- Inoue, K., et al. Congenital hypomyelinating neuropathy, central dysmyelination, and Waardenburg-Hirschsprung disease: phenotypes linked by SOX10 mutation. Annals of Neurology. 52, 836-842 (2002).
- Kim, H., Kang, K., Ekram, M. B., Roh, T. Y., Kim, J. Aebp2 as an epigenetic regulator for neural crest cells. PloS one. 6, e25174 (2011).
- Barriga, E. H., Trainor, P. A., Bronner, M., Mayor, R. Animal models for studying neural crest development: is the mouse different?. Development. 142, 1555-1560 (2015).
- Bronner, M. E., LeDouarin, N. M. Development and evolution of the neural crest: an overview. Developmental Biology. 366, 2-9 (2012).
- De Calisto, J., Araya, C., Marchant, L., Riaz, C. F., Mayor, R. Essential role of non-canonical Wnt signalling in neural crest migration. Development. 132, 2587-2597 (2005).
- Carmona-Fontaine, C., et al. Contact inhibition of locomotion in vivo controls neural crest directional migration. Nature. 456, 957-961 (2008).
- Matthews, H. K., et al. Directional migration of neural crest cells in vivo is regulated by Syndecan-4/Rac1 and non-canonical Wnt signaling/RhoA. Development. 135, 1771-1780 (2008).
- Banerjee, S., et al. A novel role for MuSK and non-canonical Wnt signaling during segmental neural crest cell migration. Development. 138, 3287-3296 (2011).
- Pryor, S. E., et al. Vangl-dependent planar cell polarity signalling is not required for neural crest migration in mammals. Development. 141, 3153-3158 (2014).
- Serbedzija, G. N., Bronner-Fraser, M., Fraser, S. E. Vital dye analysis of cranial neural crest cell migration in the mouse embryo. Development. 116, 297-307 (1992).
- Kawakami, M., Umeda, M., Nakagata, N., Takeo, T., Yamamura, K. Novel migrating mouse neural crest cell assay system utilizing P0-Cre/EGFP fluorescent time-lapse imaging. BMC Developmental Biology. 11, 68 (2011).
- Stemple, D. L., Anderson, D. J. Isolation of a stem cell for neurons and glia from the mammalian neural crest. Cell. 71, 973-985 (1992).
- Etchevers, H. Primary culture of chick, mouse or human neural crest cells. Nature protocols. 6, 1568-1577 (2011).
- Ishii, M., et al. A stable cranial neural crest cell line from mouse. Stem Cells and Development. 21, 3069-3080 (2012).
- Gonzalez Malagon, S. G., Liu, K. J. ALK and GSK3: Shared Features of Neuroblastoma and Neural Crest Cells. Journal of Experimental Neuroscience. 12, 1179069518792499 (2018).
- Gonzalez Malagon, S. G., et al. Glycogen synthase kinase 3 controls migration of the neural crest lineage in mouse and Xenopus. Nature Communications. 9, 1126 (2018).
- Danielian, P. S., Muccino, D., Rowitch, D. H., Michael, S. K., McMahon, A. P. Modification of gene activity in mouse embryos in utero by a tamoxifen-inducible form of Cre recombinase. Current Biology : CB. 8, 1323-1326 (1998).
- Laranjeira, C., et al. Glial cells in the mouse enteric nervous system can undergo neurogenesis in response to injury. Journal of Clinical Investigation. 121, 3412-3424 (2011).
- Pfaltzgraff, E. R., Mundell, N. A., Labosky, P. A. Isolation and culture of neural crest cells from embryonic murine neural tube. Journal Of Visualized Experiments : JoVE. , e4134 (2012).
- Sternberg, J., Kimber, S. J. The relationship between emerging neural crest cells and basement membranes in the trunk of the mouse embryo: a TEM and immunocytochemical study. Journal of Embryology and Experimental Morphology. 98, 251-268 (1986).
- Sternberg, J., Kimber, S. J. Distribution of fibronectin, laminin and entactin in the environment of migrating neural crest cells in early mouse embryos. Journal of Embryology and Experimental Morphology. 91, 267-282 (1986).
- George, E. L., Georges-Labouesse, E. N., Patel-King, R. S., Rayburn, H., Hynes, R. O. Defects in mesoderm, neural tube and vascular development in mouse embryos lacking fibronectin. Development. 119, 1079-1091 (1993).
- Henderson, D. J., Copp, A. J. Role of the extracellular matrix in neural crest cell migration. Journal of Anatomy. 191 (Pt 4), 507-515 (1997).
- Thomas, S., et al. Human neural crest cells display molecular and phenotypic hallmarks of stem cells. Human Molecular Genetics. 17 (21), 3411-3425 (2008).
- Greenberg, J. H., Seppa, S., Seppa, H., Hewitt, T. Role of collagen and fibronectin in neural crest cell adhesion and migration. Developmental Biology. 87 (2), 259-266 (1981).
- Leung, A. W., et al. WNT/β-catenin signaling mediates human neural crest induction via a pre-neural border intermediate. Development. 143 (3), 398-410 (2016).
- Zhu, Q., Lu, Q., Gao, R., Cao, T. Prospect of Human Pluripotent Stem Cell-Derived Neural Crest Stem Cells in Clinical Application. Stem Cells International. 2016, 7695836 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon