Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Tissu conjonctif humain dérivé de fibroblastes pour les applications de dépistage
Dans cet article
Résumé
Présenté ici est un protocole pour générer des tissus conjonctifs modifiés pour une culture parallèle de 48 tissus dans une plaque multi-puits avec des doubles pôles, adapté aux études mécanistes, à la modélisation des maladies et aux applications de dépistage. Le protocole est compatible avec les fibroblastes de différents organes et espèces et est illustré ici avec les fibroblastes cardiaques primaires humains.
Résumé
Les fibroblastes sont des cellules phénotypiquement très dynamiques, qui se transdifférencient rapidement en myofibroblastes en réponse à des stimuli biochimiques et biomécaniques. La compréhension actuelle des processus fibrotiques, y compris la fibrose cardiaque, reste médiocre, ce qui entrave le développement de nouvelles thérapies anti-fibrotiques. Des systèmes modèles humains contrôlables et fiables sont essentiels pour une meilleure compréhension de la pathologie de la fibrose. Il s’agit d’un protocole hautement reproductible et évolutif pour générer des tissus conjonctifs (ECT) dans une plaque de coulée de 48 puits afin de faciliter l’étude des fibroblastes et la physiopathologie du tissu fibrotique dans un environnement en 3 dimensions (3D). Les ECT sont générés autour des pôles avec une rigidité accordable, ce qui permet des études sous une charge biomécanique définie. Dans les conditions de charge définies, les adaptations phénotypiques contrôlées par les interactions cellule-matrice peuvent être étudiées. Les tests parallèles sont réalisables dans le format 48 puits avec la possibilité d’analyser le temps de plusieurs paramètres, tels que le compactage et la contraction des tissus par rapport à la charge. À partir de ces paramètres, des propriétés biomécaniques telles que la rigidité et l’élasticité des tissus peuvent être étudiées.
Introduction
Un obstacle majeur dans l’étude des maladies fibrotiques est le manque de modèles de tissus 3D humains représentatifs qui fournissent un aperçu du comportement des fibroblastes et de leurs dérivés pathologiques. Pour étudier les processus fibrotiques, les systèmes de culture 2D standard sont sous-optimaux puisque les fibroblastes isolés se transdifférencient rapidement en myofibroblastes exprimant l’actine musculaire α lisse (SMA) lorsqu’ils sont cultivés sur des substrats 2D non conformes1,2,3. Ainsi, les fibroblastes de la culture 2D standard ne reflètent pas un phénotype tissulaire « sain » régulier3,4,5,6. Des cultures sur substrats souples ont été introduites pour simuler des environnements tissulaires non fibrotiques (10 kPa) et fibrotiques (35 kPa)7, mais ceux-ci n’ont pas la troisième dimension, ce qui est très important en ce qui concerne la physiopathologie. L’ingénierie tissulaire offre la possibilité de surmonter cette limitation en permettant la culture de fibroblastes dans un contexte défini et expérimentalement accordable de matrice extracellulaire (ECM), par exemple, par des altérations de la cellularité, de la composition ecm et de la concentration ECM, qui peuvent toutes déterminer la biomécanique tissulaire.
Divers modèles 3D ont été générés à l’aide de fibroblastes. Les disques flottants et les microsphères ont été parmi les premiers et démontrent que le collagène est remodelé et compacté en fonction du temps. Les fibroblastes exercent des forces de traction sur les fibrilles de collagène, un processus qui peut être facilité par l’ajout d’agents pro-fibrotiques tels que le facteur de croissance transformant bêta 1 (TGF-β1)8,9,10,11,12,13,14,15,16. Cependant, les cultures flottant librement ne permettent pas la charge externe contrôlée et constituent donc des modèles de rétrécissement ou de compactage continus. Les tissus modifiés en forme de feuille ont ouvert la possibilité d’étudier la régulation homéostatique des propriétés biomécaniques des tissus, notamment par des tests de déformation uni, bi, multiaxiale ou cyclique.17,18,19,20. Ces modèles ont été utilisés, par exemple, pour démontrer l’influence du nombre de cellules sur la rigidité tissulaire, qui s’est avérée être en corrélation positive avec l’intégrité du cytosquelette et la contractilité du cytosquelette d’actomyosine.18,19. Cependant, il est important de noter que les conversions force-déformation sont compliquées par la déformation tissulaire non uniforme autour des points de serrage des transducteurs de force et des points d’ancrage. Cette limitation inhérente peut être contournée par des tissus en forme d’os de chien ou d’anneau, offrant une certaine application tissulaire aux points d’ancrage.21,22,23. Les tissus en forme d’anneau peuvent être préparés en distribuant un hydrogel de collagène cellulaire dans des moules en forme d’anneau. Au fur et à mesure que l’hydrogel se compacte, un tissu se forme autour de la tige interne incompressible du moule, ce qui offre une résistance à la contraction ultérieure des tissus.24,25,26,27. Après un compactage initial et généralement maximal, les tissus peuvent également être transférés à des entretoises réglables pour restreindre davantage l’ECT circulaire à une longueur de tissu définie.3,24,25,26,27,28,29,30. Les propriétés biophysiques peuvent être évaluées dans des dispositifs de contrainte de déformation horizontaux ou verticaux standard avec des capteurs de pesage appropriés sous déformation unidirectionnelle ou dynamique3. Comme les tissus ont une structure circulaire largement uniforme et peuvent être maintenus sur des barres / crochets (points d’ancrage et / ou transducteurs de force), bien que ceux-ci puissent encore entourer des zones de compression autour des barres de chargement, ce format permet une variation de déformation plus uniforme par rapport au serrage3. De plus, les tissus ancrés provoquent une forme de cellule bipolaire, et les cellules s’adaptent aux forces tissulaires en s’allongeant le long des lignes de force favorisant la traction anisotrope31,32,33,34,35,36. Nous avons déjà appliqué de l’ECT en forme d’anneau à partir de fibroblastes cardiaques (FK) de rats et d’humains autour d’un seul pôle rigide dans des expériences de contrainte fonctionnelle et effectué des études de gain et de perte de fonction en utilisant des fibroblastes transductés viralement24,25,26 et études pharmacologiques37. De plus, nous avons pu identifier les différences entre les sexes dans la fibrose médiée par la mucoviscidose dans le modèle ECT27.
Le protocole suivant pour la génération d’ECT humain, illustré par la mucoviscidose humaine primaire obtenue sous forme de mucoviscidose cryoconservée auprès de fournisseurs commerciaux (voir Tableau des matériaux), combine les avantages des tissus en forme d’anneau avec un moyen simple et rapide de produire des tissus macroscopiques pour une plate-forme de 48 puits conçue pour des tests parallèles à haute teneur.
Il est important de noter que le modèle ECT ne se limite pas à un type spécifique de fibroblastes, avec l’utilisation documentée dans l’étude d’autres fibroblastes, par exemple les fibroblastes de la peau38,39. De plus, les fibroblastes issus des biopsies des patients fonctionnent tout aussi bien, et le choix des fibroblastes dépend en fin de compte de la question scientifique à aborder.
La plate-forme utilisée pour la génération d’ECT décrite dans ce protocole est une plaque de culture cellulaire/tissulaire 3D de 48 puits disponible dans le commerce (Figure 1A). Les méthodes de préparation, de culture et de surveillance de la formation et du fonctionnement de l’ECT sous une géométrie et une charge mécanique définies à l’aide de la plaque de 48 puits sont décrites. Les ECT formés sont maintenus par des poteaux flexibles intégrés et la charge mécanique peut être ajustée en fonction de l’objectif final en utilisant des poteaux de dureté différente (valeur Shore A 36-89), influençant leurs rigidités en flexion. Les poteaux avec un rivage Une valeur de 46 sont recommandés. Le protocole est, en outre, compatible avec un moule circulaire personnalisé décrit précédemment, où l’ECT est maintenu autour d’une seule tige rigide37. Les dimensions de ce moule sont données à la figure 1B.
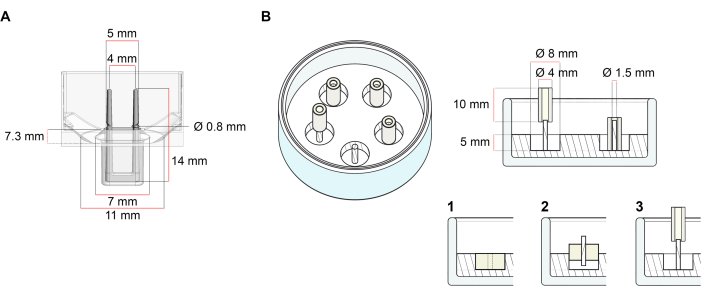
Figure 1 : Représentation schématique des moules de coulée. (A) Dessin technique et dimensions d’un moule de coulée avec deux pôles flexibles. Le moule comprend une circonférence intérieure délimitée par une paroi courte qui maintient des doubles pôles de retenue au corps principal du moule. Les pôles flexibles ont une distance horizontale libre les uns par rapport aux autres et sont connectés à la base. Le moule permet un volume de coulée de 180 μL. Le puits de chaque moule permet une capacité de volume d’au moins 600 μL de milieux de culture. Différentes compositions de matériaux peuvent être utilisées pour produire des pôles avec des rigidités spécifiques (par exemple, TM5MED-TM9MED). (B) Dessin technique et dimensions d’un moule en forme d’anneau avec une seule tige rigide. Il s’agit d’un moule alternatif avec une géométrie et un environnement mécanique distincts, qui peut être utilisé avec le protocole de coulée ECT37. La méthode d’assemblage du moule en forme d’anneau a été adaptée à partir de formats plus grands publiés28,41. En bref, la méthode comprend (1) l’impression d’entretoises de moulage en polytétrafluoroéthylène (PTFE) (8 mm de diamètre) dans du polydiméthylsiloxane (PDMS, silicone) versé dans des boîtes en verre (diamètre 60 mm), et (2) la fixation d’un support de poteau PDMS (1,5 mm de diamètre) concentriquement à l’intérieur de la cavité creuse formée, qui sert à (3) maintenir un pôle amovible (tube en silicone de 4 mm de diamètre). L’espace creux résultant permet 180 μL de volume de coulée. Chaque plat en verre peut contenir plusieurs moules imprimés (illustrés de manière exemplaire avec 5 moules) et a une capacité allant jusqu’à 5 mL de milieu de culture. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Toutes les étapes doivent être entreprises dans une hotte de biosécurité de classe II installée dans des laboratoires sous le niveau de confinement 1. Selon les réglementations locales et le type de manipulations à effectuer, telles que le transfert de gènes à médiation virale, le niveau de confinement doit être porté au niveau de biosécurité 2 ou 3. Toutes les cultures sont maintenues à 37 °C dans un incubateur de culture cellulaire avec une atmosphère humidifiée de 5 % de CO2 dans l’air. Notez que les volumes (étapes 1 et 2) sont fournis pour une fiole de culture cellulaire T75. Ajustez les volumes à différents formats de culture en fonction des recommandations de culture cellulaire standard.
1. Décongélation et pré-placage de fibroblastes cardiaques primaires (FK) pour culture monocouche (5-12 jours)
REMARQUE: Comme alternative, les cellules HFF-1 peuvent être utilisées en suivant le protocole de sous-culture standard tel que conseillé par le fournisseur.
- Préparez le milieu de croissance des fibroblastes (MGF) conformément aux instructions du fabricant. Éventuellement, ajoutez des antibiotiques tels que 100 U / mL de pénicilline et 100 mg / mL de streptomycine. Permettre le mélange complet de tous les composants avant utilisation. Conserver à 4 °C jusqu’à 14 jours.
- MGF chaudes à 20-25 °C.
- Décongeler la mucoviscidose cryoconservée (contenant idéalement 1 x 106 à 2 x 106/mL de cellules par cryovial) au bain-marie à 37 °C pendant environ 2 min, jusqu’à ce qu’il ne reste qu’une petite quantité de glace dans le flacon.
- À l’aide d’une pipette sérologique de 2 mL, transférer la suspension cellulaire goutte à goutte dans un tube de centrifugeuse stérile approprié contenant 10 mL de MGF. Pour une récupération optimale des cellules, rincez le cryovial avec 1 mL de MGF et transférez-le dans le tube de centrifugeuse. Comme les cellules sont très sensibles à ce stade, remettez en suspension à l’aide d’une pipette sérologique avec une pointe d’alésage pour minimiser les dommages cellulaires causés par la contrainte de cisaillement.
REMARQUE: Si le milieu de cryoconservation contient un pourcentage élevé de DMSO, assurez-vous qu’après la remise en suspension cellulaire dans les MGF, la teneur en DMSO est inférieure à 1 %. Alternativement, centrifugez les cellules remises en suspension à 300 x g pendant 5 min à 20-25 °C pour l’échange de milieu. Ensuite, aspirez soigneusement le surnageant, faites tourbillonner le tube pour déloger les cellules granulées et remettez-les ensaison dans le volume souhaité de MGF pour l’ensemencement. - Ensemencez 0,5 x 106 cellules dans 12 mL de MGF dans une fiole de culture cellulaire T75. Si d’autres logiciels de laboratoire sont utilisés, ajustez le nombre de cellules pour maintenir une densité d’ensemencement de 6,7 x 103/cm2.
- Remplacez les MGF tous les deux jours pendant 5 jours ou jusqu’à ce que les cellules atteignent 80 % de confluence.
REMARQUE: Le rendement cellulaire après l’expansion dépend principalement de la taille des cellules et du taux de prolifération, qui peuvent différer d’un donneur à l’autre. En règle générale, cette procédure de culture standard permet de récupérer 4 x 106 à 5 x 106 CF d’une fiole de culture cellulaire T75 après 5 jours de culture.
2. Dispersion enzymatique de la mucoviscidose humaine (10-20 min)
REMARQUE: Cette étape vise à établir une suspension unicellulaire de la mucoviscidose humaine pour la sous-culture de cellules monocouches et la préparation de l’ECT. Ce protocole a été optimisé pour les cultures monocouches de mucoviscidose humaine dans les passages 3 à 4. Pour une standardisation optimale, il est recommandé de sous-cultiver la mucoviscidose en monocouche, au moins une fois avant la préparation de l’ECT. Ce protocole doit être optimisé pour les fibroblastes provenant de différents donneurs et fournisseurs. D’autres protocoles de détachement peuvent impliquer le remplacement des réactifs de dissociation recombinants à base de sérine protéase par, par exemple, ceux contenant des enzymes protéolytiques et collagénolytiques.
- MGF chaudes, PBS (sans Ca2+/Mg2+) et réactif de dissociation cellulaire à 20-25 °C.
- Aspirer le milieu des cellules cultivées.
- Laver les cellules avec 6 mL de PBS et aspirer.
- Ajouter 6 mL du réactif de dissociation cellulaire aux cellules et incuber pendant 3 min à 20-25 °C jusqu’à ce que les cellules commencent à se détacher visiblement.
REMARQUE : Selon la source des FC, cela peut prendre plusieurs minutes de plus. Alternativement, si les cellules ne se détachent pas à température ambiante, incuber à 37 °C pour améliorer l’activité des enzymes. Pour assurer une viabilité cellulaire optimale, surveillez le détachement cellulaire au microscope. - Neutraliser l’activité enzymatique en ajoutant 6 à 12 mL de MGF aux cellules délogées dans le réactif de dissociation cellulaire. Pipettez doucement de haut en bas 4 à 8 fois à l’aide d’une pipette sérologique de 10 mL pour assurer une suspension à cellule unique et transférer les cellules dans un tube de collecte frais de 50 mL. Vérifiez le rendement à l’aide d’un microscope et d’un hémocytomètre ou d’un compteur de cellules automatisé selon les instructions du fabricant.
- Centrifuger la suspension de la cellule à 300 x g pendant 5 min à 20-25 °C.
REMARQUE: Afin d’atteindre un rendement de cellules suffisant pour la génération de la quantité souhaitée d’ECT, les cellules peuvent être sous-cultivées dans une dilution allant jusqu’à 1: 6 pour une expansion ultérieure. Laissez les cellules se développer jusqu’à ce que 80 % de confluence soit atteint (environ 5 à 6 jours), le milieu changeant tous les deux jours. Répétez ensuite la dispersion enzymatique et passez à l’étape 2.7. pour poursuivre la préparation à l’ECT. - Aspirez le surnageant et faites glisser le tube pour déloger la pastille. Remettre en suspension les cellules dans les MGF à 20-25 °C pour obtenir une suspension cellulaire de ≥ 15 x 106/mL (environ 40 % de cellules de plus que ce qui est nécessaire pour l’étape 3.3.). Cela tient compte de la perte de cellules due à la tension à l’étape suivante.
- Filtrer la suspension cellulaire à travers une passoire cellulaire maillée de 40 μm.
ATTENTION: Les agglomérats cellulaires nuisent à la formation optimale de l’ECT. Lors de l’utilisation de la dispersion enzymatique du protocole de FK humaine pour la coulée directe de l’ECT, la contrainte de la suspension cellulaire garantit l’absence d’amas cellulaires majeurs qui interfèrent avec la formation de tissus homogènes. Les hétérogénéités compromettront des analyses contraintes-déformations fiables. - Recompter le nombre de cellules et évaluer la viabilité des cellules pour garantir un nombre de cellules fiable dans une suspension avec une viabilité de ≥80 % pour procéder à la préparation de l’ECT.
- Utilisez un compteur de cellules automatisé pour évaluer le nombre de cellules et la viabilité par exclusion du courant électrique.
- Alternativement, utilisez le test d’exclusion de colorant bleu trypan (cancérogène, catégorie de danger 2 - prendre des mesures de précaution), à l’aide d’un microscope et d’un hémocytomètre pour l’identification directe et le dénombrement des cellules vivantes (membranes cellulaires intactes qui excluent le colorant) et mortes (membranes cellulaires compromises qui permettent la liaison du colorant aux protéines intracellulaires).
- Réservez le tube de collecte avec une suspension cellulaire à 20-25 °C et passez immédiatement à l’étape 3.
3. Préparation au TCE (1 h)
REMARQUE : Un aperçu schématique de la génération d’ECT est décrit à la figure 2.
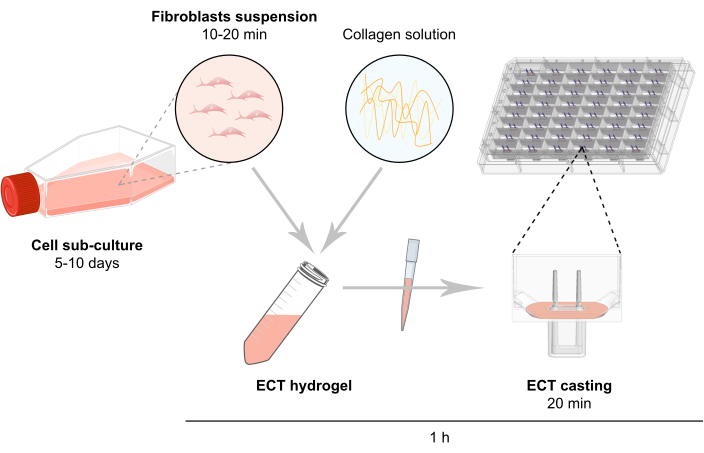
Figure 2 : Vue d’ensemble schématique de la génération d’ECT. Les fibroblastes sont développés en culture 2D avant d’être utilisés dans la génération d’ECT. Après 5 à 10 jours, les cellules sont dispersées enzymatiquement et la suspension cellulaire est reconstituée dans un mélange tamponné contenant du collagène bovin de type 1. Le mélange d’hydrogel cellule-collagène est pipeté dans des puits individuels dans une plaque de 48 puits pour la culture tissulaire 3D, conçue comme des moules de coulée avec deux pôles flexibles pour permettre la suspension ECT à une longueur et une charge définies. Les ECT sont généralement cultivés pendant 1 à 20 jours avant les mesures. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Préparer une solution mère de DMEM 10x en dissolvant la poudre de DMEM dans du ddH2O (134 mg/mL pour la formulation spécifiée dans le Tableau des matériaux) sous une rotation constante à 37 °C pendant 1 h. Stériliser par filtration. Le stock est stable jusqu’à 14 jours à 4 °C ou -20 °C jusqu’à 12 mois.
- Préparez fraîchement 2x DMEM en diluant une solution mère de 10x DMEM et en ajoutant 20 % (v/v) de FCS dans du ddH2O stérile. Éventuellement, utilisez des antibiotiques tels que 200 U/mL de pénicilline et 200 mg/mL de streptomycine. Consultez le tableau 1 pour connaître les volumes de pipetage. Le stock est stable jusqu’à 14 jours à 4 °C.
REMARQUE : Effectuez les étapes 3.1. et 3.2. avant de commencer la dispersion enzymatique des cellules (étape 2.) pour la préparation de l’ECT.
| Réactif | Concentration finale | Volume (mL) |
| 10× DMEM | n/a | 2 |
| FCS | 20 % (v/v) | 2 |
| Pénicilline | 200 U/mL | 0.2 |
| Streptomycine | 200 mg/mL | 0.2 |
| ddH2O | n/a | 5.6 |
| Total | n/a | 10 |
Tableau 1 : Composition de 2x DMEM.
ATTENTION : Tous les composants du mélange d’hydrogel cellule-collagène et des tubes centrifuges doivent être conservés sur de la glace avant l’utilisation. Cela aidera à empêcher l’auto-assemblage du collagène de se produire avant de distribuer le mélange d’hydrogel cellule-collagène dans les moules de coulée.
- Sur la base du tableau 2, ajuster la suspension cellulaire à une densité de 8,88 x 106 cellules/mL en ajoutant des MGF à 20-25 °C à la suspension cellulaire à partir de l’étape 2.10. Ensuite, déplacez le tube de collecte avec la suspension cellulaire sur la glace.
- Pour préparer le mélange d’hydrogel ECT, pré-refroidir un tube de centrifugeuse de 50 mL sur de la glace et y ajouter les différents composants énumérés dans le tableau 2 dans l’ordre suivant, en évitant la formation de bulles d’air.
REMARQUE: Le nombre maximal d’ECT à préparer dépend du nombre total de cellules déterminé à l’étape 2.9. Utiliser 0,3 mg de collagène par ECT, obtenu à partir d’une solution mère contenant 6-7 mg/mL. La concentration de la solution mère de collagène détermine le volume nécessaire pour obtenir une teneur optimale en collagène ECT. Les volumes des autres composants de l’hydrogel ECT doivent être adaptés en conséquence. Voir le tableau 2 pour les volumes ajustés selon une solution mère de collagène de 6,49 mg/mL. Les volumes décrits dans le tableau 2 sont utilisés dans ce protocole à titre indicatif.- Pipette l’hydrogel acide soluble-collagène de type 1 à l’aide d’une pipette sérologique avec une pointe d’alésage large.
- Ajustez la teneur en sel de la solution de collagène en ajoutant le 2x DMEM tout en mélangeant doucement en faisant tourbillonner le tube.
- Neutraliser le pH en ajoutant 0,2 M de NaOH tout en mélangeant doucement en faisant tourbillonner le tube. L’indicateur rouge phénol passera du jaune au rouge.
REMARQUE: Le volume de NaOH doit être titré pour chaque lot de collagène individuel pour la neutralisation optimale du pH. La neutralisation dépend de facteurs tels que le type de tampon et la préparation, ainsi que la concentration absolue de collagène, et elle affecte l’assemblage de la matrice de collagène et la viabilité cellulaire23,40. Une fois que la teneur en ionique est augmentée par l’ajout de DMEM et que le pH est neutralisé, l’auto-assemblage du collagène suit et ne doit pas être perturbé. Par conséquent, effectuez ce qui suit rapidement et sans pauses. - Ajouter la suspension de la cellule (à partir de l’étape 3.3) goutte à goutte tout en mélangeant doucement en faisant tourbillonner le tube.
| Numéro ECT: | 1 | 6 | 24 | 48 | |
| dont 10 % d’excédent | |||||
| Composants de l’hydrogel de collagène cellulaire: | (μL) | (μL) | (μL) | (μL) | |
| Stock de collagène (6,49 mg/mL) | 46.2 | 305.1 | 1220.2 | 2440.4 | |
| 2× DMEM | 46.2 | 305.1 | 1220.2 | 2440.4 | |
| 0,2 M NaOH | 3.1 | 20.5 | 81.8 | 163.7 | |
| Mélange cellulaire dans les MGF (8,88×106 cellules/mL) | 84.5 | 557.4 | 2229.7 | 4459.5 | |
| Volume total (μL) | 180.0 | 1188.0 | 4752.0 | 9504.0 | |
| Il s’agit d’un tableau exemplaire pour préparer un volume de coulée de 180 μL par ECT, contenant un total de 750 000 cellules et 0,3 mg de collagène par ECT. | |||||
Tableau 2: Préparation de l’hydrogel ECT (y compris un excédent de 10 % tenant compte des erreurs de pipetage).
- Mélangez toute la suspension en pipetant doucement de haut en bas une seule fois, en utilisant une pipette sérologique avec une pointe d’alésage large pour éviter la formation de bulles et minimiser les contraintes de cisaillement. Assurer un mélange complet en faisant doucement tourbillonner le tube 10 fois et garder le tube centrifuge de 50 mL contenant le mélange d’hydrogel ECT sur la glace tout au long du processus de coulée.
- Pré-mouillez une pointe de pipette de 1 mL avec un mélange d’hydrogel ECT et répartissez-en 180 μL uniformément dans chaque moule de la plaque de coulée à 48 puits, en évitant les forces de cisaillement excessives qui peuvent affecter l’intégrité de l’assemblage de la matrice de collagène et en veillant à ce que la plaque entière soit réalisée en 15-20 min.
REMARQUE: Le volume de coulée recommandé est de 180 μL, mais il peut être étendu à 200 μL38. Par conséquent, si vous préférez, les volumes du tableau 2 peuvent être adaptés à 200 μL de manière à conserver les mêmes concentrations et le même rapport entre les cellules et le collagène.- Assurez-vous qu’une boucle complète est formée dans le moule (Figure 3A). Si le mélange d’hydrogel ECT est appliqué de manière discontinue, une formation complète d’anneau ECT sera évitée (Figure 3B).
- Éviter le pipetage dans le puits intérieur (Figure 3C) et la formation de bulles pendant le pipetage (Figure 3D), pour assurer une formation tissulaire homogène et fonctionnelle.
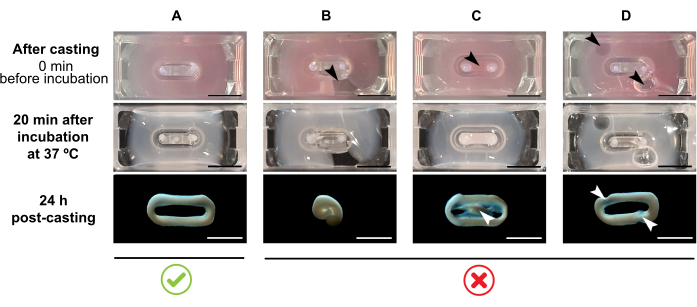
Figure 3 : Coulée, formation d’hydrogel et condensation ECT au format multi-puits. Les panneaux supérieurs illustrent l’apparence de l’ECT directement après la coulée. Les panneaux du milieu illustrent l’apparence de l’ECT après incubation pendant 20 minutes à 37 °C. Les panneaux inférieurs illustrent l’état de compactage de l’ECT 24 h après préparation, retiré des poteaux. (A) Formation correcte de l’ECT entre deux pôles au cours des 24 premières heures. (B-D) Exemples d’erreurs de pipetage qui empêchent la formation correcte de l’ECT. Les flèches blanches et noires indiquent des défauts structurels de l’ECT dus à une mauvaise coulée. Barre d’échelle: 5 mm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
- Placez soigneusement la plaque de coulée à 48 puits à l’intérieur de l’incubateur de culture cellulaire et laissez le mélange d’hydrogel ECT se reconstituer pendant 15 à 30 minutes. Après l’incubation, il apparaîtra comme un gel et opaque (Figure 3, panneau du milieu).
- Ajouter 600 μL de MGF chaudes à 37 °C par puits, sans pipeter le milieu de culture directement sur l’ECT en formation, car cela pourrait entraîner une perturbation tissulaire. Ajouter doucement le milieu de culture le long de la paroi du puits, car à ce stade, l’ECT ne doit pas non plus être détaché du fond (Figure 4).
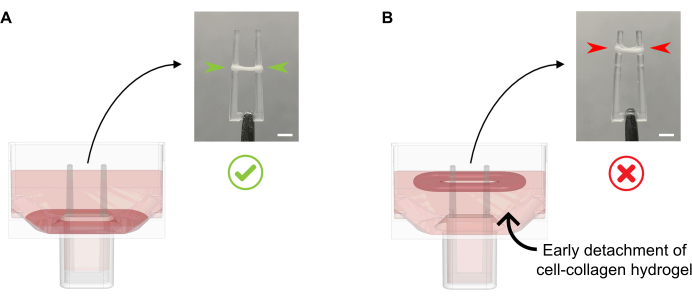
Figure 4 : Ajout correct et inapproprié d’un milieu de culture à l’ECT fraîchement coulé. (A) Lors de l’ajout du milieu de culture après solidification initiale de l’ECT (20 min après la coulée), l’ECT de condensation doit être laissé intact au fond du puits. Au cours des 24 prochaines heures, le compactage de la matrice entraînée par cellules fera glisser l’ECT vers le haut de la rampe. La position finale de l’ECT est contrôlée par des cavités concaves à une hauteur de pôle définie; cela garantit que tous les ECT s’installent à la même position pour permettre une comparaison de l’activité de flexion des pôles dans la culture ECT parallèle. (B) Former l’ECT détaché du fond tout en ajoutant le milieu de culture trop rapidement. L’ECT flottant se compactera au niveau du milieu de culture supérieur. Les forces contractantes des pôles ne seront pas directement comparables si l’ECT s’installe à des positions différentes. Barre d’échelle: 2 mm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
- Incuber pendant 24 h.
- Remplacer le milieu tous les jours par la suite, avec 500 μL de MGF jusqu’à l’analyse.
REMARQUE: Après la phase initiale de gélification indépendante des cellules, la mucoviscidose humaine commence à compacter davantage le mélange d’hydrogel ECT. Dans les 24 heures, l’ECT devrait apparaître nettement compacté et élevé au niveau où il est maintenu sur les pôles flexibles (Figure 3 et Figure 4A).
4. Évaluation du compactage ect en mesurant la section transversale (CSA) (5 min par ECT).
REMARQUE: Le compactage des tissus commence immédiatement après l’assemblage du collagène et est particulièrement important pendant les premières heures. Le compactage décrit les changements principalement déclenchés par la compression cellulaire de la matrice perpendiculairement à l’axe long du tissu. Ce paramètre est évalué en déterminant la section transversale (CSA) du TCE.
- Aux points temporels souhaités, utilisez un stéréomicroscope pour enregistrer des images macroscopiques des vues supérieures et latérales de l’ECT (Figure 5C).
REMARQUE: ECT peut être imagé à l’intérieur des puits de culture de la plaque de coulée de 48 puits. Alternativement, transférez l’ECT sur une plaque multi-puits à fond transparent pour l’imagerie. Il est conseillé d’imager l’ECT sur les pôles car le retrait de ceux-ci entraîne la perte de précharge et, par conséquent, dans un court laps de temps, le tissu peut se contracter davantage avec une torsion éventuelle en raison de la libération de tension, ce qui peut entraver une imagerie appropriée pour les analyses de dimensions. - Utilisez un programme de traitement d’image pour effectuer une analyse de balayage linéaire. Définissez une échelle et utilisez l’outil Ligne droite pour tracer et mesurer les diamètres ECT à un minimum de 6 positions par bras dans chaque plan d’imagerie (Figure 5B, C).
- Calculez le diamètre moyen à partir des plans de vue supérieur et latéral et calculez l’ASC selon une équation d’aire elliptique :


Figure 5 : Surveillance du compactage de l’ECT au fil du temps par analyse de la zone transversale (CSA). Les ECT ont été générés à l’aide de mucoviscidose humaine et de collagène de type I et cultivés autour de deux pôles flexibles pendant 5 jours. (A) Des images représentatives de l’ECT de contrôle placées dans des moules flexibles sur une période de 5 jours sont présentées. Barre d’échelle = 5 mm. De telles images en champ lumineux peuvent également être utilisées pour déterminer la variation de la déviation des pôles afin d’estimer la contraction des tissus. (B) Représentation schématique de la section transversale d’un ECT (diamètre de la vue de dessus en vert et diamètre de la vue latérale en rose). (C) Images macroscopiques de vues supérieures et latérales d’un ECT obtenues avec un stéréomicroscope et exemple correspondant d’analyse par balayage linéaire des diamètres des tissus à l’aide d’un programme de traitement d’image. Barre d’échelle = 2 mm. Les diamètres moyens sont calculés à partir de la moyenne de toutes les longueurs de ligne mesurées sur chaque plan de vue. Veuillez cliquer ici pour voir une version agrandie de cette figure.
5. Surveillance de la contraction de l’ECT par analyse de déviation des pôles (15 min par plaque de coulée de 48 puits).
REMARQUE: La culture ECT est généralement effectuée pendant 5 jours, mais elle peut être prolongée au moins jusqu’à 20 jours. La déviation des pôles se produit en raison de la contraction tissulaire entraînée par la force de contraction cellulaire dans la direction de la tension le long axe du tissu. L’évaluation de la contraction ect peut être effectuée par imagerie n’importe quel jour pendant la culture.
- Imagez la plaque de coulée de 48 puits sous un appareil d’enregistrement avec une caméra de balayage de zone intégrée placée à une distance fixe, équipée d’un capteur d’image monochrome haute résolution (≥ 5 méga pixels).
- Utilisez une source de lumière proche uv (~390 nm) pour maximiser le contraste et faciliter ainsi la détection automatisée des pointes des pôles car ils contiennent un colorant fluorescent (Figure 6A,C). S’ils sont disponibles, les lentilles télécentriques sont recommandées pour l’imagerie car elles minimisent les distorsions de l’image.
REMARQUE: Alternativement, des images macroscopiques en champ lumineux provenant de puits individuels ou de la plaque complète accompagnées d’une barre d’échelle peuvent être utilisées pour l’analyse (Figure 5A).
- Utilisez une source de lumière proche uv (~390 nm) pour maximiser le contraste et faciliter ainsi la détection automatisée des pointes des pôles car ils contiennent un colorant fluorescent (Figure 6A,C). S’ils sont disponibles, les lentilles télécentriques sont recommandées pour l’imagerie car elles minimisent les distorsions de l’image.
- Mesurez la distance entre les pôles à partir des enregistrements quotidiens (Figure 6C,D) à l’aide d’un programme de traitement d’image ou d’une analyse automatisée en exécutant des images enregistrées sur un logiciel capable de détecter des pixels lumineux à contraste élevé sur un fond sombre.
- Calculez la déviation des pôles par la variation de la distance des pôles par rapport à la distance initiale au jour zéro.
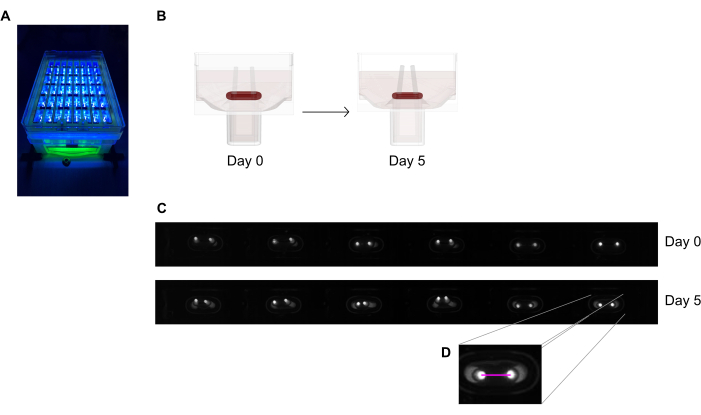
Figure 6: Aperçu schématique de l’évaluation de la contraction tissulaire en fonction de la déviation des pôles. (A) Enregistrement à haute résolution des pôles fluorescents dans la plaque de coulée de 48 puits sous excitation de lumière proche uv. Cette méthode est préférée aux images en champ lumineux pour un traçage automatisé plus précis de la pointe des pôles. (B) Les dessins schématiques montrent comment le compactage et la contraction de l’ECT conduisent à la flexion des pôles. (C) Une rangée exemplaire de la même plaque enregistre au jour 0 et au jour 5 après la coulée. D. Le gros plan montre comment mesurer la distance (ligne rose) entre les pôles à l’aide d’un programme de traitement d’image. Veuillez cliquer ici pour voir une version agrandie de cette figure.
REMARQUE: Considérez que la déviation des pôles mesurée par une image de pointe lumineuse n’est qu’une estimation de la contraction tissulaire en raison de la différence dans les plans d’imagerie. Notez également que l’application de substances pro-fibrotiques telles que le TGF-β1 pendant la culture tissulaire améliore le compactage et la contraction de l’ECT et peut finalement entraîner une perturbation précoce des tissus.
6. Évaluation de la rigidité et d’autres propriétés biomécaniques de l’ECT par mesure de traction destructive et analyse contrainte-déformation (20 min par ECT)
REMARQUE: Une courbe contrainte-déformation optimale peut afficher trois régions: région de l’orteil, région élastique et région plastique. Un exemple de courbe contrainte-déformation ECT est illustré à la figure 7. L’analyse d’une courbe contrainte-déformation permet d’extraire des paramètres biomécaniques importants du tissu tels que, par exemple, la rigidité, la résistance maximale, l’élasticité, la plasticité, l’extensibilité, la résilience et la ténacité.
- Récoltez l’ECT en tirant d’abord la civière, y compris l’ECT, hors de son puits, à l’aide d’une pince. La civière peut ensuite être maintenue sur sa base, et l’ECT a glissé sur les extrémités de la civière à l’aide d’un crochet fin ou d’une pointe de pipette.
- Transférer l’ECT sur deux crochets fixés au bras stationnaire et au bras du transducteur d’un rhéomètre d’analyse mécanique dynamique (DMA) d’extension équipé d’un bain d’orgue tempéré à 37 °C (sur mesure) rempli de PBS (Figure 7A).
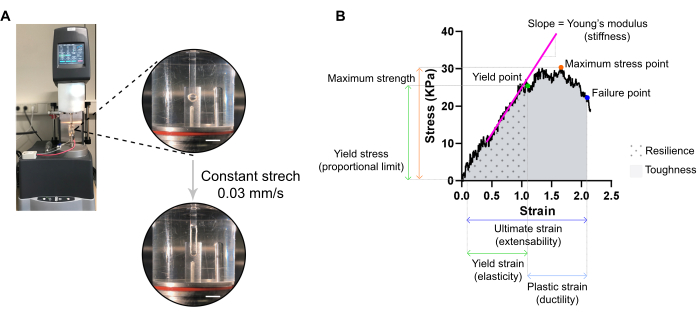
Figure 7 : Analyse de mesure de traction destructive ECT. (A) Mesure de traction rhéologique destructive sur un rhéomètre à analyse mécanique dynamique d’extension (DMA). Vue haute puissance supérieure: ECT après montage à L0 dans une chambre environnementale et connecté à un pôle supérieur et inférieur pour les analyses contrainte-déformation. Vue basse haute puissance: ECT tendu à une vitesse constante de 0,03 mm / s jusqu’au point de défaillance à la déformation ultime. Barres d’échelle = 5 mm. (B) Diagramme contrainte-déformation d’un ECT montrant les principaux paramètres mesurés. La limite supérieure de la région élastique correspond à la limite d’élasticité et la région plastique est comprise entre la limite d’élasticité et le point de défaillance (ductilité). La pente de la phase linéaire de la région élastique correspond au module de Young reflétant la rigidité tissulaire. La résistance maximale correspond à la contrainte de traction maximale qu’un tissu peut supporter. En raison de la microfracturation des fibres, le stress diminue jusqu’à ce que le tissu atteigne le point de défaillance. Cela se produit à la tension ultime (extensibilité) où une chute soudaine du stress est observée en raison de la rupture du tissu. La résilience correspond à l’énergie (kJ/m3) absorbée par le tissu avant la déformation permanente (jusqu’au point d’élasticité) et est donnée par la zone sous la courbe (ASC) jusqu’à la déformation du point d’élasticité. La ténacité correspond à l’énergie totale (kJ/m3) que le tissu peut absorber jusqu’à la rupture et est donnée par l’ASC jusqu’à la déformation ultime. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Réglez le rhéomètre pour appliquer une tension uniaxiale à une vitesse linéaire constante d’environ 1 % de la distance initiale entre les crochets par seconde. Un taux d’étirement constant de 0,03 mm/s peut être utilisé avec les dimensions ECT typiques. Tare le transducteur, initie l’étirement et enregistre jusqu’au point de rupture ect.
ATTENTION : Les images macroscopiques de l’ECT (étape 4.1.) doivent être enregistrées avant l’essai de traction, car le CSA est requis pour la normalisation des données.
REMARQUE: L’analyse contrainte-déformation, y compris le calcul CSA, peut être traitée plus tard dans le temps lors des essais de traction. Utilisez un tableur et un logiciel d’analyse statistique pour analyser les données. - Normaliser les valeurs de force mesurées (mN) par ECT par son CSA (mm2) pour obtenir des valeurs de contrainte (kPa).
- Tracer les valeurs de contrainte par rapport à la déformation (une mesure géométrique de la déformation tissulaire donnée par la distance relative entre le crochet supérieur et inférieur) sur un graphique XY.
REMARQUE: La longueur initiale du tissu (distance entre le crochet supérieur et inférieur) immédiatement avant l’étirement qui s’ensuit, L0, doit être ajustée manuellement et correspond au début de la région de l’orteil (la région de l’orteil peut être absente en fonction des propriétés du tissu). Chaque valeur de point de déformation doit être calculée selon l’équation, dans laquelle Ltotal est l’écart total à chaque point de mesure: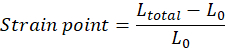
Lorsque vous tracez les données, utilisez la valeur de contrainte à L0 sélectionnée pour la soustraction d’arrière-plan. - Déterminer différents paramètres biomécaniques à partir de la courbe contrainte-déformation (utilisez la figure 7B comme exemple).
REMARQUE : Une courbe contrainte-déformation peut afficher trois régions : les régions des orteils, des élastiques et des plastiques. La limite supérieure de la région élastique, avant que le tissu ne commence à microfracturer, correspond à la limite d’élasticité, et sa déformation est une mesure de l’élasticité du tissu. La région plastique est comprise entre la limite d’élasticité et le point de défaillance. Le dernier point correspond à une chute soudaine du stress due à la rupture du tissu, définissant la tache ultime, qui est une mesure de l’extensibilité tissulaire. Le troisième point de mesure correspond à la force maximale, qui est définie par la contrainte la plus élevée que le tissu peut supporter sans se casser pendant l’étirement. La résilience et la ténacité, données par la zone sous la courbe, correspondent à l’énergie absorbée par le tissu jusqu’au point d’élasticité et au point de rupture, respectivement. Pour chaque courbe obtenue, la pente de la partie linéaire de la région élastique correspond au module de Young, également appelé module élastique, et est une propriété mécanique qui mesure la rigidité du tissu.- Extrayez de chaque courbe les valeurs XY (déformation et contrainte, respectivement) de la limite d’élasticité, du point de défaillance et du point de contrainte maximal.
- Évaluer le module de Young (rigidité en kPa = mN·mm-2) de chaque ECT à partir de la pente de la partie linéaire de la région élastique en traçant une régression linéaire de cette région.
- Utilisez un programme statistique pour calculer l’aire sous la courbe (ASC) afin de déterminer à la fois la résilience et la ténacité, jusqu’au point d’élasticité et au point de défaillance, respectivement. Calculer l’ASC par la méthode trapézoïdale. Définissez la ligne de base sur zéro et ne considérez que les pics au-dessus de la ligne de base, qui représentent au moins 10 % de la distance entre la valeur minimale et la valeur maximale dans l’axe des Y.
REMARQUE: Les modules de résilience et de ténacité sont donnés par σ × ε, où σ est la contrainte (kPa) et ε est la déformation (L / ΔL, mm / mm). Ainsi, la résilience et la ténacité sont l’énergie en kJ/m3 (kPa = kN·m-2 = kN·m·m-3 = kJ/m·m·m-3 = kJ/m3) absorbée par le tissu avant la déformation permanente et jusqu’à la rupture, respectivement.
Résultats
Les ECT atteignent environ 95 % de compactage par rapport au volume initial d’hydrogel de collagène cellulaire dans les 24 premières heures. Le compactage et la contraction des tissus dans des conditions de contrôle et en présence de FCS s’ensuivent quelques heures après la coulée et augmentent notablement jusqu’au jour 5 (Figure 5A). La déviation des pôles peut encore augmenter au cours des 15 jours suivants (20 jours ont été la plus longue période testée). L’ampleur de ...
Discussion
Le protocole présenté décrit la génération d’ECT à partir de la mucoviscidose humaine primaire, ce qui permet d’étudier l’impact mécanique de ces cellules sur leur environnement de matrice extracellulaire et vice versa.
Les fibroblastes doivent être élargis pour produire suffisamment de cellules pour les expériences ECT prévues (0,75 x 106 cellules / ECT). Pour une meilleure reproductibilité, il est conseillé de pré-cultiver des fibroblastes congelés ou dériv?...
Déclarations de divulgation
GLS et SL ont rédigé le manuscrit. Tous les auteurs ont contribué à l’élaboration du protocole et ont édité le manuscrit. TM, MT et WHZ sont les conseillers scientifiques de myriamed GmbH. WHZ est le fondateur et actionnaire de myriamed GmbH.
Remerciements
Ce travail a été soutenu par la Société allemande de cardiologie (bourse de recherche DGK pour GLS) et par la Fondation allemande pour la recherche (DFG à travers le projet IRTG 1816 pour GLS et AD; DFG 417880571 et DFG TI 956/1-1 pour MT; SFB 1002 TP C04 pour MT et WHZ; SFB 1002 TP S01 pour WHZ; et EXC 2067/1-390729940J pour WHZ). WHZ est soutenu par le ministère fédéral allemand de la Science et de l’Éducation (BMBF à travers le projet IndiHEART) et la Fondation Leducq (20CVD04). MT, WHZ et SL sont soutenus par le Centre allemand de recherche cardiovasculaire (DZHK).
matériels
| Name | Company | Catalog Number | Comments |
| Cell culture reagents: | |||
| Accutase Solution | Merk Millipore | SCR005 | |
| Dissociation reagent – TrypLE Express | Gibco | 12604013 | |
| Dulbecco's Modified Eagle Medium (DMEM) powder, high glucose | Gibco | 12100061 | |
| Dulbecco’s phosphate buffered saline (DPBS), pH 7.2, -Ca2+, -Mg2+ | Gibco | 14190144 | |
| FGM-2 Fibroblast Growth Medium-2 BulletKit | Lonza | CC-3132 | |
| FBM Fibroblast Growth Basal Medium | Lonza | CC-3131 | |
| FGM-2 Fibroblast Growth Medium-2 SingleQuots, Supplements and Growth Factors | Lonza | CC-4126 | |
| Fibroblast Growth Medium 3 KIT | PromoCell | C-23130 | |
| Fibroblast Basal Medium 3 | PromoCell | C-23230 | |
| Growth Medium 3 SupplementPack | PromoCell | C-39350 | |
| Penicillin (10000 U/mL)/ Streptomycin (10000 μL/mL) | Gibco | 15140122 | |
| Sodium hydroxide solution (NaOH) 1.0 N | Sigma-Aldrich | S2770-100ML | |
| Cell sources: | |||
| Normal human cardiac fibroblasts from the ventricle (NHCF-V) | Lonza | CC-2904 | |
| Human Cardiac Fibroblasts (HCF-c) | PromoCell | C-12375 | |
| Human Cardiac Fibroblasts (HCF-p) | PromoCell | C-12377 | |
| Primary human foreskin fibroblasts-1 (HFF-1) | ATCC | SCRC- 1041 | |
| Collagen sourses: | |||
| Collagen Type I (bovine) in 0.01 M HCl | LLC Collagen Solutions | FS22024 | 6-7 mg/mL |
| Collagen Type I (rat tail) in 0.02 M HCl | Corning | 354236 | ~4 mg/mL |
| Drugs: | |||
| Latrunculin-A (Lat-A) | Enzo Life Sciences | BML-T119-0100 | |
| Plastic ware: | |||
| Cell culture plastic ware | Sarstedt and Starlab | ||
| Mesh cell strainer (Nylon, pore size 40 μm) | Falcon | 352340 | |
| myrPlate-uniform | myriamed GmbH | TM5 med | |
| Serological pipettes wide opening, sterile (10 mL) | Corning | 07-200-619 | |
| Specific instruments: | |||
| Bi-telecentric CORE lens for 1/2″ detectors | OptoEngineering | TCCR12096 | |
| Area scan camera Basler ace acA4024 | Basler | 107404 |
Références
- Driesen, R. B., et al. Reversible and irreversible differentiation of cardiac fibroblasts. Cardiovascular Research. 101 (3), 411-422 (2014).
- Shi, X., et al. Elasticity of cardiac cells on the polymer substrates with different stiffness: an atomic force microscopy study. Physical Chemistry Chemical Physics. 13 (16), 7540-7545 (2011).
- Elson, E. L., Genin, G. M. Tissue constructs: platforms for basic research and drug discovery. Interface Focus. 6 (1), 20150095 (2016).
- Cho, N., Razipour, S. E., McCain, M. L. TGF-beta1 dominates extracellular matrix rigidity for inducing differentiation of human cardiac fibroblasts to myofibroblasts. Experimental Biology and Medicine. 243 (7), 601-612 (2018).
- Cucoranu, I., et al. NAD(P)H oxidase 4 mediates transforming growth factor-beta1-induced differentiation of cardiac fibroblasts into myofibroblasts. Circulation Research. 97 (9), 900-907 (2005).
- Peng, H., Carretero, O. A., Peterson, E. L., Rhaleb, N. E. Ac-SDKP inhibits transforming growth factor-beta1-induced differentiation of human cardiac fibroblasts into myofibroblasts. American Journal of Physiology-Heart and Circulatory Physiology. 298 (5), 1357-1364 (2010).
- Ribeiro, A. J., et al. Contractility of single cardiomyocytes differentiated from pluripotent stem cells depends on physiological shape and substrate stiffness. Proceedings of the National Academy of Sciences of the United States of America. 112 (41), 12705-12710 (2015).
- Tranquillo, R. T., Durrani, M. A., Moon, A. G. Tissue engineering science: consequences of cell traction force. Cytotechnology. 10 (3), 225-250 (1992).
- Barocas, V. H., Moon, A. G., Tranquillo, R. T. The fibroblast-populated collagen microsphere assay of cell traction force--Part 2: Measurement of the cell traction parameter. Journal of Biomechanical Engineering. 117 (2), 161-170 (1995).
- Lijnen, P., Petrov, V., Rumilla, K., Fagard, R. Stimulation of collagen gel contraction by angiotensin II and III in cardiac fibroblasts. Journal of the Renin-Angiotensin-Aldosterone System. 3 (3), 160-166 (2002).
- Baxter, S. C., Morales, M. O., Goldsmith, E. C. Adaptive changes in cardiac fibroblast morphology and collagen organization as a result of mechanical environment. Cell Biochemistry and Biophysics. 51 (1), 33-44 (2008).
- Zhou, Y., et al. Inhibition of mechanosensitive signaling in myofibroblasts ameliorates experimental pulmonary fibrosis. Journal of Clinical Investigation. 123 (3), 1096-1108 (2013).
- Lijnen, P., Petrov, V., Fagard, R. In vitro assay of collagen gel contraction by cardiac fibroblasts in serum-free conditions. Methods and Findings in Experimental and Clinical Pharmacology. 23 (7), 377-382 (2001).
- Burgess, M. L., et al. Integrin-mediated collagen gel contraction by cardiac fibroblasts. Effects of angiotensin II. Circulation Research. 74 (2), 291-298 (1994).
- Nunohiro, T., Ashizawa, N., Graf, K., Hsueh, W. A., Yano, K. Angiotensin II promotes integrin-mediated collagen gel contraction by adult rat cardiac fibroblasts. Japanese Heart Journal. 40 (4), 461-469 (1999).
- Ngu, J. M., et al. Human cardiac fibroblast extracellular matrix remodeling: Dual effects of tissue inhibitor of metalloproteinase-2. Cardiovascular Pathology. 23 (6), 335-343 (2014).
- Knezevic, V., Sim, A. J., Borg, T. K., Holmes, J. W. Isotonic biaxial loading of fibroblast-populated collagen gels: a versatile, low-cost system for the study of mechanobiology. Biomechanics and Modeling in Mechanobiology. 1 (1), 59-67 (2002).
- Delvoye, P., Wiliquet, P., Leveque, J. L., Nusgens, B. V., Lapiere, C. M. Measurement of mechanical forces generated by skin fibroblasts embedded in a three-dimensional collagen gel. Journal of Investigative Dermatology. 97 (5), 898-902 (1991).
- Kolodney, M. S., Elson, E. L. Correlation of myosin light chain phosphorylation with isometric contraction of fibroblasts. Journal of Biological Chemistry. 268 (32), 23850-23855 (1993).
- Bell, B. J., Nauman, E., Voytik-Harbin, S. L. Multiscale strain analysis of tissue equivalents using a custom-designed biaxial testing device. Biophysical Journal. 102 (6), 1303-1312 (2012).
- Wakatsuki, T., Kolodney, M. S., Zahalak, G. I., Elson, E. L. Cell mechanics studied by a reconstituted model tissue. Biophysical Journal. 79 (5), 2353-2368 (2000).
- Thomopoulos, S., et al. Fibrocartilage tissue engineering: The role of the stress environment on cell morphology and matrix expression. Tissue Engineering Part A. 17 (7-8), 1039-1053 (2011).
- Roeder, B. A., Kokini, K., Sturgis, J. E., Robinson, J. P., Voytik-Harbin, S. L. Tensile mechanical properties of three-dimensional type I collagen extracellular matrices with varied microstructure. Journal of Biomechanical Engineering. 124 (2), 214-222 (2002).
- Ongherth, A., et al. p63RhoGEF regulates auto- and paracrine signaling in cardiac fibroblasts. Journal of Molecular and Cellular Cardiology. 88, 39-54 (2015).
- Vettel, C., et al. PDE2-mediated cAMP hydrolysis accelerates cardiac fibroblast to myofibroblast conversion and is antagonized by exogenous activation of cGMP signaling pathways. American Journal of Physiology-Heart and Circulatory Physiology. 306 (8), 1246-1252 (2014).
- Jatho, A., et al. RhoA Ambivalently Controls Prominent Myofibroblast Characteritics by Involving Distinct Signaling Routes. PLoS One. 10 (10), 0137519 (2015).
- Dworatzek, E., et al. Sex-specific regulation of collagen I and III expression by 17beta-Estradiol in cardiac fibroblasts: role of estrogen receptors. Cardiovascular Research. 115 (2), 315-327 (2019).
- Tiburcy, M., Meyer, T., Soong, P. L., Zimmermann, W. H. Collagen-based engineered heart muscle. Methods in Molecular Biology. 1181, 167-176 (2014).
- Schlick, S. F., et al. Agonistic and antagonistic roles of fibroblasts and cardiomyocytes on viscoelastic stiffening of engineered human myocardium. Progress in Biophysics and Molecular Biology. 144, 51-60 (2019).
- Wille, J. J., Elson, E. L., Okamoto, R. J. Cellular and matrix mechanics of bioartificial tissues during continuous cyclic stretch. Annals of Biomedical Engineering. 34 (11), 1678-1690 (2006).
- Berry, C. C., Shelton, J. C., Bader, D. L., Lee, D. A. Influence of external uniaxial cyclic strain on oriented fibroblast-seeded collagen gels. Tissue Engineering. 9 (4), 613-624 (2003).
- Stopak, D., Harris, A. K. Connective tissue morphogenesis by fibroblast traction. I. Tissue culture observations. Developmental Biology. 90 (2), 383-398 (1982).
- Bellows, C. G., Melcher, A. H., Aubin, J. E. Association between tension and orientation of periodontal ligament fibroblasts and exogenous collagen fibres in collagen gels in vitro. Journal of Cell Science. 58 (1), 125-138 (1982).
- Tranquillo, R. T. Self-organization of tissue-equivalents: the nature and role of contact guidance. Biochemical Society Symposia. 65, 27-42 (1999).
- Barocas, V. H., Tranquillo, R. T. An anisotropic biphasic theory of tissue-equivalent mechanics: the interplay among cell traction, fibrillar network deformation, fibril alignment, and cell contact guidance. Journal of Biomechanical Engineering. 119 (2), 137-145 (1997).
- Yip, A. K., et al. Anisotropic traction stresses and focal adhesion polarization mediates topography-induced cell elongation. Biomaterials. 181, 103-112 (2018).
- Santos, G. L., Hartmann, S., Zimmermann, W. H., Ridley, A., Lutz, S. Inhibition of Rho-associated kinases suppresses cardiac myofibroblast function in engineered connective and heart muscle tissues. Journal of Molecular and Cellular Cardiology. 134, 13-28 (2019).
- Kittana, N., et al. Modulating the biomechanical properties of engineered connective tissues by chitosan-coated multiwall carbon nanotubes. International Journal of Nanomedicine. 16, 989-1000 (2021).
- Kittana, N., et al. Enhancement of wound healing by single-wall/multi-wall carbon nanotubes complexed with chitosan. International Journal of Nanomedicine. 13, 7195-7206 (2018).
- Antoine, E. E., Vlachos, P. P., Rylander, M. N. Review of collagen I hydrogels for bioengineered tissue microenvironments: characterization of mechanics, structure, and transport. Tissue Engineering Part B: Reviews. 20 (6), 683-696 (2014).
- Holder, A. J., et al. Control of collagen gel mechanical properties through manipulation of gelation conditions near the sol-gel transition. Soft Matter. 14 (4), 574-580 (2018).
- Zimmermann, W. H., et al. Tissue engineering of a differentiated cardiac muscle construct. Circulation Research. 90 (2), 223-230 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon