Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Criblage à haut débit d’isolats microbiens ayant un impact sur la santé de Caenorhabditis elegans
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les microbes intestinaux peuvent avoir un impact positif ou négatif sur la santé de leur hôte via des mécanismes spécifiques ou conservés. Caenorhabditis elegans est une plate-forme pratique pour dépister de tels microbes. Le présent protocole décrit le dépistage à haut débit de 48 isolats bactériens pour déterminer leur impact sur la résistance au stress des nématodes, utilisés comme indicateur de la santé des vers.
Résumé
Avec sa petite taille, sa courte durée de vie et sa génétique facile, Caenorhabditis elegans offre une plate-forme pratique pour étudier l’impact des isolats microbiens sur la physiologie de l’hôte. Il devient également fluorescent en bleu lors de la mort, fournissant un moyen pratique de localiser la mort. Cette propriété a été exploitée pour développer des tests de survie de C. elegans sans marquage à haut débit (LFASS). Celles-ci impliquent un enregistrement de fluorescence en accéléré des populations de vers placées dans des plaques multipuits, à partir desquelles le temps médian de mort de la population peut être déduit. La présente étude adopte l’approche LFASS pour dépister simultanément plusieurs isolats microbiens afin de déterminer les effets sur la sensibilité de C. elegans à la chaleur sévère et aux stress oxydatifs. Un tel pipeline de dépistage microbien, qui peut notamment être utilisé pour présélectionner les probiotiques, en utilisant une résistance sévère au stress comme indicateur de la santé de l’hôte, est rapporté ici. Le protocole décrit comment cultiver à la fois des collections d’isolats de microbiote intestinal de C. elegans et des populations de vers synchrones dans des réseaux multipuits avant de les combiner pour les essais. L’exemple fourni couvre l’analyse de 47 isolats bactériens et d’une souche témoin sur deux souches de vers, dans deux essais de stress en parallèle. Cependant, le pipeline d’approche est facilement évolutif et applicable à l’examen de nombreuses autres modalités. Ainsi, il fournit une configuration polyvalente pour étudier rapidement un paysage multiparamétrique de conditions biologiques et biochimiques qui ont un impact sur la santé de C. elegans.
Introduction
Le corps humain abrite environ 10 à 100 billions de cellules microbiennes vivantes (bactéries, champignons archées), qui se trouvent principalement dans les environnements intestinaux, cutanés et muqueux1. Dans un état sain, ceux-ci offrent des avantages à leur hôte, y compris la production de vitamines, la maturation du système immunitaire, la stimulation des réponses immunitaires innées et adaptatives aux agents pathogènes, la régulation du métabolisme des graisses, la modulation des réponses au stress, etc., avec un impact sur la croissance et le développement, l’apparition de la maladie et le vieillissement 2,3,4,5 . Le microbiote intestinal évolue également considérablement tout au long de la vie. L’évolution la plus radicale se produit pendant la petite enfance et la petite enfance6, mais des changements significatifs se produisent également avec l’âge, notamment une diminution de l’abondance de Bifidobacterium et une augmentation des espèces de Clostridium, Lactobacillus, Enterobacteriaceae et Enterococcus 7. Le mode de vie peut altérer davantage la composition microbienne intestinale conduisant à une dysbiose (perte de bactéries bénéfiques, prolifération de bactéries opportunistes), entraînant diverses pathologies telles que les maladies inflammatoires de l’intestin, le diabète et l’obésité5, mais contribuant également aux maladies d’Alzheimer et de Parkinson 8,9,10,11.
Cette prise de conscience a contribué de manière critique à affiner le concept de l’axe intestin-cerveau (ACS), où les interactions entre la physiologie intestinale (y compris maintenant les microbes qu’elle contient) et le système nerveux sont considérées comme le principal régulateur du métabolisme animal et des fonctions physiologiques12. Cependant, le rôle précis du microbiote dans la signalisation intestin-cerveau et les mécanismes d’action associés sont loin d’être entièrement compris13. Le microbiote intestinal étant un déterminant clé du vieillissement en bonne santé, la façon dont les bactéries modulent le processus de vieillissement est devenue un sujet de recherche intense et de controverse 6,14,15.
Avec la démonstration que le ver rond Caenorhabditis elegans héberge un véritable microbiote intestinal dominé - comme chez d’autres espèces - par Bacteroidetes, Firmicutes, et Actinobacteria 16,17,18,19,20, son ascension rapide en tant que plate-forme expérimentale pour étudier les interactions commensales hôte-intestin21,22,23,24 ,25,26 a considérablement élargi notre arsenal d’enquête26,27,28,29. En particulier, les approches expérimentales à haut débit disponibles pour C. elegans pour étudier les interactions gène-alimentation, gène-médicament, gène-pathogène, etc., peuvent être adaptées pour explorer rapidement l’impact des isolats et des cocktails bactériens sur la santé et le vieillissement de C. elegans.
Le présent protocole décrit un pipeline expérimental permettant de dépister immédiatement des réseaux d’isolats ou de mélanges bactériens placés dans des plaques multipuits pour déterminer les effets sur la résistance au stress de C. elegans en tant qu’indicateur de la santé, ce qui peut être utilisé pour identifier les probiotiques. Il explique comment développer de grandes populations de vers et manipuler des réseaux bactériens dans des formats de plaques à 96 et 384 puits avant de traiter les vers pour une analyse automatisée de la résistance au stress à l’aide d’un lecteur de plaque de fluorescence (Figure 1). L’approche est basée sur des tests de survie automatisés sans marquage (LFASS)30 qui exploitent le phénomène de fluorescence de mort31, par lequel les vers mourants produisent une explosion de fluorescence bleue qui peut être utilisée pour déterminer le moment de la mort. La fluorescence bleue est émise par les esters glucosyles de l’acide anthranilique stockés dans les granules intestinaux de C. elegans (un type d’organite apparenté au lysosome), qui éclatent lorsqu’une cascade nécrotique est déclenchée dans l’intestin du ver à la mort31.
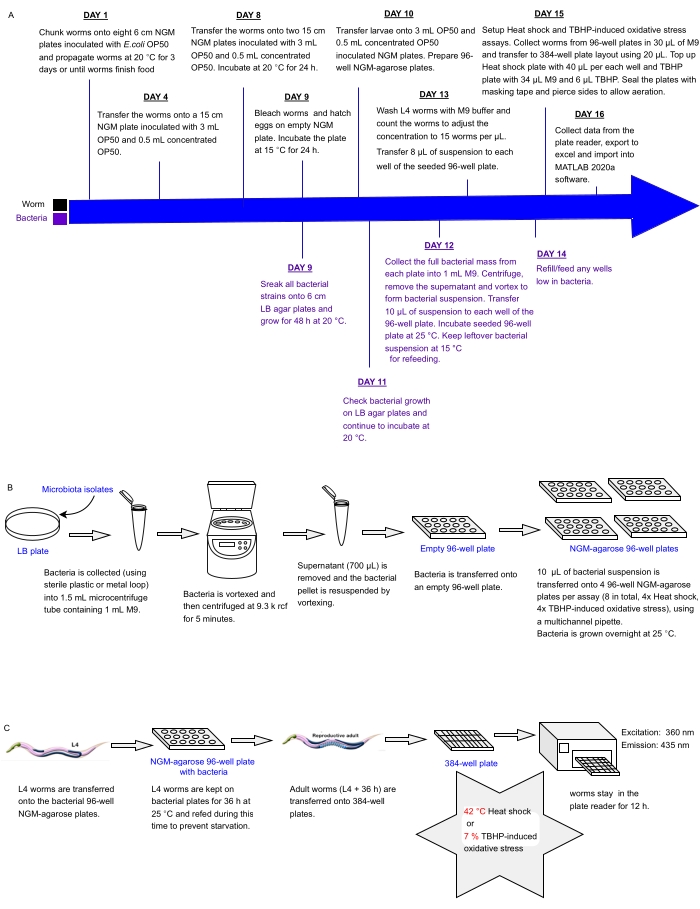
Figure 1 : Flux de travail expérimental pour le criblage à haut débit d’isolats bactériens ayant un impact sur la résistance de C. elegans au stress. (A) Calendrier pour l’entretien et la mise en place des tests vers et bactériens. (B) Installation et manipulation de réseaux de plaques bactériennes à 96 puits. (C) Installation de plaques à vis sans fin à 384 puits. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
Les deux souches de C. elegans utilisées en parallèle pour la présente étude étaient Bristol N2 de type sauvage et HT1890: daf-16 (mgDf50), qui croissent à des taux similaires. Cependant, le protocole peut être reproduit avec n’importe quelle combinaison de deux souches qui ont des taux de croissance similaires. Notez que, lors de l’essai d’autres souches en parallèle (par exemple, les mutants daf-2 de type sauvage et à croissance lente), des taux de croissance différents doivent être pris en compte et, par conséquent, le protocole doit être ajusté. Les échelles de temps et les quantités de vers et de bactéries dans le protocole suivant sont optimisées pour les tests parallèles de 48 isolats bactériens sur deux souches de vers dans deux tests LFASS dans des tétraplicates. Des ajustements seront nécessaires si d’autres conditions doivent être testées en parallèle. Escherichia coli La souche de bactéries OP50 a été obtenue auprès du Caenorhabditis Genetics Center (CGC) de l’Université du Minnesota. Les 48 isolats bactériens ont été obtenus du laboratoire de Schulenburg et conservés sur gélose LB.
1. C. elegans cultivant sur OP50 (jours 1 à 8)
REMARQUE : L’approche actuelle vise à cultiver C. elegans hermaphrodites sur un milieu solide à tous les stades et à éviter les changements alimentaires inutiles (c.-à-d. en utilisant d’autres souches d’E. coli à croissance plus rapide comme NA22 ou des milieux de croissance plus riches comme les plaques d’œufs) pour rester aussi près que possible des conditions de croissance standard32,33 qui sont encore largement utilisées. La température de croissance du ver (ici fixée à 15 °C) dépend de la ou des souches de C. elegans utilisées et peut nécessiter des ajustements (par exemple, pour éviter ou déclencher l’expression d’un phénotype ou d’un biomarqueur sensible à la température). Pour plus d’informations sur l’élevage des vers, veuillez consulter la référence33.
- Préparer huit plaques de NGM de 6 cm de diamètre (10 mL de gélose milieu de croissance des nématodes, NGM, dossier supplémentaire 1)32,33 par souche de ver et laisser sécher pendant 1 jour à température ambiante.
- Préparer une culture liquide saturée de bactéries E. coli OP50 en ensemençant un seul clone bactérien à partir d’une plaque de gélose au bouillon de lysogénie fraîchement cultivée (gélose LB, dossier supplémentaire 1) dans 25 mL de milieu OP50 (dossier supplémentaire 1) dans un tube conique de 50 mL. Cultiver la culture pendant la nuit à 37 °C dans un incubateur vibreur.
- Inoculer les huit plaques de NGM de 6 cm par souche avec 100 μL de culture liquide saturée d’E. coli OP50 par boîte et conserver les plaques à 20 °C pendant 2 jours avant utilisation.
- À l’aide d’un scalpel, couper et transférer un morceau de gélose carrée de 0,5 cm avec des vers d’une plaque NGM récemment affamée sur chacune des huit plaques de NGM inoculées de 6 cm et incuber ces plaques à 20 °C pendant 3 jours (ou jusqu’à ce que les vers aient fini la nourriture).
- Préparer cinq plaques de NGM de 15 cm par souche de vers (30 mL de milieu NGM par plaque) et inoculer avec 3 mL d’OP50. Laisser sécher les assiettes avant d’incuber à 37 °C pendant une nuit. Maintenir les plaques à 20 °C jusqu’à ce qu’elles soient utilisées ultérieurement.
- À l’aide d’une pipette P-1000, ajouter jusqu’à 3 mL de tampon M9 stérile (dossier supplémentaire 1) aux plaques de NGM de 6 cm (étape 1.1.) pour remettre les vers en suspension et recueillir la solution de vers des huit plaques par souche dans un seul tube conique de 15 mL.
- Centrifuger à 142 x g pendant 2 min à 4 °C. Retirez délicatement le surnageant à l’aide d’une pipette P-5000 ou d’une pompe à eau équipée d’une pipette ou d’un embout Pasteur stérile. Ajouter 10 ml de tampon M9 stérile pour laver la pastille de vers. Répétez 2x.
- Retirer le surnageant (autant que possible) et transférer les vers sur une plaque de NGM inoculée OP50 de 15 cm (étape 1.5.) à l’aide d’une pipette. Ajouter 0,5 mL de culture OP50 concentrée.
- Pour faire de l’OP50 concentré, inoculer chacune des quatre bouteilles de 1 L de LB avec 2 mL de culture de démarrage OP50 (préparée à l’étape 1.2.) et cultiver dans un incubateur à agitation pendant 6 h à 37 °C et 160 x g. Enduire les bactéries en granulés à 3057 x g et 20 °C pendant 15 min. Jeter le surnageant, remettre en suspension les pastilles bactériennes avec 6 mL de milieu OP50 et recueillir dans un tube conique stérile de 50 mL.
REMARQUE: Les bactéries peuvent être conservées à 4 ° C jusqu’à 1 semaine.
- Cultiver chaque souche de vers sur une plaque NGM de 15 cm de diamètre pendant 3-4 jours à 15 °C en réalimentant les vers avec 0,5 mL d’OP50 concentré par jour.
- Une fois que les vers ont presque fini la nourriture, recueillir et laver dans un tampon M9 (étape 1.6.1.), transférer chaque culture de souche de vers sur deux plaques NGM de 15 cm (étape 1.5.) et propager les vers à 20 °C jusqu’à ~95% de la population soient des adultes gravides (il faudra environ 24 heures pour le type sauvage Bristol N2).
NOTE: Les adultes gravides sont caractérisés par la présence d’œufs dans le ver, et la plaque idéale devrait également avoir une abondance d’œufs non éclos pondus sur l’assiette sans trop de larves33.
- Une fois que les vers ont presque fini la nourriture, recueillir et laver dans un tampon M9 (étape 1.6.1.), transférer chaque culture de souche de vers sur deux plaques NGM de 15 cm (étape 1.5.) et propager les vers à 20 °C jusqu’à ~95% de la population soient des adultes gravides (il faudra environ 24 heures pour le type sauvage Bristol N2).
2. Maintien des collections d’isolat du microbiote intestinal (Jour 9)
- Étaler les 48 isolats bactériens sur des plaques individuelles de gélose LB de 6 cm et croiser pendant 48 h à 20 °C.
REMARQUE: Les bactéries peuvent être cultivées à 25 ° C pendant 24-36 h si nécessaire plus tôt, mais la croissance plus longue de 20 ° C permet de repérer les contaminants potentiels. - Synchronisez un grand nombre de C. elegans.
- Blanchir les vers adultes gravides en suivant la méthode standard de préparation des œufs33 et transférer les œufs sur deux plaques NGM de 15 cm non ensemencées pendant 24 h à 15 °C pour permettre à toutes les larves L1 d’éclore et de croître de manière synchrone dans les étapes suivantes.
ATTENTION : Soyez prudent lorsque vous manipulez des solutions d’eau de Javel.
- Blanchir les vers adultes gravides en suivant la méthode standard de préparation des œufs33 et transférer les œufs sur deux plaques NGM de 15 cm non ensemencées pendant 24 h à 15 °C pour permettre à toutes les larves L1 d’éclore et de croître de manière synchrone dans les étapes suivantes.
3. Culture de grandes cultures de C. elegans (Jour 10)
- Une fois éclos, recueillir les larves L1 (à partir de l’étape 2.2.1.) dans 3-4 mL de M9 dans un tube conique propre de 15 mL. Pipeter quatre gouttes de 10 μL de solution de vers sur une lame ou une plaque et compter le nombre de vers dans chaque goutte sous un stéréomicroscope à un grossissement de 16x. Déterminer la concentration de vers de la solution en faisant la moyenne du nombre de larves de toutes les gouttes de solution de vers. Multipliez cette valeur par le volume restant et estimez le nombre total de vers pour chaque souche.
REMARQUE : De 46 000 à 50 000 larves L1 par souche sont nécessaires à ce stade pour remplir ultérieurement une plaque de 384 puits ou deux demi-plaques.- Pour chaque souche, transférer toutes les larves L1 sur deux plaques NGM de 15 cm (23 000-25 000 L1 par plaque) préalablement inoculées avec 3 mL d’OP50 (étape 1.5.) et réensemencées avec 0,5 mL d’OP50 concentré.
- Incuber à 15 °C, en complétant avec 0,5 mL d’OP50 concentré par jour au besoin jusqu’à ce que les vers atteignent le stade L4.
NOTE: Le stade L4 est caractérisé par un intestin légèrement plus foncé et une tache blanche en demi-disque ou en forme de croissant où la vulve finira par former32,33. - Préparez des plaques d’agarose NGM à 96 puits en suivant les étapes ci-dessous.
- Préparer huit plaques d’agarose NGM à 96 puits en remplissant chaque puits avec 125 μL de NGM-agarose (quatre plaques par essai).
REMARQUE: Il est recommandé de plaquer quelques plaques supplémentaires si certaines sont contaminées dans les étapes suivantes. Deux lecteurs de plaques seront nécessaires pour effectuer des essais en parallèle, mais ils peuvent également être exécutés successivement en commençant par le test de contrainte thermique, car il peut être exécuté pendant aussi peu que 6 heures. Pour ces plaques, la gélose de cendres à <4 % est remplacée par de l’agarose (voir le tableau des matériaux), ce qui permet un séchage plus lent et plus uniforme sur les bouchons NGM et réduit l’enfouissement des vers pour une meilleure récupération. - Assurez-vous que les puits sont remplis uniformément et sans bulles. Utilisez un bloc thermique réglé à 70 °C (avec un transfert de chaleur lent à travers le plastique de la plaque multipuits, l’agarose NGM ne peut chauffer que jusqu’à environ 55-60 °C) pour empêcher le mélange de se solidifier pendant le processus. Pour enlever les bulles dans les puits, utilisez une aiguille stérile chauffée à la flamme.
- Laisser les plaques à 96 puits reposer à température ambiante dans un environnement stérile avant de les retourner (couvercle vers le bas pour éviter la condensation) et conserver à 4 °C dans une boîte propre jusqu’à ce que vous en ayez besoin.
- Préparer huit plaques d’agarose NGM à 96 puits en remplissant chaque puits avec 125 μL de NGM-agarose (quatre plaques par essai).
- Le jour 11, vérifiez les vers de l’étape 3.2., en vous assurant qu’aucune contamination n’est apparue et que les vers sont toujours pleins.
- Le jour 12, vérifiez les vers de l’étape 3.1., en vous assurant qu’aucune contamination n’est apparue et que les vers sont toujours remplis. Vérifiez également le stade de développement des vers.
REMARQUE: Le sexe/souche et le stade de développement du ver, comme les vers L4 ou L4 + 24 h utilisés, dépendent des traitements auxquels les vers sont soumis. Ici, des hermaphrodites de type sauvage ont été exposés à des isolats bactériens de L4 pendant 36 heures.
4. Préparation des collections d’isolats du microbiote intestinal pour la réalimentation des vers
- Surveillez la croissance bactérienne sur les plaques de gélose LB à partir de l’étape 2.1. et continuer à incuber à 20 °C.
NOTE: Bien que ce ne soit pas idéal, dans le cas où certains clones ne se développent pas ou ne révèlent pas de contaminations, les bactéries peuvent être re-striées à partir de stocks propres sur des plaques de 6 cm LB et cultivées à 25-28 ° C pendant 24 heures pour être prêtes pour l’expérience. - Définir une disposition de réseau de 96 puits pour la collection bactérienne testée, facilitant l’ensemencement systématique des plaques et l’analyse des données dans les étapes suivantes (tableau supplémentaire 1).
- Prélever la masse bactérienne de chaque plaque bactérienne de 6 cm (étape 4.1.) et la transférer dans un tube microcentrifuge marqué de 1,5 mL contenant 1 mL de tampon M9. Pour ce faire, utilisez une boucle en plastique stérile à usage unique de 2 mm de diamètre ou une boucle métallique de 5 mm de diamètre. Stériliser la boucle métallique entre les souches bactériennes en la trempant dans de l’éthanol à 100%, en la flambant et en la refroidissant pendant 5 s.
- Vortex les tubes de microcentrifugation jusqu’à ce que les pastilles bactériennes soient complètement remises en suspension (selon la souche bactérienne, cela peut prendre ~1-10 s).
- Faire tourner à 9 300 x g pendant 5 minutes à température ambiante, retirer 700 μL de surnageant et remettre en suspension la pastille bactérienne par vortex.
- Transférer 200 μL de chaque suspension bactérienne dans un seul puits d’une plaque stérile vide de 96 puits selon la disposition décrite à l’étape 4.2.
- À partir de cette plaque, inoculer huit plaques d’agarose NGM à 96 puits (préparées à l’étape 3.3.) avec 10 μL de solution bactérienne à l’aide d’une pipette multicanal et incuber avec le couvercle à 25 °C pendant 24 h. Ne pas sceller les plaques pour permettre le séchage des plaques et la croissance aérobie bactérienne et pour éviter l’excès de condensation.
- Sceller la plaque de suspension à 96 puits préparée à l’étape 4.6. avec un film adhésif propre (voir le tableau des matériaux) et conserver à 15 °C jusqu’à 5 jours. Cela sera utilisé pour la réalimentation des vers au besoin.
5. Configuration du choc thermique et des tests oxydatifs LFASS (jours 13 - 14)
- En regardant les plaques de l’étape 3.5., évaluez le stade de développement des vers. Une fois que >90 % des vers ont atteint L434, recueillir les vers dans jusqu’à 10 mL de solution M9 stérile dans des tubes coniques de 15 mL.
- Laver abondamment les vers (au moins 4x) en tournant vers le bas à 142 x g pendant 2 minutes à 4 °C, en retirant le surnageant et en ajoutant 10 ml de M9 stérile frais entre chaque lavage pour éliminer les bactéries OP50. Remettez en suspension la pastille de ver dans 10 mL de M9.
- Transférer 50 μL de solution de vis sans fin dans un tube de liaison à faible surface (voir le tableau des matériaux) contenant 950 μL de M9. Après avoir mélangé doucement le contenu du tube pour éviter la sédimentation par les vers, utilisez rapidement une pointe de pipette humide à faible liaison pour transférer 3 à 4 gouttes distinctes de 10 μL sur une lame de verre ou une plaque NGM, et compter le nombre de vers au stéréomicroscope (voir le tableau des matériaux) à un grossissement de 16x. Faites la moyenne des comptes à partir des 3-4 gouttes et déterminez le nombre de vers par microlitre dans la solution de vers (voir étape 3.1.).
- Ajustez la concentration du ver dans le tube de 10 mL pour atteindre ~120 vers dans 8 μL. Si la solution préparée à l’étape 5.2. n’est pas assez concentré, faites tourner les vers vers vers et retirez M9 en conséquence pour atteindre 120 vers par 8 μL.
- Transférer 8 μL de solution de vers (~120 vers) dans chacun des puits des huit plaques NGM-agarose à 96 puits de l’étape 4.7., à l’aide d’une pipette multicanal ou d’une pipette répétée. Assurez-vous d’utiliser des conseils de faible rétention pour limiter la perte de vers. Il peut également être nécessaire de couper les extrémités pour permettre aux grands vers adultes de limiter le stress mécanique sur les vers adultes.
REMARQUE: Le test nécessite un minimum de 30 vers sains vivants pour fonctionner de manière fiable, mais fonctionne mieux avec environ 100 vers par puits. - Incuber les plaques d’agarose NGM à 96 puits ensemencées par le ver et la bactérie à 25 °C pendant 36 h.
- Vérifiez les plaques entre 12 et 24 h, en vous assurant que les vers restent remplis partout. Si une réalimentation est nécessaire, remettre les bactéries en suspension dans la plaque de réseau bactérienne à 96 puits stockée à 15 °C à l’étape 4.8 et ajouter jusqu’à 10 μL de la solution bactérienne correspondante aux plaques d’agarose NGM à 96 puits où les vers risquent de mourir de faim avant la fin de la période d’incubation de 36 heures (les vers affamés produiront des résultats très différents, C’est donc très important).
REMARQUE: Les étapes suivantes doivent être effectuées le jour 15. Avant de commencer le test, il peut être nécessaire d’optimiser la hauteur de lecture. La lecture optimale sera atteinte à 20-50 μm au-dessus du fond du puits. Cela dépendra du modèle du lecteur de plaques. Certains offrent la possibilité d’un Z-scan, tandis que d’autres permettent une saisie manuelle de la hauteur. Réglez la hauteur optimale au niveau où le signal de fluorescence bleue le plus élevé (365 nm/430 nm) est détecté. Certains lecteurs de plaques peuvent fonctionner à une hauteur fixe optimisée pour les tests cellulaires adhérents et peuvent ne pas être idéaux pour les tests LFASS. - Après 36 h, distribuer 30 μL de M9 dans chaque puits de la plaque de 96 puits.
REMARQUE: Pour les tests de contrainte thermique, le lecteur de plaque doit avoir atteint la température requise pour effectuer le test et peut avoir besoin d’être allumé à l’avance. Le protocole actuel utilise 42 °C pour maximiser la vitesse de destruction, mais l’approche s’applique à d’autres températures supérieures à 30 °C. - Transfère les vers (environ 20 μL) dans la plaque de 384 puits selon les dispositions définies, en utilisant des pointes à faible rétention (envisagez de couper l’extrémité des pointes pour permettre aux gros vers de réduire le stress mécanique des vers adultes).
NOTE: Pour la présente étude, deux réglages différents de lecteur de plaques sont utilisés pour les deux essais décrits ici (contrainte thermique et stress oxydatif), et donc les échantillons destinés à ces deux essais ne doivent pas être plaqués dans la même plaque de 384 puits. - Assurez-vous que les lecteurs de plaques sont correctement configurés (tableau 1).
- Complétez les plaques de 384 puits avec plus de M9, en visant un volume final de 60 μL par puits. Pour l’essai du stress thermique, ajouter 40 μL de M9, et pour le stress oxydatif induit par la t-BHP, ajouter 34 μl de M9 dans 6 μl de t-BHP (voir le tableau des matériaux).
- Commencez l’essai dans les 2 minutes suivant l’ajout de t-BHP (idéalement, tous les vers doivent être exposés simultanément à la t-BHP, la résolution du temps d’essai étant de 2 min). Si ce n’est pas possible, utilisez une minuterie pour estimer le temps passé à pipeter la t-BHP avant le début de l’essai afin de permettre un ajustement ultérieur du temps médian de décès.
- Fermez les plaques avec leur couvercle transparent. Scellez les bords des plaques de 384 puits avec du ruban de masquage (ruban adhésif sur la plaque et le couvercle), en veillant à ce que le ruban ne passe pas sur le couvercle ou sous la plaque. Couper le ruban entre le couvercle et la plaque à intervalles réguliers à l’aide d’un scalpel pour permettre l’échange d’air tout en minimisant l’évaporation pendant l’essai.
- Insérez la plaque dans le lecteur de plaques (voir le tableau des matériaux) et commencez la course. Viser à exciter à 365 nm et détecter les émissions à 435 nm toutes les 2 minutes pendant 6 à 12 h (tableau 1).
REMARQUE : En règle générale, 6 h suffisent pour les dosages de stress thermique à 42 °C et 8 h pour les dosages de stress oxydatif à 7 % de la t-BHP.
6. Traitement des données des lecteurs de plaques
- Enregistrez les données de fluorescence brutes du lecteur de plaques au format .txt, .csv ou .xls /.xlsx séparés par des virgules ou des tabulations, puis convertissez-les au format xls /.xlsx. Selon le format des données, réorganisez-les pour qu’elles correspondent à la mise en page de feuille Excel nécessaire à l’analyse LFASS. Suivez les instructions détaillées fournies dans la référence30.
REMARQUE: Alors que les données peuvent être analysées manuellement, en normalisant chaque série chronologique et en recherchant le moment où la fluorescence de mort atteint la moitié du maximum, une analyse automatisée peut être effectuée dans Matlab exécutant la routine LFASS30. - Téléchargez et installez Matlab (version 2014a ou supérieure) et le progiciel LFASS de https://github.com/ABA80/LFASS. Suivez les instructions et les annotations qui y sont fournies.
REMARQUE : La figure 1C donne une brève description de l’approche. Matlab est requis pour exécuter la routine LFASS. Alternativement, le code Matlab peut être traduit en Oracle, à l’exception de la fonction d’ajustement, qui est propriétaire. De nouvelles fonctions de lissage et de sigmoïde peuvent être réécrites pour permettre une utilisation dans une plate-forme entièrement open-source. - Entre les analyses LFASS, déplacez les données et les résultats vers un nouvel emplacement, car l’analyse LFASS traitera tous les fichiers du dossier de données et remplacera les fichiers du dossier Résultats.
7. Contrôle des données
- Ouvrez le fichier Excel et étiquetez les lignes en fonction de la position du puits sur la plaque de 384 puits. Le fichier supplémentaire 2 montre un exemple du fichier Excel des données brutes de fluorescence générées pour le test de choc thermique. Utilisez la position du puits sur la plaque de 384 puits pour étiqueter les souches de vers et de bactéries.
- Avant l’analyse Matlab, inspectez visuellement les données dans Excel, en traçant l’intensité de fluorescence au fil du temps pour un puits représentatif. Selon le lecteur de plaques utilisé, les données peuvent être bruyantes mais doivent afficher un pic clair. En particulier:
- Déterminer une valeur de fluorescence en dessous de laquelle un pic ne serait pas significativement différent du bruit (l’établissement d’un tel seuil dans LFASS accélérera l’analyse en excluant les puits vides).
- Notez le premier moment où les fluctuations de fluorescence s’atténuent avant de se lever (les animaux peuvent battre vigoureusement jusqu’à 30 minutes, ce qui entraîne des lectures de fluorescence bleue fluctuantes rapidement).
REMARQUE : Le raccord de crête peut être amélioré en excluant ces premiers points temporels de la fenêtre d’ajustement de la courbe. - Notez les points temporels entre lesquels les valeurs de fluorescence minimale et maximale devraient chuter (examinez plusieurs puits pour identifier ces plages) car ils seront utilisés pour l’ajustement de la courbe.
- Vérifiez si les amplitudes des pics de fluorescence varient de manière significative entre les puits, normalisez les données avant une analyse plus approfondie à l’aide de la formule suivante:
Puits de fluorescence normalisé n (t) = (Puitsde fluorescencen [t] - puits de fluorescence minimale [Dt]) / (puits de fluorescence maximale [Dt] - puits de fluorescence minimale [Dt])
où « n » est le numéro de puits actuel, « t » est le point temporel et « Dt » est la série de points de temps pour l’essai.
8. Traitement des données LFASS
NOTA : Des détails sont fournis au https://github.com/ABA80/LFASS et dans les documents supplémentaires de la référence30.
- Créez deux sous-dossiers dans le dossier LFASS, un pour les données à analyser et un pour les résultats, par exemple, « mes données » et « résultats ».
- Copiez le fichier de données Excel du test dans le sous-dossier LFASS « mes données » après l’inspection des données.
- Lancez MATLAB, accédez au dossier LFASS , tapez et exécutez fitfolder dans la fenêtre de commande (Fichier supplémentaire 3). Suivez ensuite les instructions à l’écran.
- Après avoir tapé « fitfolder », le système demande le nom du dossier dans lequel se trouve le fichier Excel, par exemple, « mes données ». Tapez le nom de votre dossier de données (dans cet exemple, « mes données »).
- Suivez les instructions à l’écran, en fournissant les différents paramètres demandés.
- Entrez « 2 » pour l’intervalle de temps entre les mesures successives dans le protocole actuel (en spécifiant cela, les résultats peuvent être exprimés en minutes au lieu d’unités de point de temps).
REMARQUE: L’intervalle de temps peut être modifié pour effectuer des mesures de fluorescence plus ou moins fréquemment (pour diminuer ou augmenter la résolution temporelle) et également en fonction des capacités du lecteur de plaques (c’est-à-dire que l’intervalle de temps peut devoir être augmenté pour les lecteurs de plaques qui ne peuvent pas effectuer des mesures assez rapides). Assurez-vous de toujours faire correspondre l’intervalle de temps expérimental avec la routine LFASS. - Attribuez le seuil de tolérance supérieur en tapant « 0,95 » (cela peut être modifié au besoin pour améliorer l’ajustement) et le seuil de tolérance inférieur en tapant « 0,05 » (cela peut être modifié au besoin pour améliorer l’ajustement) pour limiter l’ajustement sigmoïde.
REMARQUE : Les autres paramètres de temps sont basés sur les notes de l’utilisateur de l’inspection des données (étape 7.2.).
- Entrez « 2 » pour l’intervalle de temps entre les mesures successives dans le protocole actuel (en spécifiant cela, les résultats peuvent être exprimés en minutes au lieu d’unités de point de temps).
- Choisissez d’afficher ou non les courbes ajustées et lissées en tapant « y » pour OUI ou « n » pour NON lorsque vous y êtes invité. Pour inspecter visuellement les ajustements convergents, sélectionnez le premier.
REMARQUE: Ce dernier est utile pour visualiser toutes les données lissées mais n’est généralement pas sélectionné car il génère trop de graphiques pop-up. Ensuite, Matlab exécutera la routine LFASS, ce qui peut prendre 1 à 10 minutes si plusieurs fichiers Excel sont traités en une seule fois. Des fenêtres contextuelles avec des courbes apparaîtront en fonction de la sélection de l’étape 8.6. Le dossier supplémentaire 4A montre un exemple de courbe ajustée. - Choisissez de (1) analyser les courbes identifiées comme du bruit ou (2) réajuster les courbes mal ajustées avec une option [y/n]. Tapez y pour approuver et n pour rejeter.
REMARQUE: Il est recommandé d’approuver le réaménagement, surtout s’il y a beaucoup de courbes mal ajustées ou non ajustées. Cela permettra à l’utilisateur de fournir des paramètres d’ajustement de courbe adaptés pour chaque courbe telle qu’elle apparaît à l’écran et de ne demander que des limites antérieures et ultérieures pour l’ajustement sigmoïde. Il peut être tenté autant de fois que nécessaire. - Une fois les données analysées, fermez Matlab et ouvrez le dossier LFASS .
- Cliquez sur le sous-dossier LFASS Mes résultats, car les fichiers de résultats sont enregistrés automatiquement dans le dossier Résultats en tant que .txt.
REMARQUE: Matlab génère trois fichiers .txt: « Batch-fitted.txt », « Batch and noise-fitted.txt » et « Refitted.txt ». Les deux premiers sont enregistrés par précaution en cas de panne informatique ou d’erreur de l’utilisateur lors du réaménagement. Le fichier contenant l’analyse complète la plus précise est « Refitted.txt ». - Ouvrez le fichier Réajusté.txt avec Microsoft Excel et enregistrez-le en tant que .xls pour un traitement ultérieur. Le fichier supplémentaire 4B montre un exemple d’un tel fichier de résultats.
REMARQUE : Pour chaque puits (organisé en rangées), trois valeurs sont fournies dans les colonnes qui donnent des estimations du temps médian de mort de la population de vers : « Raw » : indique le temps qui se croise au demi-maximum du pic de données expérimentales; « Batch-fitted »: indique le temps d’intersection au demi-maximum de la courbe ajustée par lot; « Réaménagé »: indique le temps de croisement au demi-maximum de la courbe réaménagée. - Enregistrez le fichier au format .xls en tant que copie dans un emplacement sûr. Si vous ne le faites pas, les fichiers risquent d’être écrasés lors de la prochaine exécution de la routine LFASS.
REMARQUE: Les résultats peuvent ensuite être traités ultérieurement pour une analyse graphique ou statistique.
Access restricted. Please log in or start a trial to view this content.
Résultats
Les tests LFASS fournissent un criblage robuste, à haut débit et rapide de plusieurs conditions de test à la fois, telles que le criblage de nombreux paramètres génétiques et microbiotiques qui contribuent à la résistance au stress et au vieillissement. Il ne faut que 2 à 3 semaines à l’expérience pour acquérir un vaste ensemble de données de plusieurs conditions de test. Les populations adultes de vers de type sauvage L4 + 36 h ont été exposées à un stress thermique de 42 °C et à un stress ...
Access restricted. Please log in or start a trial to view this content.
Discussion
C. elegans offre de nombreux avantages pour le criblage rapide de plusieurs paramètres expérimentaux à la fois, en raison de sa petite taille, de sa transparence, de son développement rapide, de sa courte durée de vie, de son faible coût et de sa facilité de manipulation. Son génome, son plan corporel, son système nerveux, son intestin et son microbiome considérablement plus simples, mais suffisamment complexes et similaires à ceux des humains, en font un modèle préclinique puissant, où des informa...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions la CCG Minnesota (Madison, États-Unis, NIH - P40 OD010440) pour avoir fourni des souches de vers et OP50 et le Pr. Hinrich Schulenburg (CAU, Kiel, Allemagne) pour avoir fourni tous les isolats microbiens environnementaux représentés ici. Ce travail a été financé par une subvention UKRI-BBSRC à AB (BB/S017127/1). JM est financé par une bourse de doctorat FHM de l’Université de Lancaster.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
Références
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood - Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977(2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer's disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98(2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson's disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921(2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095(2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323(2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364(2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545(2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849(2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998(2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613(2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019(2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer's disease: a systematic review. Nutrients. 14 (1), 20(2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson's disease. Ageing Research Reviews. 69, 101347(2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -E. H., Jung, Y., Lee, S. -J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190(2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652(2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583(2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398(2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448(2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35(2016).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon