Method Article
Tri négatif des neurones par noyaux activés par fluorescence combiné au séquençage de l’ARN à noyau unique pour étudier la niche neurogène de l’hippocampe
Dans cet article
Résumé
Présenté ici est une méthode pour séquencer des noyaux uniques isolés du gyrus denté de la souris qui exclut la plupart des neurones par tri des noyaux activés par fluorescence (FAN). Cette approche génère des profils d’expression de haute qualité et facilite l’étude de la plupart des autres types de cellules représentées dans la niche, y compris les populations rares telles que les cellules souches neurales.
Résumé
La neurogenèse hippocampique adulte (AHN), qui consiste en un maintien à vie des cellules souches neurales (CSN) prolifératives et quiescentes dans la zone sous-granulaire (SGZ) du gyrus denté (DG) et leur différenciation des neurones nouvellement nés en cellules granulaires dans la couche cellulaire granulaire, est bien validée par de nombreuses études. L’utilisation d’animaux génétiquement modifiés, en particulier de rongeurs, est un outil précieux pour étudier les voies de signalisation régulant l’AHN et pour étudier le rôle de chaque type de cellule qui compose la niche neurogène de l’hippocampe. Pour remédier à ce dernier problème, les méthodes combinant l’isolement d’un seul noyau et le séquençage de nouvelle génération ont eu un impact significatif dans le domaine de l’AHN pour identifier les signatures génétiques de chaque population cellulaire. Un perfectionnement supplémentaire de ces techniques est toutefois nécessaire pour établir le profil phénotypique des populations de cellules plus rares au sein du DG. Ici, nous présentons une méthode qui utilise le tri des noyaux activés par fluorescence (FANS) pour exclure la plupart des populations neuronales d’une suspension de noyaux unique isolée de DG fraîchement disséquée, en sélectionnant des noyaux non colorés pour l’antigène NeuN, afin d’effectuer le séquençage de l’ARN à noyau unique (snRNA-seq). Cette méthode est un tremplin potentiel pour étudier plus avant la régulation intercellulaire de l’AHN et pour découvrir de nouveaux marqueurs et mécanismes cellulaires à travers les espèces.
Introduction
La génération continue de neurones de l’hippocampe à l’âge adulte, également connue sous le nom de neurogenèse hippocampique adulte (AHN), est associée à des fonctions cognitives telles que l’apprentissage, l’acquisition et la clairance de la mémoire et la séparation des modèles et semble être un mécanisme important de résilience dans le vieillissement et les maladies neurodégénératives pour prévenir les déficits cognitifs 1,2,3 . Les rongeurs ont été le modèle de choix pour étudier l’AHN en utilisant plusieurs méthodes, y compris l’immunocytochimie et les méthodes de séquençage de nouvelle génération (NGS). La traduction de ces résultats à d’autres espèces reste controversée. En effet, le AHN a été observé chez la plupart des espèces mais la mesure dans laquelle il persiste tout au long de la vie, en particulier chez l’homme 4,5,6,7,8, est régulièrement débattue.
À ce jour, diverses voies de signalisation intrinsèques et extrinsèques ont été confirmées pour moduler AHN1. Cependant, l’impact de la communication intercellulaire sur l’AHN n’en est qu’à ses débuts9. Cela pourrait d’abord être attribué à la spécificité insuffisante des marqueurs cellulaires actuellement connus pour effectuer des analyses in vivo avec des animaux génétiquement modifiés. En effet, de nombreuses études se sont appuyées sur des marqueurs tels que la doublecortine ou la protéine acide fibrillaire gliale (GFAP) qui sont exprimés dans plusieurs types cellulaires1. Deuxièmement, la complexité et le degré élevé de diversité cellulaire dans la niche hippocampique adulte10 apportent des défis techniques pour profiler chaque type de cellule. C’est particulièrement le cas pour l’analyse bioinformatique avec des marqueurs cellulaires qui se chevauchent utilisés dans les pipelines analytiques pour différentes populations, telles que les CSN ou les cellules gliales, ce qui entraîne des conclusions controversées lors de l’évaluation de l’AHN 7,11. Troisièmement, le grand nombre de neurones nuit à l’étude de populations cellulaires moins abondantes, telles que les astrocytes, les oligodendrocytes ou les cellules épendymaires, même si leur rôle dans la régulation fine de l’AHN devient important9. Ensemble, ces limitations ont une incidence sur la capacité de traduire les résultats des rongeurs vers d’autres espèces. Ceci est particulièrement amplifié par la difficulté de récapituler in vitro un tissu complexe, tel que la niche neurogène hippocampique, et par les nombreux obstacles à l’accès à des tissus de haute qualité ainsi que par l’absence de protocoles standardisés pour le traitement des tissus dans les études impliquant des tissus humains12,13. Il est donc essentiel de développer de nouvelles approches pour profiler les populations cellulaires et identifier de nouveaux marqueurs cellulaires dans le gyrus denté (DG) qui conduiront finalement à une meilleure compréhension des différentes contributions de chaque type de cellule à la régulation de l’AHN.
Pour ce faire, l’isolement d’une cellule unique (sc) et d’un seul noyau (sn) combiné au séquençage de l’ARN est devenu essentiel pour étudier des tissus complexes tels que le DG14. En tant que tel, des stratégies d’enrichissement cellulaire pour isoler des cellules individuelles de la niche hippocampique adulte de souris ont été réalisées principalement pour examiner les CSN15,16. Une stratégie intéressante pour enrichir les cellules non neuronales du DG a été appliquée en séquençant des cellules simples doublement négatives GluR1 / Cd24 qui ont abouti à 1 408 cellules séquencées sans grappes distinctes entre les astrocytes et les CSN après analyse bioinformatique17. Cela pourrait être dû à la digestion enzymatique sévère requise pour la préparation d’une seule cellule qui endommage l’intégrité cellulaire et l’ARN. Pour contourner ce problème technique, plusieurs méthodes utilisant l’isolement d’un seul noyau ont été développées et sont particulièrement adaptées aux tissus complexes11,18. Cependant, la prédominance des neurones au sein du DG ou plus largement au sein du système hippocampique-entorhinal génère un biais d’échantillonnage pour étudier l’ensemble des populations cellulaires présentes dans ces zones cérébrales. En outre, le nombre limité de cellules à charger pour la préparation de bibliothèques de cellules uniques accentue la présence de la population cellulaire majeure dans les pipelines analytiques de noyaux uniques séquencés. En effet, les grands amas neuronaux sont souvent annotés et analysés alors que d’autres populations cellulaires sont sous-représentées ou manquées 5,11.
Dans une tentative de surmonter ces biais et de pouvoir profiler des types de cellules autres que les neurones présents dans le DG de la souris, une méthode a été conçue dans cette étude en utilisant le principe de tri des noyaux activés par fluorescence (FANS)18 qui exclut la plupart des populations neuronales par sélection négative de noyaux uniques colorés avec antigène nucléaire neuronal (NeuN, également connu sous le nom de Rbfox3). Ce choix d’antigène a été guidé par la littérature décrivant NeuN comme un marqueur neuronal fiable19 et par la nécessité d’utiliser une protéine nucléaire pour cette approche. Des cellules triées par des NEN négatifs au FACS ont ensuite été préparées pour le séquençage de l’ARN sur une plateforme génomique 10x. Les résultats démontrent que l’exclusion des cellules exprimant les NeuN permet un profilage transcriptomique de haute qualité spécifique au type cellulaire des populations de cellules gliales et rares.
Protocole
Les soins aux animaux et les procédures expérimentales ont été effectués conformément aux directives du Francis Crick Institute, ainsi qu’aux directives et aux lois du ministère de l’Intérieur du Royaume-Uni.
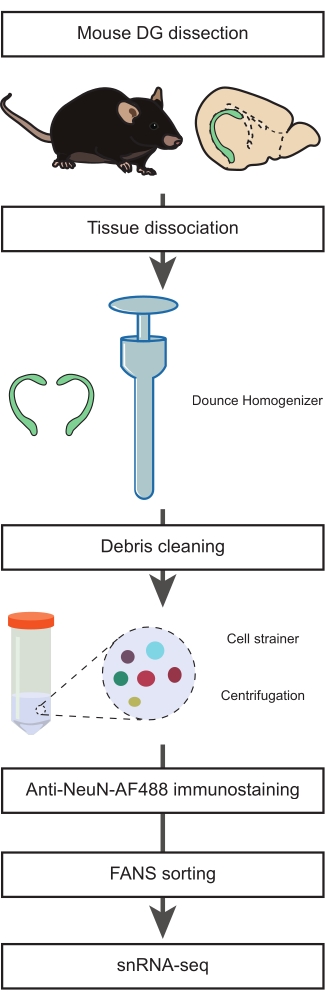
Figure 1 : Préparation d’une suspension de noyaux uniques à partir du DG disséqué de souris adultes pour le séquençage de l’ARNn de populations non neuronales. Organigramme décrivant les principales étapes du protocole qui comprennent la dissection de la DG de souris, la préparation d’une suspension de noyaux simples, l’immunomarquage NeuN et le tri négatif NeuN-FANS avant de procéder au snRNA-seq. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Dissection du DG (Durée : 15 min)
- Préparer les milieux d’isolement des noyaux 1 et 2 (NIM1 et NIM2), le tampon d’homogénéisation (HB) et le milieu de lavage (WM) (tableau supplémentaire 1). Placez tous les tampons, les milieux, les réactifs et les outils sur la glace jusqu’à ce que vous en ayez besoin. Mettez l’homogénéisateur Dounce (voir le tableau des matériaux) sur la glace pendant la préparation (minimum 1 h avant l’étape d’homogénéisation).
REMARQUE: NIM1 peut être préparé et conservé à 4 ° C jusqu’à 6 mois. NIM2, HB et WM doivent être fraîchement préparés.
ATTENTION : Manipulez le DTT, l’inhibiteur de protéase et le Triton X-100 avec précaution. Ces composés sont irritants pour la peau et les yeux, extrêmement toxiques et dangereux pour le milieu aquatique. Lorsque vous utilisez ces produits chimiques, portez des gants de protection, des vêtements, une protection des yeux et du visage, lavez-vous soigneusement les mains après les avoir manipulés et évitez tout rejet dans l’environnement. - Euthanasier une souris C57Bl/6J mâle de 22 mois par luxation cervicale en suivant la procédure20 de l’annexe 1 du ministère de l’Intérieur.
REMARQUE : Voir la discussion pour la justification de l’utilisation de souris de 22 mois dans cette étude. Cependant, ce protocole peut être effectué à tout âge tout au long de la vie. - Disséquez le cerveau d’une souris euthanasiée et transférez-le dans une boîte de Petri de 10 cm remplie de 1x PBS glacé (Figure 1). Placez la boîte de Petri sur de la glace. Retirez le cervelet à l’aide d’un scalpel et coupez le cerveau en deux entre les deux hémisphères (le long de l’axe sagittal).
- Remplissez une nouvelle boîte de Petri de 10 cm avec du PBS glacé et placez-la sur de la glace. Transférer la moitié du cerveau dans la nouvelle boîte de Pétri. À l’aide de jumelles, disséquez le DG et répétez cette étape pour obtenir le deuxième DG de la seconde moitié du cerveau.
NOTE: Ces étapes (étapes 1.2-1.4) ont été adaptées d’une procédure décrite précédemment21. Il est important de procéder le plus rapidement possible à ce stade afin de préserver l’intégrité des cellules. - Transférer les deux DG dans l’homogénéisateur Dounce prérefroidi et ajouter 1 mL de HB froid.
2. Dissociation tissulaire, isolement d’un seul noyau et immunomarquage anti-NeuN (Chronométrage : 2 h)
- Homogénéiser le tissu avec 10 coups du pilon « A » lâche, suivis de 15 coups du pilon « B » serré.
REMARQUE: L’homogénéisation des rebonds doit être effectuée avec le mortier sur glace avec des mouvements doux pour réduire la chaleur causée par la friction et le moussage. Tous les tampons et l’équipement doivent être prérefroidis et conservés sur la glace pendant la procédure. - Transférer l’homogénat dans un tube prérefroidi de 15 mL; rincer l’homogénéisateur Dounce avec 1 mL de HB froid et combiner dans le même tube. Ajouter 3 mL de HB dans le tube de 15 mL et incuber 5 min sur de la glace. Mélanger 2x en retournant doucement le tube.
- Prémouiller un bouchon de crépine de 70 μm avec 0,5 mL de HB sur un tube à essai de 50 mL. Filtrer la suspension de noyaux de l’étape 2.2 en basculant doucement le tube de 15 mL dans la crépine cellulaire. Lavez la crépine cellulaire avec 0,5 mL de HB.
- Retirer la crépine cellulaire et centrifuger le tube à essai à 500 x g pendant 5 min à 4 °C, à l’aide d’une centrifugeuse à godet pivotant. Jetez le surnageant.
REMARQUE: Forcer l’homogénat aidera à réduire les débris, ce qui est essentiel pour la cytométrie de flux et les étapes en aval du séquençage de l’ARNn. - Remettez doucement la pastille en suspension dans 4 mL de HB à l’aide d’une pipette P1000. Incuber sur la glace pendant 5 min. Faire tourner à 500 x g pendant 10 min à 4 °C. Jeter le surnageant et remettre en suspension la pastille dans 3 mL de WM.
- Prémouiller un capuchon de crépine de 35 μm sur un tube à essai de 15 mL avec 0,5 mL de WM. Filtrer la suspension de noyaux de l’étape 2.5 à travers la crépine cellulaire, en pipetant doucement 0,5 mL à la fois à l’aide d’une pipette P1000.
- Laver le capuchon de la crépine avec 0,5 mL de WM et placer le tube sur de la glace. Transférer le filtrat dans un nouveau tube de 15 mL et centrifuger pendant 5 min et 4 °C à 500 x g. Jeter le surnageant et remettre en suspension la pastille dans 3 mL de WM.
- Faire tourner à 500 x g pendant 5 min à 4 °C. Jeter le surnageant et remettre la pastille en suspension dans 1 mL de WM avec l’anticorps conjugué anti-NeuN de souris, Alexa Fluor 488 (anti-NeuN-AF488, 1:32 000) et 1 μg/mL DAPI. Incuber pendant 45 min sur de la glace dans l’obscurité.
NOTE: Pour optimiser l’immunomarquage des noyaux isolés, il est recommandé de titrer l’anticorps afin de déterminer la dilution optimale pour l’analyse et le tri par cytométrie de flux. Ensuite, exécutez des contrôles adéquats pour confirmer que les conditions de coloration sont optimales. Par exemple, avec l’anticorps conjugué anti-NeuN-AF488, un témoin négatif (c.-à-d. aucun ajout de l’anticorps, figure supplémentaire 1A) et un témoin positif (c.-à-d. coloration avec l’anticorps, figure supplémentaire 1B) ont été exécutés pour évaluer la ségrégation des populations non colorées et colorées. Lorsque vous commencez à travailler avec un anticorps conjugué AF488, il est recommandé d’exécuter un contrôle d’isotype conjugué AF488 pour évaluer la spécificité. Si un anticorps non conjugué est utilisé, un contrôle supplémentaire tel que l’ajout d’un anticorps secondaire uniquement à une préparation de noyaux peut être nécessaire pour évaluer la liaison non spécifique de l’anticorps secondaire.
3. Tri des noyaux activés par fluorescence (FANS) pour exclure les populations neuronales (Durée : 45 min)
- Transférer la suspension de noyaux immunocolorés dans un tube à essai de 5 mL et la garder sur la glace jusqu’au début de la procédure de cytométrie en flux.
NOTE: Si vous travaillez avec des morceaux de tissu plus gros que deux DG de souris, une dilution supplémentaire avec le tampon WM peut être nécessaire pour éviter de colmater le FACS si la densité des noyaux dans la solution devient élevée. - Vortex des échantillons pendant 3 s à basse vitesse avant de placer les tubes dans l’instrument FACS (voir le tableau des matériaux).
REMARQUE: (configuration FACS) Les machines de tri doivent être alignées au début de la procédure avec les particules d’étalonnage suivant les recommandations du fabricant. Le délai de chute a été calibré avec des billes ou des microsphères (voir Tableau des matériaux) selon le modèle FACS. Les échantillons ont été triés à 4 °C en mode pureté. Pour réduire le volume de collecte, les noyaux ont été triés à travers une buse de 70 μm à la pression recommandée pour le cytomètre en flux. Les noyaux ont été triés dans des tubes à faible liaison de 1,5 mL (voir le tableau des matériaux) contenant 50 μL de WM. Tous les tubes collecteurs ont été recouverts de PBS + 5 % de BSA à 4 °C pendant une nuit afin de réduire le risque que les noyaux adhèrent aux parois du tube. - Pour obtenir les données d’un échantillon de la suspension de noyaux colorés, placez les portes en hauteur DAPI et en zone DAPI afin d’exclure les débris cellulaires et les noyaux agrégés (Figure 2A). De plus, séparer les noyaux simples de tout agrégat ou débris cellulaire coloré par le DAPI restant en plaçant les portes dans la zone de diffusion latérale logarithmique (SSC) et dans la zone de diffusion vers l’avant (FCS) (figure 2B).
- Placez les portes de l’anti-NeuN-AF488 et de la zone FSC, afin d’isoler la population négative NeuN-AF488, comme le montre la figure 2C.
- Après analyse, à l’aide de la stratégie de déclenchement décrite ci-dessus, trier la population négative NeuN-AF488 dans un tube de collecte de 1,5 mL rempli de 50 μL de WM.
REMARQUE : En suivant la stratégie de contrôle décrite ci-dessus et la procédure de dissection visant à isoler le DG du cerveau d’une souris adulte, la population négative NeuN-AF488 devrait représenter ~14 % des noyaux individuels.
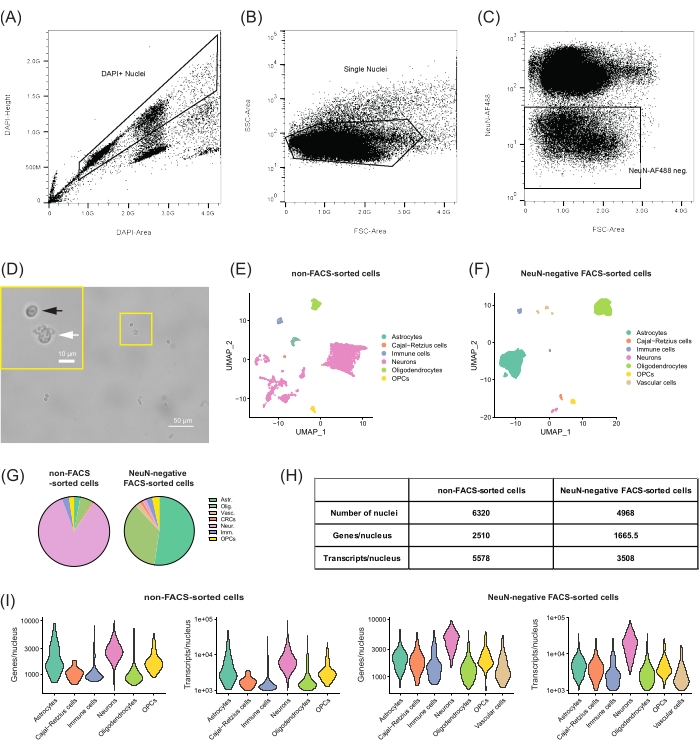
Figure 2 : Isolement et profilage transcriptomique de populations de cellules non neuronales à partir de la stratégie de déclenchement DG. (A-C) pour isoler les noyaux uniques négatifs NeuN-AF488 et exclure les débris cellulaires. (A) Diagramme à points FANS d’un échantillon représentatif de noyaux isolés, représentant le réglage de la porte pour la sélection des noyaux DAPI+ et l’exclusion des débris et agrégats cellulaires. B) Sélection plus poussée des noyaux uniques pertinents en utilisant la zone FSC et la zone SSC. (C) Les portes pour NeuN-AF488 pour exclure la population positive et trier les noyaux uniques négatifs. (D) Micrographie d’une bonne suspension de noyaux simples avec une quantité minimale de débris et une proportion plus élevée de noyaux de bonne qualité (forme ronde, flèche noire) par rapport aux noyaux de mauvaise qualité (flèche blanche). Barres d’échelle = 50 μm, 10 μm (encadré). (E,F) Analyse des données de séquençage de l’ARNn et profilage des populations cellulaires distinctes isolées à partir du DG de souris mâles C57BL/6J âgées de 22 mois. Uniform Manifold Approximation and Projection for Dimension Reduction (UMAP) diagrammes de profils de noyaux simples à partir des cellules (E) non triées FACS et (F) des cellules triées par FACS négatifs NeuN, colorées par type de cellule. (G) Diagrammes circulaires comparant les fréquences des types de cellules identifiés dans les deux échantillons. (H) Mesures respectives pour les échantillons séquencés : nombre de noyaux, nombre médian de gènes et transcrits par noyau. (I) Tracés de violon montrant la distribution du nombre de gènes et de transcrits détectés pour chaque type de cellule dans les deux échantillons. Astr. = astrocytes, Olig. = oligodendrocytes, Vasc. = cellules vasculaires, CRC = cellules de Cajal-Retzius, Neur. = neurones, Imm. = cellules immunitaires, OPC = cellules précurseurs d’oligodendrocytes . Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Préparation de la suspension mononucléiforme pour effectuer le séquençage de l’ARN mononucléien (Chronométrage : 30 min)
- Après le tri, ajouter 1 mL de PBS contenant 1% de BSA dans le tube collecteur afin de recueillir les gouttelettes sur la paroi du tube et faire tourner à 500 x g pendant 5 min à 4 °C. Jeter le surnageant, en laissant 50 μL.
REMARQUE: Manipulez les noyaux centrifugés avec précaution car il peut être difficile d’observer une pastille au fond du tube. L’utilisation d’une centrifugeuse à godet oscillant aidera à jeter le surnageant sans perturber la pastille. - Pipeter doucement pour remettre en suspension les noyaux centrifugés. Ajouter 5 μL de la suspension de noyaux à 5 μL de bleu de trypan dans un microtube de 0,5 mL.
ATTENTION: Manipulez le bleu de trypan avec précaution car il est dangereux pour la santé, peut causer le cancer et est soupçonné de nuire à la fertilité ou à l’enfant à naître. Portez des gants de protection, des vêtements et une protection des yeux et du visage. Ne pas manipuler tant que toutes les mesures de sécurité n’ont pas été lues et comprises. - Mesurer la concentration et évaluer la viabilité de la suspension unicellulaire à l’aide d’un hémocytomètre ou d’un compteur de cellules automatisé (voir le tableau des matériaux). Effectuez la préparation de la bibliothèque et le séquençage des noyaux comme détaillé à l’étape 5.
REMARQUE : Les échantillons jugés de bonne qualité pour le séquençage présentaient des noyaux ronds et réguliers au microscope, sans débris cellulaires (figure 2D). La présence d’un halo autour de la membrane nucléaire ou l’agrégation de plusieurs noyaux ensemble sont des signes de noyaux endommagés et de telles suspensions cellulaires ne devraient pas être envisagées pour le séquençage de l’ARNn (Figure 2D). La concentration mesurée des noyaux se situait entre 300 et 700 noyaux/μL.
5. Préparation et séquençage de la bibliothèque
REMARQUE : La description des étapes suivantes est basée sur la plateforme de séquençage interne utilisée dans cette étude (voir le tableau des matériaux). Par conséquent, certains paramètres peuvent différer lors de l’utilisation d’une plate-forme différente. Ici, seules les étapes clés sont décrites et chaque paramètre doit être déterminé en suivant les directives et les protocoles du fabricant choisi, mais avec une optimisation avant la première utilisation. Il est essentiel de s’assurer que la préparation des banques est effectuée le plus rapidement possible après la concentration des suspensions de noyaux triés afin d’éviter la dégradation de l’ARN et d’assurer une qualité optimale du séquençage.
- Chargez entre 7 000 et 10 000 noyaux dans une puce microfluidique unicellulaire.
- Partitionner les noyaux chargés dans des gouttelettes à l’échelle nanolitre à l’aide du contrôleur fourni et des réactifs du fournisseur choisi. Noyaux de lyse dans chaque gouttelette et transcrire l’ARN inverse.
REMARQUE: Dans une gouttelette, tout l’ADNc résultant partageait le même code-barres cellulaire. - Préparer les bibliothèques pour le snRNA-seq en suivant les directives du fournisseur choisi et en assurant la compatibilité avec la plate-forme de séquençage. Vérifier la qualité et la concentration des bibliothèques finales à l’aide de méthodes basées sur l’électrophorèse, la fluorométrie ou la qPCR et, le cas échéant, les regrouper de manière équimolarisée avant le séquençage.
- Denature a regroupé des bibliothèques d’expression génique 3' et les a diluées conformément aux recommandations du fabricant.
- Effectuez un séquençage d’indexation par paires, simple ou double sur une plateforme de séquençage de nouvelle génération avec une profondeur de séquençage de 50 000 paires de lecture par cellule.
Résultats
Le protocole présenté ici décrit une méthode pour préparer une suspension de noyaux uniques non neuronaux isolés du DG pour effectuer un snRNA-seq. Avec ou sans FANS, le regroupement bioinformatique a révélé des groupes de noyaux bien séparés correspondant à des types cellulaires connus au sein du DG (Figure 2E,F). Dans l’échantillon non trié par FACS, la majorité des noyaux de haute qualité qui ont été séquencés comprenaient trois groupes de neurones (84,9% du total des noyaux pour cet échantillon, Figure 2E, G, H). De tels résultats sont attendus, étant donné que les populations cellulaires les plus représentées dans le DG sont les neurones granulaires, les autres neurones excitateurs (neurones excitateurs marqués) et les neurones inhibiteurs10. Les amas non neuronaux identifiés étaient principalement constitués de types de cellules gliales (11,1%), y compris les astrocytes, les oligodendrocytes et les cellules précurseurs des oligodendrocytes (OPC), les cellules immunitaires (3,3%) et les cellules de Cajal-Retzius (0,6%). Lors de l’exécution de FANS pour exclure les populations NeuN positives (échantillon trié par le FACS négatif aux NeuN; Figure 2F, G, H), les amas de cellules gliales sont devenus prédominants (81,3%). L’isolement d’un plus grand nombre de noyaux gliaux permet une meilleure segmentation des différentes populations qui se regrouperaient sans FANS. En effet, lors du regroupement et de l’analyse de gènes spécifiques exprimés dans les CSN ou dans les astrocytes, quatre sous-groupes se sont séparés (Figure supplémentaire 2A,B). En examinant des marqueurs cellulaires plus spécifiques et en évaluant les niveaux d’expression génique dans les types cellulaires, un petit groupe de CSN a été détecté séparément des principales populations astrocytaires avec une expression plus élevée de Hopx et Notch2 et presque aucune expression d’Aldh1a1 ou Aqp4 (Figure supplémentaire 2C). Cependant, en raison du chevauchement de l’expression génique entre les astrocytes et les CSN, une analyse plus approfondie serait nécessaire pour profiler et identifier spécifiquement différents sous-types de cellules. De plus, l’échantillon FANS négatif aux NénN présentait des grappes supplémentaires marquées comme cellules vasculaires (2,3%) qui englobent les cellules endothéliales, les péricytes et les cellules leptoméningées vasculaires lorsqu’elles sont croisées pour l’expression de marqueurs spécifiques aux cellules (données non présentées).
Suivant les instructions du protocole choisi pour générer des bibliothèques pour le séquençage, des profils d’expression de haute qualité ont été obtenus avec ou sans FANS. Pour les échantillons séquencés à 50 000 lectures/noyau, 2 510 gènes ont été détectés en moyenne par noyau pour l’échantillon non trié par FACS (5 578 transcrits de la figure 2H) et 1 665,5 gènes (3 508 transcrits pour l’échantillon FANS négatif aux NEN), après filtrage des noyaux de faible qualité (Figure 2H,I). Ces mesures confirment que ce protocole génère un profilage transcriptomique de haute qualité des noyaux uniques comparable aux études utilisant différentes approches22,23 et que le processus de tri FACS n’endommage pas les noyaux pour le séquençage ultérieur de l’ARNn. Notamment, la différence dans le nombre de gènes et de transcrits par noyau entre les deux échantillons n’est pas due à une qualité de données inférieure, mais à la proportion élevée de neurones dans l’échantillon non trié par FACS (84,9% contre 1,7% dans l’échantillon FANS négatif NeuN), qui ont une activité transcriptionnelle plus élevée (2 660 gènes/noyau et 6 170 transcrits / noyau dans un échantillon non trié par FACS) que l’activité transcriptionnelle moyenne de tous les types de cellules non neuronales (1 090 gènes/noyau et 1 785 transcriptions/noyau, figure 2I).
Ensemble, ces résultats représentatifs montrent que la sélection de noyaux négatifs NeuN à l’aide de FANS est un outil puissant pour isoler des types de cellules de faible abondance à partir de tissus cérébraux fraîchement disséqués et effectuer un profilage transcriptomique de haute qualité de ces populations cellulaires distinctes via des méthodes de séquençage de l’ARNn.
Figure supplémentaire 1 : Validation de l’immunocoloration pour FANS. La suspension de noyaux a été incubée (A) sans l’anticorps anti-NeuN-AF 488 en tant que témoin négatif ou (B) avec l’anticorps et passée par le trieur FACS pour valider les conditions d’immunomarquage. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Analyse de l’expression génique et regroupement de l’amas d’astrocytes. (A) Diagramme UMAP (Uniform Manifold Approximation and Projection for Dimension Reduction) montrant le regroupement de 4968 noyaux sur la base des profils d’expression à l’échelle du génome de la figure 2F. Les appels de type cellulaire ont été effectués en fonction de marqueurs de type cellulaire. (B) Amas d’astrocytes composé de 2579 noyaux choisis parmi (A) pour un sous-ensemble supplémentaire afin d’étudier les sous-types cellulaires potentiels. Quatre sous-types ont été détectés par regroupement de Seurat (0-3), représentés par différentes couleurs. (C) Niveaux d’expression génique de marqueurs cellulaires spécifiques dans les quatre types de cellules. Toutes les placettes ont été obtenues à l’aide du package SeuratR 24. Brièvement, les comptes de séquençage de l’ARN ont été normalisés pour chaque cellule par l’expression totale et multipliés par le facteur d’échelle (10 000). Ce résultat a ensuite été transformé en log. Les valeurs transformées ont été mises à l’échelle (variance mise à l’échelle à un) et centrées (moyenne fixée à zéro) dans chaque cellule avant que UMAP ne soit appliqué pour calculer les intégrations, qui ont été utilisées comme valeurs sur les axes x et y. Les graphiques représentent le résultat d’une technique de réduction dimensionnelle sur un nuage de points 2D où chaque point représente une cellule avec les coordonnées x et y respectives basées sur les intégrations de cellules déterminées par la technique de réduction. Les cellules ayant des signatures génétiques similaires sont positionnées à proximité les unes des autres par les encastrements. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3 : Analyse de l’expression génique de NeuN dans la lignée neurogène. (A) Graphique UMAP montrant le regroupement de la lignée neurogène à partir de l’ensemble de données accessible au public15. Les UMAP ont été générés comme dans la figure supplémentaire 2. (B) Niveaux d’expression génique de marqueurs cellulaires spécifiques à travers la lignée neurogène montrant Astrocyte (Aquaporin 4 = Aqp4), NSCs (Homeodomain-only protein = Hopx), NeuN/Rbfox3 (NSC et cellules progénitrices intermédiaires [IPC]) et cellules cycliques (kinase cycline-dépendante 6 = Cdk6). Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire 1 : Compositions des milieux et des tampons utilisés dans l’étude. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Pour exécuter avec succès ce protocole, la dissection du DG est la première étape critique, qui nécessite une certaine pratique pour le garder intact et limiter la contamination par les tissus environnants. Par expérience, la séparation du DG de l’hippocampe pourrait être acquise très rapidement par un chercheur qualifié qui pourrait alors travailler à affiner sa technique pour augmenter la rapidité de dissection et donc améliorer la fraîcheur du tissu pour générer des données de haute qualité. Dans le même ordre d’idées, la préparation et la remise en suspension de noyaux uniques exigent une cohérence entre les différentes conditions utilisées dans une seule expérience, mais aussi l’évitement d’un pipetage excessif qui pourrait perturber la libération de la membrane nucléaire d’ARN ambiants qui biaiserait les résultats du séquençage. En plus des recommandations mentionnées précédemment pour préparer des noyaux de haute qualité, la concentration de la suspension de noyaux simples doit également être prise en compte avant de procéder au séquençage. En effet, selon les directives du fabricant, une préparation avec une concentration supérieure à 1 200 nuc/μL doit être diluée, car ce niveau de concentration de noyaux aura un risque plus élevé de formation de multiplets ayant un impact sur les analyses bioinformatiques en aval. Il convient de noter que le séquençage des échantillons avec des concentrations de noyaux inférieures à 500 nuc/μL pourrait ne pas valoir la peine en raison du coût impliqué. Il est également recommandé de suivre les conseils d’un utilisateur avancé du FACS pour mettre en place tous les points de contrôle et rester cohérent avec les paramètres entre les échantillons et les répétitions biologiques. De même, la préparation des bibliothèques pour le séquençage de l’ARN implique une certaine formation pour obtenir des résultats de haute qualité et la plupart des fournisseurs disposent d’un excellent soutien pour y parvenir efficacement. Cette méthode n’a été testée qu’avec des tissus frais dans cette étude; cependant, FANS a également été réalisé avec des tissus congelés25. Il est donc raisonnable de supposer que ce protocole pourrait être effectué avec des tissus congelés, bien qu’avec une optimisation mineure.
Ce protocole a été développé avec une application particulière en aval à l’esprit, qui est d’étudier les populations cellulaires autres que les neurones dans la niche neurogène de l’hippocampe. En effet, de plus en plus de sources de données indiquent que la déficience de l’AHN au cours du vieillissement pourrait être attribuée aux cellules environnantes dans le créneau 1,2,3,9. En particulier, les astrocytes et les oligodendrocytes apparaissent comme des régulateurs clés de l’AHN; cependant, leur isolement du DG couplé au séquençage de l’ARN a généré des résultats mitigés, ce qui rend cette hypothèse difficile à évaluer avec cette technique 1,17. Cette approche de tri FACS des noyaux NeuN-négatifs a permis d’isoler plus d’astrocytes et d’oligodendrocytes par rapport aux échantillons qui n’ont pas été triés par FACS, ce qui permet une meilleure analyse bioinformatique. Ce protocole est applicable à tous les âges de la vie et les données représentatives présentées ici avec des tissus d’animaux âgés fournissent une preuve de concept que cette méthode est robuste pour étudier la niche neurogène de l’hippocampe vieillissant. Pour étendre l’utilisation de cette méthode et l’adapter à différentes questions biologiques, il est important de considérer que d’autres antigènes de membrane nucléaire neuronale pourraient être testés avec un titrage approfondi des meilleurs anticorps validés pour ces marqueurs. Par exemple, lors de l’étude du processus de différenciation neuronale des CSN dans le DG, certains types de cellules telles que les cellules de type 2 ou les neuroblastes commencent à exprimer NeuN (Figure supplémentaire 3). Par conséquent, un autre antigène serait nécessaire pour étudier spécifiquement ces types de cellules. Inversement, certains neurones ont encore été identifiés dans cette étude après tri FACS négatif NeuN, peut-être en raison d’une expression faible ou nulle de NeuN dans ces populations (par exemple, les neurones corticaux Cajal-Retzius19). De plus, il a été rapporté que NeuN était exprimé dans des sous-populations d’oligodendrocytes26, ce qui pourrait donner des résultats biaisés si ces sous-populations étaient intéressantes. Ainsi, le choix de l’antigène lors du début de l’utilisation de FANS doit être soigneusement examiné afin d’éviter l’inclusion ou l’exclusion de populations cellulaires qui empêcheraient une réponse précise à une question biologique spécifique. En accord avec cela, il est également recommandé que chaque résultat de séquençage soit validé par des tests orthogonaux (par exemple, immunohistochimie ou portée ARN) avant de valider ou de réfuter l’hypothèse testée avec ce protocole. Enfin, l’étape impliquant FANS pourrait être développée davantage pour inclure plus d’un anticorps avec une stratégie de tri plus élaborée pour exclure et / ou inclure les populations cellulaires souhaitées.
En fin de compte, les technologies décrites dans ce protocole pourraient avoir certaines limites lorsqu’elles sont utilisées avec d’autres espèces. Par exemple, la niche est très bien définie chez les rongeurs avec la présence de CSN prolifératifs et quiescents ou de neurones nouveau-nés restreints dans des sous-régions spécifiques de la DG, mais on ne sait toujours pas comment la niche neurogène de l’hippocampe devrait être délimitée chez d’autres espèces. En effet, les cellules prolifératives ne sont pas alignées dans une zone continue du DG chez les primates non humains et les humains mais sont plutôt dispersées autour de celui-ci et pourraient également être présentes dans l’amygdale7. Par conséquent, la dissection et l’isolement de zones plus vastes que le DG chez d’autres espèces pourraient avoir une incidence sur l’utilisation de ce protocole. En particulier, les étapes de dissociation et de trituration pour la préparation des tissus devront être optimisées lors du travail avec de plus gros morceaux de tissu27,28. En ce qui concerne l’analyse bioinformatique, bien que les rongeurs consanguins aient un génome très homogène et très bien annoté, la variabilité génétique du génome humain combinée à un nombre insuffisant de marqueurs cellulaires pour distinguer clairement les différentes populations cellulaires (par exemple, les CSN et les astrocytes) nécessite beaucoup de normalisation pour l’analyse qui pourrait conduire à des conclusions différentes lorsqu’un petit groupe de cellules est identifié7, 11. Dans de telles situations, l’enrichissement cellulaire pourrait encore être une option préférée ou devrait être utilisé parallèlement à d’autres stratégies pour augmenter la puissance analytique.
Néanmoins, l’approche actuelle peut permettre d’étudier le rôle de populations cellulaires sous-étudiées mais potentiellement importantes dans la régulation de l’AHN. Cela pourrait notamment être le cas pour les populations d’astrocytes, qui jouent un rôle central dans l’apparition et la progression des maladies neurodégénératives29,30. Cette étude a démontré que les astrocytes et autres populations de cellules rares peuvent être identifiés et profilés simplement en excluant la grande majorité des neurones présents dans le DG. D’autres études utilisant des approches différentes n’ont pas été en mesure d’obtenir une récupération similaire des noyaux de la même gamme de populations cellulaires 5,11,17. De plus, les résultats de cette étude démontrent qu’il est possible avec cette approche d’isoler un cluster NSC sans enrichissement spécifique de cette population cellulaire15.
En conclusion, suivre et améliorer cette méthode serait un pas en avant pour répondre aux questions en suspens liées au rôle contextuel de la niche neurogène hippocampique pour la modulation de l’AHN. En particulier, il pourrait apporter de nouvelles informations sur les niveaux d’expression génique dans les cerveaux âgés et malades dans les populations cellulaires associées à la régulation de l’AHN9, soutenir l’identification d’une hétérogénéité potentielle des CSN1 ou aborder le rôle du système vasculaire dans l’AHN. En fin de compte, cette méthode pourrait être adaptée à d’autres niches de cellules souches adultes ayant des questions et des problèmes similaires.
Déclarations de divulgation
SG, TL et SK sont des employés de Merck Sharp & Dohme LLC, une filiale de Merck & Co., Inc., Rahway, NJ, États-Unis, connue sous le nom de MSD en dehors des États-Unis et du Canada. SG est actionnaire de Merck & Co., Inc., Rahway, NJ, USA.
Remerciements
Les auteurs tiennent à remercier Lachlan Harris et Piero Rigo pour leur soutien technique, ainsi que Jason M. Uslaner et Ditte Lovatt pour leurs commentaires sur le manuscrit. Ce travail a été soutenu par une subvention du MRC et une collaboration de recherche précompétitive avec MSD, le Francis Crick Institute, qui reçoit son financement de Cancer Research UK (FC0010089), du UK Medical Research Council (FC0010089), du Wellcome Trust (FC0010089) et par une bourse de chercheur du Wellcome Trust à FG (106187/Z/14/Z). Nous nous excusons auprès des nombreux auteurs dont nous n’avons pas pu discuter et citer le travail par manque de place.
matériels
| Name | Company | Catalog Number | Comments |
| 0.5ml microtube | Eppendorf | 30124537 | |
| 10.00µm Flouresbrite YG Carboxylate Microspheres | Polysciences | 15700-10 | |
| 15 mL polypropylene centrifuge tubes | Corning | 430052 | |
| 2 pairs of sterile Dumont #5 forceps | Fine Science Tools | 11252-30 | |
| 4′,6-diamidino-2-phenylindole (DAPI) | Sigma Aldrich | D9564-10MG | |
| 4150 TapeStation System | Agilent | N/A | |
| 5 mL round bottom high clarity polypropylene test tube with snap cap | Falcon | 352063 | |
| 5 mL round bottom polystyrene test tube with cell strainer snap cap | Falcon | 352235 | |
| 50 mL polypropylene centrifuge tubes | Corning | 430829 | |
| 70 µm cell strainer | Falcon | 352350 | |
| 8 peak SPHERO Rainbow Calibration Particles | BD Biosciences | RCP-30-5A | |
| Accudrop Beads | BD Biosciences | N/A | |
| Allegra X-30R Centrifuge | Beckman Coulter | N/A | |
| Anti-NeuN antibody, clone A60, Alexa Fluor 488 conjugated | Millipore | MAB377X | |
| BD FACSAria Fusion Flow Cytometer | BD Biosciences | N/A | |
| Beckman Coulter MoFlo XDP | Beckman Coulter | N/A | |
| Chromium Controller | 10x Genomics | N/A | |
| Chromium Next GEM Single Cell 3' Reagent Kits v3.1 | 10x Genomics | PN-1000121; PN-1000120; PN-1000213 | |
| BSA 7.5% | Gibco | 15260037 | |
| Dithiothreitol (DTT) | Thermo Scientific | R0861 | |
| Dounce tissue grinder set: mortar, loose pestle (A) and tight pestle (B) | KIMBLE | D8938-1SET | |
| Eppendorf Tubes Protein LoBind 1.5ml | Eppendorf | 30108116 | |
| Halt, 100x Protease inhibitor | ThermoFisher | 78429 | |
| HiSeq 4000 Sequencing System | Illumina | N/A | Sequencing configuration: 28-8-0-91 |
| KCl | Any chemical supplier | Laboratory made | |
| LUNA-FX7 Automated Cell counter | Logos Biosystems | N/A | |
| MgCl2 | Any chemical supplier | Laboratory made | |
| N°10 guarded sterile disposable scalpels | Swann-Morton | 6601 | |
| Nuclease-free water | Sigma Aldrich | W4502-1L | |
| Pair of sterile student surgical scissors | Fine Science Tools | 91401-12 | |
| PBS | Any chemical supplier | Laboratory made | |
| RNase Inhibitor 40 U µl-1 | Ambion | AM2684 | |
| RNasin 40 U µl-1 | Promega | N211A | |
| Sterile Petri dish | Corning | 430167 | |
| Sucrose | Sigma Aldrich | 59378-500G | |
| Tris buffer, pH 8.0 | Any chemical supplier | Laboratory made | |
| Triton X-100 10% (v/v) | Sigma Aldrich | T8787-250ML | |
| Trypan blue | Invitrogen | T10282 |
Références
- Gillotin, S. Targeting impaired adult hippocampal neurogenesis in ageing leveraging intrinsic mechanisms regulating neural stem cell activity. Ageing Research Reviews. 71, 101447 (2021).
- Urban, N., Blomfield, I. M., Guillemot, F. Quiescence of adult mammalian neural stem cells: A highly regulated rest. Neuron. 104 (5), 834-848 (2019).
- Hanspal, M. A., Gillotin, S. A new age in understanding adult hippocampal neurogenesis in Alzheimer's disease. Neural Regeneration Research. 17 (12), 2615-2618 (2022).
- Zhang, H., et al. Single-nucleus transcriptomic landscape of primate hippocampal aging. Protein & Cell. 12 (9), 695-716 (2021).
- Franjic, D., et al. Transcriptomic taxonomy and neurogenic trajectories of adult human, macaque, and pig hippocampal and entorhinal cells. Neuron. 110 (3), 452-469 (2022).
- Moreno-Jimenez, E. P., Terreros-Roncal, J., Flor-Garcia, M., Rabano, A., Llorens-Martin, M. Evidences for adult hippocampal neurogenesis in humans. Journal of Neuroscience. 41 (12), 2541-2553 (2021).
- Sorrells, S. F., et al. Positive controls in adults and children support that very few, if any, new neurons are born in the adult human hippocampus. Journal of Neuroscience. 41 (12), 2554-2565 (2021).
- Zhou, Y., et al. Molecular landscapes of human hippocampal immature neurons across lifespan. Nature. 607, 527-533 (2022).
- Bonafina, A., Paratcha, G., Ledda, F. Deciphering new players in the neurogenic adult hippocampal niche. Frontiers in Cell and Developmental Biology. 8, 548 (2020).
- Amaral, D. G., Scharfman, H. E., Lavenex, P. The dentate gyrus: fundamental neuroanatomical organization (dentate gyrus for dummies). Progress in Brain Research. 163, 3-22 (2007).
- Habib, N., et al. Massively parallel single-nucleus RNA-seq with DroNc-seq. Nature Methods. 14 (10), 955-958 (2017).
- Flor-Garcia, M., et al. Unraveling human adult hippocampal neurogenesis. Nature Protocols. 15 (2), 668-693 (2020).
- Moreno-Jimenez, E. P., et al. Adult hippocampal neurogenesis is abundant in neurologically healthy subjects and drops sharply in patients with Alzheimer's disease. Nature Medicine. 25 (4), 554-560 (2019).
- Kalinina, A., Lagace, D. Single-cell and single-nucleus RNAseq analysis of adult neurogenesis. Cells. 11 (10), 1633 (2022).
- Harris, L., et al. Coordinated changes in cellular behavior ensure the lifelong maintenance of the hippocampal stem cell population. Cell Stem Cell. 28 (5), 863-876 (2021).
- Shin, J., et al. Single-cell RNA-seq with Waterfall reveals molecular cascades underlying adult neurogenesis. Cell Stem Cell. 17 (3), 360-372 (2015).
- Artegiani, B., et al. A single-cell RNA sequencing study reveals cellular and molecular dynamics of the hippocampal neurogenic niche. Cell Reports. 21 (11), 3271-3284 (2017).
- Nott, A., Schlachetzki, J. C. M., Fixsen, B. R., Glass, C. K. Nuclei isolation of multiple brain cell types for omics interrogation. Nature Protocols. 16 (3), 1629-1646 (2021).
- Sarnat, H. B., Nochlin, D., Born, D. E. Neuronal nuclear antigen (NeuN): a marker of neuronal maturation in early human fetal nervous system. Brain Development. 20 (2), 88-94 (1998).
- . Guidance on the Operation of ASPA Available from: https://assets.publishing.service.gov.uk/government/uploads/system/uploads/attachment_dat/file/662364/Guidance_on_the_Operation_of_ASPA.pdf (2022)
- Hagihara, H., Toyama, K., Yamasaki, N., Miyakawa, T. Dissection of hippocampal dentate gyrus from adult mouse. Journal of Visualized Experiments. (33), e1543 (2009).
- Habib, N., et al. Disease-associated astrocytes in Alzheimer's disease and aging. Nature Neuroscience. 23 (6), 701-706 (2020).
- Ding, J., et al. Systematic comparison of single-cell and single-nucleus RNA-sequencing methods. Nature Biotechnology. 38 (6), 737-746 (2020).
- Hao, Y., et al. Integrated analysis of multimodal single-cell data. Cell. 184 (13), 3573-3587 (2021).
- Mussa, Z., Tome-Garcia, J., Jiang, Y., Akbarian, S., Tsankova, N. M. Isolation of adult human astrocyte populations from fresh-frozen cortex using fluorescence-activated nuclei sorting. Journal of Visualized Experiments. (170), e62405 (2021).
- Zhang, Y., et al. An RNA-sequencing transcriptome and splicing database of glia, neurons, and vascular cells of the cerebral cortex. Journal of Neuroscience. 34 (36), 11929-11947 (2014).
- Marti-Mengual, U., Varea, E., Crespo, C., Blasco-Ibanez, J. M., Nacher, J. Cells expressing markers of immature neurons in the amygdala of adult humans. European Journal of Neuroscience. 37 (1), 10-22 (2013).
- Zhang, X. M., et al. Doublecortin-expressing cells persist in the associative cerebral cortex and amygdala in aged nonhuman primates. Frontiers in Neuroanatomy. 3, 17 (2009).
- Ding, Z. B., et al. Astrocytes: a double-edged sword in neurodegenerative diseases. Neural Regeneration Research. 16 (9), 1702-1710 (2021).
- Phatnani, H., Maniatis, T. Astrocytes in neurodegenerative disease. Cold Spring Harbor Perspectives in Biology. 7 (6), 020628 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon