Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Caractérisation d’une souche pathogène d’Escherichia coli dérivée d’Oreochromis spp. Fermes utilisant le séquençage du génome entier
Dans cet article
Résumé
La faisabilité de stratégies de séquençage du génome entier (SGE) à l’aide d’instruments de paillasse a simplifié l’interrogation du génome de chaque microbe pertinent pour la santé publique en laboratoire. Une adaptation méthodologique du flux de travail pour le séquençage bactérien est décrite et un pipeline bioinformatique pour l’analyse est également présenté.
Résumé
L’aquaculture est l’un des secteurs de production alimentaire à la croissance la plus rapide au monde et l’élevage du tilapia (Oreochromis spp.) constitue la principale variété de poissons d’eau douce cultivée. Étant donné que les pratiques aquacoles sont sensibles à la contamination microbienne provenant de sources anthropiques, l’utilisation intensive d’antibiotiques est nécessaire, ce qui fait que les systèmes aquacoles deviennent une source importante de bactéries résistantes aux antibiotiques et pathogènes d’importance clinique, telles que Escherichia coli (E. coli). Ici, la résistance aux antimicrobiens, la virulence et les caractéristiques de mobilome d’une souche pathogène d’E. coli , récupérée chez Oreochromis spp. d’élevage intérieur, ont été élucidées par séquençage du génome entier (WGS) et analyse in silico . Des tests de sensibilité aux antimicrobiens (AST) et un séquençage pangénomique ont été effectués. De plus, le groupe phylogénétique, le sérotype, le typage de séquences multilocus (MLST), la résistance acquise aux antimicrobiens, la virulence, le plasmide et le contenu en prophages ont été déterminés à l’aide de divers outils Web disponibles. L’isolat d’E. coli ne présentait qu’une sensibilité intermédiaire à l’ampicilline et a été caractérisé comme étant la souche ONT:H21-B1-ST40 par typage à base de WGS. Bien qu’un seul gène lié à la résistance aux antimicrobiens ait été détecté [mdf(A)], plusieurs gènes associés à la virulence (VAG) du pathotype atypique E. coli entéropathogène (aEPEC) ont été identifiés . De plus, la cargaison de réplicons plasmidiques provenant de grands groupes plasmidiques et de 18 régions associées aux prophages a été détectée. En conclusion, la caractérisation WGS d’un isolat d’aEPEC, récupéré dans une ferme piscicole à Sinaloa, au Mexique, permet de mieux comprendre son potentiel pathogène et le risque possible pour la santé humaine de la consommation de produits aquacoles crus. Il est nécessaire d’exploiter les techniques de séquençage de nouvelle génération (NGS) pour étudier les micro-organismes environnementaux et d’adopter un cadre « Une seule santé » pour apprendre comment les problèmes de santé apparaissent.
Introduction
L’aquaculture est l’un des secteurs de production alimentaire à la croissance la plus rapide au monde, et ses pratiques de production visent à satisfaire la demande alimentaire croissante pour la consommation humaine. La production aquacole mondiale a triplé, passant de 34 millions de tonnes (Mt) en 1997 à 112 Mt en 20171. Les principaux groupes d’espèces, contribuant à près de 75% de la production, étaient les algues, les carpes, les bivalves, les poissons-chats et les tilapias (Oreochromis spp.) 1. Cependant, l’apparition de maladies causées par des entités microbiennes est inévitable en raison de la pisciculture intensive, entraînant des pertes économiques potentielles2.
L’utilisation d’antibiotiques dans les pratiques piscicoles est bien connue pour prévenir et traiter les infections bactériennes, principal facteur limitant la productivité 3,4. Néanmoins, les antibiotiques résiduels s’accumulent dans les sédiments et l’eau de l’aquaculture, exerçant une pression sélective et modifiant les communautés bactériennes associées aux poissons et résidentes 5,6,7,8. Par conséquent, l’environnement aquacole sert de réservoir pour les gènes de résistance aux antimicrobiens (ARG) et la poursuite de l’émergence et de la propagation de bactéries résistantes aux antibiotiques (ARA) dans le milieu environnant9. En plus des bactéries pathogènes couramment observées affectant les pratiques piscicoles, des membres de la famille des Enterobacteriaceae sont souvent rencontrés, y compris des souches d’agents pathogènes humains d’Enterobacter spp., Escherichia coli, Klebsiella spp. et Salmonella spp.10. E. coli est le microorganisme le plus commun isolé de la farine de poisson et de l’eau dans la pisciculture 11,12,13,14,15.
E. coli est une bactérie à Gram négatif polyvalente qui habite le tractus gastro-intestinal des mammifères et des oiseaux en tant que membre commensale de leur microbiote intestinal. Cependant, E. coli possède une capacité d’adaptation élevée pour coloniser et persister dans différentes niches environnementales, y compris le sol, les sédiments, les aliments et l’eau16. En raison du gain et de la perte de gènes par le phénomène de transfert horizontal de gènes (HGT), E. coli a rapidement évolué pour devenir un agent pathogène résistant aux antibiotiques bien adapté, capable de causer un large éventail de maladies chez les humains et les animaux17,18. Sur la base de l’origine de l’isolement, les variantes pathogènes sont définies comme E. coli pathogène intestinal (InPEC) ou E. coli pathogène extra-intestinal (ExPEC). De plus, l’InPEC et l’ExPEC sont subdivisés en pathotypes bien définis en fonction de la manifestation de la maladie, du bagage génétique, des traits phénotypiques et des facteurs de virulence (VF)16,17,19.
La culture traditionnelle et les techniques moléculaires pour les souches pathogènes d’E. coli ont permis la détection et l’identification rapides de différents pathotypes. Cependant, ils peuvent être longs, laborieux et nécessiter souvent une formation technique élevée19. De plus, aucune méthode unique ne peut être utilisée pour étudier de manière fiable toutes les variantes pathogènes d’E. coli en raison de la complexité de leur bagage génétique. Actuellement, ces inconvénients ont été surmontés avec l’avènement des technologies de séquençage à haut débit (HTS). Les approches de séquençage du génome entier (SGE) et les outils bioinformatiques ont amélioré l’exploration de l’ADN microbien à un coût abordable et à grande échelle, facilitant la caractérisation approfondie des microbes en une seule fois, y compris les variantes pathogènes étroitement apparentées20,21,22. Selon les questions biologiques, plusieurs outils bioinformatiques, algorithmes et bases de données peuvent être utilisés pour effectuer l’analyse des données. Par exemple, si l’objectif principal est d’évaluer la présence d’ARG, de VF et de plasmides, des outils tels que ResFinder, VirulenceFinder et PlasmidFinder, ainsi que leurs bases de données associées, peuvent être un bon point de départ. Carriço et coll.22 ont donné un aperçu détaillé des différents logiciels bioinformatiques et des bases de données connexes appliqués à l’analyse microbienne du séquençage pangénomique, du prétraitement des données brutes à l’inférence phylogénétique.
Plusieurs études ont démontré la grande utilité du séquençage pangénomique pour l’interrogation du génome concernant les attributs de résistance aux antimicrobiens, le potentiel pathogène et le suivi de l’émergence et des relations évolutives de variantes cliniquement pertinentes d’E. coli provenant de diverses origines23,24,25,26 . Le séquençage pangénomique a permis d’identifier les mécanismes moléculaires sous-jacents à la résistance phénotypique aux antimicrobiens, y compris les mécanismes de résistance rares ou complexes. Cela se fait en détectant des variantes ARG acquises, de nouvelles mutations dans les gènes cibles du médicament ou des régions promotrices27,28. De plus, le séquençage pangénomique offre la possibilité d’inférer des profils de résistance aux antimicrobiens sans nécessiter de connaissances préalables sur le phénotype de résistance d’une souche bactérienne29. Alternativement, le séquençage pangénomique a permis la caractérisation des éléments génétiques mobiles (MGE) porteurs à la fois de résistance aux antimicrobiens et de virulence, ce qui a entraîné l’évolution du génome bactérien des agents pathogènes existants. Par exemple, l’application du séquençage pangénomique lors de l’enquête sur l’éclosion d’E. coli en Allemagne en 2011 a permis de découvrir les caractéristiques génomiques uniques d’un pathotype apparemment nouveau d’E. coli; Il est intéressant de noter que ces souches d’éclosion provenaient du groupe E. coli entéroaggrégatif (EAEC), qui a acquis le prophage codant pour la toxine Shiga à partir du pathotype30 d’E. coli entérohémorragique (EHEC).
Ce travail présente une adaptation méthodologique du flux de travail pour le séquenceur bactérien à l’aide d’un séquenceur de paillasse. De plus, un pipeline bioinformatique est fourni à l’aide d’outils Web pour analyser les séquences résultantes et soutenir davantage les chercheurs ayant peu ou pas d’expertise en bioinformatique. Les méthodes décrites ont permis d’élucider la résistance aux antimicrobiens, la virulence et les caractéristiques de mobilome d’une souche pathogène d’E. coli ACM5, isolée en 2011 chez Oreochromis spp. d’élevage intérieur à Sinaloa, au Mexique12.
Protocole
REMARQUE : La souche ACM5 d’E. coli a été récupérée par traitement et culture de l’échantillon de poisson pour la détermination des coliformes fécaux (FC)12. Au cours de l’échantillonnage des poissons, les poissons n’ont pas présenté de signes cliniques de maladie, d’infection bactérienne ou fongique, et une température moyenne de 22,3 °C a prévalu. Après isolement, l’isolat d’E. coli a été soumis à des tests biochimiques et cryoconservé dans un bouillon de perfusion cœur cérébral (BHI) avec du DMSO (8% v/v) comme agent cryoprotecteur.
1. Réactivation de la culture mère congelée d’E. coli ACM5
- À partir du stock de bactéries congelées, ouvrez le tube et utilisez une boucle stérile, une pointe de pipette ou un cure-dent pour gratter la surface de la culture bactérienne congelée.
- Étaler les bactéries sur une plaque de gélose Luria-Bertani (LB) et incuber à 37 ± 2 °C pendant 24 h.
2. Détermination de la sensibilité aux antibactériens
REMARQUE : Les tests de sensibilité aux antimicrobiens décrits ici correspondent à la méthode de diffusion sur disque basée sur les lignes directrices du Clinical and Laboratory Standards Institute (CLSI) (M02 Ed13:2018)31. E. coli La souche ATCC 25922 est requise à des fins de contrôle de la qualité.
- Préparation de l’inoculum par la méthode de suspension de colonie
- À partir de la plaque LB incubée à l’étape 1.2, étaler une ou deux colonies sur une plaque de gélose Mueller-Hinton (MH) et incuber à 37 ± 2 °C pendant 18 à 24 h.
- Utilisez une boucle stérile pour prélever deux ou trois colonies de l’isolat d’E. coli fraîchement sous-cultivé sur une plaque de gélose MH et remettez-les en suspension dans 3 ml de bouillon MH stérile ou de solution saline à 0,85 % (p/v), en mélangeant soigneusement par vortex pour obtenir une suspension bactérienne uniforme.
- À l’aide d’un spectrophotomètre UV-visible (voir le tableau des matériaux), ajuster la suspension bactérienne à une turbidité comparable à une norme MacFarland de 0,5 (équivalant à environ 1-2 x 108 cellules/mL). Si la suspension bactérienne est trop légère ou trop lourde, ajouter plus de colonies d’E. coli ou de bouillon MH, selon le cas. Utilisez l’inoculum préparé dans les 15 minutes suivant la préparation.
- Inoculation des plaques de gélose d’essai
- Trempez un coton-tige stérile dans la suspension bactérienne ajustée. Faites pivoter l’écouvillon plusieurs fois et appuyez fermement sur la paroi intérieure du tube pour éliminer l’excès de liquide de l’écouvillon.
- Inoculer une plaque de gélose MH en striant l’écouvillon 3x sur toute la surface de la gélose, en faisant pivoter la plaque de 60° à chaque fois. Frottez également le bord de la gélose pour assurer une répartition uniforme de l’inoculum.
- Laissez le couvercle de la boîte de Petri entrouverte pendant 3 à 5 minutes pour permettre à l’humidité de s’évaporer.
- Application du disque antimicrobien sur des plaques de gélose inoculée
- Répartir uniformément et presser chaque disque antimicrobien (voir le tableau des matières) sur la surface des plaques de gélose inoculée pour assurer un contact complet. Retourner les plaques dans les 15 minutes suivant l’application du disque et incuber à 35 ± 2 °C pendant 16-18 h.
NOTE: Un maximum de 12 et six disques doit être utilisé dans les boîtes de Petri de 150 mm et 100 mm, respectivement.
- Répartir uniformément et presser chaque disque antimicrobien (voir le tableau des matières) sur la surface des plaques de gélose inoculée pour assurer un contact complet. Retourner les plaques dans les 15 minutes suivant l’application du disque et incuber à 35 ± 2 °C pendant 16-18 h.
- Après incubation, mesurer la zone d’inhibition à l’aide d’un pied à coulisse Vernier et interpréter les diamètres obtenus selon les critères du point de rupture CLSI (M100 - Tableau 2A)32.
3. Extraction et quantification de l’ADN génomique (ADNg)
- Extraction d’ADN génomique
- Resuspendre une boucle de colonies d’E. coli fraîchement cultivées dans 5 ml de bouillon LB et incuber pendant la nuit dans un incubateur à agitation (180 rpm) à 37 ± 2 °C.
- Centrifuger la suspension bactérienne à 3 500 x g pendant 5 min et jeter soigneusement le surnageant.
- Extraire l’ADNg en suivant les lignes directrices de la trousse d’extraction d’ADN (voir le tableau des matériaux).
- Vérifiez la pureté de l’ADNg en mesurant la densité optique à 260/280 nm (rapport: >1,8) et 260/230 nm (rapport: 2,0-2,2) à l’aide d’un spectrophotomètre UV-visible. Effectuer une électrophorèse sur gel d’agarose à 0,8% pour vérifier l’intégrité de l’ADNg.
- Quantification de l’ADN génomique
REMARQUE : N’utilisez que des tubes PCR transparents à paroi mince de 0,5 mL approuvés par le fabricant de la trousse d’essai de fluorescence (voir le tableau des matériaux).- Établir le nombre requis de tubes pour les échantillons et les étalons de dosage. Préparer la solution d’essai de travail en mélangeant le composant A et le composant B fournis dans la trousse, en suivant les directives du fabricant.
- Préparer les étalons de dosage en ajoutant 190 μL de la solution de travail et 10 μL de chaque étalon au tube approprié (deux étalons sont requis).
- Ajouter 198 μL de la solution de travail et 2 μL de l’échantillon d’ADN dans le tube approprié. Mélanger vigoureusement en faisant tourbillonner tous les tubes pendant 5 s. Attention à ne pas créer de bulles.
- Incuber tous les tubes pendant 2 min à température ambiante, à l’abri de la lumière. Mesurer la fluorescence de tous les tubes en fonction des directives du fabricant à l’aide du fluoromètre (voir le tableau des matériaux).
- Ajustez correctement la concentration de l’échantillon d’ADNg pour le séquençage.
4. Préparation de la banque d’ADN
REMARQUE : La préparation et le séquençage de la banque d’ADN ont été effectués conformément aux lignes directrices et aux protocoles du fabricant (voir le tableau des matériaux). La concentration initiale d’ADNg est de 4,0 ng.
- Tagmentation, amplification PCR et indexation
- Dans un tube de 0,2 mL, ajouter 2,5 μL de tampon de marquage et 2 μL d’ADNg d’entrée (2,0 ng/μL). Mélanger doucement par pipetage.
- Ajouter 1 μL de tampon d’amplification et mélanger délicatement par pipetage. Faire tourner vers le bas à 280 x g pendant 1 min à température ambiante.
- Placez les échantillons dans un thermocycleur (voir le tableau des matériaux) et exécutez le programme de PCR suivant : 55 °C pendant 5 min, puis maintenir à 10 °C. Lorsque les échantillons atteignent 10 °C, procéder immédiatement à la neutralisation de la réaction.
- Ajouter 1 μL de tampon neutralisant dans les tubes à échantillon et mélanger délicatement par pipetage. Faire tourner à 280 x g pendant 1 min et incuber pendant 5 min à température ambiante.
- Ajouter 1,7 μL de chaque adaptateur d’index (c.-à-d. indices i7 et i5) et 3 μL de mélange-maître PCR d’indexation. Mélanger doucement par pipetage. Faire tourner vers le bas à 280 x g pendant 1 min à température ambiante.
- Placer les échantillons dans le thermocycleur et effectuer une deuxième réaction de PCR comme suit: 72 °C pendant 3 min, 95 °C pendant 30 s, 18 cycles de 95 °C pendant 10 s, 55 °C pendant 30 s, 72 °C pendant 30 s, 72 °C pendant 5 min et maintenir à 10 °C.
- Nettoyage amplifié de la bibliothèque
- Mélanger en vortex la solution commerciale de billes magnétiques (voir le tableau des matériaux) en fonction des lignes directrices du fabricant.
- Transférer l’échantillon d’ADNg marqué/indexé dans un nouveau tube de 1,5 mL et ajouter 0,6 μL de billes magnétiques pour chaque μL du volume final de l’échantillon d’ADNg (≈13 μL). Mélanger délicatement par pipetage et incuber pendant 5 min à température ambiante.
- Placer les tubes à échantillon sur une grille magnétique (voir le tableau des matériaux) pendant 2 minutes jusqu’à ce que le surnageant soit éliminé. Retirez et jetez soigneusement le surnageant sans déranger les perles.
- Ajouter 200 μL d’éthanol à 80 % fraîchement préparé sans mélanger. Incuber pendant 30 s jusqu’à ce que l’éluat se dissipe et retirer et jeter soigneusement le surnageant sans déranger les perles.
- Effectuez une deuxième étape de lavage. Ajouter 200 μL d’éthanol à 80% aux billes et incuber pendant 30 s. Une fois l’éluat effacé, retirer et jeter le surnageant et sécher les perles à l’air libre pendant 10 min.
- Ajouter 15 μL de tampon tris 10 mM (pH 8) aux billes et mélanger délicatement par pipetage. Incuber pendant 2 min à température ambiante. Replacez les tubes d’échantillon sur le support magnétique pendant 2 min, en permettant au surnageant de s’effacer.
- Transférer délicatement 14 μL du surnageant du tube à échantillon vers un nouveau tube de 0,2 mL. C’est la bibliothèque nettoyée.
REMARQUE : À ce stade, la bibliothèque nettoyée peut être stockée à -20 °C pendant 7 jours maximum.
- Normalisation de la bibliothèque
- Procéder à la quantification des bibliothèques nettoyées par le test de fluorescence décrit à l’étape 3.2, à l’exception de l’étape 3.2.5.
- Visualisez les bibliothèques nettoyées sur une électrophorèse sur gel d’agarose à 1 % pour déterminer la taille moyenne des fragments.
- Calculez la valeur de molarité de la bibliothèque à l’aide de l’équation suivante :
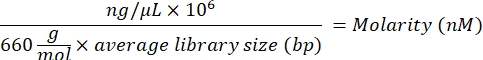
- À l’aide de la valeur de molarité, calculer les volumes appropriés de tampon de remise en suspension (RSB) et de bibliothèques nettoyées pour diluer la bibliothèque individuelle à une concentration initiale de 10 nM.
5. Mise en commun des bibliothèques, dénaturalisation et lancement du séquenceur
- Décongeler la cartouche de réactif en suivant les instructions du fabricant. Extraire la cellule d’écoulement du réfrigérateur (4 °C) et la porter à température ambiante avant le séquençage.
- Regrouper 5 μL de chaque bibliothèque normalisée (10 nM) dans un tube de 1,5 mL à faible liaison. Diluer la bibliothèque mise en commun à 4 nM avec le volume approprié de RSB.
- Ajouter 5 μL de la bibliothèque groupée (4 nM) et 5 μL de NaOH 0,2 N dans un nouveau tube de 1,5 mL à faible liaison. Mélanger brièvement par tourbillon, faire tourner vers le bas à 280 x g pendant 1 min et incuber pendant 5 min à température ambiante pour dénaturer la bibliothèque mise en commun en brins simples.
- Mélanger délicatement 10 μL de bibliothèque dénaturée et 990 μL de tampon d’hybridation prérefroidi par pipetage et placer sur de la glace jusqu’à ce que la dilution finale pour le chargement du séquenceur soit effectuée. La concentration de la bibliothèque commune dénaturée est de 20 pM.
- Décongeler et préparer une bibliothèque de contrôle (voir le tableau des matériaux) à une concentration de 4 nM en mélangeant 2 μL de bibliothèque de contrôle (10 nM) et 3 μL d’eau exempte de nucléase.
- Mélanger 5 μL de la bibliothèque de contrôle (4 nM) et 5 μL de NaOH 0,2 N fraîchement préparé. Mélanger brièvement par tourbillon, faire tourner vers le bas à 280 x g pendant 1 min et incuber pendant 5 min à température ambiante pour dénaturer la bibliothèque de contrôle en brins simples.
- Mélanger délicatement 10 μL de bibliothèque de contrôle dénaturée et 990 μL de tampon d’hybridation prérefroidi par pipetage et placer sur glace jusqu’à ce que la dilution finale pour le chargement du séquenceur soit terminée. La concentration de la bibliothèque de contrôle dénaturée est de 20 pM.
- Dans un nouveau tube à faible liaison de 1,5 mL, combiner 594 μL de la bibliothèque groupée dénaturée à 20 pM et 6 μL de la bibliothèque de contrôle dénaturée à 20 pM. Mélangez correctement.
- Diluer le mélange final de la bibliothèque (20 pM) à une concentration de charge finale de 1,2 pM dans un volume de 600 μL à l’aide du tampon d’hybridation prérefroidi.
- Avant de charger le mélange final de la bibliothèque sur la cartouche de réactif, effectuez un prétraitement thermique supplémentaire pour obtenir un chargement efficace dans la cellule d’écoulement. Incuber le mélange final de la bibliothèque pendant 2 min à 96 °C. Retourner le tube pour mélanger et placer le tube sur de la glace pendant 5 min.
- Charger 500 μL du mélange final de la bibliothèque dans le réservoir conçu sur la cartouche de réactif. Lancez le séquençage en suivant les directives. Chargez la cellule d’écoulement et la cartouche de réactif et configurez l’exécution du séquençage.
6. Analyse des données de séquence
REMARQUE : Consultez le fichier supplémentaire 1 pour obtenir une description plus détaillée du prétraitement général des données du SGE, du logiciel, des paramètres et de l’analyse des séquences du génome d’E. coli.
- Connectez-vous au serveur de données de séquençage et téléchargez les fichiers FASTQ.
- Évaluez la qualité initiale des données de séquence brutes avec un logiciel tiers. Supprimez les séquences d’adaptateurs résiduelles, les bases de faible qualité (Fichier supplémentaire 1).
- Assemblez les données de séquençage dont la qualité a été vérifiée au niveau du contig ou de l’échafaudage à l’aide d’un logiciel tiers (voir le fichier supplémentaire 1).
- Effectuer l’annotation du génome en soumettant le fichier FASTA contenant le génome assemblé au serveur RAST (https://rast.nmpdr.org/).
- Téléchargez le fichier FASTA sur les plateformes Web Center for Genome Epidemiology (CGE) (http://www.genomicepidemiology.org/services/) et ClermonTyping (http://clermontyping.iame-research.center/index.php) pour identifier les caractéristiques épidémiologiques, les ARG, les VAG et les plasmides (voir le dossier supplémentaire 1).
- Téléchargez le fichier FASTA sur le serveur PHASTER pour identifier les séquences de prophages (https://phaster.ca/).
Résultats
La sensibilité aux antimicrobiens a été déterminée par la méthode de diffusion du disque et interprétée par les critères de seuil CLSI pour 12 antibiotiques couvrant six classes antimicrobiennes distinctes, c’est-à-dire les aminoglycosides, les β-lactamines, les fluoroquinolones, les nitrofuranes, les phénicols et les antagonistes des voies folates. L’ACM5 d’E. coli a montré une sensibilité à tous les antibiotiques, sauf un médicament β-lactamines. Quatre médicaments à base de β-lactami...
Discussion
Cette étude présente une adaptation du flux de travail du séquenceur bactérien WGS à l’aide d’un séquenceur de paillasse et d’un pipeline pour la caractérisation génomique d’une variante pathogène d’E. coli. Selon la plateforme de séquençage utilisée, les délais d’exécution (TATs) pour les procédures de laboratoire humide (culture bactérienne, extraction d’ADNg, préparation de la bibliothèque et séquençage) et l’analyse de séquence peuvent varier, en particulier si des bacté...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Au Conseil national de la science et de la technologie du Mexique (CONACyT par son acronyme en espagnol) pour la bourse de doctorat attribuée à José Antonio Magaña-Lizárraga [n ° 481143].
matériels
| Name | Company | Catalog Number | Comments |
| Accublock Mini digital dry bath | Labnet | D0100 | Dry bath for incubation of tubes |
| Agencourt AMPure XP | Beckman Coulter | A63881 | Magnetic beads in solution for DNA library purification |
| DeNovix DS-11 | DeNovix Inc. | UV-Vis spectophotometer to check the quality of the gDNA extracted | |
| DNA LoBind Tubes | Eppendorf | 0030108418 | 1.5 mL PCR tubes for DNA library pooling |
| DynaMag-2 Magnet | Invitrogen, Thermo Fisher Scientific | 12321D | Magnetic microtube rack used during magnetic beads-based DNA purification |
| Gram-negative Multibac I.D. | Diagnostic reseach (Mexico) | PT-35 | Commercial standard antibiotic disks for antimicrobial susceptibility testing |
| MiniSeq Mid Output Kit (300-cycles) | Illumina | FC-420-1004 | Reagent cartdrige for paired-end sequencing (2x150) |
| MiniSeq System Instrument | Illumina | SY-420-1001 | Benchtop sequencer used for Next-generation sequencing |
| MiniSpin centrifuge | Eppendorf | 5452000816 | Standard centrifuge for tubes |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1024 | Reagents to perform DNA libraries for sequencing. Includes Box 1 and Box 2 reagents for 24 samples |
| Nextera XT Index Kit v2 | Illumina | FC-131-2001, FC-131-2002, FC-131-2003, FC-131-2004 | Index set A, B, C, D |
| PhiX Control v3 | Illumina | FC-110-3001 | DNA library control for sequencing |
| Precision waterbath | LabCare America | 51221081 | Water bath shaker used for bacterial culture |
| Qubit 1X dsDNA HS Assay Kit | Invitrogen, Thermo Fisher Scientific | Q33231 | Reagents for fluorescence-based DNA quantification assay |
| Qubit 2.0 Fluorometer | Invitrogen, Thermo Fisher Scientific | Q32866 | Fluorometer used for fluorescence assay |
| Qubit Assay tubes | Invitrogen, Thermo Fisher Scientific | Q32856 | 0.5 mL PCR tubes for fluorescence-based DNA quantification assay |
| SimpliAmp Thermal Cycler | Applied Biosystems, Thermo Fisher Scientific | A24811 | Thermocycler used for DNA library amplification |
| Spectronic GENESYS 10 Vis | Thermo | 335900 | Spectophotometer used for bacterial suspension in antimicrobial susceptibility testing |
| ZymoBIOMICS DNA Miniprep Kit | Zymo Research Inc. | D4300 | Kit for genomic DNA extraction (50 preps) |
Références
- Naylor, R. L., et al. A 20-year retrospective review of global aquaculture. Nature. 591 (7851), 551-563 (2021).
- Quesada, S. P., Paschoal, J. A. R., Reyes, F. G. R. Considerations on the aquaculture development and on the use of veterinary drugs: special issue for fluoroquinolones-a review. Journal of Food Science. 78 (9), 1321-1333 (2013).
- Defoirdt, T., Sorgeloos, P., Bossier, P. Alternatives to antibiotics for the control of bacterial disease in aquaculture. Current Opinion in Microbiology. 14 (3), 251-258 (2011).
- Stentiford, G. D., et al. New paradigms to help solve the global aquaculture disease crisis. PLOS Pathogens. 13 (2), 1006160 (2017).
- Chen, H., et al. Tissue distribution, bioaccumulation characteristics and health risk of antibiotics in cultured fish from a typical aquaculture area. Journal of Hazardous Materials. 343, 140-148 (2018).
- Zhou, M., et al. Antibiotics control in aquaculture requires more than antibiotic-free feeds: A Tilapia farming case. Environmental Pollution. 268, 115854 (2021).
- Feng, Y., et al. Ecological effects of antibiotics on aquaculture ecosystems based on microbial community in sediments. Ocean & Coastal Management. 224, 106173 (2022).
- Shen, X., Jin, G., Zhao, Y., Shao, X. Prevalence and distribution analysis of antibiotic resistance genes in a large-scale aquaculture environment. Science of The Total Environment. 711, 134626 (2020).
- Su, H., et al. Contamination of antibiotic resistance genes (ARGs) in a typical marine aquaculture farm: source tracking of ARGs in reared aquatic organisms. Journal of Environmental Science and Health, Part B. 55 (3), 220-229 (2020).
- Oliveira, R. V., Oliveira, M. C., Pelli, A. Disease infection by Enterobacteriaceae family in fishes: a review. Journal of Microbiology & Experimentation. 4 (5), 00128 (2017).
- Barbosa, M. M. C., et al. Sorologia e suscetibilidade antimicrobiana em isolados de Escherichia coli de pesque-pagues. Arquivos do Instituto Biológico. 81 (1), 43-48 (2014).
- Valenzuela-Armenta, J. A., et al. Microbiological analysis of Tilapia and water in aquaculture farms from Sinaloa. Biotecnia. 20 (1), 20-26 (2018).
- Reza, R. H., Shipa, S. A., Naser, M. N., Miah, M. F. Surveillance of Escherichia coli in a fish farm of Sylhet, Bangladesh. Bangladesh Journal of Zoology. 48 (2), 335-346 (2021).
- Liao, C. -. Y., et al. Antimicrobial resistance of Escherichia coli From aquaculture farms and their environment in Zhanjiang, China. Frontiers in Veterinary Science. 8, 806653 (2021).
- Dewi, R. R., et al. Prevalence and antimicrobial resistance of Escherichia coli, Salmonella and Vibrio derived from farm-raised Red Hybrid Tilapia (Oreochromis spp.) and Asian Sea Bass (Lates calcarifer, Bloch 1970) on the west coast of Peninsular Malaysia. Antibiotics. 11 (2), 136 (2022).
- Leimbach, A., Hacker, J., Dobrindt, U. E. coli as an all-rounder: the thin line between commensalism and pathogenicity. Current Topics in Microbiology and Immunology. 358, 3-32 (2013).
- Kaper, J. B., Nataro, J. P., Mobley, H. L. T. Pathogenic Escherichia coli. Nature Reviews Microbiology. 2 (2), 123-140 (2004).
- Croxen, M. A., Finlay, B. B. Molecular mechanisms of Escherichia coli pathogenicity. Nature Reviews Microbiology. 8 (1), 26-38 (2010).
- Croxen, M. A., et al. Recent advances in understanding enteric pathogenic Escherichia coli. Clinical Microbiology Reviews. 26 (4), 822-880 (2013).
- Bertelli, C., Greub, G. Rapid bacterial genome sequencing: methods and applications in clinical microbiology. Clinical Microbiology and Infection. 19 (9), 803-813 (2013).
- Lynch, T., Petkau, A., Knox, N., Graham, M., Van Domselaar, G. A primer on infectious disease bacterial genomics. Clinical Microbiology Reviews. 29 (4), 881-913 (2016).
- Carriço, J. A., Rossi, M., Moran-Gilad, J., Van Domselaar, G., Ramirez, M. A primer on microbial bioinformatics for nonbioinformaticians. Clinical Microbiology and Infection. 24 (4), 342-349 (2018).
- Magaña-Lizárraga, J. A., et al. Draft genome sequence of Escherichia coli M51-3: a multidrug-resistant strain assigned as ST131-H30 recovered from infant diarrheal infection in Mexico. Journal of Global Antimicrobial Resistance. 19, 311-312 (2019).
- Pérez-Vázquez, M., et al. Emergence of NDM-producing Klebsiella pneumoniae and Escherichia coli in Spain: phylogeny, resistome, virulence and plasmids encoding blaNDM-like genes as determined by WGS. Journal of Antimicrobial Chemotherapy. 74 (12), 3489-3496 (2019).
- Massella, E., et al. Snapshot study of whole genome sequences of Escherichia coli from healthy companion animals, livestock, wildlife, humans and food in Italy. Antibiotics. 9 (11), 782 (2020).
- Magaña-Lizárraga, J. A., et al. Genomic profiling of antibiotic-resistant Escherichia coli isolates from surface water of agricultural drainage in north-western Mexico: detection of the international high-risk lineages ST410 and ST617. Microorganisms. 10 (3), 662 (2022).
- Saracino, I. M., et al. Next Generation sequencing for the prediction of the antibiotic resistance in Helicobacter pylori: a literature review. Antibiotics. 10 (4), 437 (2021).
- Ghosh, A., Saha, S. Survey of drug resistance associated gene mutations in Mycobacterium tuberculosis, ESKAPE and other bacterial species. Scientific Reports. 10 (1), 8957 (2020).
- Su, M., Satola, S. W., Read, T. D. Genome-based prediction of bacterial antibiotic resistance. Journal of Clinical Microbiology. 57 (3), 01405-01418 (2019).
- Brzuszkiewicz, E., et al. Genome sequence analyses of two isolates from the recent Escherichia coli outbreak in Germany reveal the emergence of a new pathotype: Entero-Aggregative-Haemorrhagic Escherichia coli (EAHEC). Archives of Microbiology. 193 (12), 883-891 (2011).
- . CLSI Performance Standards for Antimicrobial Disk Susceptibility Tests. 13th ed. CLSI standard M02. Wayne, PA: Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m02/ (2018)
- CLSI Performance Standards for Antimicrobial Susceptibility Testing. 31st ed. CLSI supplement M100. Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m100/ (2021)
- Ewing, B., Green, P. Base-calling of automated sequencer traces using phred. II. Error probabilities. Genome Research. 8 (3), 186-194 (1998).
- Quainoo, S., et al. Whole-genome sequencing of bacterial pathogens: the future of nosocomial outbreak analysis. Clinical Microbiology Reviews. 30 (4), 1015-1063 (2017).
- Desai, A., et al. Identification of optimum sequencing depth especially for de novo genome assembly of small genomes using next generation sequencing data. PLoS ONE. 8 (4), 60204 (2013).
- Nishino, K., Yamada, J., Hirakawa, H., Hirata, T., Yamaguchi, A. Roles of TolC-dependent multidrug transporters of Escherichia coli in resistance to β-lactams. Antimicrobial Agents and Chemotherapy. 47 (9), 3030-3033 (2003).
- Li, M., et al. The resistance mechanism of Escherichia coli induced by ampicillin in laboratory. Infection and Drug Resistance. 12, 2853-2863 (2019).
- Ménard, L. -. P., Dubreuil, J. D. Enteroaggregative Escherichia coli heat-stable enterotoxin 1 (EAST1): a new toxin with an old twist. Critical Reviews in Microbiology. 28 (1), 43-60 (2002).
- Dubreuil, J. D. EAST1 toxin: An enigmatic molecule associated with sporadic episodes of diarrhea in humans and animals. Journal of Microbiology. 57 (7), 541-549 (2019).
- Goldstein, S., Beka, L., Graf, J., Klassen, J. L. Evaluation of strategies for the assembly of diverse bacterial genomes using MinION long-read sequencing. BMC Genomics. 20 (1), 23 (2019).
- Guerrero, A., Gomez-Gil, B., Lizarraga-Partida, M. L. Genomic stability among O3:K6 V. parahaemolyticus pandemic strains isolated between 1996 to 2012 in American countries. BMC Genomic Data. 22 (1), 38 (2021).
- FAO Applications of Whole Genome Sequencing (WGS) in food safety management. Food and Agriculture Organization of the United Nations Available from: https://www.fao.org/documents/card/es/c/61e44b34-b328-4239-b59c-a9e926e327b4/ (2016)
- Rantsiou, K., et al. Next generation microbiological risk assessment: opportunities of whole genome sequencing (WGS) for foodborne pathogen surveillance, source tracking and risk assessment. International Journal of Food Microbiology. 287, 3-9 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon