È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Caratterizzazione di un ceppo patogeno di Escherichia coli derivato da allevamenti di Oreochromis spp. che utilizzano il sequenziamento dell'intero genoma
In questo articolo
Riepilogo
La fattibilità di strategie di sequenziamento dell'intero genoma (WGS) utilizzando strumenti da banco ha semplificato l'interrogazione del genoma di ogni microbo di rilevanza per la salute pubblica in un ambiente di laboratorio. Viene descritto un adattamento metodologico del flusso di lavoro per WGS batterico e viene inoltre presentata una pipeline bioinformatica per l'analisi.
Abstract
L'acquacoltura è uno dei settori di produzione alimentare in più rapida crescita in tutto il mondo e l'allevamento di tilapia (Oreochromis spp.) costituisce la principale varietà di pesce d'acqua dolce coltivata. Poiché le pratiche di acquacoltura sono suscettibili alla contaminazione microbica derivata da fonti antropogeniche, è necessario un ampio uso di antibiotici, portando i sistemi di acquacoltura a diventare un'importante fonte di batteri resistenti agli antibiotici e patogeni di rilevanza clinica come Escherichia coli (E. coli). Qui, la resistenza antimicrobica, la virulenza e le caratteristiche del mobiloma di un ceppo patogeno di E. coli , recuperato da Oreochromis spp. allevato nell'entroterra, sono state chiarite attraverso il sequenziamento dell'intero genoma (WGS) e l'analisi in silico . Sono stati eseguiti test di sensibilità antimicrobica (AST) e WGS. Inoltre, il gruppo filogenetico, il sierotipo, la tipizzazione della sequenza multilocus (MLST), la resistenza antimicrobica acquisita, la virulenza, il contenuto di plasmidi e profagi sono stati determinati utilizzando diversi strumenti web disponibili. L'isolato di E. coli ha mostrato solo una suscettibilità intermedia all'ampicillina ed è stato caratterizzato come ceppo ONT: H21-B1-ST40 dalla tipizzazione basata su WGS. Sebbene sia stato rilevato un solo gene correlato alla resistenza antimicrobica [mdf(A)], sono stati identificati diversi geni associati alla virulenza (VAG) dal patotipo enteropatogeno atipico E. coli (aEPEC). Inoltre, è stato rilevato il carico di replicons plasmidi da grandi gruppi plasmidi e 18 regioni associate ai profagi. In conclusione, la caratterizzazione WGS di un isolato aEPEC, recuperato da un allevamento ittico a Sinaloa, in Messico, consente di comprendere il suo potenziale patogeno e il possibile rischio per la salute umana derivante dal consumo di prodotti di acquacoltura crudi. È necessario sfruttare le tecniche di sequenziamento di nuova generazione (NGS) per studiare i microrganismi ambientali e adottare un quadro sanitario unico per imparare come hanno origine i problemi di salute.
Introduzione
L'acquacoltura è uno dei settori di produzione alimentare in più rapida crescita in tutto il mondo e le sue pratiche di produzione sono destinate a soddisfare la crescente domanda alimentare per il consumo umano. La produzione mondiale dell'acquacoltura è triplicata da 34 milioni di tonnellate (Mt) nel 1997 a 112 Mt nel 20171. I principali gruppi di specie, che hanno contribuito a quasi il 75% della produzione, sono stati alghe, carpe, bivalvi, pesci gatto e tilapia (Oreochromis spp.) 1. Tuttavia, la comparsa di malattie causate da entità microbiche è inevitabile a causa dell'allevamento ittico intensivo, con conseguenti potenziali perdite economiche2.
L'uso di antibiotici nelle pratiche di allevamento ittico è ben noto per prevenire e curare le infezioni batteriche, il principale fattore limitante della produttività 3,4. Tuttavia, gli antibiotici residui si accumulano nei sedimenti di acquacoltura e nell'acqua, esercitando una pressione selettiva e modificando le comunità batteriche associate ai pesci e residenti 5,6,7,8. Di conseguenza, l'ambiente dell'acquacoltura funge da serbatoio per i geni di resistenza antimicrobica (ARG) e per l'ulteriore comparsa e diffusione di batteri resistenti agli antibiotici (ARB) nell'ambiente circostante9. Oltre ai patogeni batterici comunemente osservati che colpiscono le pratiche di allevamento ittico, si incontrano spesso membri della famiglia delle Enterobacteriaceae, compresi ceppi patogeni umani di Enterobacter spp., Escherichia coli, Klebsiella spp. e Salmonella spp.10. E. coli è il microrganismo più comune isolato dalla farina di pesce e dall'acqua negli allevamenti ittici 11,12,13,14,15.
E. coli è un versatile batterio gram-negativo che abita il tratto gastrointestinale di mammiferi e uccelli come membro commensale del loro microbiota intestinale. Tuttavia, E. coli possiede una capacità altamente adattativa di colonizzare e persistere in diverse nicchie ambientali, tra cui suolo, sedimenti, cibo e acqua16. A causa del guadagno e della perdita genica attraverso il fenomeno del trasferimento genico orizzontale (HGT), E. coli si è rapidamente evoluto in un patogeno resistente agli antibiotici ben adattato, in grado di causare un ampio spettro di malattie nell'uomo e negli animali17,18. In base all'origine dell'isolamento, le varianti patogene sono definite come E. coli patogeno intestinale (InPEC) o E. coli patogeno extra-intestinale (ExPEC). Inoltre, InPEC ed ExPEC sono sottoclassificati in patotipi ben definiti in base alla manifestazione della malattia, al background genetico, ai tratti fenotipici e ai fattori di virulenza (VF)16,17,19.
La coltura tradizionale e le tecniche molecolari per i ceppi patogeni di E. coli hanno permesso la rapida individuazione e identificazione di diversi patotipi. Tuttavia, possono richiedere molto tempo, essere laboriosi e spesso richiedere un'elevata formazione tecnica19. Inoltre, nessun singolo metodo può essere utilizzato per studiare in modo affidabile tutte le varianti patogene di E. coli a causa della complessità del loro background genetico. Attualmente, questi inconvenienti sono stati superati con l'avvento delle tecnologie HTS (High-Throughput Sequencing). Gli approcci di sequenziamento dell'intero genoma (WGS) e gli strumenti bioinformatici hanno migliorato l'esplorazione del DNA microbico in modo economico e su larga scala, facilitando la caratterizzazione approfondita dei microbi in un'unica corsa, comprese le varianti patogene strettamente correlate20,21,22. A seconda delle domande biologiche, diversi strumenti bioinformatici, algoritmi e database possono essere utilizzati per eseguire l'analisi dei dati. Ad esempio, se l'obiettivo principale è valutare la presenza di ARG, VF e plasmidi, strumenti come ResFinder, VirulenceFinder e PlasmidFinder, insieme ai loro database associati, potrebbero essere un buon punto di partenza. Carriço et al.22 hanno fornito una panoramica dettagliata dei diversi software bioinformatici e dei relativi database applicati per l'analisi microbica WGS, dalla pre-elaborazione dei dati grezzi all'inferenza filogenetica.
Diversi studi hanno dimostrato l'ampia utilità del WGS per l'interrogazione del genoma per quanto riguarda gli attributi di resistenza antimicrobica, il potenziale patogeno e il monitoraggio dell'emergenza e delle relazioni evolutive di varianti clinicamente rilevanti di E. coli provenienti da diverse origini23,24,25,26 . WGS ha permesso l'identificazione dei meccanismi molecolari alla base della resistenza fenotipica agli antimicrobici, compresi quei meccanismi di resistenza rari o complessi. Ciò avviene attraverso la rilevazione di varianti ARG acquisite, nuove mutazioni nei geni bersaglio dei farmaci o regioni promotrici27,28. Inoltre, WGS offre il potenziale per dedurre profili di resistenza antimicrobica senza richiedere conoscenze preliminari sul fenotipo di resistenza di un ceppo batterico29. In alternativa, WGS ha permesso la caratterizzazione degli elementi genetici mobili (MGE) che portano sia la resistenza antimicrobica che le caratteristiche di virulenza, che ha guidato l'evoluzione del genoma batterico dei patogeni esistenti. Ad esempio, l'applicazione di WGS durante l'indagine sull'epidemia tedesca di E. coli nel 2011 ha portato alla scoperta delle caratteristiche genomiche uniche di un patotipo di E. coli apparentemente nuovo; È interessante notare che questi ceppi epidemici hanno avuto origine dal gruppo enteroaggregativo E. coli (EAEC), che ha acquisito il profago che codifica la tossina Shiga dal patotipo enteroemorragico E. coli (EHEC)30.
Questo lavoro presenta un adattamento metodologico del flusso di lavoro per WGS batterico utilizzando un sequenziatore da banco. Inoltre, viene fornita una pipeline bioinformatica utilizzando strumenti basati sul web per analizzare le sequenze risultanti e supportare ulteriormente i ricercatori con competenze bioinformatiche limitate o assenti. I metodi descritti hanno permesso di chiarire la resistenza antimicrobica, la virulenza e le caratteristiche del mobiloma di un ceppo patogeno di E. coli ACM5, isolato nel 2011 da Oreochromis spp. nell'entroterra coltivato a Sinaloa, Messico12.
Protocollo
NOTA: Il ceppo ACM5 di E. coli è stato recuperato elaborando e coltivando il campione di pesce per la determinazione dei coliformi fecali (FC)12. Durante il campionamento del pesce, i pesci non hanno mostrato segni clinici di malattia, infezione batterica o fungina e ha prevalso una temperatura media di 22,3 ° C. Dopo l'isolamento, l'isolato di E. coli è stato sottoposto a test biochimici e crioconservato in brodo di infusione cardiaca cerebrale (BHI) con DMSO (8% v / v) come agente crioprotettivo.
1. Riattivazione della coltura madre congelata di E. coli ACM5
- Dal brodo batterico congelato, aprire il tubo e utilizzare un anello sterile, una punta di pipetta o uno stuzzicadenti per raschiare la superficie della coltura batterica congelata.
- Striare i batteri su una piastra di agar Luria-Bertani (LB) e incubare a 37 ± 2 °C per 24 ore.
2. Determinazione della suscettibilità antibatterica
NOTA: Il test di sensibilità antimicrobica qui descritto corrisponde al metodo di diffusione del disco basato sulle linee guida del Clinical and Laboratory Standards Institute (CLSI) (M02 Ed13:2018)31. Coli Il ceppo ATCC 25922 è richiesto ai fini del controllo qualità.
- Preparazione dell'inoculo con il metodo della sospensione della colonia
- Dalla piastra LB incubata nella fase 1.2, strisciare una o due colonie su una piastra di agar Mueller-Hinton (MH) e incubare a 37 ± 2 °C per 18-24 ore.
- Utilizzare un anello sterile per prelevare due o tre colonie dall'isolato di E. coli appena sub-coltivato su piastra di agar MH e risospenderle in 3 ml di brodo MH sterile o soluzione salina allo 0,85% (p/v), mescolando accuratamente per vortice per ottenere una sospensione batterica uniforme.
- Utilizzando uno spettrofotometro UV-visibile (vedi Tabella dei materiali), regolare la sospensione batterica su torbidità paragonabile a uno standard di 0,5 MacFarland (equivalente a circa 1-2 x 108 celle / ml). Se la sospensione batterica è troppo leggera o troppo pesante, aggiungere più colonie di E. coli o brodo MH a seconda dei casi. Utilizzare l'inoculo preparato entro 15 minuti dalla preparazione.
- Inoculazione delle piastre di agar di prova
- Immergere un batuffolo di cotone sterile nella sospensione batterica regolata. Ruotare il tampone più volte e premerlo saldamente sulla parete interna del tubo per rimuovere il liquido in eccesso dal tampone.
- Inoculare una piastra di agar MH strisciando il tampone 3 volte su tutta la superficie dell'agar, ruotando la piastra di 60 ° ogni volta. Tamponare anche il bordo dell'agar per garantire una distribuzione uniforme dell'inoculo.
- Lasciare il coperchio della capsula di Petri socchiuso per 3-5 minuti per consentire all'umidità di evaporare.
- Applicazione del disco antimicrobico su piastre di agar inoculate
- Distribuire uniformemente e premere ciascun disco antimicrobico (vedere Tabella dei materiali) sulla superficie delle piastre di agar inoculate per garantire un contatto completo. Capovolgere le piastre entro 15 minuti dall'applicazione del disco e incubare a 35 ± 2 °C per 16-18 ore.
NOTA: Un massimo di 12 e sei dischi devono essere utilizzati in piastre di Petri rispettivamente di 150 mm e 100 mm.
- Distribuire uniformemente e premere ciascun disco antimicrobico (vedere Tabella dei materiali) sulla superficie delle piastre di agar inoculate per garantire un contatto completo. Capovolgere le piastre entro 15 minuti dall'applicazione del disco e incubare a 35 ± 2 °C per 16-18 ore.
- Dopo l'incubazione, misurare la zona delle dimensioni di inibizione utilizzando una pinza Vernier e interpretare i diametri risultanti secondo i criteri del punto di rottura CLSI (M100 - Tabella 2A)32.
3. Estrazione e quantificazione del DNA genomico (gDNA)
- Estrazione del DNA genomico
- Risospendere un ciclo di colonie di E. coli appena coltivate in 5 ml di brodo LB e incubare per una notte in un'incubatrice agitata (180 giri / min) a 37 ± 2 ° C.
- Centrifugare la sospensione batterica a 3.500 x g per 5 minuti ed eliminare con attenzione il surnatante.
- Estrarre il gDNA seguendo le linee guida del kit di estrazione del DNA (vedi Tabella dei materiali).
- Verificare la purezza del gDNA misurando la densità ottica a 260/280 nm (rapporto: >1,8) e 260/230 nm (rapporto: 2,0-2,2) utilizzando uno spettrofotometro UV-visibile. Eseguire l'elettroforesi su gel di agarosio allo 0,8% per verificare l'integrità del gDNA.
- Quantificazione del DNA genomico
NOTA: Utilizzare solo provette PCR a parete sottile, trasparenti, da 0,5 mL approvate dal produttore del kit di dosaggio a fluorescenza (vedere la tabella dei materiali).- Impostare il numero richiesto di provette per i campioni e gli standard di analisi. Preparare la soluzione di analisi di lavoro miscelando il componente A e il componente B forniti nel kit, seguendo le linee guida del produttore.
- Preparare gli standard di analisi aggiungendo 190 μL della soluzione di lavoro e 10 μL di ogni standard al tubo appropriato (sono necessari due standard).
- Aggiungere 198 μL della soluzione di lavoro e 2 μL del campione di DNA alla provetta appropriata. Mescolare energicamente vorticando tutti i tubi per 5 s. Fai attenzione a non creare bolle.
- Incubare tutti i tubi per 2 minuti a temperatura ambiente, al riparo dalla luce. Misurare la fluorescenza di tutti i tubi in base alle linee guida del produttore utilizzando il fluorometro (vedere la tabella dei materiali).
- Regolare correttamente la concentrazione del campione di gDNA per procedere al sequenziamento.
4. Preparazione della libreria del DNA
NOTA: la preparazione e il sequenziamento della libreria del DNA sono stati eseguiti seguendo le linee guida e i protocolli del produttore (vedere la tabella dei materiali). La concentrazione iniziale di gDNA è di 4,0 ng.
- Tagmentation, amplificazione PCR e indicizzazione
- In una provetta da 0,2 ml, aggiungere 2,5 μL di tampone di tagmentazione e 2 μL di gDNA di ingresso (2,0 ng/μL). Mescolare delicatamente mediante pipettaggio.
- Aggiungere 1 μL di buffer di amplificazione e mescolare delicatamente mediante pipettaggio. Girare a 280 x g per 1 minuto a temperatura ambiente.
- Posizionare i campioni in un termociclatore (vedere Tabella dei materiali) ed eseguire il seguente programma PCR: 55 °C per 5 minuti, quindi tenere a 10 °C. Quando i campioni raggiungono i 10 °C, procedere immediatamente a neutralizzare la reazione.
- Aggiungere 1 μL di tampone neutralizzante alle provette e mescolare delicatamente mediante pipettaggio. Centrifugare a 280 x g per 1 minuto e incubare per 5 minuti a temperatura ambiente.
- Aggiungere 1,7 μL di ciascun adattatore indice (cioè indici i7 e i5) e 3 μL di master mix PCR indicizzato. Mescolare delicatamente mediante pipettaggio. Girare a 280 x g per 1 minuto a temperatura ambiente.
- Posizionare i campioni nel termociclatore ed eseguire una seconda reazione PCR come segue: 72 °C per 3 min, 95 °C per 30 s, 18 cicli di 95 °C per 10 s, 55 °C per 30 s, 72 °C per 30 s, 72 °C per 5 minuti e tenere a 10 °C.
- Pulizia amplificata della libreria
- Miscelare vorticando la soluzione commerciale di sfere magnetiche (vedere Tabella dei materiali) in base alle linee guida del produttore.
- Trasferire il campione di gDNA etichettato/indicizzato in una nuova provetta da 1,5 mL e aggiungere 0,6 μL di sfere magnetiche per ogni μL del volume finale del campione di gDNA (≈13 μL). Mescolare delicatamente mediante pipettaggio e incubare per 5 minuti a temperatura ambiente.
- Posizionare le provette su un rack magnetico (vedere Tabella dei materiali) per 2 minuti fino a quando il surnatante non si è liberato. Rimuovere e scartare accuratamente il surnatante senza disturbare le perline.
- Aggiungere 200 μL di etanolo all'80% appena preparato senza mescolare. Incubare per 30 s fino a quando l'eluato si schiarisce e rimuovere e scartare con cura il surnatante senza disturbare le perline.
- Eseguire una seconda fase di lavaggio. Aggiungere 200 μL di etanolo all'80% alle perle e incubare per 30 s. Dopo che l'eluato si è schiarito, rimuovere e scartare il surnatante e asciugare all'aria le perline per 10 minuti.
- Aggiungere 15 μL di tampone tris da 10 mM (pH 8) alle sfere e mescolare delicatamente mediante pipettaggio. Incubare per 2 minuti a temperatura ambiente. Posizionare nuovamente le provette di campionamento sul rack magnetico per 2 minuti, consentendo al surnatante di liberarsi.
- Trasferire con cautela 14 μL di surnatante dalla provetta a una nuova provetta da 0,2 ml. Questa è la libreria ripulita.
NOTA: A questo punto, la libreria ripulita può essere conservata a -20 °C per un massimo di 7 giorni.
- Normalizzazione della libreria
- Procedere alla quantificazione delle librerie ripulite mediante saggio di fluorescenza come descritto al punto 3.2, ad eccezione del punto 3.2.5.
- Visualizza le librerie ripulite su un'elettroforesi su gel di agarosio all'1% per determinare le dimensioni medie dei frammenti.
- Calcolare il valore di molarità della libreria utilizzando la seguente equazione:
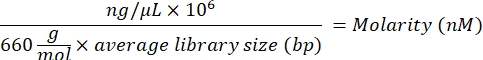
- Utilizzando il valore di molarità, calcolare i volumi appropriati di buffer di risospensione (RSB) e librerie ripulite per diluire la singola libreria a una concentrazione iniziale di 10 nM.
5. Pool di librerie, denaturalizzazione e avvio del sequencer
- Scongelare la cartuccia del reagente seguendo le istruzioni del produttore. Estrarre la cella di flusso dal frigorifero (4 °C) e portarla a temperatura ambiente prima del sequenziamento.
- Raggruppa 5 μL di ciascuna libreria normalizzata (10 nM) in un tubo a basso legame da 1,5 ml. Diluire la libreria in pool a 4 nM con il volume appropriato di RSB.
- Aggiungere 5 μL della libreria in pool (4 nM) e 5 μL di 0,2 N NaOH in una nuova provetta da 1,5 mL a basso legame. Mescolare brevemente a vortice, girare a 280 x g per 1 minuto e incubare per 5 minuti a temperatura ambiente per denaturare la libreria in pool in singoli filamenti.
- Miscelare delicatamente 10 μL di libreria in pool denaturata e 990 μL di tampone di ibridazione prerefrigerato mediante pipettaggio e posizionare su ghiaccio fino a quando non viene eseguita la diluizione finale per il caricamento del sequenziatore. La concentrazione della biblioteca in pool denaturata è di 20 pM.
- Scongelare e preparare una libreria di controllo (vedi Tabella dei materiali) ad una concentrazione di 4 nM mescolando 2 μL di libreria di controllo (10 nM) e 3 μL di acqua priva di nucleasi.
- Mescolare 5 μL della libreria di controllo (4 nM) e 5 μL di NaOH 0,2 N appena preparato. Mescolare brevemente a vortice, girare a 280 x g per 1 minuto e incubare per 5 minuti a temperatura ambiente per denaturare la libreria di controllo in singoli filamenti.
- Miscelare delicatamente 10 μL di libreria di controllo denaturata e 990 μL di tampone di ibridazione prerefrigerato mediante pipettaggio e posizionare su ghiaccio fino al completamento della diluizione finale per il caricamento del sequenziatore. La concentrazione della libreria di controllo denaturata è di 20 pM.
- In un nuovo tubo a basso legame da 1,5 mL, combinare 594 μL della libreria in pool denaturata a 20 pM e 6 μL della libreria di controllo denaturata a 20 pM. Mescolare correttamente.
- Diluire la miscela finale della libreria (20 pM) a una concentrazione di carico finale di 1,2 pM in un volume di 600 μL utilizzando il tampone di ibridazione prerefrigerato.
- Prima di caricare la miscela finale della libreria sulla cartuccia del reagente, eseguire un ulteriore pretrattamento termico per ottenere un carico efficiente nella cella di flusso. Incubare la miscela finale della libreria per 2 minuti a 96 °C. Capovolgere il tubo per mescolare e posizionare il tubo sul ghiaccio per 5 minuti.
- Caricare 500 μL della miscela di libreria finale nel serbatoio progettato sulla cartuccia del reagente. Avviare la sequenziazione seguendo le linee guida. Caricare la cella di flusso e la cartuccia del reagente e impostare l'esecuzione del sequenziamento.
6. Analisi dei dati di sequenza
NOTA: Controllare il file supplementare 1 per ulteriori informazioni sulla pre-elaborazione generale dei dati WGS, sul software, sulle impostazioni dei parametri e sull'analisi della sequenza del genoma di E. coli.
- Connettersi al server di sequenziamento dei dati e scaricare i file FASTQ.
- Valuta la qualità iniziale dei dati di sequenza grezzi con software di terze parti. Rimuovere le sequenze residue dell'adattatore, le basi di bassa qualità (File supplementare 1).
- Assemblare i dati di sequenziamento di qualità verificata a livello continuo o di scaffold utilizzando software di terze parti (vedere File supplementare 1).
- Eseguire l'annotazione del genoma inviando il file FASTA contenente il genoma assemblato al server RAST (https://rast.nmpdr.org/).
- Caricare il file FASTA sulle piattaforme web del Center for Genome Epidemiology (CGE) (http://www.genomicepidemiology.org/services/) e ClermonTyping (http://clermontyping.iame-research.center/index.php) per identificare caratteristiche epidemiologiche, ARG, VAG e plasmidi (vedi file supplementare 1).
- Caricare il file FASTA sul server PHASTER per identificare le sequenze di profagi (https://phaster.ca/).
Risultati
La suscettibilità antimicrobica è stata determinata dal metodo di diffusione del disco e interpretata dai criteri di breakpoint CLSI per 12 antibiotici che coprono sei distinte classi antimicrobiche, ovvero aminoglicosidi, β-lattamici, fluorochinoloni, nitrofurani, fenioli e antagonisti della via dei folati. L'E. coli ACM5 ha mostrato sensibilità a tutti gli antibiotici tranne un farmaco β-lattamico. Sono stati testati quattro farmaci β-lattamici: ampicillina, carbenicillina, cefalotina e cefotaxime. Tra q...
Discussione
Questo studio presenta un adattamento del flusso di lavoro batterico WGS utilizzando un sequenziatore da banco e una pipeline per la caratterizzazione genomica di una variante patogena di E. coli. A seconda della piattaforma di sequenziamento utilizzata, i tempi di consegna (TAT) per le procedure di laboratorio umide (coltivazione batterica, estrazione del gDNA, preparazione della libreria e sequenziamento) e l'analisi della sequenza potrebbero variare, in particolare se vengono studiati batteri a crescita lenta...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Al Consiglio Nazionale della Scienza e della Tecnologia del Messico (CONACyT con il suo acronimo in spagnolo) per la borsa di dottorato assegnata a José Antonio Magaña-Lizárraga [n. 481143].
Materiali
| Name | Company | Catalog Number | Comments |
| Accublock Mini digital dry bath | Labnet | D0100 | Dry bath for incubation of tubes |
| Agencourt AMPure XP | Beckman Coulter | A63881 | Magnetic beads in solution for DNA library purification |
| DeNovix DS-11 | DeNovix Inc. | UV-Vis spectophotometer to check the quality of the gDNA extracted | |
| DNA LoBind Tubes | Eppendorf | 0030108418 | 1.5 mL PCR tubes for DNA library pooling |
| DynaMag-2 Magnet | Invitrogen, Thermo Fisher Scientific | 12321D | Magnetic microtube rack used during magnetic beads-based DNA purification |
| Gram-negative Multibac I.D. | Diagnostic reseach (Mexico) | PT-35 | Commercial standard antibiotic disks for antimicrobial susceptibility testing |
| MiniSeq Mid Output Kit (300-cycles) | Illumina | FC-420-1004 | Reagent cartdrige for paired-end sequencing (2x150) |
| MiniSeq System Instrument | Illumina | SY-420-1001 | Benchtop sequencer used for Next-generation sequencing |
| MiniSpin centrifuge | Eppendorf | 5452000816 | Standard centrifuge for tubes |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1024 | Reagents to perform DNA libraries for sequencing. Includes Box 1 and Box 2 reagents for 24 samples |
| Nextera XT Index Kit v2 | Illumina | FC-131-2001, FC-131-2002, FC-131-2003, FC-131-2004 | Index set A, B, C, D |
| PhiX Control v3 | Illumina | FC-110-3001 | DNA library control for sequencing |
| Precision waterbath | LabCare America | 51221081 | Water bath shaker used for bacterial culture |
| Qubit 1X dsDNA HS Assay Kit | Invitrogen, Thermo Fisher Scientific | Q33231 | Reagents for fluorescence-based DNA quantification assay |
| Qubit 2.0 Fluorometer | Invitrogen, Thermo Fisher Scientific | Q32866 | Fluorometer used for fluorescence assay |
| Qubit Assay tubes | Invitrogen, Thermo Fisher Scientific | Q32856 | 0.5 mL PCR tubes for fluorescence-based DNA quantification assay |
| SimpliAmp Thermal Cycler | Applied Biosystems, Thermo Fisher Scientific | A24811 | Thermocycler used for DNA library amplification |
| Spectronic GENESYS 10 Vis | Thermo | 335900 | Spectophotometer used for bacterial suspension in antimicrobial susceptibility testing |
| ZymoBIOMICS DNA Miniprep Kit | Zymo Research Inc. | D4300 | Kit for genomic DNA extraction (50 preps) |
Riferimenti
- Naylor, R. L., et al. A 20-year retrospective review of global aquaculture. Nature. 591 (7851), 551-563 (2021).
- Quesada, S. P., Paschoal, J. A. R., Reyes, F. G. R. Considerations on the aquaculture development and on the use of veterinary drugs: special issue for fluoroquinolones-a review. Journal of Food Science. 78 (9), 1321-1333 (2013).
- Defoirdt, T., Sorgeloos, P., Bossier, P. Alternatives to antibiotics for the control of bacterial disease in aquaculture. Current Opinion in Microbiology. 14 (3), 251-258 (2011).
- Stentiford, G. D., et al. New paradigms to help solve the global aquaculture disease crisis. PLOS Pathogens. 13 (2), 1006160 (2017).
- Chen, H., et al. Tissue distribution, bioaccumulation characteristics and health risk of antibiotics in cultured fish from a typical aquaculture area. Journal of Hazardous Materials. 343, 140-148 (2018).
- Zhou, M., et al. Antibiotics control in aquaculture requires more than antibiotic-free feeds: A Tilapia farming case. Environmental Pollution. 268, 115854 (2021).
- Feng, Y., et al. Ecological effects of antibiotics on aquaculture ecosystems based on microbial community in sediments. Ocean & Coastal Management. 224, 106173 (2022).
- Shen, X., Jin, G., Zhao, Y., Shao, X. Prevalence and distribution analysis of antibiotic resistance genes in a large-scale aquaculture environment. Science of The Total Environment. 711, 134626 (2020).
- Su, H., et al. Contamination of antibiotic resistance genes (ARGs) in a typical marine aquaculture farm: source tracking of ARGs in reared aquatic organisms. Journal of Environmental Science and Health, Part B. 55 (3), 220-229 (2020).
- Oliveira, R. V., Oliveira, M. C., Pelli, A. Disease infection by Enterobacteriaceae family in fishes: a review. Journal of Microbiology & Experimentation. 4 (5), 00128 (2017).
- Barbosa, M. M. C., et al. Sorologia e suscetibilidade antimicrobiana em isolados de Escherichia coli de pesque-pagues. Arquivos do Instituto Biológico. 81 (1), 43-48 (2014).
- Valenzuela-Armenta, J. A., et al. Microbiological analysis of Tilapia and water in aquaculture farms from Sinaloa. Biotecnia. 20 (1), 20-26 (2018).
- Reza, R. H., Shipa, S. A., Naser, M. N., Miah, M. F. Surveillance of Escherichia coli in a fish farm of Sylhet, Bangladesh. Bangladesh Journal of Zoology. 48 (2), 335-346 (2021).
- Liao, C. -. Y., et al. Antimicrobial resistance of Escherichia coli From aquaculture farms and their environment in Zhanjiang, China. Frontiers in Veterinary Science. 8, 806653 (2021).
- Dewi, R. R., et al. Prevalence and antimicrobial resistance of Escherichia coli, Salmonella and Vibrio derived from farm-raised Red Hybrid Tilapia (Oreochromis spp.) and Asian Sea Bass (Lates calcarifer, Bloch 1970) on the west coast of Peninsular Malaysia. Antibiotics. 11 (2), 136 (2022).
- Leimbach, A., Hacker, J., Dobrindt, U. E. coli as an all-rounder: the thin line between commensalism and pathogenicity. Current Topics in Microbiology and Immunology. 358, 3-32 (2013).
- Kaper, J. B., Nataro, J. P., Mobley, H. L. T. Pathogenic Escherichia coli. Nature Reviews Microbiology. 2 (2), 123-140 (2004).
- Croxen, M. A., Finlay, B. B. Molecular mechanisms of Escherichia coli pathogenicity. Nature Reviews Microbiology. 8 (1), 26-38 (2010).
- Croxen, M. A., et al. Recent advances in understanding enteric pathogenic Escherichia coli. Clinical Microbiology Reviews. 26 (4), 822-880 (2013).
- Bertelli, C., Greub, G. Rapid bacterial genome sequencing: methods and applications in clinical microbiology. Clinical Microbiology and Infection. 19 (9), 803-813 (2013).
- Lynch, T., Petkau, A., Knox, N., Graham, M., Van Domselaar, G. A primer on infectious disease bacterial genomics. Clinical Microbiology Reviews. 29 (4), 881-913 (2016).
- Carriço, J. A., Rossi, M., Moran-Gilad, J., Van Domselaar, G., Ramirez, M. A primer on microbial bioinformatics for nonbioinformaticians. Clinical Microbiology and Infection. 24 (4), 342-349 (2018).
- Magaña-Lizárraga, J. A., et al. Draft genome sequence of Escherichia coli M51-3: a multidrug-resistant strain assigned as ST131-H30 recovered from infant diarrheal infection in Mexico. Journal of Global Antimicrobial Resistance. 19, 311-312 (2019).
- Pérez-Vázquez, M., et al. Emergence of NDM-producing Klebsiella pneumoniae and Escherichia coli in Spain: phylogeny, resistome, virulence and plasmids encoding blaNDM-like genes as determined by WGS. Journal of Antimicrobial Chemotherapy. 74 (12), 3489-3496 (2019).
- Massella, E., et al. Snapshot study of whole genome sequences of Escherichia coli from healthy companion animals, livestock, wildlife, humans and food in Italy. Antibiotics. 9 (11), 782 (2020).
- Magaña-Lizárraga, J. A., et al. Genomic profiling of antibiotic-resistant Escherichia coli isolates from surface water of agricultural drainage in north-western Mexico: detection of the international high-risk lineages ST410 and ST617. Microorganisms. 10 (3), 662 (2022).
- Saracino, I. M., et al. Next Generation sequencing for the prediction of the antibiotic resistance in Helicobacter pylori: a literature review. Antibiotics. 10 (4), 437 (2021).
- Ghosh, A., Saha, S. Survey of drug resistance associated gene mutations in Mycobacterium tuberculosis, ESKAPE and other bacterial species. Scientific Reports. 10 (1), 8957 (2020).
- Su, M., Satola, S. W., Read, T. D. Genome-based prediction of bacterial antibiotic resistance. Journal of Clinical Microbiology. 57 (3), 01405-01418 (2019).
- Brzuszkiewicz, E., et al. Genome sequence analyses of two isolates from the recent Escherichia coli outbreak in Germany reveal the emergence of a new pathotype: Entero-Aggregative-Haemorrhagic Escherichia coli (EAHEC). Archives of Microbiology. 193 (12), 883-891 (2011).
- . CLSI Performance Standards for Antimicrobial Disk Susceptibility Tests. 13th ed. CLSI standard M02. Wayne, PA: Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m02/ (2018)
- CLSI Performance Standards for Antimicrobial Susceptibility Testing. 31st ed. CLSI supplement M100. Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m100/ (2021)
- Ewing, B., Green, P. Base-calling of automated sequencer traces using phred. II. Error probabilities. Genome Research. 8 (3), 186-194 (1998).
- Quainoo, S., et al. Whole-genome sequencing of bacterial pathogens: the future of nosocomial outbreak analysis. Clinical Microbiology Reviews. 30 (4), 1015-1063 (2017).
- Desai, A., et al. Identification of optimum sequencing depth especially for de novo genome assembly of small genomes using next generation sequencing data. PLoS ONE. 8 (4), 60204 (2013).
- Nishino, K., Yamada, J., Hirakawa, H., Hirata, T., Yamaguchi, A. Roles of TolC-dependent multidrug transporters of Escherichia coli in resistance to β-lactams. Antimicrobial Agents and Chemotherapy. 47 (9), 3030-3033 (2003).
- Li, M., et al. The resistance mechanism of Escherichia coli induced by ampicillin in laboratory. Infection and Drug Resistance. 12, 2853-2863 (2019).
- Ménard, L. -. P., Dubreuil, J. D. Enteroaggregative Escherichia coli heat-stable enterotoxin 1 (EAST1): a new toxin with an old twist. Critical Reviews in Microbiology. 28 (1), 43-60 (2002).
- Dubreuil, J. D. EAST1 toxin: An enigmatic molecule associated with sporadic episodes of diarrhea in humans and animals. Journal of Microbiology. 57 (7), 541-549 (2019).
- Goldstein, S., Beka, L., Graf, J., Klassen, J. L. Evaluation of strategies for the assembly of diverse bacterial genomes using MinION long-read sequencing. BMC Genomics. 20 (1), 23 (2019).
- Guerrero, A., Gomez-Gil, B., Lizarraga-Partida, M. L. Genomic stability among O3:K6 V. parahaemolyticus pandemic strains isolated between 1996 to 2012 in American countries. BMC Genomic Data. 22 (1), 38 (2021).
- FAO Applications of Whole Genome Sequencing (WGS) in food safety management. Food and Agriculture Organization of the United Nations Available from: https://www.fao.org/documents/card/es/c/61e44b34-b328-4239-b59c-a9e926e327b4/ (2016)
- Rantsiou, K., et al. Next generation microbiological risk assessment: opportunities of whole genome sequencing (WGS) for foodborne pathogen surveillance, source tracking and risk assessment. International Journal of Food Microbiology. 287, 3-9 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon