JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
전체 게놈 시퀀싱을 사용하는 Oreochromis spp. 농장에서 파생된 병원성 대장균 균주의 특성 분석
요약
벤치탑 기기를 사용한 전체 게놈 시퀀싱(WGS) 전략의 실현 가능성은 실험실 환경에서 공중 보건과 관련된 모든 미생물의 게놈 조사를 단순화했습니다. 박테리아 WGS에 대한 워크 플로우의 방법 론적 적응이 설명되고 분석을위한 생물 정보학 파이프 라인도 제시됩니다.
초록
양식업은 전 세계적으로 가장 빠르게 성장하는 식품 생산 분야 중 하나이며 틸라피아 (Oreochromis spp.) 양식은 양식되는 주요 민물 고기 품종을 구성합니다. 양식 관행은 인위적 원인에서 파생 된 미생물 오염에 취약하기 때문에 광범위한 항생제 사용이 필요하며, 이로 인해 양식 시스템은 대장균 (E. coli)과 같은 임상 적 관련성의 항생제 내성 및 병원성 박테리아의 중요한 공급원이됩니다. 여기에서 내륙 양식 Oreochromis spp.에서 회수된 병원성 대장균 균주의 항균 내성, 독성, 동원체 특징은 전체 게놈 시퀀싱(WGS) 및 인실리코 분석을 통해 해명되었습니다. 항균 감수성 시험 (AST) 및 WGS를 수행하였다. 또한, 계통발생기, 혈청형, 다중위치 서열형(MLST), 획득된 항균 내성, 독성, 플라스미드 및 프로파지 함량을 다양한 사용 가능한 웹 도구를 사용하여 측정했습니다. 분리 된 대장균은 암피실린에 대한 중간 감수성만을 나타 냈으며 WGS 기반 타이핑에 의해 ONT : H21-B1-ST40 균주로 특성화되었습니다. 단일 항생제 내성 관련 유전자 만 검출되었지만 [mdf (A)], 비정형 장 병원성 대장균 (aEPEC) 병리학 형에서 여러 독성 관련 유전자 (VAG)가 확인되었습니다. 추가적으로, 큰 플라스미드 그룹 및 18개의 프로파지 관련 영역으로부터의 플라스미드 레플리콘의 화물이 검출되었다. 결론적으로, 멕시코 시날로아의 양식장에서 회수된 분리균주(aEPEC)의 WGS 특성 분석을 통해 병원성 잠재력과 무가공 양식 제품 섭취의 인체 건강 위험에 대한 통찰력을 얻을 수 있습니다. 환경 미생물을 연구하기 위해 차세대 염기서열분석(NGS) 기술을 활용하고 건강 문제가 어떻게 발생하는지 알아보기 위해 하나의 건강 프레임워크를 채택해야 합니다.
서문
양식업은 전 세계적으로 가장 빠르게 성장하는 식량 생산 분야 중 하나이며, 그 생산 관행은 인간 소비에 대한 증가하는 식량 수요를 충족시키기위한 것입니다. 전 세계 양식 생산량은 1997년 3,400만 톤(Mt)에서 2017년 112Mt로 3배 증가했습니다1. 생산량의 거의 75 %를 차지하는 주요 종 그룹은 해초, 잉어, 이매패 류, 메기 및 틸라피아 (Oreochromis spp.) 1. 그러나 집중적 인 어류 양식으로 인해 미생물 개체로 인한 질병의 출현은 불가피하여 잠재적 인 경제적 손실을 초래합니다2.
어류 양식 관행에서 항생제 사용은 생산성의 주요 제한 요소 인 박테리아 감염을 예방하고 치료하는 것으로 잘 알려져 있습니다 3,4. 그럼에도 불구하고 잔류 항생제는 양식 퇴적물과 물에 축적되어 선택적 압력을 가하고 물고기 관련 및 상주 박테리아 군집 5,6,7,8을 변형시킵니다. 결과적으로, 양식 환경은 항생제 내성 유전자 (ARGs)의 저장소로서 작용하고, 주변 환경에서 항생제 내성 박테리아 (ARB)의 추가 출현 및 확산9. 어류 양식 관행에 영향을 미치는 일반적으로 관찰되는 박테리아 병원체 외에도 Enterobacter spp., Escherichia coli, Klebsiella spp. 및 Salmonella spp.10의 인간 병원균 균주를 포함하여 장내 세균과 가족의 구성원이 종종 발생합니다. 대장균은 어류 양식 11,12,13,14,15에서 어분과 물에서 분리 된 가장 일반적인 미생물입니다.
E. coli는 포유류와 조류의 위장관에 장내 미생물총의 공생 구성원으로 서식하는 다목적 그람 음성 박테리아입니다. 그러나 대장균은 토양, 퇴적물, 음식 및 물을 포함한 다양한 환경 틈새에서 식민지화하고 지속할 수 있는 고도로 적응할 수 있는 능력을 가지고 있습니다16. 수평 유전자 전달 (HGT) 현상을 통한 유전자 이득과 손실로 인해 대장균은 잘 적응 된 항생제 내성 병원체로 빠르게 진화하여 인간과 동물에서 광범위한 질병을 일으킬 수 있습니다17,18. 분리 기원에 따라 병원성 변이체는 장내 병원성 대장균(InPEC) 또는 장외 병원성 대장균(ExPEC)으로 정의됩니다. 또한 InPEC와 ExPEC는 질병 증상, 유전적 배경, 표현형 특성 및 독성 인자(VF)16,17,19에 따라 잘 정의된 병원형으로 하위 분류됩니다.
병원성 대장균 균주에 대한 전통적인 배양 및 분자 기술은 다양한 병원형의 신속한 검출 및 식별을 가능하게 했습니다. 그러나 시간이 많이 걸리고 힘들며 종종 높은 기술 교육이 필요할 수 있습니다19. 또한, 유전 적 배경의 복잡성 때문에 대장균의 모든 병원성 변이체를 안정적으로 연구하는 데 단일 방법을 사용할 수 없습니다. 현재 이러한 단점은 고처리량 시퀀싱(HTS) 기술의 출현으로 극복되었습니다. 전체 게놈 시퀀싱(WGS) 접근 방식과 생물정보학적 도구는 미생물 DNA의 탐색을 저렴하고 대규모로 개선하여 밀접하게 관련된 병원성 변이체20,21,22를 포함하여 한 번의 실행으로 미생물의 심층 특성 분석을 용이하게 합니다. 생물학적 질문에 따라 여러 생물 정보학 도구, 알고리즘 및 데이터베이스를 사용하여 데이터 분석을 수행 할 수 있습니다. 예를 들어, 주요 목표가 ARG, VF 및 플라스미드의 존재를 평가하는 것이라면 ResFinder, VirulenceFinder, PlasmidFinder와 같은 도구가 관련 데이터베이스와 함께 좋은 출발점이 될 수 있습니다. Carriço et al.22는 원시 데이터 전처리에서 계통발생 추론에 이르기까지 미생물 WGS 분석에 적용되는 다양한 생물정보학 소프트웨어 및 관련 데이터베이스에 대한 자세한 개요를 제공했습니다.
여러 연구에서 항생제 내성 특성, 병원성 잠재력, 다양한 기원에서 유래한 임상적으로 관련된 대장균 변이체의 출현 및 진화적 관계 추적에 관한 게놈 심문에 대한 WGS의 광범위한 유용성을 입증했습니다.23,24,25,26 . WGS는 희귀하거나 복잡한 내성 메커니즘을 포함하여 항균제에 대한 표현형 내성의 기초가되는 분자 메커니즘의 식별을 가능하게했습니다. 이는 획득된 ARG 변이체, 약물-표적 유전자의 신규한 돌연변이, 또는 프로모터 영역27,28을 검출하는 것이다. 더욱이, WGS는 박테리아 균주29의 내성 표현형에 대한 사전 지식 없이도 항미생물 내성 프로파일을 추론할 수 있는 잠재력을 제공한다. 또는 WGS는 항생제 내성과 독성 특징을 모두 지닌 이동성 유전 요소 (MGE)의 특성화를 허용하여 기존 병원체의 박테리아 게놈 진화를 주도했습니다. 예를 들어, 2011 년 독일 대장균 발병을 조사하는 동안 WGS를 적용한 결과 분명히 새로운 대장균 병리형의 고유 한 게놈 특징이 밝혀졌습니다. 흥미롭게도, 이러한 발병 균주는 장 출혈성 대장균 (EHEC) 병리학30에서 시가 독소를 암호화하는 프로파지를 획득 한 장 응집성 대장균 (EAEC) 그룹에서 유래했습니다.
이 작업은 벤치탑 시퀀서를 사용하여 박테리아 WGS에 대한 워크플로우의 방법론적 적응을 제시합니다. 또한 웹 기반 도구를 사용하여 생물 정보학 파이프 라인을 제공하여 결과 시퀀스를 분석하고 생물 정보학 전문 지식이 제한적이거나 전혀없는 연구자를 추가로 지원합니다. 설명 된 방법은 2011 년 멕시코 시날로아의 내륙 양식 Oreochromis spp.에서 분리 된 병원성 대장균 균주 ACM5의 항생제 내성, 독성 및 동원체 특징을 해명 할 수있었습니다12.
프로토콜
참고: 대장균 균 주 ACM5는 분변 대장균군(FC) 결정을 위해 어류 샘플을 처리 및 배양하여 회수되었습니다12. 어류 샘플링 동안 물고기는 질병, 박테리아 또는 곰팡이 감염의 임상 징후를 보이지 않았으며 평균 기온은 22.3 ° C였습니다. 분리 후, 분리 대장 균 을 생화학 적 테스트를 거쳐 DMSO (8 % v / v)를 냉동 보호제로 사용하여 뇌 심장 주입 (BHI) 배양액에서 동결 보존했습니다.
1. 냉동 대장균 ACM5 주식 배양의 재활성화
- 냉동 박테리아 스톡에서 튜브를 열고 멸균 루프, 피펫 팁 또는 이쑤시개를 사용하여 냉동 박테리아 배양의 표면을 긁어냅니다.
- 박테리아를 Luria-Bertani (LB) 한천 플레이트에 뿌리고 37 ± 2 ° C에서 24 시간 동안 배양하십시오.
2. 항균 감수성의 결정
참고: 여기에 설명된 항균 감수성 테스트는 임상 및 실험실 표준 연구소(CLSI) 지침(M02 Ed13:2018)31에 기반한 디스크 확산 방법에 해당합니다. 대장균 ATCC 25922 균주는 품질 관리 목적으로 필요합니다.
- 콜로니 현탁법에 의한 접종 준비
- 단계 1.2에서 배양된 LB 플레이트로부터, 하나 또는 두 개의 콜로니를 Mueller-Hinton (MH) 한천 플레이트 상에 줄무늬 및 37 ± 2°C에서 18-24시간 동안 인큐베이션한다.
- 멸균 루프를 사용하여 MH 한천 플레이트에서 갓 계대 배양된 대장균 분리물에서 2개 또는 3개의 콜로니를 선택하고 3mL의 멸균 MH 브로스 또는 0.85%(w/v) 식염수에 재현탁하고 와동으로 완전히 혼합하여 균일한 박테리아 현탁액을 얻습니다.
- UV 가시광선 분광 광도계( 재료 표 참조)를 사용하여 박테리아 현탁액을 0.5 MacFarland 표준(약 1-2 x 108 cells/mL에 해당)에 필적하는 탁도로 조정합니다. 박테리아 현탁액이 너무 가볍거나 너무 무거우면 대장균 콜로니 또는 MH 국물을 적절하게 추가하십시오. 준비 후 15 분 이내에 준비된 접종 물을 사용하십시오.
- 시험 한천 플레이트의 접종
- 멸균 된 면봉을 조정 된 박테리아 현탁액에 담그십시오. 면봉을 여러 번 돌리고 튜브 내벽을 단단히 눌러 면봉에서 과도한 액체를 제거합니다.
- 전체 한천 표면에 면봉을 3배 걸치고 매번 플레이트를 60° 회전시켜 MH 한천 플레이트를 접종합니다. 접종원의 균일한 분포를 보장하기 위해 한천의 가장자리도 면봉으로 닦습니다.
- 수분이 증발할 수 있도록 페트리 접시 뚜껑을 3-5분 동안 열어 둡니다.
- 접종 된 한천 플레이트에 항균 디스크 적용
- 균등하게 분배하고 각 항균 디스크( 재료 표 참조)를 접종된 한천 플레이트의 표면에 눌러 완전한 접촉을 보장합니다. 디스크 도포 후 15분 이내에 플레이트를 뒤집고 35-2°C에서 16-18시간 동안 배양±.
참고: 각각 150mm 및 100mm의 페트리 접시에는 최대 12개 및 6개의 디스크를 사용해야 합니다.
- 균등하게 분배하고 각 항균 디스크( 재료 표 참조)를 접종된 한천 플레이트의 표면에 눌러 완전한 접촉을 보장합니다. 디스크 도포 후 15분 이내에 플레이트를 뒤집고 35-2°C에서 16-18시간 동안 배양±.
- 배양 후 버니어 캘리퍼스를 사용하여 억제 크기 영역을 측정하고 CLSI 중단점 기준(M100 - 표 2A)32에 따라 결과 직경을 해석합니다.
3. 게놈 DNA(gDNA) 추출 및 정량화
- 게놈 DNA 추출
- 갓 배양한 대장균 콜로 니를 LB 브로스 5mL에 재현탁하고 37 ± 2°C의 진탕 인큐베이터(180rpm)에서 밤새 배양합니다.
- 박테리아 현탁액을 3,500 x g 에서 5분 동안 원심분리하고 상청액을 조심스럽게 버립니다.
- DNA 추출 키트 지침에 따라 gDNA를 추출합니다( 재료 표 참조).
- UV 가시광선 분광광도계를 사용하여 260/280nm(비율: >1.8) 및 260/230nm(비율: 2.0-2.2)에서 광학 밀도를 측정하여 gDNA의 순도를 확인합니다. gDNA 무결성을 확인하기 위해 0.8% 아가로스 겔 전기영동을 수행한다.
- 게놈 DNA 정량화
참고: 형광 분석 키트 제조업체에서 승인한 얇은 벽의 투명한 0.5mL PCR 튜브만 사용하십시오( 재료 표 참조).- 샘플 및 분석 표준에 필요한 수의 튜브를 설정합니다. 제조업체의 지침에 따라 키트에 제공된 성분 A와 성분 B를 혼합하여 작업 분석 용액을 준비합니다.
- 190 μL의 작업 용액과 10 μL의 각 표준물질을 적절한 튜브에 추가하여 분석 표준물질을 준비합니다(두 가지 표준물질 필요).
- 198 μL의 작업 용액과 2 μL의 DNA 샘플을 적절한 튜브에 추가합니다. 모든 튜브를 5 초 동안 소용돌이 치면서 격렬하게 혼합하십시오. 거품이 생기지 않도록 주의하십시오.
- 빛으로부터 보호되는 실온에서 2 분 동안 모든 튜브를 배양하십시오. 형광계를 사용하여 제조업체의 지침에 따라 모든 튜브의 형광을 측정합니다( 재료 표 참조).
- 시퀀싱 진행을 위해 gDNA 샘플 농도를 적절하게 조정한다.
4. DNA 라이브러리 준비
참고: DNA 라이브러리 준비 및 시퀀싱은 제조업체의 지침 및 프로토콜에 따라 수행되었습니다( 재료 표 참조). 시작 gDNA 농도는 4.0ng입니다.
- 태깅, PCR 증폭 및 인덱싱
- 0.2mL 튜브에 2.5μL의 태깅 버퍼와 2μL의 입력 gDNA(2.0ng/μL)를 추가합니다. 피펫팅으로 부드럽게 섞습니다.
- 1 μL의 증폭 버퍼를 추가하고 피펫팅으로 부드럽게 혼합합니다. 실온에서 280 x g 에서 1분 동안 회전시킵니다.
- 샘플을 열순환기( 재료 표 참조)에 넣고 다음 PCR 프로그램을 실행합니다: 55°C에서 5분 동안 유지한 다음 10°C에서 유지합니다. 샘플이 10 ° C에 도달하면 즉시 반응을 중화하십시오.
- 1 μL의 중화 버퍼를 샘플 튜브에 추가하고 피펫팅으로 부드럽게 혼합합니다. 280 x g 에서 1분 동안 스핀다운하고 실온에서 5분 동안 배양합니다.
- 1.7μL의 각 인덱스 어댑터(즉, 인덱스 i7 및 i5)와 3μL의 인덱싱 PCR 마스터 믹스를 추가합니다. 피펫팅으로 부드럽게 섞습니다. 실온에서 280 x g 에서 1분 동안 회전시킵니다.
- 샘플을 써모사이클러에 넣고 다음과 같이 두 번째 PCR 반응을 수행한다: 3분 동안 72°C, 30초 동안 95°C, 10초 동안 95°C의 18사이클, 30초 동안 55°C, 30초 동안 72°C, 5분 동안 72°C, 및 10°C에서 유지.
- 증폭된 라이브러리 정리
- 상업용 마그네틱 비드 용액( 재료 표 참조)을 제조업체의 지침에 따라 와동하여 혼합합니다.
- 태그/색인 gDNA 샘플을 새 1.5mL 튜브로 옮기고 gDNA 샘플의 최종 부피(≈13μL)의 각 μL에 대해 0.6μL의 자성 비드를 추가합니다. 피펫팅으로 부드럽게 혼합하고 실온에서 5분 동안 배양합니다.
- 상청액이 제거될 때까지 2분 동안 마그네틱 랙( 재료 표 참조)에 샘플 튜브를 놓습니다. 구슬을 방해하지 않고 상청액을 조심스럽게 제거하고 폐기하십시오.
- 혼합하지 않고 새로 준비한 80% 에탄올 200μL를 추가합니다. 용출액이 깨끗해질 때까지 30초 동안 배양하고 비드를 방해하지 않고 상청액을 조심스럽게 제거하고 버립니다.
- 두 번째 세척 단계를 수행하십시오. 비드에 80% 에탄올 200μL를 넣고 30초 동안 배양합니다. 용출액이 깨끗해지면 상청액을 제거하고 버리고 비드를 10분 동안 자연 건조합니다.
- 15μL의 10mM 트리스 완충액(pH 8)을 비드에 추가하고 피펫팅으로 부드럽게 혼합합니다. 실온에서 2 분 동안 배양하십시오. 샘플 튜브를 마그네틱 랙에 다시 2분 동안 놓고 상층액이 깨끗해지도록 합니다.
- 14 μL의 상청액을 샘플 튜브에서 새 0.2 mL 튜브로 조심스럽게 옮깁니다. 정리된 라이브러리입니다.
참고: 이 시점에서 정리된 라이브러리는 -20°C에서 최대 7일 동안 보관할 수 있습니다.
- 라이브러리 정규화
- 단계 3.2.5를 제외하고 단계 3.2에 기재된 바와 같이 형광 분석에 의해 클린업된 라이브러리를 정량화하는 것을 진행한다.
- 1% 아가로스 겔 전기영동에서 클린업된 라이브러리를 시각화하여 평균 단편 크기를 결정합니다.
- 다음 방정식을 사용하여 라이브러리의 몰 농도 값을 계산합니다.
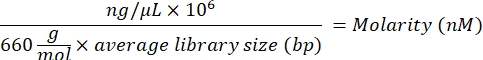
- 몰 농도 값을 사용하여 적절한 부피의 재현탁 버퍼(RSB) 및 클린업된 라이브러리를 계산하여 개별 라이브러리를 10nM의 시작 농도로 희석합니다.
5. 라이브러리 풀링, 비자연화 및 시퀀서 시작
- 제조업체의 지침에 따라 시약 카트리지를 해동하십시오. 냉장고(4°C)에서 플로우 셀을 추출하고 시퀀싱하기 전에 실온으로 가져옵니다.
- 5μL의 각 정규화된 라이브러리(10nM)를 저결합 1.5mL 튜브에 넣습니다. 풀링된 라이브러리를 적절한 부피의 RSB로 4nM으로 희석합니다.
- 새로운 저결합 1.5mL 튜브에 풀링된 라이브러리 5μL(4nM)와 0.2N NaOH 5μL를 추가합니다. 와동으로 간단히 혼합하고 280 x g 에서 1분 동안 스핀다운하고 실온에서 5분 동안 배양하여 풀링된 라이브러리를 단일 가닥으로 변성시킵니다.
- 피펫팅을 통해 변성 풀 라이브러리 10μL와 사전 냉장 하이브리드화 버퍼 990μL를 부드럽게 혼합하고 시퀀서 로딩을 위한 최종 희석이 수행될 때까지 얼음 위에 놓습니다. 변성 풀링된 라이브러리의 농도는 20pM이다.
- 해동하고 2 μL의 대조 라이브러리 (10 nM) 및 3 μL의 뉴클레아제 비함유 물을 혼합하여 4 nM의 농도로 대조 라이브러리 ( 재료 표 참조)를 제조한다.
- 5μL의 제어 라이브러리(4nM)와 5μL의 새로 준비된 0.2N NaOH를 혼합합니다. vortexing으로 간단히 혼합하고 280 x g 에서 1분 동안 스핀다운하고 실온에서 5분 동안 배양하여 제어 라이브러리를 단일 가닥으로 변성시킵니다.
- 피펫팅을 통해 변성 대조군 라이브러리 10μL와 사전 냉장 하이브리드화 버퍼 990μL를 부드럽게 혼합하고 시퀀서 로딩을 위한 최종 희석이 완료될 때까지 얼음 위에 놓습니다. 변성 제어 라이브러리의 농도는 20 pM이다.
- 새로운 저결합 1.5mL 튜브에서 20pM에서 594μL의 변성 풀 라이브러리와 20pM의 변성 제어 라이브러리 6μL를 결합합니다. 제대로 섞는다.
- 최종 라이브러리 믹스(20 pM)를 예냉된 혼성화 완충액을 사용하여 600 μL의 부피로 1.2 pM의 최종 로딩 농도로 희석한다.
- 최종 라이브러리 혼합물을 시약 카트리지에 로딩하기 전에 추가 열 전처리를 수행하여 플로우 셀에 효율적으로 로딩하십시오. 최종 라이브러리 믹스를 96°C에서 2분 동안 인큐베이션합니다. 튜브를 뒤집어 혼합하고 튜브를 얼음 위에 5분 동안 놓습니다.
- 최종 라이브러리 혼합물 500μL를 시약 카트리지의 설계된 저장소에 로드합니다. 지침에 따라 시퀀싱을 시작합니다. 플로우 셀과 시약 카트리지를 로드하고 시퀀싱 실행을 설정합니다.
6. 시퀀스 데이터 분석
참고: 대장균 게놈의 일반적인 WGS 데이터 전처리, 소프트웨어, 파라미터 설정 및 서열 분석에 대한 자세한 설명은 보충 파일 1을 확인하십시오.
- 시퀀싱 데이터 서버에 연결하고 FASTQ 파일을 다운로드합니다.
- 타사 소프트웨어로 원시 시퀀스 데이터의 초기 품질을 평가합니다. 원시 시퀀싱 데이터에서 나머지 어댑터 시퀀스, 저품질 베이스(보충 파일 1 참조).
- 타사 소프트웨어를 사용하여 품질 검사를 거친 시퀀싱 데이터를 연속 또는 스캐폴드 수준으로 조합합니다( 보충 파일 1 참조).
- 조립된 게놈을 포함하는 FASTA 파일을 RAST 서버(https://rast.nmpdr.org/)에 제출하여 게놈 주석을 수행한다.
- FASTA 파일을 게놈 역학 센터(CGE)(http://www.genomicepidemiology.org/services/) 및 ClermonTyping(http://clermontyping.iame-research.center/index.php) 웹 플랫폼에 업로드하여 역학적 특징, ARG, VAG 및 플라스미드를 식별합니다(보충 파일 1 참조).
- FASTA 파일을 PHASTER 서버에 업로드하여 프로파지 서열(https://phaster.ca/)을 식별합니다.
결과
항균 감수성은 디스크 확산 방법에 의해 결정되고 6 가지 별개의 항균 클래스, 즉 아미노 글리코 시드, β- 락탐, 플루오로 퀴놀론, 니트로 퓨란, 페니콜 및 엽산 경로 길항제에 걸친 12 가지 항생제에 대한 CLSI 중단 점 기준에 의해 해석되었습니다. 대장균 ACM5는 하나의 β-락탐 약물을 제외한 모든 항생제에 민감성을 나타냈다. 4 가지 β 락탐 약물이 테스트되었습니다 : 암피실린, 카르 베니 실?...
토론
이 연구는 병원성 대장균 변이체의 게놈 특성화를 위한 벤치탑 시퀀서와 파이프라인을 사용하여 박테리아 WGS 워크플로우의 적응을 제시합니다. 사용된 염기서열분석 플랫폼에 따라 습식 실험실 절차(박테리아 배양, gDNA 추출, 라이브러리 준비 및 염기서열분석) 및 서열 분석의 처리 시간(TAT)이 달라질 수 있으며, 특히 느리게 성장하는 박테리아를 연구하는 경우 더욱 그렇습니다. 위에서 설...
공개
저자는 공개 할 것이 없습니다.
감사의 말
호세 안토니오 마가냐-리자라가 [No. 481143]에게 수여되는 박사 장학금을 위해 멕시코 국립 과학 기술위원회 (CONACyT)에 수여됩니다.
자료
| Name | Company | Catalog Number | Comments |
| Accublock Mini digital dry bath | Labnet | D0100 | Dry bath for incubation of tubes |
| Agencourt AMPure XP | Beckman Coulter | A63881 | Magnetic beads in solution for DNA library purification |
| DeNovix DS-11 | DeNovix Inc. | UV-Vis spectophotometer to check the quality of the gDNA extracted | |
| DNA LoBind Tubes | Eppendorf | 0030108418 | 1.5 mL PCR tubes for DNA library pooling |
| DynaMag-2 Magnet | Invitrogen, Thermo Fisher Scientific | 12321D | Magnetic microtube rack used during magnetic beads-based DNA purification |
| Gram-negative Multibac I.D. | Diagnostic reseach (Mexico) | PT-35 | Commercial standard antibiotic disks for antimicrobial susceptibility testing |
| MiniSeq Mid Output Kit (300-cycles) | Illumina | FC-420-1004 | Reagent cartdrige for paired-end sequencing (2x150) |
| MiniSeq System Instrument | Illumina | SY-420-1001 | Benchtop sequencer used for Next-generation sequencing |
| MiniSpin centrifuge | Eppendorf | 5452000816 | Standard centrifuge for tubes |
| Nextera XT DNA Library Preparation Kit | Illumina | FC-131-1024 | Reagents to perform DNA libraries for sequencing. Includes Box 1 and Box 2 reagents for 24 samples |
| Nextera XT Index Kit v2 | Illumina | FC-131-2001, FC-131-2002, FC-131-2003, FC-131-2004 | Index set A, B, C, D |
| PhiX Control v3 | Illumina | FC-110-3001 | DNA library control for sequencing |
| Precision waterbath | LabCare America | 51221081 | Water bath shaker used for bacterial culture |
| Qubit 1X dsDNA HS Assay Kit | Invitrogen, Thermo Fisher Scientific | Q33231 | Reagents for fluorescence-based DNA quantification assay |
| Qubit 2.0 Fluorometer | Invitrogen, Thermo Fisher Scientific | Q32866 | Fluorometer used for fluorescence assay |
| Qubit Assay tubes | Invitrogen, Thermo Fisher Scientific | Q32856 | 0.5 mL PCR tubes for fluorescence-based DNA quantification assay |
| SimpliAmp Thermal Cycler | Applied Biosystems, Thermo Fisher Scientific | A24811 | Thermocycler used for DNA library amplification |
| Spectronic GENESYS 10 Vis | Thermo | 335900 | Spectophotometer used for bacterial suspension in antimicrobial susceptibility testing |
| ZymoBIOMICS DNA Miniprep Kit | Zymo Research Inc. | D4300 | Kit for genomic DNA extraction (50 preps) |
참고문헌
- Naylor, R. L., et al. A 20-year retrospective review of global aquaculture. Nature. 591 (7851), 551-563 (2021).
- Quesada, S. P., Paschoal, J. A. R., Reyes, F. G. R. Considerations on the aquaculture development and on the use of veterinary drugs: special issue for fluoroquinolones-a review. Journal of Food Science. 78 (9), 1321-1333 (2013).
- Defoirdt, T., Sorgeloos, P., Bossier, P. Alternatives to antibiotics for the control of bacterial disease in aquaculture. Current Opinion in Microbiology. 14 (3), 251-258 (2011).
- Stentiford, G. D., et al. New paradigms to help solve the global aquaculture disease crisis. PLOS Pathogens. 13 (2), 1006160 (2017).
- Chen, H., et al. Tissue distribution, bioaccumulation characteristics and health risk of antibiotics in cultured fish from a typical aquaculture area. Journal of Hazardous Materials. 343, 140-148 (2018).
- Zhou, M., et al. Antibiotics control in aquaculture requires more than antibiotic-free feeds: A Tilapia farming case. Environmental Pollution. 268, 115854 (2021).
- Feng, Y., et al. Ecological effects of antibiotics on aquaculture ecosystems based on microbial community in sediments. Ocean & Coastal Management. 224, 106173 (2022).
- Shen, X., Jin, G., Zhao, Y., Shao, X. Prevalence and distribution analysis of antibiotic resistance genes in a large-scale aquaculture environment. Science of The Total Environment. 711, 134626 (2020).
- Su, H., et al. Contamination of antibiotic resistance genes (ARGs) in a typical marine aquaculture farm: source tracking of ARGs in reared aquatic organisms. Journal of Environmental Science and Health, Part B. 55 (3), 220-229 (2020).
- Oliveira, R. V., Oliveira, M. C., Pelli, A. Disease infection by Enterobacteriaceae family in fishes: a review. Journal of Microbiology & Experimentation. 4 (5), 00128 (2017).
- Barbosa, M. M. C., et al. Sorologia e suscetibilidade antimicrobiana em isolados de Escherichia coli de pesque-pagues. Arquivos do Instituto Biológico. 81 (1), 43-48 (2014).
- Valenzuela-Armenta, J. A., et al. Microbiological analysis of Tilapia and water in aquaculture farms from Sinaloa. Biotecnia. 20 (1), 20-26 (2018).
- Reza, R. H., Shipa, S. A., Naser, M. N., Miah, M. F. Surveillance of Escherichia coli in a fish farm of Sylhet, Bangladesh. Bangladesh Journal of Zoology. 48 (2), 335-346 (2021).
- Liao, C. -. Y., et al. Antimicrobial resistance of Escherichia coli From aquaculture farms and their environment in Zhanjiang, China. Frontiers in Veterinary Science. 8, 806653 (2021).
- Dewi, R. R., et al. Prevalence and antimicrobial resistance of Escherichia coli, Salmonella and Vibrio derived from farm-raised Red Hybrid Tilapia (Oreochromis spp.) and Asian Sea Bass (Lates calcarifer, Bloch 1970) on the west coast of Peninsular Malaysia. Antibiotics. 11 (2), 136 (2022).
- Leimbach, A., Hacker, J., Dobrindt, U. E. coli as an all-rounder: the thin line between commensalism and pathogenicity. Current Topics in Microbiology and Immunology. 358, 3-32 (2013).
- Kaper, J. B., Nataro, J. P., Mobley, H. L. T. Pathogenic Escherichia coli. Nature Reviews Microbiology. 2 (2), 123-140 (2004).
- Croxen, M. A., Finlay, B. B. Molecular mechanisms of Escherichia coli pathogenicity. Nature Reviews Microbiology. 8 (1), 26-38 (2010).
- Croxen, M. A., et al. Recent advances in understanding enteric pathogenic Escherichia coli. Clinical Microbiology Reviews. 26 (4), 822-880 (2013).
- Bertelli, C., Greub, G. Rapid bacterial genome sequencing: methods and applications in clinical microbiology. Clinical Microbiology and Infection. 19 (9), 803-813 (2013).
- Lynch, T., Petkau, A., Knox, N., Graham, M., Van Domselaar, G. A primer on infectious disease bacterial genomics. Clinical Microbiology Reviews. 29 (4), 881-913 (2016).
- Carriço, J. A., Rossi, M., Moran-Gilad, J., Van Domselaar, G., Ramirez, M. A primer on microbial bioinformatics for nonbioinformaticians. Clinical Microbiology and Infection. 24 (4), 342-349 (2018).
- Magaña-Lizárraga, J. A., et al. Draft genome sequence of Escherichia coli M51-3: a multidrug-resistant strain assigned as ST131-H30 recovered from infant diarrheal infection in Mexico. Journal of Global Antimicrobial Resistance. 19, 311-312 (2019).
- Pérez-Vázquez, M., et al. Emergence of NDM-producing Klebsiella pneumoniae and Escherichia coli in Spain: phylogeny, resistome, virulence and plasmids encoding blaNDM-like genes as determined by WGS. Journal of Antimicrobial Chemotherapy. 74 (12), 3489-3496 (2019).
- Massella, E., et al. Snapshot study of whole genome sequences of Escherichia coli from healthy companion animals, livestock, wildlife, humans and food in Italy. Antibiotics. 9 (11), 782 (2020).
- Magaña-Lizárraga, J. A., et al. Genomic profiling of antibiotic-resistant Escherichia coli isolates from surface water of agricultural drainage in north-western Mexico: detection of the international high-risk lineages ST410 and ST617. Microorganisms. 10 (3), 662 (2022).
- Saracino, I. M., et al. Next Generation sequencing for the prediction of the antibiotic resistance in Helicobacter pylori: a literature review. Antibiotics. 10 (4), 437 (2021).
- Ghosh, A., Saha, S. Survey of drug resistance associated gene mutations in Mycobacterium tuberculosis, ESKAPE and other bacterial species. Scientific Reports. 10 (1), 8957 (2020).
- Su, M., Satola, S. W., Read, T. D. Genome-based prediction of bacterial antibiotic resistance. Journal of Clinical Microbiology. 57 (3), 01405-01418 (2019).
- Brzuszkiewicz, E., et al. Genome sequence analyses of two isolates from the recent Escherichia coli outbreak in Germany reveal the emergence of a new pathotype: Entero-Aggregative-Haemorrhagic Escherichia coli (EAHEC). Archives of Microbiology. 193 (12), 883-891 (2011).
- . CLSI Performance Standards for Antimicrobial Disk Susceptibility Tests. 13th ed. CLSI standard M02. Wayne, PA: Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m02/ (2018)
- CLSI Performance Standards for Antimicrobial Susceptibility Testing. 31st ed. CLSI supplement M100. Clinical and Laboratory Standards Institute Available from: https://clsi.org/standards/products/microbiology/documents/m100/ (2021)
- Ewing, B., Green, P. Base-calling of automated sequencer traces using phred. II. Error probabilities. Genome Research. 8 (3), 186-194 (1998).
- Quainoo, S., et al. Whole-genome sequencing of bacterial pathogens: the future of nosocomial outbreak analysis. Clinical Microbiology Reviews. 30 (4), 1015-1063 (2017).
- Desai, A., et al. Identification of optimum sequencing depth especially for de novo genome assembly of small genomes using next generation sequencing data. PLoS ONE. 8 (4), 60204 (2013).
- Nishino, K., Yamada, J., Hirakawa, H., Hirata, T., Yamaguchi, A. Roles of TolC-dependent multidrug transporters of Escherichia coli in resistance to β-lactams. Antimicrobial Agents and Chemotherapy. 47 (9), 3030-3033 (2003).
- Li, M., et al. The resistance mechanism of Escherichia coli induced by ampicillin in laboratory. Infection and Drug Resistance. 12, 2853-2863 (2019).
- Ménard, L. -. P., Dubreuil, J. D. Enteroaggregative Escherichia coli heat-stable enterotoxin 1 (EAST1): a new toxin with an old twist. Critical Reviews in Microbiology. 28 (1), 43-60 (2002).
- Dubreuil, J. D. EAST1 toxin: An enigmatic molecule associated with sporadic episodes of diarrhea in humans and animals. Journal of Microbiology. 57 (7), 541-549 (2019).
- Goldstein, S., Beka, L., Graf, J., Klassen, J. L. Evaluation of strategies for the assembly of diverse bacterial genomes using MinION long-read sequencing. BMC Genomics. 20 (1), 23 (2019).
- Guerrero, A., Gomez-Gil, B., Lizarraga-Partida, M. L. Genomic stability among O3:K6 V. parahaemolyticus pandemic strains isolated between 1996 to 2012 in American countries. BMC Genomic Data. 22 (1), 38 (2021).
- FAO Applications of Whole Genome Sequencing (WGS) in food safety management. Food and Agriculture Organization of the United Nations Available from: https://www.fao.org/documents/card/es/c/61e44b34-b328-4239-b59c-a9e926e327b4/ (2016)
- Rantsiou, K., et al. Next generation microbiological risk assessment: opportunities of whole genome sequencing (WGS) for foodborne pathogen surveillance, source tracking and risk assessment. International Journal of Food Microbiology. 287, 3-9 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유